Method Article
유전적으로 재프로그래밍된 쥐 흑색종의 FFPE 검체를 분석하기 위한 DNA 바코드 기반 다중 면역형광 이미징
* 이 저자들은 동등하게 기여했습니다
요약
이 프로토콜은 쥐 FFPE 흑색종 조직의 DNA 바코드 기반 이미징을 위한 다중 면역형광 항체 패널을 설계하기 위한 단계별 가이드를 제공합니다. 또한 쥐 흑색종 종양 면역 미세환경에 대한 공간 단백질체학 통찰력을 생성하기 위해 오픈 소스 도구를 사용하는 이미지 분석 파이프라인에 대해 설명합니다.
초록
여기에 제시된 것은 조직 미세환경의 공간 단백질체학을 분석하는 Co-Detection-by-indEXing을 기반으로 하는 새로운 DNA-바코드 기반 멀티플렉스 이미징 기술입니다. 성공적인 이미징을 위해서는 잘 설계되고 적절하게 검증된 항체 패널의 레퍼토리가 필요하지만, 현재 포르말린 고정 파라핀 포매(FFPE) 샘플에 대한 항체는 거의 없습니다. FFPE는 갓 동결된 표본에 비해 광범위한 가용성, 취급 및 보관 용이성, 조직 마이크로어레이(TMA)를 만들 수 있는 능력과 같은 몇 가지 이점을 제공합니다. 여기에서는 종양 미세환경 재프로그래밍을 위한 면역학적 신호를 암호화하는 플라스미드 DNA를 전달하는 나노입자로 처리된 쥐 흑색종 모델로부터 FFPE 조직을 시각화하고 분석하기 위한 항체 패널을 개발하기 위한 프로토콜을 제시합니다. 또한 조직에 주석을 달고, 세포를 분할하고, 단백질체학 데이터를 처리하고, 세포 집단을 표현형화하고, 공간 메트릭을 정량화하기 위한 오픈 소스 컴퓨팅 도구를 사용하는 이미지 분석 파이프라인에 대해서도 설명합니다. 이 프로토콜은 쥐 FFPE에서 항체 패널을 설계하고 복잡한 조직 미세환경의 공간 단백질체학에 대한 새로운 통찰력을 생성하기 위한 응용 프로그램을 제공합니다.
서문
피부 흑색종은 가장 흔한 피부암으로, 진단 시점과 1차 진료에 따라 전 세계적으로 다양한 질병과 사망률을 보입니다1. 지난 10년 동안 흑색종에 대한 생물학적 이해가 높아짐에 따라 고형 종양을 치료하기 위한 새로운 암 모델 개발이 촉진되었습니다2. 최근 면역요법이 부상하면서 내인성 면역 체계의 활성화에 기초한 암 치료의 혁신적인 개념이 생겨났습니다 3,4.
종양 미세환경(TME)은 매우 복잡하며 다양한 면역세포, 암 관련 섬유아세포, 주피세포, 내피 세포 및 다양한 조직 상주 세포로 구성되어 있습니다5. TME를 연구하기 위해 과거에는 유세포 분석 및 단세포 염기서열분석과 같은 여러 기술이 적용되었는데, 이는 종양 조직을 파괴하는 데 필요하기 때문에 공간적 맥락을 손상시킵니다. 면역형광(IF) 및 면역조직화학(IHC)과 같은 기존 현미경 이미징을 통해 샘플 조직을 파괴하지 않고 단백질 바이오마커를 시각화할 수 있습니다. 그러나 이러한 접근법은 2개 또는 3개의 바이오마커로 제한되며 복잡한 TME 6 내의 공간적 및 구조적 관계에 대한 완전한 이해를 제공할 수 없습니다.
이러한 문제를 해결하기 위해, 복잡한 TME를 공간적으로 시각화하기 위해 여러 멀티플렉스 이미징 기술이 개발되었다 7,8,9,10. 그 중 하나는 DNA 올리고뉴클레오티드 접합 항체11을 기반으로 PhenoCycler 시스템으로 이름이 변경된 CoDetection-by-in-DEXing입니다. 이 시스템은 인간 표본에 대한 100개 이상의 바이오마커에 대한 단일 세포 이미징 및 분석을 제공할 수 있습니다. 그러나 쥐 표본, 특히 포르말린 고정 파라핀 포매드(FFPE) 샘플을 시각화하고 분석할 수 있는 인벤토리 항체는 거의 없습니다12. FFPE는 취급 및 보관의 용이성, 시간 경과에 따른 잘 보존된 형태, 그리고 가장 중요한 것은 단일 슬라이드에서 여러 표본을 시각화할 수 있는 조직/종양 마이크로어레이(TMA)를 준비할 수 있는 기능과 같은 신선 냉동(FF) 보존에 비해 여러 가지 이점을 제공합니다. 우리는 최근 쥐 FFPE CODEX/PhenoCycler 항체 패널을 설계 및 개발하여 유전적으로 재프로그래밍된 쥐 흑색종 표본13의 공간 단백질체학을 시각화하고 분석하는 데 성공적으로 적용했습니다.
이 프로토콜의 전반적인 목표는 쥐 FFPE 항체 패널을 설계하기 위한 단계별 가이드를 제공하고 항체-바코드 접합, 조직 염색 및 이미징 프로세스를 설명하는 것입니다. 또한 QuPath 및 R 패키지와 같은 오픈 소스 도구를 활용하는 상세한 이미지 분석 파이프라인을 제공합니다. 이 프로토콜을 따른 후 연구원들은 맞춤형 접합 항체 패널을 설계하고, Phenocycler-Fusion 장치를 사용하여 다중 이미징을 수행하고, 흑색종 TME의 공간 단백질체학에 대한 새로운 통찰력을 얻는 방법을 배우게 됩니다. 또한 이 프로토콜은 다양한 종양 면역 미세환경을 연구하기 위해 조정될 수 있으며 기존 공간 전사체학 기술과 결합될 수 있습니다.
프로토콜
모든 동물 작업은 존스 홉킨스 동물 관리 및 사용 위원회(Johns Hopkins Animal Care and Use Committee)가 정한 지침에 따라 승인된 프로토콜 번호 MO18M388 및 MO21M384를 사용하여 수행되었습니다.
1. 항체 선택
- 관심 조직과 특정 바이오마커의 풍부함을 기반으로 항체 패널을 설계합니다. 문헌에서 면역형광(IF) 및 면역조직화학(IHC) 염색의 성공적인 이전 응용 프로그램을 기반으로 항체 클론을 선택하십시오. 이러한 항체의 안전한 무운반 버전.
참고: 캐리어가 없는 정제된 항체는 바코드 접합에 중요합니다. BSA, 글루텐, 글리세롤 등과 같은 단백질 기반 방부제는 피하십시오. 무운반 항체를 사용할 수 없는 경우, 공급업체에 맞춤형 항체를 요청하거나 BSA 제거 키트를 사용하여 무운반 항체를 만드십시오. 아지드화나트륨은 항체 접합을 방해하지 않습니다. IgG 이소형은 IgM 이소형보다 권장됩니다. - 기존의 면역형광(IF) 이미징11을 사용하여 관심 조직에서 이러한 선택된 클론을 확인합니다. 이중 IF 이미징을 사용하여 특정 클론의 특이성을 확인합니다(예: 조절 T 세포에 대한 염색을 확인하기 위해 FOXP3 및 CD4로 이중 IF 이미징 수행).
참고: 가능하면 염색에 사용할 IF 검증에 동일한 조직을 사용하는 것이 중요합니다. 적절한 항원 회수 솔루션을 선택합니다. 이 프로토콜에서는 Akoya Biosciences에서 제공하는 AR9 완충액을 사용하여 모든 쥐 흑색종 FFPE 조직 항원결정기를 회수했습니다. AR6 또는 Universal과 같은 다른 항원 회수 솔루션은 에피토프 요구 사항에 따라 사용할 수 있습니다.
2. 항체 접합 및 확인
- ATTO550(Cy3), AF647(Cy5) 또는 AF750(Cy7) 형광단에 무료 리포터가 부착된 바코드에 검증된 항체를 할당합니다. 풍부하지 않은 항원은 일반적으로 더 낮은 신호를 생성합니다. 이러한 항체를 AF647(Cy5)과 같은 낮은 자가형광 채널에 접합합니다. 자가형광 가능성으로 인해 발현이 높은 항원을 ATTO550(Cy3) 및 AF750(Cy7)에 접합합니다.
참고: 이 중요한 단계는 채널 민감도와 항원 풍부도를 고려합니다. FFPE 조직에 AF488 채널을 사용하는 것은 자가형광이 높기 때문에 권장되지 않습니다. - 항체-바코드 접합(4.5시간)의 경우 항체 접합 키트를 구하십시오. 환원 용액 1을 제외한 모든 시약은 4°C에서 보관하며, 환원 용액 1은 작은 일회용 바이알로 -20°C에서 보관해야 합니다.
- 분광 광도계를 사용하여 항체 농도를 측정합니다.
- 500 μL의 필터 차단 용액을 50kDa MWCO 필터 컬럼에 적용하여 항체 결합을 시작하여 필터에 대한 항체의 비특이적 결합을 차단합니다. 실온(RT)에서 12,000 x g 로 2분 동안 스핀다운합니다. 200μL 마이크로피펫을 사용하여 컬럼에서 여분의 액체를 제거합니다.
참고: 환원 용액 1은 일회용 용액 바이알로, 한 번에 3개의 항체 접합체를 사용할 수 있습니다. 해동 후 용액을 재사용하지 마십시오. 남은 시약은 모두 버리십시오. 50kDa MWCO 필터 컬럼 위에 필터를 사용하면 정제 및 접합이 불량해질 수 있으므로 필터를 사용하지 마십시오. - 50μg 상당량의 항체 용액을 사용하여 50kDa MWCO 필터에 추가합니다. 필요한 경우 PBS를 사용하여 총 부피를 100μL로 조정합니다. 12,000 x g 에서 4°C에서 8분 동안 스핀다운합니다. 이 시간 동안 3개의 항체 접합체에 대해 19.8 μL의 환원 용액 1 + 825 μL의 환원 용액 2를 사용하여 환원 마스터 믹스를 준비합니다.
- 플로우 스루(flow through)를 버리고 260 μL의 환원 마스터 믹스를 각 필터 장치의 상단에 추가합니다. 필터 장치에 와류 용액을 2-3 초 동안 넣고 RT에서 30 분 동안 배양합니다.
참고: 항체의 과도한 감소를 피하기 위해 30분을 초과하지 않는 것이 중요하며, 이는 항체를 손상시키고 접합에 실패할 수 있기 때문입니다. - 30분 배양 후 12,000 x g 에서 4°C에서 8분 동안 스핀다운하고 바닥에서 흐름을 버립니다. 컬럼 상단에 450μL의 접합 용액을 추가하고 4°C에서 8분 동안 12,000 x g 에서 스핀다운합니다. 원심분리 중에 CODEX 바코드 솔루션을 준비합니다. -20 °C에서 바코드를 검색한 후 바코드가 열화되기 시작하므로 빠르게 움직입니다.
참고: 바코드는 일반적으로 -20°C에서 보관된 동결건조 제품(플레이크 또는 분말)이 들어 있는 작은 유리 바이알에 공급됩니다. 이 바이알은 한 번에 50μg의 항체를 접합하는 데 사용할 수 있습니다. 한 번에 3개 이상의 항체를 접합하지 않는 것이 좋습니다. - 동결건조된 바코드를 유리 바이알 바닥에 조심스럽게 배치합니다(벽에 붙어 있을 수도 있음). 테이블 위의 유리 바이알을 두드려 고형물을 바닥으로 가져옵니다. 동결건조된 생성물을 찾는 것은 10μL의 뉴클레아제가 없는 분자생물학 등급의 물에 용해시킬 때 필수적입니다. 10μL의 뉴클레아제가 없는 물을 첨가한 후 각 바코드에 210μL의 접합 용액을 추가합니다. 동결건조된 모든 물질을 용해하고 위아래로 피펫팅하여 부드럽게 혼합합니다. 곁.
- 2.2.5단계에서 스핀을 완료한 후 플로우 스루를 폐기하고 2.2.6단계의 해당 바코드 솔루션을 각 필터 장치의 맨 위에 추가합니다. 초기 접합되지 않은 항체의 1 μg을 저장합니다. 이는 겔 전기영동 검증 중에 공액 5μL 항체 샘플과 함께 실행됩니다.
- 각 튜브에 해당 항체 및 바코드 이름을 표시합니다. 뚜껑을 닫고 2-3초 동안 볼텍싱하여 용액을 혼합합니다. RT에서 2시간 동안 항체-바코드 접합 반응을 배양합니다.
- 2시간 배양 후 5μL의 접합 항체를 제거하고 0.2mL PCR 튜브에 보관하여 겔 전기영동으로 접합을 확인합니다.
- 남은 접합 항체 용액을 12,000 x g 에서 4°C에서 8분 동안 스핀다운합니다. 컬럼 상단에 450μL의 정제 용액을 추가하고 4°C에서 8분 동안 12,000 x g 로 회전한 다음 플로우 스루를 버립니다. 이 정제 단계를 2회 반복하여 매번 450μL 정제 용액을 추가하고 스핀다운합니다.
- 3번째 원심분리 후 플로우 스루를 폐기합니다. 상단 필터에는 바코드 접합 항체가 포함됩니다.
- 접합 항체를 수집하려면 필터 컬럼을 고정하는 새 외부 튜브에 해당 항체와 바코드를 표시합니다. 원심분리하기 전에 외부 튜브의 뚜껑을 잘라냅니다. 각 필터 장치에 100μL의 항체 보관 용액을 추가하고 새로 라벨링된 외부 튜브를 필터 컬럼 위에 거꾸로 놓습니다.
- 필터 컬럼을 뒤집어 접합 항체를 외부 튜브에 모으고 RT에서 2분 동안 3,000 x g 로 스핀 다운합니다. 이를 통해 약 120μL의 접합 항체 용액을 수집해야 합니다. 멸균 마이크로 원심분리기 튜브로 옮기고 4°C에서 18-24개월 동안 보관합니다.
참고: 접합 후 높은 백그라운드 핵 염색을 방지하려면 이러한 항체를 최소 2일 동안 사용하지 않는 것이 좋습니다.
- 이미 발표된 프로토콜13에 설명된 대로 겔 전기영동에 의한 접합 확인을 수행합니다.
참고: 이 프로세스는 바코드를 항체와 접합하는 데 사용되는 화학 접합 반응의 성공 여부만 확인합니다. 항체 검증은 조직의 성공적인 염색 및 이미징 후에만 가능하기 때문에 선택 사항입니다.
3. 쥐 FFPE 표본 준비
- B16F10 옆구리 종양이 이식된 C57BL/6J 암컷 마우스에서 FFPE 조직 샘플을 얻습니다. 이식 후 9일, 11일, 16일 및 18일에 항-PD1의 복강 내 전달과 함께 4-1BBL 및 IL-12 플라스미드를 함유하는 폴리(β-아미노 에스테르) 기반 나노입자의 종양 내 주사로 마우스 치료14. 20일째에 생쥐를 희생하고 파라핀에 박힌 10% 포르말린으로 종양을 고정한 다음 Johns Hopkins Oncology Tissue Services 코어에서 절편화합니다.
- 사용 설명서에 제공된 이미징 영역 사양을 신중하게 고려하고 이미징 영역 내부의 슬라이드에 5μm 표본을 장착합니다. 우리는 하나의 슬라이드에 4개의 서로 다른 종양 표본을 장착할 수 있었습니다.
참고: Leica Apex 접착 슬라이드 또는 Fisherbrand Superfrost Plus 슬라이드와 같이 이미징에 사용할 수 있는 권장 슬라이드를 사용하는 것이 중요합니다. 이렇게 하면 플로우 셀이 슬라이드에 단단히 부착되는 데 도움이 됩니다.
4. FFPE 조직 염색 및 이미징
- 항체 농도 및 노출 시간의 검증 및 최적화: 항체-바코드 접합의 성공 여부를 확인하기 위해 관심 조직을 6-9개의 접합 항체로 염색합니다. 멀티플렉스 이미징 장치를 실행하고 각 항체 농도와 노출 시간을 기록합니다. 필요에 따라 희석 및 노출 시간을 조정한 다음 이전에 확인된 항체로 6-9개의 새로운 항체를 실행하고 조정 사항을 기록하십시오. 염색 및 이미징을 위해 아래 단계를 따르십시오.
- 조직 염색 및 이미징
- 염색하기 전날 FFPE 티슈 슬라이드를 60°C 오븐에서 밤새 굽습니다. 다음 날, 탈파라핀화 및 조직 수분 보충 단계를 시작하기 전에 RT에서 10분 동안 슬라이드를 식힙니다.
- 자일렌 용액에서 각각 5분 동안 2회 배양하여 조직의 파라핀 제거를 시작합니다.
- 조직을 재수화하려면 100% 에탄올 용액을 각각 5분 동안 두 번 움직입니다. 다음으로, 일련의 알코올 용액을 90%에서 70%, 50%, 30%로 각각 5분 동안 이동합니다. 마지막으로 증류수로 티슈를 5분씩 두 번씩 씻습니다.
참고: Neoclear 용액과 같은 자일렌에 대한 독성이 적은 대안은 탈파라핀화 단계에 사용할 수 있습니다. 흄 후드 아래에서 탈파라핀화 및 재수화를 수행합니다. - 증류수를 사용하여 10x AR9 버퍼를 1x로 희석합니다. Coplin 용기에 50mL의 1x AR9 완충액을 채우고 슬라이드가 항원 회수 완충액에 완전히 잠겼는지 확인합니다.
- 압력솥에 물을 채워 수위가 Coplin 용기의 절반이 되도록 한 다음 고압 설정에서 20분 동안 배양합니다.
참고: 더 나은 항원 회수를 위해 특수 압력솥 또는 은폐 챔버를 120°C에서 10분 동안 설정하여 사용할 수 있습니다. - 압력솥 단계를 완료한 후 슬라이드를 45-60분 동안 식힙니다. 슬라이드를 증류수로 2회씩 각각 2분씩 헹굽니다. 그런 다음 1x PBS가 들어 있는 용기로 슬라이드를 옮깁니다.
- 자가형광을 최소화하려면 항체로 염색하기 전에 조직에 표백 단계를 수행하십시오. 권장 시약과 농도를 사용하여 표백 용액(25x PBS 1mL + 0.8mL 1M NaOH + 4.5mL H2O2)을 갓 준비합니다. 실온에서 45분 동안 두 개의 LED 램프 사이에 표백 용액이 들어 있는 플라스틱 용기에 슬라이드를 배양합니다. 새 표백 용액으로 45분 더 반복합니다.
- 이 표백 단계에서 항체 칵테일 용액을 준비하기 시작하며, 특히 패널에 다양한 희석액이 있는 항체가 여러 개 있는 경우 더욱 그렇습니다. 4개의 차단제(G, S, J, N)를 모두 회수하여 얼음 위에 올려놓으며, 일부는 -20°C에서 해동하는 데 시간이 걸립니다.
- 광표백 후 PBS 1개, 2개씩 2분씩 조직을 세척합니다. 티슈를 수분 완충액이 들어 있는 병으로 옮기고 각각 2분씩 2회씩 씻습니다.
- 조직 슬라이드를 염색 완충액으로 옮기고 30분 동안 평형을 이룹니다. 표백 단계에서 준비되지 않은 경우 이 기간 동안 항체 칵테일 용액을 준비합니다.
- 두 개의 슬라이드를 염색하기 위해 300 μL의 항체 칵테일 용액을 준비합니다(일반적으로 혼성화 플라스틱 챔버를 사용하는 경우 100-120 μL의 항체 칵테일로 각 슬라이드를 염색하기에 충분합니다).
- 4°C에서 밤새 습도 챔버 내부에 항체 칵테일 용액을 도포하여 슬라이드를 염색합니다.
참고: 여기에 있는 모든 항체는 4°C에서 밤새 염색할 때 긍정적인 신호를 나타냅니다. 그러나 각 항체의 염색 시간과 온도는 다를 수 있으므로 최적화해야 합니다. 가장 일반적으로 사용되는 염색 조건은 RT에서 3시간 또는 4°C에서 밤새 염색하는 것입니다. 필요한 경우 처음에는 다른 온도에서 몇 가지 항체를 염색한 다음 4°C에서 밤새 나머지 항체를 염색할 수도 있습니다. 순차적인 염색이 필요한 경우 나중에 PBS로 조직을 세척하십시오. - 다음날, 염색 후 고정을 수행합니다. 플라스틱 챔버를 제거하고 수집된 항체 칵테일을 4°C에서 보관합니다. 염색 버퍼에서 슬라이드를 각각 2분씩 2회 세척합니다. 다음으로, 슬라이드를 40mL 염색 후 고정 용액(16% 파라포름알데히드 4mL + 보관 완충액 36mL)으로 옮기고 10분 동안 배양합니다. PBS로 3회씩 2분씩 헹굽니다.
- 슬라이드를 얼음처럼 차가운 메탄올에 5분 동안 넣은 다음 PBS 3x로 각각 2분 동안 헹굽니다. 최종 정착 용액(20μL, 튜브 1개 + PBS 1mL)을 습도 챔버 아래의 슬라이드에 도포합니다. 일반적으로 슬라이드당 200μL의 고착 용액으로 충분합니다. RT에서 고정액을 20분 동안 그대로 둡니다. PBS에서 각각 2분씩 3번의 최종 세척을 수행합니다.
알림: 미리 -20 °C에서 고정 튜브를 제거하지 마십시오. 적용하기 전에 만 해동하십시오. - 즉시 이미징하는 경우 보푸라기가 없는 티슈를 사용하여 조직 주위의 슬라이드를 닦고 플로우 셀 조립 장비를 사용하여 30초 동안 눌러 플로우 셀을 적용합니다. 나중에 이미징하는 경우 플로우 셀을 적용하지 않고 슬라이드를 4°C의 저장 버퍼에 보관하십시오. 이미징할 준비가 되면 플로우 셀을 적용하기 전에 10분 동안 스토리지 버퍼에서 PBS로 슬라이드를 전송합니다.
- 실험에서 원하는 사이클을 기반으로 첨가제가 포함된 실행 버퍼 1x 준비를 시작합니다. 원하는 사이클에 필요한 1-파트 러닝 버퍼와 4-파트 러닝 버퍼와 9-파트 DMSO로 혼합하여 낮은(1:4) 및 높은 DMSO 버퍼(9:1)를 준비합니다. 리포터 플레이트를 만들기 위한 첨가제와 함께 약 20mL의 1x 버퍼를 절약하십시오. 첨가제 및 고/저 DMSO 버퍼를 사용하여 필요한 1x 버퍼를 계산하려면 기기 관리자에게 문의하십시오.
- 리포터 플레이트를 준비하기 시작합니다. 먼저 총 사이클 수에 필요한 리포터 재고 솔루션을 만듭니다. 검은색 또는 호박색 1mL 마이크로 원심분리기 튜브에 사이클 이름을 표시합니다. 해당 튜브에 250μL의 리포터 스톡 용액과 5μL의 각 리포터를 추가합니다. 그런 다음 용액을 검은색 96웰 플레이트로 옮기고 접착 호일로 밀봉합니다.
- 장치를 시작하고 기기 관리자를 사용하여 매개변수와 노출 시간(표 1 참조)을 설정하고 이미지를 획득합니다. 그림 1 은 염색 전과 후 염색 과정을 요약한 것입니다.
5. 조직 주석 및 세포 세분화
- 프로젝트 및 작업 영역을 설정합니다.
- 최신 버전의 디지털 병리학 분석 소프트웨어(예: QuPath 버전 0.5.1 이상)를 설치합니다. 프로젝트 작성 을 클릭하여 프로젝트 공간의 대상 폴더를 선택합니다.
- Add images(이미지 추가) > Choose files(파일 선택)를 클릭한 다음 QPTIFF 파일(다중 면역형광 이미징에서 생성됨)로 이동합니다. 이미지 유형을 형광(Fluorescence)으로 설정하고 다른 모든 기본 설정은 유지한 다음 가져오기(Import)를 클릭합니다.
- QuPath를 사용하는 경우 새 이미지를 두 번 클릭하여 작업 공간을 엽니다. 파일 > 저장을 주기적으로 클릭하여 프로젝트의 변경 사항을 추적합니다. 툴바 중앙 근처에 있는 Brightness & contrast (half-moon 아이콘)를 사용하여 마커 가시성 및 보기 설정을 전환합니다.
참고: 이미지 목록에서 이미지를 마우스 오른쪽 버튼으로 클릭하여 이름을 바꿉니다. 이것은 나중에 표현형 분류를 시각화할 때 중요합니다.
- 전체 조직 절편, 종양 내 구획 및 기질 구획에 주석을 답니다.
- 브러시 및/또는 지팡이 도구를 사용하여 전체 조직 섹션 주위에 주석을 그립니다. 이 주석을 Full_Tissue로 정의합니다. 위에 있는 피부 및/또는 분석해서는 안 되는 부위는 제외합니다. 음수 주석을 작성하거나 주석 경계를 축소하려면 Alt 키를 누른 상태에서 주석 도구 중 하나를 사용합니다.
- 전체 조직 주석을 복제합니다. Annotations 탭에서 Annotations 를 선택한 다음 Objects > Annotations... > 선택한 주석을 복제합니다.
- SOX10 채널을 켭니다. 복제된 주석에서 Alt + 브러시/지팡이 도구를 사용하여 종양 내 구획을 캡처하도록 주석 경계를 축소합니다. 이 주석을 종양(Tumor)으로 정의합니다(그림 2).
- 전체 조직 주석을 선택하고 Objects > Annotations... > 주석을 확장합니다. 확장 반경을 1μm로 정의하고 실행을 클릭합니다.
- 새 주석의 이름을 Full_Tissue_Expansion로 바꿉니다. Tumor Annotation을 선택하고 마우스 오른쪽 버튼을 클릭한 다음 Insert in hierarchy를 선택합니다.
- Tumor Annotation을 다시 선택하고 Objects > Annotations... > 반전을 만듭니다. 이 새 주석을 Stroma로 정의합니다. 모든 조직 섹션에 대해 반복합니다.
- 셀 분할을 실행하고 결과를 내보냅니다.
- Image 탭으로 이동하여 이미지의 픽셀 너비와 높이를 찾습니다(그림 3A).
- https://github.com/qupath/qupath-extension-stardist/releases 에서 StarDist 확장 프로그램을 다운로드하십시오. qupath-extension-stardist-[version].jar 파일을 QuPath 창으로 드래그한 다음 QuPath 창의 오른쪽 상단 모서리에 있는 톱니바퀴 아이콘을 클릭합니다. 확장에서 QuPath 사용자 디렉터리를 StarDist 폴더의 위치로 정의합니다.
- https://github.com/xzhou125/JOVE_QuPath_Spatial_Analysis 에서 StarDist 그루비 스크립트 파일과 StarDist 모델 파일을 다운로드합니다(StarDist 스크립트는 원래 Akoya Biosciences에서 구성되었습니다).
- 각 조직 절편에 대해 Tumor 및 Stroma 주석을 모두 선택합니다(Ctrl을 사용하여 여러 주석 선택). 상단 설정 표시줄에서 > 스크립트 편집기 자동화를 클릭하여 스크립트 인터페이스를 엽니다. 이미지 픽셀 크기에 해당하는 적절한 StarDist 셀 분할 스크립트(groovy 파일)를 엽니다. 셀 구분 파일을 선택하라는 메시지가 표시되면 stardist_cell_seg_model.pb 파일을 엽니다.
- 셀 분할 후 이미지를 저장합니다. Measure > Export measurements(측정 내보내기)로 이동한 다음 해당 이미지를 선택합니다. Export type(내보내기 유형)을 Cells(셀)로 할당하고 Separator(구분 기호)를 .csv로 할당한 다음 Output file location(출력 파일 위치)을 선택합니다. Columns 옆에 있는 Populate 를 클릭하여 관심 있는 메트릭을 포함합니다. 내보낼 중요한 열은 이미지(Image), 개체 ID(Object ID), 상위(Parent), 중심 X(Centroid X), 중심 Y(Centroid Y) 및 핵 영역(Nucleus Area)입니다.
참고: 개체 ID는 디지털 병리학 분석 소프트웨어의 다중 면역형광 이미지로 분류를 다시 매핑하기 위해 후속 단계에서 사용됩니다. Parent 열은 셀이 속한 주석 이름을 나타냅니다. X 및 Y 데이터는 공간 분석에 사용됩니다. - 클러스터링/표현형에 사용할 각 계통 마커에 대해 내보낼 평균값을 하나 선택합니다. 핵 마커(예: SOX10 및 FOXP3)의 경우 Nucleus: Mean 옵션을 내보냅니다. 세포질/멤브레인 마커(예: CD45, CD3, CD4)의 경우 Cytoplasm: Mean 옵션을 내보냅니다.
6. 단백질체학 데이터 전처리 및 정규화
- 내보낸 CSV 파일을 엽니다. 클러스터링/표현형에 사용할 각 마커에 대해 마커만 포함하도록 열 머리글의 이름을 자릅니다. 예를 들어 SOX10: Nucleus: Mean을 SOX10으로 바꾸고 CD3: Cytoplasm: Mean을 CD3로 이름을 바꿉니다.
- 최신 버전의 R 및 RStudio를 설치합니다. https://github.com/xzhou125/JOVE_QuPath_Spatial_Analysis 에서 Marker Normalization.R 파일을 다운로드하고 스크립트를 실행합니다. 이것은 먼저 핵 크기별로 세포를 필터링하는 것과 관련이 있습니다.
- 그런 다음 각 마커에 대해 최소-최대 정규화가 수행되어 표현식 범위에서 가장 낮은 MFI 값이 0으로 설정되고 99.7번째 백분위수의 MFI 값이 1로 설정됩니다. 99.7번째 백분위수 이상의 모든 농도는 1에서 잘립니다. 완료되면 정규화된 데이터로 생성된 새 CSV 파일을 저장합니다.
7. 클러스터링 및 표현형 분석
- marker expression profiles에 따라 셀을 클러스터링하기 위한 알고리즘을 선택합니다. https://github.com/xzhou125/JOVE_QuPath_Spatial_Analysis 에서는 Seurat v4.415 및 FlowSOM v2.13.9(클러스터링 및 시각화 도구)16을 사용하여 클러스터링을 위한 R 스크립트가 제공됩니다.
- key lineage markers의 marker expression profiles에 따른 표현형 세포. 여기에 제시된 프로토콜은 클러스터링 및 표현형을 위해 14개의 계통 마커(SOX10, CD45, CD3, CD4, CD8, FOXP3, CD11C, F4/80, CD68, CD86, CD163, CD206, NK1.1 및 CD31)를 사용했습니다.
- 종양 세포를 SOX10hi로 정의합니다. CD4 T cell을 CD45mod-hi/ CD4hi/CD8low/FOXP3low로 정의합니다. CD8 T cell을 CD45mod-hi/ CD8hi/CD4low로 정의합니다. Tregs를 CD45mod-hi/CD4hi/CD8low/FOXP3hi로 정의합니다. 다른 T cell을 CD45mod-hi/CD3hi/CD4low/CD8low로 정의합니다.
- M1 대식세포를 CD45,mod-hi/F4/80hi/CD68hi/CD86hi/CD163low , /CD206low로 정의합니다. M2 대식세포를 CD45mod-hi/F4/80hi/CD68mod-hi/CD86low/CD163hi/CD206low, CD45mod-hi/F4/80hi/CD68mod-hi/CD86low/CD163low/CD206hi 또는 CD45mod-hi/F4/80hi/CD68mod-hi/CD86low/CD163hi/CD206hi로 정의합니다. 다른 대식세포를 CD45,mod-hi/F4/80,hi/CD68,mod-hi/CD86low/CD163low/CD206low , 또는 CD45,mod-hi/F4/80hi/CD68,mod-hi/CD86hi/CD163,hi/CD206hi로 정의합니다.
- 수지상 세포를 CD45mod-hi/CD11chi/CD86mod-hi로 정의합니다. NK 셀을 CD45mod-hi/NK1.1hi로 정의합니다. 다른 면역 세포를 위에 설명된 조건을 충족하지 않는 CD45mod-hi 로 정의합니다. 내피 세포를 CD31mod-hi로 정의합니다. 다른 모든 표현식 프로파일을 제거합니다.
8. 표현형 분류 재매핑 및 품질 관리 수행
- https://github.com/xzhou125/JOVE_QuPath_Spatial_Analysis 에서 Remapping Classifications.groovy 스크립트를 다운로드합니다(이 스크립트는 petebankhead의 https://forum.image.sc/ 게시물에서 수정됨).
- 표현형이 지정된 CSV 파일을 열고 이미지 탭 아래의 모든 레이블이 다시 매핑에 사용할 디지털 병리학 분석 소프트웨어의 이미지 파일 이름과 정확히 일치하는지 확인합니다. CSV 파일에서 많은 양의 레이블을 빠르게 변경하려면 모두 바꾸기 기능을 사용합니다.
- QuPath를 사용하는 경우 > 스크립트 편집기 자동화를 클릭하여 분류 재매핑 groovy 파일을 실행합니다.
- Annotations 탭에서 Auto set 버튼 옆에 있는 Three Dots 를 클릭한 다음 Populate from existing objects > All classes (including subclasses) > Yes를 클릭합니다.
- 마커 목록에서 아래로 스크롤하여 다양한 표현형 분류를 보고 필요에 따라 색 구성표를 조정합니다. 아무 셀이나 두 번 클릭하면 해당 표현형(Classification 태그 옆)과 클러스터(Name 태그 옆)를 볼 수 있습니다. 대표 시야를 확인하여 표현형 할당의 정확도를 결정하고 필요에 따라 표현형 분석 전략을 수정합니다(그림 3B).
9. 밀도 정량화 및 공간 분석
- 표현형 분석이 완료되면 Annotations 탭 아래의 디지털 병리학 분석 소프트웨어에서 주석 영역을 가져옵니다. 면적 값을 mm2로 변환한 다음 개수를 주석 영역으로 나누어 밀도를 계산합니다.
- https://github.com/xzhou125/JOVE_QuPath_Spatial_Analysis 에서 SPIAT Spatial Analysis.R 스크립트를 다운로드하여 실행합니다. 평균 최소 거리(AMD), 이웃 구성 및 정규화된 혼합 점수(NMS)에 대한 데이터를 수집합니다.
- 통계 분석 소프트웨어에서 수치를 생성하여 결과를 제시합니다. 치료 조건의 평균 최소 거리에 대한 단일 히트맵을 생성할 때 다른 조직 절편에서 얻은 결과의 평균을 구합니다.
결과
여기에서는 쥐 FFPE 조직에 대한 항체 패널을 설계하고, 다중 면역형광 이미징을 수행하고, 단백질체학 정량화 및 공간 관계를 위한 이미지를 분석하기 위한 프로토콜을 제시합니다. 검증된 패널에는 흑색종 세포(SOX10), 백혈구(CD45), T 세포(CD3, CD4, CD8, FOXP3), B 세포(CD20), 대식세포 및 아형(F4/80, CD68, CD86, CD163, CD206), 수지상 세포(CD11c), NK 세포(NK1.1) 및 내피 세포(CD31)를 시각화하기 위한 마커를 제공하는 27개의 항체가 포함되어 있습니다. 전체 패널에는 다른 면역 집단(CD11b, CD38), 증식 활성(Ki67), T 세포 기능(T-bet, Eomesodermin, granzyme B), 항원 발현(LMP2, beta-2 microglobulin, MHC II) 및 관문 발현(TIM3, LAG3, PD-L1)에 대한 마커도 포함되어 있습니다12. 대표적인 겔 전기영동 이미지는 그림 4와 같이 DNA 올리고뉴클레오티드 바코드가 캐리어가 없는 항체에 성공적으로 접합되었음을 확인합니다. 이 단계는 화학 반응만 확인하며, 이미지 확인은 관심 조직에서 멀티플렉스 이미징 장치로 이러한 항체를 확인한 후에만 수행할 수 있습니다. 패널13에서 27개의 마커 모두의 검증된 이미지를 보려면 다음 참조를 참조하십시오. 종양내 4-1BBL/IL-12 나노입자 주사와 전신 항-PD1로 처리된 3개의 쥐 흑색종 조직 절편을 염색하는 주요 계통 마커의 융합 이미지가 그림 5에 제시되어 있습니다. 이 섹션은 이 프로토콜에서 후속 이미지 분석에 사용되었습니다.
조직 주석, 세포 세분화 및 단백질체학 데이터 전처리 후, 이전에 단일 세포 전사체 및/또는 단백질체학 분석에 적용된 두 가지 클러스터링 알고리즘을 비교합니다17,18. 표현형화된 집단에 대한 발현 프로파일은 두 가지 접근 방식 모두에 대해 그림 6에 제시되어 있습니다. 우리는 FlowSOM이 대식세포 아형과 같은 유사한 세포 집단 간의 차이를 식별하기 위해 더 넓은 범위의 강도 값(~0.7 대 ~0.5)을 제공한다는 것을 보여줍니다. Seurat는 클러스터링 중에 Louvain 알고리즘을 적용하여 29개의 클러스터를 생성했습니다(보충 그림 1). 이에 비해 FlowSOM은 자기 조직화 맵을 적용했으며 100개의 클러스터를 생성할 수 있습니다(보충 그림 2). 클러스터 수가 많을수록 표현형에 더 많은 시간이 필요하지만, 이 후자의 FlowSOM 접근 방식은 유사한 세포 집단을 분류할 때 더 많은 뉘앙스를 제공합니다. 질적으로, FlowSOM은 동일한 시야에서 Seurat 표현형과 비교할 때 더 많은 종양내 대식세포를 M1 또는 M2 아형으로 분류할 수 있음을 알 수 있습니다(그림 7). 대식세포 밀도를 정량화할 때도 동일한 결과가 나타나는데, FlowSOM은 Seurat에 비해 M1 및 M2 대식세포의 밀도가 더 높고(그림 8A-B) 그 결과 다른/분류되지 않은 대식세포의 밀도가 현저히 낮아졌습니다(p = 0.0028; 그림 8C). 그럼에도 불구하고 두 가지 분석 접근 방식은 CD4 T 세포, CD8 T 세포 및 내피 세포와 같은 다른 세포 집단 밀도를 설명할 때도 유사한 결과를 생성했습니다(그림 8D-F).
또한 클러스터링 및 표현형 분석 후의 다운스트림 공간 분석 결과를 제시합니다. 그림 9 는 이 프로토콜을 사용하여 생성할 수 있는 몇 가지 공간 메트릭을 보여 줍니다. 모든 표현형 집단에 비해 M2 대식세포와 NK 세포는 4-1BBL/IL-12 나노입자 및 항-PD1 처리 후 AMD가 가장 높았습니다(그림 9A). 마찬가지로, 종양내 CD8 T 세포와 M1 대식세포 간의 NMS는 CD8 T 세포와 M2 대식세포 간의 NMS보다 훨씬 높았습니다(그림 9B). 또한, M2 대식세포는 종양내 CD8 T 세포를 둘러싼 100μm 인근에서 ~1%를 기여한 반면, M1 대식세포는 이러한 인근의 9%-13%를 차지했습니다(그림 9C). 이러한 결과를 종합해 볼 때, 4-1BBL/IL-12 치료 요법은 종양 관련 대식세포를 M1 아형으로 편극화하고 M2 대식세포를 종양 면역 미세환경에서 배제했습니다.
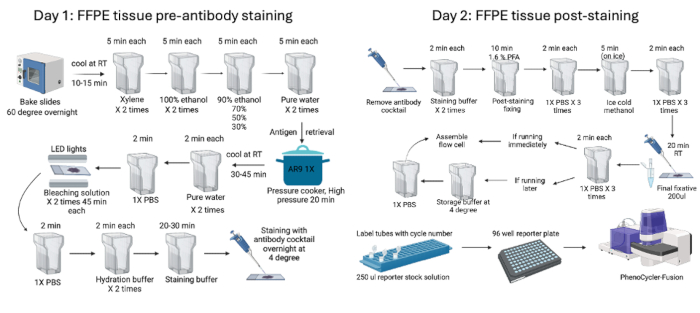
그림 1: FFPE 조직 염색 및 이미징 워크플로우 요약. 쥐 FFPE 조직은 2일간의 사전 염색(1일차) 및 염색 후(2일차) 공정을 시작하기 전에 밤새 조직을 베이킹하는 것으로 시작하는 사전 염색 절차를 사용하여 처리되었습니다. 마지막으로, 장치로 이미징하기 전에 리포터 플레이트를 준비했습니다. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
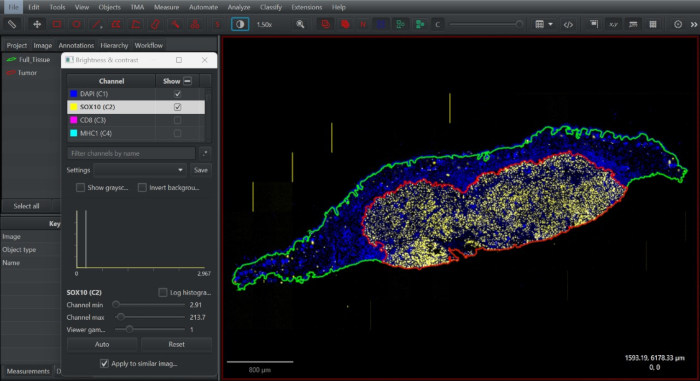
그림 2: 오픈 액세스 디지털 병리학 소프트웨어 QuPath의 조직 주석 스크린샷. 종양 경계(빨간색)에서 SOX10+ 멜라닌 세포의 분포에 따라 종양 내 구획을 정의하기 위해 전체 조직 주석을 그리고(녹색), 복제하고 최소화했습니다. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
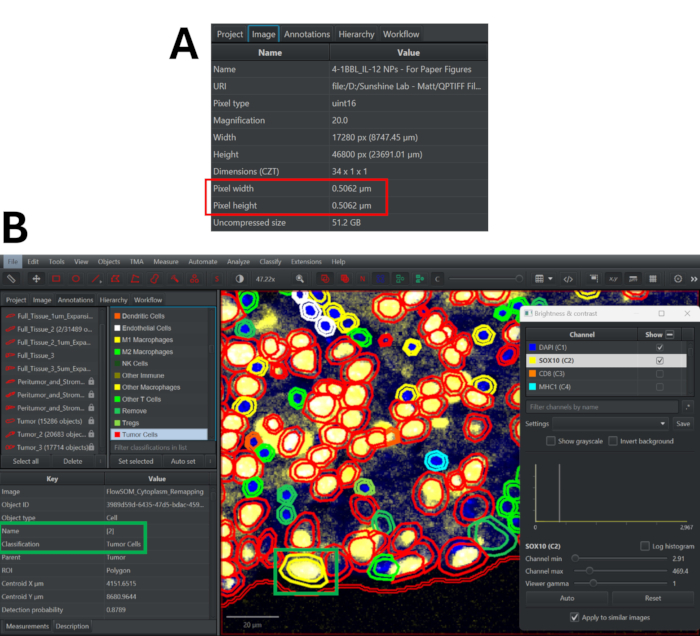
그림 3: QuPath의 StarDist 알고리즘을 사용한 세포 세분화 및 표현형 분류 검토를 위한 품질 관리 프로세스. (A) 이미지 탭으로 이동하여 멀티플렉스 면역형광 이미지의 픽셀 너비와 높이를 확인합니다. (B) 분류를 다시 매핑한 후 아무 셀(노란색으로 강조 표시됨)을 두 번 클릭하여 클러스터 할당 및 표현형을 확인합니다. 패널 마커를 켜거나 끄면 이 분류 방법이 정확한지 확인할 수 있습니다. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
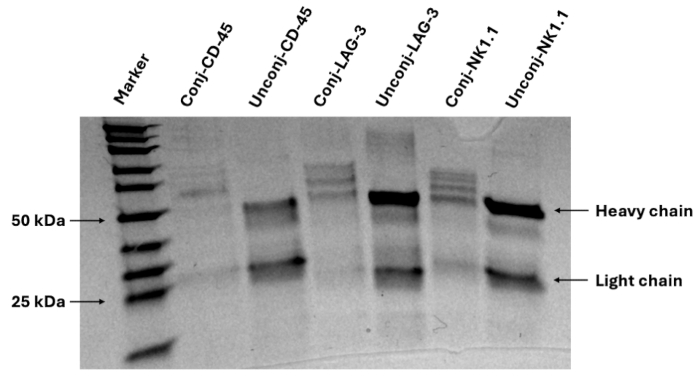
그림 4: 항체-DNA 바코드 접합 확인의 대표적인 겔 이미지. 단백질 겔 전기영동은 중쇄 부위의 추가 밴드에 의해 관찰되는 DNA 올리고뉴클레오티드 바코드와의 항체 접합을 확인합니다. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
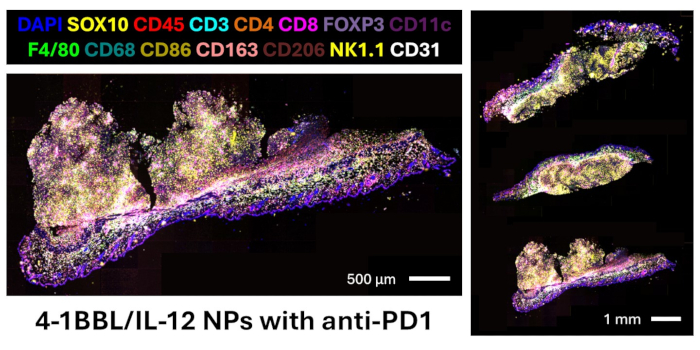
그림 5: 전신 항-PD1을 이용한 4-1BBL/IL-12 나노입자의 종양내 주사로 치료한 B16F10 옆구리 종양의 DNA 바코드 기반 멀티플렉스 이미징. 패널의 마커가 표시되지 않음: CD11b, CD20, CD38, TIM3, LAG3, T-bet, Eomesodermin, granzyme B, Ki67, LMP2, beta-2 microglobulin, MHC II, PD-L1. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
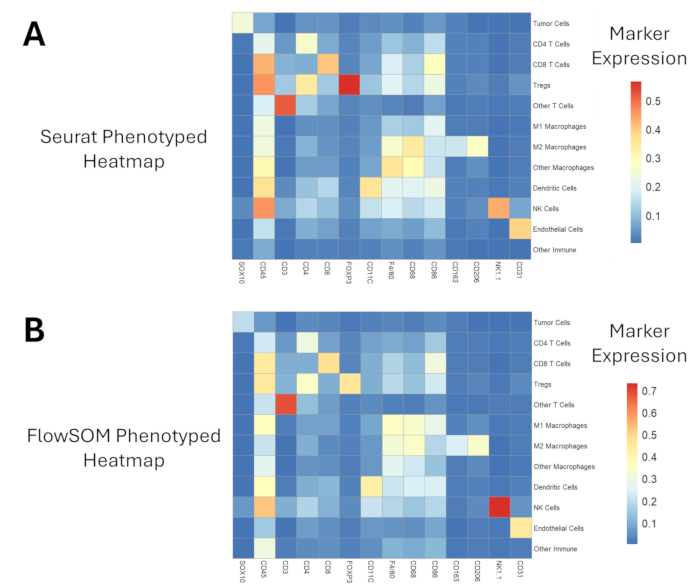
그림 6: 표현형화된 세포 집단의 단백질체학 발현 히트맵. (A) Seurat 클러스터링 및 표현형 분석 접근 방식은 (B) FlowSOM 클러스터링 및 표현형에 비해 약간 더 작은 범위의 마커 발현 값을 생성합니다. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
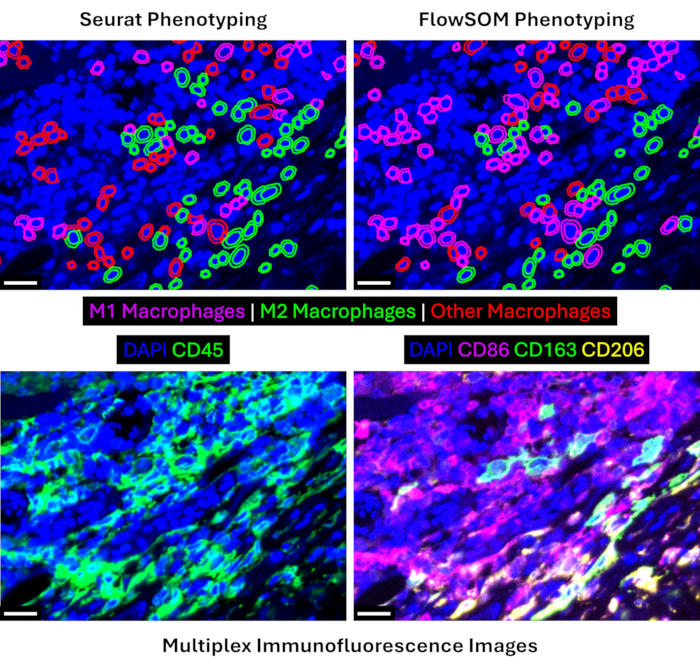
그림 7: FlowSOM 표현형은 더 넓은 범위의 다양한 대식세포 아형을 식별합니다. 상단 패널은 동일한 시야에 대한 Seurat(왼쪽) 및 FlowSOM(오른쪽) 대식세포 표현형을 보여줍니다. 하단 패널은 동일한 시야에서 주요 대식세포 계통 마커의 다중 면역 형광 이미지를 보여줍니다. 모든 스케일 바는 20μm입니다. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
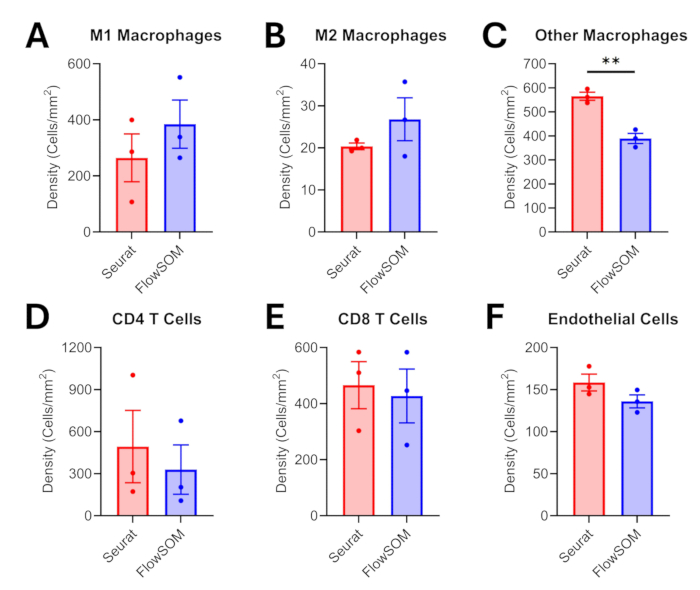
그림 8: 클러스터링/표현형 분석 후 서로 다른 세포 집단의 종양 내 밀도 비교. 종양내 (A) M1 대식세포, (B) M2 대식세포, (C) 기타 대식세포, (D) CD4 T 세포, (E) CD8 T 세포 및 (F) 내피 세포에 대한 밀도 비교가 표시됩니다. 오차 막대는 평균의 표준 오차(SEM)이며, 유의성은 쌍을 이루지 않은 t-검정(**p ≤ 0.01)을 사용하여 검정되었습니다. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
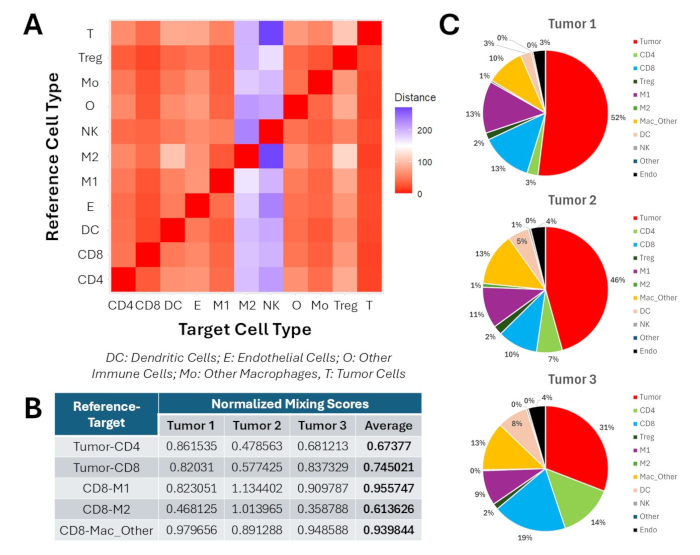
그림 9: 4-1BBL/IL-12 종양내 나노입자 주사 및 전신 항-PD1로 치료한 3개의 옆구리 종양에 대한 종양내 공간 메트릭 프로파일링. (A) 종양내 표현형 집단 간의 평균 최소 거리의 히트맵. 측정 단위는 μm입니다. (B) 주요 종양내 표현형 쌍 간의 정규화된 혼합 점수. (C) 종양내 CD8 T 세포 집단 주변 100μm 반경 내 이웃 조성의 분석. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
| 항체 | 희석 | 노출 시간(ms) |
| 삭스-10 | 50 | 450 |
| CD8 | 100 | 450 |
| CD3 | 100 | 450 |
| 폭스P3 | 50 | 350 |
| CD4 | 50 | 450 |
| MHC-II | 100 | 450 |
| PDL1 | 50 | 450 |
| CD45 | 100 | 450 |
| 기-67 | 100 | 300 |
| F4/80 | 100 | 150 |
| CD20 | 100 | 450 |
| NK1.1/CD161 | 50 | 450 |
| CD206 | 100 | 450 |
| CD68 | 100 | 450 |
| 그랑자임-B | 50 | 450 |
| CD86 | 100 | 150 |
| CD31 | 100 | 450 |
| CD11C | 50 | 450 |
| CD11b | 50 | 450 |
| EOMES | 50 | 450 |
| 팀-3 | 50 | 300 |
| CD38 | 50 | 200 |
| 지연3 | 50 | 450 |
| CD163 | 50 | 200 |
| T-벳 | 50 | 300 |
| LMP2 | 100 | 100 |
| 베타2 MG | 200 | 50 |
표 1: 항체 희석 및 노출 시간 설정.
보충 그림 1: Seurat 클러스터링 후 생성된 초기 단백질체학 발현 히트맵. 이 그림을 다운로드하려면 여기를 클릭하십시오.
보충 그림 2: FlowSOM 클러스터링 후 생성된 초기 단백질체학 발현 히트맵. 이 그림을 다운로드하려면 여기를 클릭하십시오.
토론
이미징의 성공 여부는 잘 설계되고 검증된 항체 패널에 달려 있습니다. FFPE 샘플의 다중 면역형광 이미징은 높은 자가형광과 파라핀 포매링으로 가려진 에피토프를 회수하는 어려움으로 인해 어려움을 겪습니다. 그러나 FFPE가 FF 표본에 비해 몇 가지 이점을 제공한다는 점을 감안할 때 FFPE 항체 패널을 설계하고 검증하는 것이 필수적입니다. 면역형광(IF) 이미징 중에 양성 신호를 보이는 항체 클론을 마무리하는 것이 첫 번째 단계입니다. 그런 다음 DNA 바코드와 조심스럽게 결합하는 것이 중요합니다. 항체 접합은 SH 결합을 생성하기 위해 항체의 부분적인 감소를 필요로 하며, 이는 바코드를 통한 말레이미드기 반응 중에 사용됩니다. 모든 항체 클론이 이 단계를 견딜 수 있는 것은 아니며, 일부 반응은 항체에 돌이킬 수 없는 손상을 일으켜 성공적인 접합에도 불구하고 이미징 실패를 초래할 수 있습니다. 이러한 이유로, 일부 항체가 기존 IF 검증 중에 긍정적인 신호를 보일 수 있지만, 항체 접합의 최종 성공 여부를 평가하기 위해서는 실제 관심 조직에서 각 항체를 검증하고 해당 조직에 대해 원하는 노출 시간을 기록하는 것이 중요합니다. 향후 응용 분야에서는 이 기술을 연속 슬라이드/동일한 슬라이드에 대한 기존 공간 전사체학 분석과 결합하여 추가 통찰력을 생성할 수 있습니다. 이 방법의 한계 중 하나는 표적과 조직 유형에 따라 패널의 각 항체를 신중하게 선택하고 검증해야 한다는 것입니다.
이미지 분석과 관련하여 QuPath는 단백질체학 마커의 고품질 시각화, 강도 측정 내보내기 및 표현형 분류를 위한 품질 관리 수행을 위한 광범위한 기능, 사용자 생성 스크립트에 대한 우수한 유연성을 갖춘 귀중한 개방형 액세스 도구를 제공합니다. https://forum.image.sc/ 와 같은 온라인 포럼은 특정 분석 작업을 수행하는 방법을 논의하고 다른 사용자와 스크립트를 공유할 수 있는 추가 리소스입니다. 이 프로토콜에서는 Seurat 및 FlowSOM을 사용하여 두 가지 클러스터링 및 표현형 분석 접근 방식을 비교합니다. FlowSOM은 TME의 면역 세포 하위 집단에 대한 보다 세분화된 통찰력을 생성할 수 있는 능력 때문에 선호될 수 있지만, 단백질체학 분석에 필요한 시간도 고려해야 합니다. 사용자가 하나 또는 두 개의 조직 샘플 내에서 세포를 표현형으로 분석해야 하는 경우 100개의 클러스터를 생성하는 것이 불필요할 수 있습니다. 이러한 상황에서 Seurat는 이미지 분석을 위한 더 빠르고 효율적인 파이프라인을 제공할 수 있습니다. 대조적으로, 40개 또는 50개 이상의 조직 절편이 있는 TMA를 분석하면 두 분석 접근 방식 모두에서 더 많은 수의 세포 클러스터가 생성될 가능성이 더 높으며, FlowSOM은 보다 미묘한 표현형 분류를 생성하기 위해 선호되는 방법론일 수 있습니다.
세포 클러스터링/표현형 분석 및 모든 후속 이미지 분석 단계는 세포 분할에 크게 의존합니다. 우리의 현재 연구는 HALO (Indica Labs) 및 StarDist 알고리즘의 세포 분할을 탐구했으며, 두 접근 방식 모두 핵 DAPI 신호를 기반으로 세포를 과도하게 분할하는 경향이 있음을 발견했습니다. Mesmer19 및 InstanSeg20과 같은 많은 대체 세분화 알고리즘도 사용할 수 있습니다. 이것은 추가 탐색과 최적화가 필요한 컴퓨팅 연구의 성장 분야입니다.
공개
J.C.S.는 Emerson Collective LLC와 미국 국립보건원(National Institutes of Health)의 재정적 지원을 인정합니다. J.J.G.는 또한 미국 국립보건원(National Institutes of Health)의 자금 지원도 인정합니다. J.C.S.는 Palleon Pharmaceuticals Inc.와 관계를 맺고 있으며, 여기에는 기금 지원금이 포함됩니다. S.Y.T. 및 J.J.G.는 주식 또는 주식과 관련된 OncoSwitch Therapeutics와 관계를 맺고 있습니다. S.Y.T., J.J.G., J.C.S. 및 K.M.L.은 특허 출원 중입니다. 다른 모든 저자는 이 논문에 제시된 연구에 영향을 미친 것으로 인식될 수 있는 경쟁적인 재정적 이해관계나 개인적 관계를 알려진 바가 없다고 진술합니다.
감사의 말
J.C.S.는 저자의 경력을 발전시킨 공로로 Dermatology Foundation과 Dermatopathology Career Development Award를 인정합니다. 저자는 컴퓨터 분석 기술에 대한 도움을 준 미국 국립 암 연구소(National Cancer Institute)의 암 데이터 과학 연구소(Cancer Data Science Laboratory)의 Hsin-Pei Lee에게 감사를 표합니다. 이 연구는 Emerson Collective와 National Institutes of Health(R37CA246699, P41EB028239 및 R01CA228133)로부터 자금을 지원받았습니다. 또한 존스 홉킨스 종양학 조직 서비스(OTS) 핵심은 미국 국립보건원(P30CA006973)의 지원을 받습니다.
자료
| Name | Company | Catalog Number | Comments |
| 16% paraformaldehyde (PFA) | Electron Microscopy Sciences | PN# 15710 | Required during tissue staining process |
| 50kDa MWCO filter | Millipore | UFC505096 | 25 kDa and 100 kDa result in failure |
| Akoya barcodes and reporters | Akoya Biosciences | Varied | Each barcode can be used to conjugate 50 ug of carrier free antibody and comes with two vials of reporters |
| Antibody conjugation kit | Akoya Biosciences | 7000009 | A conjugation kit is used to create custom barcode-conjugated antibodies. Each kit contains sufficient reagents for ten conjugations. The kit consists of one subkit box stored at 4 °C and one subkit box stored at -20 °C. The 4 °C subkit includes Filter Blocking Solution, Reduction Solution 2, Conjugation Solution, Purification Solution, and Antibody Storage Solution. The -20 °C subkit includes Reduction Solution 1. |
| Beta2MG | Abcam | ab214769 | |
| CD11b | CST | #41028 | |
| CD11C | CST | #39143SF | |
| CD163 | CST | #68922BF | |
| CD20 | CST | #45839 | |
| CD206 | CST | #87887 | |
| CD3 | CST | #24581 | |
| CD31 | CST | #92841 | |
| CD38 | CST | #68336BF | |
| CD4 | Abcam | ab271945 | |
| CD45 | CST | #98819 | |
| CD68 | CST | #29176 | |
| CD8 | Invitrogen | #14-0808-82 | |
| CD86 | CST | #20018 | |
| Dimethyl sulfoxide | Avantor/VWR | BDH1115-4LP | |
| EOMES | Abcam | ab222226 | |
| Eppendorf PCR tubes | Eppendorf | E0030124286 | Required store 5 μL conjugated antibody for conjugation confirmation by gel electrophoresis |
| Ethanol 200 proof | |||
| F4/80 | CST | # 25514 | |
| FoxP3 | CST | #72338 | |
| Gran-B | CST | #79903SF | |
| Heating oven | |||
| Hybridization chamber | Used to stain tissue with antibody cocktail | ||
| Hydrogen peroxide | Sigma | #216763 | Required for preparing bleaching solution |
| Instant pot pressure cooker or Decloaking Chamber ARC | Instant pot or Biocare Medical | ||
| Ki-67 | Akoya Biosciences | ||
| LAG3 | CST | #80282BF | |
| LMP2 | Abcam | ab243556 | |
| Methanol | |||
| MHC-II | eBioscience | #14-5321-82 | |
| NK1.1 | CST | #24395SF | |
| Nuclear Stain | Akoya Biosciences | 7000003 | |
| PDL1 | CST | #85095 | |
| Salmon Sperm DNA, sheared (10 mg/mL) | Invitrogen | AM9680 | Can be used as an alternative to assay reagent (Akoya) during reporter plate preparation step. |
| SOX-10 | Abcam | ab245760 | |
| Staining kit for PhenoCycler-Fusion | Akoya Biosciences | 7000017 | The PhenoCycler-Fusion Sample Kit includes the buffers and reagents necessary for tissue staining using antibodies conjugated with PhenoCycler barcodes, along with flow cells for whole-slide imaging. Each kit is sufficient for 10 PhenoCycler-Fusion experiments. It consists of one subkit stored at 4 °C, another at -20 °C, and a package of 10 flow cells kept at room temperature. The subkit stored at 4 °C contains Hydration Buffer, Staining Buffer, Storage Buffer, N Blocker, and J Blocker. The subkit stored at -20 °C contains G Blocker, S Blocker, and Fixative Reagent. |
| T-bet | CST | #53753 | |
| TIM-3 | CST | #72911 | |
| UltraPure DNase/RNase-free distilled water | Invitrogen | 10977015 | Required to dissolve barcodes during antibody conjugation |
참고문헌
- Schadendorf, D., et al. Melanoma. Lancet. 392 (10151), 971-984 (2018).
- Taube, J. M., et al. Implications of the tumor immune microenvironment for staging and therapeutics. Modern Pathol. 31 (2), 214-234 (2018).
- Marzagalli, M., Ebelt, N. D., Manuel, E. R. Seminars in cancer biology. 59, 236-250 (2019).
- De Visser, K. E., Joyce, J. A. The evolving tumor microenvironment: From cancer initiation to metastatic outgrowth. Cancer cell. 41 (3), 374-403 (2023).
- Balkwill, F. R., Capasso, M., Hagemann, T. The tumor microenvironment at a glance. J Cell Sci. 125 (23), 5591-5596 (2012).
- Phillips, D., et al. Highly multiplexed phenotyping of immunoregulatory proteins in the tumor microenvironment by codex tissue imaging. Front Immunol. 12, 687673(2021).
- Giesen, C., et al. Highly multiplexed imaging of tumor tissues with subcellular resolution by mass cytometry. Nat Methods. 11 (4), 417-422 (2014).
- Keren, L., et al. Mibi-tof: A multiplexed imaging platform relates cellular phenotypes and tissue structure. Sci Adv. 5 (10), eaax5851(2019).
- Lin, J. R., Fallahi-Sichani, M., Sorger, P. K. Highly multiplexed imaging of single cells using a high-throughput cyclic immunofluorescence method. Nat Comm. 6 (1), 8390(2015).
- Radtke, A. J., et al. Ibex: An iterative immunolabeling and chemical bleaching method for high-content imaging of diverse tissues. Nat Protoc. 17 (2), 378-401 (2022).
- Black, S., et al. Codex multiplexed tissue imaging with DNA-conjugated antibodies. Nat Protoc. 16 (8), 3802-3835 (2021).
- Aung, T. N., Bates, K. M., Rimm, D. L. High-plex assessment of biomarkers in tumors. Mod Pathol. 37 (3), 100425(2024).
- Surwase, S. S., et al. Highly-multiplexed immunofluorescence phenocycler panel for murine ffpe yields insight into tumor microenvironment immunoengineering. Lab Invest. 105 (1), 102165(2024).
- Luly, K. M., Green, J. J., Sunshine, J. C., Tzeng, S. Y. Biomaterial-mediated genetic reprogramming of merkel cell carcinoma and melanoma leads to targeted cancer cell killing in vitro and in vivo. ACS Biomater Sci Eng. 9 (11), 6438-6450 (2023).
- Satija, R., Farrell, J. A., Gennert, D., Schier, A. F., Regev, A. Spatial reconstruction of single-cell gene expression data. Nat Biotechnol. 33 (5), 495-502 (2015).
- Van Gassen, S., et al. Flowsom: Using self-organizing maps for visualization and interpretation of cytometry data. Cytometry Part A. 87 (7), 636-645 (2015).
- Butler, A., Hoffman, P., Smibert, P., Papalexi, E., Satija, R. Integrating single-cell transcriptomic data across different conditions, technologies, and species. Nat Biotechnol. 36 (5), 411-420 (2018).
- Watson, S. S., et al. Microenvironmental reorganization in brain tumors following radiotherapy and recurrence revealed by hyperplexed immunofluorescence imaging. Nat Comm. 15 (1), 3226(2024).
- Greenwald, N. F., et al. Whole-cell segmentation of tissue images with human-level performance using large-scale data annotation and deep learning. Nat Biotechnol. 40 (4), 555-565 (2022).
- Goldsborough, T., et al. Instanseg: An embedding-based instance segmentation algorithm optimized for accurate, efficient and portable cell segmentation. arXiv. , (2024).
재인쇄 및 허가
JoVE'article의 텍스트 или 그림을 다시 사용하시려면 허가 살펴보기
허가 살펴보기더 많은 기사 탐색
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. 판권 소유