Method Article
DNA-Barcode-basierte Multiplex-Immunfluoreszenz-Bildgebung zur Analyse von FFPE-Proben von genetisch umprogrammierten murinen Melanomen
* Diese Autoren haben gleichermaßen beigetragen
In diesem Artikel
Zusammenfassung
Dieses Protokoll bietet eine Schritt-für-Schritt-Anleitung für die Entwicklung eines Multiplex-Immunfluoreszenz-Antikörper-Panels für die DNA-Barcode-basierte Bildgebung von murinen FFPE-Melanomgeweben. Wir beschreiben auch eine Bildanalyse-Pipeline mit Open-Source-Tools zur Generierung von räumlichen Proteomik-Einblicken in die Immunmikroumgebung des Mausmelanomtumors.
Zusammenfassung
Vorgestellt wird eine aufstrebende DNA-Barcode-basierte Multiplex-Bildgebungstechnik, die auf Co-Detection-by-indEXing basiert und die räumliche Proteomik von Gewebemikroumgebungen analysiert. Für eine erfolgreiche Bildgebung ist ein Repertoire an gut konzipierten und ordnungsgemäß validierten Antikörper-Panels erforderlich, aber für formalinfixierte paraffineingebettete (FFPE) Proben gibt es derzeit nur sehr wenige. FFPE bietet mehrere Vorteile gegenüber frisch gefrorenen Proben, wie z. B. die weit verbreitete Verfügbarkeit, die einfache Handhabung und Lagerung sowie die Möglichkeit, Gewebe-Microarrays (TMAs) herzustellen. Hier stellen wir ein Protokoll zur Entwicklung eines Antikörper-Panels zur Visualisierung und Analyse von FFPE-Geweben aus einem murinen Melanommodell vor, das mit Nanopartikeln behandelt wurde, die Plasmid-DNA liefern, die für immunologische Signale für die Reprogrammierung der Tumormikroumgebung kodiert. Wir beschreiben auch eine Bildanalyse-Pipeline mit Open-Source-Computerwerkzeugen für die Annotation von Geweben, die Segmentierung von Zellen, die Verarbeitung von Proteomik-Daten, die Phänotypisierung von Zellpopulationen und die Quantifizierung räumlicher Metriken. Das Protokoll bietet Anwendungen für das Design von Antikörper-Panels in muriner FFPE und die Generierung neuer Einblicke in die räumliche Proteomik komplexer Gewebemikroumgebungen.
Einleitung
Das kutane Melanom ist der häufigste Hautkrebs mit weltweit unterschiedlichen Krankheits- und Sterblichkeitsraten, je nach Zeitpunkt der Diagnose und der Primärversorgung1. In den letzten zehn Jahren hat ein verbessertes biologisches Verständnis des Melanoms dazu beigetragen, die Entwicklung neuer Krebsmodelle zur Behandlung solider Tumore voranzutreiben2. Das jüngste Aufkommen der Immuntherapie hat zu einem revolutionären Konzept der Krebsbehandlung geführt, das auf der Aktivierung des endogenen Immunsystems basiert 3,4.
Die Tumormikroumgebung (TME) ist hochkomplex und besteht aus verschiedenen Immunzellen, krebsassoziierten Fibroblasten, Perizyten, Endothelzellen und verschiedenen geweberesidenten Zellen5. In der Vergangenheit wurden verschiedene Techniken zur Untersuchung der TME angewendet, wie z. B. die Durchflusszytometrie und die Einzelzellsequenzierung, die den räumlichen Kontext beeinträchtigen, da sie zur Zerstörung des Tumorgewebes erforderlich sind. Traditionelle mikroskopische Bildgebung wie Immunfluoreszenz (IF) und Immunhistochemie (IHC) ermöglichen die Visualisierung von Protein-Biomarkern, ohne das Probengewebe zu zerstören. Diese Ansätze sind jedoch auf zwei oder drei Biomarker beschränkt und können kein vollständiges Verständnis der räumlichen und strukturellen Zusammenhänge innerhalb des Komplexes TME 6 liefern.
Um dieses Problem zu lösen, wurden mehrere Multiplex-Bildgebungsverfahren entwickelt, um die komplexe TME räumlich sichtbar zu machen 7,8,9,10. Eines davon ist CoDetection-by-inDEXing, umbenannt in PhenoCycler-System, das auf DNA-Oligonukleotid-konjugierten Antikörpernbasiert 11. Das System kann die Einzelzellbildgebung und die Analyse von über 100 Biomarkern für menschliche Proben ermöglichen. Es stehen jedoch nur sehr wenige inventarisierte Antikörper zur Verfügung, um murine Proben zu visualisieren und zu analysieren, insbesondere Formalin-fixierte Paraffin-eingebettete (FFPE) Proben12. FFPE bietet mehrere Vorteile gegenüber der Konservierung von Fresh Frozen (FF), wie z. B. eine einfache Handhabung und Lagerung, eine gut erhaltene Morphologie im Laufe der Zeit und vor allem die Möglichkeit, Gewebe-/Tumor-Microarrays (TMA) herzustellen, die die Visualisierung mehrerer Proben auf einem einzigen Objektträger ermöglichen. Wir haben kürzlich ein murines FFPE CODEX/PhenoCycler-Antikörperpanel entworfen und entwickelt und es erfolgreich eingesetzt, um die räumliche Proteomik von genetisch reprogrammierten murinen Melanomproben zu visualisieren und zu analysieren13.
Das übergeordnete Ziel dieses Protokolls ist es, eine Schritt-für-Schritt-Anleitung für das Design eines murinen FFPE-Antikörper-Panels bereitzustellen und den Prozess der Antikörper-Barcode-Konjugation, der Gewebefärbung und der Bildgebung zu beschreiben. Darüber hinaus stellen wir eine detaillierte Bildanalyse-Pipeline vor, die Open-Source-Tools wie QuPath und R-Pakete verwendet. Nach dem Befolgen dieses Protokolls lernen die Forscher, wie sie ein individuell konjugiertes Antikörperpanel entwerfen, Multiplex-Bildgebung mit einem Phenocycler-Fusion-Gerät durchführen und neue Einblicke in die räumliche Proteomik des Melanom-TME gewinnen. Darüber hinaus kann dieses Protokoll angepasst werden, um verschiedene Mikroumgebungen des Tumorimmungewebes zu untersuchen und mit bestehenden räumlichen Transkriptomik-Techniken kombiniert zu werden.
Protokoll
Alle Tierarbeiten wurden in Übereinstimmung mit den Richtlinien des Johns Hopkins Animal Care and Use Committee unter Verwendung der genehmigten Protokollnummern MO18M388 und MO21M384 durchgeführt.
1. Auswahl der Antikörper
- Entwerfen Sie ein Antikörper-Panel basierend auf dem interessierenden Gewebe und der Häufigkeit des jeweiligen Biomarkers. Wählen Sie Antikörperklone aus, die auf erfolgreichen früheren Anwendungen der Immunfluoreszenz (IF) und Immunhistochemie (IHC) in der Literatur basieren. Sichern Sie sich trägerfreie Versionen dieser Antikörper.
HINWEIS: Ein gereinigter, trägerfreier Antikörper ist wichtig für die Barcode-Konjugation. Vermeiden Sie proteinbasierte Konservierungsstoffe wie BSA, Gluten, Glycerin usw. Wenn der trägerfreie Antikörper nicht verfügbar ist, fordern Sie einen kundenspezifischen Antikörper beim Lieferanten an oder verwenden Sie ein BSA-Entfernungskit, um den trägerfreien Antikörper herzustellen. Natriumazid beeinträchtigt die Antikörperkonjugation nicht. IgG-Isotypen werden gegenüber IgM-Isotypen empfohlen. - Verifizieren Sie diese ausgewählten Klone auf dem interessierenden Gewebe mit konventioneller Immunfluoreszenz-Bildgebung (IF)11. Verwenden Sie die doppelte ZF-Bildgebung, um die Spezifität bestimmter Klone zu bestätigen (z. B. führen Sie eine doppelte ZF-Bildgebung mit FOXP3 und CD4 durch, um die Färbung für regulatorische T-Zellen zu bestätigen).
HINWEIS: Wann immer möglich, ist es wichtig, für die IF-Validierung dasselbe Gewebe zu verwenden, das auch für die Färbung verwendet wird. Wählen Sie die geeignete Antigen-Retrieval-Lösung. In diesem Protokoll verwendeten wir einen von Akoya Biosciences gelieferten AR9-Puffer, um alle FFPE-Gewebeepitope des murinen Melanoms zu gewinnen. Andere Antigen-Retrieval-Lösungen, wie z. B. AR6 oder Universal, können je nach Epitopanforderungen verwendet werden.
2. Antikörperkonjugation und -bestätigung
- Ordnen Sie verifizierte Antikörper Barcodes zu, an die komplementäre Reporter an ATTO550 (Cy3), AF647 (Cy5) oder AF750 (Cy7) Fluorophoren angehängt sind. Antigene mit geringer Häufigkeit erzeugen in der Regel geringere Signale; konjugieren Sie solche Antikörper an Kanäle mit niedriger Autofluoreszenz, wie z. B. AF647 (Cy5). Konjugieren Sie hochexprimierte Antigene an ATTO550 (Cy3) und AF750 (Cy7) aufgrund der Möglichkeit der Autofluoreszenz.
HINWEIS: Dieser kritische Schritt berücksichtigt die Kanalempfindlichkeit und die Antigenhäufigkeit. Die Verwendung des AF488-Kanals für FFPE-Gewebe wird aufgrund seiner höheren Autofluoreszenz nicht empfohlen. - Für die Antikörper-Barcode-Konjugation (4,5 h) besorgen Sie sich das Antikörper-Konjugationskit. Lagern Sie alle Reagenzien bei 4 °C, mit Ausnahme von Reduktionslösung 1, die bei -20 °C als kleine Durchstechflaschen zum Einmalgebrauch gelagert werden muss.
- Messen Sie die Antikörperkonzentration mit einem Spektralphotometer.
- Beginnen Sie die Antikörperkonjugation, indem Sie 500 μl der Filterblocklösung auf eine 50 kDa MWCO-Filtersäule aufbringen, um die unspezifische Bindung von Antikörpern an den Filter zu blockieren. Bei 12.000 x g für 2 min bei Raumtemperatur (RT) herunterschleudern. Entfernen Sie überschüssige Flüssigkeit mit einer 200-μl-Mikropipette aus der Säule.
HINWEIS: Bei Reduktionslösung 1 handelt es sich um eine Durchstechflasche mit Einweglösung, die für drei Antikörperkonjugationen gleichzeitig ausreicht. Verwenden Sie die Lösung nach dem Auftauen nicht wieder. Verwerfen Sie alle verbleibenden Reagenzien. Verwenden Sie keinen Filter über der 50 kDa MWCO-Filtersäule, da dies zu einer schlechten Reinigung und Konjugation führen kann. - Verwenden Sie ein äquivalentes Volumen von 50 μg der Antikörperlösung und geben Sie es in einen 50 kDa MWCO-Filter. Stellen Sie das Gesamtvolumen bei Bedarf mit PBS auf 100 μl ein. Bei 12.000 x g 8 min bei 4 °C runterschleudern. Während dieser Zeit bereiten Sie den Reduktions-Mastermix mit 19,8 μl Reduktionslösung 1 + 825 μl Reduktionslösung 2 für drei Antikörperkonjugationen vor.
- Verwerfen Sie den Durchfluss und geben Sie 260 μl Reduktions-Mastermix auf die Oberseite jeder Filtereinheit. Die Lösung 2-3 Sekunden in der Filtereinheit vortexen und 30 min bei RT inkubieren.
HINWEIS: Es ist wichtig, die Inkubationszeit von 30 Minuten nicht zu überschreiten, um eine Überreduktion der Antikörper zu vermeiden, da dies diese schädigt und zu einer fehlgeschlagenen Konjugation führen kann. - Nach 30 Minuten Inkubation bei 12.000 x g für 8 Minuten bei 4 °C herunterschleudern und den Durchfluss am Boden entsorgen. Geben Sie 450 μl Konjugationslösung auf die Oberseite der Säule und schleudern Sie sie 8 Minuten lang bei 4 °C bei 12.000 x g . Bereiten Sie während der Zentrifugation die CODEX-Barcode-Lösung vor. Bewegen Sie sich schnell, nachdem Sie den Barcode aus -20 °C abgerufen haben, da sich der Barcode zu verschlechtern beginnt.
HINWEIS: Barcodes werden in der Regel in kleinen Glasfläschchen geliefert, die lyophilisierte Produkte (Flocken oder Pulver) enthalten, die bei -20 °C gelagert werden. Diese Fläschchen können verwendet werden, um 50 μg Antikörper auf einmal zu konjugieren. Es wird empfohlen, nicht mehr als drei Antikörper gleichzeitig zu konjugieren. - Platzieren Sie den lyophilisierten Barcode vorsichtig am Boden des Glasfläschchens (er kann auch an den Wänden kleben). Klopfen Sie das Glasfläschchen auf einen Tisch, um die Feststoffe nach unten zu bringen. Die Lokalisierung des lyophilisierten Produkts ist wichtig, wenn es in 10 μl nukleasefreiem Wasser in molekularbiologischer Qualität gelöst wird. Nachdem Sie 10 μl nukleasefreies Wasser hinzugefügt haben, fügen Sie jedem Barcode 210 μl Konjugationslösung hinzu. Lösen Sie das gesamte lyophilisierte Material auf und mischen Sie es vorsichtig durch Auf- und Abpipettieren. Verwerfen.
- Nachdem Sie das Schleudern in Schritt 2.2.5 abgeschlossen haben, verwerfen Sie den Durchfluss und fügen Sie die entsprechende Barcode-Lösung aus Schritt 2.2.6 oben auf jeder Filtereinheit hinzu. Bewahren Sie 1 μg des ursprünglichen unkonjugierten Antikörpers auf; Diese wird zusammen mit der konjugierten 5-μl-Antikörperprobe während der Validierung der Gelelektrophorese durchgeführt.
- Beschriften Sie jedes Röhrchen mit dem jeweiligen Antikörper- und Barcode-Namen. Schließen Sie den Deckel und mischen Sie die Lösung, indem Sie sie 2-3 s lang vortexen. Inkubieren Sie die Antikörper-Barcode-Konjugationsreaktion für 2 Stunden bei RT.
- Nach 2-stündiger Inkubation werden 5 μl des konjugierten Antikörpers entnommen und in einem 0,2-ml-PCR-Röhrchen gelagert, um die Konjugation durch Gelelektrophorese zu bestätigen.
- Die restliche konjugierte Antikörperlösung bei 12.000 x g für 8 min bei 4 °C herunterschleudern. Geben Sie 450 μl Reinigungslösung auf die Oberseite der Säule, drehen Sie sie 8 Minuten lang bei 4 °C bei 12.000 x g und verwerfen Sie den Durchfluss. Wiederholen Sie diesen Reinigungsschritt 2x, indem Sie jedes Mal 450 μl Reinigungslösung hinzufügen und herunterschleudern.
- Nach der 3. Zentrifugation den Durchfluss verwerfen. Der obere Filter enthält die Barcode-konjugierten Antikörper.
- Um den konjugierten Antikörper zu entnehmen, beschriften Sie das neue äußere Röhrchen, in dem sich die Filtersäule befindet, mit dem entsprechenden Antikörper und Barcode. Schneiden Sie vor dem Zentrifugieren den Deckel vom Außenröhrchen ab. Geben Sie 100 μl Antikörperspeicherlösung in jede Filtereinheit und legen Sie das neu markierte Außenröhrchen kopfüber auf die Filtersäule.
- Drehen Sie die Filtersäule um, um den konjugierten Antikörper in das äußere Röhrchen zu sammeln, und schleudern Sie bei RT 2 Minuten lang bei 3.000 x g nach unten. Dadurch sollten etwa 120 μl konjugierte Antikörperlösung gesammelt werden. In ein steriles Mikrozentrifugenröhrchen umfüllen und 18-24 Monate bei 4 °C lagern.
HINWEIS: Um eine nukleare Färbung mit hohem Hintergrund nach der Konjugation zu vermeiden, wird empfohlen, diese Antikörper mindestens 2 Tage lang nicht zu verwenden.
- Führen Sie die Konjugationsbestätigung durch Gelelektrophorese durch, wie im bereits veröffentlichten Protokoll13 beschrieben.
HINWEIS: Dieser Prozess bestätigt nur den Erfolg der chemischen Konjugationsreaktion, die zur Konjugation der Barcodes mit Antikörpern verwendet wird. Sie ist optional, da eine Antikörpervalidierung erst nach erfolgreicher Färbung und Bildgebung des Gewebes möglich ist.
3. Vorbereitung der FFPE-Proben aus Murine
- Gewinnung von FFPE-Gewebeproben von weiblichen C57BL/6J-Mäusen, denen B16F10-Flankentumoren implantiert wurden. Behandlung von Mäusen mit intratumoralen Injektionen von Nanopartikeln auf Poly(β-Aminoester)-Basis, die 4-1BBL- und IL-12-Plasmide enthalten, zusammen mit intraperitonealer Verabreichung von Anti-PD1 9, 11, 16 und 18 Tage nach der Implantation14. Opfern Sie Mäuse am Tag 20 und fixieren Sie Tumore in 10% Formalin, eingebettet in Paraffin, dann schneiden Sie im Kern der Johns Hopkins Oncology Tissue Services.
- Berücksichtigen Sie sorgfältig die in der Bedienungsanleitung angegebenen Spezifikationen für den Bildgebungsbereich und montieren Sie 5-μm-Proben auf einem Objektträger innerhalb des Bildgebungsbereichs. Wir konnten vier verschiedene Tumorproben auf einem einzigen Objektträger montieren.
HINWEIS: Es ist wichtig, die empfohlenen Objektträger zu verwenden, die für die Bildgebung verfügbar sind, wie z. B. Leica Apex Klebedias oder Fisherbrand Superfrost Plus Objektträger. Dies hilft der Durchflusszelle, fest auf dem Objektträger zu haften.
4. FFPE-Gewebefärbung und Bildgebung
- Verifizierung und Optimierung der Antikörperkonzentration und der Expositionszeiten: Um den Erfolg der Antikörper-Barcode-Konjugation zu überprüfen, färben Sie das gewünschte Gewebe mit 6-9 konjugierten Antikörpern. Führen Sie das Multiplex-Bildgebungsgerät aus und notieren Sie sich jede Antikörperkonzentration und Expositionszeit. Passen Sie die Verdünnungen und Expositionszeiten nach Bedarf an, führen Sie dann 6-9 neue Antikörper mit den zuvor verifizierten Antikörpern durch und notieren Sie alle Anpassungen. Führen Sie die folgenden Schritte für die Färbung und Bildgebung aus.
- Gewebefärbung und Bildgebung
- Am Tag vor der Färbung die FFPE-Objektträger über Nacht im 60 °C heißen Ofen backen. Kühlen Sie die Objektträger am nächsten Tag 10 Minuten lang bei RT ab, bevor Sie mit den Schritten zur Entparaffinisierung und Geweberehydrierung beginnen.
- Beginnen Sie mit der Entparaffinisierung des Gewebes, indem Sie 2x für jeweils 5 min in einer Xylollösung inkubieren.
- Um das Gewebe zu rehydrieren, bewegen Sie es durch zwei Runden mit 100%iger Ethanollösung für jeweils 5 Minuten. Bewegen Sie es anschließend durch eine Reihe von Alkohollösungen von 90 % auf 70 %, 50 % und 30 % für jeweils 5 Minuten. Zum Schluss das Tuch zweimal für jeweils 5 min mit destilliertem Wasser waschen.
HINWEIS: Weniger toxische Alternativen zu Xylol, wie z. B. eine Neoclear-Lösung, können für Entparaffinisierungsschritte verwendet werden. Entparaffinisierung und Rehydrierung unter einem Abzug durchführen. - 10x AR9-Puffer mit destilliertem Wasser auf 1x verdünnen. Füllen Sie ein Coplin-Glas mit 50 mL 1x AR9-Puffer und stellen Sie sicher, dass die Objektträger vollständig in den Antigen-Retrieval-Puffer eingetaucht sind.
- Füllen Sie einen Schnellkochtopf mit Wasser, so dass sich der Wasserstand auf halber Höhe des Coplin-Glases befindet, und inkubieren Sie dann 20 Minuten lang unter hohem Druck.
HINWEIS: Es ist möglich, einen speziellen Schnellkochtopf oder eine Enttarnungskammer für eine bessere Antigenrückgewinnung zu verwenden, indem man ihn 10 Minuten lang auf 120 °C einstellt. - Nachdem Sie den Schnellkochtopfschritt abgeschlossen haben, kühlen Sie die Objektträger 45-60 Minuten lang ab. Spülen Sie den Objektträger 2x für jeweils 2 min mit destilliertem Wasser ab. Übertragen Sie dann den Objektträger in das Glas mit 1x PBS.
- Um die Autofluoreszenz zu minimieren, führen Sie vor dem Färben mit Antikörpern einen Bleichschritt auf dem Gewebe durch. Bereiten Sie eine Bleichlösung (25 mL 1x PBS + 0,8 mL 1M NaOH + 4,5 mL H2O2) unter Verwendung der empfohlenen Reagenzien und Konzentrationen frisch vor. Inkubieren Sie die Objektträger in einem Kunststoffbehälter mit der Bleichlösung zwischen zwei LED-Lampen für 45 Minuten bei Raumtemperatur. Wiederholen Sie den Vorgang mit frischer Bleichlösung für weitere 45 Minuten.
- Beginnen Sie während dieses Bleichschritts mit der Zubereitung der Antikörper-Cocktail-Lösung, insbesondere wenn sich mehrere Antikörper mit unterschiedlichen Verdünnungen im Panel befinden. Holen Sie alle vier Blocker (G, S, J und N) heraus und legen Sie sie auf Eis, da einige Zeit brauchen, um ab -20 °C aufzutauen.
- Waschen Sie das Tuch nach dem Photobleaching mit 1x PBS, 2x für jeweils 2 min. Füllen Sie das Tuch in Gläser mit Hydratationspuffer und waschen Sie es 2x für jeweils 2 Minuten.
- Übertragen Sie die Gewebeobjektträger in den Färbepuffer und lassen Sie sie 30 Minuten lang äquilibrieren. Wenn die Antikörper-Cocktail-Lösung während des Bleichschritts nicht vorbereitet wurde, bereiten Sie während dieser Zeit die Antikörper-Cocktail-Lösung vor.
- Bereiten Sie 300 μl Antikörper-Cocktail-Lösung vor, um zwei Objektträger zu färben (normalerweise reichen bei Verwendung einer Hybridisierungs-Kunststoffkammer 100-120 μ0 μl Antikörper-Cocktail aus, um jeden Objektträger zu färben).
- Färben Sie die Objektträger, indem Sie eine Antikörper-Cocktail-Lösung über Nacht bei 4 °C in die Feuchtigkeitskammer auftragen.
HINWEIS: Alle Antikörper hier zeigen ein positives Signal, wenn sie über Nacht bei 4 °C gefärbt werden. Die Färbezeit und -temperatur jedes Antikörpers kann jedoch variieren und muss optimiert werden. Die am häufigsten verwendeten Färbebedingungen sind 3 h bei RT oder über Nacht bei 4 °C. Bei Bedarf ist es auch möglich, einige Antikörper zunächst bei unterschiedlichen Temperaturen und dann die restlichen Antikörper über Nacht bei 4 °C zu färben. Waschen Sie das Tuch anschließend mit PBS, wenn eine sequentielle Färbung erforderlich ist. - Führen Sie am nächsten Tag eine Fixierung nach der Färbung durch. Entfernen Sie die Kunststoffkammer und lagern Sie den gesammelten Antikörpercocktail bei 4 °C. Waschen Sie die Objektträger 2x im Färbepuffer für jeweils 2 min. Geben Sie die Objektträger anschließend in eine 40 mL Fixierlösung nach der Färbung (4 mL 16 % Paraformaldehyd + 36 mL Lagerpuffer) und inkubieren Sie sie 10 Minuten lang. 3x für jeweils 2 min mit PBS spülen.
- Schieben Sie die Objektträger für 5 min in eiskaltes Methanol, spülen Sie dann mit PBS 3x für jeweils 2 min. Tragen Sie eine endgültige Fixierlösung (20 μl ein Röhrchen + 1 ml PBS) auf die Objektträger unter einer Feuchtigkeitskammer auf. In der Regel reichen 200 μl der Fixierlösung pro Objektträger aus. Lassen Sie das Fixiermittel 20 Minuten bei RT einwirken. Führen Sie drei letzte Wäschen in PBS für jeweils 2 Minuten durch.
HINWEIS: Entfernen Sie den Fixierschlauch nicht vorher bei -20 °C; Tauen Sie nur vor dem Auftragen auf. - Wenn Sie sofort eine Bildgebung durchführen, wischen Sie den Objektträger mit einem fusselfreien Gewebe um das Gewebe herum und setzen Sie die Flusszelle auf, indem Sie sie 30 s lang mit einem Durchflusszellen-Montagegerät drücken. Wenn Sie später ein Bild machen, lagern Sie den Objektträger (ohne Anwendung der Durchflusszelle) im Lagerpuffer bei 4 °C. Wenn die Objektträger bereit sind, übertragen Sie sie für 10 Minuten aus dem Speicherpuffer auf PBS, bevor Sie die Durchflusszelle einsetzen.
- Beginnen Sie mit der Vorbereitung des Laufpuffers 1x mit Additiv basierend auf den gewünschten Zyklen im Experiment. Bereiten Sie niedrige (1:4) und hohe DMSO-Puffer (9:1) vor, indem Sie als 1-teiliges DMSO mit 4-teiligem Laufpuffer und 9-teiligem DMSO mit 1-teiligem Laufpuffer mischen, der für die gewünschten Zyklen erforderlich ist. Sparen Sie ca. 20 mL 1x Puffer mit Additiv für die Herstellung einer Reporterplatte. Um den erforderlichen 1x-Puffer mit additiven und hohen/niedrigen DMSO-Puffern zu berechnen, wenden Sie sich an den Gerätemanager.
- Beginnen Sie mit der Vorbereitung der Reporterplatte. Stellen Sie zunächst fest, dass die Reporter-Stammlösung für die Gesamtzahl der Zyklen erforderlich ist. Beschriften Sie schwarze oder bernsteinfarbene 1-ml-Mikrozentrifugenröhrchen mit Zyklusnamen. Geben Sie 250 μl Reporterstammlösung und 5 μl jedes Reporters in das entsprechende Röhrchen. Gib dann die Lösung auf eine schwarze 96-Well-Platte und versiegele sie mit Klebefolie.
- Starten Sie das Gerät, stellen Sie mit dem Gerätemanager die Parameter und Belichtungszeiten ein (gemäß Tabelle 1) und erfassen Sie die Bilder. Abbildung 1 fasst die Vor- und Nachfärbungsprozesse zusammen.
5. Gewebeannotation und Zellsegmentierung
- Richten Sie ein Projekt und einen Arbeitsbereich ein.
- Installieren Sie die neueste Version der Software zur Analyse der digitalen Pathologie (z. B. QuPath Version 0.5.1 oder höher). Klicken Sie auf Projekt erstellen , um einen Zielordner für den Projektraum auszuwählen.
- Klicken Sie auf Bilder hinzufügen > Dateien auswählen, und navigieren Sie dann zur QPTIFF-Datei (erstellt aus Multiplex-Immunfluoreszenz-Bildgebung). Stellen Sie den Bildtyp auf Fluoreszenz ein, behalten Sie alle anderen Standardeinstellungen bei und klicken Sie auf Importieren.
- Wenn Sie QuPath verwenden, doppelklicken Sie auf das neue Bild, um einen Arbeitsbereich zu öffnen. Klicken Sie regelmäßig auf Datei > Speichern , um die am Projekt vorgenommenen Änderungen zu verfolgen. Schalten Sie die Sichtbarkeit der Markierung und die Anzeigeeinstellungen mit dem Symbol für Helligkeit und Kontrast (Halbmondsymbol) in der Mitte der Symbolleiste um.
HINWEIS: Klicken Sie mit der rechten Maustaste auf ein Bild in der Bildliste, um es umzubenennen. Dies wird später bei der Visualisierung von Phänotyp-Klassifikationen wichtig sein.
- Annotation des gesamten Gewebeschnitts, des intratumoralen Kompartiments und des stromalen Kompartiments.
- Verwenden Sie den Pinsel und/oder den Zauberstab, um eine Anmerkung um den gesamten Gewebeabschnitt zu zeichnen. Definieren Sie diese Anmerkung als Full_Tissue. Schließen Sie die darüber liegende Haut und/oder alle Regionen aus, die nicht analysiert werden sollen. Um eine negative Anmerkung zu erstellen und/oder die Anmerkungsgrenze zu verkleinern, halten Sie die Alt-Taste gedrückt, während Sie eines der Anmerkungswerkzeuge verwenden.
- Duplizieren Sie die vollständige Gewebeanmerkung. Wählen Sie die Anmerkung auf der Registerkarte Anmerkungen aus, und gehen Sie dann zu Objekte > Anmerkungen... > Ausgewählte Anmerkungen duplizieren.
- Schalten Sie den SOX10-Kanal ein. Verkleinern Sie in der duplizierten Annotation die Annotationsgrenze, um das intratumorale Kompartiment mit dem Alt + Pinsel-/Zauberstab-Werkzeug zu erfassen. Definieren Sie diese Annotation als Tumor (Abbildung 2).
- Wählen Sie die vollständige Gewebeanmerkung aus und gehen Sie zu Objekte > Anmerkungen... > Anmerkungen erweitern. Definieren Sie den Erweiterungsradius auf 1 μm und klicken Sie auf Ausführen.
- Benennen Sie die neue Anmerkung in Full_Tissue_Expansion um. Wählen Sie die Tumoranmerkung aus, klicken Sie mit der rechten Maustaste, und wählen Sie In Hierarchie einfügen aus.
- Wählen Sie die Tumor-Anmerkung erneut aus, gehen Sie zu Objekte > Anmerkungen... > Umkehrung machen. Definieren Sie diese neue Annotation als Stroma. Wiederholen Sie dies für alle Gewebeabschnitte.
- Führen Sie die Zellsegmentierung durch und exportieren Sie die Ergebnisse.
- Wechseln Sie zur Registerkarte Bild , um die Pixelbreite und -höhe des Bildes zu ermitteln (Abbildung 3A).
- Laden Sie die StarDist-Erweiterung bei https://github.com/qupath/qupath-extension-stardist/releases herunter. Ziehen Sie die Datei qupath-extension-stardist-[version].jar in das QuPath-Fenster und klicken Sie dann auf das Zahnradsymbol in der oberen rechten Ecke des QuPath-Fensters. Definieren Sie unter Erweiterungen das QuPath-Benutzerverzeichnis als Speicherort des Ordners StarDist.
- Laden Sie die StarDist-Groovy-Skriptdateien und die StarDist-Modelldatei von https://github.com/xzhou125/JOVE_QuPath_Spatial_Analysis herunter (StarDist-Skripte wurden ursprünglich von Akoya Biosciences organisiert).
- Wählen Sie für jeden Gewebeabschnitt sowohl die Tumor- als auch die Stroma-Annotation aus (verwenden Sie Strg, um mehrere Annotationen auszuwählen). Klicken Sie in der oberen Einstellungsleiste auf Automatisieren > Skripteditor , um die Skriptoberfläche zu öffnen. Öffnen Sie das entsprechende StarDist-Zellensegmentierungsskript (Groovy-Datei), das der Pixelgröße des Bildes entspricht. Wenn Sie aufgefordert werden, die Zellensegmentierungsdatei auszuwählen, öffnen Sie die Datei stardist_cell_seg_model.pb.
- Speichern Sie das Bild nach der Zellensegmentierung. Gehen Sie zu Messen > Messungen exportieren und wählen Sie dann die entsprechenden Bilder aus. Weisen Sie dem Exporttyp "Zellen" und dem Trennzeichen ".csv" zu und wählen Sie dann den Speicherort der Ausgabedatei aus. Klicken Sie auf Füllen neben Spalten, um die gewünschten Metriken einzuschließen. Wichtige Spalten, die exportiert werden sollen, sind "Image", "Object ID", "Parent", "Centroid X", "Centroid Y" und "Nucleus Area".
HINWEIS: Die Objekt-ID wird in nachfolgenden Schritten verwendet, um die Klassifizierungen wieder dem Multiplex-Immunfluoreszenzbild in der digitalen Pathologie-Analysesoftware zuzuordnen. Die Spalte Übergeordnet gibt den Namen der Anmerkung an, zu der die Zelle gehört. X- und Y-Daten werden für die räumliche Analyse verwendet. - Wählen Sie für jeden Abstammungsmarker, der bei der Clusterbildung/Phänotypisierung verwendet werden soll, einen Mittelwert aus, der exportiert werden soll. Für nukleare Marker (z. B. SOX10 und FOXP3) exportieren Sie die Option Nucleus: Mean. Für zytoplasmatische/membranöse Marker (z. B. CD45, CD3, CD4) exportieren Sie die Option Zytoplasma: Mittelwert.
6. Vorverarbeitung und Normalisierung von Proteomik-Daten
- Öffnen Sie die exportierte CSV-Datei. Kürzen Sie für jeden Marker, der beim Clustering/Phänotypisieren verwendet werden soll, den Namen der Spaltenüberschrift so, dass nur der Marker enthalten ist. Benennen Sie z. B. SOX10: Nucleus: Mean in SOX10 und CD3: Cytoplasm: Mean in CD3 um.
- Installieren Sie die neueste Version von R und RStudio. Laden Sie die Datei Marker Normalization.R von https://github.com/xzhou125/JOVE_QuPath_Spatial_Analysis herunter, und führen Sie das Skript aus. Dabei werden zunächst Zellen nach Kerngröße herausgefiltert.
- Für jeden Marker wird dann eine Min-Max-Normalisierung durchgeführt, sodass der niedrigste MFI-Wert im Ausdrucksbereich auf 0 und der MFI-Wert am 99,7. Perzentil auf 1 gesetzt wird. Alle Intensitäten oberhalb des 99,7. Perzentils werden bei 1 abgeschnitten. Speichern Sie nach Abschluss des Vorgangs die neue CSV-Datei, die mit den normalisierten Daten generiert wurde.
7. Clustering und Phänotypisierung
- Wählen Sie einen Algorithmus für die Gruppierung von Zellen gemäß Markerausdrucksprofilen aus. Auf https://github.com/xzhou125/JOVE_QuPath_Spatial_Analysis werden R-Skripte für das Clustering mit Seurat v4.415 und FlowSOM v2.13.9 (Clustering- und Visualisierungstool)16 bereitgestellt.
- Phänotypisierung der Zellen gemäß den Markerexpressionsprofilen der wichtigsten Abstammungsmarker. Das hier vorgestellte Protokoll verwendete 14 Linienmarker für das Clustering und die Phänotypisierung: SOX10, CD45, CD3, CD4, CD8, FOXP3, CD11C, F4/80, CD68, CD86, CD163, CD206, NK1.1 und CD31.
- Definieren Sie Tumorzellen als SOX10hi. Definieren SieCD4-T-Zellen als CD45 mod-hi/ CD4hi/CD8low/FOXP3low. Definieren Sie CD8 T-Zellen als CD45mod-hi/ CD8hi/CD4low. Definieren Sie Tregs als CD45mod-hi/CD4hi/CD8low/FOXP3hi. Definieren Sie andereT-Zellen als CD45 mod-hi/CD3hi/CD4low/CD8low.
- Definieren Sie M1-Makrophagen als CD45mod-hi/F4/80hi/CD68hi/CD86hi/CD163niedrig /CD206niedrig. Definieren Sie M2-Makrophagen als CD45mod-hi/F4/80hi/CD68mod-hi/CD86low/CD163hi/CD206low, CD45mod-hi/F4/80hi/CD68mod-hi/CD86low/CD163low/CD206hi, oder CD45mod-hi/F4/80hi/CD68mod-hi/CD86low/CD163hi/CD206hi. Definieren Sie andere Makrophagen als CD45mod-hi/F4/80hi/CD68mod-hi/CD86low/ CD163low/CD206low oder CD45mod-hi/F4/80hi/CD68mod-hi/CD86hi/CD163hi/CD206hi.
- Definieren Sie dendritische Zellen als CD45mod-hi/CD11chi/CD86mod-hi. Definieren Sie NK-Zellen als CD45mod-hi/NK1.1hi. Definieren Sie andere Immunzellen als CD45mod-hi , die die oben genannten Bedingungen nicht erfüllten. Definieren Sie Endothelzellen als CD31mod-hi. Entfernen Sie alle anderen Ausdrucksprofile.
8. Neuzuordnung von Phänotyp-Klassifikationen und Durchführung von Qualitätskontrollen
- Laden Sie das Skript Remapping Classifications.groovy von https://github.com/xzhou125/JOVE_QuPath_Spatial_Analysis herunter (dieses Skript wurde aus einem Beitrag auf https://forum.image.sc/ von petebankhead übernommen).
- Öffnen Sie die phänotypisierte CSV-Datei und stellen Sie sicher, dass alle Beschriftungen auf der Registerkarte Bild genau mit dem Namen der Bilddatei in der digitalen Pathologie-Analysesoftware übereinstimmen, die für die Neuzuordnung verwendet wird. Um große Mengen an Beschriftungen in der CSV-Datei schnell zu ändern, verwenden Sie die Funktion Alle ersetzen.
- Wenn Sie QuPath verwenden, führen Sie die Klassifizierungs-Neuzuordnung der Groovy-Datei aus, indem Sie auf Automatisieren > Skript-Editor klicken.
- Klicken Sie auf der Registerkarte Anmerkungen auf die drei Punkte neben der Schaltfläche Automatisch festlegen und klicken Sie dann auf Aus vorhandenen Objekten auffüllen > Alle Klassen (einschließlich Unterklassen) > Ja.
- Scrollen Sie in der Marker-Liste nach unten, um die verschiedenen Phänotyp-Klassifizierungen anzuzeigen und das Farbschema nach Bedarf anzupassen. Doppelklicken Sie auf eine beliebige Zelle, um ihren Phänotyp (neben dem Klassifikations-Tag) und den Cluster (neben dem Name-Tag) anzuzeigen. Überprüfen Sie repräsentative Sichtfelder, um die Genauigkeit der Phänotyp-Zuordnungen zu bestimmen und die Phänotypisierungsstrategie bei Bedarf zu überarbeiten (Abbildung 3B).
9. Dichtequantifizierung und räumliche Analyse
- Sobald die Phänotypisierung abgeschlossen ist, können Sie die Anmerkungsbereiche in der digitalen Pathologie-Analysesoftware auf der Registerkarte "Anmerkungen" abrufen. Konvertieren Sie Flächenwerte in mm2, und dividieren Sie dann die Anzahl durch Annotationsflächen, um die Dichte zu berechnen.
- Laden Sie das Skript SPIAT Spatial Analysis.R von https://github.com/xzhou125/JOVE_QuPath_Spatial_Analysis herunter, und führen Sie es aus. Sammeln Sie Daten zu durchschnittlichen Mindestabständen (AMD), Nachbarschaftskompositionen und normalisierten Mixing-Scores (NMS).
- Generieren Sie Zahlen in einer statistischen Analysesoftware, um die Ergebnisse zu präsentieren. Wenn Sie eine einzelne Heatmap für durchschnittliche Mindestabstände der Behandlungsbedingungen erstellen, mitteln Sie die Ergebnisse aus verschiedenen Gewebeschnitten.
Ergebnisse
Hier stellen wir ein Protokoll für das Design eines Antikörper-Panels für murines FFPE-Gewebe, die Durchführung von Multiplex-Immunfluoreszenz-Bildgebung und die Analyse von Bildern für die Proteomik-Quantifizierung und räumliche Beziehungen vor. Das validierte Panel enthält 27 Antikörper, die Marker für die Visualisierung von Melanomzellen (SOX10), Leukozyten (CD45), T-Zellen (CD3, CD4, CD8, FOXP3), B-Zellen (CD20), Makrophagen und Subtypen (F4/80, CD68, CD86, CD163, CD206), dendritischen Zellen (CD11c), NK-Zellen (NK1.1) und Endothelzellen (CD31) liefern. Das vollständige Panel enthält auch Marker für andere Immunpopulationen (CD11b, CD38), Proliferationsaktivität (Ki67), T-Zellfunktionalität (T-bet, Eomesodermin, Granzym B), Antigenpräsentation (LMP2, Beta-2-Mikroglobulin, MHC II) und Checkpoint-Expression (TIM3, LAG3, PD-L1)12. Ein repräsentatives Bild der Gelelektrophorese bestätigt die erfolgreiche Konjugation von DNA-Oligonukleotid-Barcodes an die trägerfreien Antikörper, wie in Abbildung 4 dargestellt. Dieser Schritt bestätigt nur die chemische Reaktion, und die Bildbestätigung kann erst erfolgen, nachdem diese Antikörper mit dem Multiplex-Bildgebungsgerät am interessierenden Gewebe überprüft wurden. In der folgenden Referenz finden Sie validierte Bilder aller 27 Marker in Panel13. Abbildung 5 zeigt ein Fusionsbild der wichtigsten Linienmarker, die drei murine Melanom-Gewebeschnitte färben, die mit intratumoralen 4-1BBL/IL-12-Nanopartikel-Injektionen und systemischem Anti-PD1 behandelt wurden. Diese Schnitte wurden für die anschließende Bildanalyse in diesem Protokoll verwendet.
Nach der Gewebeannotation, der Zellsegmentierung und der Vorverarbeitung der Proteomikdaten präsentieren wir einen Vergleich zwischen zwei Clustering-Algorithmen, die zuvor für die transkriptomische und/oder proteomische Einzelzellanalyse angewendet wurden17,18. Expressionsprofile für phänotypisierte Populationen sind in Abbildung 6 für beide Ansätze dargestellt. Wir zeigen, dass FlowSOM einen breiteren Bereich von Intensitätswerten (~0,7 vs. ~0,5) bietet, um Unterschiede zwischen ähnlichen Zellpopulationen, wie z.B. Makrophagen-Subtypen, zu erkennen. Seurat wandte den Louvain-Algorithmus während des Clusterings an und generierte 29 Cluster (Ergänzende Abbildung 1). Im Vergleich dazu verwendete FlowSOM selbstorganisierende Karten und kann 100 Cluster generieren (Ergänzende Abbildung 2). Eine größere Anzahl von Clustern führt zu mehr Zeit, die für die Phänotypisierung benötigt wird, aber dieser letztgenannte FlowSOM-Ansatz bietet auch mehr Nuancen bei der Klassifizierung ähnlicher Zellpopulationen. Qualitativ sehen wir, dass FlowSOM in der Lage war, mehr intratumorale Makrophagen entweder als M1- oder M2-Subtyp zu klassifizieren, verglichen mit der Seurat-Phänotypisierung im gleichen Sichtfeld (Abbildung 7). Das gleiche Ergebnis zeigt sich bei der Quantifizierung der Makrophagendichten, wobei FlowSOM im Vergleich zu Seurat eine höhere Dichte sowohl von M1- als auch von M2-Makrophagen erfasst (Abbildung 8A-B) und anschließend eine signifikant geringere Dichte anderer/nicht klassifizierter Makrophagen (p = 0,0028; Abbildung 8C). Nichtsdestotrotz lieferten die beiden Analyseansätze auch bei der Beschreibung anderer Zellpopulationsdichten wie CD4-T-Zellen, CD8-T-Zellen und Endothelzellen ähnliche Ergebnisse (Abbildung 8D-F).
Wir präsentieren auch die Ergebnisse der nachgelagerten räumlichen Analyse nach Clustering und Phänotypisierung. Abbildung 9 zeigt einige der räumlichen Metriken, die mit diesem Protokoll generiert werden können. Bezogen auf alle phänotypisierten Populationen wiesen M2-Makrophagen und NK-Zellen nach Behandlung mit 4-1BBL/IL-12-Nanopartikeln und Anti-PD1 die höchsten AMDs auf (Abbildung 9A). In ähnlicher Weise war die NMS zwischen intratumoralen CD8-T-Zellen und M1-Makrophagen viel höher als zwischen CD8-T-Zellen und M2-Makrophagen (Abbildung 9B). Darüber hinaus trugen M2-Makrophagen ~1% in der 100-μm-Nachbarschaft bei, die intratumorale CD8-T-Zellen umgibt, während M1-Makrophagen 9%-13% dieser Nachbarschaften ausmachten (Abbildung 9C). Zusammengenommen deuten diese Ergebnisse darauf hin, dass das 4-1BBL/IL-12-Behandlungsschema tumorassoziierte Makrophagen in Richtung eines M1-Subtyps polarisierte und M2-Makrophagen aus der immunen Mikroumgebung des Tumors ausschloss.
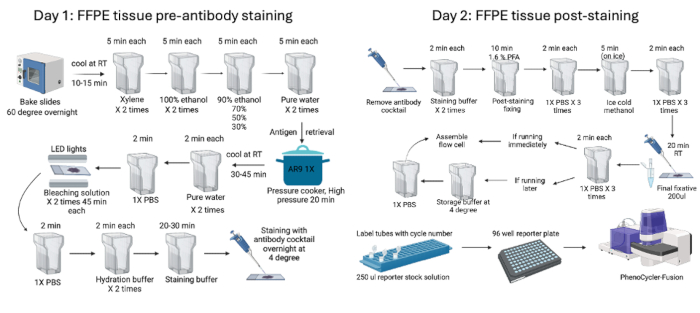
Abbildung 1: Zusammenfassung des Arbeitsablaufs für die FFPE-Gewebefärbung und -bildgebung. Maus-FFPE-Gewebe wurden mit Vorfärbeverfahren verarbeitet, die mit dem Einbrennen des Gewebes über Nacht begannen, bevor mit der zweitägigen Vorfärbung (Tag 1) und der Nachfärbung (Tag 2) begonnen wurde. Abschließend wurde die Reporterplatte vorbereitet, bevor mit dem Gerät abgebildet wurde. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
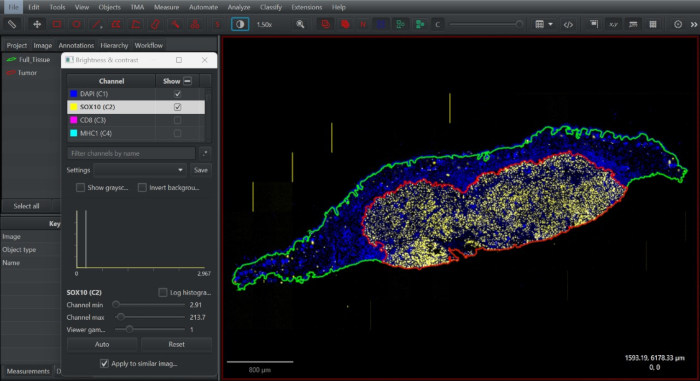
Abbildung 2: Screenshot der Gewebeannotation in der frei zugänglichen digitalen Pathologiesoftware QuPath. Die vollständige Gewebeannotation wurde gezeichnet (grün), dupliziert und minimiert, um das intratumorale Kompartiment entsprechend der Verteilung der SOX10+-Melanozyten an der Tumorgrenze (rot) zu definieren. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
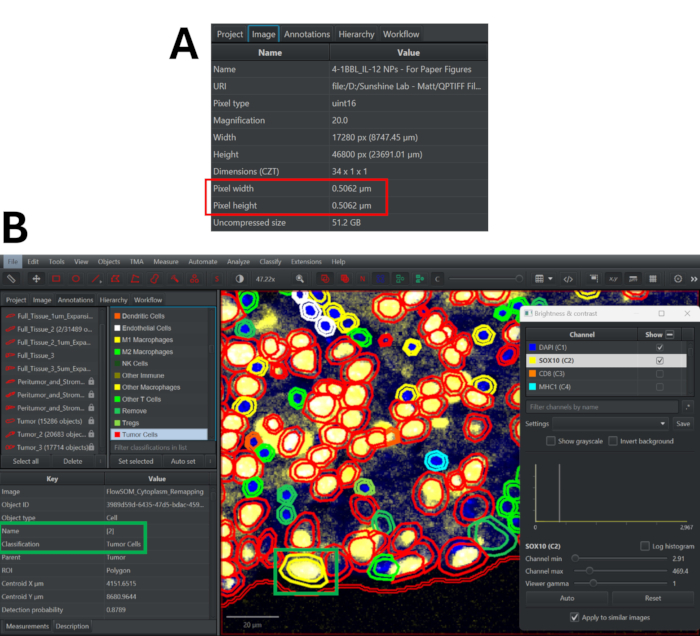
Abbildung 3: Zellsegmentierung mit dem StarDist-Algorithmus in QuPath und Qualitätskontrollprozess zur Überprüfung von Phänotyp-Klassifikationen. (A) Navigieren Sie zur Registerkarte Bild, um die Pixelbreite und -höhe des Multiplex-Immunfluoreszenzbildes zu bestimmen. (B) Doppelklicken Sie nach der Neuzuordnung der Klassifikationen auf eine beliebige Zelle (wird gelb hervorgehoben), um die Clusterzuordnung und den Phänotyp anzuzeigen. Schalten Sie die Panel-Markierungen ein/aus, um zu bestimmen, ob dieser Klassifizierungsansatz korrekt ist. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
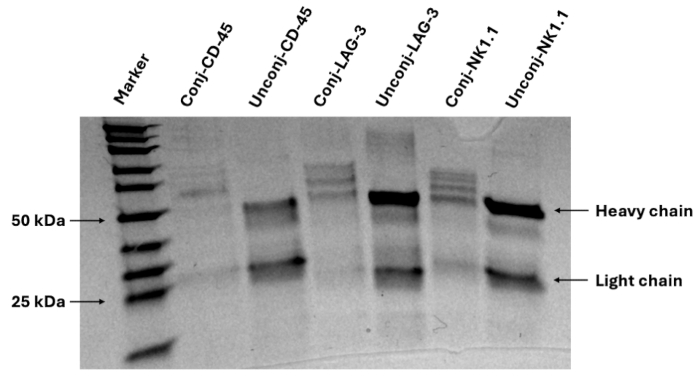
Abbildung 4: Repräsentatives Gelbild der Bestätigung der Antikörper-DNA-Barcode-Konjugation. Die Protein-Gel-Elektrophorese bestätigt die Antikörperkonjugation mit DNA-Oligonukleotid-Barcodes, die durch zusätzliche Banden an der Stelle der schweren Kette beobachtet werden. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
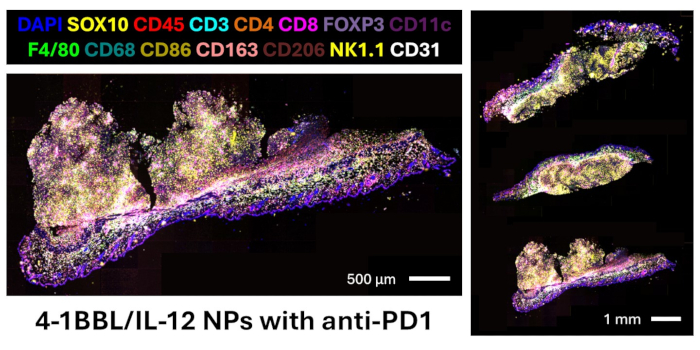
Abbildung 5: DNA-Barcode-basierte Multiplex-Bildgebung von B16F10-Flankentumoren, die mit intratumoralen Injektionen von 4-1BBL/IL-12-Nanopartikeln mit systemischem Anti-PD1 behandelt wurden. Marker in unserem Panel nicht gezeigt: CD11b, CD20, CD38, TIM3, LAG3, T-bet, Eomesodermin, Granzym B, Ki67, LMP2, Beta-2-Mikroglobulin, MHC II, PD-L1. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
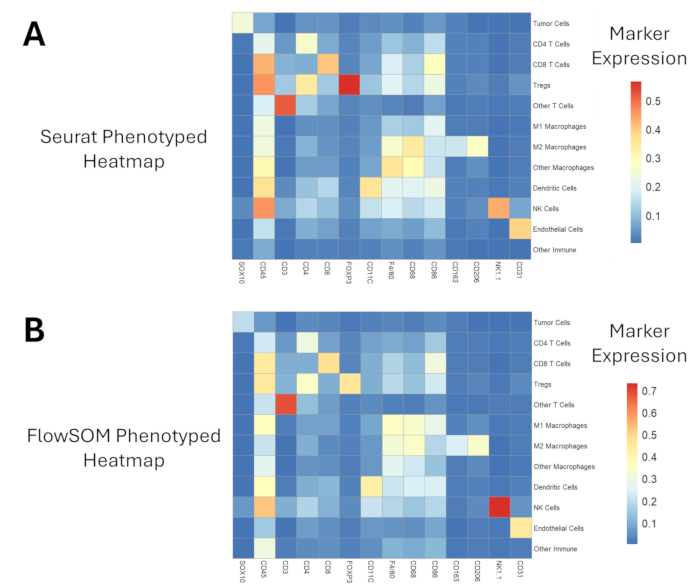
Abbildung 6: Proteomik-Expressions-Heatmaps von phänotypisierten Zellpopulationen. (A) Der Seurat-Clustering- und Phänotypisierungsansatz erzeugt einen etwas kleineren Bereich von Markerexpressionswerten im Vergleich zu (B) FlowSOM-Clustering und Phänotypisierung. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
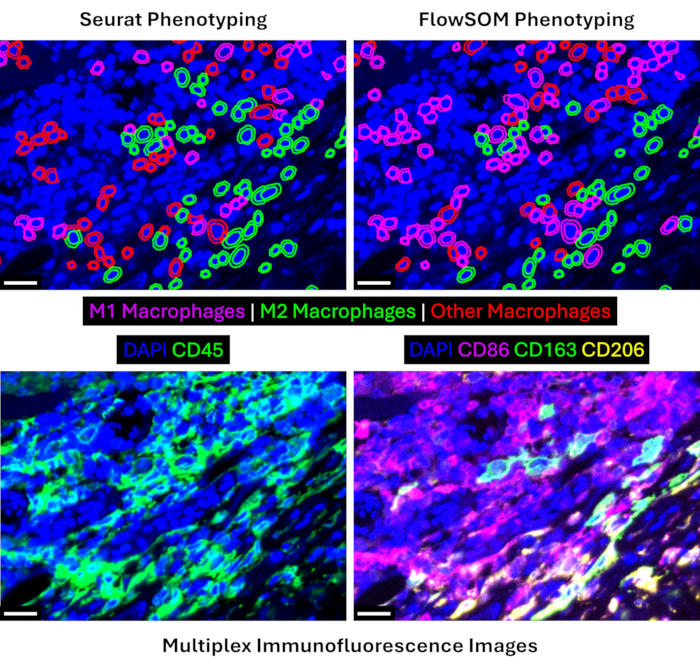
Abbildung 7: Die FlowSOM-Phänotypisierung identifiziert eine größere Bandbreite verschiedener Makrophagen-Subtypen. Der obere Bereich zeigt die Makrophagen-Phänotypisierung von Seurat (links) und FlowSOM (rechts) für dasselbe Sichtfeld. Der untere Bereich zeigt Multiplex-Immunfluoreszenzbilder von wichtigen Markern der Makrophagen-Abstammungslinie im selben Sichtfeld. Alle Maßstabsleisten sind 20 μm. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
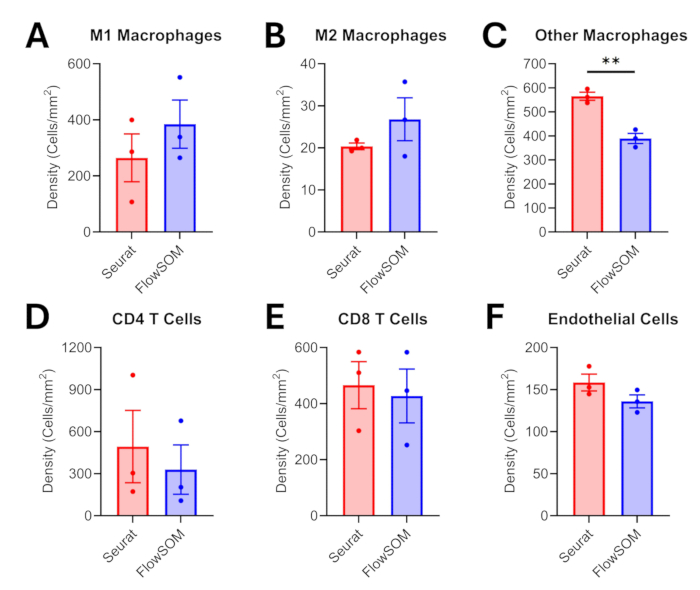
Abbildung 8: Vergleich der intratumoralen Dichten verschiedener Zellpopulationen nach Clustering/Phänotypisierung. Dichtevergleiche werden für intratumorale (A) M1-Makrophagen, (B) M2-Makrophagen, (C) andere Makrophagen, (D) CD4-T-Zellen, (E) CD8-T-Zellen und (F) Endothelzellen gezeigt. Bei den Fehlerbalken handelt es sich um Standardfehler des Mittelwerts (SEM), und die Signifikanz wurde mit ungepaarten t-Tests (**p ≤ 0,01) getestet. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
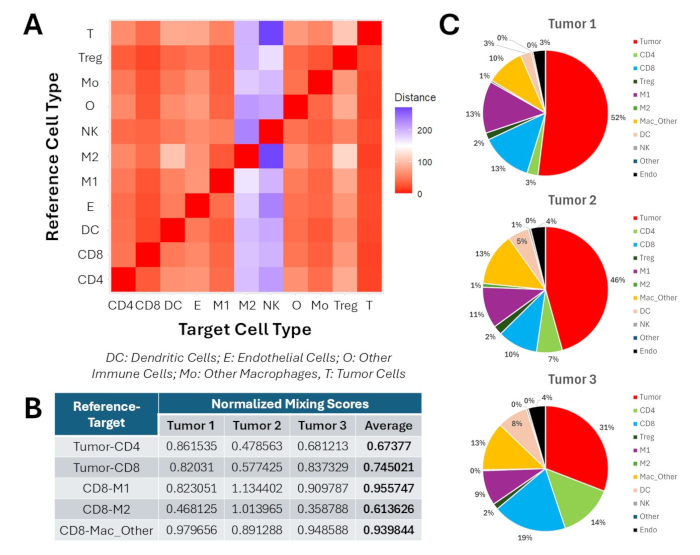
Abbildung 9: Profilierung intratumoraler räumlicher Metriken für Dreiflankentumoren, die mit intratumoralen 4-1BBL/IL-12-Nanopartikel-Injektionen und systemischem Anti-PD1 behandelt wurden. (A) Heatmap der durchschnittlichen Mindestabstände zwischen intratumoralen phänotypisierten Populationen. Die Messungen erfolgen in μm. (B) Normalisierte Mixing-Scores zwischen wichtigen intratumoralen Phänotyppaaren. (C) Aufschlüsselung der Zusammensetzung der Nachbarschaft in einem Radius von 100 μm um intratumorale CD8-T-Zellpopulationen. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
| Antikörper | Verdünnung | Belichtungszeit (ms) |
| SOX-10 | 50 | 450 |
| CD8 | 100 | 450 |
| CD3 | 100 | 450 |
| FoxP3 | 50 | 350 |
| CD4 | 50 | 450 |
| MHC-II | 100 | 450 |
| PDL1 | 50 | 450 |
| CD45 | 100 | 450 |
| Ki-67 | 100 | 300 |
| F4/80 | 100 | 150 |
| CD20 (Englisch) | 100 | 450 |
| NK1.1/CD161 | 50 | 450 |
| Nr. CD206 | 100 | 450 |
| CD68 | 100 | 450 |
| Granzyme-B | 50 | 450 |
| CD86 | 100 | 150 |
| CD31 | 100 | 450 |
| CD11C | 50 | 450 |
| CD11b | 50 | 450 |
| EOMES | 50 | 450 |
| TIM-3 | 50 | 300 |
| CD38 | 50 | 200 |
| LAG3 | 50 | 450 |
| Nr. CD163 | 50 | 200 |
| T-Wette | 50 | 300 |
| LMP2-Liga | 100 | 100 |
| Beta2 MG | 200 | 50 |
Tabelle 1: Einstellungen für Antikörperverdünnung und Expositionszeit.
Ergänzende Abbildung 1: Anfängliche Heatmap der Proteomik-Expression, die nach dem Seurat Clustering erstellt wurde. Bitte klicken Sie hier, um diese Abbildung herunterzuladen.
Ergänzende Abbildung 2: Anfängliche Heatmap der Proteomik-Expression, die nach dem FlowSOM-Clustering erstellt wurde. Bitte klicken Sie hier, um diese Abbildung herunterzuladen.
Diskussion
Der Erfolg der Bildgebung hängt von einem gut konzipierten und validierten Antikörperpanel ab. Die Multiplex-Immunfluoreszenz-Bildgebung von FFPE-Proben stellt aufgrund der hohen Autofluoreszenz und der Schwierigkeit, Epitope zu gewinnen, die durch Paraffineinbettung maskiert sind, eine Herausforderung dar. Da FFPE jedoch mehrere Vorteile im Vergleich zu FF-Proben bietet, ist es unerlässlich, FFPE-Antikörper-Panels zu entwerfen und zu validieren. Die Fertigstellung der Antikörperklon, die während der Immunfluoreszenz (IF)-Bildgebung positive Signale zeigen, ist der erste Schritt. Anschließend ist es wichtig, diese sorgfältig mit DNA-Barcodes zu konjugieren. Die Antikörperkonjugation erfordert eine teilweise Reduktion des Antikörpers, um SH-Bindungen zu erzeugen, die während der Maleimidgruppenreaktion mit einem Barcode verwendet werden. Nicht jeder Antikörperklon kann diesem Schritt standhalten, und einige Reaktionen können den Antikörper irreversibel schädigen, was trotz erfolgreicher Konjugation zu einem Versagen der Bildgebung führt. Auch wenn einige Antikörper während der konventionellen IF-Validierung positive Signale zeigen können, ist es aus diesem Grund wichtig, jeden Antikörper an dem tatsächlich interessierenden Gewebe zu validieren und die gewünschten Expositionszeiten für dieses Gewebe aufzuzeichnen, auch wenn einige Antikörper während der konventionellen IF-Validierung positive Signale zeigen können. In zukünftigen Anwendungen kann diese Technik mit bestehenden räumlichen Transkriptomik-Analysen auf aufeinanderfolgenden Objektträgern/demselben Objektträger kombiniert werden, um zusätzliche Erkenntnisse zu gewinnen. Eine der Einschränkungen dieser Methode besteht darin, dass sie eine sorgfältige Auswahl und Validierung jedes Antikörpers im Panel auf der Grundlage des Ziels und der Art des Gewebes erfordert.
In Bezug auf die Bildanalyse bietet QuPath ein wertvolles Open-Access-Tool mit hochwertiger Visualisierung von Proteomik-Markern, breiter Funktionalität für den Export von Intensitätsmessungen und die Durchführung von Qualitätskontrollen für Phänotyp-Klassifizierungen sowie eine gute Flexibilität für benutzergenerierte Skripte. Online-Foren wie https://forum.image.sc/ sind eine zusätzliche Ressource, um zu diskutieren, wie bestimmte Analyseaufgaben ausgeführt werden können, und um Skripte mit anderen Benutzern zu teilen. In diesem Protokoll vergleichen wir zwei Clustering- und Phänotypisierungsansätze mit Seurat und FlowSOM. Während FlowSOM aufgrund seiner Fähigkeit, detailliertere Einblicke in Immunzell-Subpopulationen des TME zu gewinnen, bevorzugt werden kann, muss auch die für die Proteomik-Analyse erforderliche Zeit berücksichtigt werden. Die Generierung von 100 Clustern kann unnötig sein, wenn ein Benutzer nur Zellen innerhalb einer oder zwei Gewebeproben phänotypisieren muss. In diesen Situationen kann Seurat eine schnellere und effizientere Pipeline für die Bildanalyse bieten. Im Gegensatz dazu ist es bei beiden Analyseansätzen wahrscheinlicher, dass die Analyse einer TMA mit mehr als 40 oder 50 Gewebeschnitten eine größere Anzahl von Zellclustern erzeugt, und FlowSOM kann die bevorzugte Methode zur Generierung nuancierterer Phänotyp-Klassifikationen sein.
Die Zellclusterbildung/Phänotypisierung und alle nachfolgenden Schritte der Bildanalyse hängen weitgehend von der Zellsegmentierung ab. Unsere aktuelle Arbeit hat die Zellsegmentierung in den Algorithmen HALO (Indica Labs) und StarDist untersucht, und wir haben festgestellt, dass beide Ansätze dazu neigen, Zellen basierend auf nukleären DAPI-Signalen zu übersegmentieren. Es stehen auch viele alternative Segmentierungsalgorithmen zur Verfügung, wie z. B. Mesmer19 und InstanSeg20. Dies ist ein wachsendes Feld der computergestützten Forschung, das weitere Untersuchungen und Optimierungen erfordert.
Offenlegungen
J.C.S. bedankt sich für die finanzielle Unterstützung von Emerson Collective LLC und den National Institutes of Health. J.J.G. bedankt sich auch für die Finanzierung durch die National Institutes of Health. J.C.S. unterhält eine Beziehung zu Palleon Pharmaceuticals Inc., die Finanzierungszuschüsse beinhaltet. S.Y.T. und J.J.G. haben eine Beziehung zu OncoSwitch Therapeutics, die Aktien oder Aktien umfasst. S.Y.T., J.J.G., J.C.S. und K.M.L. haben ein angemeldetes Patent. Alle anderen Autoren geben an, dass ihnen keine konkurrierenden finanziellen Interessen oder persönlichen Beziehungen bekannt sind, die als Einfluss auf die in dieser Arbeit vorgestellte Forschung wahrgenommen werden könnten.
Danksagungen
J.C.S. dankt der Dermatology Foundation und dem Dermatopathology Career Development Award für die Förderung der Karriere des Autors. Die Autoren danken Hsin-Pei Lee vom Cancer Data Science Laboratory des National Cancer Institute für die Unterstützung bei computergestützten Analysetechniken. Diese Forschung wurde vom Emerson Collective und den National Institutes of Health (R37CA246699, P41EB028239 und R01CA228133) finanziert. Darüber hinaus wird der Kern der Johns Hopkins Oncology Tissue Services (OTS) von den National Institutes of Health (P30CA006973) unterstützt.
Materialien
| Name | Company | Catalog Number | Comments |
| 16% paraformaldehyde (PFA) | Electron Microscopy Sciences | PN# 15710 | Required during tissue staining process |
| 50kDa MWCO filter | Millipore | UFC505096 | 25 kDa and 100 kDa result in failure |
| Akoya barcodes and reporters | Akoya Biosciences | Varied | Each barcode can be used to conjugate 50 ug of carrier free antibody and comes with two vials of reporters |
| Antibody conjugation kit | Akoya Biosciences | 7000009 | A conjugation kit is used to create custom barcode-conjugated antibodies. Each kit contains sufficient reagents for ten conjugations. The kit consists of one subkit box stored at 4 °C and one subkit box stored at -20 °C. The 4 °C subkit includes Filter Blocking Solution, Reduction Solution 2, Conjugation Solution, Purification Solution, and Antibody Storage Solution. The -20 °C subkit includes Reduction Solution 1. |
| Beta2MG | Abcam | ab214769 | |
| CD11b | CST | #41028 | |
| CD11C | CST | #39143SF | |
| CD163 | CST | #68922BF | |
| CD20 | CST | #45839 | |
| CD206 | CST | #87887 | |
| CD3 | CST | #24581 | |
| CD31 | CST | #92841 | |
| CD38 | CST | #68336BF | |
| CD4 | Abcam | ab271945 | |
| CD45 | CST | #98819 | |
| CD68 | CST | #29176 | |
| CD8 | Invitrogen | #14-0808-82 | |
| CD86 | CST | #20018 | |
| Dimethyl sulfoxide | Avantor/VWR | BDH1115-4LP | |
| EOMES | Abcam | ab222226 | |
| Eppendorf PCR tubes | Eppendorf | E0030124286 | Required store 5 μL conjugated antibody for conjugation confirmation by gel electrophoresis |
| Ethanol 200 proof | |||
| F4/80 | CST | # 25514 | |
| FoxP3 | CST | #72338 | |
| Gran-B | CST | #79903SF | |
| Heating oven | |||
| Hybridization chamber | Used to stain tissue with antibody cocktail | ||
| Hydrogen peroxide | Sigma | #216763 | Required for preparing bleaching solution |
| Instant pot pressure cooker or Decloaking Chamber ARC | Instant pot or Biocare Medical | ||
| Ki-67 | Akoya Biosciences | ||
| LAG3 | CST | #80282BF | |
| LMP2 | Abcam | ab243556 | |
| Methanol | |||
| MHC-II | eBioscience | #14-5321-82 | |
| NK1.1 | CST | #24395SF | |
| Nuclear Stain | Akoya Biosciences | 7000003 | |
| PDL1 | CST | #85095 | |
| Salmon Sperm DNA, sheared (10 mg/mL) | Invitrogen | AM9680 | Can be used as an alternative to assay reagent (Akoya) during reporter plate preparation step. |
| SOX-10 | Abcam | ab245760 | |
| Staining kit for PhenoCycler-Fusion | Akoya Biosciences | 7000017 | The PhenoCycler-Fusion Sample Kit includes the buffers and reagents necessary for tissue staining using antibodies conjugated with PhenoCycler barcodes, along with flow cells for whole-slide imaging. Each kit is sufficient for 10 PhenoCycler-Fusion experiments. It consists of one subkit stored at 4 °C, another at -20 °C, and a package of 10 flow cells kept at room temperature. The subkit stored at 4 °C contains Hydration Buffer, Staining Buffer, Storage Buffer, N Blocker, and J Blocker. The subkit stored at -20 °C contains G Blocker, S Blocker, and Fixative Reagent. |
| T-bet | CST | #53753 | |
| TIM-3 | CST | #72911 | |
| UltraPure DNase/RNase-free distilled water | Invitrogen | 10977015 | Required to dissolve barcodes during antibody conjugation |
Referenzen
- Schadendorf, D., et al. Melanoma. Lancet. 392 (10151), 971-984 (2018).
- Taube, J. M., et al. Implications of the tumor immune microenvironment for staging and therapeutics. Modern Pathol. 31 (2), 214-234 (2018).
- Marzagalli, M., Ebelt, N. D., Manuel, E. R. Seminars in cancer biology. 59, 236-250 (2019).
- De Visser, K. E., Joyce, J. A. The evolving tumor microenvironment: From cancer initiation to metastatic outgrowth. Cancer cell. 41 (3), 374-403 (2023).
- Balkwill, F. R., Capasso, M., Hagemann, T. The tumor microenvironment at a glance. J Cell Sci. 125 (23), 5591-5596 (2012).
- Phillips, D., et al. Highly multiplexed phenotyping of immunoregulatory proteins in the tumor microenvironment by codex tissue imaging. Front Immunol. 12, 687673(2021).
- Giesen, C., et al. Highly multiplexed imaging of tumor tissues with subcellular resolution by mass cytometry. Nat Methods. 11 (4), 417-422 (2014).
- Keren, L., et al. Mibi-tof: A multiplexed imaging platform relates cellular phenotypes and tissue structure. Sci Adv. 5 (10), eaax5851(2019).
- Lin, J. R., Fallahi-Sichani, M., Sorger, P. K. Highly multiplexed imaging of single cells using a high-throughput cyclic immunofluorescence method. Nat Comm. 6 (1), 8390(2015).
- Radtke, A. J., et al. Ibex: An iterative immunolabeling and chemical bleaching method for high-content imaging of diverse tissues. Nat Protoc. 17 (2), 378-401 (2022).
- Black, S., et al. Codex multiplexed tissue imaging with DNA-conjugated antibodies. Nat Protoc. 16 (8), 3802-3835 (2021).
- Aung, T. N., Bates, K. M., Rimm, D. L. High-plex assessment of biomarkers in tumors. Mod Pathol. 37 (3), 100425(2024).
- Surwase, S. S., et al. Highly-multiplexed immunofluorescence phenocycler panel for murine ffpe yields insight into tumor microenvironment immunoengineering. Lab Invest. 105 (1), 102165(2024).
- Luly, K. M., Green, J. J., Sunshine, J. C., Tzeng, S. Y. Biomaterial-mediated genetic reprogramming of merkel cell carcinoma and melanoma leads to targeted cancer cell killing in vitro and in vivo. ACS Biomater Sci Eng. 9 (11), 6438-6450 (2023).
- Satija, R., Farrell, J. A., Gennert, D., Schier, A. F., Regev, A. Spatial reconstruction of single-cell gene expression data. Nat Biotechnol. 33 (5), 495-502 (2015).
- Van Gassen, S., et al. Flowsom: Using self-organizing maps for visualization and interpretation of cytometry data. Cytometry Part A. 87 (7), 636-645 (2015).
- Butler, A., Hoffman, P., Smibert, P., Papalexi, E., Satija, R. Integrating single-cell transcriptomic data across different conditions, technologies, and species. Nat Biotechnol. 36 (5), 411-420 (2018).
- Watson, S. S., et al. Microenvironmental reorganization in brain tumors following radiotherapy and recurrence revealed by hyperplexed immunofluorescence imaging. Nat Comm. 15 (1), 3226(2024).
- Greenwald, N. F., et al. Whole-cell segmentation of tissue images with human-level performance using large-scale data annotation and deep learning. Nat Biotechnol. 40 (4), 555-565 (2022).
- Goldsborough, T., et al. Instanseg: An embedding-based instance segmentation algorithm optimized for accurate, efficient and portable cell segmentation. arXiv. , (2024).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenWeitere Artikel entdecken
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten