Method Article
Imagerie par immunofluorescence multiplex basée sur un code-barres d’ADN pour analyser des échantillons FFPE provenant d’un mélanome murin génétiquement reprogrammé
* Ces auteurs ont contribué à parts égales
Dans cet article
Résumé
Ce protocole fournit un guide étape par étape pour la conception d’un panel d’anticorps d’immunofluorescence multiplex pour l’imagerie basée sur un code-barres d’ADN des tissus murins de mélanome FFPE. Nous décrivons également un pipeline d’analyse d’images utilisant des outils open-source pour générer des informations protéomiques spatiales sur le microenvironnement immunitaire de la tumeur du mélanome murin.
Résumé
Nous présentons ici une technique émergente d’imagerie multiplex basée sur la co-détection par indEXing qui analyse la protéomique spatiale des microenvironnements tissulaires. Une imagerie réussie nécessite un répertoire de panels d’anticorps bien conçus et correctement validés, mais il en existe très peu à l’heure actuelle pour les échantillons fixés au formol et inclus dans la paraffine (FFPE). Le FFPE offre plusieurs avantages par rapport aux échantillons fraîchement congelés, tels qu’une grande disponibilité, une facilité de manipulation et de stockage, et la possibilité de fabriquer des microréseaux de tissus (TMA). Ici, nous présentons un protocole pour développer un panel d’anticorps pour visualiser et analyser les tissus FFPE à partir d’un modèle de mélanome murin traité avec des nanoparticules, qui délivrent de l’ADN plasmidique codant des signaux immunologiques pour la reprogrammation du microenvironnement tumoral. Nous décrivons également un pipeline d’analyse d’images utilisant des outils informatiques open source pour annoter les tissus, segmenter les cellules, traiter les données protéomiques, phénotyper les populations cellulaires et quantifier les métriques spatiales. Le protocole offre des applications pour la conception de panels d’anticorps dans le FFPE murin et la génération de nouvelles connaissances sur la protéomique spatiale de microenvironnements tissulaires complexes.
Introduction
Le mélanome cutané est le cancer de la peau le plus courant, avec des taux de maladie et de mortalité variables à travers le monde en fonction du moment du diagnostic et des soins primaires1. Au cours de la dernière décennie, une meilleure compréhension biologique du mélanome a contribué à propulser le développement de nouveaux modèles de cancer pour traiter les tumeurs solides2. L’essor récent de l’immunothérapie a conduit à un concept révolutionnaire de traitement du cancer basé sur l’activation du système immunitaire endogène 3,4.
Le microenvironnement tumoral (TME) est très complexe, composé de diverses cellules immunitaires, de fibroblastes associés au cancer, de péricytes, de cellules endothéliales et de diverses cellules tissulaires5. Plusieurs techniques ont été appliquées dans le passé pour étudier l’EUT, telles que la cytométrie en flux et le séquençage unicellulaire, qui compromettent le contexte spatial car elles sont nécessaires pour détruire le tissu tumoral. L’imagerie microscopique traditionnelle, telle que l’immunofluorescence (IF) et l’immunohistochimie (IHC), permet de visualiser des biomarqueurs protéiques sans détruire les tissus de l’échantillon. Cependant, ces approches sont limitées à deux ou trois biomarqueurs et ne sont pas en mesure de fournir une compréhension complète des relations spatiales et structurelles au sein du complexe TME 6.
Pour résoudre ce problème, plusieurs techniques d’imagerie multiplex ont été développées pour visualiser spatialement le TME complexe 7,8,9,10. L’un d’entre eux est la codétection par inDEXing, rebaptisée système PhenoCycler, basée sur des anticorps conjugués à l’ADN oligonucléotidique11. Le système peut fournir une imagerie unicellulaire et l’analyse de plus de 100 biomarqueurs pour des échantillons humains. Cependant, très peu d’anticorps inventoriés sont disponibles pour visualiser et analyser des échantillons murins, en particulier des échantillons fixés au formol et inclus dans de la paraffine (FFPE)12. Le FFPE offre plusieurs avantages par rapport à la conservation des produits frais congelés (FF), tels que la facilité de manipulation et de stockage, une morphologie bien préservée au fil du temps et, surtout, la possibilité de préparer des microréseaux de tissus/tumeurs (TMA) qui permettent de visualiser plusieurs échantillons sur une seule lame. Nous avons récemment conçu et développé un panel d’anticorps murins FFPE CODEX/PhenoCycler et l’avons appliqué avec succès pour visualiser et analyser la protéomique spatiale d’échantillons de mélanome murin génétiquement reprogrammés13.
L’objectif général de ce protocole est de fournir un guide étape par étape pour la conception d’un panel d’anticorps FFPE murin et de décrire le processus de conjugaison anticorps-code-barres, de coloration tissulaire et d’imagerie. De plus, nous présentons un pipeline d’analyse d’images détaillé utilisant des outils open source tels que les packages QuPath et R. Après avoir suivi ce protocole, les chercheurs apprendront à concevoir un panel d’anticorps conjugués personnalisés, à effectuer une imagerie multiplex à l’aide d’un dispositif Phenocycler-Fusion et à acquérir de nouvelles connaissances sur la protéomique spatiale de l’EUT du mélanome. De plus, ce protocole peut être adapté pour étudier divers microenvironnements immunitaires tumoraux et combiné avec des techniques de transcriptomique spatiale existantes.
Protocole
Tous les travaux sur les animaux ont été effectués conformément aux lignes directrices établies par le Comité de protection et d’utilisation des animaux de Johns Hopkins, en utilisant les numéros de protocole approuvés MO18M388 et MO21M384.
1. Sélection des anticorps
- Concevez un panel d’anticorps en fonction du tissu d’intérêt et de l’abondance du biomarqueur particulier. Choisissez des clones d’anticorps en fonction des applications précédentes réussies de l’immunofluorescence (IF) et de l’immunohistochimie (IHC) dans la littérature. Versions sécurisées sans transporteur de ces anticorps.
REMARQUE : Un anticorps purifié sans support est important pour la conjugaison du code-barres. Évitez tout conservateur à base de protéines tels que le BSA, le gluten, le glycérol, etc. Si l’anticorps sans porteur n’est pas disponible, demandez un anticorps personnalisé au fournisseur ou utilisez un kit de retrait BSA pour fabriquer l’anticorps sans porteur. L’azoture de sodium n’interfère pas avec la conjugaison des anticorps. Les isotypes IgG sont recommandés par rapport aux isotypes IgM. - Vérifiez ces clones sélectionnés sur le tissu d’intérêt à l’aide de l’imagerie conventionnelle par immunofluorescence (IF)11. Utiliser l’imagerie double FI pour confirmer la spécificité de certains clones (p. ex., effectuer une imagerie double FI avec FOXP3 et CD4 pour confirmer la coloration des lymphocytes T régulateurs).
REMARQUE : Dans la mesure du possible, il est important d’utiliser le même tissu pour la validation de la FI que celui qui sera utilisé pour la coloration. Sélectionnez la solution de récupération d’antigène appropriée. Dans ce protocole, nous avons utilisé un tampon AR9 fourni par Akoya Biosciences pour récupérer tous les épitopes tissulaires FFPE du mélanome murin. D’autres solutions de récupération d’antigènes, telles que AR6 ou Universal, peuvent être utilisées en fonction des exigences de l’épitope.
2. Conjugaison et confirmation des anticorps
- Attribuez des anticorps vérifiés à des codes-barres dotés de rapporteurs complémentaires attachés aux fluorophores ATTO550 (Cy3), AF647 (Cy5) ou AF750 (Cy7). Les antigènes faiblement abondants produisent généralement des signaux plus faibles ; conjuguer ces anticorps à des canaux de faible autofluorescence tels que AF647 (Cy5). Conjuguez des antigènes fortement exprimés à ATTO550 (Cy3) et AF750 (Cy7) en raison de la possibilité d’autofluorescence.
REMARQUE : Cette étape critique tient compte de la sensibilité du canal et de l’abondance de l’antigène. L’utilisation du canal AF488 pour les tissus FFPE n’est pas recommandée en raison de son autofluorescence plus élevée. - Pour la conjugaison anticorps-code-barres (4,5 h), procurez-vous le kit de conjugaison d’anticorps. Conserver tous les réactifs à 4 °C, à l’exception de la solution de réduction 1, qui doit être conservée à -20 °C sous forme de petits flacons à usage unique.
- Mesurez la concentration d’anticorps à l’aide d’un spectrophotomètre.
- Démarrez la conjugaison des anticorps en appliquant 500 μL de la solution de blocage du filtre dans une colonne de filtre MWCO de 50 kDa pour bloquer la liaison non spécifique des anticorps au filtre. Essorer à 12 000 x g pendant 2 min à température ambiante (RT). Retirez le liquide supplémentaire de la colonne à l’aide d’une micropipette de 200 μL.
REMARQUE : La solution de réduction 1 est un flacon de solution à usage unique, ce qui est suffisant pour trois conjugaisons d’anticorps à la fois. Ne réutilisez pas la solution après la décongélation ; Jetez tout réactif restant. N’utilisez pas de filtre sur la colonne de filtre MWCO de 50 kDa, car cela peut entraîner une mauvaise purification et une mauvaise conjugaison. - Utilisez un volume équivalent de 50 μg de la solution d’anticorps et ajoutez-le à un filtre MWCO de 50 kDa. Ajustez le volume total à 100 μL à l’aide de PBS si nécessaire. Essorage à 12 000 x g pendant 8 min à 4 °C. Pendant ce temps, préparez le mélange maître de réduction en utilisant 19,8 μL de solution de réduction 1 + 825 μL de solution de réduction 2 pour trois conjugaisons d’anticorps.
- Jeter l’écoulement continu et ajouter 260 μL de mélange maître de réduction sur le dessus de chaque unité de filtration. Solution vortex dans l’unité de filtration pendant 2-3 secondes et incubation pendant 30 min à RT.
REMARQUE : Il est essentiel de ne pas dépasser 30 minutes d’incubation pour éviter une réduction excessive des anticorps, car cela les endommage et peut entraîner un échec de la conjugaison. - Après 30 min d’incubation, essorer à 12 000 x g pendant 8 min à 4 °C et jeter couler par le bas. Ajouter 450 μL de solution de conjugaison sur le haut de la colonne et faire tourner vers le bas à 12 000 x g pendant 8 min à 4 °C. Pendant la centrifugation, préparez la solution de code-barres CODEX. Déplacez-vous rapidement après avoir récupéré le code-barres à -20 °C, car le code-barres commence à se dégrader.
REMARQUE : Les codes-barres sont généralement fournis dans de petits flacons en verre contenant des produits lyophilisés (flocons ou poudre) stockés à -20 °C. Ces flacons peuvent être utilisés pour conjuguer 50 μg d’anticorps à la fois. Il est recommandé de ne pas conjuguer plus de trois anticorps à la fois. - Localisez soigneusement le code-barres lyophilisé au fond du flacon en verre (il peut également être collé aux parois). Tapotez le flacon en verre sur une table pour ramener les solides au fond. La localisation du produit lyophilisé est essentielle lors de sa dissolution dans 10 μL d’eau de biologie moléculaire sans nucléases. Après avoir ajouté 10 μL d’eau exempte de nucléases, ajoutez 210 μL de solution de conjugaison à chaque code-barres. Dissoudre tout le matériau lyophilisé et mélanger doucement en pipetant de haut en bas. Réserver.
- Après avoir terminé l’essorage à l’étape 2.2.5, jetez le flux et ajoutez la solution de code-barres correspondante de l’étape 2.2.6 en haut de chaque unité de filtration. Économisez 1 μg de l’anticorps non conjugué initial ; il sera effectué avec l’échantillon d’anticorps conjugué de 5 μL lors de la validation de l’électrophorèse sur gel.
- Étiquetez chaque tube avec le nom de l’anticorps et du code-barres correspondant. Fermez le couvercle et mélangez la solution en tourbillonnant pendant 2-3 s. Incuber la réaction de conjugaison anticorps-code-barres pendant 2 h à RT.
- Après 2 h d’incubation, prélever 5 μL d’anticorps conjugués et les conserver dans un tube PCR de 0,2 mL pour confirmer la conjugaison par électrophorèse sur gel.
- Faire tourner la solution d’anticorps conjugués restante à 12 000 x g pendant 8 min à 4 °C. Ajouter 450 μL de solution de purification sur le dessus de la colonne, faire tourner à 12 000 x g pendant 8 min à 4 °C et jeter l’écoulement. Répétez cette étape de purification 2 fois, en ajoutant 450 μL de solution de purification à chaque fois et en la faisant tourner.
- Après la 3ème centrifugation, jeter le flux. Le filtre supérieur contiendra les anticorps conjugués au code-barres.
- Pour recueillir l’anticorps conjugué, étiquetez le nouveau tube extérieur qui maintient la colonne de filtre avec l’anticorps et le code-barres correspondants. Coupez le couvercle du tube extérieur avant la centrifugation. Ajoutez 100 μL de solution de stockage d’anticorps dans chaque unité de filtrage et placez le tube extérieur nouvellement étiqueté à l’envers sur le dessus de la colonne de filtre.
- Inversez la colonne filtrante pour recueillir l’anticorps conjugué dans le tube extérieur et tournez à 3 000 x g pendant 2 min à RT. Celui-ci devrait recueillir environ 120 μL de solution d’anticorps conjugués. Transférer dans un tube de microcentrifugation stérile et conserver à 4 °C pendant 18 à 24 mois.
REMARQUE : Pour éviter une coloration nucléaire de fond élevée après la conjugaison, il est recommandé de ne pas utiliser ces anticorps pendant au moins 2 jours.
- Effectuer la confirmation de la conjugaison par électrophorèse sur gel comme décrit dans le protocole13 déjà publié.
REMARQUE : Ce processus ne fait que confirmer le succès de la réaction de conjugaison chimique utilisée pour conjuguer les codes-barres avec des anticorps. Elle est facultative, car la validation des anticorps n’est possible qu’après une coloration et une imagerie réussies du tissu.
3. Préparation de l’échantillon FFPE murin
- Obtenez des échantillons de tissus FFPE de souris femelles C57BL/6J implantées avec des tumeurs de flanc B16F10. Traitez les souris avec des injections intratumorales de nanoparticules à base de poly(β-amino ester) contenant des plasmides 4-1BBL et IL-12, ainsi qu’une administration intrapéritonéale d’anti-PD1 à 9, 11, 16 et 18 jours après l’implantation14. Sacrifiez des souris le 20e jour et réparez les tumeurs dans du formol à 10 %, incorporé dans de la paraffine, puis sectionnez au cœur des services tissulaires d’oncologie de Johns Hopkins.
- Examinez attentivement les spécifications de la zone d’imagerie fournies dans le manuel d’utilisation et montez des échantillons de 5 μm sur une lame à l’intérieur de la zone d’imagerie. Nous avons pu monter quatre échantillons tumoraux différents sur une seule lame.
REMARQUE : Il est essentiel d’utiliser les lames recommandées disponibles pour l’imagerie, telles que les lames adhésives Leica Apex ou les diapositives Fisherbrand Superfrost Plus. Cela aidera la cellule d’écoulement à adhérer fermement à la lame.
4. Coloration et imagerie tissulaire FFPE
- Vérification et optimisation de la concentration des anticorps et des temps d’exposition : Pour vérifier le succès de la conjugaison anticorps-code-barres, colorez le tissu d’intérêt avec 6 à 9 anticorps conjugués. Exécutez l’appareil d’imagerie multiplex et notez chaque concentration d’anticorps et temps d’exposition. Ajustez les dilutions et les temps d’exposition au besoin, puis analysez 6 à 9 nouveaux anticorps avec ceux précédemment vérifiés et notez tous les ajustements. Suivez les étapes ci-dessous pour la coloration et l’imagerie.
- Coloration et imagerie des tissus
- La veille de la coloration, faites cuire les lames de tissu FFPE dans un four à 60 °C pendant la nuit. Le lendemain, refroidissez les lames pendant 10 min à RT avant de commencer les étapes de déparaffinisation et de réhydratation des tissus.
- Commencer la déparaffinisation des tissus en les incubant dans une solution de xylène 2 fois pendant 5 minutes chacune.
- Pour réhydrater le tissu, faites-le passer à travers deux cycles de solution d’éthanol à 100 % pendant 5 minutes chacun. Ensuite, passez-le dans une série de solutions d’alcool de 90 % à 70 %, 50 % et 30 % pendant 5 minutes chacune. Enfin, lavez le mouchoir à l’eau distillée deux fois pendant 5 minutes chacune.
REMARQUE : Des alternatives moins toxiques au xylène, telles qu’une solution Neoclear, peuvent être utilisées pour les étapes de déparaffinisation. Effectuer la déparaffinisation et la réhydratation sous une hotte. - Diluez 10x tampon AR9 à 1x avec de l’eau distillée. Remplissez un bocal Coplin avec 50 ml de 1 tampon AR9, en vous assurant que les lames sont entièrement immergées dans le tampon de récupération d’antigène.
- Remplissez un autocuiseur d’eau de manière à ce que le niveau soit à mi-hauteur du bocal Coplin, puis incubez à haute pression pendant 20 min.
REMARQUE : Il est possible d’utiliser un autocuiseur spécialisé ou une chambre de démasquage pour une meilleure récupération de l’antigène en le réglant à 120 °C pendant 10 min. - Une fois l’étape de cocotte-minute terminée, refroidissez les lames pendant 45 à 60 minutes. Rincez la lame à l’eau distillée 2 fois pendant 2 minutes chacune. Ensuite, transférez la lame dans le bocal contenant 1x PBS.
- Pour minimiser l’autofluorescence, effectuez une étape de blanchiment sur le tissu avant de colorer avec des anticorps. Préparez fraîchement une solution de blanchiment (25 ml de 1x PBS + 0,8 ml de 1M NaOH + 4,5 ml de H2O2) en utilisant les réactifs et les concentrations recommandés. Incuber les lames dans un récipient en plastique contenant la solution de blanchiment entre deux lampes LED pendant 45 min à température ambiante. Répétez l’opération avec une solution de blanchiment fraîche pendant encore 45 min.
- Commencez à préparer la solution cocktail d’anticorps pendant cette étape de blanchiment, surtout s’il y a plusieurs anticorps dans le panel avec différentes dilutions. Récupérez les quatre bloqueurs (G, S, J et N) et placez-les sur de la glace, car certains mettent du temps à décongeler à partir de -20 °C.
- Après le photoblanchiment, lavez le mouchoir avec 1x PBS, 2x pendant 2 min chacun. Déplacez le mouchoir dans des bocaux contenant un tampon d’hydratation et lavez-le 2 fois pendant 2 minutes chacun.
- Transférez les lames de tissu dans le tampon de coloration et laissez-les s’équilibrer pendant 30 min. S’il n’est pas préparé pendant l’étape de blanchiment, préparez la solution de cocktail d’anticorps pendant ce temps.
- Préparez 300 μL de solution cocktail d’anticorps pour colorer deux lames (habituellement, si vous utilisez une chambre en plastique d’hybridation, 100 à 120 μL de cocktail d’anticorps suffisent pour colorer chaque lame).
- Colorez les lames en appliquant une solution cocktail d’anticorps à l’intérieur de la chambre d’humidité pendant une nuit à 4 °C.
REMARQUE : Tous les anticorps ici montrent un signal positif lorsqu’ils sont colorés pendant la nuit à 4 °C. Cependant, le temps et la température de coloration de chaque anticorps peuvent varier et doivent être optimisés. Les conditions de coloration les plus couramment utilisées sont 3 h à RT ou une nuit à 4 °C. Si nécessaire, il est également possible de colorer quelques anticorps d’abord à différentes températures, puis les anticorps restants pendant la nuit à 4 °C. Lavez ensuite les mouchoirs avec du PBS si une coloration séquentielle est nécessaire. - Le lendemain, effectuez une fixation post-coloration. Retirez la chambre en plastique et conservez le cocktail d’anticorps collecté à 4 °C. Lavez les lames 2 fois dans le tampon de coloration pendant 2 minutes chacune. Ensuite, placez les lames dans une solution de fixation post-coloration de 40 ml (4 ml de paraformaldéhyde à 16 % + 36 ml de tampon de stockage) et incubez pendant 10 min. Rincer au PBS 3x pendant 2 min chacun.
- Placez les lames dans du méthanol glacé pendant 5 min, puis rincez avec du PBS 3x pendant 2 min chacune. Appliquer une solution fixatrice finale (20 μL un tube + 1 mL de PBS) sur les lames sous une chambre d’humidité. Habituellement, 200 μL de solution fixatrice suffisent par lame. Laissez le fixateur reposer pendant 20 minutes à RT. Effectuez trois derniers lavages dans PBS pendant 2 minutes chacun.
REMARQUE : Ne retirez pas le tube fixateur de -20 °C avant ; Décongeler seulement avant l’application. - Si l’imagerie est immédiate, essuyez la lame autour du tissu à l’aide d’un mouchoir non pelucheux et appliquez la cellule d’écoulement en appuyant dessus pendant 30 secondes à l’aide d’un équipement d’assemblage de cellules d’écoulement. Si l’imagerie est ultérieure, stockez la lame (sans appliquer la cellule d’écoulement) dans la mémoire tampon de stockage à 4 °C. Lorsque vous êtes prêt à prendre l’image, transférez les diapositives de la mémoire tampon de stockage vers PBS pendant 10 minutes avant d’appliquer la cellule d’écoulement.
- Commencez à préparer le tampon de fonctionnement 1x avec un additif basé sur les cycles souhaités dans l’expérience. Préparez des tampons DMSO faibles (1:4) et élevés (9:1) en les mélangeant en tant que DMSO 1 partie avec 4 parties de tampon de fonctionnement et 9 parties de DMSO avec 1 partie de tampon de fonctionnement requis pour les cycles souhaités. Économisez environ 20 ml de 1x tampon avec additif pour la fabrication d’une plaque rapporteure. Pour calculer le tampon 1x requis avec des tampons DMSO additifs et haut/bas, consultez le gestionnaire de l’instrument.
- Commencez à préparer la plaque de reporter. Tout d’abord, préparez la solution mère rapporteur requise pour le nombre total de cycles. Étiquetez les tubes de microcentrifugation de 1 ml de couleur noire ou ambrée avec les noms de cycle. Ajouter 250 μL de solution mère de rapporteur et 5 μL de chaque rapporteur dans le tube correspondant. Ensuite, transférez la solution dans une plaque noire à 96 puits et scellez-la avec une feuille adhésive.
- Démarrez l’appareil, utilisez le gestionnaire d’instruments pour régler les paramètres et les temps d’exposition (comme indiqué dans le tableau 1) et acquérez les images. La figure 1 résume les processus de précoloration et de post-coloration.
5. Annotation tissulaire et segmentation cellulaire
- Configurer un projet et un espace de travail.
- Installez la dernière version du logiciel d’analyse de pathologie numérique (par exemple, QuPath version 0.5.1 ou ultérieure). Cliquez sur Créer un projet pour choisir un dossier de destination pour l’espace projet.
- Cliquez sur Ajouter des images > Choisissez des fichiers, puis naviguez jusqu’au fichier QPTIFF (produit à partir de l’imagerie d’immunofluorescence multiplexe). Définissez le type d’image sur Fluorescence, conservez tous les autres paramètres par défaut, puis cliquez sur Importer.
- Si vous utilisez QuPath, double-cliquez sur la nouvelle image pour ouvrir un espace de travail. Cliquez sur Fichier > Enregistrer régulièrement pour suivre les modifications apportées au projet. Basculez la visibilité des marqueurs et les paramètres d’affichage à l’aide de la luminosité et du contraste (icône en forme de demi-lune) près du milieu de la barre d’outils.
REMARQUE : cliquez avec le bouton droit de la souris sur une image dans la liste des images pour la renommer. Cela sera important plus tard lors de la visualisation des classifications de phénotypes.
- Annoter la coupe complète du tissu, le compartiment intratumoral et le compartiment stromal.
- Utilisez l’outil pinceau et/ou baguette pour dessiner une annotation autour de toute la section du tissu. Définissez cette annotation comme Full_Tissue. Excluez la peau sus-jacente et/ou les régions qui ne doivent pas être analysées. Pour créer une annotation négative et/ou réduire la limite d’annotation, maintenez la touche Alt enfoncée tout en utilisant l’un des outils d’annotation.
- Dupliquez l’annotation complète du tissu. Sélectionnez l’annotation sous l’onglet Annotations, puis allez dans Objets > Annotations... > Dupliquer les annotations sélectionnées.
- Activez le canal SOX10. Sur l’annotation dupliquée, réduisez la limite de l’annotation pour capturer le compartiment intratumoral à l’aide de l’outil Alt + pinceau/baguette. Définissez cette annotation comme une tumeur (Figure 2).
- En sélectionnant l’annotation complète du tissu, allez dans Objets > Annotations... > Développez les annotations. Définissez le rayon d’expansion sur 1 μm et cliquez sur Exécuter.
- Renommez la nouvelle annotation en Full_Tissue_Expansion. Sélectionnez l’annotation de tumeur, cliquez avec le bouton droit de la souris et sélectionnez Insérer dans la hiérarchie.
- Sélectionnez à nouveau l’annotation de tumeur, allez dans Objets > Annotations... > Faites l’inverse. Définissez cette nouvelle annotation comme Stroma. Répétez l’opération pour toutes les sections de tissus.
- Exécutez la segmentation des cellules et exportez les résultats.
- Accédez à l’onglet Image pour connaître la largeur et la hauteur en pixels de l’image (Figure 3A).
- Téléchargez l’extension StarDist à l’https://github.com/qupath/qupath-extension-stardist/releases. Faites glisser le fichier qupath-extension-stardist-[version].jar dans la fenêtre QuPath, puis cliquez sur l’icône d’engrenage dans le coin supérieur droit de la fenêtre QuPath. Sous Extensions, définissez le répertoire utilisateur QuPath comme emplacement du dossier StarDist.
- Téléchargez les fichiers de script groovy StarDist et le fichier de modèle StarDist à partir de https://github.com/xzhou125/JOVE_QuPath_Spatial_Analysis (les scripts StarDist ont été initialement organisés à partir d’Akoya Biosciences).
- Pour chaque section de tissu, sélectionnez les annotations Tumeur et Stroma (utilisez Ctrl pour sélectionner plusieurs annotations). Dans la barre de paramètres supérieure, cliquez sur Automatiser > éditeur de script pour ouvrir l’interface des scripts. Ouvrez le script de segmentation de cellule StarDist approprié (fichier groovy) qui correspond à la taille des pixels de l’image. Lorsque vous êtes invité à sélectionner le fichier de segmentation de cellule, ouvrez le fichier stardist_cell_seg_model.pb.
- Après la segmentation de la cellule, enregistrez l’image. Allez dans Mesure > Exporter les mesures, puis sélectionnez la ou les images correspondantes. Attribuez le type d’exportation Cellules et Séparateur .csv, puis choisissez l’emplacement du fichier de sortie . Cliquez sur Remplir à côté de Colonnes pour inclure les indicateurs qui vous intéressent. Les colonnes importantes à exporter sont Image, ID d’objet, Parent, Centroïde X, Centroïde Y et Nucleus Area.
REMARQUE : L’ID d’objet est utilisé dans les étapes suivantes pour remapper les classifications sur l’image d’immunofluorescence multiplex dans le logiciel d’analyse de pathologie numérique. La colonne Parent indique le nom de l’annotation à laquelle appartient la cellule. Les données X et Y sont utilisées pour l’analyse spatiale. - Pour chaque marqueur de lignage à utiliser dans le clustering/phénotypage, sélectionnez une valeur moyenne à exporter. Pour les marqueurs nucléaires (par exemple, SOX10 et FOXP3), exportez l’option Nucleus : Mean. Pour les marqueurs cytoplasmiques/membraneux (par exemple, CD45, CD3, CD4), exportez l’option Cytoplasme : Moyenne.
6. Prétraitement et normalisation des données protéomiques
- Ouvrez le fichier CSV exporté. Pour chaque marqueur à utiliser dans le clustering/phénotypage, tronquez le nom de l’en-tête de colonne pour n’inclure que le marqueur. Par exemple, renommez SOX10 : Noyau : Moyenne en SOX10 et CD3 : Cytoplasme : Moyenne en CD3.
- Installez la dernière version de R et RStudio. Téléchargez le fichier Marker Normalization.R à partir de https://github.com/xzhou125/JOVE_QuPath_Spatial_Analysis et exécutez le script. Il s’agit d’abord de filtrer les cellules par taille nucléaire.
- Pour chaque marqueur, une normalisation min-max est ensuite effectuée de sorte que la valeur MFI la plus basse dans la plage d’expression soit définie sur 0 et que la valeur MFI au 99,7e centile soit définie sur 1. Toutes les intensités supérieures au 99,7e centile sont coupées à 1. Une fois terminé, enregistrez le nouveau fichier CSV généré avec les données normalisées.
7. Regroupement et phénotypage
- Choisissez un algorithme de regroupement des cellules en fonction des profils d’expression des marqueurs. Au https://github.com/xzhou125/JOVE_QuPath_Spatial_Analysis, des scripts R sont fournis pour le clustering à l’aide de Seurat v4.415 et FlowSOM v2.13.9 (outil de clustering et de visualisation)16.
- Cellules phénotypiques selon les profils d’expression des marqueurs clés de la lignée. Le protocole présenté ici a utilisé 14 marqueurs de lignage pour le clustering et le phénotypage : SOX10, CD45, CD3, CD4, CD8, FOXP3, CD11C, F4/80, CD68, CD86, CD163, CD206, NK1.1 et CD31.
- Définissez les cellules tumorales comme SOX10hi. Définissez les lymphocytes T CD4 comme CD45mod-hi/ CD4hi/CD8low/FOXP3low. Définissez les lymphocytes T CD8 comme CD45mod-hi/ CD8hi/CD4low. Définissez les Tregs comme CD45mod-hi/CD4hi/CD8low/FOXP3hi. Définissez les autres lymphocytes T comme CD45mod-hi/CD3hi/CD4low/CD8low.
- Définissez les macrophages M1 comme CD45mod-hi/F4/80hi/CD68hi/CD86hi/CD163low /CD206low. Définissez les macrophages M2 comme CD45mod-hi/F4/80hi/CD68mod-hi/CD86low/CD163hi/CD206low, CD45mod-hi/F4/80hi/CD68mod-hi/CD86low/CD163low/CD206hi, ou CD45mod-hi/F4/80hi/CD68mod-hi/CD86low/CD163hi/CD206hi. Définissez les autres macrophages comme CD45mod-hi/F4/80hi/CD68mod-hi/CD86low/ CD163low/CD206low ou CD45mod-hi/F4/80hi/CD68mod-hi/CD86hi/CD163hi/CD206hi.
- Définissez les cellules dendritiques comme CD45mod-hi/CD11chi/CD86mod-hi. Définissez les cellules NK comme CD45mod-hi/NK1.1hi. Définir les autres cellules immunitaires comme CD45mod-hi qui n’ont pas satisfait aux conditions détaillées ci-dessus. Définir les cellules endothéliales comme CD31mod-hi. Supprimez tous les autres profils d’expression.
8. Remappage des classifications de phénotypes et contrôle de la qualité
- Téléchargez le script Remapping Classifications.groovy à partir de https://github.com/xzhou125/JOVE_QuPath_Spatial_Analysis (ce script a été adapté d’un article sur https://forum.image.sc/ par petebankhead).
- Ouvrez le fichier CSV phénotypé et assurez-vous que toutes les étiquettes sous l’onglet Image correspondent exactement au nom du fichier d’image dans le logiciel d’analyse de pathologie numérique qui sera utilisé pour le remappage. Pour modifier rapidement un grand nombre d’étiquettes dans le fichier CSV, utilisez la fonction Remplacer tout.
- Si vous utilisez QuPath, exécutez le fichier groovy de remappage des classifications en cliquant sur Automatiser > éditeur de script.
- Dans l’onglet Annotations, cliquez sur les trois points à côté du bouton Réglage automatique, puis cliquez sur Remplir à partir d’objets existants > Toutes les classes (y compris les sous-classes) > Oui.
- Faites défiler la liste des marqueurs vers le bas pour afficher les différentes classifications de phénotypes et ajustez le schéma de couleurs si nécessaire. Double-cliquez sur n’importe quelle cellule pour afficher son phénotype (à côté de la balise Classification) et son cluster (à côté de la balise Nom). Vérifiez les champs de vision représentatifs pour déterminer la précision des affectations de phénotypes et révisez la stratégie de phénotypage si nécessaire (Figure 3B).
9. Quantification de la densité et analyse spatiale
- Une fois le phénotypage finalisé, obtenez les zones d’annotation dans le logiciel d’analyse de pathologie numérique sous l’onglet Annotations. Convertissez les valeurs de surface en mm2, puis divisez les nombres par les zones d’annotation pour calculer les densités.
- Téléchargez le script SPIAT Spatial Analysis.R à partir de https://github.com/xzhou125/JOVE_QuPath_Spatial_Analysis et exécutez-le. Collectez des données sur les distances minimales moyennes (DMLA), les compositions de quartier et les scores de mélange normalisés (NMS).
- Générer des chiffres dans un logiciel d’analyse statistique pour présenter les résultats. Lors de la génération d’une seule carte thermique pour les distances minimales moyennes des conditions de traitement, faites la moyenne des résultats obtenus à partir de différentes coupes de tissus.
Résultats
Ici, nous présentons un protocole pour la conception d’un panel d’anticorps pour le tissu FFPE murin, la réalisation d’une imagerie d’immunofluorescence multiplex et l’analyse d’images pour la quantification protéomique et les relations spatiales. Le panel validé contient 27 anticorps qui fournissent des marqueurs pour visualiser les cellules de mélanome (SOX10), les leucocytes (CD45), les cellules T (CD3, CD4, CD8, FOXP3), les cellules B (CD20), les macrophages et sous-types (F4/80, CD68, CD86, CD163, CD206), les cellules dendritiques (CD11c), les cellules NK (NK1.1) et les cellules endothéliales (CD31). Le panel complet contient également des marqueurs pour d’autres populations immunitaires (CD11b, CD38), l’activité de prolifération (Ki67), la fonctionnalité des lymphocytes T (T-bet, Eomesodermin, granzyme B), la présentation de l’antigène (LMP2, bêta-2 microglobuline, CMH II) et l’expression du point de contrôle (TIM3, LAG3,-L1)12. Une image représentative de l’électrophorèse sur gel confirme la conjugaison réussie des codes-barres d’oligonucléotides d’ADN aux anticorps sans porteur, comme le montre la figure 4. Cette étape ne permet que de confirmer la réaction chimique, et la confirmation de l’image ne peut être effectuée qu’après vérification de ces anticorps avec le dispositif d’imagerie multiplex sur le tissu d’intérêt. Voir la référence suivante pour voir les images validées des 27 marqueurs du panneau13. La figure 5 présente une image de fusion de marqueurs de lignage clés colorant trois sections de tissu de mélanome murin traitées par des injections intratumorales de nanoparticules 4-1BBL/IL-12 et d’anti-PD1 systémique. Ces sections ont été utilisées pour l’analyse ultérieure des images dans ce protocole.
Après l’annotation tissulaire, la segmentation cellulaire et le prétraitement des données protéomiques, nous présentons une comparaison entre deux algorithmes de clustering qui ont déjà été appliqués pour l’analyse transcriptomique et/ou protéomique sur cellule unique17,18. Les profils d’expression des populations phénotypées sont présentés à la figure 6 pour les deux approches. Nous montrons que FlowSOM offre une gamme plus large de valeurs d’intensité (~0,7 vs ~0,5) pour discerner les différences entre des populations cellulaires similaires, telles que les sous-types de macrophages. Seurat a appliqué l’algorithme de Louvain pendant le clustering et a généré 29 clusters (figure supplémentaire 1). En comparaison, FlowSOM a appliqué des cartes auto-organisées et peut générer 100 clusters (figure supplémentaire 2). Un plus grand nombre de grappes se traduit par plus de temps nécessaire pour le phénotypage, mais cette dernière approche FlowSOM offre également plus de nuances lors de la classification de populations cellulaires similaires. Qualitativement, nous voyons que FlowSOM a été capable de classer plus de macrophages intratumoraux comme un sous-type M1 ou M2 par rapport au phénotypage Seurat dans le même champ de vision (Figure 7). Le même résultat est observé lorsque nous quantifions les densités de macrophages, FlowSOM capturant une densité plus élevée de macrophages M1 et M2 par rapport à Seurat (Figure 8A-B) et par conséquent une densité significativement plus faible d’autres macrophages non classifiés (p = 0,0028 ; Figure 8C). Néanmoins, les deux approches d’analyse ont également généré des résultats similaires lors de la description d’autres densités de population cellulaire, telles que les cellules T CD4, les cellules T CD8 et les cellules endothéliales (Figure 8D-F).
Nous présentons également les résultats de l’analyse spatiale en aval après le regroupement et le phénotypage. La figure 9 illustre certaines des métriques spatiales qui peuvent être générées à l’aide de ce protocole. Par rapport à toutes les populations phénotypées, les macrophages M2 et les cellules NK présentaient les DMLA les plus élevées après un traitement avec des nanoparticules 4-1BBL/IL-12 et des anti-PD1 (Figure 9A). De même, la NMS entre les lymphocytes T CD8 intratumoraux et les macrophages M1 était beaucoup plus élevée qu’entre les lymphocytes T CD8 et les macrophages M2 (Figure 9B). De plus, les macrophages M2 représentaient ~1 % dans le voisinage de 100 μm entourant les lymphocytes T CD8 intratumoraux, tandis que les macrophages M1 représentaient 9 à 13 % de ces quartiers (Figure 9C). Pris ensemble, ces résultats suggèrent que le schéma de traitement 4-1BBL/IL-12 a polarisé les macrophages associés à la tumeur vers un sous-type M1 et exclu les macrophages M2 du microenvironnement immunitaire tumoral.
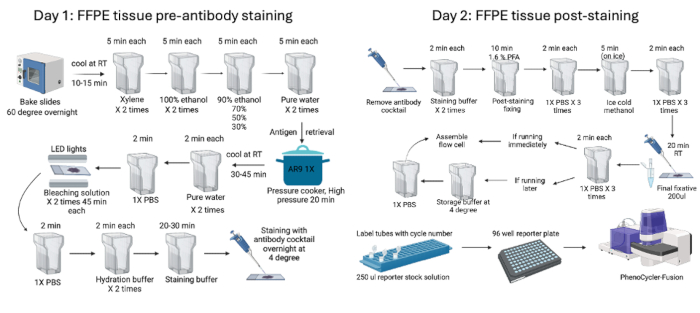
Figure 1 : Résumé du flux de travail de coloration et d’imagerie des tissus FFPE. Les tissus murins FFPE ont été traités à l’aide de procédures de pré-coloration qui ont commencé par la cuisson du tissu pendant la nuit avant de commencer les processus de pré-coloration (jour 1) et de post-coloration (jour 2) de deux jours. Enfin, la plaque rapporteure a été préparée avant l’imagerie avec l’appareil. Veuillez cliquer ici pour voir une version agrandie de cette figure.
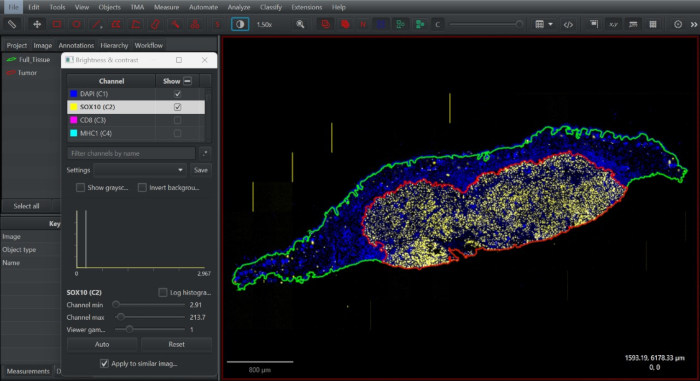
Figure 2 : Capture d’écran d’une annotation tissulaire dans le logiciel de pathologie numérique en libre accès QuPath. L’annotation tissulaire complète a été dessinée (vert), dupliquée et minimisée pour définir le compartiment intratumoral en fonction de la distribution des mélanocytes SOX10+ à la limite de la tumeur (rouge). Veuillez cliquer ici pour voir une version agrandie de cette figure.
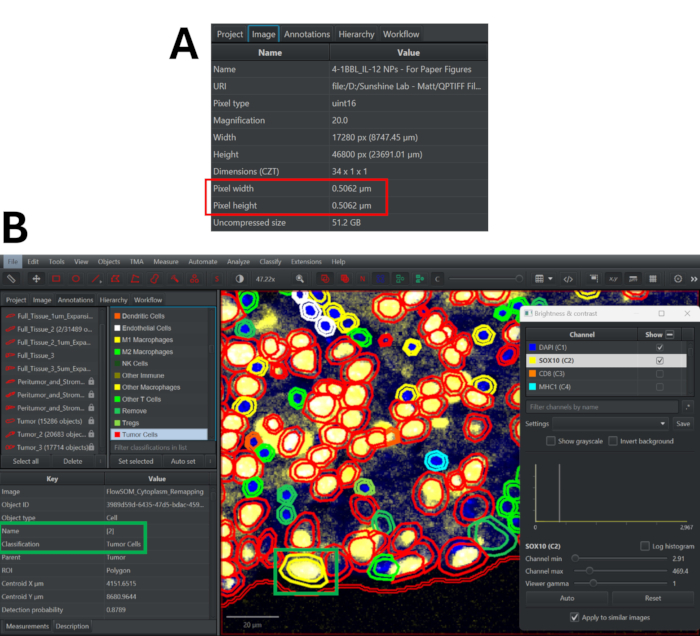
Figure 3 : Segmentation cellulaire à l’aide de l’algorithme StarDist dans QuPath et processus de contrôle qualité pour l’examen des classifications de phénotypes. (A) Accédez à l’onglet Image pour déterminer la largeur et la hauteur des pixels de l’image d’immunofluorescence multiplexe. (B) Après avoir remappé les classifications, double-cliquez sur n’importe quelle cellule (elle est mise en surbrillance en jaune) pour voir son affectation de cluster et son phénotype. Activez/désactivez les marqueurs de panneau pour déterminer si cette approche de classification est exacte. Veuillez cliquer ici pour voir une version agrandie de cette figure.
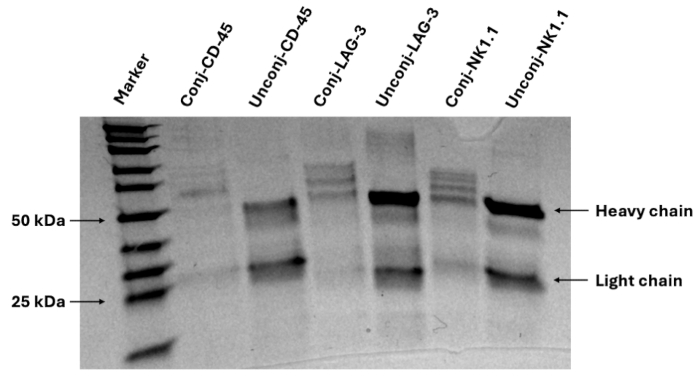
Figure 4 : Image de gel représentative de la confirmation de la conjugaison de codes-barres anticorps-ADN. L’électrophorèse sur gel de protéines confirme la conjugaison des anticorps avec les codes-barres d’oligonucléotides d’ADN, observée par des bandes supplémentaires au site de la chaîne lourde. Veuillez cliquer ici pour voir une version agrandie de cette figure.
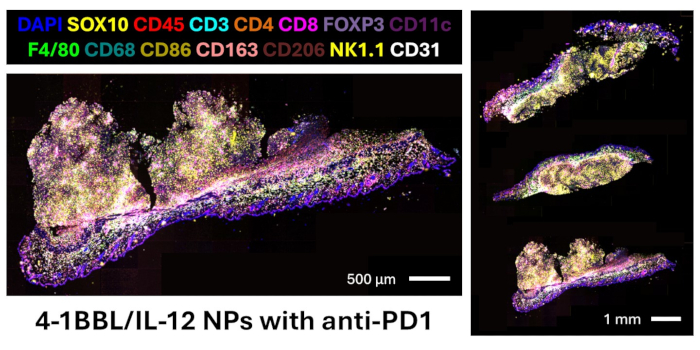
Figure 5 : Imagerie multiplex basée sur un code-barres d’ADN de tumeurs du flanc B16F10 traitées par injections intratumorales de nanoparticules 4-1BBL/IL-12 avec anti-PD1 systémique. Marqueurs de notre panel non représentés :CD11b, CD20, CD38, TIM3, LAG3, T-bet, Eomesodermin, granzyme B, Ki67, LMP2, bêta-2 microglobuline, MHC II,-L1. Veuillez cliquer ici pour voir une version agrandie de cette figure.
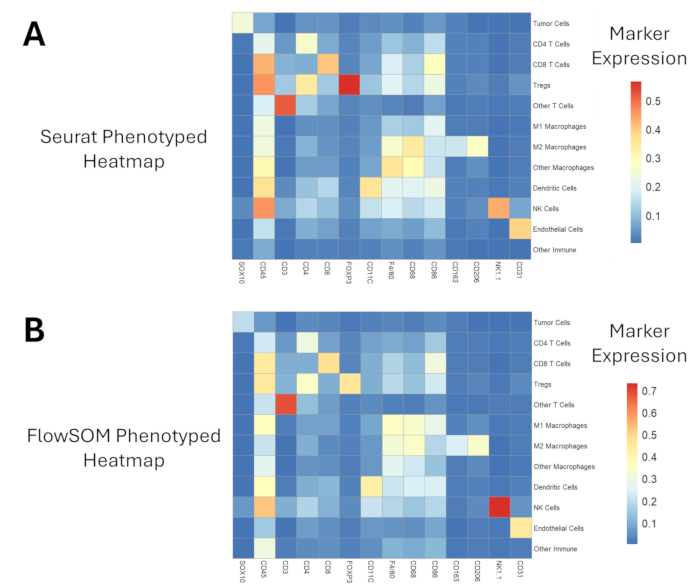
Figure 6 : Cartes thermiques d’expression protéomique de populations cellulaires phénotypées. (A) L’approche de clustering et de phénotypage de Seurat génère une gamme légèrement plus petite de valeurs d’expression de marqueurs par rapport à (B) le clustering et le phénotypage FlowSOM. Veuillez cliquer ici pour voir une version agrandie de cette figure.
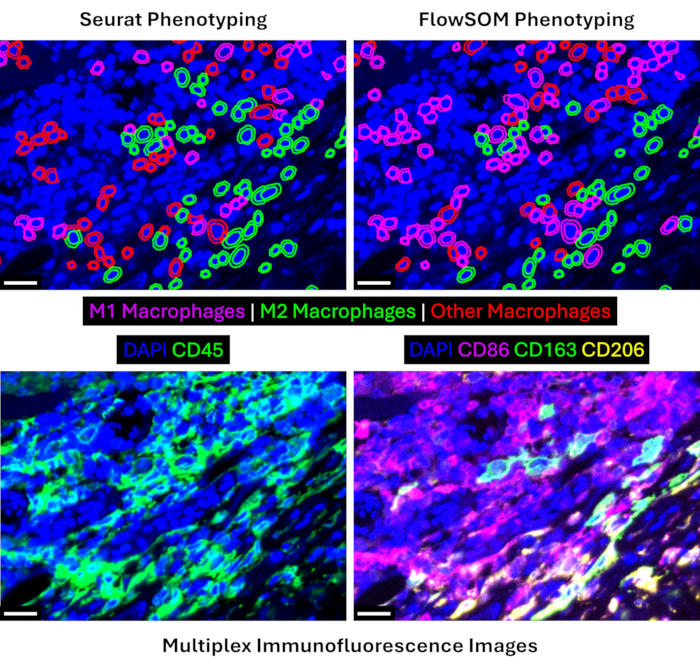
Figure 7 : Le phénotypage FlowSOM identifie un plus grand nombre de sous-types de macrophages différents. Le panneau supérieur montre le phénotypage des macrophages Seurat (à gauche) et FlowSOM (à droite) pour le même champ de vision. Le panneau inférieur montre des images d’immunofluorescence multiplex des principaux marqueurs de la lignée des macrophages dans le même champ de vision. Toutes les barres d’échelle mesurent 20 μm. Veuillez cliquer ici pour voir une version agrandie de cette figure.
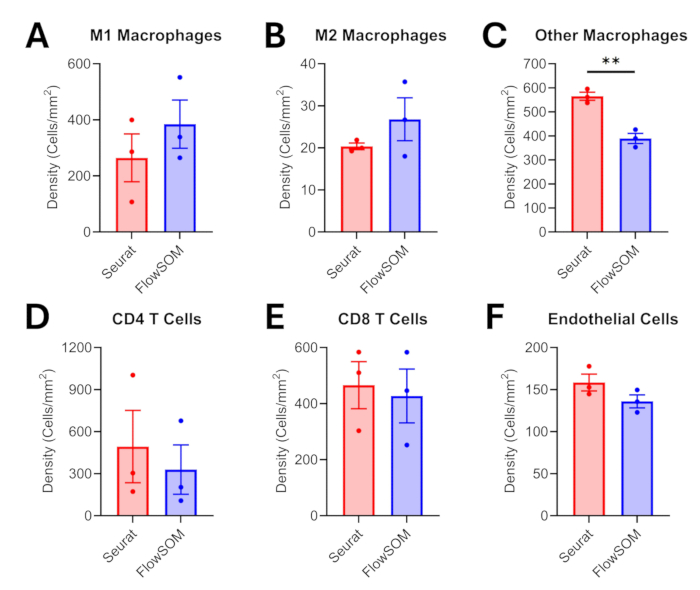
Figure 8 : Comparaison des densités intratumorales de différentes populations cellulaires après regroupement/phénotypage. Des comparaisons de densité sont présentées pour les macrophages intratumoraux (A) M1, (B) les macrophages M2, (C) les autres macrophages, (D) les lymphocytes T CD4, (E) les lymphocytes T CD8 et (F) les cellules endothéliales. Les barres d’erreur sont des erreurs types de la moyenne (MEB), et la signification a été testée à l’aide de tests t non appariés (**p ≤ 0,01). Veuillez cliquer ici pour voir une version agrandie de cette figure.
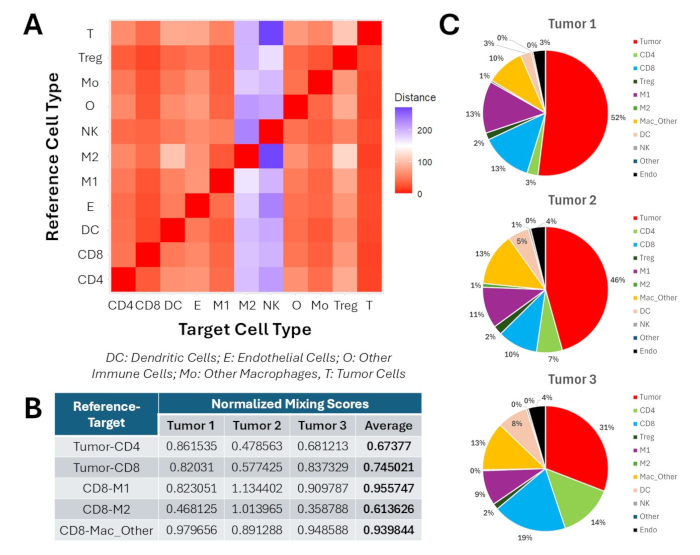
Figure 9 : Profilage des métriques spatiales intratumorales pour trois tumeurs du flanc traitées avec des injections de nanoparticules intratumorales 4-1BBL/IL-12 et un anti-PD1 systémique. (A) Carte thermique des distances minimales moyennes entre les populations phénotypées intratumorales. Les mesures sont exprimées en μm. (B) Scores de mélange normalisés entre les paires de phénotypes intratumoraux clés. (C) Répartition de la composition du voisinage dans un rayon de 100 μm autour des populations de lymphocytes T CD8 intratumoraux. Veuillez cliquer ici pour voir une version agrandie de cette figure.
| Anticorps | Dilution | Temps d’exposition (ms) |
| SOX-10 | 50 | 450 |
| CD8 | 100 | 450 |
| CD3 | 100 | 450 |
| RenardP3 | 50 | 350 |
| CD4 | 50 | 450 |
| CMH-II | 100 | 450 |
| PDL1 | 50 | 450 |
| CD45 | 100 | 450 |
| Ki-67 | 100 | 300 |
| F4/80 | 100 | 150 |
| CD20 | 100 | 450 |
| NK1.1/CD161 | 50 | 450 |
| CD206 | 100 | 450 |
| CD68 | 100 | 450 |
| Granzyme-B | 50 | 450 |
| CD86 | 100 | 150 |
| CD31 | 100 | 450 |
| CD11C | 50 | 450 |
| CD11b | 50 | 450 |
| Émomes | 50 | 450 |
| TIM-3 | 50 | 300 |
| CD38 | 50 | 200 |
| GAL3 | 50 | 450 |
| CD163 (en anglais) | 50 | 200 |
| T-bet | 50 | 300 |
| LMP2 | 100 | 100 |
| Bêta2 MG | 200 | 50 |
Tableau 1 : Réglages de la dilution des anticorps et du temps d’exposition.
Figure supplémentaire 1 : Carte thermique initiale de l’expression protéomique générée après le clustering de Seurat. Veuillez cliquer ici pour télécharger cette figure.
Figure supplémentaire 2 : Carte thermique d’expression protéomique initiale générée après le clustering FlowSOM. Veuillez cliquer ici pour télécharger cette figure.
Discussion
Le succès de l’imagerie dépend d’un panel d’anticorps bien conçu et validé. L’imagerie par immunofluorescence multiplex d’échantillons FFPE présente des défis en raison de l’autofluorescence élevée et de la difficulté de récupérer les épitopes masqués par l’enrobage de paraffine. Cependant, étant donné que le FFPE offre plusieurs avantages par rapport aux échantillons FF, il est essentiel de concevoir et de valider des panels d’anticorps FFPE. La première étape consiste à finaliser les clones d’anticorps qui présentent des signaux positifs lors de l’imagerie par immunofluorescence (IF). par la suite, il est important de les conjuguer soigneusement avec des codes-barres ADN. La conjugaison des anticorps nécessite une réduction partielle de l’anticorps pour créer des liaisons SH, qui sont utilisées lors de la réaction du groupe maléimide avec un code-barres. Tous les clones d’anticorps ne peuvent pas résister à cette étape, et certaines réactions peuvent causer des dommages irréversibles à l’anticorps, entraînant un échec de l’imagerie malgré une conjugaison réussie. Pour cette raison, même si certains anticorps peuvent présenter des signaux positifs lors de la validation conventionnelle de la FI, pour évaluer le succès final de la conjugaison des anticorps, il est important de valider chaque anticorps sur le tissu d’intérêt réel et d’enregistrer les temps d’exposition souhaités pour ce tissu. Dans les applications futures, cette technique pourra être combinée à l’analyse transcriptomique spatiale existante sur des lames consécutives/la même lame pour générer des informations supplémentaires. L’une des limites de cette méthode est qu’elle nécessite une sélection et une validation minutieuses de chaque anticorps du panel en fonction de la cible et du type de tissu.
En ce qui concerne l’analyse d’images, QuPath offre un outil précieux en libre accès avec une visualisation de haute qualité des marqueurs protéomiques, de nombreuses fonctionnalités pour l’exportation des mesures d’intensité et le contrôle de la qualité pour les classifications de phénotypes, et une bonne flexibilité pour les scripts générés par l’utilisateur. Les forums en ligne tels que https://forum.image.sc/ constituent une ressource supplémentaire pour discuter de la façon d’accomplir des tâches d’analyse spécifiques et pour partager des scripts avec d’autres utilisateurs. Dans ce protocole, nous comparons deux approches de clustering et de phénotypage à l’aide de Seurat et FlowSOM. Bien que FlowSOM puisse être préféré pour sa capacité à générer des informations plus granulaires sur les sous-populations de cellules immunitaires du TME, le temps nécessaire à l’analyse protéomique doit également être pris en compte. Générer 100 grappes peut être inutile si un utilisateur n’a besoin de phénotyper que des cellules dans un ou deux échantillons de tissus. Dans ces situations, Seurat peut offrir un pipeline plus rapide et plus efficace pour l’analyse d’images. En revanche, l’analyse d’une TMA avec plus de 40 ou 50 sections de tissus est plus susceptible de produire un plus grand nombre de grappes de cellules dans les deux approches d’analyse, et FlowSOM peut être la méthodologie préférée pour générer des classifications de phénotypes plus nuancées.
Le regroupement et le phénotypage des cellules et toutes les étapes d’analyse d’images ultérieures dépendent en grande partie de la segmentation cellulaire. Nos travaux actuels ont exploré la segmentation cellulaire dans les algorithmes HALO (Indica Labs) et StarDist, et nous avons constaté que les deux approches ont tendance à sursegmenter les cellules en fonction des signaux DAPI nucléaires. De nombreux algorithmes de segmentation alternatifs sont également disponibles, tels que Mesmer19 et InstanSeg20. Il s’agit d’un domaine de recherche informatique en pleine croissance qui nécessite une exploration et une optimisation plus poussées.
Déclarations de divulgation
J.C.S. reconnaît le soutien financier d’Emerson Collective LLC et des National Institutes of Health. J.J.G. reconnaît également le financement des National Institutes of Health. J.C.S. entretient une relation avec Palleon Pharmaceuticals Inc., qui implique des subventions de financement. S.Y.T. et J.J.G. ont une relation avec OncoSwitch Therapeutics qui implique des actions ou des actions. S.Y.T., J.J.G., J.C.S. et K.M.L. ont un brevet en instance. Tous les autres auteurs affirment qu’ils n’ont pas d’intérêts financiers concurrents connus ou de relations personnelles qui pourraient être perçus comme influençant la recherche présentée dans cet article.
Remerciements
J.C.S. remercie la Fondation de dermatologie et le Prix de développement de carrière en dermatopathologie pour l’avancement de la carrière de l’auteur. Les auteurs remercient Hsin-Pei Lee du Cancer Data Science Laboratory du National Cancer Institute pour son aide avec les techniques d’analyse informatique. Cette recherche a reçu un financement de l’Emerson Collective et des National Institutes of Health (R37CA246699, P41EB028239 et R01CA228133). De plus, le noyau des Johns Hopkins Oncology Tissue Services (OTS) est soutenu par les National Institutes of Health (P30CA006973).
matériels
| Name | Company | Catalog Number | Comments |
| 16% paraformaldehyde (PFA) | Electron Microscopy Sciences | PN# 15710 | Required during tissue staining process |
| 50kDa MWCO filter | Millipore | UFC505096 | 25 kDa and 100 kDa result in failure |
| Akoya barcodes and reporters | Akoya Biosciences | Varied | Each barcode can be used to conjugate 50 ug of carrier free antibody and comes with two vials of reporters |
| Antibody conjugation kit | Akoya Biosciences | 7000009 | A conjugation kit is used to create custom barcode-conjugated antibodies. Each kit contains sufficient reagents for ten conjugations. The kit consists of one subkit box stored at 4 °C and one subkit box stored at -20 °C. The 4 °C subkit includes Filter Blocking Solution, Reduction Solution 2, Conjugation Solution, Purification Solution, and Antibody Storage Solution. The -20 °C subkit includes Reduction Solution 1. |
| Beta2MG | Abcam | ab214769 | |
| CD11b | CST | #41028 | |
| CD11C | CST | #39143SF | |
| CD163 | CST | #68922BF | |
| CD20 | CST | #45839 | |
| CD206 | CST | #87887 | |
| CD3 | CST | #24581 | |
| CD31 | CST | #92841 | |
| CD38 | CST | #68336BF | |
| CD4 | Abcam | ab271945 | |
| CD45 | CST | #98819 | |
| CD68 | CST | #29176 | |
| CD8 | Invitrogen | #14-0808-82 | |
| CD86 | CST | #20018 | |
| Dimethyl sulfoxide | Avantor/VWR | BDH1115-4LP | |
| EOMES | Abcam | ab222226 | |
| Eppendorf PCR tubes | Eppendorf | E0030124286 | Required store 5 μL conjugated antibody for conjugation confirmation by gel electrophoresis |
| Ethanol 200 proof | |||
| F4/80 | CST | # 25514 | |
| FoxP3 | CST | #72338 | |
| Gran-B | CST | #79903SF | |
| Heating oven | |||
| Hybridization chamber | Used to stain tissue with antibody cocktail | ||
| Hydrogen peroxide | Sigma | #216763 | Required for preparing bleaching solution |
| Instant pot pressure cooker or Decloaking Chamber ARC | Instant pot or Biocare Medical | ||
| Ki-67 | Akoya Biosciences | ||
| LAG3 | CST | #80282BF | |
| LMP2 | Abcam | ab243556 | |
| Methanol | |||
| MHC-II | eBioscience | #14-5321-82 | |
| NK1.1 | CST | #24395SF | |
| Nuclear Stain | Akoya Biosciences | 7000003 | |
| PDL1 | CST | #85095 | |
| Salmon Sperm DNA, sheared (10 mg/mL) | Invitrogen | AM9680 | Can be used as an alternative to assay reagent (Akoya) during reporter plate preparation step. |
| SOX-10 | Abcam | ab245760 | |
| Staining kit for PhenoCycler-Fusion | Akoya Biosciences | 7000017 | The PhenoCycler-Fusion Sample Kit includes the buffers and reagents necessary for tissue staining using antibodies conjugated with PhenoCycler barcodes, along with flow cells for whole-slide imaging. Each kit is sufficient for 10 PhenoCycler-Fusion experiments. It consists of one subkit stored at 4 °C, another at -20 °C, and a package of 10 flow cells kept at room temperature. The subkit stored at 4 °C contains Hydration Buffer, Staining Buffer, Storage Buffer, N Blocker, and J Blocker. The subkit stored at -20 °C contains G Blocker, S Blocker, and Fixative Reagent. |
| T-bet | CST | #53753 | |
| TIM-3 | CST | #72911 | |
| UltraPure DNase/RNase-free distilled water | Invitrogen | 10977015 | Required to dissolve barcodes during antibody conjugation |
Références
- Schadendorf, D., et al. Melanoma. Lancet. 392 (10151), 971-984 (2018).
- Taube, J. M., et al. Implications of the tumor immune microenvironment for staging and therapeutics. Modern Pathol. 31 (2), 214-234 (2018).
- Marzagalli, M., Ebelt, N. D., Manuel, E. R. Seminars in cancer biology. 59, 236-250 (2019).
- De Visser, K. E., Joyce, J. A. The evolving tumor microenvironment: From cancer initiation to metastatic outgrowth. Cancer cell. 41 (3), 374-403 (2023).
- Balkwill, F. R., Capasso, M., Hagemann, T. The tumor microenvironment at a glance. J Cell Sci. 125 (23), 5591-5596 (2012).
- Phillips, D., et al. Highly multiplexed phenotyping of immunoregulatory proteins in the tumor microenvironment by codex tissue imaging. Front Immunol. 12, 687673(2021).
- Giesen, C., et al. Highly multiplexed imaging of tumor tissues with subcellular resolution by mass cytometry. Nat Methods. 11 (4), 417-422 (2014).
- Keren, L., et al. Mibi-tof: A multiplexed imaging platform relates cellular phenotypes and tissue structure. Sci Adv. 5 (10), eaax5851(2019).
- Lin, J. R., Fallahi-Sichani, M., Sorger, P. K. Highly multiplexed imaging of single cells using a high-throughput cyclic immunofluorescence method. Nat Comm. 6 (1), 8390(2015).
- Radtke, A. J., et al. Ibex: An iterative immunolabeling and chemical bleaching method for high-content imaging of diverse tissues. Nat Protoc. 17 (2), 378-401 (2022).
- Black, S., et al. Codex multiplexed tissue imaging with DNA-conjugated antibodies. Nat Protoc. 16 (8), 3802-3835 (2021).
- Aung, T. N., Bates, K. M., Rimm, D. L. High-plex assessment of biomarkers in tumors. Mod Pathol. 37 (3), 100425(2024).
- Surwase, S. S., et al. Highly-multiplexed immunofluorescence phenocycler panel for murine ffpe yields insight into tumor microenvironment immunoengineering. Lab Invest. 105 (1), 102165(2024).
- Luly, K. M., Green, J. J., Sunshine, J. C., Tzeng, S. Y. Biomaterial-mediated genetic reprogramming of merkel cell carcinoma and melanoma leads to targeted cancer cell killing in vitro and in vivo. ACS Biomater Sci Eng. 9 (11), 6438-6450 (2023).
- Satija, R., Farrell, J. A., Gennert, D., Schier, A. F., Regev, A. Spatial reconstruction of single-cell gene expression data. Nat Biotechnol. 33 (5), 495-502 (2015).
- Van Gassen, S., et al. Flowsom: Using self-organizing maps for visualization and interpretation of cytometry data. Cytometry Part A. 87 (7), 636-645 (2015).
- Butler, A., Hoffman, P., Smibert, P., Papalexi, E., Satija, R. Integrating single-cell transcriptomic data across different conditions, technologies, and species. Nat Biotechnol. 36 (5), 411-420 (2018).
- Watson, S. S., et al. Microenvironmental reorganization in brain tumors following radiotherapy and recurrence revealed by hyperplexed immunofluorescence imaging. Nat Comm. 15 (1), 3226(2024).
- Greenwald, N. F., et al. Whole-cell segmentation of tissue images with human-level performance using large-scale data annotation and deep learning. Nat Biotechnol. 40 (4), 555-565 (2022).
- Goldsborough, T., et al. Instanseg: An embedding-based instance segmentation algorithm optimized for accurate, efficient and portable cell segmentation. arXiv. , (2024).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationExplorer plus d’articles
This article has been published
Video Coming Soon