Method Article
Imaging multiplex a immunofluorescenza basato su codice a barre del DNA per analizzare campioni FFPE da melanoma murino geneticamente riprogrammato
* Questi autori hanno contribuito in egual misura
In questo articolo
Riepilogo
Questo protocollo fornisce una guida passo passo per la progettazione di un pannello di anticorpi in immunofluorescenza multiplex per l'imaging basato su codici a barre del DNA di tessuti murini di melanoma FFPE. Descriviamo anche una pipeline di analisi delle immagini utilizzando strumenti open-source per generare approfondimenti di proteomica spaziale nel microambiente immunitario del tumore del melanoma murino.
Abstract
Qui viene presentata una tecnica emergente di imaging multiplex basata su codici a barre del DNA basata sul Co-Detection-by-indEXing che analizza la proteomica spaziale dei microambienti tissutali. Il successo dell'imaging richiede un repertorio di pannelli anticorpali ben progettati e adeguatamente convalidati, ma attualmente ne esistono pochissimi per i campioni fissati in formalina e inclusi in paraffina (FFPE). L'FFPE offre diversi vantaggi rispetto ai campioni freschi congelati, come l'ampia disponibilità, la facilità di manipolazione e conservazione e la capacità di realizzare microarray di tessuti (TMA). Qui, presentiamo un protocollo per sviluppare un pannello di anticorpi per la visualizzazione e l'analisi di tessuti FFPE da un modello di melanoma murino trattato con nanoparticelle, che forniscono DNA plasmidico codificante segnali immunologici per la riprogrammazione del microambiente tumorale. Descriviamo anche una pipeline di analisi delle immagini utilizzando strumenti computazionali open source per l'annotazione dei tessuti, la segmentazione delle cellule, l'elaborazione dei dati proteomici, la fenotipizzazione delle popolazioni cellulari e la quantificazione delle metriche spaziali. Il protocollo offre applicazioni per la progettazione di pannelli anticorpali in FFPE murino e per la generazione di nuove intuizioni sulla proteomica spaziale di microambienti tissutali complessi.
Introduzione
Il melanoma cutaneo è il tumore della pelle più comune, con tassi di malattia e mortalità variabili in tutto il mondo a seconda del momento della diagnosi e delle cure primarie1. Nell'ultimo decennio, una maggiore comprensione biologica del melanoma ha contribuito a promuovere lo sviluppo di nuovi modelli di cancro per il trattamento dei tumori solidi2. La recente ascesa dell'immunoterapia ha portato a un concetto rivoluzionario di trattamento del cancro basato sull'attivazione del sistema immunitario endogeno 3,4.
Il microambiente tumorale (TME) è molto complesso e consiste di diverse cellule immunitarie, fibroblasti associati al cancro, periciti, cellule endoteliali e varie cellule residenti nei tessuti5. In passato sono state applicate diverse tecniche per studiare il TME, come la citometria a flusso e il sequenziamento di singole cellule, che compromettono il contesto spaziale in quanto sono necessarie per distruggere il tessuto tumorale. L'imaging al microscopio tradizionale, come l'immunofluorescenza (IF) e l'immunoistochimica (IHC), consente la visualizzazione di biomarcatori proteici senza distruggere i tessuti del campione. Tuttavia, questi approcci sono limitati a due o tre biomarcatori e non sono in grado di fornire una comprensione completa delle relazioni spaziali e strutturali all'interno del complesso TME 6.
Per affrontare questo problema, sono state sviluppate diverse tecniche di imaging multiplex per visualizzare spazialmente il complesso TME 7,8,9,10. Uno di questi è il CoDetection-by-inDEXing, rinominato sistema PhenoCycler, basato su anticorpi coniugati con oligonucleotidi di DNA11. Il sistema è in grado di fornire l'imaging e l'analisi di una singola cellula di oltre 100 biomarcatori per campioni umani. Tuttavia, sono disponibili pochissimi anticorpi inventariati per visualizzare e analizzare campioni murini, in particolare campioni di FFPE (Formalin-Fixed Paraffin-Embedded)12. L'FFPE offre diversi vantaggi rispetto alla conservazione Fresh Frozen (FF), come la facilità di manipolazione e conservazione, la morfologia ben conservata nel tempo e, soprattutto, la capacità di preparare microarray di tessuti/tumori (TMA) che consentono la visualizzazione di diversi campioni su un singolo vetrino. Di recente abbiamo progettato e sviluppato un pannello di anticorpi murini FFPE CODEX/PhenoCycler e lo abbiamo applicato con successo per visualizzare e analizzare la proteomica spaziale di campioni di melanoma murino geneticamente riprogrammati13.
L'obiettivo generale di questo protocollo è fornire una guida passo passo per la progettazione di un pannello di anticorpi FFPE murini e descrivere il processo di coniugazione anticorpo-codice a barre, colorazione dei tessuti e imaging. Inoltre, presentiamo una pipeline di analisi delle immagini dettagliata che utilizza strumenti open source come i pacchetti QuPath e R. Dopo aver seguito questo protocollo, i ricercatori impareranno come progettare un pannello di anticorpi coniugati su misura, eseguire l'imaging multiplex utilizzando un dispositivo Phenocycler-Fusion e ottenere nuove informazioni sulla proteomica spaziale del melanoma TME. Inoltre, questo protocollo può essere adattato per studiare vari microambienti immunitari tumorali e combinato con le tecniche di trascrittomica spaziale esistenti.
Protocollo
Tutto il lavoro sugli animali è stato eseguito in conformità con le linee guida stabilite dal Johns Hopkins Animal Care and Use Committee, utilizzando i numeri di protocollo approvati MO18M388 e MO21M384.
1. Selezione degli anticorpi
- Progettare un pannello di anticorpi basato sul tessuto di interesse e sull'abbondanza del particolare biomarcatore. Scegliere i cloni di anticorpi in base alle precedenti applicazioni di successo della colorazione in immunofluorescenza (IF) e immunoistochimica (IHC) presenti in letteratura. Versioni sicure di questi anticorpi prive di portatore.
NOTA: Un anticorpo purificato privo di vettore è importante per la coniugazione del codice a barre. Evita i conservanti a base di proteine come BSA, glutine, glicerolo, ecc. Se l'anticorpo senza vettore non è disponibile, richiedere un anticorpo personalizzato al fornitore o utilizzare un kit di rimozione BSA per produrre l'anticorpo senza portatore. L'azide di sodio non interferisce con la coniugazione degli anticorpi. Gli isotipi IgG sono raccomandati rispetto agli isotipi IgM. - Verificare questi cloni selezionati sul tessuto di interesse utilizzando l'imaging a immunofluorescenza convenzionale (IF)11. Utilizzare l'imaging a doppia IF per confermare la specificità di alcuni cloni (ad esempio, eseguire l'imaging a doppia IF con FOXP3 e CD4 per confermare la colorazione per le cellule T regolatorie).
NOTA: Quando possibile, è importante utilizzare lo stesso tessuto per la convalida IF che verrà utilizzato per la colorazione. Selezionare la soluzione appropriata per il recupero dell'antigene. In questo protocollo, abbiamo utilizzato un tampone AR9 fornito da Akoya Biosciences per recuperare tutti gli epitopi tissutali FFPE del melanoma murino. Altre soluzioni per il recupero dell'antigene, come AR6 o Universal, possono essere utilizzate in base ai requisiti dell'epitopo.
2. Coniugazione e conferma degli anticorpi
- Assegna anticorpi verificati ai codici a barre con reporter complementari collegati ai fluorofori ATTO550 (Cy3), AF647 (Cy5) o AF750 (Cy7). Gli antigeni poco abbondanti di solito producono segnali più bassi; coniugare tali anticorpi a canali a bassa autofluorescenza come AF647 (Cy5). Coniugare antigeni altamente espressi a ATTO550 (Cy3) e AF750 (Cy7) a causa della possibilità di autofluorescenza.
NOTA: Questo passaggio critico considera la sensibilità del canale e l'abbondanza dell'antigene. L'uso del canale AF488 per il tessuto FFPE non è raccomandato a causa della sua maggiore autofluorescenza. - Per la coniugazione anticorpo-codice a barre (4,5 ore), procurarsi il kit di coniugazione anticorpale. Conservare tutti i reagenti a 4 °C tranne la soluzione di riduzione 1, che deve essere conservata a -20 °C in piccoli flaconcini monouso.
- Misurare la concentrazione di anticorpi utilizzando uno spettrofotometro.
- Avviare la coniugazione degli anticorpi applicando 500 μl della soluzione di blocco del filtro a una colonna filtrante MWCO da 50 kDa per bloccare il legame non specifico degli anticorpi al filtro. Centrifugare a 12.000 x g per 2 minuti a temperatura ambiente (RT). Rimuovere il liquido in eccesso dalla colonna utilizzando una micropipetta da 200 μl.
NOTA: La soluzione di riduzione 1 è un flaconcino di soluzione monouso, sufficiente per tre coniugazioni di anticorpi alla volta. Non riutilizzare la soluzione dopo lo scongelamento; Eliminare il reagente rimanente. Non utilizzare un filtro sopra la colonna filtrante MWCO da 50 kDa, poiché ciò può comportare una scarsa purificazione e coniugazione. - Utilizzare un volume equivalente a 50 μg della soluzione anticorpale e aggiungerlo a un filtro MWCO da 50 kDa. Se necessario, regolare il volume totale a 100 μl utilizzando PBS. Centrifugare a 12.000 x g per 8 minuti a 4 °C. Durante questo periodo, preparare la miscela master di riduzione utilizzando 19,8 μl di soluzione di riduzione 1 + 825 μl di soluzione di riduzione 2 per tre coniugazioni di anticorpi.
- Scartare il flusso continuo e aggiungere 260 μl di miscela master di riduzione sulla parte superiore di ciascuna unità filtrante. Agitare la soluzione nell'unità filtrante per 2-3 secondi e incubare per 30 minuti a RT.
NOTA: È fondamentale non superare i 30 minuti di incubazione per evitare un'eccessiva riduzione degli anticorpi, poiché ciò li danneggia e può causare una coniugazione fallita. - Dopo 30 minuti di incubazione, centrifugare a 12.000 x g per 8 minuti a 4 °C e gettare il flusso sul fondo. Aggiungere 450 μl di soluzione di coniugazione alla sommità della colonna e centrifugare a 12.000 x g per 8 minuti a 4 °C. Durante la centrifugazione, preparare la soluzione di codice a barre CODEX. Muoviti rapidamente dopo aver recuperato il codice a barre da -20 °C, poiché il codice a barre inizia a degradarsi.
NOTA: I codici a barre sono solitamente forniti in piccole fiale di vetro contenenti prodotti liofilizzati (scaglie o polvere) conservati a -20 °C. Queste fiale possono essere utilizzate per coniugare 50 μg di anticorpi in una sola volta. Si raccomanda di non coniugare più di tre anticorpi alla volta. - Individuare con cura il codice a barre liofilizzato nella parte inferiore della fiala di vetro (potrebbe anche essere attaccato alle pareti). Battere la fiala di vetro su un tavolo per portare i solidi sul fondo. L'individuazione del prodotto liofilizzato è essenziale per la sua dissoluzione in 10 μL di acqua di biologia molecolare priva di nucleasi. Dopo aver aggiunto 10 μl di acqua priva di nucleasi, aggiungere 210 μl di soluzione di coniugazione a ciascun codice a barre. Sciogliere tutto il materiale liofilizzato e mescolare delicatamente pipettando su e giù. Accantonare.
- Dopo aver completato la centrifuga al punto 2.2.5, scartare il flusso e aggiungere la rispettiva soluzione di codice a barre dal passaggio 2.2.6 alla parte superiore di ciascuna unità filtrante. Conservare 1 μg dell'anticorpo iniziale non coniugato; Questo verrà eseguito insieme al campione di anticorpi coniugati da 5 μL durante la convalida dell'elettroforesi su gel.
- Etichettare ogni provetta con il rispettivo nome dell'anticorpo e del codice a barre. Chiudere il coperchio e mescolare la soluzione agitando per 2-3 s. Incubare la reazione di coniugazione anticorpo-codice a barre per 2 ore a RT.
- Dopo 2 ore di incubazione, rimuovere 5 μL di anticorpo coniugato e conservarlo in una provetta PCR da 0,2 mL per confermare la coniugazione mediante elettroforesi su gel.
- Centrifugare la soluzione di anticorpi coniugati rimanente a 12.000 x g per 8 minuti a 4°C. Aggiungere 450 μl di soluzione di purificazione sulla parte superiore della colonna, centrifugare a 12.000 x g per 8 minuti a 4 °C ed eliminare il flusso. Ripetere questo passaggio di purificazione 2 volte, aggiungendo ogni volta 450 μl di soluzione di purificazione e facendola girare verso il basso.
- Dopo la 3acentrifugazione , scartare il flusso attraverso. Il filtro superiore conterrà gli anticorpi coniugati con codice a barre.
- Per raccogliere l'anticorpo coniugato, etichettare il nuovo tubo esterno che contiene la colonna filtrante con l'anticorpo e il codice a barre corrispondenti. Tagliare il coperchio del tubo esterno prima della centrifugazione. Aggiungere 100 μl di soluzione di conservazione degli anticorpi a ciascuna unità filtrante e posizionare il tubo esterno appena etichettato capovolto sopra la colonna filtrante.
- Capovolgere la colonna filtrante per raccogliere l'anticorpo coniugato nel tubo esterno e ruotare verso il basso a 3.000 x g per 2 minuti a RT. Questo dovrebbe raccogliere circa 120 μL di soluzione di anticorpi coniugati. Trasferire in una provetta sterile per microcentrifuga e conservare a 4 °C per 18-24 mesi.
NOTA: Per evitare un'elevata colorazione nucleare di fondo dopo la coniugazione, si raccomanda di non utilizzare questi anticorpi per almeno 2 giorni.
- Eseguire la conferma della coniugazione mediante elettroforesi su gel come descritto nel protocollo13 già pubblicato.
NOTA: Questo processo conferma solo il successo della reazione di coniugazione chimica utilizzata per coniugare i codici a barre con gli anticorpi. È facoltativo, poiché la convalida degli anticorpi è possibile solo dopo la colorazione e l'imaging del tessuto.
3. Preparazione del campione FFPE murino
- Ottenere campioni di tessuto FFPE da topi femmina C57BL/6J impiantati con tumori del fianco B16F10. Trattare i topi con iniezioni intratumorali di nanoparticelle a base di poli(β-amminoestere) contenenti plasmidi 4-1BBL e IL-12, insieme alla somministrazione intraperitoneale di anti-PD1 a 9, 11, 16 e 18 giorni dopo l'impianto14. Sacrificare i topi il giorno 20 e fissare i tumori in formalina al 10%, incorporata in paraffina, quindi sezionare il nucleo dei servizi tissutali oncologici della Johns Hopkins.
- Considerare attentamente le specifiche dell'area di imaging fornite nel manuale dell'utente e montare campioni da 5 μm su un vetrino all'interno dell'area di imaging. Siamo stati in grado di montare quattro diversi campioni di tumore su un unico vetrino.
NOTA: È essenziale utilizzare i vetrini consigliati disponibili per l'imaging, come i vetrini adesivi Leica Apex o i vetrini Fisherbrand Superfrost Plus. Questo aiuterà la cella di flusso ad aderire saldamente al vetrino.
4. Colorazione e imaging dei tessuti FFPE
- Verifica e ottimizzazione della concentrazione anticorpale e dei tempi di esposizione: Per verificare il successo della coniugazione anticorpo-codice a barre, colorare il tessuto di interesse con 6-9 anticorpi coniugati. Eseguire il dispositivo di imaging multiplex e annotare ogni concentrazione di anticorpi e il tempo di esposizione. Regolare le diluizioni e i tempi di esposizione secondo necessità, quindi eseguire 6-9 nuovi anticorpi con quelli precedentemente verificati e annotare eventuali regolazioni. Seguire i passaggi seguenti per la colorazione e l'imaging.
- Colorazione e imaging dei tessuti
- Il giorno prima della colorazione, cuocere i vetrini di tessuto FFPE in forno a 60 °C per una notte. Il giorno successivo, raffreddare i vetrini per 10 minuti a RT prima di iniziare le fasi di deparaffinazione e reidratazione dei tessuti.
- Iniziare la deparaffinizzazione del tessuto incubando in una soluzione di xilene 2 volte per 5 minuti ciascuna.
- Per reidratare il tessuto, farlo passare attraverso due cicli di soluzione di etanolo al 100% per 5 minuti ciascuno. Quindi, passalo attraverso una serie di soluzioni alcoliche dal 90% al 70%, 50% e 30% per 5 minuti ciascuna. Infine, lavare il fazzoletto con acqua distillata due volte per 5 minuti ciascuno.
NOTA: Per le fasi di deparaffinazione possono essere utilizzate alternative meno tossiche allo xilene, come una soluzione Neoclear. Eseguire la deparaffinazione e la reidratazione sotto una cappa aspirante. - Diluire 10 tampone AR9 a 1 x utilizzando acqua distillata. Riempire un barattolo Coplin con 50 ml di 1 tampone AR9, assicurandosi che i vetrini siano completamente immersi nel tampone di recupero dell'antigene.
- Riempi una pentola a pressione con acqua in modo che il livello sia a metà del barattolo Coplin, quindi incuba ad alta pressione per 20 minuti.
NOTA: È possibile utilizzare una pentola a pressione specializzata o una camera di decloaking per un migliore recupero dell'antigene impostandola a 120 °C per 10 min. - Dopo aver completato la fase della pentola a pressione, raffreddare i vetrini per 45-60 minuti. Sciacquare il vetrino con acqua distillata 2 volte per 2 minuti ciascuno. Quindi, trasferisci il vetrino nel barattolo contenente 1x PBS.
- Per ridurre al minimo l'autofluorescenza, eseguire una fase di sbiancamento sul tessuto prima di colorare con gli anticorpi. Preparare una soluzione sbiancante (25 mL di 1x PBS + 0,8 mL di 1M NaOH + 4,5 mL di H2O2) utilizzando i reagenti e le concentrazioni raccomandate. Incubare i vetrini in un contenitore di plastica contenente la soluzione sbiancante tra due lampade a LED per 45 minuti a temperatura ambiente. Ripetere con soluzione decolorante fresca per altri 45 minuti.
- Iniziare a preparare la soluzione cocktail di anticorpi durante questa fase di sbiancamento, soprattutto se nel pannello sono presenti diversi anticorpi con varie diluizioni. Recupera tutti e quattro i bloccanti (G, S, J e N) e mettili sul ghiaccio, poiché alcuni impiegano tempo a scongelarsi da -20 °C.
- Dopo il fotosbiancamento, lavare il fazzoletto con 1x PBS, 2x per 2 minuti ciascuno. Spostare il fazzoletto in barattoli contenenti tampone di idratazione e lavare 2 volte per 2 minuti ciascuno.
- Trasferire i vetrini di tessuto nel tampone di colorazione e lasciarli equilibrare per 30 minuti. Se non preparato durante la fase di sbiancamento, preparare la soluzione di cocktail di anticorpi durante questo periodo.
- Preparare 300 μL di soluzione di cocktail di anticorpi per colorare due vetrini (di solito, se si utilizza una camera di plastica di ibridazione, 100-120 μL di cocktail di anticorpi sono sufficienti per colorare ogni vetrino).
- Colorare i vetrini applicando una soluzione cocktail di anticorpi all'interno della camera di umidità per una notte a 4 °C.
NOTA: Tutti gli anticorpi qui mostrano un segnale positivo quando vengono colorati durante la notte a 4 °C. Tuttavia, il tempo di colorazione e la temperatura di ciascun anticorpo possono variare e devono essere ottimizzati. Le condizioni di colorazione più comunemente utilizzate sono 3 ore a RT o durante la notte a 4 °C. Se necessario, è anche possibile colorare alcuni anticorpi inizialmente a temperature diverse e poi gli anticorpi rimanenti durante la notte a 4 °C. Lavare successivamente il fazzoletto con PBS se è necessaria una colorazione sequenziale. - Il giorno successivo, eseguire la fissazione post-colorazione. Rimuovere la camera di plastica e conservare il cocktail di anticorpi raccolto a 4 °C. Lavare i vetrini 2 volte nel tampone di colorazione per 2 minuti ciascuno. Successivamente, spostare i vetrini su una soluzione di fissaggio post-colorazione da 40 mL (4 mL di Paraformaldeide al 16% + 36 mL di tampone di conservazione) e incubare per 10 min. Risciacquare con PBS 3 volte per 2 minuti ciascuno.
- Immergere i vetrini nel metanolo ghiacciato per 5 minuti, quindi risciacquare con PBS 3 volte per 2 minuti ciascuno. Applicare una soluzione fissativa finale (20 μL, una provetta + 1 mL di PBS) sui vetrini sotto una camera di umidità. Di solito, 200 μL di soluzione fissativa sono sufficienti per vetrino. Lasciare riposare il fissativo per 20 minuti a RT. Eseguire tre lavaggi finali in PBS per 2 minuti ciascuno.
NOTA: Non rimuovere preventivamente il tubo di fissaggio da -20 °C; Scongelare solo prima dell'applicazione. - In caso di imaging immediato, pulire il vetrino attorno al tessuto utilizzando un panno privo di lanugine e applicare la cella di flusso premendola per 30 s utilizzando l'attrezzatura di assemblaggio della cella di flusso. In caso di imaging successivo, conservare il vetrino (senza applicare la cella di flusso) nel buffer di conservazione a 4 °C. Quando si è pronti per l'imaging, trasferire i vetrini dal buffer di archiviazione al PBS per 10 minuti prima di applicare la cella di flusso.
- Inizia a preparare il buffer in esecuzione 1x con l'additivo in base ai cicli desiderati nell'esperimento. Preparare buffer DMSO bassi (1:4) e alti (9:1) mescolandoli come DMSO a 1 parte con buffer di funzionamento a 4 parti e DMSO a 9 parti con buffer di funzionamento a 1 parte necessari per i cicli desiderati. Risparmia circa 20 ml di tampone 1x con additivo per realizzare una piastra reporter. Per calcolare il buffer 1x richiesto con buffer additivi e buffer DMSO alto/basso, consultare il gestore degli strumenti.
- Inizia a preparare la piastra reporter. Innanzitutto, crea la soluzione stock reporter richiesta per il numero totale di cicli. Etichettare le provette per microcentrifuga da 1 ml di colore nero o ambrato con i nomi dei cicli. Aggiungere 250 μl di soluzione madre reporter e 5 μl di ciascun reporter alla provetta corrispondente. Quindi, trasferire la soluzione in una piastra nera a 96 pozzetti e sigillarla con un foglio adesivo.
- Avviare il dispositivo, utilizzare il gestore degli strumenti per impostare i parametri e i tempi di esposizione (come da Tabella 1) e acquisire le immagini. La Figura 1 riassume i processi pre e post colorazione.
5. Annotazione tissutale e segmentazione cellulare
- Configura un progetto e un'area di lavoro.
- Installa l'ultima versione del software di analisi patologica digitale (ad esempio, QuPath versione 0.5.1 o successiva). Fare clic su Crea progetto per scegliere una cartella di destinazione per lo spazio del progetto.
- Fare clic su Aggiungi immagini > Scegli file, quindi accedere al file QPTIFF (prodotto dall'imaging a immunofluorescenza multiplex). Impostate il tipo di immagine su Fluorescenza, mantenete tutte le altre impostazioni predefinite e fate clic su Importa.
- Se si utilizza QuPath, fare doppio clic sulla nuova immagine per aprire un'area di lavoro. Fare clic su File > Salva periodicamente per tenere traccia delle modifiche apportate al progetto. Attiva/disattiva le impostazioni di visibilità e visualizzazione dei marcatori utilizzando Luminosità e contrasto (icona a forma di mezzaluna) vicino al centro della barra degli strumenti.
NOTA: Fare clic con il pulsante destro del mouse su un'immagine nell'elenco delle immagini per rinominarla. Questo sarà importante in seguito quando si visualizzeranno le classificazioni dei fenotipi.
- Annotare l'intera sezione di tessuto, il compartimento intratumorale e il compartimento stromale.
- Utilizzare lo strumento pennello e/o bacchetta per disegnare un'annotazione attorno all'intera sezione di tessuto. Definire questa annotazione come Full_Tissue. Escludere la pelle sovrastante e/o le regioni che non devono essere analizzate. Per creare un'annotazione negativa e/o ridurre il limite dell'annotazione, tenere premuto il tasto Alt mentre si utilizza uno degli strumenti di annotazione.
- Duplicare l'annotazione completa del tessuto. Seleziona l'annotazione nella scheda Annotazioni, quindi vai su Oggetti > Annotazioni... > Duplica le annotazioni selezionate.
- Attiva il canale SOX10. Sull'annotazione duplicata, ridurre il limite dell'annotazione per acquisire il compartimento intratumorale utilizzando lo strumento Alt + pennello/bacchetta. Definisci questa annotazione come un tumore (Figura 2).
- Selezionando l'annotazione completa del tessuto, vai su Oggetti > Annotazioni... > Espandi le annotazioni. Definite il raggio di espansione come 1 μm e fate clic su Esegui (Run).
- Rinominare la nuova annotazione come Full_Tissue_Expansion. Selezionare l'annotazione del tumore, fare clic con il pulsante destro del mouse e selezionare Inserisci nella gerarchia.
- Riseleziona l'annotazione del tumore, vai su Oggetti > Annotazioni... > Fai l'inverso. Definire questa nuova annotazione come Stroma. Ripetere per tutte le sezioni di tessuto.
- Esegui la segmentazione delle celle ed esporta i risultati.
- Vai alla scheda Immagine per trovare la larghezza e l'altezza in pixel dell'immagine (Figura 3A).
- Scarica l'estensione StarDist su https://github.com/qupath/qupath-extension-stardist/releases. Trascina il file qupath-extension-stardist-[version].jar nella finestra di QuPath, quindi fai clic sull'icona a forma di ingranaggio nell'angolo in alto a destra della finestra di QuPath. In Estensioni, definire la directory utente QuPath come percorso della cartella StarDist.
- Scaricare i file di script groovy di StarDist e il file del modello di StarDist da https://github.com/xzhou125/JOVE_QuPath_Spatial_Analysis (gli script di StarDist sono stati originariamente organizzati da Akoya Biosciences).
- Per ogni sezione di tessuto, selezionare entrambe le annotazioni Tumore e Stroma (utilizzare Ctrl per selezionare più annotazioni). Nella barra delle impostazioni in alto, fai clic su Automatizza >editor di script per aprire l'interfaccia degli script. Aprire lo script di segmentazione delle celle StarDist appropriato (file groovy) che corrisponde alla dimensione in pixel dell'immagine. Quando viene richiesto di selezionare il file di segmentazione delle celle, aprire il file stardist_cell_seg_model.pb.
- Dopo la segmentazione delle celle, salvare l'immagine. Vai su Misura > Esporta misurazioni, quindi seleziona l'immagine o le immagini corrispondenti. Assegna il tipo di esportazione come Celle e Separatore come .csv, quindi scegli la posizione del file di output . Fai clic su Compila accanto a Colonne per includere le metriche di interesse. Le colonne importanti da esportare sono Immagine, ID oggetto, Padre, Centroide X, Centroide Y e Area del nucleo.
NOTA: L'ID oggetto viene utilizzato nei passaggi successivi per rimappare le classificazioni all'immagine di immunofluorescenza multiplex nel software di analisi patologica digitale. La colonna Genitore indica il nome dell'annotazione a cui appartiene la cella. I dati X e Y vengono utilizzati per l'analisi spaziale. - Per ogni marcatore di derivazione da utilizzare nel clustering/fenotipizzazione, selezionare un valore medio da esportare. Per i marcatori nucleari (ad esempio, SOX10 e FOXP3), esportare l'opzione Nucleus: Media. Per i marcatori citoplasmatici/membranosi (ad es. CD45, CD3, CD4), esportare l'opzione Citoplasma: Media.
6. Pre-elaborazione e normalizzazione dei dati in proteomica
- Apri il file CSV esportato. Per ogni marcatore da utilizzare nel clustering/fenotipizzazione, troncare il nome dell'intestazione della colonna per includere solo il marcatore. Ad esempio, rinominare SOX10: Nucleus: Mean in SOX10 e CD3: Cytoplasma: Mean to CD3.
- Installare la versione più recente di R e RStudio. Scaricare il file Marker Normalization.R da https://github.com/xzhou125/JOVE_QuPath_Spatial_Analysis ed eseguire lo script. Questo comporta innanzitutto il filtraggio delle cellule in base alle dimensioni nucleari.
- Per ogni marcatore, viene quindi eseguita una normalizzazione min-max in modo che il valore MFI più basso nell'intervallo di espressione sia impostato su 0 e il valore MFI al 99,7° percentile sia impostato su 1. Tutte le intensità superiori al 99,7° percentile vengono tagliate a 1. Una volta completato, salva il nuovo file CSV generato con i dati normalizzati.
7. Clustering e fenotipizzazione
- Scegliere un algoritmo per il clustering delle celle in base ai profili di espressione dei marcatori. Al https://github.com/xzhou125/JOVE_QuPath_Spatial_Analysis, vengono forniti script R per il clustering utilizzando Seurat v4.415 e FlowSOM v2.13.9 (strumento di clustering e visualizzazione)16.
- Fenotipo delle cellule in base ai profili di espressione dei marcatori chiave del lignaggio. Il protocollo qui presentato ha utilizzato 14 marcatori di linea per il clustering e la fenotipizzazione: SOX10, CD45, CD3, CD4, CD8, FOXP3, CD11C, F4/80, CD68, CD86, CD163, CD206, NK1.1 e CD31.
- Definisci le cellule tumorali come SOX10hi. Definisci le cellule T CD4 come CD45mod-hi/ CD4hi/CD8low/FOXP3low. Definisci le cellule T CD8 come CD45mod-hi/ CD8hi/CD4low. Definisci le Treg come CD45mod-hi/CD4hi/CD8low/FOXP3hi. Definisci le altre cellule T come CD45mod-hi/CD3hi/CD4low/CD8low.
- Definisci i macrofagi M1 come CD45mod-hi/F4/80hi/CD68hi/CD86hi/CD163low /CD206low. Definisci i macrofagi M2 come CD45mod-hi/F4/80hi/CD68mod-hi/CD86low/CD163hi/CD206low, CD45mod-hi/F4/80hi/CD68mod-hi/CD86low/CD163low/CD206hi, o CD45mod-hi/F4/80hi/CD68mod-hi/CD86low/CD163hi/CD206hi. Definisci gli altri macrofagi come CD45mod-hi/F4/80hi/CD68mod-hi/CD86low/ CD163low/CD206low o CD45mod-hi/F4/80hi/CD68mod-hi/CD86hi/CD163hi/CD206hi.
- Definisci le cellule dendritiche come CD45mod-hi/CD11chi/CD86mod-hi. Definisci le cellule NK come CD45mod-hi/NK1.1hi. Definisci altre cellule immunitarie come CD45mod-hi che non soddisfano le condizioni descritte sopra. Definisci le cellule endoteliali come CD31mod-hi. Rimuovere tutti gli altri profili di espressione.
8. Rimappatura delle classificazioni dei fenotipi ed esecuzione del controllo di qualità
- Scarica lo script Remapping Classifications.groovy da https://github.com/xzhou125/JOVE_QuPath_Spatial_Analysis (questo script è stato adattato da un post su https://forum.image.sc/ da petebankhead).
- Apri il file CSV fenotipato e assicurati che tutte le etichette nella scheda Immagine corrispondano esattamente al nome del file immagine nel software di analisi patologica digitale che verrà utilizzato per la rimappatura. Per modificare rapidamente grandi quantità di etichette nel file CSV, utilizzare la funzione Sostituisci tutto.
- Se si utilizza QuPath, eseguire il remapping delle classificazioni del file groovy facendo clic su Automatizza >editor di script.
- Nella scheda Annotazioni, fare clic sui tre punti accanto al pulsante Impostazione automatica, quindi fare clic su Popola da oggetti esistenti > Tutte le classi (incluse le sottoclassi) > Sì.
- Scorri verso il basso nell'elenco dei marcatori per visualizzare le diverse classificazioni dei fenotipi e regolare la combinazione di colori secondo necessità. Fare doppio clic su qualsiasi cella per visualizzarne il fenotipo (accanto al tag Classificazione) e il cluster (accanto al tag Nome). Controllare i campi visivi rappresentativi per determinare l'accuratezza delle assegnazioni dei fenotipi e rivedere la strategia di fenotipizzazione secondo necessità (Figura 3B).
9. Quantificazione della densità e analisi spaziale
- Una volta completata la fenotipizzazione, ottieni le aree di annotazione nel software di analisi patologica digitale nella scheda Annotazioni. Converti i valori dell'area in mm2, quindi dividi i conteggi per le aree di annotazione per calcolare le densità.
- Scaricare lo script SPIAT Spatial Analysis.R da https://github.com/xzhou125/JOVE_QuPath_Spatial_Analysis ed eseguirlo. Raccogli dati sulle distanze minime medie (AMD), sulle composizioni dei quartieri e sui punteggi di miscelazione normalizzati (NMS).
- Genera cifre nel software di analisi statistica per presentare i risultati. Quando si genera un'unica heatmap per le distanze minime medie delle condizioni di trattamento, calcolare la media dei risultati ottenuti da diverse sezioni di tessuto.
Risultati
Qui, presentiamo un protocollo per la progettazione di un pannello di anticorpi per il tessuto FFPE murino, l'esecuzione di imaging in immunofluorescenza multiplex e l'analisi di immagini per la quantificazione proteomica e le relazioni spaziali. Il pannello convalidato contiene 27 anticorpi che forniscono marcatori per la visualizzazione di cellule di melanoma (SOX10), leucociti (CD45), cellule T (CD3, CD4, CD8, FOXP3), cellule B (CD20), macrofagi e sottotipi (F4/80, CD68, CD86, CD163, CD206), cellule dendritiche (CD11c), cellule NK (NK1.1) e cellule endoteliali (CD31). L'intero pannello contiene anche marcatori per altre popolazioni immunitarie (CD11b, CD38), attività di proliferazione (Ki67), funzionalità delle cellule T (T-bet, eomesodermina, granzima B), presentazione dell'antigene (LMP2, beta-2 microglobulina, MHC II) ed espressione del checkpoint (TIM3, LAG3, PD-L1)12. Un'immagine rappresentativa dell'elettroforesi su gel conferma il successo della coniugazione dei codici a barre degli oligonucleotidi del DNA con gli anticorpi privi di carrier, come mostrato nella Figura 4. Questo passaggio conferma solo la reazione chimica e la conferma dell'immagine può essere eseguita solo dopo aver controllato questi anticorpi con il dispositivo di imaging multiplex sul tessuto di interesse. Vedere il seguente riferimento per visualizzare le immagini convalidate di tutti i 27 marcatori nel pannello13. Nella Figura 5 è presentata un'immagine di fusione dei marcatori chiave del lignaggio che colorano tre sezioni di tessuto di melanoma murino trattate con iniezioni intratumorali di nanoparticelle 4-1BBL/IL-12 e anti-PD1 sistemico. Queste sezioni sono state utilizzate per la successiva analisi delle immagini in questo protocollo.
Dopo l'annotazione tissutale, la segmentazione cellulare e la pre-elaborazione dei dati proteomici, presentiamo un confronto tra due algoritmi di clustering che sono stati precedentemente applicati per l'analisi trascrittomica e/o proteomica di singole cellule17,18. I profili di espressione per le popolazioni fenotipizzate sono presentati nella Figura 6 per entrambi gli approcci. Mostriamo che FlowSOM offre una gamma più ampia di valori di intensità (~0,7 vs ~0,5) per discernere le differenze tra popolazioni cellulari simili, come i sottotipi di macrofagi. Seurat ha applicato l'algoritmo di Louvain durante il clustering e ha generato 29 cluster (Figura supplementare 1). In confronto, FlowSOM ha applicato mappe auto-organizzanti ed è in grado di generare 100 cluster (Figura 2 supplementare). Un numero maggiore di cluster si traduce in più tempo necessario per la fenotipizzazione, ma quest'ultimo approccio FlowSOM offre anche più sfumature nella classificazione di popolazioni cellulari simili. Qualitativamente, vediamo che FlowSOM è stato in grado di classificare più macrofagi intratumorali come sottotipo M1 o M2 rispetto alla fenotipizzazione di Seurat nello stesso campo visivo (Figura 7). Lo stesso risultato si vede quando quantifichiamo le densità dei macrofagi, con FlowSOM che cattura una densità più elevata di macrofagi M1 e M2 rispetto a Seurat (Figura 8A-B) e successivamente una densità significativamente più bassa di altri/non classificati macrofagi (p = 0,0028; Figura 8C). Tuttavia, i due approcci di analisi hanno anche generato risultati simili quando descrivono altre densità di popolazione cellulare, come le cellule T CD4, le cellule T CD8 e le cellule endoteliali (Figura 8D-F).
Presentiamo anche i risultati dell'analisi spaziale a valle dopo il clustering e la fenotipizzazione. Nella Figura 9 vengono illustrate alcune delle metriche spaziali che possono essere generate utilizzando questo protocollo. Rispetto a tutte le popolazioni fenotipizzate, i macrofagi M2 e le cellule NK avevano le AMD più alte dopo il trattamento con nanoparticelle 4-1BBL/IL-12 e anti-PD1 (Figura 9A). Allo stesso modo, la NMS tra le cellule T CD8 intratumorali e i macrofagi M1 era molto più alta rispetto a quella tra le cellule T CD8 e i macrofagi M2 (Figura 9B). Inoltre, i macrofagi M2 hanno contribuito ~1% nell'area di 100 μm che circonda le cellule T CD8 intratumorali, mentre i macrofagi M1 costituivano il 9%-13% di questi quartieri (Figura 9C). Nel loro insieme, questi risultati suggeriscono che il regime di trattamento 4-1BBL/IL-12 ha polarizzato i macrofagi associati al tumore verso un sottotipo M1 ed ha escluso i macrofagi M2 dal microambiente immunitario del tumore.
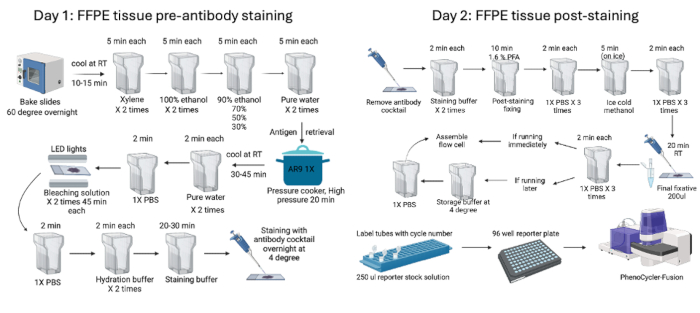
Figura 1: Riepilogo del flusso di lavoro di colorazione e imaging dei tessuti FFPE. I tessuti FFPE murini sono stati lavorati utilizzando procedure di pre-colorazione che sono iniziate con la cottura del tessuto durante la notte prima di iniziare i processi di pre-colorazione (giorno 1) e post-colorazione (giorno 2) di due giorni. Infine, la piastra reporter è stata preparata prima dell'imaging con il dispositivo. Clicca qui per visualizzare una versione più grande di questa figura.
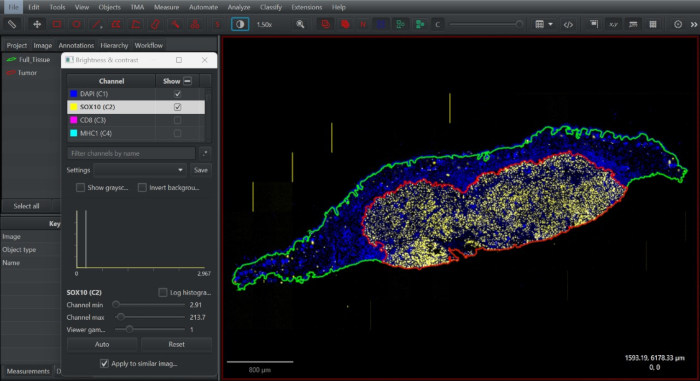
Figura 2: Screenshot dell'annotazione dei tessuti nel software di patologia digitale ad accesso aperto QuPath. L'annotazione completa del tessuto è stata disegnata (verde), duplicata e minimizzata per definire il compartimento intratumorale in base alla distribuzione dei melanociti SOX10+ al confine tumorale (rosso). Clicca qui per visualizzare una versione più grande di questa figura.
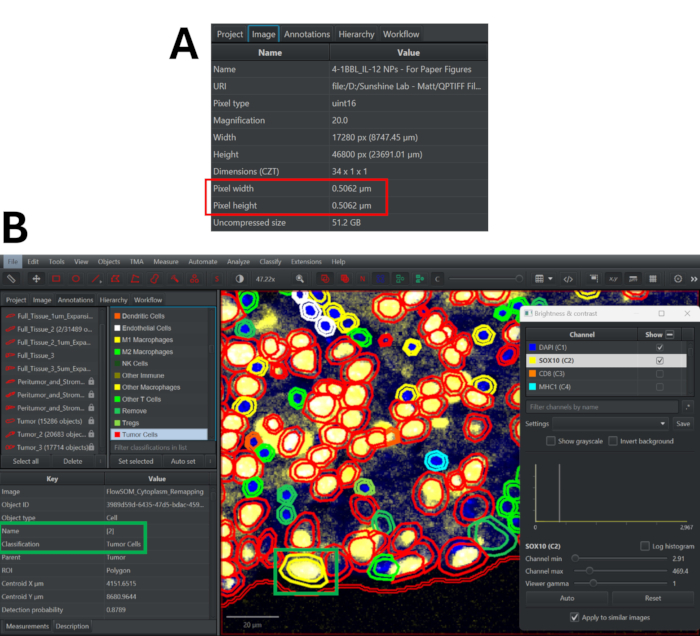
Figura 3: Segmentazione cellulare utilizzando l'algoritmo StarDist in QuPath e il processo di controllo qualità per la revisione delle classificazioni dei fenotipi. (A) Passare alla scheda Immagine per determinare la larghezza e l'altezza dei pixel dell'immagine multiplex in immunofluorescenza. (B) Dopo aver rimappato le classificazioni, fare doppio clic su qualsiasi cella (viene evidenziata in giallo) per vedere l'assegnazione del cluster e il fenotipo. Attiva/disattiva i marcatori del pannello per determinare se questo approccio di classificazione è accurato. Clicca qui per visualizzare una versione più grande di questa figura.
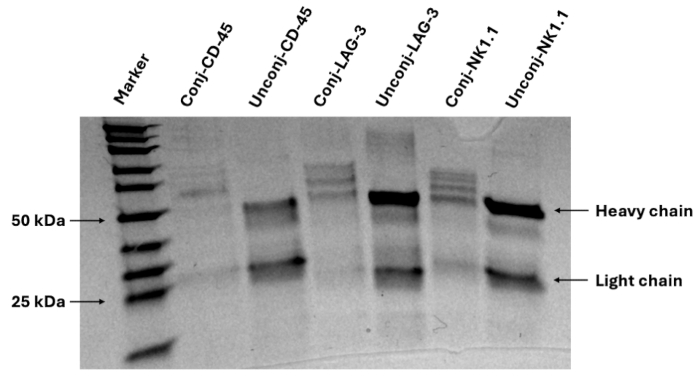
Figura 4: Immagine gel rappresentativa della conferma della coniugazione del codice a barre anticorpo-DNA. L'elettroforesi su gel proteico conferma la coniugazione degli anticorpi con codici a barre di oligonucleotidi del DNA, osservata da bande aggiuntive nel sito della catena pesante. Clicca qui per visualizzare una versione più grande di questa figura.
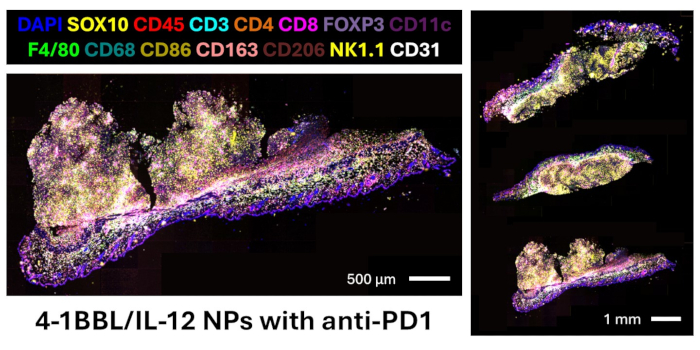
Figura 5: Imaging multiplex basato su codice a barre del DNA di tumori del fianco B16F10 trattati con iniezioni intratumorali di nanoparticelle 4-1BBL/IL-12 con anti-PD1 sistemico. Marker nel nostro pannello non mostrati: CD11b, CD20, CD38, TIM3, LAG3, T-bet, Eomesodermin, granzima B, Ki67, LMP2, beta-2 microglobulina, MHC II, PD-L1. Clicca qui per visualizzare una versione più grande di questa figura.
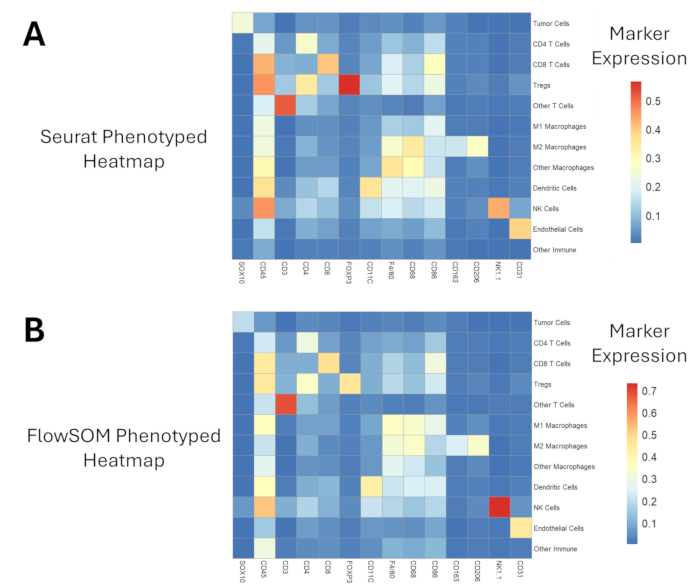
Figura 6: Mappe di calore di espressione proteomica di popolazioni cellulari fenotipizzate. (A) L'approccio di clustering e fenotipizzazione di Seurat genera un intervallo leggermente più piccolo di valori di espressione dei marcatori rispetto a (B) Clustering e fenotipizzazione di FlowSOM. Clicca qui per visualizzare una versione più grande di questa figura.
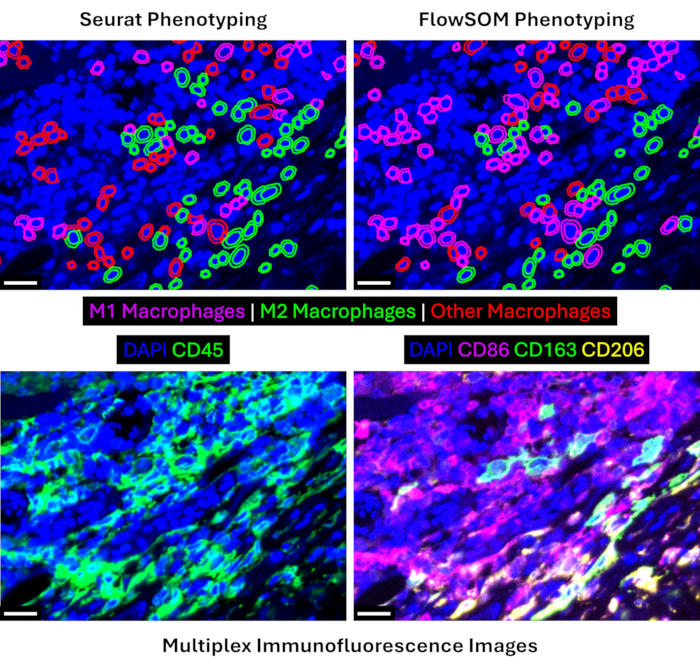
Figura 7: La fenotipizzazione FlowSOM identifica una gamma più ampia di diversi sottotipi di macrofagi. Il pannello superiore mostra la fenotipizzazione dei macrofagi Seurat (a sinistra) e FlowSOM (a destra) per lo stesso campo visivo. Il pannello inferiore mostra le immagini multiplex in immunofluorescenza dei marcatori chiave del lignaggio dei macrofagi nello stesso campo visivo. Tutte le barre della scala sono da 20 μm. Clicca qui per visualizzare una versione più grande di questa figura.
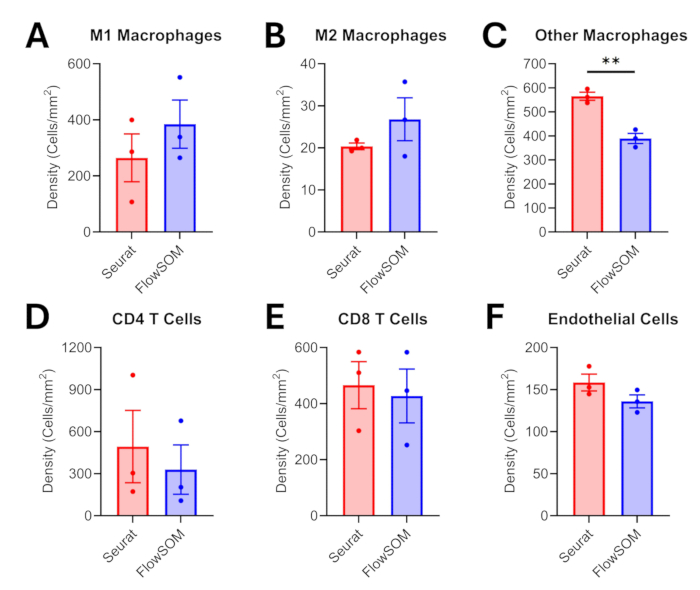
Figura 8: Confronto delle densità intratumorali di diverse popolazioni cellulari dopo il clustering/fenotipizzazione. I confronti di densità sono mostrati per i macrofagi intratumorali (A) M1, (B) macrofagi M2, (C) altri macrofagi, (D) cellule T CD4, (E) cellule T CD8 e (F) cellule endoteliali. Le barre di errore sono errori standard della media (SEM) e la significatività è stata testata utilizzando test t non appaiati (**p ≤ 0,01). Clicca qui per visualizzare una versione più grande di questa figura.
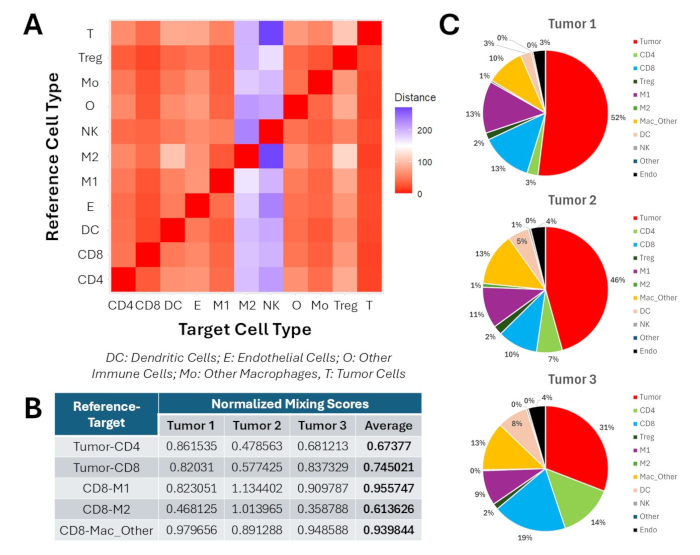
Figura 9: Profilazione delle metriche spaziali intratumorali per tre tumori del fianco trattati con iniezioni intratumorali di nanoparticelle 4-1BBL/IL-12 e anti-PD1 sistemico. (A) Heatmap delle distanze minime medie tra le popolazioni fenotipizzate intratumorali. Le misure sono in μm. (B) Punteggi di miscelazione normalizzati tra coppie chiave di fenotipi intratumorali. (C) Scomposizione delle composizioni di quartiere in un raggio di 100 μm attorno a popolazioni intratumorali di cellule T CD8. Clicca qui per visualizzare una versione più grande di questa figura.
| Anticorpo | Diluizione | Tempo di esposizione (ms) |
| SOX-10 | 50 | 450 |
| CD8 | 100 | 450 |
| CD3 | 100 | 450 |
| VolpeP3 | 50 | 350 |
| CD4 | 50 | 450 |
| MHC-II | 100 | 450 |
| PDL1 | 50 | 450 |
| CD45 | 100 | 450 |
| Ki-67 | 100 | 300 |
| F4/80 | 100 | 150 |
| CD20 | 100 | 450 |
| NK1.1/CD161 | 50 | 450 |
| CD206 | 100 | 450 |
| CD68 | 100 | 450 |
| Granzima-B | 50 | 450 |
| CD86 | 100 | 150 |
| CD31 | 100 | 450 |
| CD11C | 50 | 450 |
| CD11b | 50 | 450 |
| EOMES | 50 | 450 |
| TIM-3 | 50 | 300 |
| CD38 | 50 | 200 |
| LAG3 | 50 | 450 |
| CD163 | 50 | 200 |
| Scommessa T | 50 | 300 |
| Motore LMP2 | 100 | 100 |
| Beta2 MG | 200 | 50 |
Tabella 1: Impostazioni della diluizione degli anticorpi e del tempo di esposizione.
Figura 1 supplementare: Heatmap iniziale dell'espressione proteomica generata dopo il clustering di Seurat. Clicca qui per scaricare questa figura.
Figura 2 supplementare: Heatmap iniziale dell'espressione proteomica generata dopo il clustering di FlowSOM. Clicca qui per scaricare questa figura.
Discussione
Il successo dell'imaging dipende da un pannello di anticorpi ben progettato e convalidato. L'imaging multiplex in immunofluorescenza di campioni FFPE presenta sfide a causa dell'elevata autofluorescenza e della difficoltà di recuperare gli epitopi mascherati dall'inclusione di paraffina. Tuttavia, dato che FFPE offre diversi vantaggi rispetto ai campioni FF, è essenziale progettare e convalidare pannelli anticorpali FFPE. Il primo passo è finalizzare i cloni di anticorpi che mostrano segnali positivi durante l'imaging in immunofluorescenza (IF); successivamente, è importante coniugarli accuratamente con codici a barre del DNA. La coniugazione dell'anticorpo richiede una riduzione parziale dell'anticorpo per creare legami SH, che vengono utilizzati durante la reazione del gruppo maleimmide con un codice a barre. Non tutti i cloni di anticorpi possono resistere a questo passaggio e alcune reazioni possono causare danni irreversibili all'anticorpo, con conseguente fallimento dell'imaging nonostante la coniugazione riuscita. Per questo motivo, anche se alcuni anticorpi possono mostrare segnali positivi durante la validazione convenzionale dell'IF, per valutare il successo finale della coniugazione anticorpale, è importante convalidare ogni anticorpo sul tessuto di interesse e registrare i tempi di esposizione desiderati per quel tessuto. Nelle applicazioni future, questa tecnica può essere combinata con l'analisi trascrittomica spaziale esistente su vetrini consecutivi/lo stesso vetrino per generare ulteriori informazioni. Uno dei limiti di questo metodo è che richiede un'attenta selezione e convalida di ogni anticorpo nel pannello in base al bersaglio e al tipo di tessuto.
Per quanto riguarda l'analisi delle immagini, QuPath offre un prezioso strumento ad accesso aperto con una visualizzazione di alta qualità dei marcatori proteomici, un'ampia funzionalità per l'esportazione delle misurazioni dell'intensità e l'esecuzione del controllo di qualità per le classificazioni dei fenotipi e una buona flessibilità per gli script generati dagli utenti. I forum online come https://forum.image.sc/ sono una risorsa aggiuntiva per discutere su come eseguire attività di analisi specifiche e per condividere script con altri utenti. In questo protocollo, confrontiamo due approcci di clustering e fenotipizzazione utilizzando Seurat e FlowSOM. Sebbene FlowSOM possa essere preferito per la sua capacità di generare informazioni più granulari sulle sottopopolazioni di cellule immunitarie del TME, è necessario prendere in considerazione anche il tempo necessario per l'analisi proteomica. La generazione di 100 cluster potrebbe non essere necessaria se un utente ha bisogno solo di fenotipizzare le cellule all'interno di uno o due campioni di tessuto. In queste situazioni, Seurat può offrire una pipeline più rapida ed efficiente per l'analisi delle immagini. Al contrario, l'analisi di un TMA con più di 40 o 50 sezioni di tessuto ha maggiori probabilità di produrre un numero maggiore di cluster cellulari in entrambi gli approcci di analisi e FlowSOM può essere la metodologia preferita per generare classificazioni fenotipiche più sfumate.
Il clustering/fenotipizzazione cellulare e tutte le successive fasi di analisi delle immagini dipendono in gran parte dalla segmentazione cellulare. Il nostro lavoro attuale ha esplorato la segmentazione cellulare negli algoritmi HALO (Indica Labs) e StarDist, e abbiamo scoperto che entrambi gli approcci tendono a sovrasegmentare le cellule in base ai segnali nucleari DAPI. Sono disponibili anche molti algoritmi di segmentazione alternativi, come Mesmer19 e InstanSeg20. Si tratta di un campo in crescita della ricerca computazionale che richiede ulteriori esplorazioni e ottimizzazioni.
Divulgazioni
J.C.S. riconosce il sostegno finanziario di Emerson Collective LLC e del National Institutes of Health. J.J.G. riconosce anche i finanziamenti del National Institutes of Health. J.C.S. ha un rapporto con Palleon Pharmaceuticals Inc., che prevede il finanziamento di sovvenzioni. S.Y.T. e J.J.G. hanno una relazione con OncoSwitch Therapeutics che coinvolge azioni o azioni. S.Y.T., J.J.G., J.C.S. e K.M.L. hanno un brevetto in corso di registrazione. Tutti gli altri autori affermano di non avere interessi finanziari o relazioni personali che potrebbero essere percepiti come influenzanti la ricerca presentata in questo articolo.
Riconoscimenti
J.C.S. riconosce la Dermatology Foundation e il Dermatopathology Career Development Award per aver fatto avanzare la carriera dell'autore. Gli autori ringraziano Hsin-Pei Lee del Cancer Data Science Laboratory del National Cancer Institute per l'assistenza con le tecniche di analisi computazionale. Questa ricerca ha ricevuto finanziamenti dall'Emerson Collective e dal National Institutes of Health (R37CA246699, P41EB028239 e R01CA228133). Inoltre, il nucleo del Johns Hopkins Oncology Tissue Services (OTS) è supportato dal National Institutes of Health (P30CA006973).
Materiali
| Name | Company | Catalog Number | Comments |
| 16% paraformaldehyde (PFA) | Electron Microscopy Sciences | PN# 15710 | Required during tissue staining process |
| 50kDa MWCO filter | Millipore | UFC505096 | 25 kDa and 100 kDa result in failure |
| Akoya barcodes and reporters | Akoya Biosciences | Varied | Each barcode can be used to conjugate 50 ug of carrier free antibody and comes with two vials of reporters |
| Antibody conjugation kit | Akoya Biosciences | 7000009 | A conjugation kit is used to create custom barcode-conjugated antibodies. Each kit contains sufficient reagents for ten conjugations. The kit consists of one subkit box stored at 4 °C and one subkit box stored at -20 °C. The 4 °C subkit includes Filter Blocking Solution, Reduction Solution 2, Conjugation Solution, Purification Solution, and Antibody Storage Solution. The -20 °C subkit includes Reduction Solution 1. |
| Beta2MG | Abcam | ab214769 | |
| CD11b | CST | #41028 | |
| CD11C | CST | #39143SF | |
| CD163 | CST | #68922BF | |
| CD20 | CST | #45839 | |
| CD206 | CST | #87887 | |
| CD3 | CST | #24581 | |
| CD31 | CST | #92841 | |
| CD38 | CST | #68336BF | |
| CD4 | Abcam | ab271945 | |
| CD45 | CST | #98819 | |
| CD68 | CST | #29176 | |
| CD8 | Invitrogen | #14-0808-82 | |
| CD86 | CST | #20018 | |
| Dimethyl sulfoxide | Avantor/VWR | BDH1115-4LP | |
| EOMES | Abcam | ab222226 | |
| Eppendorf PCR tubes | Eppendorf | E0030124286 | Required store 5 μL conjugated antibody for conjugation confirmation by gel electrophoresis |
| Ethanol 200 proof | |||
| F4/80 | CST | # 25514 | |
| FoxP3 | CST | #72338 | |
| Gran-B | CST | #79903SF | |
| Heating oven | |||
| Hybridization chamber | Used to stain tissue with antibody cocktail | ||
| Hydrogen peroxide | Sigma | #216763 | Required for preparing bleaching solution |
| Instant pot pressure cooker or Decloaking Chamber ARC | Instant pot or Biocare Medical | ||
| Ki-67 | Akoya Biosciences | ||
| LAG3 | CST | #80282BF | |
| LMP2 | Abcam | ab243556 | |
| Methanol | |||
| MHC-II | eBioscience | #14-5321-82 | |
| NK1.1 | CST | #24395SF | |
| Nuclear Stain | Akoya Biosciences | 7000003 | |
| PDL1 | CST | #85095 | |
| Salmon Sperm DNA, sheared (10 mg/mL) | Invitrogen | AM9680 | Can be used as an alternative to assay reagent (Akoya) during reporter plate preparation step. |
| SOX-10 | Abcam | ab245760 | |
| Staining kit for PhenoCycler-Fusion | Akoya Biosciences | 7000017 | The PhenoCycler-Fusion Sample Kit includes the buffers and reagents necessary for tissue staining using antibodies conjugated with PhenoCycler barcodes, along with flow cells for whole-slide imaging. Each kit is sufficient for 10 PhenoCycler-Fusion experiments. It consists of one subkit stored at 4 °C, another at -20 °C, and a package of 10 flow cells kept at room temperature. The subkit stored at 4 °C contains Hydration Buffer, Staining Buffer, Storage Buffer, N Blocker, and J Blocker. The subkit stored at -20 °C contains G Blocker, S Blocker, and Fixative Reagent. |
| T-bet | CST | #53753 | |
| TIM-3 | CST | #72911 | |
| UltraPure DNase/RNase-free distilled water | Invitrogen | 10977015 | Required to dissolve barcodes during antibody conjugation |
Riferimenti
- Schadendorf, D., et al. Melanoma. Lancet. 392 (10151), 971-984 (2018).
- Taube, J. M., et al. Implications of the tumor immune microenvironment for staging and therapeutics. Modern Pathol. 31 (2), 214-234 (2018).
- Marzagalli, M., Ebelt, N. D., Manuel, E. R. Seminars in cancer biology. 59, 236-250 (2019).
- De Visser, K. E., Joyce, J. A. The evolving tumor microenvironment: From cancer initiation to metastatic outgrowth. Cancer cell. 41 (3), 374-403 (2023).
- Balkwill, F. R., Capasso, M., Hagemann, T. The tumor microenvironment at a glance. J Cell Sci. 125 (23), 5591-5596 (2012).
- Phillips, D., et al. Highly multiplexed phenotyping of immunoregulatory proteins in the tumor microenvironment by codex tissue imaging. Front Immunol. 12, 687673(2021).
- Giesen, C., et al. Highly multiplexed imaging of tumor tissues with subcellular resolution by mass cytometry. Nat Methods. 11 (4), 417-422 (2014).
- Keren, L., et al. Mibi-tof: A multiplexed imaging platform relates cellular phenotypes and tissue structure. Sci Adv. 5 (10), eaax5851(2019).
- Lin, J. R., Fallahi-Sichani, M., Sorger, P. K. Highly multiplexed imaging of single cells using a high-throughput cyclic immunofluorescence method. Nat Comm. 6 (1), 8390(2015).
- Radtke, A. J., et al. Ibex: An iterative immunolabeling and chemical bleaching method for high-content imaging of diverse tissues. Nat Protoc. 17 (2), 378-401 (2022).
- Black, S., et al. Codex multiplexed tissue imaging with DNA-conjugated antibodies. Nat Protoc. 16 (8), 3802-3835 (2021).
- Aung, T. N., Bates, K. M., Rimm, D. L. High-plex assessment of biomarkers in tumors. Mod Pathol. 37 (3), 100425(2024).
- Surwase, S. S., et al. Highly-multiplexed immunofluorescence phenocycler panel for murine ffpe yields insight into tumor microenvironment immunoengineering. Lab Invest. 105 (1), 102165(2024).
- Luly, K. M., Green, J. J., Sunshine, J. C., Tzeng, S. Y. Biomaterial-mediated genetic reprogramming of merkel cell carcinoma and melanoma leads to targeted cancer cell killing in vitro and in vivo. ACS Biomater Sci Eng. 9 (11), 6438-6450 (2023).
- Satija, R., Farrell, J. A., Gennert, D., Schier, A. F., Regev, A. Spatial reconstruction of single-cell gene expression data. Nat Biotechnol. 33 (5), 495-502 (2015).
- Van Gassen, S., et al. Flowsom: Using self-organizing maps for visualization and interpretation of cytometry data. Cytometry Part A. 87 (7), 636-645 (2015).
- Butler, A., Hoffman, P., Smibert, P., Papalexi, E., Satija, R. Integrating single-cell transcriptomic data across different conditions, technologies, and species. Nat Biotechnol. 36 (5), 411-420 (2018).
- Watson, S. S., et al. Microenvironmental reorganization in brain tumors following radiotherapy and recurrence revealed by hyperplexed immunofluorescence imaging. Nat Comm. 15 (1), 3226(2024).
- Greenwald, N. F., et al. Whole-cell segmentation of tissue images with human-level performance using large-scale data annotation and deep learning. Nat Biotechnol. 40 (4), 555-565 (2022).
- Goldsborough, T., et al. Instanseg: An embedding-based instance segmentation algorithm optimized for accurate, efficient and portable cell segmentation. arXiv. , (2024).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneEsplora altri articoli
This article has been published
Video Coming Soon