Method Article
DNAバーコードを用いたマルチプレックス免疫蛍光イメージングによる、遺伝的に再プログラムされたマウス黒色腫のFFPE検体を解析
* これらの著者は同等に貢献しました
要約
このプロトコールは、マウスFFPE黒色腫組織のDNAバーコードベースのイメージングのためのマルチプレックス免疫蛍光抗体パネルを設計するためのステップバイステップガイドを提供します。また、マウスの黒色腫腫瘍免疫微小環境に対する空間的プロテオミクスの洞察を生成するためのオープンソースツールを使用した画像解析パイプラインについても説明します。
要約
ここで紹介するのは、組織微小環境の空間プロテオミクスを解析するCo-Detection-by-indEXingに基づく新しいDNAバーコードベースのマルチプレックスイメージング技術です。イメージングを成功させるには、適切に設計され、適切に検証された抗体パネルのレパートリーが必要ですが、現在、ホルマリン固定パラフィン包埋(FFPE)サンプルにはごくわずかしか存在しません。FFPEは、広く入手可能であること、取り扱いと保管が容易であること、組織マイクロアレイ(TMA)を作成できることなど、新鮮凍結検体に比べていくつかの利点があります。ここでは、ナノ粒子で処理したマウス黒色腫モデルからFFPE組織を可視化および分析するための抗体パネルを開発するためのプロトコルを紹介します。これは、腫瘍微小環境リプログラミングのための免疫学的シグナルをコードするプラスミドDNAを送達します。また、組織のアノテーション、細胞のセグメンテーション、プロテオミクスデータの処理、細胞集団の表現型決定、および空間メトリクスの定量化のためのオープンソースの計算ツールを使用した画像解析パイプラインについても説明します。このプロトコルは、マウスFFPEで抗体パネルを設計し、複雑な組織微小環境の空間的プロテオミクスに関する新たな洞察を生成するためのアプリケーションを提供します。
概要
皮膚黒色腫は最も一般的な皮膚がんであり、診断時期やプライマリケアの時期によって、世界中で疾患率や死亡率にばらつきがあります1。過去10年間で、黒色腫の生物学的理解が深まったことで、固形腫瘍を治療するための新しいがんモデルの開発が推進されました2。近年の免疫療法の台頭により、内因性免疫系の活性化に基づくがん治療の革新的な概念が生まれました3,4。
腫瘍微小環境(TME)は非常に複雑で、多様な免疫細胞、がん関連線維芽細胞、周皮細胞、内皮細胞、およびさまざまな組織常在細胞5で構成されています。TMEの研究には、フローサイトメトリーやシングルセルシーケンシングなど、過去にいくつかの技術が適用されてきましたが、腫瘍組織を破壊する必要があるため、空間的な状況が損なわれます。免疫蛍光法(IF)や免疫組織化学法(IHC)などの従来の顕微鏡イメージングでは、サンプル組織を破壊することなくタンパク質バイオマーカーを可視化することができます。しかし、これらのアプローチは2つまたは3つのバイオマーカーに限定されており、複雑な TME6内の空間的および構造的関係を完全に理解することはできません。
この問題に対処するために、複雑なTMEを空間的に視覚化するためのいくつかのマルチプレックスイメージング技術が開発されました7,8,9,10。その1つが、DNAオリゴヌクレオチド標識抗体11に基づくPhenoCyclerシステムとして改名されたCoDetection-by-inDEXingです。このシステムは、ヒト検体の100を超えるバイオマーカーのシングルセルイメージングと分析を提供できます。しかし、マウス検体、特にホルマリン固定パラフィン包埋(FFPE)サンプル12を可視化および分析するために利用できるインベントリされた抗体は非常に少ない。FFPEは、取り扱いと保管の容易さ、長期にわたる良好な形態の保存、そして最も重要なこととして、1枚のスライドで複数の標本を視覚化できる組織/腫瘍マイクロアレイ(TMA)を調製する能力など、フレッシュフローズン(FF)の保存に比べていくつかの利点があります。私たちは最近、マウスFFPE CODEX / PhenoCycler抗体パネルを設計および開発し、遺伝的に再プログラムされたマウス黒色腫検体の空間プロテオミクスを視覚化および分析するためにそれを適用することに成功しました13。
このプロトコールの全体的な目標は、マウスFFPE抗体パネルを設計するためのステップバイステップのガイドを提供し、抗体バーコードの標識、組織染色、およびイメージングのプロセスを説明することです。さらに、QuPathやRパッケージなどのオープンソースツールを活用した詳細な画像解析パイプラインも紹介します。このプロトコルに従った後、研究者はカスタム標識抗体パネルの設計方法、Phenocycler-Fusionデバイスを使用したマルチプレックスイメージングの実行方法、およびメラノーマTMEの空間プロテオミクスに関する新たな洞察を得ることができます。さらに、このプロトコルは、さまざまな腫瘍免疫微小環境の研究に適合させ、既存の空間的トランスクリプトミクス技術と組み合わせることができます。
プロトコル
すべての動物作業は、ジョンズホプキンス大学動物管理および使用委員会によって設定されたガイドラインに従って、承認されたプロトコル番号MO18M388およびMO21M384を使用して行われました。
1. 抗体の選択
- 目的の組織と特定のバイオマーカーの存在量に基づいて抗体パネルを設計します。文献に記載されている免疫蛍光染色(IF)および免疫組織化学染色(IHC)の過去の成功したアプリケーションに基づいて、抗体クローンを選択してください。これらの抗体の安全なキャリアフリーバージョン。
注:キャリアを含まない精製された抗体は、バーコード標識にとって重要です。BSA、グルテン、グリセロールなどのタンパク質ベースの防腐剤は避けてください。キャリアフリー抗体が入手できない場合は、サプライヤーにカスタム抗体をリクエストするか、BSA除去キットを使用してキャリアフリー抗体を作製してください。アジ化ナトリウムは抗体の結合を妨げません。IgMアイソタイプよりもIgGアイソタイプが推奨されます。 - これらの選択されたクローンを、従来の免疫蛍光(IF)イメージング11を用いて目的の組織で検証する。ダブルIFイメージングを使用して、特定のクローンの特異性を確認します(例えば、FOXP3およびCD4によるダブルIFイメージングを行い、制御性T細胞の染色を確認します)。
注:可能な限り、染色に使用されるのと同じ組織をIFバリデーションに使用することが重要です。適切な抗原賦活化溶液を選択します。このプロトコルでは、Akoya Biosciencesから提供されたAR9バッファーを使用して、すべてのマウス黒色腫FFPE組織エピトープを取得しました。AR6やUniversalなどの他の抗原賦活化溶液は、エピトープの要件に基づいて使用できます。
2. 抗体の標識と確認
- ATTO550(Cy3)、AF647(Cy5)、またはAF750(Cy7)蛍光色素に相補レポーターが付着したバーコードに検証済みの抗体を割り当てます。存在量が少ない抗原は、通常、シグナルの生成が少なくなります。このような抗体をAF647(Cy5)などの低自家蛍光チャネルに結合させます。高発現抗原をATTO550(Cy3)およびAF750(Cy7)に結合させると、自家蛍光の可能性があります。
注:この重要なステップでは、チャネル感度と抗原存在量を考慮します。FFPE組織にAF488チャンネルを使用することは、自家蛍光が高いため推奨されません。 - 抗体-バーコード標識(4.5時間)の場合は、抗体標識キットを入手してください。すべての試薬は、小さな1回限りの使用バイアルとして-20°Cで保存する必要がある還元液1を除いて、4°Cで保存してください。
- 分光光度計を使用して抗体濃度を測定します。
- 500 μLのフィルターブロッキング溶液を50 kDa MWCOフィルターカラムに適用して抗体の標識を開始し、抗体のフィルターへの非特異的結合をブロックします。12,000 x g で室温(RT)で2分間スピンダウンします。200 μL マイクロピペットを使用してカラムから余分な液体を取り除きます。
注:還元液1は使い捨ての溶液バイアルであり、一度に3つの抗体標識に十分です。解凍後は溶液を再利用しないでください。残っている試薬は廃棄してください。50 kDa MWCOフィルターカラムにフィルターを使用すると、精製と結合が不十分になる可能性があるため、使用しないでください。 - 50 μg相当量の抗体溶液を使用し、50 kDaのMWCOフィルターに添加します。必要に応じて、PBSを使用して総容量を100μLに調整します。12,000 x g で4°Cで8分間スピンダウンします。 この間に、19.8 μL の還元液 1 + 825 μL の還元液 2 を使用して、3 つの抗体標識に対して還元マスターミックスを調製します。
- フロースルーを捨て、各フィルターユニットの上部に260μLのリダクションマスターミックスを追加します。フィルターユニット内で溶液を2〜3秒間ボルテックスし、室温で30分間インキュベートします。
注:抗体の過剰還元を避けるためには、30分を超えるインキュベーションを行わないことが重要です。これは抗体を損傷し、結合に失敗する可能性があるためです。 - 30分間のインキュベーション後、12,000 x g で4°Cで8分間スピンダウンし、底部でフロースルーを廃棄します。450 μLの標識溶液をカラムの上部に加え、12,000 x g で4°Cで8分間スピンダウンします。 遠心分離中に、CODEXバーコード溶液を調製します。-20°Cからバーコードを取り出した後、バーコードが劣化し始めるため、すばやく移動してください。
注:バーコードは通常、-20°Cで保存された凍結乾燥製品(フレークまたは粉末)が入った小さなガラスバイアルで提供されます。 これらのバイアルは、一度に50 μgの抗体を標識するために使用できます。一度に3つ以上の抗体を標識しないことが推奨されます。 - ガラスバイアルの下部にある凍結乾燥バーコードを慎重に見つけます(壁に貼り付けられている場合もあります)。テーブルの上のガラスバイアルを軽くたたいて、固形物を底に持ってきます。凍結乾燥産物をヌクレアーゼフリーの分子生物学グレードの水10 μLに溶解する際には、凍結乾燥産物の位置を特定することが不可欠です。10 μLのヌクレアーゼフリー水を加えた後、各バーコードに210 μLの抱合溶液を加えます。凍結乾燥された材料をすべて溶解し、ピペッティングで上下させて穏やかに混合します。扠置く。
- ステップ2.2.5でスピンが完了したら、フロースルーを破棄し、ステップ2.2.6のそれぞれのバーコード溶液を各フィルターユニットの上部に追加します。最初の非標識抗体を1 μg保存します。これは、ゲル電気泳動の検証中に、標識された5 μL抗体サンプルと一緒に実行されます。
- 各チューブにそれぞれの抗体とバーコード名をラベル付けします。蓋を閉め、2〜3秒間ボルテックスして溶液を混合します。抗体-バーコード標識反応をRTで2時間インキュベートします。
- 2時間のインキュベーション後、5 μLの標識抗体を取り出し、0.2 mLのPCRチューブに保存して、ゲル電気泳動による標識を確認します。
- 残りの標識抗体溶液を12,000 x g で4°Cで8分間スピンダウンします。 カラムの上部に450 μLの精製溶液を加え、12,000 x g で4°Cで8分間遠心し、フロースルーを廃棄します。この精製ステップを2回繰り返し、毎回450μLの精製溶液を加えてスピンダウンします。
- 3回目の 遠心分離後、フロースルーを廃棄します。上部のフィルターには、バーコード標識抗体が含まれます。
- 標識抗体を採取するには、フィルターカラムを保持する新しいアウターチューブに対応する抗体とバーコードをラベル付けします。遠心分離する前に、アウターチューブの蓋を切り取ります。各フィルターユニットに100 μLの抗体保存溶液を加え、新しく標識したアウターチューブを逆さまにしてフィルターカラムの上に置きます。
- フィルターカラムを反転させて標識抗体をアウターチューブに集め、3,000 x g で室温で2分間スピンダウンします。これにより、約120 μLの標識抗体溶液が収集されます。滅菌マイクロ遠心チューブに移し、4°Cで18〜24ヶ月間保存します。
注:標識後の高バックグラウンド核染色を避けるために、これらの抗体を少なくとも2日間使用しないことが推奨されます。
- すでに公開されているプロトコル13に記載されているように、ゲル電気泳動による標識確認を行います。
注:このプロセスは、バーコードを抗体と結合するために使用される化学標識反応の成功を確認するだけです。抗体の検証は、組織の染色とイメージングが成功した後にのみ可能となるため、オプションです。
3. マウスFFPE試料作製
- B16F10側面腫瘍を移植したC57BL/6J雌マウスからFFPE組織サンプルを採取します。4-1BBLおよびIL-12プラスミドを含むポリ(β-アミノエステル)ベースのナノ粒子の腫瘍内注射でマウスを治療し、移植後9、11、16、および18日で抗PD1の腹腔内送達を行います14。20日目にマウスを犠牲にし、パラフィンに埋め込まれた10%ホルマリンで腫瘍を固定し、次にジョンズホプキンス腫瘍学組織サービスのコアで切片を作ります。
- ユーザーマニュアルに記載されているイメージングエリアの仕様を慎重に検討し、イメージングエリア内のスライドに5μmの試料をマウントします。1枚のスライドに4種類の腫瘍標本を載せることができました。
注:イメージングには、Leica Apex Adhesive SlidesやFisherbrand Superfrost Plus Slidesなどの推奨スライドを使用することが不可欠です。これにより、フローセルがスライドにしっかりと接着します。
4. FFPE組織染色とイメージング
- 抗体濃度と曝露時間の検証と最適化:抗体-バーコード標識の成功を検証するには、目的の組織を6-9標識抗体で染色します。マルチプレックスイメージングデバイスを実行し、各抗体濃度と露光時間を記録します。必要に応じて希釈と曝露時間を調整し、次に以前に検証された抗体で6〜9個の新しい抗体を実行し、調整内容に注意してください。染色とイメージングについては、以下の手順に従ってください。
- 組織染色とイメージング
- 染色の前日に、FFPEティッシュスライドを60°Cのオーブンで一晩焼きます。翌日、スライドをRTで10分間冷却してから、脱パラフィンと組織の再水和のステップを開始します。
- 組織の脱パラフィン化を開始するには、キシレン溶液に2回、それぞれ5分間インキュベートします。
- 組織を再水和するには、100%エタノール溶液を2回、それぞれ5分間動かします。次に、90%から70%、50%、30%までの一連のアルコール溶液をそれぞれ5分間移動させます。最後に、ティッシュを蒸留水で5分間ずつ2回洗います。
注:ネオクリア溶液など、キシレンの毒性の低い代替品を脱パラフィンステップに使用できます。ヒュームフードの下で脱パラフィンと水分補給を行います。 - 蒸留水を使用して10x AR9バッファーを1xに希釈します。Coplinジャーに50 mLの1x AR9バッファーを充填し、スライドが抗原賦活化バッファーに完全に浸されていることを確認します。
- レベルがCoplinジャーの半分になるように圧力鍋に水を入れ、高圧設定で20分間インキュベートします。
注:120°Cで10分間設定することで、抗原の賦活化を向上させるために、専用の圧力鍋またはDecloaking Chamberを使用することが可能です。 - 圧力鍋のステップが完了したら、スライドを45〜60分間冷却します。スライドを蒸留水で2回ずつ2分間すすぎます。次に、スライドを1x PBSが入った瓶に移します。
- 自家蛍光を最小限に抑えるには、抗体で染色する前に組織で漂白ステップを実行します。推奨試薬と濃度を使用して、漂白液(25 mL 1x PBS + 0.8 mL 1M NaOH + 4.5 mL H2O2)を新たに調製します。漂白液を入れたプラスチック容器内で、2つのLEDランプの間にスライドを室温で45分間インキュベートします。新鮮な漂白液でさらに45分間繰り返します。
- この漂白ステップ中に、特にパネル内にさまざまな希釈度の抗体がいくつかある場合は、抗体カクテル溶液の調製を開始します。4つのブロッカー(G、S、J、N)をすべて回収し、-20°Cから解凍するのに時間がかかるものもあるため、氷の上に置きます。
- 光退色後、ティッシュを1x PBSで、2回ずつ2分間洗浄します。ティッシュをハイドレーションバッファーの入った瓶に移し、それぞれ2分間2回洗浄します。
- 組織スライドを染色バッファーに移し、30分間平衡化します。漂白ステップで調製しなかった場合、この間に抗体カクテル溶液を調製してください。
- 2枚のスライドを染色するために、300 μLの抗体カクテル溶液を調製します(通常、ハイブリダイゼーションプラスチックチャンバーを使用する場合、各スライドを染色するには100〜120 μLの抗体カクテルで十分です)。
- スライドを染色するには、抗体カクテル溶液を湿度チャンバー内に4°Cで一晩塗布します。
注:ここに記載されているすべての抗体は、4°Cで一晩染色すると陽性のシグナルを示します。 ただし、各抗体の染色時間と温度は異なる場合があり、最適化する必要があります。最も一般的に使用される染色条件は、室温で3時間、または4°Cで一晩です。 必要に応じて、いくつかの抗体を最初に異なる温度で染色し、次に残りの抗体を4°Cで一晩染色することも可能です。 その後、逐次染色が必要な場合は、PBSで組織を洗浄します。 - 翌日、染色後の固定を行います。プラスチックチャンバーを取り外し、回収した抗体カクテルを4°Cで保存します。 スライドを染色バッファーで2回ずつ2分間洗浄します。次に、スライドを40 mLの染色後固定溶液(16%パラホルムアルデヒド4 mL+保存バッファー36 mL)に移し、10分間インキュベートします。PBSで3回、各2分間すすぎます。
- スライドを氷冷したメタノールに5分間移動させ、次にPBSで3回、それぞれ2分間すすぎます。最終固定液(チューブ1本20μL+PBS1mL)を湿度チャンバー下のスライドに塗布します。通常、スライド1枚につき200μLの固定液で十分です。固定剤をRTで20分間放置し、PBSでそれぞれ2分間の最終洗浄を3回行います。
注意: 事前に-20°Cから固定チューブを取り外さないでください。適用する前にのみ解凍してください。 - すぐにイメージングする場合は、糸くずの出ないティッシュを使用してスライドを組織の周りで拭き取り、フローセルアセンブリ装置を使用してフローセルを30秒間押して塗布します。後でイメージングする場合は、スライドを(フローセルを適用せずに)4°Cの保存バッファーに保存します。 イメージングの準備ができたら、スライドをストレージバッファーからPBSに10分間移してから、フローセルを適用します。
- 実験で希望するサイクルに基づいて、添加剤を含むランニングバッファー1xの調製を開始します。低DMSOバッファ(1:4)と高DMSOバッファ(9:1)を調製するには、1液型DMSOと4液性ランニングバッファー、および9液型DMSOと1液性ランニングバッファーを所望のサイクルに混合します。レポータープレートを作るための添加剤を含む1xバッファーを約20 mL保存してください。添加剤および高/低DMSOバッファーを含む必要な1xバッファーを計算するには、機器マネージャーにご相談ください。
- レポータープレートの準備を始めます。まず、総サイクル数に必要なレポーターストックソリューションを作成します。黒色または琥珀色の1 mL微量遠心チューブにサイクル名をラベル付けします。250 μLのレポーターストック溶液と各レポーター5 μLを対応するチューブに加えます。次に、溶液を黒色の96ウェルプレートに移し、接着ホイルで密封します。
- デバイスを起動し、機器マネージャーを使用してパラメータと露光時間(表1を参照)を設定し、画像を取得します。 図1 は、染色前と染色後のプロセスをまとめたものです。
5. 組織アノテーションと細胞セグメンテーション
- プロジェクトとワークスペースを設定します。
- 最新バージョンのデジタルパソロジー解析ソフトウェア(QuPathバージョン0.5.1以降など)をインストールします。 [プロジェクトの作成 ] をクリックして、プロジェクト スペースの保存先フォルダーを選択します。
- 「 Add images」>「Choose files」をクリックし、QPTIFFファイル(マルチプレックス免疫蛍光イメージングから生成)に移動します。画像の種類を [蛍光] に設定し、他のすべてのデフォルト設定をそのままにして、[ インポート] をクリックします。
- QuPathを使用している場合は、新しい画像をダブルクリックしてワークスペースを開きます。 「ファイル」>「保存 」を定期的にクリックして、プロジェクトに加えられた変更を追跡します。マーカーの表示設定と表示設定を切り替えるには、ツールバーの中央付近にある Brightness & contrast (明るさとコントラスト ) (半月形のアイコン) を使用します。
注: 画像リスト内の画像を右クリックして、名前を変更します。これは、後で表現型の分類を視覚化するときに重要になります。
- 全組織切片、腫瘍内コンパートメント、および間質コンパートメントに注釈を付けます。
- ブラシやワンドツールを使用して、ティッシュ部分全体に注釈を描きます。このアノテーションを Full_Tissue として定義します。その上にある皮膚や分析すべきでない領域は除外します。ネガティブアノテーションを作成したり、アノテーションの境界を縮小したりするには、 Alt キーを押しながらアノテーションツールの1つを使用します。
- 完全な組織アノテーションを複製します。「Annotations」タブで 「Annotations 」を選択し、「 Objects > Annotations...」に移動します。> 選択した注釈を複製します。
- SOX10チャンネルをオンにします。複製された注釈で、注釈の境界を縮小し、Alt + ブラシ/ワンドツールを使用して腫瘍内コンパートメントをキャプチャします。このアノテーションを腫瘍として定義します(図2)。
- 完全な組織アノテーションを選択し、[ オブジェクト] > [アノテーション...] に移動します。> 注釈を展開します。 Expansion radius を1 μmとして定義し 、Runをクリックします。
- 新しい注釈の名前を Full_Tissue_Expansion に変更します。 腫瘍注釈を選択し、右クリックして、 階層に挿入を選択します。
- 腫瘍の注釈を再度選択し、[オブジェクト] > [注釈...] に移動します。> 逆にします。この新しいアノテーションを Stroma として定義します。すべての組織切片について繰り返します。
- セルセグメンテーションを実行し、結果をエクスポートします。
- [Image] タブに移動して、画像の幅と高さのピクセルを確認します (図 3A)。
- StarDist拡張機能を https://github.com/qupath/qupath-extension-stardist/releases でダウンロードします。qupath-extension-stardist-[version].jarファイルをQuPathウィンドウにドラッグし、QuPathウィンドウの右上隅にある歯車のアイコンをクリックします。 [拡張機能]で、 QuPathユーザーディレクトリ をStarDistフォルダーの場所として定義します。
- StarDist groovy スクリプト ファイルと StarDist モデル ファイルを https://github.com/xzhou125/JOVE_QuPath_Spatial_Analysis からダウンロードします (StarDist スクリプトはもともと Akoya Biosciences から編成されました)。
- 各組織切片について、Tumor と Stroma の両方のアノテーションを選択します (複数のアノテーションを選択するには Ctrl キーを使用します)。上部の設定バーで、[スクリプトエディタの自動化]をクリックして>スクリプトインターフェイスを開きます。画像のピクセルサイズに対応する適切なStarDistセルセグメンテーションスクリプト(Groovyファイル)を開きます。セル セグメンテーション ファイルを選択するように求められたら、stardist_cell_seg_model.pb ファイルを開きます。
- セルセグメンテーション後、画像を保存します。 [測定] > [測定値のエクスポート] に移動し、 対応する画像を選択します。[エクスポートの種類] を [セル] に、[区切り記号] を [.csv] に割り当て、[ 出力ファイル の場所] を選択します。[列] の横にある [入力] をクリックして、関心のあるメトリックを含めます。エクスポートする重要な列は、画像、オブジェクト ID、親、重心 X、重心 Y、および核領域です。
注:オブジェクトIDは、後続のステップで、デジタルパソロジー解析ソフトウェアのマルチプレックス免疫蛍光画像に分類を再マッピングするために使用されます。「親」列は、セルが属する注釈名を示します。X データと Y データは、空間解析に使用されます。 - クラスタリング/表現型解析で使用する系統マーカーごとに、エクスポートする平均値を1つ選択します。核マーカー(SOX10やFOXP3など)の場合は、Nucleus:Meanオプションをエクスポートします。細胞質/膜性マーカー(CD45、CD3、CD4など)の場合は、Cytoplasm:Meanオプションをエクスポートします。
6. プロテオミクスデータの前処理と正規化
- エクスポートしたCSVファイルを開きます。クラスタリング/表現型分析で使用するマーカーごとに、列見出しの名前を切り捨ててマーカーのみを含めます。たとえば、名前を SOX10: Nucleus: Mean に変更し、名前を [CD3: Cytoplasm: Mean] に変更します。
- 最新バージョンの R と RStudio をインストールします。https://github.com/xzhou125/JOVE_QuPath_Spatial_Analysis から Marker Normalization.R ファイルをダウンロードし、スクリプトを実行します。これには、まず、核サイズによる細胞のフィルタリングが含まれます。
- 次に、マーカーごとに最小-最大正規化が実行され、式の範囲内の最小の MFI 値が 0 に設定され、99.7パー センタイルの MFI 値が 1 に設定されます。99.7パーセン タイルを超えるすべての強度は1でクリップされます。完了したら、正規化されたデータで生成された新しい CSV ファイルを保存します。
7. クラスタリングと表現型
- マーカー発現プロファイルに従ってセルをクラスタリングするアルゴリズムを選択します。https://github.com/xzhou125/JOVE_QuPath_Spatial_Analysis では、Seurat v4.415 およびFlowSOM v2.13.9(クラスタリングおよび視覚化ツール)16を使用したクラスタリング用のRスクリプトが提供されています。
- 主要な系統マーカーのマーカー発現プロファイルに従った細胞表現型。ここで紹介するプロトコルでは、クラスタリングと表現型解析に14の系統マーカーを使用しました:SOX10、CD45、CD3、CD4、CD8、FOXP3、CD11C、F4/80、CD68、CD86、CD163、CD206、NK1.1、CD31。
- 腫瘍細胞をSOX10hiと定義します。CD4 T細胞をCD45mod-hi/CD4hi/CD8low/FOXP3lowと定義します。CD8 T細胞をCD45mod-hi/CD8hi/CD4lowと定義します。制御性をCD45mod-hi/CD4hi/CD8low/FOXP3hiと定義します。他のT細胞をCD45mod-hi/CD3hi/CD4low/CD8lowと定義します。
- M1マクロファージをCD45mod-hi/F4/80hi/CD68hi/CD86hi/CD163low /CD206lowと定義します。M2マクロファージをCD45mod-hi/F4/80hi/CD68mod-hi/CD86low/CD163hi/CD206low、CD45mod-hi/F4/80hi/CD68mod-hi/CD86low/CD163low/CD206hi、またはCD45mod-hi/F4/80hi/CD68mod-hi/CD86low/CD163hi/CD206hiと定義します。他のマクロファージをCD45mod-hi/F4/80hi/CD68mod-hi/CD86low/CD163low/CD206low またはCD45mod-hi/F4/80hi/CD68mod-hi/CD86hi/CD163hi/CD206hiと定義します。
- 樹状細胞をCD45mod-hi/CD11chi/CD86mod-hiと定義します。NK細胞をCD45mod-hi/NK1.1hiと定義します。他の免疫細胞を、上記の条件を満たさなかったCD45mod-hi と定義します。内皮細胞をCD31mod-hiと定義します。他のすべての発現プロファイルを削除します。
8. 表現型分類の再マッピングと品質管理の実施
- https://github.com/xzhou125/JOVE_QuPath_Spatial_Analysis から Remapping Classifications.groovy スクリプトをダウンロードします (このスクリプトは petebankhead による https://forum.image.sc/ の投稿から改作されました)。
- 表現型が設定されたCSVファイルを開き、[Image]タブのすべてのラベルが、再マッピングに使用されるデジタルパソロジー解析ソフトウェアの画像ファイルの名前と完全に一致していることを確認します。CSV ファイル内の大量のラベルをすばやく変更するには、[すべて置換] 機能を使用します。
- QuPathを使用している場合は、 スクリプトエディターで「自動化」をクリックして、groovyファイルの再マッピング>分類を実行します。
- [注釈] タブで、[自動設定] ボタンの横にある 3 つのドット をクリックし、[ 既存のオブジェクトから入力] > [すべてのクラス (サブクラスを含む)] > [はい] をクリックします。
- マーカーリストを下にスクロールして、さまざまな表現型分類を表示し、必要に応じて配色を調整します。任意のセルをダブルクリックすると、その表現型(Classificationタグの横)とクラスター(Nameタグの横)が表示されます。代表的な視野を確認して、表現型の割り当ての精度を判断し、必要に応じて表現型戦略を修正します(図3B)。
9. 密度定量化と空間分析
- 表現型が確定したら、デジタルパソロジー解析ソフトウェアの「アノテーション」タブでアノテーション領域を取得します。面積の値を mm2 に変換し、カウントを注釈面積で除算して密度を計算します。
- https://github.com/xzhou125/JOVE_QuPath_Spatial_Analysis から SPIAT Spatial Analysis.R スクリプトをダウンロードして実行します。平均最小距離 (AMD)、近傍構成、正規化混合スコア (NMS) に関するデータを収集します。
- 統計分析ソフトウェアで数値を生成して、調査結果を提示します。治療条件の平均最小距離について単一のヒートマップを生成する場合は、異なる組織切片から得られた結果を平均化します。
結果
ここでは、マウスFFPE組織用の抗体パネルを設計し、マルチプレックス免疫蛍光イメージングを実施し、プロテオミクスの定量化と空間的関係のための画像解析を行うためのプロトコルを紹介します。検証済みのパネルには、黒色腫細胞(SOX10)、白血球(CD45)、T細胞(CD3、CD4、CD8、FOXP3)、B細胞(CD20)、マクロファージおよびサブタイプ(F4/80、CD68、CD86、CD163、CD206)、樹状細胞(CD11c)、NK細胞(NK1.1)、および内皮細胞(CD31)を視覚化するためのマーカーを提供する27の抗体が含まれています。全パネルには、他の免疫集団(CD11b、CD38)、増殖活性(Ki67)、T細胞機能(T-bet、Eomesodermin、granzyme B)、抗原提示(LMP2、β-2ミクログロブリン、MHC II)、およびチェックポイント発現(TIM3、LAG3、PD-L1)12のマーカーも含まれています。代表的なゲル電気泳動画像は、 図4に示すように、DNAオリゴヌクレオチドバーコードのキャリアフリー抗体への結合が成功したことを確認しています。このステップは化学反応を確認するだけであり、画像の確認は、これらの抗体を目的の組織でマルチプレックスイメージングデバイスを使用して確認した後にのみ行うことができます。 パネル13内の27個のマーカーすべての検証済み画像を表示するには、次のリファレンスを参照してください。腫瘍内4-1BBL/IL-12ナノ粒子注射と全身性抗PD1で治療した3つのマウス黒色腫組織切片を染色した主要な系統マーカーの融合画像を 図5に示します。これらの切片は、このプロトコルでのその後の画像解析に使用されました。
組織アノテーション、細胞セグメンテーション、およびプロテオミクスデータの前処理の後、シングルセルトランスクリプトームおよび/またはプロテオミクス解析17,18に以前に適用された2つのクラスタリングアルゴリズムの比較を提示する。表現型集団の発現プロファイルを、両方のアプローチについて図6に示します。FlowSOMは、マクロファージのサブタイプなど、類似した細胞集団間の違いを識別するために、より広い範囲の強度値(~0.7対~0.5)を提供することを示しています。Seurat はクラスタリング中に Louvain アルゴリズムを適用し、29 個のクラスターを生成しました (補足図 1)。これに対し、FlowSOMは自己組織化マップを適用し、100個のクラスターを生成することができます(補足図2)。クラスターの数が多いほど、表現型解析に必要な時間が長くなりますが、この後者のFlowSOMアプローチでは、類似した細胞集団を分類する際に、より微妙なニュアンスが得られます。定性的には、同じ視野で FlowSOM は、Seurat 表現型と比較して、より多くの腫瘍内マクロファージを M1 または M2 サブタイプとして分類できたことがわかります (図 7)。マクロファージ密度を定量化しても同じ結果が得られ、FlowSOMはSeuratと比較してM1マクロファージとM2マクロファージの両方の密度が高く(図8A-B)、その後、他の/未分類のマクロファージの密度が大幅に低下しました(p = 0.0028;図8C)。それにもかかわらず、2つの解析アプローチは、CD4 T細胞、CD8 T細胞、内皮細胞などの他の細胞集団密度を説明する際にも同様の結果をもたらしました(図8D-F)。
また、クラスタリングと表現型解析後の下流の空間解析結果も示します。 図 9 は、このプロトコルを使用して生成できる空間メトリックの一部を示しています。すべての表現型集団と比較して、M2マクロファージとNK細胞は、4-1BBL/IL-12ナノ粒子と抗PD1による処理後にAMDが最も高かった(図9A)。同様に、腫瘍内CD8 T細胞とM1マクロファージとの間のNMSは、CD8 T細胞とM2マクロファージとの間のNMSよりもはるかに高かった(図9B)。さらに、M2マクロファージは、腫瘍内CD8 T細胞を囲む100μm近傍に~1%寄与したのに対し、M1マクロファージはこれらの近傍の9%〜13%を占めていました(図9C)。まとめると、これらの結果は、4-1BBL/IL-12治療レジメンが腫瘍関連マクロファージをM1サブタイプに分極させ、M2マクロファージを腫瘍免疫微小環境から除外したことを示唆しています。
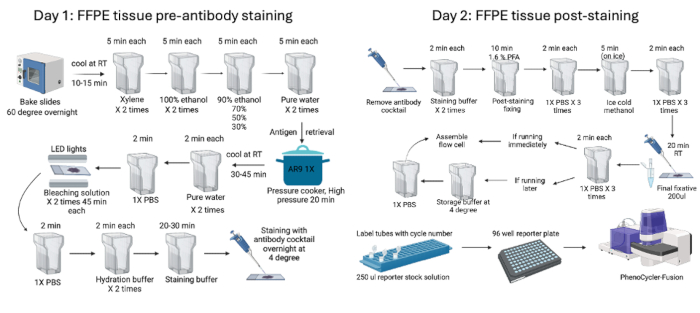
図1:FFPE組織染色およびイメージングワークフローの要約。 マウスFFPE組織は、組織を一晩焼くことから始まり、2日間の染色前(1日目)および染色後(2日目)のプロセスを開始する前に、染色前手順を使用して処理されました。最後に、レポータープレートを調製してから、装置でイメージングしました。 この図の拡大版を表示するには、ここをクリックしてください。
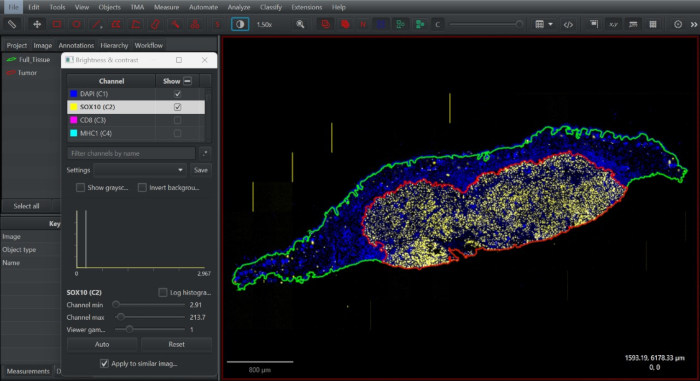
図2:オープンアクセスのデジタルパソロジーソフトウェアQuPathの組織アノテーションのスクリーンショット。 全組織アノテーションを描画(緑)し、複製し、最小化して、腫瘍境界(赤)でのSOX10+メラノサイトの分布に従って腫瘍内コンパートメントを定義しました。 この図の拡大版を表示するには、ここをクリックしてください。
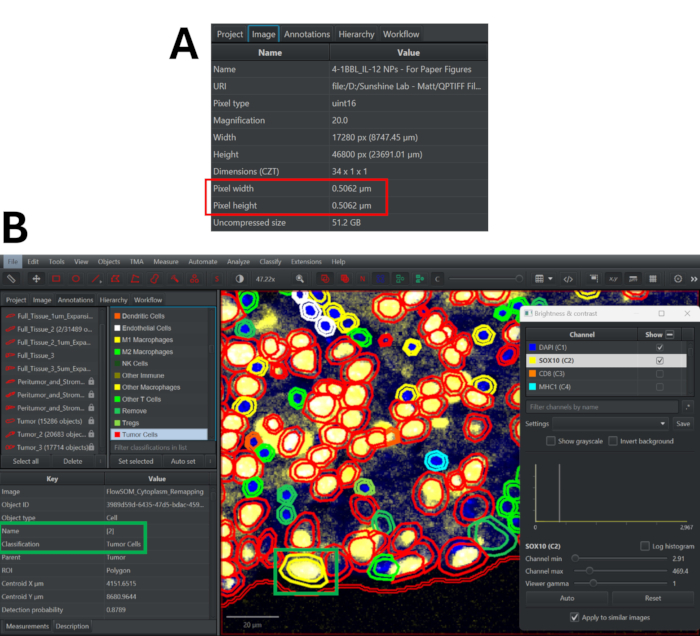
図3:QuPathのStarDistアルゴリズムを使用した細胞セグメンテーションと、表現型分類のレビューのための品質管理プロセス (A)[Image]タブに移動して、マルチプレックス免疫蛍光画像のピクセル幅と高さを決定します。(B)分類を再マッピングした後、任意のセル(黄色で強調表示されている)をダブルクリックして、そのクラスターの割り当てと表現型を確認します。パネルマーカーのオン/オフを切り替えて、この分類アプローチが正確かどうかを判断します。 この図の拡大版を表示するには、ここをクリックしてください。
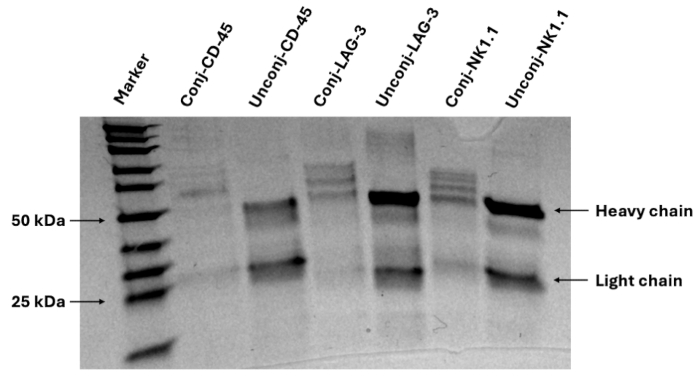
図4:抗体-DNAバーコード標識確認の代表的なゲル画像。 タンパク質ゲル電気泳動により、DNAオリゴヌクレオチドバーコードによる抗体の結合が確認され、重鎖部位に追加のバンドが観察されます。 この図の拡大版を表示するには、ここをクリックしてください。
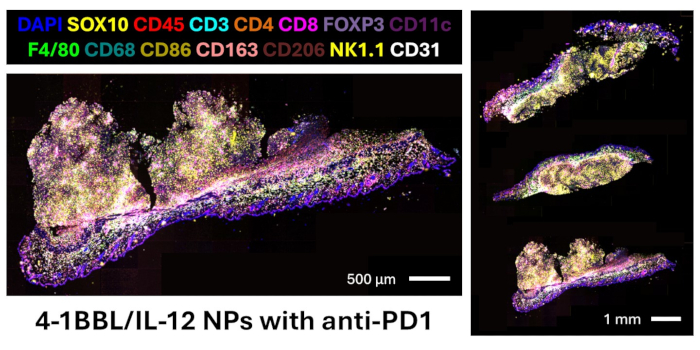
図5:全身性抗PD1を用いた4-1BBL/IL-12ナノ粒子の腫瘍内注射で治療したB16F10側面腫瘍のDNAバーコードベースのマルチプレックスイメージング。 私たちのパネルのマーカーは示されていません:CD11b、CD20、CD38、TIM3、LAG3、T-bet、エオメソデルミン、グランザイムB、Ki67、LMP2、ベータ-2ミクログロブリン、MHC II、PD-L1。 この図の拡大版を表示するには、ここをクリックしてください。
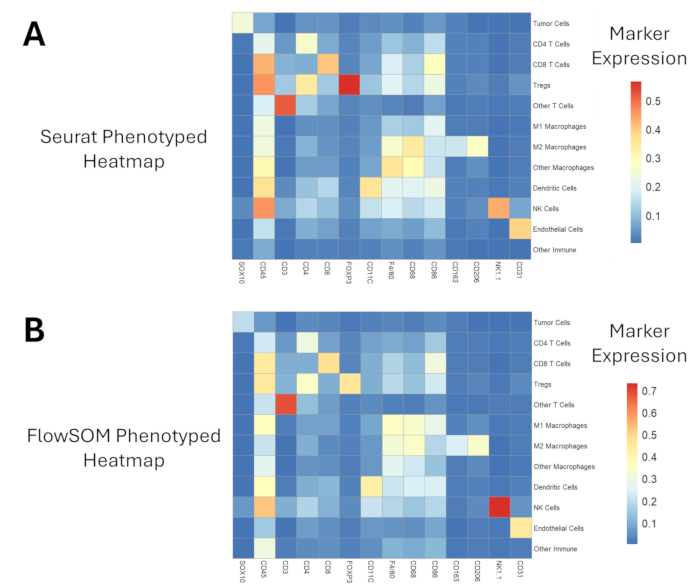
図6:表現型が付けられた細胞集団のプロテオミクス発現ヒートマップ。 (A)Seuratクラスタリングおよび表現型解析アプローチは、(B)FlowSOMクラスタリングおよび表現型解析と比較して、マーカー発現値の範囲がわずかに小さくなります。 この図の拡大版を表示するには、ここをクリックしてください。
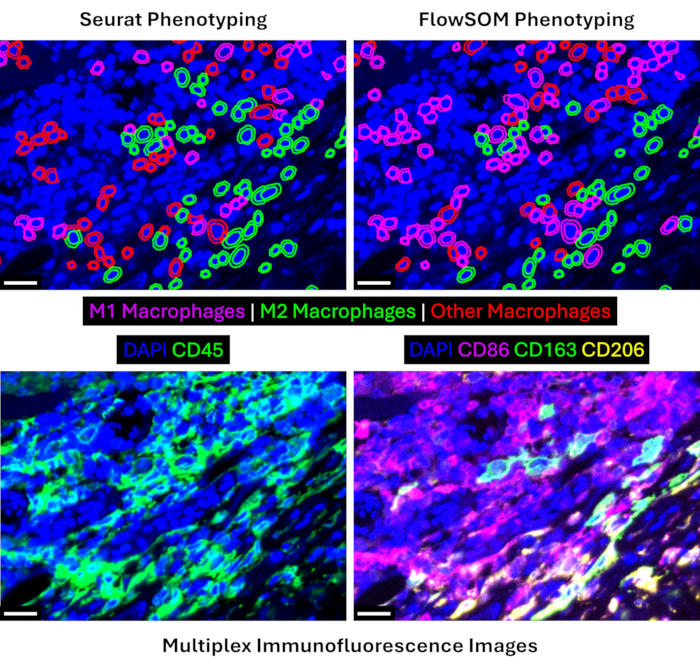
図7:FlowSOMの表現型は、より広範なマクロファージの異なるサブタイプを同定します。 トップパネルは、同じ視野でのSeurat(左)とFlowSOM(右)のマクロファージ表現型を示しています。下のパネルは、同じ視野での主要なマクロファージ系統マーカーのマルチプレックス免疫蛍光画像を示しています。すべてのスケールバーは20μmです。 この図の拡大版を表示するには、ここをクリックしてください。
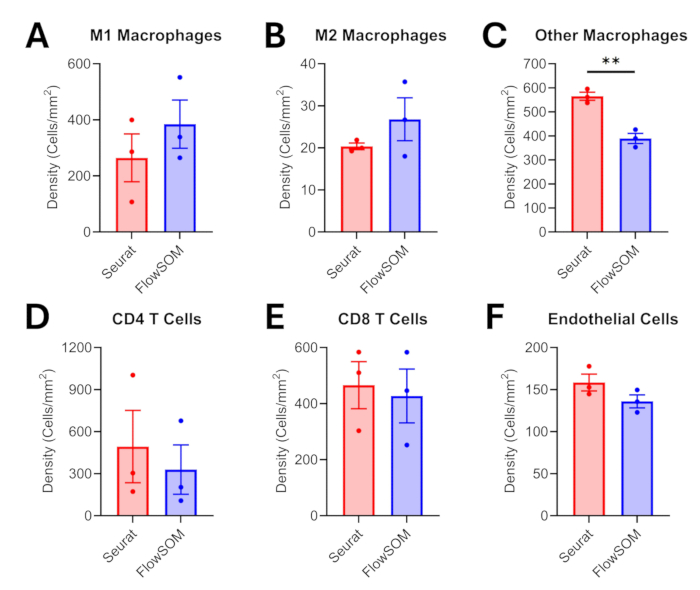
図8:クラスタリング/表現型解析後の異なる細胞集団の腫瘍内密度の比較。 腫瘍内(A)M1マクロファージ、(B)M2マクロファージ、(C)その他のマクロファージ、(D)CD4 T細胞、(E)CD8 T細胞、および(F)内皮細胞について密度比較が示されています。エラーバーは平均の標準誤差(SEM)であり、有意性は対応のないt検定(**p ≤ 0.01)を使用して検定されました。 この図の拡大版を表示するには、ここをクリックしてください。
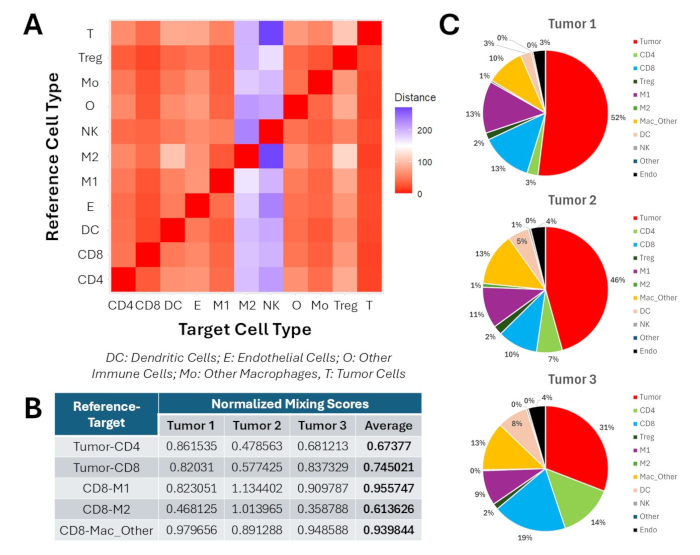
図9:4-1BBL / IL-12腫瘍内ナノ粒子注射と全身性抗PD1で治療された3つの側面腫瘍の腫瘍内空間メトリックのプロファイリング (A)腫瘍内表現型集団間の平均最小距離のヒートマップ。測定値はμmです。(B)主要な腫瘍内表現型ペア間の正規化された混合スコア。(C)腫瘍内CD8 T細胞集団の半径100μmにおける近傍組成の内訳。 この図の拡大版を表示するには、ここをクリックしてください。
| 抗体 | 希釈 | 露光時間(ms) |
| ソックス-10 | 50 | 450 |
| CD8 | 100 | 450 |
| CD3 | 100 | 450 |
| フォックスP3 | 50 | 350 |
| CD4の | 50 | 450 |
| MHC-II.(英語) | 100 | 450 |
| PDL1の | 50 | 450 |
| CD45の | 100 | 450 |
| キ67 | 100 | 300 |
| F4/80 | 100 | 150 |
| CD20の | 100 | 450 |
| NK1.1/CD161 | 50 | 450 |
| CD206の | 100 | 450 |
| CD68 | 100 | 450 |
| グランザイム-B | 50 | 450 |
| CD86の | 100 | 150 |
| CD31 | 100 | 450 |
| CD11C | 50 | 450 |
| CD11bの | 50 | 450 |
| EOMES(エオメス) | 50 | 450 |
| ティム-3 | 50 | 300 |
| CD38の | 50 | 200 |
| ラグ3 | 50 | 450 |
| CD163の | 50 | 200 |
| T-ベット | 50 | 300 |
| LMP2の | 100 | 100 |
| ベータ2 MG | 200 | 50 |
表1:抗体の希釈と曝露時間の設定。
補足 図1:Seuratクラスタリング後に生成された初期プロテオミクス発現ヒートマップ。 この図をダウンロードするには、ここをクリックしてください。
補足図2:FlowSOMクラスタリング後に生成された初期プロテオミクス発現ヒートマップ この図をダウンロードするには、ここをクリックしてください。
ディスカッション
イメージングの成功は、適切に設計され、検証された抗体パネルにかかっています。FFPEサンプルのマルチプレックス免疫蛍光イメージングは、高い自家蛍光とパラフィン包埋によってマスクされたエピトープの取得が困難であるため、課題があります。しかし、FFPEはFF検体と比較していくつかの利点があるため、FFPE抗体パネルの設計と検証が不可欠です。免疫蛍光(IF)イメージング中に陽性のシグナルを示す抗体クローンを最終決定することが最初のステップです。その後、それらをDNAバーコードで慎重に結合することが重要です。抗体の標識では、抗体を部分的に還元してSH結合を作製する必要があり、バーコードによるマレイミド基反応中にSH結合が利用されます。すべての抗体クローンがこのステップに耐えられるわけではなく、一部の反応は抗体に不可逆的な損傷を引き起こし、標識が成功したにもかかわらずイメージングが失敗する可能性があります。このため、従来のIFバリデーションでは一部の抗体が陽性のシグナルを示すことがありますが、抗体標識の最終的な成功を評価するためには、実際の対象組織で各抗体を検証し、その組織に対する所望の曝露時間を記録することが重要です。将来のアプリケーションでは、この手法を既存の空間トランスクリプトミクス解析と連続したスライド/同じスライドで組み合わせて、さらなる洞察を生み出すことができます。この方法の限界の1つは、ターゲットと組織の種類に基づいて、パネル内の各抗体を慎重に選択し、検証する必要があることです。
画像解析に関しては、QuPathは、プロテオミクスマーカーの高品質な可視化、強度測定値のエクスポートや表現型分類の品質管理のための幅広い機能、ユーザー生成スクリプトの優れた柔軟性を備えた貴重なオープンアクセスツールを提供しています。https://forum.image.sc/ などのオンラインフォーラムは、特定の解析タスクの実行方法について話し合ったり、他のユーザーとスクリプトを共有したりするための追加のリソースです。このプロトコルでは、Seurat と FlowSOM を使用した 2 つのクラスタリングおよび表現型分析アプローチを比較します。FlowSOMは、TMEの免疫細胞亜集団についてより詳細な洞察を得ることができるため、好ましいかもしれませんが、プロテオミクス解析に必要な時間も考慮する必要があります。ユーザーが1つまたは2つの組織サンプル内の細胞を表現型決定するだけでよい場合、100個のクラスターを生成する必要はないかもしれません。このような状況では、Seuratは画像分析のためのより高速で効率的なパイプラインを提供する場合があります。対照的に、40または50以上の組織切片を持つTMAを分析すると、どちらの分析アプローチでもより多くの細胞クラスターが生成される可能性が高く、FlowSOMはより微妙な表現型の分類を生成するための好ましい方法論になる可能性があります。
細胞のクラスタリング/表現型解析とその後の画像解析ステップは、細胞のセグメンテーションに大きく依存します。私たちの現在の研究では、HALO(Indica Labs)アルゴリズムとStarDistアルゴリズムでの細胞セグメンテーションを調査しており、どちらのアプローチも核DAPIシグナルに基づいて細胞を過剰にセグメント化する傾向があることがわかりました。また、Mesmer19 や InstanSeg20 など、多くの代替セグメンテーションアルゴリズムも利用できます。これは、さらなる調査と最適化を必要とする成長中の計算研究分野です。
開示事項
J.C.S.は、Emerson Collective LLCおよび国立衛生研究所からの財政的支援を認めています。J.J.G.は、国立衛生研究所からの資金提供も認めています。J.C.S.はPalleon Pharmaceuticals Inc.と関係があり、助成金の資金提供を行っています。S.Y.T.とJ.J.G.は、OncoSwitch Therapeuticsと株式または株式を含む関係にあります。S.Y.T.、J.J.G.、J.C.S.、およびKMLは特許出願中です。他のすべての著者は、この論文で提示された研究に影響を与えると認識される可能性のある既知の競合する金銭的利益や個人的な関係はないと述べています。
謝辞
J.C.S.は、著者のキャリアを前進させたDermatology FoundationとDermatopathology Career Development Awardを表彰します。著者らは、米国国立がん研究所のがんデータサイエンス研究所のHsin-Pei Lee氏に、計算解析技術の支援に感謝しています。この研究は、エマソンコレクティブと国立衛生研究所(R37CA246699、P41EB028239、およびR01CA228133)から資金提供を受けました。さらに、ジョンズ・ホプキンス大学腫瘍組織サービス(OTS)の中核は、国立衛生研究所(P30CA006973)によってサポートされています。
資料
| Name | Company | Catalog Number | Comments |
| 16% paraformaldehyde (PFA) | Electron Microscopy Sciences | PN# 15710 | Required during tissue staining process |
| 50kDa MWCO filter | Millipore | UFC505096 | 25 kDa and 100 kDa result in failure |
| Akoya barcodes and reporters | Akoya Biosciences | Varied | Each barcode can be used to conjugate 50 ug of carrier free antibody and comes with two vials of reporters |
| Antibody conjugation kit | Akoya Biosciences | 7000009 | A conjugation kit is used to create custom barcode-conjugated antibodies. Each kit contains sufficient reagents for ten conjugations. The kit consists of one subkit box stored at 4 °C and one subkit box stored at -20 °C. The 4 °C subkit includes Filter Blocking Solution, Reduction Solution 2, Conjugation Solution, Purification Solution, and Antibody Storage Solution. The -20 °C subkit includes Reduction Solution 1. |
| Beta2MG | Abcam | ab214769 | |
| CD11b | CST | #41028 | |
| CD11C | CST | #39143SF | |
| CD163 | CST | #68922BF | |
| CD20 | CST | #45839 | |
| CD206 | CST | #87887 | |
| CD3 | CST | #24581 | |
| CD31 | CST | #92841 | |
| CD38 | CST | #68336BF | |
| CD4 | Abcam | ab271945 | |
| CD45 | CST | #98819 | |
| CD68 | CST | #29176 | |
| CD8 | Invitrogen | #14-0808-82 | |
| CD86 | CST | #20018 | |
| Dimethyl sulfoxide | Avantor/VWR | BDH1115-4LP | |
| EOMES | Abcam | ab222226 | |
| Eppendorf PCR tubes | Eppendorf | E0030124286 | Required store 5 μL conjugated antibody for conjugation confirmation by gel electrophoresis |
| Ethanol 200 proof | |||
| F4/80 | CST | # 25514 | |
| FoxP3 | CST | #72338 | |
| Gran-B | CST | #79903SF | |
| Heating oven | |||
| Hybridization chamber | Used to stain tissue with antibody cocktail | ||
| Hydrogen peroxide | Sigma | #216763 | Required for preparing bleaching solution |
| Instant pot pressure cooker or Decloaking Chamber ARC | Instant pot or Biocare Medical | ||
| Ki-67 | Akoya Biosciences | ||
| LAG3 | CST | #80282BF | |
| LMP2 | Abcam | ab243556 | |
| Methanol | |||
| MHC-II | eBioscience | #14-5321-82 | |
| NK1.1 | CST | #24395SF | |
| Nuclear Stain | Akoya Biosciences | 7000003 | |
| PDL1 | CST | #85095 | |
| Salmon Sperm DNA, sheared (10 mg/mL) | Invitrogen | AM9680 | Can be used as an alternative to assay reagent (Akoya) during reporter plate preparation step. |
| SOX-10 | Abcam | ab245760 | |
| Staining kit for PhenoCycler-Fusion | Akoya Biosciences | 7000017 | The PhenoCycler-Fusion Sample Kit includes the buffers and reagents necessary for tissue staining using antibodies conjugated with PhenoCycler barcodes, along with flow cells for whole-slide imaging. Each kit is sufficient for 10 PhenoCycler-Fusion experiments. It consists of one subkit stored at 4 °C, another at -20 °C, and a package of 10 flow cells kept at room temperature. The subkit stored at 4 °C contains Hydration Buffer, Staining Buffer, Storage Buffer, N Blocker, and J Blocker. The subkit stored at -20 °C contains G Blocker, S Blocker, and Fixative Reagent. |
| T-bet | CST | #53753 | |
| TIM-3 | CST | #72911 | |
| UltraPure DNase/RNase-free distilled water | Invitrogen | 10977015 | Required to dissolve barcodes during antibody conjugation |
参考文献
- Schadendorf, D., et al. Melanoma. Lancet. 392 (10151), 971-984 (2018).
- Taube, J. M., et al. Implications of the tumor immune microenvironment for staging and therapeutics. Modern Pathol. 31 (2), 214-234 (2018).
- Marzagalli, M., Ebelt, N. D., Manuel, E. R. Seminars in cancer biology. 59, 236-250 (2019).
- De Visser, K. E., Joyce, J. A. The evolving tumor microenvironment: From cancer initiation to metastatic outgrowth. Cancer cell. 41 (3), 374-403 (2023).
- Balkwill, F. R., Capasso, M., Hagemann, T. The tumor microenvironment at a glance. J Cell Sci. 125 (23), 5591-5596 (2012).
- Phillips, D., et al. Highly multiplexed phenotyping of immunoregulatory proteins in the tumor microenvironment by codex tissue imaging. Front Immunol. 12, 687673(2021).
- Giesen, C., et al. Highly multiplexed imaging of tumor tissues with subcellular resolution by mass cytometry. Nat Methods. 11 (4), 417-422 (2014).
- Keren, L., et al. Mibi-tof: A multiplexed imaging platform relates cellular phenotypes and tissue structure. Sci Adv. 5 (10), eaax5851(2019).
- Lin, J. R., Fallahi-Sichani, M., Sorger, P. K. Highly multiplexed imaging of single cells using a high-throughput cyclic immunofluorescence method. Nat Comm. 6 (1), 8390(2015).
- Radtke, A. J., et al. Ibex: An iterative immunolabeling and chemical bleaching method for high-content imaging of diverse tissues. Nat Protoc. 17 (2), 378-401 (2022).
- Black, S., et al. Codex multiplexed tissue imaging with DNA-conjugated antibodies. Nat Protoc. 16 (8), 3802-3835 (2021).
- Aung, T. N., Bates, K. M., Rimm, D. L. High-plex assessment of biomarkers in tumors. Mod Pathol. 37 (3), 100425(2024).
- Surwase, S. S., et al. Highly-multiplexed immunofluorescence phenocycler panel for murine ffpe yields insight into tumor microenvironment immunoengineering. Lab Invest. 105 (1), 102165(2024).
- Luly, K. M., Green, J. J., Sunshine, J. C., Tzeng, S. Y. Biomaterial-mediated genetic reprogramming of merkel cell carcinoma and melanoma leads to targeted cancer cell killing in vitro and in vivo. ACS Biomater Sci Eng. 9 (11), 6438-6450 (2023).
- Satija, R., Farrell, J. A., Gennert, D., Schier, A. F., Regev, A. Spatial reconstruction of single-cell gene expression data. Nat Biotechnol. 33 (5), 495-502 (2015).
- Van Gassen, S., et al. Flowsom: Using self-organizing maps for visualization and interpretation of cytometry data. Cytometry Part A. 87 (7), 636-645 (2015).
- Butler, A., Hoffman, P., Smibert, P., Papalexi, E., Satija, R. Integrating single-cell transcriptomic data across different conditions, technologies, and species. Nat Biotechnol. 36 (5), 411-420 (2018).
- Watson, S. S., et al. Microenvironmental reorganization in brain tumors following radiotherapy and recurrence revealed by hyperplexed immunofluorescence imaging. Nat Comm. 15 (1), 3226(2024).
- Greenwald, N. F., et al. Whole-cell segmentation of tissue images with human-level performance using large-scale data annotation and deep learning. Nat Biotechnol. 40 (4), 555-565 (2022).
- Goldsborough, T., et al. Instanseg: An embedding-based instance segmentation algorithm optimized for accurate, efficient and portable cell segmentation. arXiv. , (2024).
転載および許可
このJoVE論文のテキスト又は図を再利用するための許可を申請します
許可を申請さらに記事を探す
This article has been published
Video Coming Soon
Copyright © 2023 MyJoVE Corporation. All rights reserved