Method Article
Полногеномный CRISPR-скрининг для выявления радиочувствительных и радиорезистентных генов
* Эти авторы внесли равный вклад
В этой статье
Резюме
Тщательный и структурированный подход применяется к отбору устойчивых и чувствительных генов радиации с помощью метода полногеномного скрининга CRISPR/Cas9. Этот протокол также может послужить универсальной основой для других исследований, изучающих механизмы устойчивости к клинически вводимым химическим препаратам.
Аннотация
Система CRISPR-Cas9 была использована и перепрофилирована в мощный инструмент редактирования генома. Используя эту технологию, исследователи могут точно вырезать, вставлять и даже переписывать последовательности ДНК в живых клетках. Тем не менее, применение технологии экранов CRISPR выходит далеко за рамки простых экспериментов. Он служит ключевым инструментом в борьбе с генетическими заболеваниями, систематически анализируя сложные генетические ландшафты, позволяя исследователям раскрывать молекулярные механизмы, лежащие в основе биологических явлений, и позволяя ученым выявлять и устранять первопричины таких заболеваний, как рак, муковисцидоз и серповидноклеточная анемия. Помимо всего прочего, рак представляет собой сложнейшую проблему для медицины, стимулируя усилия по его ликвидации. Лучевая терапия, как и традиционное лечение, дает результаты, но имеет ограничения. Он уничтожает раковые клетки, но также повреждает здоровые ткани, вызывая неблагоприятные последствия, снижающие качество жизни. Кроме того, не все раковые клетки реагируют на лучевую терапию, а некоторые могут развить резистентность, что ухудшает состояние. Для решения этой проблемы внедряется комплексная технология полногеномного скрининга CRISPR, которая позволяет эффективно идентифицировать радиочувствительные и радиорезистентные гены, тем самым продвигая область исследований и лечения рака. В клетках аденокарциномы легкого, подвергшихся облучению, был проведен полногеномный скрининг CRISPR в соответствии с описанным протоколом, с помощью которого были идентифицированы гены, ассоциированные с радиорезистентностью и радиочувствительностью.
Введение
Изучение биологических явлений неразрывно связано с изучением клеточного поведения, и, в свою очередь, изучение клеточного поведения фундаментально связано с исследованием его генома. По мере того, как современные технологии продолжают развиваться, медицинские исследователи все больше перенаправляют свое внимание на изменение клеточного поведения с помощью редактирования генов, чтобы улучшить результаты лечения различных заболеваний. В связи с этим технология кластеризованных регулярных чередующихся коротких палиндромных повторов (CRISPR) стала революционным инструментом редактирования генома благодаря своей относительнопростой области применения. Система CRISPR-Cas9 состоит из нуклеазы Cas9 и однонаправляющей РНК (sgRNA), которая специфически распознает и связывается с целевой последовательностью ДНК, направляя нуклеазу Cas9 на разрезание в этом месте, что приводит к двухцепочечному разрыву (DSB) в ДНК генома 2,3,4. Кроме того, введение других веществ может привести к специфическим вставкам, делециям или мутациям в геноме, что позволяет целенаправленно редактировать гены.
В исследованиях функциональной геномики скрининг РНК-интерференции (РНК-интерференции) когда-то был широко используемым методом для проведения крупномасштабных экспериментов по потере функций для изучения роли генов в развитии рака. Технология РНК-интерференции изучает функцию генов, целенаправленно подавляя целевые гены, помогая исследователям выявлять критические онкогенные факторы. Однако он ограничен нецелевыми эффектами и неполной эффективностью нокдауна генов. Побочные эффекты могут приводить к подавлению других нецелевых генов, тем самым ставя под угрозу точность и надежность экспериментальных результатов 1,2. Кроме того, РНК-интерференция демонстрирует низкую эффективность нокдауна для определенных генов, потенциально не в состоянии полностью подавить экспрессию генов-мишеней. В отличие от традиционных РНК-интерференционных экранов, CRISPR-скрининг демонстрирует более высокую специфичность и эффективность3. Эта технология не только позволяет точно редактировать определенные гены, но и позволяет проводить широкомасштабный скрининг по всему геному, обеспечивая надежную поддержку для исследований функций генов. Технология скрининга CRISPR, мощный инструмент редактирования генов на основе системы CRISPR-Cas9, используется для эффективного скрининга и выявления неизвестных функций конкретных генов в клетках 5,6,7,8. Исследователи проектируют sgРНК партиями для конкретных генов или областей генов и готовят соответствующие библиотеки sgRNA с точностью и строгостью, обеспечивая их целостностьи функциональность. Эти библиотеки sgRNA затем инкапсулируются в лентивирусные частицы, которые используются для эффективного заражения клеток-хозяев. После успешной инфекции инфицированные клетки культивируются в соответствии с индивидуально определенными условиями скрининга. После скрининга извлекается геномная ДНК прошедших скрининг клеток, при этом поддерживаются высокие стандарты чистоты и количества. Впоследствии целевые участки, представляющие интерес для sgRNA, подвергают амплификации ПЦР, процессу, который точно воспроизводит желаемые сегменты нуклеиновых кислот 3,9. Наконец, высокопроизводительное секвенирование амплифицированных фрагментов ДНК выполняется с высокой пропускной способностью, что позволяет проводить всесторонний и эффективный анализ целевых областей, что позволяет получить ценную информацию о функции иповедении изучаемых генов.
Рак представляет собой огромную угрозу для здоровья человека как сложное заболевание. Во всем мире исследователи и клиницисты прилагают согласованные усилия для разгадки молекулярных механизмов канцерогенеза и разработки новых терапевтических стратегий. Международное сотрудничество было налажено для ускорения преобразования результатов фундаментальных исследований в клиническое применение с конечной целью улучшения результатов лечения пациентов. Сасмал и др. предложили биоортогональную стратегию сборки, основанную на синтетической системе хозяин-гость для точного нацеливания на метастатические раковые клетки, что значительно помогло десяткам ученых в развитии медицинских технологий. Их выдающаяся исследовательская работа отличается высокой инновационностью и уникальными идеями, внося значимый вклад в научное сообщество10. Рак характеризуется бурным состоянием геномной нестабильности, возникающим в результате беспорядочной регуляции реакций на повреждение ДНК11-14. Повреждение ДНК включает однонуклеотидные дефекты, одноцепочечные разрывы и DSB. Гомологичная рекомбинация (HR) и негомологичное соединение концов (NHEJ) участвуют в репарации DSB на разных стадиях 15,16,17. Исходя из этого, лучевая терапия стала жизнеспособным вариантом лечения, в котором используются высокоэнергетические лучи (такие как рентгеновские и γ-лучи) для облучения опухолевой ткани, вызывающей повреждение ДНК в опухолевых клетках, тем самым нарушая их рости пролиферацию. Тем не менее, лучевая терапия не всегда дает желаемые эффекты у значительной части онкологических больных, что может быть результатом повреждения парараковых тканей и ограничений, налагаемых внутренними характеристиками опухоли, такими как низкая чувствительность клучевой терапии.
Теоретически для скрининга CRISPR можно использовать любой тип клеток. Однако для поддержания достаточного представительства в мутировавших популяциях требуется большое количество исходных клеток. Типы клеток с низкой численностью не особенно подходят для полногеномного скрининга. Что касается выбора библиотеки, то большинство библиотек содержат 3-6 гРНК на ген-мишень, и поддержание распределения каждой гРНК в популяции имеет решающее значение. Потеря репрезентации из-за обогащения или истощения специфических гРНК может привести к неравномерному распределению результатов. Для решения этой проблемы предпочтительным выбором может быть выбор коммерчески доступных библиотек CRISPR, которые были протестированы на рынке. In vitro Скрининг CRISPR с использованием гомогенных линий раковых клеток может не полностью отражать генетическую и эпигенетическую гетерогенность опухолей in vivo . В то время как скрининг in vitro выявил ключевые гены, участвующие в репарации повреждений ДНК и радиационно-индуцированной аутокринной сигнализации, он не полностью воспроизвел микроокружение опухоли, включая индуцированную гипоксией радиорезистентность (через АФК, метаболическую адаптацию и аутофагию), иммуноопосредованные паракринные эффекты и ЭКМ-зависимую модуляцию цитокинов. Прежде чем использовать скрининг CRISPR для изучения генов, связанных с чувствительностью к излучению или резистентностью, эти факторы должны быть тщательно изучены. В свете текущей ситуации в области лечения необходимо срочно выявить и глубоко изучить факторы, связанные с радиорезистентностью и радиочувствительностью, для эффективного повышения эффективности лучевой терапии. Учитывая ключевое преимущество CRISPR-скрининга в изучении функций неизвестных генов, для эффективной идентификации радиочувствительных и радиорезистентных генов предоставляется систематически детализированная технология полногеномного скрининга CRISPR.
протокол
Реагенты и оборудование, использованные в данном исследовании, перечислены в Таблице материалов.
1. Выбор подходящей дозы облучения
- Подготовка и нанесение покрытий адгезивных клеток
- Отрегулируйте плотность клеток до 5 x 105 клеток на мл с помощью полной среды для культивирования клеток RPMI 1640, содержащей 10% FBS для прохождения и роста клеток. Распределите клетки по 3,5-сантиметровым культуральным чашкам для облучения в разных дозах. Добавьте по 2 мл (1 x 106 ячеек) в каждую чашку и инкубируйте в течение ночи при 37 °C с 5%CO2.
- Применение различных доз облучения
- Пронумеруйте посуду для культуры 3,5 см от 1 до 5. Используйте группу #1 в качестве контрольной группы, а остальные 4 группы обозначаются как группы лечения.
- Края 6-луночных планшетов герметизируют герметизирующей мембраной для групп обработки и вводят дозы облучения 2, 4, 6 и 8 Гр соответственно. После лечения верните посуду в инкубатор.
- Осаждение клеточных линий после облучения
- Подготовьте 6-луночные и 96-луночные планшеты. Вымойте клетки в чашках для культивирования диаметром 3,5 см один раз PBS, расщепите логарифмически растущие клетки с 0,25% трипсина и отрегулируйте плотность клеток до 1 x 105 клеток на мл с помощью полной среды RPMI 1640, содержащей 10% FBS.
- Засейте 10 μл/лунку (1000 клеток/100 μл) в 6-луночные планшеты с 3 повторениями на каждую дозу облучения. Добавьте по 2 мл готовой среды в каждую лунку и отсчитайте через 14 дней (когда в каждой группе клонов будет примерно 50 клеток).
- Засейте 30 μл/лунку (3000 клеток/100 μл) в 96-луночные планшеты с 5 повторениями для каждой дозы облучения. Добавьте 70 мкл готовой среды в каждую лунку и измерьте жизнеспособность клеток через 72 ч.
- Измерение жизнеспособности клеток
Примечание: Коэффициент клоногенного ингибирования = 1 - (число клонов в группе лечения/количество клонов в контрольной группе) x 100%. В анализе CCK-8 используется водорастворимая соль тетразолия (WST-8), которая может быть восстановлена клеточными дегидрогеназами для получения высокорастворимого в воде желтого формазана11. Количество вырабатываемого формазана прямо пропорционально количеству жизнеспособных клеток, а его оптическая плотность (измеренная на длине волны 450 нм) точно отражает клеточную метаболическую активность и пролиферативный статус11,13. Основываясь на этом хорошо зарекомендовавшем себя принципе, анализ CCK-8 получил широкое применение в различных областях, включая анализы пролиферации клеток и тесты на чувствительность к опухолевым препаратам. В этом протоколе анализ CCK-8 используется для систематической оценки жизнеспособности клеток при различных дозах облучения.- Смешайте реагент CCK8 со средой RPMI 1640 без FBS в соотношении 1:9. Выбросьте среду из 96-луночных планшетов и добавьте по 100 мкл среды, содержащей CCK8, в каждую лунку.
- Инкубируйте в темноте в течение 1 ч и измерьте значение OD на длине волны 450 нм с помощью считывателя микропланшетов.
ПРИМЕЧАНИЕ: Выживаемость клеток = [(Значение ОД группы лечения - Значение ОД пустой лунки) / (Значение ОД контрольной группы - Величина ОД пустой лунки)] x 100%
- Подбор дозы облучения
- Интегрируйте скорость клоногенного ингибирования с выживаемостью клеток, чтобы выбрать подходящую дозу облучения на основе исследовательских потребностей. Выберите дозу облучения со степенью ингибирования 50% для скрининга как радиорезистентных, так и радиочувствительных генов.
2. Выбор подходящей концентрации MOI и пуромицина
- Покрытие адгезивных клеток
- Отрегулируйте плотность клеток до 3 x 105 клеток на мл и инокулируйте 1 мл (3 x 105 клеток) на лунку в 12-луночный планшет. Инкубируйте тарелку в течение ночи при температуре 37 °C с 5% CO2.
- Лентивирусная инфекция
- С помощью пипетки отсасывайте среду из 12-луночного планшета и замените ее 1 мл полной среды RPMI 1640, содержащей 10% FBS. Подготовьте проверенную на качество полногеномную лентивирусную библиотеку CRISPR (рекомендуется использовать коммерчески доступные библиотеки) и установите логарифмический градиент концентрации (например, 0-10-50-100-200-400-800)4,5,6,7,8.
- Добавьте соответствующее количество лентивируса в 2 μл/чашку полибрена и уравновесьте при комнатной температуре в течение 5 минут. Медленно капните смесь лентивируса и полибрена в каждую лунку, хорошо перемешайте и инкубируйте в течение ночи при температуре 37 °C с 5%CO2.
- Определение минимальной концентрации пуромицина для уничтожения клеток
- Отрегулируйте плотность родительских клеток до 3 x 105 клеток/мл и инокулируйте 1 мл (3 x 105 клеток) на лунку в 12-луночный планшет. Инкубируйте тарелку в течение ночи при температуре 37 °C с 5% CO2.
- Добавьте пуромицин в каждую лунку 12-луночного планшета с градиентом концентрации 0-0,1-0,2-0,5-1-2-4-8 μМ. Подсчитайте клетки через 72 ч. Минимальная концентрация, которая убивает все клетки в лунке, является минимальной концентрацией пуромицина для уничтожения клеток, которая будет использоваться для последующего скрининга вирусно инфицированных клеток.
- Выбор пуромицина после инфицирования
- На второй день после заражения аспирируйте среду из каждой лунки и замените ее 1 мл полной среды RPMI 1640, содержащей 10% FBS. Продолжайте культивацию в течение 48 ч.
- Через 72 ч после заражения замените существующую среду на полноценную среду, содержащую минимальную концентрацию пуромицина для уничтожения клеток. Установите 2 лунки в 12-луночном планшете без лентивирусной инфекции в качестве отрицательного и положительного контроля. Обработайте отрицательный контроль пуромицином, а положительный контроль оставьте без лечения. Продолжайте культивацию в течение 72 ч.
- Через 72 ч после отбора пуромицина все родительские клетки в отрицательном контроле будут мертвы, в то время как родительские клетки в положительном контроле демонстрируют минимальную смертность. Рассчитайте MOI для каждой скважины.
ПРИМЕЧАНИЕ: MOI = (Количество клеток в инфицированной вирусом группе / Количество клеток в положительной контрольной группе) x 100%. Для последующего скрининга используйте концентрацию вируса с MOI ~0,3.
3. Полногеномная инфекция лентивирусной библиотеки CRISPR
- Посев адгезивных клеточных линий
- Отрегулируйте плотность клеток до 1 x 107 клеток на мл в полной среде RPMI 1640, содержащей 10% FBS для пассирования и роста клеток. Инокулируйте 1 мл клеток (1 x 107 клеток) в каждую 15-сантиметровую культуральную чашку при 37 °C с 5%CO2 в течение 8 часов. Как только клетки слипались и достигали слияния на 70-80%, они были готовы к вирусной инфекции (число копий составляло примерно 500).
- Лентивирусная инфекция
- Отсадите среду из 15-сантиметровых культуральных чаш и замените ее 15 мл полной среды RPMI 1640, содержащей 10% FBS. Подготовьте лентивирусную библиотеку CRISPR с проверкой качества.
- Добавьте соответствующее количество лентивируса с MOI = 0,3 до 30 мкл/чашку полибрена и уравновесьте при комнатной температуре в течение 5 минут, медленно капните смесь лентивируса и полибрена в 15-сантиметровые чашки для культивирования, хорошо перемешайте и инкубируйте при 37 °C с 5%CO2 в течение ночи. Одновременно приготовьте 15-сантиметровую культуральную чашку с родительской клеткой в качестве контраста.
- Выбор пуромицина после инфицирования
- На второй день после вирусной инфекции аспирируйте среду из 15-сантиметровых культуральных чашек и замените ее 15 мл полной среды RPMI 1640, содержащей 10% FBS. Продолжайте культивацию в течение 48 ч.
- Через 72 ч после инфицирования замените среду на полноценную среду, содержащую минимальную летальную концентрацию пуромицина. Таким же образом обрабатывайте неинфицированные родительские клетки в качестве отрицательного контроля и продолжайте культивирование в течение 72 часов. Через 72 ч после отбора пуромицина все родительские клетки в отрицательной контрольной группе будут убиты, а выжившие клетки лентивирусной инфекции будут считаться успешно инфицированными.
- Экстракция генома Дня 0
- Расщепляют клетки из одной 15-сантиметровой чашки для культивирования с использованием 0,25% трипсина, ресуспендируют в полной среде RPMI 1640, содержащей 10% FBS, и подсчитывают количество клеток. Обозначьте этот образец как День 0.
- Центрифугируйте при 300 х г в течение 5 минут (при комнатной температуре) и выбросьте надосадочную жидкость. Повторно суспендируйте в 1 мл PBS, центрифугируйте при 300 x g в течение 5 минут и выбросьте надосадочную жидкость. Извлеките геномную ДНК Дня 0, измерьте концентрацию и чистоту ДНК с помощью нанокапельного ультрафиолетового спектрофотометра.
- Для определения целостности sgРНК используют электрофорез в агарозном геле для анализа амплифицированной библиотеки sgРНК, гарантируя, что полосы были прозрачными и не имели существенной деградации, тем самым сохраняя целостность библиотеки23. Для оценки охвата лентивирусной библиотеки используйте технологию глубокого секвенирования для проведения анализа секвенирования sgРНК в библиотеке24,25.
- В скрининге CRISPR амплификация ПЦР служит предварительной проверкой целостности фрагмента sgRNA и качества библиотеки6. Чтобы более полно проанализировать охват библиотекой и закономерности распределения sgRNA, а также обеспечить адекватное представительство sgRNA для каждого целевого гена во время скрининга, выполните NGS для более глубокого понимания надежности скрининга9.
ПРИМЕЧАНИЕ: Обнаружение изменений в распространенности sgРНК до и после скрининга, а также выявление потенциальной интеграции sgRNA вне мишени или загрязнения библиотеки, может быть дополнительно улучшена точность и надежность скрининга CRISPR. Эти образцы Дня 0 служат важнейшим негативным контролем и проходят всестороннюю оценку качества с помощью ПЦР-амплификации и NGS для достижения 2 ключевых целей: (1) подтверждение существенного истощения генов (что указывает на адекватное представление библиотеки) и (2) демонстрация стабильной экспрессии в несущественных генах (установление экспериментальных исходных условий)9. - Обеспечение того, чтобы охват достиг ожидаемого уровня, чтобы обеспечить разнообразие и репрезентативность библиотеки и избежать предвзятости в процессе отбора. Для измерения эффективности инфекции используйте флуоресцентную визуализацию для оценки эффективности инфекции, гарантируя, что она соответствует требованиям эксперимента.
4. Применение радиации в качестве условия скрининга
- Группировка
- Отрегулируйте плотность инфицированных CRISPR клеток до 1 x 10-7 клеток на мл и введите 1 мл в каждую чашку Петри диаметром 15 см. Инкубировать в течение ночи при 37 °C с 5%CO2.
- Лучевая терапия
- Случайным образом разделите клетки на 2 группы: группу лечения и контрольную группу, по 6 блюд в группе. Ввести соответствующую дозу облучения в клетки в группе лечения, в то время как клетки контрольной группы оставить необработанными для нормального размножения. После облучения продолжайте инкубацию при 37 °C с 5%CO2 в течение 7 дней.
- На второй неделе повторите введение соответствующей дозы облучения клеткам группы лечения, оставляя клетки контрольной группы необработанными и позволяя им нормально размножаться. После лучевой терапии продолжайте инкубацию при 37 °C с 5%CO2 в течение еще 7 дней.
5. Экстракция и секвенирование генома
- Извлечение из 14-го дня
- После 14 дней лечения расщепите клетки в группе лечения и контрольной группе с 0,25% трипсина, повторно суспендируйте их с помощью полной среды RPMI 1640, содержащей 10% FBS, и пометьте их как День 14-RT или День 14-NC.
- Центрифугируйте клетки при 300 x g в течение 5 минут и выбросьте надосадочную жидкость. Повторно суспендируйте в 1 мл PBS, центрифугируйте при 300 x g в течение 5 минут и снова выбросьте надосадочную жидкость. Извлеките геномную ДНК на 14-й день и определите концентрацию и чистоту ДНК с помощью нанокапельной УФ-спектроскопии.
- ПЦР-амплификация
- Приготовьте соответствующие праймеры последовательности (см. Таблицу материалов) и разбавьте их до 10 мкМ. Настройте реакционную систему объемом 20 мкл, добавив реагенты в стерильную микроцентрифужную пробирку, центрифугируйте в течение 5 с при 300 x g и хорошо перемешайте. Установите параметры прибора для ПЦР в соответствии с условием амплификации для получения продуктов ПЦР.
- Обнаружение электрофореза в агарозном геле
- Подготовьте пластину для литья гелем, запечатайте края формы агарозой, вставьте гребень и приготовьте агарозный гель соответствующей концентрации в соответствии с длиной ДНК образца.
- Точно взвесьте определенное количество порошка агарозы, добавьте соответствующее количество буфера для электрофореза, хорошо перемешайте и нагрейте в микроволновой печи, чтобы он растаял. После небольшого охлаждения добавьте соответствующее количество красителя для нуклеиновых кислот, аккуратно перемешайте и медленно вылейте в форму для литья в гель. Дайте гелю застыть в течение 30 минут.
- Снимите расческу и добавьте соответствующее количество буфера для электрофореза в резервуар для электрофореза, пока он не покроет гель. Добавьте соответствующее количество загрузочного буфера в образец ДНК, хорошо перемешайте и с помощью пипетки медленно добавьте смесь в лунку для образца.
- Установите соответствующее напряжение исходя из размера фрагмента ДНК и концентрации агарозного геля. После электрофореза осторожно извлеките гель и поместите его в формирователь изображения геля, чтобы наблюдать за результатами и проверить, была ли успешно проведена ПЦР-амплификация.
- Секвенирование Illumina
- Соберите геномную ДНК из трех групп (группа дня 0, контрольная группа и группа лечения) и отправьте ее в компанию для создания библиотеки и секвенирования Illumina. Выполнение биоинформатического анализа и визуализации результатов секвенирования с целью получения генов, чувствительных к лучевой терапии и устойчивых к лучевой терапии26.
- Постановка нескольких повторных экспериментов для анализа данных, а также проведение статистического анализа по результатам экспериментов для обеспечения согласованности и воспроизводимости полученных данных27,28.
- Оценка качества данных секвенирования
- Подвергайте необработанные данные строгим механизмам фильтрации и контроля качества, чтобы гарантировать точность информации о секвенировании5. Вычислите показатель Phred (Qphred) для каждого нуклеотида на основе частоты ошибок секвенирования, используя указанный алгоритм преобразования и модель, которая оценивает вероятность возникновения ошибки во время вызова базы28.
- Поддерживайте частоту ошибок секвенирования для каждой базовой позиции на уровне ниже 1% (что эквивалентно пороговому значению Q30) и убедитесь, что не менее 80% данных секвенирования соответствуют стандарту Q30 для поддержки последующих аналитических процедур.
- Обработка данных и контроль качества на уровне выборки
ПРИМЕЧАНИЕ: Для получения подробной информации, пожалуйста, обратитесь к ранее опубликованным отчетам29,30.- Соровняйте отфильтрованные чтения данных секвенирования с последовательностями библиотеки sgRNA. Отчетная статистика, включая количество sgРНК в библиотеке с идеальным выравниванием, среднее количество sgРНК, количество необнаруженных sgRNA и долю прочтений из отдельных образцов, успешно картированных в библиотеку sgRNA. Используйте более высокий коэффициент сопоставления, чтобы указать на большее покрытие.
- Подсчитайте обогащение прочтений для каждого гена (на который нацелены разные sgRNA) в каждом образце. Нормализуйте поддерживающие чтения для каждой гРНК в образцах с помощью метода «медианной» нормализации MAGeCK. Выполняйте контроль качества на уровне образцов путем оценки распределения количества прочтений sgРНК, построения ящичковых диаграмм, анализа главных компонент (PCA) и построения корреляционных тепловых карт.
- Предположим, что количество прочтений sgRNA соответствует распределению Пуассона. Изобразите нормализованное количество sgРНК из разных групп на ящичковых диаграммах, чтобы визуализировать общее распределение данных в выборках и сравнить распределения между группами.
- Используйте PCA для упрощения анализа данных за счет отражения различий между основными компонентами и скорости вариаций внутри каждого компонента, облегчая наблюдение за внутригрупповыми и межгрупповыми вариациями. Используйте тепловую карту корреляции, чтобы проиллюстрировать взаимосвязи между образцами.
- Базовый анализ
- Выполнение дифференциального анализа sgРНК между группами и выявление основных генов после контроля качества и предварительной обработки данных. Проведение функционального анализа обогащения соответствующих генов30. Ранжируйте основные гены на основе надежной оценки ранговой агрегации (RRA), рассчитанной с помощью MAGeCK или других статистических методов, таких как MLE, при этом более низкая оценка RRA указывает на более высокую важность.
- Используйте язык R или другие языки программирования для выполнения биоинформатического анализа ранжированных результатов и визуализируйте результаты с помощью диаграмм анализа GO и KEGG30. Связывайте гены или белки с соответствующими терминами GO (молекулярной функцией, биологическим процессом и клеточным компонентом) с помощью ID-картирования или аннотации последовательностей.
- Используйте KEGG в качестве базы данных для понимания высокоуровневых функций и биологических систем, связывая идентифицированный каталог генов с системными функциями на клеточном, видовом и экосистемном уровнях. Выберите 10 или 20 основных путей из анализов GO и KEGG для интуитивно понятного отображения направлений путей.
Результаты
Рак легких, с ведущим уровнем смертности, представляет собой очень агрессивное и распространенное заболевание. На примере клеточной линии рака легкого A549 для проведения полногеномного скрининга CRISPR с облучением в качестве условия скрининга, схематический рабочий процесс показан на рисунке 1. Во-первых, изучите чувствительность клеток A549 к различным дозам радиации посредством формирования клонов и экспериментов CCK8 (рис. 2). В клоногенном анализе количество колоний составило 140 ± 5,35 при 2 Гр против 303 ± 31,63 при 0 Гр, в то время как в анализе CCK-8 значение оптической плотности (OD) составило 0,65 ± 0,05 при 2 Гр против 1,35 ± 0,08 при 0 Гр. Чтобы исследовать гены-кандидаты на радиочувствительность и радиорезистентность, выберите 2 Гр (IC50) в качестве последующей дозы облучения. Клетки А549 обрабатывали различными дозами пуромицина и подсчитывали через 72 ч. Минимальная убивающая концентрация пуромицина для клеток А549 была обнаружена как 1 мкМ (рис. 3). Клетки А549 были инфицированы лентивирусами при различных градиентных MOI. Через 72 ч после инфицирования эффективность инфекции наблюдали с помощью флуоресцентного микроскопа и подсчитывали до и после 72 ч лечения пуромицином. Получен MOI с эффективностью заражения ~0,3 (табл. 1). Геном человека содержит 19 050 генов, из которых 6 sgРНК соответствуют каждому гену и имеют число копий 500 (19 050 x 6 x 500 = 5,7 x 107 клеток). С помощью лентивирусов с MOI = 0,3 и, соответственно, заражают 5,7 х 107 клеток А549. Через 72 ч клетки обрабатывали 1 мкМ пуромицином в течение еще 72 ч. Был собран геном Day 0 и проведена ПЦР-амплификация. Последовательности sgРНК в полногеномной библиотеке CRISPR имеют длину 231 пару оснований, что подтверждается результатами электрофореза в агарозном геле (рис. 4).
Впоследствии ПЦР-продукты были отправлены в Novogene для контроля качества данных и контроля качества на уровне образцов. Коэффициент картирования дал всеобъемлющий коэффициент охвата около 60%, что считается достаточным для процедур скрининга всего генома, учитывая огромную сложность и неизбежный отток во время обработки всего генома (рис. 5). Подсчет прочтений sgRNA соответствовал распределению Пуассона, что соответствовало теоретическим ожиданиям. Последующий анализ с помощью построения графиков PCA и корреляционного теплового картографирования ярко показал заметное несоответствие между отдельными группами, при этом межгрупповые различия заметно перевешивали внутригрупповые различия. Более того, коэффициент вариации в сгруппированных выборках находился в допустимых границах, что свидетельствует об успешности мер контроля качества на уровне выборки. Впоследствии рейтинги RRA, разработанные MAGeCK, будут использованы для проведения фундаментальной биоинформатической оценки результатов рейтинга с использованием языка R. Примечательно, что в топ-15 путей с точки зрения GO заметно фигурируют механизмы, связанные с повреждением ДНК, что органично согласуется с основными критериями скрининга эксперимента.
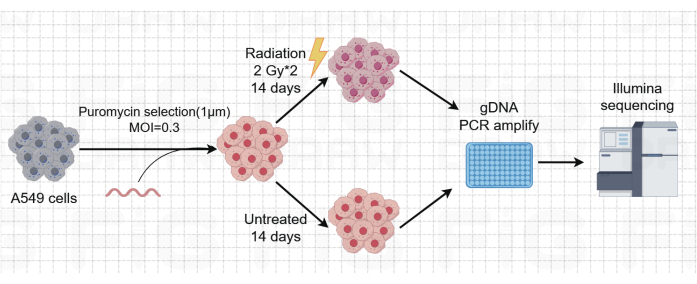
Рисунок 1: Схема полногеномного скрининга лучевой терапии CRISPR/Cas9 в клетках рака легких A549. Схематическое изображение процесса скрининга в клетках A549 с использованием полногеномного CRISPR/Cas9 и лучевой терапии. Пожалуйста, нажмите здесь, чтобы просмотреть увеличенную версию этой цифры.
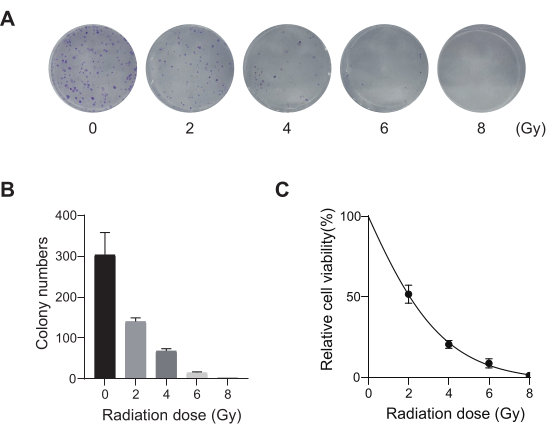
Рисунок 2: Оптимизация дозы облучения для клеток A549. (A) Клетки A549 подвергали воздействию возрастающих доз облучения (0 Гр, 2 Гр, 4 Гр, 6 Гр и 8 Гр) и давали им расти в течение 14 дней. Показаны изображения анализа образования колоний. Результаты являются репрезентативными для двух независимых биологических экспериментов. (B) Результаты количественной оценки клонированных изображений для анализа пласта. Полосы погрешностей представляют собой стандартное отклонение (n = 3). Односторонняя ANOVA дала P < 0,01. Результаты являются репрезентативными для двух независимых биологических экспериментов. (C) Кривые выживаемости «доза-реакция» клеток A549 после облучения различными дозами облучения. Полосы погрешностей представляют собой стандартное отклонение (n = 3). Односторонний ANOVA дал P < 0,01. Результаты являются репрезентативными для двух независимых биологических экспериментов. Пожалуйста, нажмите здесь, чтобы просмотреть увеличенную версию этой цифры.
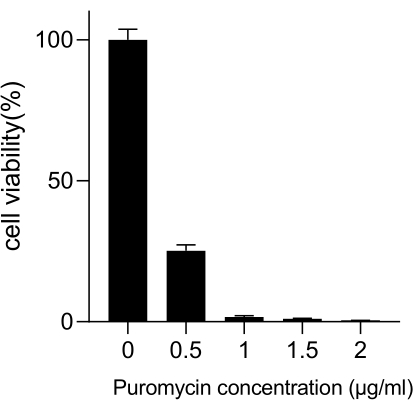
Рисунок 3: Определение минимальной концентрации пуромицина, необходимой для отбора клеток A549. Клетки А549 обрабатывали возрастающими концентрациями пуромицина и инкубировали в течение 72 ч. Полосы погрешностей представляют собой стандартное отклонение (n = 3). Односторонняя ANOVA дала P < 0,01. Результаты являются репрезентативными для двух независимых биологических экспериментов. Пожалуйста, нажмите здесь, чтобы просмотреть увеличенную версию этой цифры.
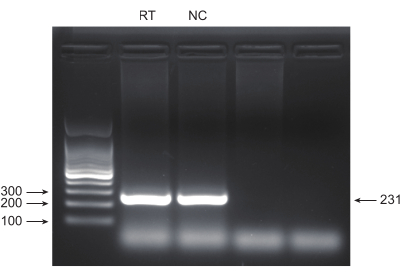
Рисунок 4: Валидация продукта ПЦР методом электрофореза в агарозном геле. Электрофорез в агарозном геле был использован для исследования наличия и качества полос продукта ПЦР. Пожалуйста, нажмите здесь, чтобы просмотреть увеличенную версию этой цифры.
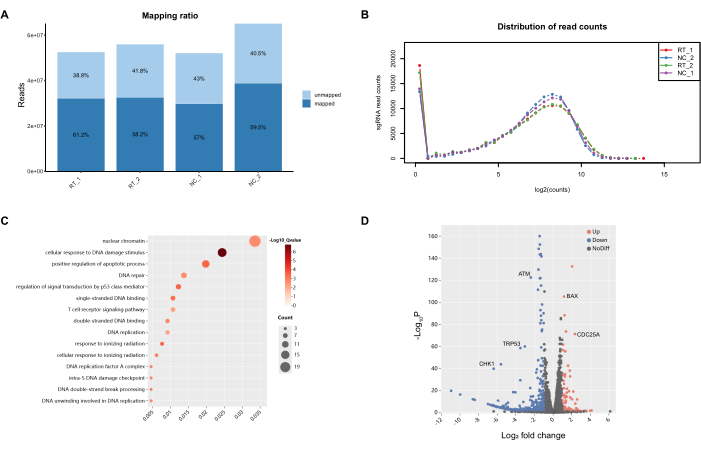
Рисунок 5: Контроль качества и базовый анализ продуктов, амплифицированных методом ПЦР. (A) Коэффициент картирования показал примерно 60% охват референсного генома. (B) Распределение сгРНК по количеству прочтений следовало распределению Пуассона. (C) Большинство из 10 основных терминов онтологии генов (GO) были связаны с реакцией на повреждение ДНК. (D) Вулканический график, показывающий паттерны обогащения генов, где отрицательно обогащенные гены выделены красным цветом, положительно обогащенные гены - синим, а незначимые гены - серым. Пожалуйста, нажмите здесь, чтобы просмотреть увеличенную версию этой цифры.
| Вирусная частица Номер (VP) | Количество кол-во (перед лечением пуромицином) | Количество кол-во (после лечения пуромицином) | Инфекция эффективность |
| 1.23 × 104 | 1.23 ×10 5 | 1.04 × 104 | 0.084552846 |
| 3.72 × 104 | 1.24 × 106 | 3.61 × 104 | 0.291129032 |
| 6.15 × 104 | 1.23 × 107 | 5.4 × 104 | 0.43902439 |
Таблица 1: Количество клеток A549 до и после отбора пуромицина под различными вирусными частицами. Подсчет клеток A549 регистрировался до и после лечения пуромицином под действием различных вирусных частиц.
Обсуждение
Являясь передовой технологией редактирования генов, скрининг CRISPR вызвал глубокие изменения в области научных исследований5. Возникшая на основе системы CRISPR-Cas9, эта технология стала важным инструментом для изучения функций генов благодаря своей высокой эффективности иточности9. Инженерный принцип CRISPR/Cas9 включает в себя разработку и введение специфических sgРНК с примерно 20 нуклеотидами, чтобы направлять нуклеазу Cas9 на точное определение местоположения и разрезание целевых последовательностей ДНК, что позволяет редактировать гены, такие как нокаут, активация или репрессия. Этот принцип лежит в основе разработки высокопроизводительного и настраиваемого экрана CRISPR. Создавая библиотеки, содержащие огромное количество sgRNA, исследователи могут одновременно редактировать и проверять несколько генов, быстро выявляя те, которые связаны с конкретными фенотипами или заболеваниями. Кроме того, скрининг CRISPR позволяет экспериментировать с различными типами клеток, тканями и даже целыми организмами, обеспечивая более полное понимание функции генов и механизмов их действия.
С ускорением развития технологий экран CRISPR показал огромный потенциал для применения в различных областях. Например, скрининг CRISPR может помочь ученым в обнаружении новых мишеней для лекарств, тем самым ускоряя процесс поиска и разработки лекарств. В исследованиях болезней эта технология выявляет механизмы патогенеза заболеваний, предлагая свежие идеи и подходы к диагностике и лечению. Скрининг CRISPR принципиально отличается от традиционных методов скрининга (таких как РНК-интерференция и низкомолекулярные ингибиторы) в нескольких ключевых аспектах: механически CRISPR обеспечивает постоянные генетические модификации за счет редактирования генов, в то время как РНК-интерференция лишь временно подавляет экспрессию генов, а низкомолекулярные ингибиторы обратимо блокируют функцию белка28,29. С точки зрения специфичности, CRISPR демонстрирует превосходную точность за счет точного нацеливания на ДНК, в то время как РНК-интерференция склонна к нецелевым эффектам 3,5. Что касается продолжительности эффекта, CRISPR создает постоянные генетические изменения по сравнению с преходящими вмешательствами традиционных методов. Кроме того, CRISPR обеспечивает возможности скрининга на уровне генома и может одновременно исследовать несколько генов/путей, сохраняя при этом высокую эффективность для различных типов клеток9. Традиционные подходы, как правило, ограничены анализом конкретных путей и часто демонстрируют сниженную эффективность в определенных клеточныхлиниях. Эти отличительные характеристики делают скрининг CRISPR особенно ценным для исследований функциональной геномики и поиска мишеней, особенно в исследованиях, требующих долгосрочного наблюдения или точной генетической регуляции, где он демонстрирует уникальные преимущества по сравнению с традиционными методологиями скрининга.
Однако специфичность этого процесса не является абсолютной, и белок Cas9 может ошибочно распознавать и расщеплять участки ДНК, которые похожи, но не полностью идентичны предполагаемой последовательности-мишени5. Это явление обычно называют нецелевыми эффектами. Это непреднамеренное расщепление ДНК может привести к геномной нестабильности и непредсказуемым генетическим последствиям, особенно при применении в терапии человека. Технология CRISPR также испытывает трудности при решении сложных задач редактирования генома. Столкнувшись с требованиями к редактированию, которые охватывают множество генов, кластеров генов или сложных генетических условий, система CRISPR может испытывать трудности в точном прогнозировании и управлении результатамипроцесса редактирования.
Основное техническое препятствие при проведении полногеномного скрининга с использованием технологии CRISPR связано с огромной сложностью генома, который охватывает десятки тысяч генов, что значительно увеличивает рискпобочных эффектов. Чтобы обеспечить целостность и надежность последующих результатов секвенирования, крайне важно, чтобы каждая клетка была трансфицирована одной sgRNA23. Это обуславливает необходимость тщательного контроля MOI со стороны экспериментатора, гарантируя, что он остается ниже 0,5, при одновременном поддержании адекватного порогового значения числа копий, которое должно составлять не менее 500 или больше24. Учитывая тот факт, что каждый из десятков тысяч генов во всем геноме требует использования 4-6 sgRNAs, весь процесс скрининга требует большого количестваклеток25. Длительная продолжительность эксперимента, значительное количество задействованных клеток и повышенный риск требуют от экспериментатора разработки стратегий для смягченияэтих опасений. Потенциальные гены должны быть дополнительно проверены с помощью РНК-секвенирования, а также анализов на усиление и потерю функции. В последующих экспериментах, после получения сверхэкспрессии и нокаутных клеток идентифицированных генов с помощью редактирования CRISPR, была проведена полногеномная геномика и транскриптомика для доступа к потенциальным внецелевым изменениям, особенно в локусах с частичной комплементарностью sgРНК, которые могут подвергаться неспецифическому расщеплению Cas9. Инструменты биоинформатического прогнозирования, такие как COSMID, могут помочь выявить участки с высоким риском отклонения от цели. Спасательные анализы также могут быть использованы для обеспечения специфичности вовлечения идентифицированных генов.
В последующем анализе критерии дифференциально экспрессируемых sgРНК основывались на оценке устойчивой ранговой агрегации (RRA), рассчитанной с помощью MAGeCK, которая включала статистическую значимость, изменение кратности и согласованность репликации31,32. Для набора данных каждый ген ранжируется в соответствии с комплексными критериями, сочетающими уровни экспрессии генов и статистическую значимость. RRA использует коэффициент ранговой корреляции Спирмена для ранга для оценки ситуации всеобъемлющего ранга каждого гена во всех наборах данных и интегрирует результаты сортировки нескольких наборов данных экспрессии генов. Ген с относительно высоким рангом в большинстве наборов данных может рассматриваться как подлинный дифференциально экспрессируемый ген33. p-значение RRA менее 0,05 считалось статистически значимым. Изменение в раз, по крайней мере, в 1,5 раза или на 33%, рассматривалось как дифференциальное выражение. Кроме того, согласованность 3 независимых реплик должна была рассматриваться как статистически различное выражение. После ранжирования RRA по основным генам более низкий показатель RRA означает более высокую важность.
Являясь мощным инструментом для редактирования генома, технология CRISPR-Cas9 демонстрирует беспрецедентный потенциал и точность в исследовании области радиорезистентности26,27. Однако его применение все еще имеет ограничения. Помимо риска побочных эффектов, тонкости, связанные с радиочувствительностью, вносят сложностив процесс скрининга. Эта чувствительность не просто регулируется одним геном, а скорее возникает как следствие сложного взаимодействия между несколькими генами и их регуляторнымисетями. Таким образом, несмотря на успешную идентификацию специфических генов, относящихся к радиочувствительности, они могут представлять лишь часть более широкой картины, что создает значительные трудности для всестороннего выяснения основных механизмов 34,35,36. Вот решения распространенных проблем в скрининге CRISPR, таких как низкая эффективность инфекции, деградация sgРНК и сбой секвенирования: поскольку для скрининга CRISPR требуется значение MOI <0,5, корректировка состояния клеток имеет решающее значение для обеспечения того, чтобы клетки находились в логарифмической фазе роста или оценки эффективности инфекции с помощью флуоресцентных анализов; потенциальные причины деградации sgRNA включают загрязнение нуклеазами или дефекты дизайна векторов, поэтому следует избегать повторных циклов замораживания-оттаивания библиотек sgRNA (хранить аликвоты при -80 °C), а для проверки целостности библиотеки sgRNA следует использовать секвенирование NGS; Неудачное секвенирование может быть вызвано недостаточной глубиной секвенирования или проблемами с дизайном праймера sgРНК, для повышения точности идентификации последовательности sgRNA рекомендуется секвенирование с парным концом, с рекомендуемой глубиной секвенирования >500× охватом 37,38,39,40,41,42.
Лучевая терапия остается краеугольным камнем в лечении рака, но при этом демонстрирует замечательную вариабельность терапевтической эффективности при различных типах опухолей и даже у пациентов с одним и тем же раком9. Эта межиндивидуальная гетерогенность часто обуславливает необходимость комбинированной терапии для достижения оптимальной эрадикации опухоли11,12. В клинической практике интеграция лучевой терапии с иммунотерапией, в частности с ингибиторами контрольных точек иммунного ответа и конъюгатами антитело-лекарство, стала заметной терапевтическойпарадигмой19. Этот протокол представляет собой скрининг CRISPR in vitro с использованием относительно однородной линии раковых клеток, которая не может полностью отразить генетическую и эпигенетическую гетерогенность опухолей in vivo. После определения подходящей дозы облучения для скрининга полученные результаты позволяют выявить потенциальные радиочувствительные или радиорезистентные гены, которые необходимо дополнительно подтвердить как в клеточных анализах in vitro, так и на животных моделях in vivo. Кроме того, скрининг in vivo CRISPR в сочетании с транскриптомикой одиночных клеток может быть полезен для расшифровки сложности гетерогенности опухоли. Скрининг CRISPR in vitro идентифицировал важные гены в восстановлении повреждений ДНК в клетках, а также в аутокринных механизмах в ответ на облучение, но действительно не полностью реплицировал гетерогенные ТМЭ. Например, гипоксия увеличивает выработку АФК, запуская петлю обратной связи, которая стимулирует метаболическую адаптацию, выработку антиоксидантов и активацию аутофагии, в конечном итоге способствуя радиорезистентности. Иммунные клетки могут активироваться в паракринном механизме после облучения, индуцируя радиочувствительность14. ВКМ служит резервуаром для факторов роста, а цитокины оказывают как положительное, так и отрицательное влияние на радиочувствительность21. Скрининг CRISPR in vivo должен быть выполнен таким образом, чтобы отразить гетерогенность TME. Кроме того, скрининг CRISPR in vitro смог выявить только важные гены в восстановлении повреждений ДНК в клетках, а также в аутокринных механизмах в ответ на излучение, но не полностью отразил влияние in vivo на радиочувствительность, включая сосудистую сеть опухоли и системные эффекты20,21. Скрининг CRISPR in vivo должен быть выполнен для выявления потенциальных факторов, влияющих на радиочувствительность in vivo.
Здесь разработан комплексный протокол, направленный на использование полногеномного скрининга CRISPR для точного выявления генов, которые участвуют либо в чувствительности, либо в резистентности к лучевой терапии. Раскрывая новые пути, эта методология стремится проложить путь к достижениям в исследованиях лучевой терапии опухолей. Этот протокол также может служить справочным материалом для изучения механизмов клинических ограничений в химиотерапии и прогрессирования альтернативных терапевтических агентов. Подводя итог, можно сказать, что технология CRISPR воплощает в себе значительный потенциал и ценность, но присущие ей ограничения подчеркивают важность трезвого признания и разумного подхода. По мере того, как будущие исследования и приложения углубляются, крайне важно неустанно стремиться к изучению и оптимизации технологии CRISPR, чтобы преодолеть ее ограничения и в полной мере использовать ее потенциал.
Раскрытие информации
Никакой.
Благодарности
Это исследование было поддержано Региональным проектом научно-технических инноваций провинции Хубэй (2024EIA001) и Проектом поддержки строительства платформы инноваций в области медицинской науки и технологий больницы Чжуннань Уханьского университета (PTXM2025001). Рисунок 1 был создан с помощью Figdraw.
Материалы
| Name | Company | Catalog Number | Comments |
| 150 mm cell and tissue culture dish | Wuxi NEST Biotechnology Co., Ltd. | 715011 | Cell culture |
| 35 mm cell and tissue culture dish | Wuxi NEST Biotechnology Co., Ltd. | 706011 | Cell culture |
| A549 cell line | ATCC | - | - |
| Cell counting kit-8 | Shanghai Beyotime Biotech Inc | C0041 | For cell viability assay |
| CO2-independent medium | PHCbi | MCO-50AIC | Cell culture |
| Countess 3 FL automated cell counter | Themo Scientific | AMQAF2001 | For cell counting |
| EDTA | Gibco | 25200056 | Cell culture |
| Fetal bovine serum | Gibco | 10099141 | Cell culture |
| Fluorescence microscopy | LEICA | ebq 100-04 | For fluorescence microscope |
| Genome-wide CRISPR lentiviral library | Shanghai OBiO Technology Co., Ltd. | H5070 | For lentiviral infection |
| MiniAmp plus PCR | Themo Scientific | C37835 | For PCR amplification |
| NEST cell culture plates, 12-well | Wuxi NEST Biotechnology Co., Ltd. | 712002 | Cell culture |
| NEST cell culture plates, 6-well | Wuxi NEST Biotechnology Co., Ltd. | 703002 | Cell culture |
| NEST cell culture plates, 96-well | Wuxi NEST Biotechnology Co., Ltd. | 701001 | Cell culture |
| PBS | Gibco | C10010500BT | Cell culture |
| PCR forward primer (AATGGACTATCATATGCTTACCGTAACTTGAAAGTATTTCG) | Beijing AuGCT Biotech Co., Ltd. | - | For PCR amplification |
| PCR reverse primer (GATGTGCGCTCTGCCCACTGACGGGCA) | Beijing AuGCT Biotech Co., Ltd. | - | For PCR amplification |
| Polybrene | Shanghai Beyotime Biotech Inc | C0351 | For lentiviral infection |
| Puromycin | MCE | HY-K1057 | For seletion post lentiviral infection |
| RPMI 1640 cell culture medium | Gibco | 23400-021 | Cell culture |
| TIANGENamp genomic DNA kit | Beijing TIANGEN Biotech Co.,Ltd. | DP304 | For genomic DNA extraction |
| X-ray powder diffractometer | PerkinElmer | For radiotherapy |
Ссылки
- Barrangou, R., Marraffini, L. A. CRISPR-Cas systems: Prokaryotes upgrade to adaptive immunity. Mol Cell. 54 (2), 234-244 (2014).
- Joung, J., et al. Genome-scale CRISPR-Cas9 knockout and transcriptional activation screening. Nat Protoc. 12 (4), 828-863 (2017).
- Sanjana, N. E., Shalem, O., Zhang, F. Improved vectors and genome-wide libraries for CRISPR screening. Nat Methods. 11 (8), 783-784 (2014).
- Arroyo, J. D., et al. A Genome-wide CRISPR death screen identifies genes essential for oxidative phosphorylation. Cell Metab. 24 (6), 875-885 (2016).
- Sidik, S. M., et al. A genome-wide CRISPR screen in toxoplasma identifies essential apicomplexan genes. Cell. 166 (6), 1423-1435 (2016).
- Jing, Y., et al. Genome-wide CRISPR activation screening in senescent cells reveals SOX5 as a driver and therapeutic target of rejuvenation. Cell Stem Cell. 30 (11), 1452-1471 (2023).
- Shifrut, E., et al. Genome-wide CRISPR screens in primary human t cells reveal key regulators of immune function. Cell. 175 (7), 1958-1971 (2018).
- Shalem, O., et al. Genome-scale CRISPR-Cas9 knockout screening in human cells. Science. 343 (6166), 84-87 (2014).
- Katti, A., Diaz, B. J., Caragine, C. M., Sanjana, N. E., Dow, L. E. CRISPR in cancer biology and therapy. Nat Rev Cancer. 22 (5), 259-279 (2022).
- Sasmal, R., et al. Synthetic host-guest assembly in cells and tissues: Fast, stable, and selective bioorthogonal imaging via molecular recognition. Anal Chem. 90 (19), 11305-11314 (2018).
- Roos, W. P., Thomas, A. D., Kaina, B. DNA damage and the balance between survival and death in cancer biology. Nat Rev Cancer. 16 (1), 20-33 (2016).
- Huang, R., Zhou, P. K. DNA damage repair: Historical perspectives, mechanistic pathways and clinical translation for targeted cancer therapy. Signal Transduct Target Ther. 6 (1), 254(2021).
- O'Connor, M. J. Targeting the DNA damage response in cancer. Mol Cell. 60 (4), 547-560 (2015).
- Lord, C. J., Ashworth, A. The DNA damage response and cancer therapy. Nature. 481 (7381), 287-294 (2012).
- Carusillo, A., Mussolino, C. DNA Damage: From threat to treatment. Cells. 9 (7), 1665(2020).
- Li, X., Heyer, W. D. Homologous recombination in DNA repair and DNA damage tolerance. Cell Res. 18 (1), 99-113 (2008).
- Chang, H., Pannunzio, N. R., Adachi, N., Lieber, M. R. Non-homologous DNA end joining and alternative pathways to double-strand break repair. Nat Rev Mol Cell Biol. 18 (8), 495-506 (2017).
- Andratschke, N., et al. European society for radiotherapy and oncology and European organization for research and treatment of cancer consensus on re-irradiation: Definition, reporting, and clinical decision making. Lancet Oncol. 23 (10), e469-e478 (2022).
- Olivares-Urbano, M. A., Grinan-Lison, C., Marchal, J. A., Nunez, M. I. CSC radioresistance: A therapeutic challenge to improve radiotherapy effectiveness in cancer. Cells. 9 (7), 1651(2020).
- Schaue, D., McBride, W. H. Opportunities and challenges of radiotherapy for treating cancer. Nat Rev Clin Oncol. 12 (9), 527-540 (2015).
- Allen, C., Her, S., Jaffray, D. A. Radiotherapy for cancer: Present and future. Adv Drug Deliv Rev. 109, 1-2 (2017).
- Xue, C., Greene, E. C. DNA repair pathway choices in CRISPR-Cas9-Mediated genome editing. Trends Genet. 37 (7), 639-656 (2021).
- Tzelepis, K., et al. A CRISPR dropout screen identifies genetic vulnerabilities and therapeutic targets in acute myeloid leukemia. Cell Rep. 17 (4), 1193-1205 (2016).
- Rottner, A. K., et al. A genome-wide CRISPR screen identifies CALCOCO2 as a regulator of beta cell function influencing type 2 diabetes risk. Nat Genet. 55 (1), 54-65 (2023).
- Chen, M., et al. CRISPR-Cas9 for cancer therapy: Opportunities and challenges. Cancer Lett. 447, 48-55 (2019).
- Gupta, D., et al. CRISPR-Cas9 system: A new-fangled dawn in gene editing. Life Sci. 232, 116636(2019).
- Li, B., et al. Genome-wide CRISPR screen identifies host dependency factors for influenza A virus infection. Nat Commun. 11 (1), 164(2020).
- Hung, K. L., et al. Targeted profiling of human extrachromosomal DNA by CRISPR-CATCH. Nat Genet. 54 (11), 1746-1754 (2022).
- Bhattacharya, D., Van Meir, E. G. A simple genotyping method to detect small CRISPR-Cas9 induced indels by agarose gel electrophoresis. Sci Rep. 9 (1), 4437(2019).
- Razavi, Z., Soltani, M., Souri, M., Van Wijnen, A. J. CRISPR innovations in tissue engineering and gene editing. Life Sci. 358, 123120(2024).
- Wang, W., et al. A genome-wide CRISPR-based screen identifies KAT7 as a driver of cellular senescence. Sci Transl Med. 13 (575), eabd2655(2021).
- Xu, X., et al. Engineered miniature CRISPR-Cas system for mammalian genome regulation and editing. Mol Cell. 81 (20), 4333-4345 (2021).
- Huang, M., et al. FACS-based genome-wide CRISPR screens define key regulators of DNA damage signaling pathways. Mol Cell. 83 (15), 2810-2828 (2023).
- Feng, J., et al. Genome-wide CRISPR screen identifies synthetic lethality between DOCK1 inhibition and metformin in liver cancer. Protein Cell. 13 (11), 825-841 (2022).
- Saurat, N., et al. Genome-wide CRISPR screen identifies neddylation as a regulator of neuronal aging and AD neurodegeneration. Cell Stem Cell. 31 (8), 1162-1174 (2024).
- Du, Y., Liu, Y., Hu, J., Peng, X., Liu, Z. CRISPR/Cas9 systems: Delivery technologies and biomedical applications. Asian J Pharm Sci. 18 (6), 100854(2023).
- Shi, J., et al. A genome-wide CRISPR screen identifies WDFY3 as a regulator of macrophage efferocytosis. Nat Commun. 13 (1), 7929(2022).
- Liu, N., Olson, E. N. CRISPR modeling and correction of cardiovascular disease. Circ Res. 130, (2022).
- Feng, X., et al. Genome-wide CRISPR screens using isogenic cells reveal vulnerabilities conferred by loss of tumor suppressors. Sci Adv. 8 (19), 6638(2022).
- Ghaemi, A., et al. CRISPR-Cas9 genome editing delivery systems for targeted cancer therapy. Life Sci. 267, 118969(2021).
- Derry, W. B. CRISPR: Development of a technology and its applications. FEBS J. 288 (2), 358-359 (2021).
- Sharma, G., Sharma, A. R., Bhattacharya, M., Lee, S. S., Chakraborty, C. CRISPR-Cas9: A preclinical and clinical perspective for the treatment of human diseases. Mol Ther. 29 (2), 571-586 (2021).
Перепечатки и разрешения
Запросить разрешение на использование текста или рисунков этого JoVE статьи
Запросить разрешениеThis article has been published
Video Coming Soon
Авторские права © 2025 MyJoVE Corporation. Все права защищены