Method Article
방사선 민감성 및 방사선 저항성 유전자 공개를 위한 게놈 와이드 CRISPR 스크리닝
* 이 저자들은 동등하게 기여했습니다
요약
게놈 차원의 CRISPR/Cas9 스크리닝 방법을 적용하여 방사선의 저항성 및 민감성 유전자를 선택하기 위한 세심하고 구조화된 접근 방식이 제공됩니다. 이 프로토콜은 또한 임상적으로 투여되는 화학 약물에 대한 내성 메커니즘을 조사하는 다른 연구 노력을 위한 다재다능한 프레임워크 역할을 할 수 있는 잠재력을 가지고 있습니다.
초록
CRISPR-Cas9 시스템은 강력한 게놈 편집 도구로 활용되고 용도가 변경되었습니다. 이 기술을 활용함으로써 연구원들은 살아있는 세포 내에서 DNA 염기서열을 정밀하게 자르고, 붙여넣고, 심지어 다시 쓸 수도 있습니다. 그럼에도 불구하고 CRISPR 스크린 기술의 적용은 단순한 실험을 훨씬 뛰어넘습니다. 이는 유전 질환과의 싸움에서 중추적인 도구 역할을 하며, 복잡한 유전 환경을 체계적으로 해부하고, 연구자들이 생물학적 현상의 기저에 있는 분자 메커니즘을 밝힐 수 있도록 하며, 과학자들이 암, 낭포성 섬유증 및 겸상 적혈구 빈혈과 같은 질병의 근본 원인을 식별하고 표적으로 삼을 수 있도록 합니다. 무엇보다도, 암은 의학계에 만만치 않은 도전이 되고 있으며, 이를 박멸하기 위한 노력에 박차를 가하고 있습니다. 방사선 요법은 전통적인 치료법으로 결과는 나오지만 한계가 있습니다. 암세포를 박멸할 뿐만 아니라 건강한 조직을 손상시켜 삶의 질을 떨어뜨리는 부작용을 일으킵니다. 또한 모든 암세포가 방사선 요법에 반응하는 것은 아니며 일부 암세포는 내성을 일으켜 상태를 악화시킬 수 있습니다. 이 문제를 해결하기 위해 방사선에 민감한 유전자와 방사선 저항성 유전자를 효율적으로 식별할 수 있는 포괄적인 전체 게놈 CRISPR 스크리닝 기술이 도입되어 암 연구 및 치료 분야를 발전시키고 있습니다. 설명된 프로토콜에 따라 방사선 조사에 노출된 폐 선암종 세포에서 게놈 차원의 CRISPR 스크리닝을 실시했으며, 이를 통해 방사선 저항성 및 방사선 민감성 관련 유전자를 모두 확인했습니다.
서문
생물학적 현상에 대한 연구는 본질적으로 세포 행동에 대한 연구와 얽혀 있으며, 세포 행동에 대한 조사는 근본적으로 게놈 탐사와 연결되어 있습니다. 현대 기술이 계속 발전함에 따라 의학 연구자들은 다양한 질병의 치료 결과를 향상시키기 위해 유전자 편집을 통해 세포 행동을 변화시키는 데 점진적으로 관심을 돌리고 있습니다. 이와 관련하여, CRISPR(Clustered Regularly Interspaced Short Palindromic Repeats) 기술은 비교적 간단한 응용 프로그램으로 인해 게놈 편집을 위한 혁신적인 도구로 부상했습니다1. CRISPR-Cas9 시스템은 Cas9 nuclease와 single-guide RNA (sgRNA)로 구성되며, 이는 표적 DNA 염기서열을 특이적으로 인식하고 결합하여 Cas9 nuclease가 해당 위치에서 절단되도록 유도하여 게놈 DNA 2,3,4에서 이중 가닥 절단(DSB)을 초래합니다. 또한 다른 물질의 도입은 게놈에 특정 삽입, 결실 또는 돌연변이로 이어져 표적 유전자 편집을 가능하게 할 수 있습니다.
기능 유전체학 연구에서 RNA 간섭(RNAi) 스크리닝은 한때 암의 유전자 역할을 조사하기 위해 대규모 기능 상실 실험을 수행하는 데 널리 사용되는 방법이었습니다. RNAi 기술은 표적 유전자를 특이적으로 침묵시켜 유전자 기능을 연구하여 연구자들이 중요한 발암 요인을 식별하는 데 도움을 줍니다. 그러나 off-target 효과와 불완전한 유전자 녹다운 효율로 인해 제한됩니다. off-target 효과는 다른 비표적 유전자의 침묵으로 이어질 수 있으며, 이로 인해 실험 결과의 정확성과 신뢰성이 손상될 수 있습니다 1,2. 또한 RNAi는 특정 유전자에 대해 낮은 knockdown 효율을 나타내어 표적 유전자 발현을 완전히 억제하지 못할 수 있습니다. 기존의 RNAi 스크리닝과 달리 CRISPR 스크리닝은 더 높은 특이성과 효율성을 보여줍니다3. 이 기술은 특정 유전자의 정밀한 편집을 가능하게 할 뿐만 아니라 게놈 전체의 대규모 스크리닝을 가능하게 하여 유전자 기능 연구를 강력하게 지원합니다. CRISPR-Cas9 시스템을 기반으로 하는 강력한 유전자 편집 도구인 CRISPR 스크 리닝 기술은 세포 5,6,7,8 내 특정 유전자의 알려지지 않은 기능을 효율적으로 스크리닝하고 밝히는 데 사용됩니다. 연구자들은 특정 유전자 또는 유전자 영역에 대해 배치로 sgRNA를 설계하고 해당 sgRNA 라이브러리를 정밀하고 엄격하게 준비하여 무결성과 기능을 보장합니다9. 그런 다음 이러한 sgRNA 라이브러리는 렌티바이러스 입자로 캡슐화되어 숙주 세포를 효율적으로 감염시키는 데 사용됩니다. 성공적인 감염 후, 감염된 세포는 개인적으로 정의된 선별 검사 조건 하에서 배양됩니다. 스크리닝 시 스크리닝된 세포의 게놈 DNA가 추출되어 높은 수준의 순도와 양을 유지합니다. 그 후, sgRNA 관심의 표적 영역은 PCR 증폭, 핵산의 원하는 세그먼트를 정확하게 복제하는 과정인 3,9를 받습니다. 마지막으로, 증폭된 DNA 단편에 대해 고처리량 염기서열분석(high-throughput sequencing)을 수행하여 표적 부위를 포괄적이고 효율적으로 분석할 수 있게 함으로써 연구 대상 유전자의 기능과 행동에 대한 귀중한 통찰력을 얻을 수 있습니다4.
암은 복잡한 질병으로서 인간의 건강에 무서운 위협이 되고 있습니다. 전 세계적으로 연구자와 임상의는 발암의 분자 메커니즘을 밝히고 새로운 치료 전략을 개발하기 위해 공동의 노력을 기울이고 있습니다. 기초 연구 결과를 임상 애플리케이션으로 빠르게 전환하기 위해 국제 협력이 구축되었으며, 궁극적인 목표는 환자 결과를 개선하는 것입니다. Sasmal 등은 전이성 암세포를 정확하게 표적화하기 위해 합성 숙주-게스트 시스템을 기반으로 하는 생체 직교 조립 전략을 제안했으며, 이는 수십 명의 과학자들이 의료 기술을 발전시키는 데 크게 도움이 되었습니다. 이들의 뛰어난 연구 작업은 높은 혁신성과 고유한 통찰력을 가지고 있어 과학계에 의미 있는 기여를 하고 있다10. 암은 DNA 손상 반응의 불규칙한 조절로 인해 발생하는 유전체 불안정성의 격동적인 상태를 특징으로 합니다11-14. DNA 손상에는 단일 뉴클레오티드 결함, 단일 가닥 절단 및 DSB가 포함됩니다. 상동 재조합(HR) 및 비상동 말단 결합(NHEJ)은 서로 다른 단계에서 DSB의 복구에 참여합니다 15,16,17. 이러한 토대에 기초하여 방사선 요법은 고에너지 광선(예: X선 및 γ선)을 사용하여 종양 조직에 방사선을 조사하여 종양 세포의 DNA 손상을 일으킴으로써 종양 세포의 성장과 증식을 방해하는 실행 가능한 치료 옵션으로 부상했습니다18. 그러나 방사선 요법이 상당수의 암 환자에서 항상 원하는 효과를 나타내는 것은 아니며, 이는 잠재적으로 파라암 조직의 손상 및 방사선 요법에 대한 낮은 민감도와 같은 종양의 고유한 특성으로 인한 한계로 인해 발생할 수 있습니다 19,20,21.
이론적으로 모든 세포 유형을 CRISPR 스크리닝에 사용할 수 있습니다. 그러나 돌연변이된 집단에서 충분한 대표성을 유지하려면 많은 수의 시작 세포가 필요합니다. 존재 비중이 낮은 세포 유형은 게놈 전체 스크리닝에 특히 적합하지 않습니다. library의 선택과 관련하여, 대부분의 library는 target gene 당 3-6개의 gRNA를 포함하고 있으며, population 내에서 각 gRNA의 분포를 유지하는 것은 매우 중요하다18. 특정 gRNA의 농축 또는 고갈로 인한 representation의 상실은 불균일한 결과 분포로 이어질 수 있습니다. 이 문제를 해결하기 위해서는 시장 테스트를 거친 상업적으로 이용 가능한 CRISPR 라이브러리를 선택하는 것이 바람직할 수 있습니다20. 시험관 내 동질적인 암 세포주를 사용하는 CRISPR 스크리닝은 in vivo 종양의 유전적 및 후성유전학적 이질성을 완전히 포착하지 못할 수 있습니다. 체외 검사는 DNA 손상 복구 및 방사선 유도 자가분비 신호전달에 관여하는 주요 유전자를 밝혀냈지만, 저산소증 유도 방사선 저항(ROS, 대사 적응 및 자가포식을 통해 ), 면역 매개 파라크린 효과 및 ECM 의존성 사이토카인 조절을 포함한 종양 미세환경을 완전히 복제하지는 못했습니다. 방사선 민감도 또는 내성과 관련된 유전자를 탐색하기 위해 CRISPR 스크리닝을 사용하기 전에 이러한 요인을 신중하게 고려해야 합니다. 현재의 치료 환경에 비추어 볼 때, 방사선 요법의 효능을 효과적으로 향상시키기 위해서는 방사선 내성 및 방사선 민감성과 관련된 요인을 파악하고 심층적으로 연구하는 것이 시급하다22. CRISPR 스크리닝이 알려지지 않은 유전자의 기능을 연구하는 데 있어 주요 이점을 감안할 때, 방사성 및 방사선 내성 유전자를 효율적으로 식별하기 위해 체계적으로 상세한 전체 게놈 CRISPR 스크리닝 기술이 제공됩니다.
프로토콜
이 연구에 사용된 시약과 장비는 재료 표에 나열되어 있습니다.
1. 적정한 방사선 선량 선택
- 부착 세포의 준비 및 도금
- 계대 및 세포 성장을 위해 10% FBS를 함유하는 RPMI 1640 완전 세포 배양 배지를 사용하여 세포 밀도를 mL당 5 x 105 cells로 조정합니다. 세포를 3.5cm 배양 접시에 분배하여 다양한 용량의 방사선을 투여합니다. 각 접시에 2mL(1 x 10,6 세포)를 추가하고 5% CO2로 37°C에서 밤새 배양합니다.
- 다양한 방사선 선량의 적용
- 3.5cm 배양 접시에 1부터 5까지 번호를 매깁니다. 그룹 #1을 대조군으로 활용하고 나머지 4개 그룹을 치료 그룹으로 지정합니다.
- 6웰 플레이트의 가장자리를 처리 그룹에 대한 밀봉 멤브레인으로 밀봉하고 각각 2, 4, 6 및 8 Gy의 방사선량을 투여합니다. 치료 후에는 접시를 인큐베이터에 다시 넣으십시오.
- 방사선 치료 후 세포주 도금
- 6웰 플레이트와 96웰 플레이트를 준비합니다. PBS로 3.5cm 배양 접시에서 세포를 한 번 세척하고, 0.25% 트립신으로 대수적으로 성장하는 세포를 분해하고, 10% FBS를 함유한 RPMI 1640 완전 배지를 사용하여 세포 밀도를 mL당 1 x 105 세포로 조정합니다.
- 10 μL/웰(1,000 세포/100 μL)을 6-웰 플레이트에 각각 방사선 선량에 대해 3회 반복하여 시딩합니다. 각 웰에 2mL의 완전한 배지를 추가하고 14일 후(각 클론 그룹에 약 50개의 세포가 있는 경우) 계수합니다.
- 30 μL/웰(3,000 세포/100 μL)을 96웰 플레이트에 각 방사선 선량에 대해 5회 반복하여 시딩합니다. 각 웰에 70μL의 완전한 배지를 추가하고 72시간 후 세포 생존율을 측정합니다.
- 세포 생존율 측정
참고: 클론 생성 억제율 = 1 - (치료군의 클론 수/대조군의 클론 수) x 100%. CCK-8 분석은 세포 탈수소 효소에 의해 환원될 수 있는 수용성 테트라졸륨 염(WST-8)을 사용하여 수용성이 높은 노란색 포르마잔 생성물을 생성합니다11. 생산된 포르마잔의 양은 생존 가능한 세포의 수에 정비례하며, 광학 밀도(450nm 파장에서 측정)는 세포 대사 활동과 증식 상태를 정확하게 반영합니다11,13. 이 잘 정립된 원리를 기반으로 하는 CCK-8 분석은 세포 증식 분석 및 종양 약물 감수성 검사를 포함한 다양한 응용 분야에 널리 채택되었습니다. 이 프로토콜에서는 CCK-8 분석을 사용하여 다양한 방사선 선량에서 세포 생존율을 체계적으로 평가합니다.- CCK8 시약과 FBS가 없는 RPMI 1640 배지를 1:9 비율로 혼합합니다. 96웰 플레이트에서 배지를 버리고 각 웰에 100μL의 CCK8 함유 배지를 추가합니다.
- 어둠 속에서 1시간 동안 배양하고 마이크로플레이트 리더를 사용하여 450nm에서 OD 값을 측정합니다.
참고: 세포 생존율 = [(처리군의 OD 값 - 블랭크 웰의 OD 값) / (대조군의 OD 값 - 블랭크 웰의 OD 값)] x 100%
- 방사선량의 선정
- 연구 요구에 따라 적절한 방사선 선량을 선택하기 위해 클론 생성 억제율과 세포 생존율을 통합합니다. 방사선 내성 및 방사선 민감성 유전자를 모두 스크리닝하기 위해 억제율이 50%인 방사선 선량을 선택하십시오.
2. 적절한 MOI 및 퓨로마이신 농도 선택
- 부착 세포의 도금
- 세포 밀도를 3 x 10 mL당5 개 세포로 조정하고 웰당 1mL(3 x 10,5 개 세포)를 12웰 플레이트에 접종합니다. 37 ° C에서 5 % CO2로 밤새 플레이트를 배양합니다.
- 렌티바이러스 감염
- 피펫을 사용하여 12웰 플레이트에서 배지를 흡입하고 10% FBS를 함유한 1mL의 RPMI 1640 완전 배지로 교체합니다. 품질이 검사된 전체 게놈 CRISPR 렌티바이러스 라이브러리(시판되는 라이브러리 권장)를 준비하고 로그 농도 구배(예: 0-10-50-100-200-400-800)를 설정합니다.4,5,6,7,8.
- 해당 양의 렌티바이러스를 2μL/접시의 폴리브레렌에 첨가하고 실온에서 5분 동안 평형을 이룹니다. 렌티바이러스와 폴리브레인 혼합물을 각 웰에 천천히 떨어뜨리고 잘 섞은 다음 37°C에서 5% CO2로 밤새 배양합니다.
- 세포 사멸을 위한 최소 퓨로마이신 농도 측정
- Parent Cell Density를 3 x 105 cells/mL로 조정하고 웰당 1mL(3 x 105 cells)를 12-well plate에 접종합니다. 37 ° C에서 5 % CO2로 밤새 플레이트를 배양합니다.
- 12웰 플레이트의 각 웰에 0-0.1-0.2-0.5-1-2-4-8 μM의 농도 구배로 퓨로마이신을 추가합니다.72시간 후에 세포를 계수합니다. 웰 내의 모든 세포를 죽이는 최소 농도는 세포 사멸을 위한 최소 퓨로마이신 농도이며, 이는 바이러스에 감염된 세포의 후속 스크리닝에 사용됩니다.
- 감염 후 퓨로마이신 선택
- 감염 후 2일째에 각 웰에서 배지를 흡인하고 10% FBS를 함유한 RPMI 1640 완전 배지 1mL로 교체합니다. 48시간 동안 계속 배양합니다.
- 감염 72시간 후, 기존 배지를 세포 사멸을 위한 최소 퓨로마이신 농도를 함유한 완전한 배지로 교체합니다. 렌티바이러스 감염이 없는 12웰 플레이트에 2개의 웰을 음성 및 양성 대조군으로 설정합니다. 음성 대조군은 퓨로마이신으로 치료하고 양성 대조군은 치료하지 않고 그대로 둡니다. 72시간 동안 배양을 계속합니다.
- 퓨로마이신 선택 72시간 후, 음성 대조군의 모든 부모 세포는 죽는 반면, 양성 대조군의 부모 세포는 최소한의 사멸을 보입니다. 각 웰에 대한 MOI를 계산합니다.
참고: MOI = (바이러스 감염 그룹의 세포 수 / 양성 대조군의 세포 수) x 100%. 후속 스크리닝을 위해 MOI가 ~0.3인 바이러스 농도를 사용합니다.
3. 게놈 전체 CRISPR 렌티바이러스 라이브러리 감염
- 부착 세포주(adherent cell line)의 파스(seeding)
- 계대 배양 및 세포 성장을 위해 10% FBS를 포함하는 RPMI 1640 완전 배지에서 세포 밀도를 1 x 10mL당 7 세포로 조정합니다. 37°C에서 각 15cm 배양 접시에 1mL의 세포(1 x 10,7 개 세포)를 5% CO2 로 8시간 동안 접종합니다. 세포가 부착되어 70%-80%의 합류점에 도달하면 바이러스 감염에 대한 준비가 된 것입니다(복제 수는 약 500개).
- 렌티바이러스 감염
- 15cm 배양 접시에서 배지를 흡입하고 10% FBS를 함유한 15mL의 RPMI 1640 완전 배지로 교체합니다. 품질이 검사된 전체 게놈 CRISPR 렌티바이러스 라이브러리를 준비합니다.
- MOI = 0.3 - 30 μL/접시의 폴리브레렌과 함께 해당 양의 렌티바이러스를 첨가하고 실온에서 5분 동안 평형을 이루고, 렌티바이러스와 폴리브레렌 혼합물을 15cm 배양 접시에 천천히 떨어뜨리고 잘 섞은 다음 37°C에서 5%CO2 로 하룻밤 동안 배양합니다. 동시에 부모 세포를 대조적으로 하는 15cm 배양 접시를 준비합니다.
- 감염 후 퓨로마이신 선택
- 바이러스 감염 후 2일째에 15cm 배양 접시에서 배지를 흡인하고 10% FBS를 함유한 15mL의 RPMI 1640 완전 배지로 교체합니다. 48시간 동안 계속 배양합니다.
- 감염 후 72시간이 지나면 배지를 퓨로마이신의 최소 치사 농도를 함유한 완전한 배지로 교체하십시오. 감염되지 않은 부모 세포를 동일한 방식으로 음성 대조군으로 취급하고 72시간 동안 배양을 계속합니다. 퓨로마이신 선택 72시간 후, 음성 대조군의 부모 세포는 모두 사멸되고 렌티바이러스 감염에서 살아남은 세포는 성공적으로 감염된 것으로 간주됩니다.
- Day 0 게놈 추출
- 0.25% 트립신을 사용하여 15cm 배양 접시 1개에서 세포를 분해하고, 10% FBS를 함유한 RPMI 1640 완전 배지에 재현탁하고, 세포 수를 계수합니다. 이 샘플에 Day 0이라는 레이블을 지정합니다.
- 300 x g 에서 5분(실온에서) 원심분리기를 하고 상층액을 버립니다. PBS 1mL에 재현탁하고 300 x g 에서 5분 동안 원심분리한 후 상층액을 버립니다. Day 0 게놈 DNA를 추출하고 나노드롭 UV 분광 광도계를 사용하여 DNA 농도와 순도를 측정합니다.
- sgRNA integrity 검출을 위해 agarose gel electrophoresis를 사용하여 증폭된 sgRNA 라이브러리를 분석하여 band가 깨끗하고 심각한 저하가 없는지 확인하여 library23의 무결성을 보존합니다. 렌티바이러스 라이브러리 커버리지 평가를 위해 심층 염기서열분석 기술을 사용하여 라이브러리 내 sgRNA에 대한 염기서열분석 분석을 수행합니다24,25.
- CRISPR 스크리닝에서 PCR 증폭은 sgRNA 단편 무결성 및 라이브러리 품질에 대한 예비 검증 역할을 합니다6. 라이브러리 커버리지 및 sgRNA 분포 패턴을 보다 종합적으로 분석하고 스크리닝 중 각 타겟 유전자에 대한 sgRNA의 적절한 표현을 보장하기 위해 NGS를 수행하여 스크리닝9의 견고성에 대한 심층적인 통찰력을 얻을 수 있습니다.
참고: 스크리닝 전후 sgRNA 함량의 변화를 감지하고 잠재적인 sgRNA off-target integration 또는 라이브러리 오염을 식별함으로써 CRISPR 스크리닝의 정확도와 신뢰성을 더욱 향상시킬 수 있습니다. 이러한 Day 0 샘플은 중요한 음성 대조군 역할을 하며 PCR 증폭 및 NGS를 통한 포괄적인 품질 평가를 거쳐 2가지 주요 목표를 달성합니다: (1) 필수 유전자 고갈 확인(적절한 라이브러리 표현 표시) 및 (2) 비필수 유전자의 안정적인 발현 입증(실험 기준선 조건 설정)9. - 도서관의 다양성과 대표성을 보장하고 심사 과정에서 편견을 피하기 위해 적용 범위가 예상 수준에 도달하도록 합니다. 감염 효율 측정의 경우 형광 이미징을 사용하여 감염 효율을 평가하고 실험 요구 사항을 충족하는지 확인합니다.
4. 방사선을 선별 조건으로 적용
- 그룹화
- CRISPR에 감염된 세포의 밀도를 mL당 1 x 107 세포로 조정하고 각 15cm 페트리 접시에 1mL를 접종합니다. 37 ° C에서 5 % CO2로 밤새 배양하십시오.
- 방사선 치료
- 세포를 무작위로 2개 그룹(치료 그룹과 대조군)으로 나누고 그룹당 6개의 접시를 제공합니다. 치료군의 세포에 적절한 선량의 방사선을 투여하고 대조군 세포는 정상적으로 증식하도록 처리하지 않은 상태로 둡니다. 방사선 투여 후 37 ° C에서 5 % CO2 로 7 일 동안 계속 배양합니다.
- 두 번째 주에는 치료군 세포에 적절한 용량의 방사선을 반복적으로 투여하여 대조군 세포는 치료하지 않고 정상적으로 증식할 수 있도록 합니다. 방사선 요법 후 37 ° C에서 5 % CO2 로 7 일 동안 계속 배양합니다.
5. 게놈 추출 및 염기서열분석
- 14일째 추출
- 처리 14일 후, 0.25% 트립신으로 처리군과 대조군의 세포를 분해하고, 10% FBS를 함유한 완전한 RPMI 1640 배지를 사용하여 재현탁한 후 Day 14-RT 또는 Day 14-NC로 라벨링합니다.
- 300 x g 에서 5분 동안 세포를 원심분리하고 상층액을 버립니다. PBS 1mL에 재현탁하고 300 x g 에서 5분 동안 원심분리한 후 상등액을 다시 버립니다. Day 14의 게놈 DNA를 추출하고 nanodrop UV 흡광도 분광법을 사용하여 DNA의 농도와 순도를 검출합니다.
- PCR 증폭
- 서열의 해당 프라이머를 준비하고( 재료 표 참조) 10μM으로 희석합니다.멸균 마이크로 원심분리기 튜브에 시약을 첨가하여 20μL 반응 시스템을 설정하고 300 x g에서 5초 동안 원심분리한 후 잘 혼합합니다. 증폭 조건에 따라 PCR 기기 매개변수를 설정하여 PCR 산물을 얻습니다.
- Agarose gel 전기영동 검출
- 겔 주조 플레이트를 준비하고, 아가로 스로의 가장자리를 밀봉하고, 빗을 삽입하고, 샘플 DNA의 길이에 따라 적절한 농도의 아가로 스 젤을 준비합니다.
- 일정량의 아가로스 분말을 정확하게 계량하고 적당량의 전기영동 버퍼를 첨가하여 잘 섞어 전자레인지에서 가열하여 녹입니다. 약간 식힌 후 적당량의 핵산 염료를 넣고 부드럽게 섞어 겔 주조 금형에 천천히 붓습니다. 젤이 30분 동안 굳어지도록 합니다.
- 빗을 제거하고 겔을 덮을 때까지 전기영동 탱크에 적절한 양의 전기영동 완충액을 추가합니다. DNA 샘플에 적절한 양의 로딩 버퍼를 추가하고 잘 혼합한 후 피펫을 사용하여 혼합물을 샘플 웰에 천천히 첨가합니다.
- DNA 단편의 크기와 아가로스 겔의 농도에 따라 적절한 전압을 설정합니다. 전기영동 후 겔을 조심스럽게 제거하고 겔 이미저에 넣어 결과를 관찰하고 PCR 증폭이 성공적이었는지 확인합니다.
- Illumina 염기서열분석
- 3개 그룹(Day 0 그룹, 대조군, 치료군)에서 유전체 DNA를 수집하여 라이브러리 구축 및 Illumina 염기서열분석을 위해 회사에 보냅니다. 생물정보학적 분석과 염기서열분석 결과의 시각화를 수행하여 방사선 치료에 민감하고 방사선 치료에 내성이 있는 유전자를 얻습니다26.
- 데이터 분석을 위해 여러 번 반복되는 실험을 설정하고 실험 결과에 대한 통계 분석을 수행하여 데이터의 일관성과 재현성을 보장합니다27,28.
- 염기서열분석 데이터의 품질 평가
- 염기서열분석 정보의 정확성을 보장하기 위해 원시 데이터에 엄격한 필터링 및 품질 관리 메커니즘을 적용합니다5. 지정된 변환 알고리즘과 염기 호출28 중에 오류가 발생할 가능성을 평가하는 모델을 사용하여 염기서열분석 오류율을 기반으로 각 뉴클레오티드에 대한 Phred 점수(Qphred)를 계산합니다.
- 각 염기 위치에 대한 염기서열분석 오류율을 1%(Q30 임계값에 해당) 미만으로 유지하고, 염기서열분석 데이터의 최소 80%가 이 Q30 표준을 달성하여 후속 분석 절차를 지원하도록 합니다.
- 데이터 처리 및 시료 수준 품질 관리
참고: 자세한 내용은 이전에 발표된 보고서29,30을 참조하십시오.- 염기서열분석 데이터에서 필터링된 read를 sgRNA 라이브러리 염기서열과 정렬합니다. 완벽하게 정렬된 라이브러리의 sgRNA 수, sgRNA의 평균 함량, 검출되지 않은 sgRNA의 수, sgRNA 라이브러리에 성공적으로 매핑된 개별 샘플의 판독 비율을 포함한 통계를 보고합니다. 더 높은 매핑 비율을 사용하여 더 큰 적용 범위를 나타냅니다.
- 각 샘플에서 각 유전자(서로 다른 sgRNA에 의해 표적화됨)에 대한 판독 농축을 계산합니다. MAGeCK의 "중앙값" 정규화 방법을 사용하여 샘플 전체에서 각 sgRNA에 대한 지원 read를 정규화합니다. sgRNA 판독 횟수의 분포를 평가하고, 상자 플롯을 생성하고, 주성분 분석(PCA)을 수행하고, 상관 관계 히트맵을 구성하여 샘플 수준의 품질 관리를 수행합니다.
- sgRNA 판독 횟수가 푸아송 분포를 따른다고 가정합니다. 서로 다른 그룹의 정규화된 sgRNA 수를 상자 그림으로 묘사하여 샘플 내의 전체 데이터 분포를 시각화하고 그룹 간의 분포를 비교합니다.
- PCA를 사용하여 주성분 간의 차이와 각 성분 내의 변동률을 반영함으로써 데이터 분석을 단순화하고 그룹 내 및 그룹 간 변동을 쉽게 관찰할 수 있습니다. 상관 관계 히트맵을 사용하여 샘플 간의 관계를 설명할 수 있습니다.
- 기본 분석
- 그룹 간 차등 sgRNA 분석을 수행하고 품질 관리 및 예비 데이터 처리 후 필수 유전자를 식별합니다. 관련 유전자의 기능적 농축 분석을 수행한다30. MAGeCK 또는 MLE와 같은 다른 통계적 방법으로 계산한 RRA(Robust Rank Aggregation) 점수를 기반으로 필수 유전자의 순위를 매기며, RRA 점수가 낮을수록 중요도가 높음을 나타냅니다.
- R 언어 또는 다른 프로그래밍 언어를 사용하여 순위가 매겨진 결과에 대한 생물정보학 분석을 수행하고, GO 및 KEGG 분석 다이어그램30을 사용하여 결과를 시각화한다. ID 매핑 또는 염기서열 주석을 통해 유전자 또는 단백질을 해당 GO 용어(molecular function, biological process 및 cellular component)에 연결합니다.
- KEGG를 데이터베이스로 사용하여 높은 수준의 기능과 생물학적 시스템을 이해하고, 식별된 유전자 카탈로그를 세포, 종 및 생태계 수준의 시스템 기능에 연결합니다. GO 및 KEGG 분석에서 상위 10개 또는 20개의 경로를 선택하여 경로 방향을 직관적으로 표시할 수 있습니다.
결과
사망률이 가장 높은 폐암은 매우 공격적이고 널리 퍼져 있는 의학적 질병입니다. 폐암 세포주 A549를 예로 들어 방사선을 스크리닝 조건으로 하여 게놈 전체 CRISPR 스크리닝을 수행하는 데 개략적인 워크플로우를 그림 1에 보여주었습니다. 먼저, 클론 형성 및 CCK8 실험을 통해 다양한 방사선 선량에 대한 A549 세포의 민감도를 조사합니다(그림 2). 클론 생성 분석에서 콜로니 수는 2 Gy에서 140 ± 5.35 대 0 Gy에서 303 ± 31.63이었던 반면, CCK-8 분석에서 광학 밀도(OD) 값은 2 Gy에서 0.65 ± 0.05 대 0 Gy에서 1.35 ± 0.08이었습니다. 방사선 민감도 및 방사선 저항에 대한 후보 유전자를 조사하려면 후속 방사선 선량으로 2 Gy(IC50)를 선택합니다. A549 세포는 다른 용량의 푸로마이신으로 처리되고 72시간 후에 계수되었습니다. A549 세포에 대한 퓨로마이신의 최소 사멸 농도는 1μM로 검출되었습니다(그림 3). A549 세포는 서로 다른 구배 MOI에서 렌티바이러스에 감염되었습니다. 감염 72시간 후, 감염 효율을 형광 현미경으로 관찰하고 퓨로마이신 치료 72시간 전후에 계수했습니다. 감염 효율이 ~0.3인 MOI를 얻었습니다(표 1). 인간 게놈에는 19,050개의 유전자가 포함되어 있으며 각 유전자에 해당하는 6개의 sgRNA와 500(19,050 x 6 x 500 = 5.7 x 107 세포)의 복제 수가 있습니다. MOI = 0.3인 렌티바이러스를 사용하여 5.7 x 107 A549 세포를 감염시킵니다. 72시간 후, 세포를 추가로 72시간 동안 1μM 퓨로마이신으로 처리했습니다. Day 0 게놈을 수확하고 PCR 증폭을 수행했습니다. CRISPR 전체 게놈 라이브러리 내의 sgRNA 염기서열은 231개의 염기쌍 길이를 나타내며, 이는 아가로스 겔 전기영동 결과에 의해 확증됩니다(그림 4).
PCR 산물은 이후 데이터 품질 관리 및 샘플 수준 품질 관리를 위해 Novogene으로 보내졌습니다. 매핑 비율은 약 60%의 포괄적인 커버리지 비율을 산출했으며, 이는 전체 게놈 처리 중 순전히 복잡성과 불가피한 감소를 감안할 때 전체 게놈 스크리닝 절차에 적합한 것으로 간주되는 지표입니다(그림 5). sgRNA 판독 횟수는 이론적 기대치에 부합하는 푸아송 분포를 고수했습니다. PCA 플로팅 및 상관 관계 히트 매핑을 통한 후속 분석은 그룹 간 변동이 그룹 내 불일치보다 현저히 크면서 뚜렷한 그룹 간의 식별 가능한 차이를 생생하게 묘사했습니다. 더욱이, 그룹화된 샘플 내의 변동률은 허용 가능한 범위 내에 있었으며, 이는 샘플 수준 품질 관리 측정의 성공을 입증했습니다. 그 후, MAGeCK이 고안한 RRA 순위는 R 언어를 사용하여 순위 결과에 대한 기초적인 생물정보학 평가에 착수하는 데 활용될 것입니다. 특히, GO 측면에서 상위 15개 경로는 DNA 손상과 관련된 메커니즘을 두드러지게 특징으로 했으며, 이는 실험의 기본 스크리닝 기준과 완벽하게 일치합니다.
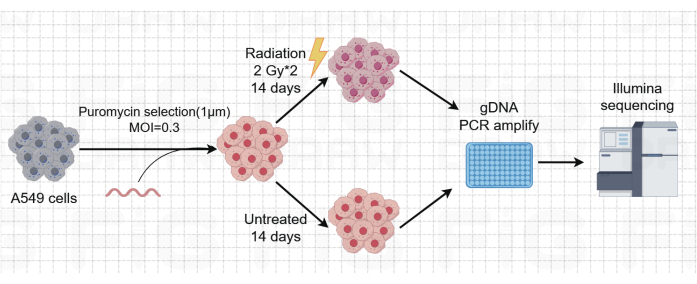
그림 1: A549 폐암 세포에서 게놈 전체 방사선 요법 CRISPR/Cas9 스크리닝의 개략도. 게놈 전체 CRISPR/Cas9 및 방사선 요법을 사용한 A549 세포의 스크리닝 워크플로우를 개략적으로 표현한 것입니다. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
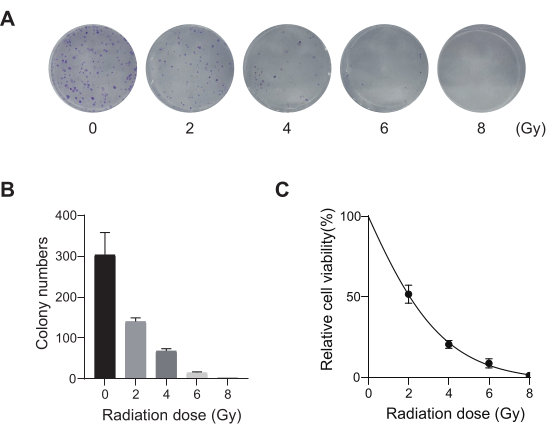
그림 2: A549 세포에 대한 방사선 선량 최적화. (A) A549 세포는 증가하는 방사선 선량(0 Gy, 2 Gy, 4 Gy, 6 Gy 및 8 Gy)에 노출되어 14일 동안 성장하도록 했습니다. 집락 형성 분석 이미지가 표시됩니다. 결과는 두 가지 독립적인 생물학적 실험을 대표합니다. (B) 클론 형성 분석 이미지의 정량화 결과. 오차 막대는 표준 편차(n = 3)를 나타냅니다. 일원 분산 분석은 P < 0.01을 산출했습니다. 결과는 두 가지 독립적인 생물학적 실험을 대표합니다. (C) 다른 방사선 선량에 노출된 후 A549 세포의 선량-반응 생존 곡선. 오차 막대는 표준 편차(n = 3)를 나타냅니다. 일원 분산 분석은 P < 0.01을 산출했습니다. 결과는 두 가지 독립적인 생물학적 실험을 대표합니다. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
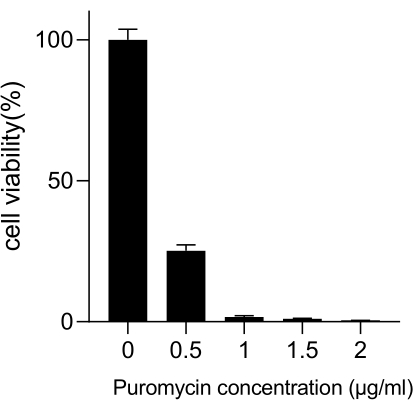
그림 3: A549 세포 선택에 필요한 최소 퓨로마이신 농도 측정. A549 세포를 증가하는 농도의 퓨로마이신으로 처리하고 72시간 동안 배양했습니다. 오차 막대는 표준 편차(n = 3)를 나타냅니다. 일원 분산 분석은 P < 0.01을 산출했습니다. 결과는 두 가지 독립적인 생물학적 실험을 대표합니다. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
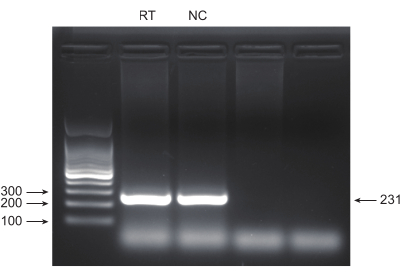
그림 4: 아가로스 겔 전기영동에 의한 PCR 산물 검증. 아가로스 겔 전기영동을 사용하여 PCR 산물 밴드의 존재 및 품질을 검사했습니다. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
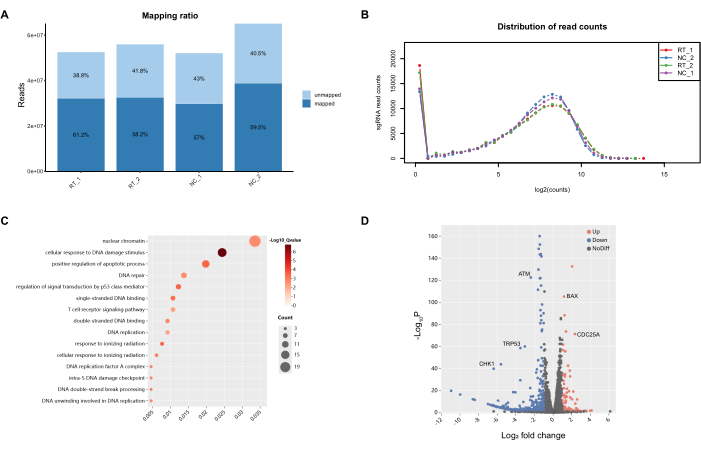
그림 5: PCR 증폭 제품의 품질 관리 및 기본 분석. (A) 매핑 비율은 참조 게놈의 약 60% 커버리지를 나타냈습니다. (B) sgRNA의 판독 횟수 분포는 푸아송 분포를 따랐습니다. (C) 상위 10개 유전자 온톨로지(GO) 용어 중 대부분은 DNA 손상 반응과 관련이 있었습니다. (D) 유전자 농축 패턴을 보여주는 화산 그래프로, 음의 농축 유전자는 빨간색, 양의 농축 유전자는 파란색, 유의하지 않은 유전자는 회색입니다. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
| 바이러스 입자 숫자(VP) | 카운트 번호 (퓨로마이신 치료 전) | 카운트 번호 (퓨로마이신 치료 후) | 감염 능률 |
| 1.23 × 104 | 1.23 × 105 | 1.04 × 104 | 0.084552846 |
| 3.72 × 104 | 1.24 × 106 | 3.61 × 104 | 0.291129032 |
| 6.15 × 104 | 1.23 × 107 | 5.4 × 104 | 0.43902439 |
표 1: 다양한 바이러스 입자에서 퓨로마이신 선택 전후의 A549 세포 수. 다양한 바이러스 입자 하에서 퓨로마이신 처리 전후에 기록된 A549 세포의 수.
토론
최첨단 유전자 편집 기술인 CRISPR 스크리닝은 과학 연구 분야에 중대한 변화를 불러일으켰습니다5. CRISPR-Cas9 시스템에서 비롯된 이 기술은 높은 효율성과 정밀도로 인해 유전자 기능을 연구하는 데 필수적인 도구가 되었습니다9. CRISPR/Cas9 엔지니어링 원리는 약 20개의 뉴클레오티드를 가진 특정 sgRNA를 설계하고 도입하여 Cas9 nuclease가 타겟 DNA 염기서열을 정확하게 찾고 절단하도록 유도하여 knockout, activation 또는 repression6과 같은 유전자 편집을 가능하게 하는 것입니다. 이 원리는 고처리량 및 맞춤형 CRISPR 스크리닝 개발의 기초가 됩니다. 방대한 수의 sgRNA를 포함하는 라이브러리를 구축함으로써 연구자들은 여러 유전자를 동시에 편집하고 스크리닝하여 특정 표현형 또는 질병 상태와 관련된 유전자를 신속하게 식별할 수 있습니다. 또한 CRISPR 스크리닝을 통해 다양한 세포 유형, 조직 및 전체 유기체에 대한 실험이 가능하여 유전자 기능과 작용 메커니즘에 대한 보다 포괄적인 이해를 제공합니다.
기술 개발이 가속화됨에 따라 CRISPR 스크린은 다양한 분야에서 적용할 수 있는 엄청난 잠재력을 보여주었습니다. 예를 들어, CRISPR 스크리닝은 과학자들이 새로운 약물 표적을 발견하는 데 도움이 될 수 있으며, 이를 통해 약물 발견 및 개발 과정을 가속화할 수 있습니다. 질병 연구에서 이 기술은 질병 발병 기전을 밝혀 진단 및 치료를 위한 새로운 통찰력과 접근 방식을 제공합니다. CRISPR 스크리닝은 몇 가지 주요 측면에서 기존 스크리닝 방법(예: RNAi 및 소분자 억제제)과 근본적으로 다릅니다: 기계론적으로 CRISPR은 유전자 편집을 통해 영구적인 유전자 변형을 달성하는 반면, RNAi는 유전자 발현을 일시적으로만 억제하고 소분자 억제제는 단백질 기능을 가역적으로 차단합니다28,29. 특이성 측면에서 CRISPR은 정확한 DNA 표적화를 통해 우수한 정밀도를 보여주는 반면 RNAi는 off-target 효과가 발생하기 쉽습니다 3,5. 효과 지속 기간과 관련하여, CRISPR은 기존 방법의 일시적인 개입에 비해 영구적인 유전적 변형을 일으킵니다. 또한 CRISPR는 게놈 전체 스크리닝 기능을 가능하게 하고 다양한 세포 유형에 걸쳐 높은 효율성을 유지하면서 여러 유전자/경로를 동시에 조사할 수 있습니다9. 기존 접근법은 일반적으로 특정 경로 분석에 국한되며 특정 세포주에서 효능이 감소하는 경우가 많습니다3. 이러한 독특한 특성으로 인해 CRISPR 스크리닝은 기능적 유전체학 연구 및 표적 발견에 특히 유용하며, 특히 장기 관찰 또는 정확한 유전자 조절이 필요한 연구에서 기존 스크리닝 방법론에 비해 고유한 이점을 보여줍니다.
그러나 이 과정의 특이성은 절대적이지 않으며, Cas9 단백질이 의도한 표적 염기서열5와 유사하지만 완전히 동일하지는 않은 DNA 영역을 잘못 인식하고 절단할 수 있습니다. 이러한 현상을 일반적으로 off-target effect라고 합니다. 이러한 의도하지 않은 DNA 절단은 특히 인간 치료에 적용될 때 게놈 불안정성과 예측할 수 없는 유전적 결과를 초래할 수 있습니다. CRISPR 기술은 복잡한 게놈 편집 작업을 처리할 때도 어려움을 겪습니다. 수많은 유전자, 유전자 클러스터 또는 복잡한 유전자 설정을 포괄하는 편집 요구에 직면할 때 CRISPR 시스템은 편집 프로세스1의 결과를 정확하게 예측하고 지시하는 데 한계가 있을 수 있습니다.
CRISPR 기술을 사용하여 게놈 전체 스크리닝을 수행하는 데 있어 가장 큰 기술적 장애물은 수만 개의 유전자를 포함하는 게놈의 방대한 복잡성으로 인해 off-target effect의 위험이 크게 증가한다는 것입니다9. 후속 염기서열분석 결과의 무결성과 신뢰성을 보장하려면 각 세포에 단일 sgRNA23을 transfection하는 것이 필수적입니다. 이를 위해서는 실험자가 MOI를 세심하게 제어하여 MOI가 0.5 미만으로 유지되도록 하는 동시에 적절한 복제 수 임계값(500 이상 24)을 유지해야 합니다. 전체 게놈에 있는 수만 개의 유전자 각각이 4-6개의 sgRNA의 활용을 필요로 한다는 사실을 감안할 때, 전체 스크리닝 과정에는 광범위한 양의 세포가 필요합니다25. 실험 기간이 길어지고, 관련된 세포의 수가 많으며, 위험이 높아짐에 따라 실험자는 이러한 우려를 완화하기 위한 전략을 고안해야 한다26. 잠재적인 유전자는 RNA-seq와 gain-of-function 및 loss-of-function assay를 통해 추가로 검증되어야 합니다. 다음 실험에서는 CRISPR 편집을 사용하여 식별된 유전자의 과발현 및 녹아웃 세포를 얻은 후, 특히 비특이적 Cas9 절단을 거칠 수 있는 부분 sgRNA 상보성이 있는 유전자좌에서 잠재적인 off-target 변형에 접근하기 위해 게놈 전체 유전체학 및 전사체학을 수행했습니다. COSMID와 같은 생물정보학 예측 도구는 고위험 비표적 부위를 식별하는 데 도움이 될 수 있습니다. 구조 분석은 또한 확인된 유전자의 관여의 특이성을 보장하는 데 사용될 수 있습니다.
후속 분석에서 차등적으로 발현된 sgRNA에 대한 기준은 MAGeCK에 의해 계산된 RRA(Robust Rank Aggregation) 점수에 의존했으며, 여기에는 통계적 유의성, 접힘 변화 및 복제 일관성31,32가 포함되었습니다. 데이터 세트의 경우 각 유전자는 유전자 발현 수준과 통계적 유의성을 결합한 포괄적인 기준에 따라 순위가 매겨집니다. RRA는 순위에 대한 Spearman의 순위 상관 계수를 활용하여 모든 데이터 세트에서 각 유전자의 포괄적인 순위 상황을 평가하고 여러 유전자 발현 데이터 세트의 정렬 결과를 통합합니다. 대부분의 데이터 세트에서 상대적으로 높은 순위를 차지한 유전자는 진정한 차등 발현 유전자로 간주될 수 있습니다33. RRA의 p-값이 0.05 미만이면 통계적으로 유의한 것으로 간주되었습니다. 최소 1.5배 증가 또는 33% 감소의 폴드 변화는 차등 표현으로 간주되었습니다. 또한, 3개의 독립적인 반복실험의 일관성은 통계적으로 다른 발현으로 간주되어야 했습니다. 필수 유전자에 대한 RRA 순위 지정 후 RRA 점수가 낮을수록 중요도가 높다는 것을 의미합니다.
게놈 편집을 위한 강력한 도구인 CRISPR-Cas9 기술은 방사선 저항 영역을 조사하는 데 있어 타의 추종을 불허하는 잠재력과 정확성을 보여줍니다26,27. 그러나 응용 프로그램에는 여전히 제한 사항이 있습니다. off-target effect의 위험은 차치하고라도, 방사선 민감도와 관련된 복잡성으로 인해 스크리닝 과정이 복잡해진다27. 이러한 민감성은 단순히 단독의 유전자에 의해 지배되는 것이 아니라, 여러 유전자와 그 조절 네트워크 사이의 복잡한 상호작용의 결과로 나타난다26. 따라서 방사선 민감성과 관련된 특정 유전자의 성공적인 확인에도 불구하고 이러한 유전자는 전체 그림의 일부에 불과할 수 있으며, 기본 메커니즘을 포괄적으로 밝히는 데 상당한 어려움을 제기할 수 있습니다 34,35,36. 낮은 감염 효율, sgRNA 저하 및 염기서열분석 실패와 같은 CRISPR 스크리닝의 일반적인 문제에 대한 해결책은 다음과 같습니다. CRISPR 스크리닝에는 <0.5의 MOI 값이 필요하기 때문에 세포가 로그 성장 단계에 있는지 확인하거나 형광 분석을 통해 감염 효율을 평가하기 위해서는 세포 상태 조정이 중요합니다. sgRNA 분해의 잠재적 원인으로는 뉴클레아제 오염 또는 벡터 설계 결함 등이 있으므로 sgRNA 라이브러리의 반복적인 동결-해동 주기는 피해야 하며(-80 °C에서 부분 표본 보관) NGS 염기서열분석을 사용하여 sgRNA 라이브러리 무결성을 확인해야 합니다. 염기서열분석 실패는 염기서열분석 깊이가 충분하지 않거나 sgRNA 프라이머 설계 문제로 인해 발생할 수 있으며, sgRNA 염기서열분석 깊이를 >500× 커버리지 37,38,39,40,41,42로 하여 sgRNA 염기서열분석 정확도를 개선하기 위해 paired-end 염기서열분석이 권장됩니다.
방사선 요법은 암 치료의 초석으로 남아 있지만, 종양 유형에 따라, 심지어 동일한 암을 앓고 있는 환자들 사이에서도 치료 효과에 있어 현저한 차이를 보인다9. 이러한 개인간 이질성으로 인해 최적의 종양 박멸을 달성하기 위해 병용 요법이 필요한 경우가 많다11,12. 임상에서 방사선 요법과 면역 요법, 특히 면역 관문 억제제와 항체-약물 접합체의 통합은 저명한 치료 패러다임으로 부상하고 있다19. 이 프로토콜은 상대적으로 균질한 암 세포주를 사용하여 in vivo 종양의 유전적 및 후성유전학적 이질성을 완전히 반영할 수 없는 in vitro CRISPR 스크리닝을 제공합니다. 스크리닝을 위한 적절한 방사선 선량을 결정한 후, 그 결과는 시험관 내 세포 분석과 생체 내 동물 모델 모두에서 추가로 확인해야 하는 잠재적인 방사선 또는 방사선 내성 유전자를 제공합니다. 또한, in vivo CRISPR 스크리닝과 단세포 전사체학은 종양 이질성의 복잡성을 해독하는 데 도움이 될 수 있습니다. 시험관 내 CRISPR 스크리닝은 세포 내 DNA 손상 복구와 방사선에 대한 반응으로 자가분비 메커니즘에서 중요한 유전자를 확인했지만 실제로 이질적인 TME를 완전히 복제하지는 않았습니다. 예를 들어, 저산소증은 ROS 생성을 증가시켜 대사 적응, 항산화 생성 및 자가포식 활성화를 자극하는 피드백 루프를 유발하여 궁극적으로 방사선 내성을 촉진합니다18. 방사선 치료 후 부분비 메커니즘에서 면역 세포가 활성화되어 방사선 민감성을 유발할 수 있습니다14. ECM은 성장 인자의 저장소 역할을 하며, 사이토카인은 방사선 민감도에 긍정적인 영향과 부정적인 영향을 모두 미칩니다21. in vivo CRISPR 스크리닝은 TME의 이질성을 반영하기 위해 수행되어야 합니다. 또한, 체외 CRISPR 스크리닝은 방사선에 대한 반응으로 자가분비 메커니즘뿐만 아니라 세포 내 DNA 손상 복구에서 중요한 유전자만 식별할 수 있었지만 종양 혈관 구조 및 전신 효과를 포함하여 방사선 민감도에 대한 생체 내 영향을 완전히 포착하지 못했습니다20,21. in vivo CRISPR 스크리닝을 수행하여 in vivo 방사선 민감도와 관련된 잠재적 요인을 식별해야 합니다.
여기서, 방사선 요법에 대한 민감성 또는 내성과 관련된 유전자를 정확히 찾아내기 위해 CRISPR 게놈 전체 스크리닝을 활용하는 것을 목표로 하는 포괄적인 프로토콜이 고안되었습니다. 이 방법론은 새로운 경로를 밝혀냄으로써 종양 방사선 요법 연구의 발전을 위한 길을 닦고자 합니다. 이 프로토콜은 또한 화학 요법의 임상적 제약 메커니즘과 대체 치료제의 진행을 탐구하기 위한 참고 자료가 될 수 있습니다. 요약하면, CRISPR 기술은 상당한 잠재력과 가치를 내포하고 있지만, 그 내재적 제약으로 인해 냉정한 인식과 신중한 접근의 중요성이 강조되고 있습니다. 향후 연구 및 응용 분야가 탐구됨에 따라 CRISPR 기술의 제약을 극복하고 잠재력을 최대한 활용하기 위해 CRISPR 기술을 탐색하고 최적화하기 위해 끊임없이 노력해야 합니다.
공개
없음.
감사의 말
본 연구는 후베이성 지역과학기술혁신사업(2024EIA001)과 우한대학교 중난병원 의료과학기술혁신플랫폼 구축지원사업(PTXM2025001)의 지원을 받았다. 그림 1 은 Figdraw를 사용하여 만들었습니다.
자료
| Name | Company | Catalog Number | Comments |
| 150 mm cell and tissue culture dish | Wuxi NEST Biotechnology Co., Ltd. | 715011 | Cell culture |
| 35 mm cell and tissue culture dish | Wuxi NEST Biotechnology Co., Ltd. | 706011 | Cell culture |
| A549 cell line | ATCC | - | - |
| Cell counting kit-8 | Shanghai Beyotime Biotech Inc | C0041 | For cell viability assay |
| CO2-independent medium | PHCbi | MCO-50AIC | Cell culture |
| Countess 3 FL automated cell counter | Themo Scientific | AMQAF2001 | For cell counting |
| EDTA | Gibco | 25200056 | Cell culture |
| Fetal bovine serum | Gibco | 10099141 | Cell culture |
| Fluorescence microscopy | LEICA | ebq 100-04 | For fluorescence microscope |
| Genome-wide CRISPR lentiviral library | Shanghai OBiO Technology Co., Ltd. | H5070 | For lentiviral infection |
| MiniAmp plus PCR | Themo Scientific | C37835 | For PCR amplification |
| NEST cell culture plates, 12-well | Wuxi NEST Biotechnology Co., Ltd. | 712002 | Cell culture |
| NEST cell culture plates, 6-well | Wuxi NEST Biotechnology Co., Ltd. | 703002 | Cell culture |
| NEST cell culture plates, 96-well | Wuxi NEST Biotechnology Co., Ltd. | 701001 | Cell culture |
| PBS | Gibco | C10010500BT | Cell culture |
| PCR forward primer (AATGGACTATCATATGCTTACCGTAACTTGAAAGTATTTCG) | Beijing AuGCT Biotech Co., Ltd. | - | For PCR amplification |
| PCR reverse primer (GATGTGCGCTCTGCCCACTGACGGGCA) | Beijing AuGCT Biotech Co., Ltd. | - | For PCR amplification |
| Polybrene | Shanghai Beyotime Biotech Inc | C0351 | For lentiviral infection |
| Puromycin | MCE | HY-K1057 | For seletion post lentiviral infection |
| RPMI 1640 cell culture medium | Gibco | 23400-021 | Cell culture |
| TIANGENamp genomic DNA kit | Beijing TIANGEN Biotech Co.,Ltd. | DP304 | For genomic DNA extraction |
| X-ray powder diffractometer | PerkinElmer | For radiotherapy |
참고문헌
- Barrangou, R., Marraffini, L. A. CRISPR-Cas systems: Prokaryotes upgrade to adaptive immunity. Mol Cell. 54 (2), 234-244 (2014).
- Joung, J., et al. Genome-scale CRISPR-Cas9 knockout and transcriptional activation screening. Nat Protoc. 12 (4), 828-863 (2017).
- Sanjana, N. E., Shalem, O., Zhang, F. Improved vectors and genome-wide libraries for CRISPR screening. Nat Methods. 11 (8), 783-784 (2014).
- Arroyo, J. D., et al. A Genome-wide CRISPR death screen identifies genes essential for oxidative phosphorylation. Cell Metab. 24 (6), 875-885 (2016).
- Sidik, S. M., et al. A genome-wide CRISPR screen in toxoplasma identifies essential apicomplexan genes. Cell. 166 (6), 1423-1435 (2016).
- Jing, Y., et al. Genome-wide CRISPR activation screening in senescent cells reveals SOX5 as a driver and therapeutic target of rejuvenation. Cell Stem Cell. 30 (11), 1452-1471 (2023).
- Shifrut, E., et al. Genome-wide CRISPR screens in primary human t cells reveal key regulators of immune function. Cell. 175 (7), 1958-1971 (2018).
- Shalem, O., et al. Genome-scale CRISPR-Cas9 knockout screening in human cells. Science. 343 (6166), 84-87 (2014).
- Katti, A., Diaz, B. J., Caragine, C. M., Sanjana, N. E., Dow, L. E. CRISPR in cancer biology and therapy. Nat Rev Cancer. 22 (5), 259-279 (2022).
- Sasmal, R., et al. Synthetic host-guest assembly in cells and tissues: Fast, stable, and selective bioorthogonal imaging via molecular recognition. Anal Chem. 90 (19), 11305-11314 (2018).
- Roos, W. P., Thomas, A. D., Kaina, B. DNA damage and the balance between survival and death in cancer biology. Nat Rev Cancer. 16 (1), 20-33 (2016).
- Huang, R., Zhou, P. K. DNA damage repair: Historical perspectives, mechanistic pathways and clinical translation for targeted cancer therapy. Signal Transduct Target Ther. 6 (1), 254(2021).
- O'Connor, M. J. Targeting the DNA damage response in cancer. Mol Cell. 60 (4), 547-560 (2015).
- Lord, C. J., Ashworth, A. The DNA damage response and cancer therapy. Nature. 481 (7381), 287-294 (2012).
- Carusillo, A., Mussolino, C. DNA Damage: From threat to treatment. Cells. 9 (7), 1665(2020).
- Li, X., Heyer, W. D. Homologous recombination in DNA repair and DNA damage tolerance. Cell Res. 18 (1), 99-113 (2008).
- Chang, H., Pannunzio, N. R., Adachi, N., Lieber, M. R. Non-homologous DNA end joining and alternative pathways to double-strand break repair. Nat Rev Mol Cell Biol. 18 (8), 495-506 (2017).
- Andratschke, N., et al. European society for radiotherapy and oncology and European organization for research and treatment of cancer consensus on re-irradiation: Definition, reporting, and clinical decision making. Lancet Oncol. 23 (10), e469-e478 (2022).
- Olivares-Urbano, M. A., Grinan-Lison, C., Marchal, J. A., Nunez, M. I. CSC radioresistance: A therapeutic challenge to improve radiotherapy effectiveness in cancer. Cells. 9 (7), 1651(2020).
- Schaue, D., McBride, W. H. Opportunities and challenges of radiotherapy for treating cancer. Nat Rev Clin Oncol. 12 (9), 527-540 (2015).
- Allen, C., Her, S., Jaffray, D. A. Radiotherapy for cancer: Present and future. Adv Drug Deliv Rev. 109, 1-2 (2017).
- Xue, C., Greene, E. C. DNA repair pathway choices in CRISPR-Cas9-Mediated genome editing. Trends Genet. 37 (7), 639-656 (2021).
- Tzelepis, K., et al. A CRISPR dropout screen identifies genetic vulnerabilities and therapeutic targets in acute myeloid leukemia. Cell Rep. 17 (4), 1193-1205 (2016).
- Rottner, A. K., et al. A genome-wide CRISPR screen identifies CALCOCO2 as a regulator of beta cell function influencing type 2 diabetes risk. Nat Genet. 55 (1), 54-65 (2023).
- Chen, M., et al. CRISPR-Cas9 for cancer therapy: Opportunities and challenges. Cancer Lett. 447, 48-55 (2019).
- Gupta, D., et al. CRISPR-Cas9 system: A new-fangled dawn in gene editing. Life Sci. 232, 116636(2019).
- Li, B., et al. Genome-wide CRISPR screen identifies host dependency factors for influenza A virus infection. Nat Commun. 11 (1), 164(2020).
- Hung, K. L., et al. Targeted profiling of human extrachromosomal DNA by CRISPR-CATCH. Nat Genet. 54 (11), 1746-1754 (2022).
- Bhattacharya, D., Van Meir, E. G. A simple genotyping method to detect small CRISPR-Cas9 induced indels by agarose gel electrophoresis. Sci Rep. 9 (1), 4437(2019).
- Razavi, Z., Soltani, M., Souri, M., Van Wijnen, A. J. CRISPR innovations in tissue engineering and gene editing. Life Sci. 358, 123120(2024).
- Wang, W., et al. A genome-wide CRISPR-based screen identifies KAT7 as a driver of cellular senescence. Sci Transl Med. 13 (575), eabd2655(2021).
- Xu, X., et al. Engineered miniature CRISPR-Cas system for mammalian genome regulation and editing. Mol Cell. 81 (20), 4333-4345 (2021).
- Huang, M., et al. FACS-based genome-wide CRISPR screens define key regulators of DNA damage signaling pathways. Mol Cell. 83 (15), 2810-2828 (2023).
- Feng, J., et al. Genome-wide CRISPR screen identifies synthetic lethality between DOCK1 inhibition and metformin in liver cancer. Protein Cell. 13 (11), 825-841 (2022).
- Saurat, N., et al. Genome-wide CRISPR screen identifies neddylation as a regulator of neuronal aging and AD neurodegeneration. Cell Stem Cell. 31 (8), 1162-1174 (2024).
- Du, Y., Liu, Y., Hu, J., Peng, X., Liu, Z. CRISPR/Cas9 systems: Delivery technologies and biomedical applications. Asian J Pharm Sci. 18 (6), 100854(2023).
- Shi, J., et al. A genome-wide CRISPR screen identifies WDFY3 as a regulator of macrophage efferocytosis. Nat Commun. 13 (1), 7929(2022).
- Liu, N., Olson, E. N. CRISPR modeling and correction of cardiovascular disease. Circ Res. 130, (2022).
- Feng, X., et al. Genome-wide CRISPR screens using isogenic cells reveal vulnerabilities conferred by loss of tumor suppressors. Sci Adv. 8 (19), 6638(2022).
- Ghaemi, A., et al. CRISPR-Cas9 genome editing delivery systems for targeted cancer therapy. Life Sci. 267, 118969(2021).
- Derry, W. B. CRISPR: Development of a technology and its applications. FEBS J. 288 (2), 358-359 (2021).
- Sharma, G., Sharma, A. R., Bhattacharya, M., Lee, S. S., Chakraborty, C. CRISPR-Cas9: A preclinical and clinical perspective for the treatment of human diseases. Mol Ther. 29 (2), 571-586 (2021).
재인쇄 및 허가
JoVE'article의 텍스트 или 그림을 다시 사용하시려면 허가 살펴보기
허가 살펴보기더 많은 기사 탐색
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. 판권 소유