Method Article
Genomweiter CRISPR-Screen zur Entschlüsselung strahlenempfindlicher und strahlenresistenter Gene
* Diese Autoren haben gleichermaßen beigetragen
In diesem Artikel
Zusammenfassung
Es wird ein akribischer und strukturierter Ansatz verfolgt, um resistente und empfindliche Strahlengene durch die Anwendung einer genomweiten CRISPR/Cas9-Screening-Methode auszuwählen. Dieses Protokoll hat auch das Potenzial, als vielseitiger Rahmen für andere Forschungsvorhaben zu dienen, die die Mechanismen der Resistenz gegen klinisch verabreichte chemische Arzneimittel untersuchen.
Zusammenfassung
Das CRISPR-Cas9-System wurde nutzbar gemacht und zu einem leistungsstarken Genom-Editing-Tool umfunktioniert. Durch den Einsatz dieser Technologie können Forscher DNA-Sequenzen in lebenden Zellen präzise ausschneiden, einfügen und sogar umschreiben. Dennoch geht die Anwendung der CRISPR-Screen-Technologie weit über das reine Experimentieren hinaus. Es dient als zentrales Instrument im Kampf gegen genetische Krankheiten, indem es komplexe genetische Landschaften systematisch seziert, Forscher in die Lage versetzt, die molekularen Mechanismen zu entschlüsseln, die biologischen Phänomenen zugrunde liegen, und Wissenschaftler in die Lage versetzt, die Ursachen von Krankheiten wie Krebs, Mukoviszidose und Sichelzellenanämie zu identifizieren und zu bekämpfen. Vor allem aber stellt Krebs eine gewaltige Herausforderung für die Medizin dar und spornt die Bemühungen um die Ausrottung an. Die Strahlentherapie als traditionelle Behandlung führt zu Ergebnissen, hat aber Grenzen. Es rottet Krebszellen aus, schädigt aber auch gesundes Gewebe und verursacht Nebenwirkungen, die die Lebensqualität beeinträchtigen. Darüber hinaus sprechen nicht alle Krebszellen auf eine Strahlentherapie an, und einige können Resistenzen entwickeln, die den Zustand verschlimmern. Um dies zu adressieren, wird eine umfassende CRISPR-Screen-Technologie für das gesamte Genom eingeführt, die die effiziente Identifizierung von strahlenempfindlichen und strahlenresistenten Genen ermöglicht und damit das Feld der Krebsforschung und -behandlung voranbringt. Ein genomweites CRISPR-Screening wurde in Lungenadenokarzinomzellen durchgeführt, die nach dem beschriebenen Protokoll bestrahlt wurden, wobei sowohl Strahlenresistenz- als auch Strahlenempfindlichkeits-assoziierte Gene identifiziert wurden.
Einleitung
Die Erforschung biologischer Phänomene ist untrennbar mit der Erforschung zellulärer Verhaltensweisen verflochten, und die Untersuchung zellulärer Verhaltensweisen ist wiederum grundlegend mit der Erforschung ihres Genoms verbunden. Mit der Weiterentwicklung der modernen Technologie richten medizinische Forscher ihre Aufmerksamkeit zunehmend auf die Veränderung zellulärer Verhaltensweisen durch Gen-Editierung aus, um die Behandlungsergebnisse verschiedener Krankheiten zu verbessern. In dieser Hinsicht hat sich die CRISPR-Technologie (Clustered Regularly Interspaced Short Palindromic Repeats) aufgrund ihrer relativ einfachen Anwendung als revolutionäres Werkzeug für die Genom-Editierung erwiesen1. Das CRISPR-Cas9-System besteht aus Cas9-Nuklease und Single-Guide-RNA (sgRNA), die spezifisch die Ziel-DNA-Sequenz erkennt und an sie bindet, wodurch die Cas9-Nuklease an dieser Stelle geschnitten wird, was zu einem Doppelstrangbruch (DSB) in der Genom-DNAführt 2,3,4. Darüber hinaus kann die Einführung anderer Substanzen zu spezifischen Insertionen, Deletionen oder Mutationen im Genom führen, die eine gezielte Gen-Editierung ermöglichen.
In der funktionellen Genomforschung war das RNA-Interferenz-Screening (RNAi) einst eine weit verbreitete Methode zur Durchführung groß angelegter Funktionsverlust-Experimente, um die Rolle von Genen bei Krebs zu untersuchen. Die RNAi-Technologie untersucht die Funktion von Genen, indem sie Zielgene spezifisch stummschaltet und den Forschern hilft, kritische onkogene Faktoren zu identifizieren. Sie ist jedoch durch Off-Target-Effekte und eine unvollständige Gen-Knockdown-Effizienz begrenzt. Off-Target-Effekte können zur Stilllegung anderer Nicht-Zielgene führen, wodurch die Genauigkeit und Zuverlässigkeit der Versuchsergebnisse beeinträchtigtwird 1,2. Darüber hinaus weist RNAi für bestimmte Gene eine geringe Knockdown-Effizienz auf, so dass die Expression des Zielgens möglicherweise nicht vollständig unterdrückt werden kann. Im Gegensatz zu herkömmlichen RNAi-Screenings weist der CRISPR-Screen eine höhere Spezifität und Effizienzauf 3. Diese Technologie ermöglicht nicht nur die präzise Editierung spezifischer Gene, sondern auch ein genomweites groß angelegtes Screening, was die Erforschung der Genfunktion robust unterstützt. Die CRISPR-Screen-Technologie, ein leistungsfähiges Gen-Editing-Tool, das auf dem CRISPR-Cas9-System basiert, wird für das effiziente Screening und die Aufdeckung unbekannter Funktionen bestimmter Gene in Zellen eingesetzt 5,6,7,8. Die Forscher entwerfen sgRNAs in Chargen für bestimmte Gene oder Genregionen und bereiten entsprechende sgRNA-Bibliotheken mit Präzision und Genauigkeit vor, um ihre Integrität und Funktionalität sicherzustellen9. Diese sgRNA-Bibliotheken werden dann in lentivirale Partikel eingekapselt, die zur effizienten Infektion von Wirtszellen verwendet werden. Nach erfolgreicher Infektion werden die infizierten Zellen unter persönlich definierten Screening-Bedingungen kultiviert. Beim Screening wird die genomische DNA der gescreenten Zellen extrahiert, wobei hohe Reinheits- und Mengenstandards eingehalten werden. Anschließend werden Zielregionen, die für sgRNA von Interesse sind, einer PCR-Amplifikation unterzogen, einem Prozess, der die gewünschten Segmente von Nukleinsäuren genau repliziert 3,9. Schließlich wird eine Hochdurchsatz-Sequenzierung an den amplifizierten DNA-Fragmenten durchgeführt, die eine umfassende und effiziente Analyse der Zielregionen ermöglicht und somit wertvolle Einblicke in die Funktion und das Verhalten der zu untersuchenden Gene liefert4.
Krebs stellt als komplexe Krankheit eine große Bedrohung für die menschliche Gesundheit dar. Weltweit arbeiten Forscher und Kliniker gemeinsam daran, die molekularen Mechanismen der Krebsentstehung zu entschlüsseln und neue therapeutische Strategien zu entwickeln. Es wurden internationale Kooperationen aufgebaut, um die Translation von Erkenntnissen aus der Grundlagenforschung in die klinische Anwendung zu beschleunigen, mit dem Ziel, die Ergebnisse für die Patienten zu verbessern. Sasmal et al. schlugen eine bioorthogonale Assemblierungsstrategie vor, die auf einem synthetischen Wirt-Gast-System für die präzise Ausrichtung auf metastasierende Krebszellen basiert und Dutzenden von Wissenschaftlern bei der Weiterentwicklung medizinischer Technologien erheblich geholfen hat. Ihre herausragende Forschungsarbeit zeichnet sich durch hohe Innovationskraft und einzigartige Erkenntnisse aus und leistet einen bedeutenden Beitrag zur wissenschaftlichen Gemeinschaft10. Krebs ist gekennzeichnet durch den turbulenten Zustand genomischer Instabilität, der sich aus der unregelmäßigen Regulation der DNA-Schadensantworten ergibt 11-14. Zu den DNA-Schäden gehören Einzelnukleotiddefekte, Einzelstrangbrüche und DSBs. Homologe Rekombination (HR) und nicht-homologe Endverbindung (NHEJ) sind an der Reparatur von DSBs in verschiedenen Stadien beteiligt15,16,17. Auf dieser Grundlage hat sich die Strahlentherapie als praktikable Behandlungsoption herausgestellt, bei der hochenergetische Strahlen (wie Röntgenstrahlen und γ-Strahlen) zur Bestrahlung des Tumorgewebes verwendet werden, wodurch DNA-Schäden in Tumorzellen verursacht werden, wodurch deren Wachstum und Proliferation gestörtwerden 18. Die Strahlentherapie führt jedoch bei einem signifikanten Teil der Krebspatienten nicht immer zu den gewünschten Effekten, was möglicherweise auf Schädigungen des parakanzerösen Gewebes und Einschränkungen zurückzuführen ist, die durch die inhärenten Eigenschaften des Tumors auferlegt werden, wie z. B. eine geringe Empfindlichkeit gegenüber der Strahlentherapie 19,20,21.
Theoretisch kann jeder Zelltyp für ein CRISPR-Screening verwendet werden. Um jedoch eine ausreichende Repräsentation in mutierten Populationen aufrechtzuerhalten, ist eine große Anzahl von Startzellen erforderlich. Zelltypen mit geringer Abundanz sind nicht besonders geeignet für ein genomweites Screening. Was die Wahl der Bibliothek betrifft, so enthalten die meisten Bibliotheken 3-6 gRNAs pro Zielgen, und die Aufrechterhaltung der Verteilung jeder gRNA innerhalb der Population ist entscheidend18. Ein Verlust der Repräsentation aufgrund von Anreicherung oder Depletion spezifischer gRNAs kann zu einer ungleichmäßigen Ergebnisverteilung führen. Um dieses Problem anzugehen, kann die Entscheidung für kommerziell erhältliche CRISPR-Bibliotheken, die auf dem Markt getestet wurden, eine bevorzugte Wahlsein 20. In vitro Das CRISPR-Screening mit homogenen Krebszelllinien kann die genetische und epigenetische Heterogenität von in vivo-Tumoren möglicherweise nicht vollständig erfassen. Während das In-vitro-Screening Schlüsselgene aufdeckte, die an der Reparatur von DNA-Schäden und der strahleninduzierten autokrinen Signalgebung beteiligt sind, replizierte es die Mikroumgebung des Tumors nicht vollständig, einschließlich Hypoxie-induzierter Strahlenresistenz (über ROS, metabolische Anpassung und Autophagie), immunvermittelte parakrine Effekte und EZM-abhängige Zytokinmodulation. Bevor der CRISPR-Screen zur Erforschung von Genen eingesetzt wird, die mit Strahlenempfindlichkeit oder -resistenz verbunden sind, müssen diese Faktoren sorgfältig abgewogen werden. Angesichts der aktuellen Behandlungslandschaft ist es dringend erforderlich, Faktoren im Zusammenhang mit Strahlenresistenz und Strahlenempfindlichkeit zu identifizieren und eingehend zu untersuchen, um die Wirksamkeit der Strahlentherapie wirksam zu verbessern22. Angesichts des entscheidenden Vorteils des CRISPR-Screenings bei der Untersuchung der Funktionen unbekannter Gene wird eine systematisch detaillierte CRISPR-Screen-Technologie für das gesamte Genom bereitgestellt, um strahlenempfindliche und strahlenresistente Gene effizient zu identifizieren.
Protokoll
Die in dieser Studie verwendeten Reagenzien und Geräte sind in der Materialtabelle aufgeführt.
1. Auswahl einer geeigneten Strahlendosis
- Präparation und Plattierung von adhärenten Zellen
- Stellen Sie die Zelldichte auf 5 x 105 Zellen pro ml mit RPMI 1640 vollständigem Zellkulturmedium mit 10 % FBS für die Passage und das Zellwachstum ein. Verteilen Sie die Zellen auf 3,5 cm große Kulturschalen für die Bestrahlung in unterschiedlichen Dosen. 2 ml (1 x 106 Zellen) in jede Schale geben und über Nacht bei 37 °C mit 5 % CO2 inkubieren.
- Anwendung unterschiedlicher Strahlendosen
- Nummerieren Sie die 3,5 cm großen Kulturschalen von 1 bis 5. Verwenden Sie Gruppe #1 als Kontrollgruppe, wobei die restlichen 4 Gruppen als Behandlungsgruppen bezeichnet werden.
- Versiegeln Sie die Ränder der 6-Well-Platten mit einer Dichtungsmembran für Behandlungsgruppen und verabreichen Sie Strahlendosen von 2, 4, 6 bzw. 8 Gy. Stellen Sie die Schalen nach der Behandlung wieder in den Inkubator.
- Plattierung von Zelllinien nach der Bestrahlung
- Bereiten Sie 6-Well- und 96-Well-Platten vor. Waschen Sie die Zellen in den 3,5-cm-Kulturschalen einmal mit PBS, verdauen Sie die logarithmisch wachsenden Zellen mit 0,25 % Trypsin und stellen Sie die Zelldichte auf 1 x 105 Zellen pro ml mit RPMI 1640 Komplettmedium mit 10 % FBS ein.
- 10 μl/Well (1.000 Zellen/100 μl) mit 3 Replikaten pro Strahlendosis in die 6-Well-Platten einfüllen. Geben Sie 2 ml vollständiges Medium in jede Vertiefung und zählen Sie nach 14 Tagen (wenn jede Klongruppe etwa 50 Zellen umfasst).
- 30 μl/Well (3.000 Zellen/100 μl) mit 5 Replikaten pro Strahlendosis in die 96-Well-Platten einfüllen. Geben Sie 70 μl vollständiges Medium in jede Vertiefung und messen Sie die Zellviabilität nach 72 Stunden.
- Messung der Zellviabilität
HINWEIS: Klonogene Hemmrate = 1 - (Anzahl der Klone in der Behandlungsgruppe/Anzahl der Klone in der Kontrollgruppe) x 100%. Der CCK-8-Assay verwendet ein wasserlösliches Tetrazoliumsalz (WST-8), das durch zelluläre Dehydrogenasen reduziert werden kann, um ein hochgradig wasserlösliches gelbes Formazanproduktzu erzeugen 11. Die Menge an produziertem Formazan ist direkt proportional zur Anzahl lebensfähiger Zellen, und seine optische Dichte (gemessen bei einer Wellenlänge von 450 nm) spiegelt die zelluläre Stoffwechselaktivität und den proliferativen Status genau wider11,13. Basierend auf diesem etablierten Prinzip wurde der CCK-8-Assay für verschiedene Anwendungen eingesetzt, darunter Zellproliferationsassays und Sensitivitätstests für Tumormedikamente. In diesem Protokoll wird der CCK-8-Assay eingesetzt, um die zelluläre Lebensfähigkeit unter verschiedenen Strahlendosen systematisch zu bewerten.- Mischen Sie das CCK8-Reagenz mit dem RPMI 1640 Medium ohne FBS im Verhältnis 1:9. Verwerfen Sie das Medium in den 96-Well-Platten und geben Sie 100 μl des CCK8-haltigen Mediums in jede Vertiefung.
- Inkubieren Sie 1 h im Dunkeln und messen Sie den OD-Wert bei 450 nm mit einem Mikroplatten-Reader.
HINWEIS: Zellüberlebensrate = [(OD-Wert der Behandlungsgruppe - OD-Wert des Blind-Wells) / (OD-Wert der Kontrollgruppe - OD-Wert des Blank-Wells)] x 100%
- Auswahl der Strahlendosis
- Integrieren Sie die klonogene Hemmrate in die Überlebensrate der Zellen, um eine geeignete Strahlendosis basierend auf den Forschungsbedürfnissen auszuwählen. Wählen Sie eine Strahlendosis mit einer Hemmrate von 50 % für das Screening sowohl strahlenresistenter als auch strahlenempfindlicher Gene.
2. Auswahl des geeigneten MOI und der Puromycin-Konzentration
- Plattierung von adhärenten Zellen
- Stellen Sie die Zelldichte auf 3 x 105 Zellen pro ml ein und inokulieren Sie 1 ml (3 x 105 Zellen) pro Vertiefung in eine 12-Well-Platte. Die Platte über Nacht bei 37 °C mit 5 % CO2 inkubieren.
- Lentivirale Infektion
- Verwenden Sie eine Pipette, um das Medium aus der 12-Well-Platte zu aspirieren, und ersetzen Sie es durch 1 ml RPMI 1640 vollständiges Medium mit 10 % FBS. Bereiten Sie eine qualitätsgeprüfte CRISPR-Lentivirusbibliothek für das gesamte Genom vor (kommerziell erhältliche Bibliotheken werden empfohlen) und richten Sie einen logarithmischen Konzentrationsgradienten ein (z. B. 0-10-50-100-200-400-800)4,5,6,7,8.
- Die entsprechende Menge Lentivirus auf 2 μl/Schale Polybren geben und 5 Minuten lang bei Raumtemperatur äquilibrieren. Die Mischung aus Lentivirus und Polybren langsam in jede Vertiefung tropfen, gut mischen und über Nacht bei 37 °C mit 5 % CO2 inkubieren.
- Bestimmung der minimalen Puromycinkonzentration für die Zellabtötung
- Stellen Sie die Dichte der elterlichen Zellen auf 3 x 105 Zellen/ml ein und inokulieren Sie 1 ml (3 x 105 Zellen) pro Well in eine 12-Well-Platte. Die Platte über Nacht bei 37 °C mit 5 % CO2 inkubieren.
- Geben Sie Puromycin in jede Vertiefung der 12-Well-Platte in einem Konzentrationsgradienten von 0-0,1-0,2-0,5-1-2-4-8 μM. Zählen Sie die Zellen nach 72 h. Die minimale Konzentration, die alle Zellen in der Vertiefung abtötet, ist die minimale Puromycin-Konzentration für die Zellabtötung, die für das anschließende Screening von viral infizierten Zellen verwendet wird.
- Puromycin-Selektion nach der Infektion
- Am zweiten Tag nach der Infektion aspirieren Sie das Medium aus jeder Vertiefung und ersetzen Sie es durch 1 ml RPMI 1640 vollständiges Medium mit 10 % FBS. Kultivieren Sie 48 Stunden lang.
- Nach 72 Stunden der Infektion ist das vorhandene Medium durch ein vollständiges Medium zu ersetzen, das die Mindestkonzentration von Puromycin für die Zellabtötung enthält. Richten Sie 2 Wells in der 12-Well-Platte ohne lentivirale Infektion als Negativ- und Positivkontrollen ein. Behandeln Sie die Negativkontrolle mit Puromycin und lassen Sie die Positivkontrolle unbehandelt. Kultivieren Sie 72 Stunden lang.
- Nach 72 Stunden Puromycin-Selektion sind alle Elternzellen in der Negativkontrolle tot, während die Elternzellen in der Positivkontrolle nur minimalen Tod zeigen. Berechnen Sie das Trägheitsmoment für jede Vertiefung.
HINWEIS: MOI = (Anzahl der Zellen in der virusinfizierten Gruppe / Anzahl der Zellen in der positiven Kontrollgruppe) x 100 %. Verwenden Sie die Viruskonzentration mit einem MOI von ~0,3 für das anschließende Screening.
3. Genomweite Infektion der lentiviralen CRISPR-Bibliothek
- Aussaat von adhärenten Zelllinien
- Stellen Sie die Zelldichte auf 1 x 107 Zellen pro ml in RPMI 1640 Komplettmedium mit 10 % FBS für die Passage und das Zellwachstum ein. 1 ml Zellen (1 x 107 Zellen) in jede 15-cm-Kulturschale bei 37 °C 8 h lang mit 5 % CO2 inokulieren. Sobald die Zellen anhafteten und eine Konfluenz von 70%-80% erreichten, waren sie bereit für eine Virusinfektion (was eine Kopienzahl von etwa 500 ergibt).
- Lentivirale Infektion
- Aspirieren Sie das Medium aus den 15-cm-Kulturschalen und ersetzen Sie es durch 15 ml RPMI 1640 vollständiges Medium mit 10 % FBS. Bereiten Sie eine qualitätsgeprüfte CRISPR-Lentivirusbibliothek des gesamten Genoms vor.
- Die entsprechende Menge Lentivirus mit dem MOI = 0,3 bis 30 μl/Schale Polybren zugeben und 5 min bei Raumtemperatur äquilibrieren, die Mischung aus Lentivirus und Polybren langsam in die 15-cm-Kulturschalen tropfen, gut mischen und bei 37 °C mit 5 % CO2 über Nacht inkubieren. Bereiten Sie gleichzeitig eine 15 cm große Kulturschale mit der Elternzelle als Kontrast vor.
- Puromycin-Selektion nach der Infektion
- Am zweiten Tag nach der Virusinfektion ist das Medium aus den 15-cm-Kulturschalen zu aspirieren und durch 15 ml RPMI 1640 vollständiges Medium mit 10 % FBS zu ersetzen. Kultivieren Sie 48 Stunden lang.
- 72 h nach der Infektion ist das Medium durch ein vollständiges Medium zu ersetzen, das die minimale letale Konzentration von Puromycin enthält. Behandeln Sie die nicht infizierten Elternzellen auf die gleiche Weise als Negativkontrolle und kultivieren Sie 72 Stunden lang. Nach 72 Stunden Puromycin-Selektion werden alle Elternzellen in der negativen Kontrollgruppe abgetötet, und die überlebenden Zellen der lentiviralen Infektion gelten als erfolgreich infiziert.
- Extraktion des Genoms von Tag 0
- Die Zellen aus einer 15-cm-Kulturschale werden mit 0,25 % Trypsin verdaut, in RPMI 1640 Komplettmedium mit 10 % FBS resuspendiert und die Zellzahl gezählt. Beschriften Sie dieses Beispiel als Tag 0.
- Bei 300 x g für 5 min (bei Raumtemperatur) zentrifugieren und den Überstand verwerfen. In 1 ml PBS resuspendieren, 5 min bei 300 x g zentrifugieren und den Überstand verwerfen. Extrahieren Sie die genomische DNA von Tag 0, messen Sie die DNA-Konzentration und -Reinheit mit einem Nanotropfen-UV-Spektralphotometer.
- Für den Nachweis der sgRNA-Integrität wird die Agarose-Gelelektrophorese verwendet, um die amplifizierte sgRNA-Bibliothek zu analysieren, wobei sichergestellt wird, dass die Banden klar und ohne signifikante Degradation sind, wodurch die Integrität der Bibliothekerhalten bleibt 23. Für die Bewertung der Abdeckung lentiviraler Bibliotheken ist die Deep-Sequencing-Technologie zu verwenden, um eine Sequenzierungsanalyse an den sgRNAs innerhalb der Bibliothekdurchzuführen 24,25.
- Im CRISPR-Screen dient die PCR-Amplifikation als vorläufige Überprüfung der Integrität des sgRNA-Fragments und der Bibliotheksqualität6. Um die Bibliotheksabdeckung und die sgRNA-Verteilungsmuster umfassender zu analysieren und eine angemessene Repräsentation von sgRNAs für jedes Zielgen während des Screenings sicherzustellen, führen Sie NGS durch, um detaillierte Einblicke in die Robustheit von Screen9 zu erhalten.
HINWEIS: Durch die Erkennung von Veränderungen in der sgRNA-Abundanz vor und nach dem Screening und die Identifizierung einer potenziellen sgRNA-Off-Target-Integration oder Bibliothekskontamination könnte die Genauigkeit und Zuverlässigkeit des CRISPR-Screenings weiter verbessert werden. Diese Tag-0-Proben dienen als wichtige Negativkontrollen und werden einer umfassenden Qualitätsbewertung durch PCR-Amplifikation und NGS unterzogen, um 2 Hauptziele zu erreichen: (1) Bestätigung der Depletion essentieller Gene (was auf eine angemessene Bibliotheksrepräsentation hinweist) und (2) Nachweis einer stabilen Expression in nicht-essentiellen Genen (Etablierung experimenteller Ausgangsbedingungen)9. - Stellen Sie sicher, dass die Abdeckung das erwartete Niveau erreicht, um die Vielfalt und Repräsentativität der Bibliothek zu gewährleisten und Verzerrungen im Screening-Prozess zu vermeiden. Verwenden Sie für die Messung der Infektionseffizienz die Fluoreszenzbildgebung, um die Infektionseffizienz zu beurteilen und sicherzustellen, dass sie den Anforderungen des Experiments entspricht.
4. Anwendung von Strahlung als Screening-Bedingung
- Gruppierung
- Stellen Sie die Dichte der CRISPR-infizierten Zellen auf 1 x 107 Zellen pro ml ein und inokulieren Sie 1 ml in jede 15-cm-Petrischale. Über Nacht bei 37 °C mit 5 % CO2 inkubieren.
- Strahlenbehandlung
- Teilen Sie die Zellen nach dem Zufallsprinzip in 2 Gruppen ein: eine Behandlungsgruppe und eine Kontrollgruppe mit 6 Gerichten pro Gruppe. Verabreichen Sie den Zellen in der Behandlungsgruppe eine angemessene Strahlendosis, während die Zellen der Kontrollgruppe unbehandelt bleiben, um sich normal zu vermehren. Nach der Bestrahlung bei 37 °C mit 5 % CO2 für 7 Tage weiter inkubieren.
- In der zweiten Woche wiederholen Sie die Verabreichung einer angemessenen Strahlendosis an die Zellen der Behandlungsgruppe, wobei die Zellen der Kontrollgruppe unbehandelt bleiben und sich normal vermehren können. Nach der Strahlentherapie die Inkubation bei 37 °C mit 5 % CO2 für weitere 7 Tage fortsetzen.
5. Genomextraktion und -sequenzierung
- Extraktion von Tag 14
- Nach 14-tägiger Behandlung verdauen Sie die Zellen in der Behandlungs- und Kontrollgruppe mit 0,25 % Trypsin, resuspendieren Sie sie mit vollständigem RPMI 1640-Medium mit 10 % FBS und markieren Sie sie als Tag 14-RT oder Tag 14-NC.
- Die Zellen werden bei 300 x g für 5 min zentrifugiert und der Überstand verworfen. In 1 mL PBS resuspendieren, 5 min bei 300 x g zentrifugieren und den Überstand wieder verwerfen. Extrahieren Sie die genomische DNA von Tag 14 und bestimmen Sie die Konzentration und Reinheit der DNA mithilfe der Nanotropfen-UV-Absorptionsspektroskopie.
- PCR-Amplifikation
- Bereiten Sie die entsprechenden Primer der Sequenz vor (siehe Materialtabelle) und verdünnen Sie sie auf 10 μM. Richten Sie ein 20 μL-Reaktionssystem ein, indem Sie die Reagenzien in ein steriles Mikrozentrifugenröhrchen geben, zentrifugieren Sie 5 s lang bei 300 x g und mischen Sie es gut. Stellen Sie die Parameter des PCR-Geräts entsprechend der Amplifikationsbedingung ein, um PCR-Produkte zu erhalten.
- Nachweis der Agarose-Gelelektrophorese
- Bereiten Sie eine Gelgussplatte vor, versiegeln Sie die Ränder der Form mit Agarose, führen Sie den Kamm ein und bereiten Sie ein Agarosegel in geeigneter Konzentration entsprechend der Länge der Proben-DNA vor.
- Wiegen Sie eine bestimmte Menge Agarosepulver genau ab, fügen Sie eine angemessene Menge Elektrophoresepuffer hinzu, mischen Sie es gut und erhitzen Sie es in der Mikrowelle, um es zu schmelzen. Nach dem Abkühlen eine angemessene Menge Nukleinsäurefarbstoff hinzufügen, vorsichtig mischen und langsam in die Gelgussform gießen. Lassen Sie das Gel 30 Minuten lang fest werden.
- Entfernen Sie den Kamm und geben Sie eine angemessene Menge Elektrophoresepuffer in den Elektrophoresetank, bis er das Gel bedeckt. Geben Sie eine angemessene Menge Ladepuffer in die DNA-Probe, mischen Sie sie gut und geben Sie die Mischung mit einer Pipette langsam in die Probenvertiefung.
- Stellen Sie die geeignete Spannung basierend auf der Größe des DNA-Fragments und der Konzentration des Agarosegels ein. Entfernen Sie das Gel nach der Elektrophorese vorsichtig und legen Sie es in einen Gel-Imager, um die Ergebnisse zu beobachten und zu überprüfen, ob die PCR-Amplifikation erfolgreich war.
- Illumina-Sequenzierung
- Sammeln Sie die genomische DNA aus den drei Gruppen (Tag-0-Gruppe, Kontrollgruppe und Behandlungsgruppe) und senden Sie sie an ein Unternehmen für den Bibliotheksaufbau und die Illumina-Sequenzierung. Durchführung einer bioinformatischen Analyse und Visualisierung der Sequenzierungsergebnisse, um strahlentherapieempfindliche und strahlentherapieresistente Gene zu erhalten26.
- Richten Sie mehrere wiederholte Experimente für die Datenanalyse ein und führen Sie statistische Analysen der Versuchsergebnisse durch, um die Konsistenz und Reproduzierbarkeit der Daten sicherzustellen27,28.
- Qualitätsbewertung von Sequenzierungsdaten
- Unterziehen Sie die Rohdaten strengen Filter- und Qualitätskontrollmechanismen, um die Präzision der Sequenzierungsinformationen zu gewährleisten5. Berechnen Sie den Phred-Score (Qphred) für jedes Nukleotid basierend auf der Sequenzierungsfehlerrate unter Verwendung eines angegebenen Konvertierungsalgorithmus und eines Modells, das die Wahrscheinlichkeit eines Fehlers beim Aufrufen der Basebewertet 28.
- Halten Sie die Sequenzierungsfehlerrate für jede Basisposition unter 1 % (entspricht einem Q30-Schwellenwert) und stellen Sie sicher, dass mindestens 80 % der Sequenzierungsdaten diesen Q30-Standard erreichen, um nachfolgende Analyseverfahren zu unterstützen.
- Datenverarbeitung und Qualitätskontrolle auf Probenebene
HINWEIS: Für Details verweisen wir auf die zuvor veröffentlichten Berichte 29,30.- Richten Sie die gefilterten Reads aus Sequenzierungsdaten mit den Sequenzen der sgRNA-Bibliothek ab. Melden Sie Statistiken, einschließlich der Anzahl der sgRNAs in der Bibliothek mit perfekter Ausrichtung, der durchschnittlichen Häufigkeit von sgRNAs, der Anzahl der nicht nachgewiesenen sgRNAs und des Anteils der Lesevorgänge aus einzelnen Proben, die erfolgreich der sgRNA-Bibliothek zugeordnet wurden. Verwenden Sie ein höheres Zuordnungsverhältnis, um eine größere Abdeckung anzuzeigen.
- Zählen Sie die Anreicherung von Reads für jedes Gen (auf das verschiedene sgRNAs abzielen) in jeder Probe. Normalisieren Sie die unterstützenden Reads für jede sgRNA über Proben hinweg mit der "medianen" Normalisierungsmethode von MAGeCK. Führen Sie eine Qualitätskontrolle auf Stichprobenebene durch, indem Sie die Verteilung der sgRNA-Lesezahlen auswerten, Boxplots erstellen, Hauptkomponentenanalysen (PCA) durchführen und Korrelations-Heatmaps erstellen.
- Nehmen wir an, dass die Anzahl der sgRNA-Lesevorgänge einer Poisson-Verteilung folgt. Stellen Sie normalisierte sgRNA-Zahlen aus verschiedenen Gruppen in Boxplots dar, um die Gesamtdatenverteilung innerhalb der Proben zu visualisieren und die Verteilungen zwischen den Gruppen zu vergleichen.
- Verwenden Sie PCA, um die Datenanalyse zu vereinfachen, indem Sie Unterschiede zwischen den Hauptkomponenten und die Variationsrate innerhalb jeder Komponente widerspiegeln und so die Beobachtung von Variationen innerhalb und zwischen Gruppen erleichtern. Verwenden Sie eine Korrelations-Heatmap, um die Beziehungen zwischen den Stichproben zu veranschaulichen.
- Grundlegende Analyse
- Führen Sie differentielle sgRNA-Analysen zwischen Gruppen durch und identifizieren Sie essentielle Gene nach Qualitätskontrolle und vorläufiger Datenverarbeitung. Durchführung einer funktionellen Anreicherungsanalyse der relevanten Gene30. Ordnen Sie essentielle Gene basierend auf dem RRA-Wert (Robust Rank Aggregation) ein, der von MAGeCK oder anderen statistischen Methoden wie MLE berechnet wird, wobei ein niedrigerer RRA-Wert auf eine höhere Wichtigkeit hinweist.
- Verwenden Sie die Sprache R oder andere Programmiersprachen, um eine bioinformatische Analyse der eingestuften Ergebnisse durchzuführen, und visualisieren Sie die Ergebnisse mithilfe von GO- und KEGG-Analysediagrammen30. Verknüpfen Sie Gene oder Proteine mit entsprechenden GO-Begriffen (molekulare Funktion, biologischer Prozess und zelluläre Komponente) durch ID-Mapping oder Sequenzannotation.
- Verwenden Sie KEGG als Datenbank, um High-Level-Funktionen und biologische Systeme zu verstehen und den identifizierten Genkatalog mit Systemfunktionen auf Zell-, Spezies- und Ökosystemebene zu verbinden. Wählen Sie die 10 oder 20 wichtigsten Pfade aus GO- und KEGG-Analysen aus, um eine intuitive Anzeige der Pfadrichtungen zu erhalten.
Ergebnisse
Lungenkrebs mit der führenden Sterblichkeitsrate stellt eine sehr aggressive und weit verbreitete medizinische Erkrankung dar. Am Beispiel der Lungenkrebszelllinie A549 für die Durchführung eines genomweiten CRISPR-Screenings mit Strahlung als Screening-Bedingung ist der schematische Arbeitsablauf in Abbildung 1 dargestellt. Zunächst soll die Empfindlichkeit von A549-Zellen gegenüber unterschiedlichen Strahlendosen durch Klonbildung und CCK8-Experimente untersucht werden (Abbildung 2). Im klonogenen Assay betrug die Koloniezahl 140 ± 5,35 bei 2 Gy gegenüber 303 ± 31,63 bei 0 Gy, während im CCK-8-Assay der Wert der optischen Dichte (OD) 0,65 ± 0,05 bei 2 Gy gegenüber 1,35 ± 0,08 bei 0 Gy betrug. Um Kandidatengene für Strahlenempfindlichkeit und Strahlenresistenz zu untersuchen, wählen Sie 2 Gy (IC50) als nachfolgende Strahlendosis. A549-Zellen wurden mit unterschiedlichen Dosen von Puromycin behandelt und nach 72 h gezählt. Die minimale Abtötungskonzentration von Puromycin für A549-Zellen wurde mit 1 μM nachgewiesen (Abbildung 3). A549-Zellen wurden mit Lentiviren an verschiedenen Gradienten-MOIs infiziert. Nach 72 Stunden der Infektion wurde die Wirksamkeit der Infektion mit dem Fluoreszenzmikroskop beobachtet und vor und nach 72 Stunden der Puromycin-Behandlung gezählt. Es wurde der MOI mit einer Infektionseffizienz von ~0,3 ermittelt (Tabelle 1). Das menschliche Genom enthält 19.050 Gene, wobei jedem Gen 6 sgRNAs entsprechen und eine Kopienzahl von 500 (19.050 x 6 x 500 = 5,7 x 107 Zellen) vorliegt. Unter Verwendung von Lentiviren mit MOI = 0,3 und dementsprechend infizieren Sie 5,7 x 107 A549-Zellen. Nach 72 h wurden die Zellen für weitere 72 h mit 1 μM Puromycin behandelt. Das Genom von Tag 0 wurde entnommen und eine PCR-Amplifikation durchgeführt. Die sgRNA-Sequenzen innerhalb der CRISPR-Gesamtgenombibliothek weisen eine Länge von 231 Basenpaaren auf, was durch die Ergebnisse der Agarose-Gelelektrophorese bestätigt wird (Abbildung 4).
Die PCR-Produkte wurden anschließend zur Datenqualitätskontrolle und Qualitätskontrolle auf Probenebene an Novogene geschickt. Das Kartierungsverhältnis ergab eine umfassende Abdeckungsrate von etwa 60 %, eine Metrik, die angesichts der schieren Komplexität und des unvermeidlichen Verschleißes während der Verarbeitung eines gesamten Genoms als angemessen für Gesamtgenom-Screening-Verfahren angesehen wird (Abbildung 5). Die sgRNA-Lesezahlen folgten einer Poisson-Verteilung, die den theoretischen Erwartungen entsprach. Die anschließende Analyse durch PCA-Plotting und Korrelations-Heatmapping stellte die erkennbare Ungleichheit zwischen den verschiedenen Gruppen anschaulich dar, wobei die Variation zwischen den Gruppen die Diskrepanzen innerhalb der Gruppe deutlich überwog. Darüber hinaus lag die Variationsrate innerhalb der gruppierten Stichproben innerhalb akzeptabler Grenzen, was den Erfolg der Qualitätskontrollmaßnahmen auf Stichprobenebene untermauert. Anschließend werden die von MAGeCK entwickelten RRA-Rankings genutzt, um eine grundlegende bioinformatische Bewertung der Ranking-Ergebnisse unter Verwendung der R-Sprache durchzuführen. Bemerkenswert ist, dass die Top 15 Signalwege in Bezug auf GO Mechanismen aufwiesen, die mit DNA-Schäden verbunden sind, was nahtlos mit den zugrunde liegenden Screening-Kriterien des Experiments übereinstimmt.
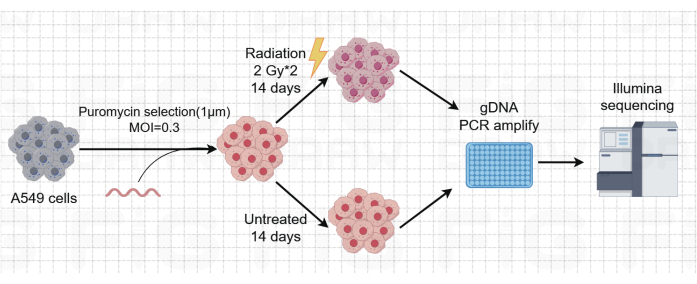
Abbildung 1: Schematische Darstellung des genomweiten CRISPR/Cas9-Screenings der Strahlentherapie in A549-Lungenkrebszellen. Eine schematische Darstellung des Screening-Workflows in A549-Zellen unter Verwendung von genomweiter CRISPR/Cas9 und Strahlentherapie. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
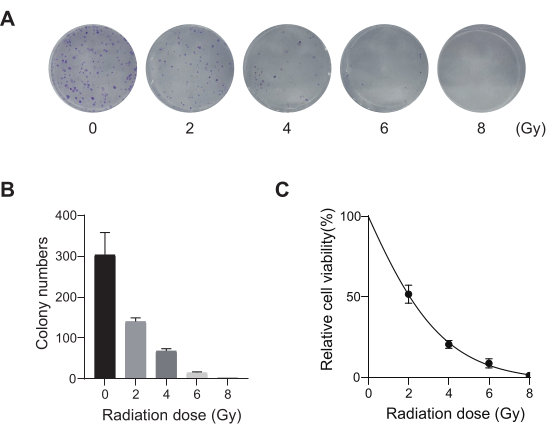
Abbildung 2: Optimierung der Strahlendosis für A549-Zellen. (A) A549-Zellen wurden steigenden Strahlendosen (0 Gy, 2 Gy, 4 Gy, 6 Gy und 8 Gy) ausgesetzt und 14 Tage lang wachsen gelassen. Es werden Assay-Bilder der Koloniebildung gezeigt. Die Ergebnisse sind repräsentativ für zwei unabhängige biologische Experimente. (B) Quantifizierungsergebnisse von geklonten Formations-Assay-Bildern. Fehlerbalken stellen die Standardabweichung (n = 3) dar. Die ANOVA in eine Richtung ergab P < 0,01. Die Ergebnisse sind repräsentativ für zwei unabhängige biologische Experimente. (C) Dosis-Wirkungs-Überlebenskurven von A549-Zellen nach Exposition gegenüber unterschiedlichen Strahlendosen. Fehlerbalken stellen die Standardabweichung (n = 3) dar. Die ANOVA in eine Richtung ergab P < 0,01. Die Ergebnisse sind repräsentativ für zwei unabhängige biologische Experimente. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
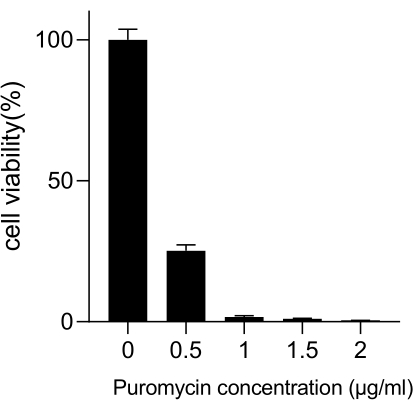
Abbildung 3: Bestimmung der minimalen Puromycin-Konzentration, die für die A549-Zellselektion erforderlich ist. A549-Zellen wurden mit steigenden Konzentrationen von Puromycin behandelt und 72 Stunden lang inkubiert. Fehlerbalken stellen die Standardabweichung (n = 3) dar. Die einfache ANOVA ergab einen P < 0,01. Die Ergebnisse sind repräsentativ für zwei unabhängige biologische Experimente. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
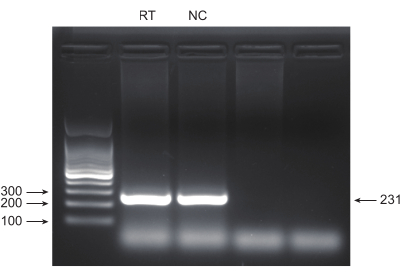
Abbildung 4: PCR-Produktvalidierung durch Agarose-Gelelektrophorese. Die Agarose-Gelelektrophorese wurde verwendet, um das Vorhandensein und die Qualität von PCR-Produktbanden zu untersuchen. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
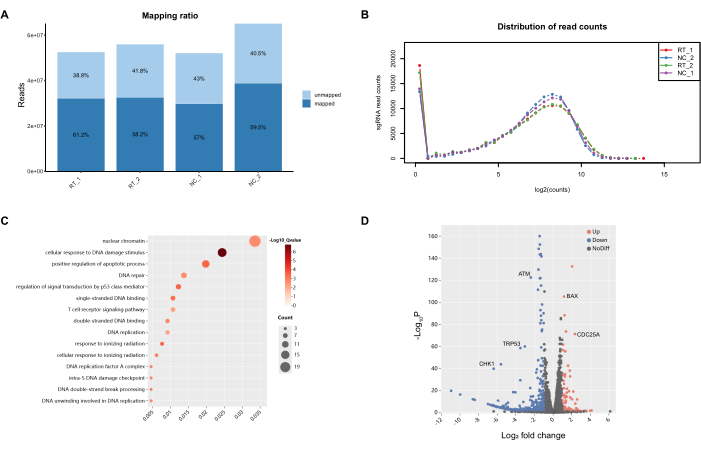
Abbildung 5: Qualitätskontrolle und Basisanalytik von PCR-amplifizierten Produkten. (A) Das Kartierungsverhältnis ergab eine Abdeckung des Referenzgenoms von etwa 60 %. (B) Die Lesezahlverteilung von sgRNAs folgte einer Poisson-Verteilung. (C) Die meisten der 10 wichtigsten Begriffe der Genontologie (GO) wurden mit der Reaktion auf DNA-Schäden in Verbindung gebracht. (D) Vulkandiagramm mit Genanreicherungsmustern mit negativ angereicherten Genen in rot, positiv angereicherten Genen in blau und nicht signifikanten Genen in grau. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
| Viruspartikel Anzahl (VP) | Anzahl der Zählungen (vor der Behandlung mit Puromycin) | Anzahl der Zählungen (nach Puromycin-Behandlung) | Infektion Effizienz |
| 1.23 × 104 | 1.23 × 105 | 1.04 × 104 | 0.084552846 |
| 3,72 × 104 | 1,24 × 106 | 3.61 × 104 | 0.291129032 |
| 6.15 × 104 | 1.23 × 107 | 5.4 × 104 | 0.43902439 |
Tabelle 1: A549-Zellzahlen vor und nach der Puromycin-Selektion unter verschiedenen Viruspartikeln. Anzahl von A549-Zellen, die vor und nach der Behandlung mit Puromycin unter unterschiedlichen Viruspartikeln aufgezeichnet wurden.
Diskussion
Als hochmoderne Technologie zur Gen-Editierung hat der CRISPR-Bildschirm tiefgreifende Veränderungen im Bereich der wissenschaftlichen Forschung ausgelöst5. Diese Technologie, die aus dem CRISPR-Cas9-System hervorgegangen ist, hat sich aufgrund ihrer hohen Effizienz und Präzision zu einem unverzichtbaren Werkzeug für die Untersuchung von Genfunktionenentwickelt 9. Das CRISPR/Cas9-Engineering-Prinzip umfasst das Design und die Einführung spezifischer sgRNAs mit etwa 20 Nukleotiden, um die Cas9-Nuklease dazu zu bringen, Ziel-DNA-Sequenzen präzise zu lokalisieren und zu schneiden, was eine Gen-Editierung wie Knockout, Aktivierung oder Repression ermöglicht6. Dieses Prinzip liegt der Entwicklung von hochdurchsatzfähigen und anpassbaren CRISPR-Bildschirmen zugrunde. Durch den Aufbau von Bibliotheken, die eine große Anzahl von sgRNAs enthalten, können Forscher mehrere Gene gleichzeitig bearbeiten und screenen und so schnell diejenigen identifizieren, die mit bestimmten Phänotypen oder Krankheitszuständen assoziiert sind. Darüber hinaus ermöglicht der CRISPR-Screen Experimente in verschiedenen Zelltypen, Geweben und sogar ganzen Organismen und ermöglicht so ein umfassenderes Verständnis der Genfunktion und der Wirkmechanismen.
Mit der beschleunigten technologischen Entwicklung hat der CRISPR-Bildschirm ein enormes Potenzial für die Anwendung in verschiedenen Bereichen gezeigt. Zum Beispiel kann das CRISPR-Screening Wissenschaftler bei der Entdeckung neuartiger Wirkstoffziele unterstützen und so den Prozess der Arzneimittelforschung und -entwicklung beschleunigen. In der Krankheitsforschung deckt diese Technologie die Mechanismen der Krankheitsentstehung auf und bietet neue Erkenntnisse und Ansätze für Diagnose und Behandlung. Das CRISPR-Screening unterscheidet sich von herkömmlichen Screening-Methoden (wie RNAi und niedermolekularen Inhibitoren) grundlegend in mehreren wichtigen Aspekten: Mechanistisch erreicht CRISPR dauerhafte genetische Veränderungen durch Geneditierung, während RNAi die Genexpression nur vorübergehend unterdrückt und niedermolekulare Inhibitoren die Proteinfunktion reversibel blockieren28,29. In Bezug auf die Spezifität zeigt CRISPR eine überlegene Präzision durch genaues DNA-Targeting, während RNAi anfällig für Off-Target-Effekte ist 3,5. Hinsichtlich der Wirkungsdauer führt CRISPR zu dauerhaften genetischen Veränderungen, verglichen mit den transienten Eingriffen herkömmlicher Methoden. Darüber hinaus ermöglicht CRISPR genomweite Screening-Funktionen und kann gleichzeitig mehrere Gene/Signalwege untersuchen, während gleichzeitig eine hohe Effizienz über verschiedene Zelltypen hinweg beibehaltenwird 9. Traditionelle Ansätze beschränken sich in der Regel auf die Analyse des spezifischen Signalwegs und zeigen bei bestimmten Zelllinien oft eine verminderte Wirksamkeit3. Diese besonderen Merkmale machen das CRISPR-Screening besonders wertvoll für die funktionelle Genomforschung und die Zielentdeckung, insbesondere in Studien, die eine Langzeitbeobachtung oder eine präzise genetische Regulation erfordern, wo es einzigartige Vorteile gegenüber herkömmlichen Screening-Methoden aufweist.
Die Spezifität dieses Prozesses ist jedoch nicht absolut, und es ist möglich, dass das Cas9-Protein fälschlicherweise DNA-Regionen erkennt und spaltet, die der beabsichtigten Zielsequenz ähnlich, aber nicht vollständig identisch sind5. Dieses Ereignis wird allgemein als Off-Target-Effekte bezeichnet. Diese unbeabsichtigte DNA-Spaltung kann zu genomischer Instabilität und unvorhersehbaren genetischen Folgen führen, insbesondere wenn sie in der Humantherapie angewendet wird. Die CRISPR-Technologie hat auch Schwierigkeiten, wenn es um komplexe Genom-Editing-Aufgaben geht. Wenn das CRISPR-System mit Editierungsanforderungen konfrontiert wird, die zahlreiche Gene, Gencluster oder komplizierte genetische Einstellungen umfassen, kann es zu Einschränkungen kommen, wenn es darum geht, die Ergebnisse des Editierungsprozesses genau zu antizipieren und zu lenken1.
Die wichtigste technische Hürde bei der Durchführung eines genomweiten Screenings mit der CRISPR-Technologie ergibt sich aus der enormen Komplexität des Genoms, das Zehntausende von Genen umfasst, wodurch das Risiko von Off-Target-Effekten erheblich erhöhtwird 9. Um die Integrität und Zuverlässigkeit der nachfolgenden Sequenzierungsergebnisse zu gewährleisten, ist es zwingend erforderlich, dass jede Zelle mit einer singulären sgRNA23 transfiziert wird. Dies erfordert eine sorgfältige Kontrolle des MOI durch den Experimentator, um sicherzustellen, dass er unter 0,5 bleibt, während gleichzeitig ein angemessener Schwellenwert für die Kopienzahl eingehalten wird, der mindestens 500 oder mehrbetragen muss 24. Angesichts der Tatsache, dass jedes der Zehntausende von Genen im gesamten Genom die Verwendung von 4-6 sgRNAs erfordert, erfordert der gesamte Screening-Prozess eine umfangreiche Anzahl von Zellen25. Die verlängerte Dauer des Experiments, die beträchtliche Anzahl der beteiligten Zellen und das erhöhte Risiko machen es erforderlich, dass der Experimentator Strategien entwickelt, um diese Bedenken zu lindern26. Die potenziellen Gene müssen mit RNA-seq sowie mit Gain- und Loss-of-Function-Assays weiter validiert werden. In den folgenden Experimenten wurden nach der Gewinnung der Überexpressions- und Knockout-Zellen identifizierter Gene mittels CRISPR-Editierung genomweite Genomik und Transkriptomik durchgeführt, um Zugang zu potenziellen Off-Target-Veränderungen zu erhalten, insbesondere an Loci mit partieller sgRNA-Komplementarität, die eine unspezifische Cas9-Spaltung durchlaufen können. Bioinformatische Vorhersagetools wie COSMID könnten dabei helfen, risikoreiche Off-Target-Stellen zu identifizieren. Rescue-Assays könnten auch verwendet werden, um die Spezifität der Beteiligung identifizierter Gene sicherzustellen.
In der anschließenden Analyse stützten sich die Kriterien für differentiell exprimierte sgRNAs auf den von MAGeCK berechneten Robust Rank Aggregation (RRA)-Score, der statistische Signifikanz, Faltungsänderung und Replikatkonsistenz umfasste31,32. Für einen Datensatz wird jedes Gen nach umfassenden Kriterien eingestuft, die Genexpressionsniveaus und statistische Signifikanz kombinieren. RRA verwendet den Rangkorrelationskoeffizienten von Spearman für den Rang, um die Situation des umfassenden Rangs jedes Gens in allen Datensätzen zu bewerten, und integriert die Sortierergebnisse mehrerer Genexpressionsdatensätze. Ein Gen mit einem relativ hohen Rang in den meisten Datensätzen könnte als echtes differentiell exprimiertes Genangesehen werden 33. Ein p-Wert von RRA kleiner als 0,05 wurde als statistisch signifikant angesehen. Eine Faltungsänderung von mindestens einer 1,5-fachen Zunahme oder 33%igen Abnahme wurde als differentielle Ausprägung angesehen. Darüber hinaus musste die Konsistenz von 3 unabhängigen Replikaten als statistisch unterschiedliche Expression betrachtet werden. Nach dem RRA-Ranking für essentielle Gene bedeutet ein niedrigerer RRA-Score eine höhere Bedeutung.
Als leistungsfähiges Werkzeug für die Genom-Editierung zeigt die CRISPR-Cas9-Technologie ein beispielloses Potenzial und eine Genauigkeit bei der Untersuchung des Bereichs der Strahlenresistenz26,27. Die Anwendung hat jedoch noch Einschränkungen. Abgesehen vom Risiko von Off-Target-Effekten führen die mit der Strahlenempfindlichkeit verbundenen Feinheiten zu einer Komplexität des Screening-Prozesses27. Diese Sensitivität wird nicht nur durch ein einzelnes Gen gesteuert, sondern entsteht als Folge des komplizierten Zusammenspiels zwischen mehreren Genen und ihren regulatorischen Netzwerken26. Trotz der erfolgreichen Identifizierung spezifischer Gene, die sich auf die Strahlenempfindlichkeit beziehen, stellen sie möglicherweise nur einen Bruchteil des Gesamtbildes dar, was eine große Herausforderung für die umfassende Aufklärung der zugrunde liegenden Mechanismen darstellt 34,35,36. Hier finden Sie Lösungen für häufige Probleme beim CRISPR-Screening, wie z. B. geringe Infektionseffizienz, sgRNA-Abbau und Sequenzierungsfehler: Da das CRISPR-Screening einen MOI-Wert <0,5 erfordert, ist die Anpassung des Zellzustands entscheidend, um sicherzustellen, dass sich die Zellen in einer logarithmischen Wachstumsphase befinden, oder um die Infektionseffizienz über Fluoreszenzassays zu bewerten. Zu den möglichen Ursachen für den sgRNA-Abbau gehören Nukleasekontamination oder Fehler im Vektordesign, so dass wiederholte Gefrier-Tau-Zyklen von sgRNA-Bibliotheken vermieden werden sollten (Aliquote bei -80 °C lagern), und NGS-Sequenzierung verwendet werden sollte, um die Integrität der sgRNA-Bibliothek zu überprüfen; Ein Sequenzierungsversagen kann auf eine unzureichende Sequenzierungstiefe oder Probleme mit dem Design des sgRNA-Primers zurückzuführen sein. Zur Verbesserung der Genauigkeit der sgRNA-Sequenzidentifikation wird eine Paired-End-Sequenzierung empfohlen, wobei eine Sequenzierungstiefe von >500 × eine Abdeckung von 37,38,39,40,41,42 empfohlen wird.
Die Strahlentherapie ist nach wie vor ein Eckpfeiler der Krebsbehandlung, weist jedoch eine bemerkenswerte Variabilität in der therapeutischen Wirksamkeit bei verschiedenen Tumorarten und sogar bei Patienten mit demselben Krebs auf9. Diese interindividuelle Heterogenität erfordert häufig Kombinationstherapien, um eine optimale Tumoreradikation zu erreichen11,12. In der klinischen Praxis hat sich die Integration der Strahlentherapie mit der Immuntherapie - insbesondere von Immun-Checkpoint-Inhibitoren und Antikörper-Wirkstoff-Konjugaten - als herausragendes therapeutisches Paradigma herauskristallisiert19. Dieses Protokoll stellt einen in vitro CRISPR-Screen unter Verwendung einer relativ homogenen Krebszelllinie dar, die die genetische und epigenetische Heterogenität von Tumoren in vivo nicht vollständig widerspiegeln konnte. Nach der Bestimmung der geeigneten Strahlendosis für das Screening liefern die Ergebnisse potenziell strahlenempfindliche oder strahlenresistente Gene, die sowohl in in vitro zellulären Assays als auch in in vivo Tiermodellen weiter bestätigt werden müssen. Darüber hinaus könnten in vivo CRISPR-Screenings plus Einzelzell-Transkriptomik hilfreich sein, um die Komplexität der Tumorheterogenität zu entschlüsseln. Das in vitro CRISPR-Screening identifizierte wichtige Gene bei der Reparatur von DNA-Schäden innerhalb von Zellen sowie bei autokrinen Mechanismen als Reaktion auf Strahlung, replizierte TME jedoch nicht vollständig heterogen. Zum Beispiel erhöht Hypoxie die ROS-Produktion und löst eine Rückkopplungsschleife aus, die die metabolische Anpassung, die Produktion von Antioxidantien und die Aktivierung der Autophagie stimuliert und letztendlich die Strahlenresistenz fördert18. Immunzellen könnten nach der Bestrahlung im parakrinen Mechanismus aktiviert werden, was zu Strahlenempfindlichkeit führt14. Die EZM dient als Reservoir für Wachstumsfaktoren, und Zytokine haben sowohl positive als auch negative Auswirkungen auf die Strahlenempfindlichkeit21. Das in vivo CRISPR-Screening sollte durchgeführt werden, um die Heterogenität der TME widerzuspiegeln. Darüber hinaus konnte das in vitro CRISPR-Screening nur wichtige Gene bei der Reparatur von DNA-Schäden innerhalb von Zellen sowie bei autokrinen Mechanismen als Reaktion auf Strahlung identifizieren, erfasste jedoch nicht vollständig den in vivo Einfluss auf die Strahlenempfindlichkeit, einschließlich Tumorgefäße und systemische Effekte20,21. Das in vivo CRISPR-Screening sollte durchgeführt werden, um potenzielle Faktoren zu identifizieren, die an der Strahlenempfindlichkeit in vivo beteiligt sind.
Hier wird ein umfassendes Protokoll entwickelt, das auf die Nutzung des genomweiten CRISPR-Screenings abzielt, um Gene zu lokalisieren, die entweder an der Empfindlichkeit oder Resistenz gegen Strahlentherapie beteiligt sind. Durch die Entschlüsselung neuer Signalwege soll diese Methodik den Weg für Fortschritte in der Forschung zur Tumorstrahlentherapie ebnen. Dieses Protokoll könnte auch als Referenz für die Erforschung der Mechanismen klinischer Einschränkungen in der Chemotherapie und des Fortschreitens alternativer Therapeutika dienen. Zusammenfassend lässt sich sagen, dass die CRISPR-Technologie ein erhebliches Potenzial und einen erheblichen Wert verkörpert, aber ihre inhärenten Einschränkungen unterstreichen die Bedeutung einer nüchternen Erkennung und eines vernünftigen Ansatzes. Während zukünftige Forschungen und Anwendungen erforscht werden, ist es unerlässlich, sich unermüdlich darum zu bemühen, die CRISPR-Technologie zu erforschen und zu optimieren, um ihre Einschränkungen zu überwinden und ihr volles Potenzial auszuschöpfen.
Offenlegungen
Nichts.
Danksagungen
Diese Studie wurde unterstützt durch das Regional Science and Technology Innovation Project der Provinz Hubei (2024EIA001) und das Medical Science and Technology Innovation Platform Construction Support Project des Zhongnan Hospital der Universität Wuhan (PTXM2025001). Abbildung 1 wurde mit Figdraw erstellt.
Materialien
| Name | Company | Catalog Number | Comments |
| 150 mm cell and tissue culture dish | Wuxi NEST Biotechnology Co., Ltd. | 715011 | Cell culture |
| 35 mm cell and tissue culture dish | Wuxi NEST Biotechnology Co., Ltd. | 706011 | Cell culture |
| A549 cell line | ATCC | - | - |
| Cell counting kit-8 | Shanghai Beyotime Biotech Inc | C0041 | For cell viability assay |
| CO2-independent medium | PHCbi | MCO-50AIC | Cell culture |
| Countess 3 FL automated cell counter | Themo Scientific | AMQAF2001 | For cell counting |
| EDTA | Gibco | 25200056 | Cell culture |
| Fetal bovine serum | Gibco | 10099141 | Cell culture |
| Fluorescence microscopy | LEICA | ebq 100-04 | For fluorescence microscope |
| Genome-wide CRISPR lentiviral library | Shanghai OBiO Technology Co., Ltd. | H5070 | For lentiviral infection |
| MiniAmp plus PCR | Themo Scientific | C37835 | For PCR amplification |
| NEST cell culture plates, 12-well | Wuxi NEST Biotechnology Co., Ltd. | 712002 | Cell culture |
| NEST cell culture plates, 6-well | Wuxi NEST Biotechnology Co., Ltd. | 703002 | Cell culture |
| NEST cell culture plates, 96-well | Wuxi NEST Biotechnology Co., Ltd. | 701001 | Cell culture |
| PBS | Gibco | C10010500BT | Cell culture |
| PCR forward primer (AATGGACTATCATATGCTTACCGTAACTTGAAAGTATTTCG) | Beijing AuGCT Biotech Co., Ltd. | - | For PCR amplification |
| PCR reverse primer (GATGTGCGCTCTGCCCACTGACGGGCA) | Beijing AuGCT Biotech Co., Ltd. | - | For PCR amplification |
| Polybrene | Shanghai Beyotime Biotech Inc | C0351 | For lentiviral infection |
| Puromycin | MCE | HY-K1057 | For seletion post lentiviral infection |
| RPMI 1640 cell culture medium | Gibco | 23400-021 | Cell culture |
| TIANGENamp genomic DNA kit | Beijing TIANGEN Biotech Co.,Ltd. | DP304 | For genomic DNA extraction |
| X-ray powder diffractometer | PerkinElmer | For radiotherapy |
Referenzen
- Barrangou, R., Marraffini, L. A. CRISPR-Cas systems: Prokaryotes upgrade to adaptive immunity. Mol Cell. 54 (2), 234-244 (2014).
- Joung, J., et al. Genome-scale CRISPR-Cas9 knockout and transcriptional activation screening. Nat Protoc. 12 (4), 828-863 (2017).
- Sanjana, N. E., Shalem, O., Zhang, F. Improved vectors and genome-wide libraries for CRISPR screening. Nat Methods. 11 (8), 783-784 (2014).
- Arroyo, J. D., et al. A Genome-wide CRISPR death screen identifies genes essential for oxidative phosphorylation. Cell Metab. 24 (6), 875-885 (2016).
- Sidik, S. M., et al. A genome-wide CRISPR screen in toxoplasma identifies essential apicomplexan genes. Cell. 166 (6), 1423-1435 (2016).
- Jing, Y., et al. Genome-wide CRISPR activation screening in senescent cells reveals SOX5 as a driver and therapeutic target of rejuvenation. Cell Stem Cell. 30 (11), 1452-1471 (2023).
- Shifrut, E., et al. Genome-wide CRISPR screens in primary human t cells reveal key regulators of immune function. Cell. 175 (7), 1958-1971 (2018).
- Shalem, O., et al. Genome-scale CRISPR-Cas9 knockout screening in human cells. Science. 343 (6166), 84-87 (2014).
- Katti, A., Diaz, B. J., Caragine, C. M., Sanjana, N. E., Dow, L. E. CRISPR in cancer biology and therapy. Nat Rev Cancer. 22 (5), 259-279 (2022).
- Sasmal, R., et al. Synthetic host-guest assembly in cells and tissues: Fast, stable, and selective bioorthogonal imaging via molecular recognition. Anal Chem. 90 (19), 11305-11314 (2018).
- Roos, W. P., Thomas, A. D., Kaina, B. DNA damage and the balance between survival and death in cancer biology. Nat Rev Cancer. 16 (1), 20-33 (2016).
- Huang, R., Zhou, P. K. DNA damage repair: Historical perspectives, mechanistic pathways and clinical translation for targeted cancer therapy. Signal Transduct Target Ther. 6 (1), 254(2021).
- O'Connor, M. J. Targeting the DNA damage response in cancer. Mol Cell. 60 (4), 547-560 (2015).
- Lord, C. J., Ashworth, A. The DNA damage response and cancer therapy. Nature. 481 (7381), 287-294 (2012).
- Carusillo, A., Mussolino, C. DNA Damage: From threat to treatment. Cells. 9 (7), 1665(2020).
- Li, X., Heyer, W. D. Homologous recombination in DNA repair and DNA damage tolerance. Cell Res. 18 (1), 99-113 (2008).
- Chang, H., Pannunzio, N. R., Adachi, N., Lieber, M. R. Non-homologous DNA end joining and alternative pathways to double-strand break repair. Nat Rev Mol Cell Biol. 18 (8), 495-506 (2017).
- Andratschke, N., et al. European society for radiotherapy and oncology and European organization for research and treatment of cancer consensus on re-irradiation: Definition, reporting, and clinical decision making. Lancet Oncol. 23 (10), e469-e478 (2022).
- Olivares-Urbano, M. A., Grinan-Lison, C., Marchal, J. A., Nunez, M. I. CSC radioresistance: A therapeutic challenge to improve radiotherapy effectiveness in cancer. Cells. 9 (7), 1651(2020).
- Schaue, D., McBride, W. H. Opportunities and challenges of radiotherapy for treating cancer. Nat Rev Clin Oncol. 12 (9), 527-540 (2015).
- Allen, C., Her, S., Jaffray, D. A. Radiotherapy for cancer: Present and future. Adv Drug Deliv Rev. 109, 1-2 (2017).
- Xue, C., Greene, E. C. DNA repair pathway choices in CRISPR-Cas9-Mediated genome editing. Trends Genet. 37 (7), 639-656 (2021).
- Tzelepis, K., et al. A CRISPR dropout screen identifies genetic vulnerabilities and therapeutic targets in acute myeloid leukemia. Cell Rep. 17 (4), 1193-1205 (2016).
- Rottner, A. K., et al. A genome-wide CRISPR screen identifies CALCOCO2 as a regulator of beta cell function influencing type 2 diabetes risk. Nat Genet. 55 (1), 54-65 (2023).
- Chen, M., et al. CRISPR-Cas9 for cancer therapy: Opportunities and challenges. Cancer Lett. 447, 48-55 (2019).
- Gupta, D., et al. CRISPR-Cas9 system: A new-fangled dawn in gene editing. Life Sci. 232, 116636(2019).
- Li, B., et al. Genome-wide CRISPR screen identifies host dependency factors for influenza A virus infection. Nat Commun. 11 (1), 164(2020).
- Hung, K. L., et al. Targeted profiling of human extrachromosomal DNA by CRISPR-CATCH. Nat Genet. 54 (11), 1746-1754 (2022).
- Bhattacharya, D., Van Meir, E. G. A simple genotyping method to detect small CRISPR-Cas9 induced indels by agarose gel electrophoresis. Sci Rep. 9 (1), 4437(2019).
- Razavi, Z., Soltani, M., Souri, M., Van Wijnen, A. J. CRISPR innovations in tissue engineering and gene editing. Life Sci. 358, 123120(2024).
- Wang, W., et al. A genome-wide CRISPR-based screen identifies KAT7 as a driver of cellular senescence. Sci Transl Med. 13 (575), eabd2655(2021).
- Xu, X., et al. Engineered miniature CRISPR-Cas system for mammalian genome regulation and editing. Mol Cell. 81 (20), 4333-4345 (2021).
- Huang, M., et al. FACS-based genome-wide CRISPR screens define key regulators of DNA damage signaling pathways. Mol Cell. 83 (15), 2810-2828 (2023).
- Feng, J., et al. Genome-wide CRISPR screen identifies synthetic lethality between DOCK1 inhibition and metformin in liver cancer. Protein Cell. 13 (11), 825-841 (2022).
- Saurat, N., et al. Genome-wide CRISPR screen identifies neddylation as a regulator of neuronal aging and AD neurodegeneration. Cell Stem Cell. 31 (8), 1162-1174 (2024).
- Du, Y., Liu, Y., Hu, J., Peng, X., Liu, Z. CRISPR/Cas9 systems: Delivery technologies and biomedical applications. Asian J Pharm Sci. 18 (6), 100854(2023).
- Shi, J., et al. A genome-wide CRISPR screen identifies WDFY3 as a regulator of macrophage efferocytosis. Nat Commun. 13 (1), 7929(2022).
- Liu, N., Olson, E. N. CRISPR modeling and correction of cardiovascular disease. Circ Res. 130, (2022).
- Feng, X., et al. Genome-wide CRISPR screens using isogenic cells reveal vulnerabilities conferred by loss of tumor suppressors. Sci Adv. 8 (19), 6638(2022).
- Ghaemi, A., et al. CRISPR-Cas9 genome editing delivery systems for targeted cancer therapy. Life Sci. 267, 118969(2021).
- Derry, W. B. CRISPR: Development of a technology and its applications. FEBS J. 288 (2), 358-359 (2021).
- Sharma, G., Sharma, A. R., Bhattacharya, M., Lee, S. S., Chakraborty, C. CRISPR-Cas9: A preclinical and clinical perspective for the treatment of human diseases. Mol Ther. 29 (2), 571-586 (2021).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenWeitere Artikel entdecken
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten