Method Article
放射線感受性および放射線耐性遺伝子を明らかにするためのゲノムワイドCRISPRスクリーニング
* これらの著者は同等に貢献しました
要約
ゲノムワイドなCRISPR/Cas9スクリーニング法の適用により、放射線耐性および感受性遺伝子を選択するために、細心の注意を払った構造化されたアプローチが与えられています。このプロトコルは、臨床投与された化学薬品に対する耐性のメカニズムを調査する他の研究努力のための汎用性の高いフレームワークとしても役立つ可能性を秘めています。
要約
CRISPR-Cas9システムは、強力なゲノム編集ツールとして活用され、再利用されています。この技術を活用することで、研究者は生細胞内のDNA配列を正確に切り取り、貼り付け、さらには書き換えることができます。それにもかかわらず、CRISPRスクリーニング技術の応用は、単なる実験をはるかに超えています。これは、遺伝病との闘いにおける極めて重要なツールとして機能し、複雑な遺伝的ランドスケープを体系的に分析し、研究者が生物学的現象の根底にある分子メカニズムを解明し、科学者ががん、嚢胞性線維症、鎌状赤血球貧血などの病気の根本原因を特定して標的にすることを可能にしています。中でも、がんは医療にとって大きな課題であり、撲滅に向けた取り組みに拍車をかけています。放射線療法は、従来の治療法として、結果をもたらしますが、限界があります。がん細胞を根絶するだけでなく、健康な組織に損傷を与え、生活の質を低下させる悪影響を引き起こします。さらに、すべてのがん細胞が放射線療法に反応するわけではなく、一部のがん細胞が耐性を獲得し、状態を悪化させる可能性があります。これに対処するために、放射線感受性および放射線耐性遺伝子の効率的な同定を可能にする包括的な全ゲノムCRISPRスクリーニング技術が導入され、がんの研究と治療の分野が進歩します。記載されたプロトコルに従って照射に曝露された肺腺癌細胞において、ゲノムワイドなCRISPRスクリーニングを実施し、それを通じて放射線抵抗性および放射線感受性関連遺伝子の両方を同定した。
概要
生命現象の解明は、細胞の振る舞いの研究と本質的に絡み合っており、細胞の振る舞いの解明は、そのゲノムの探索と根本的につながっています。現代の技術が進化し続ける中、医学研究者は、さまざまな疾患の治療結果を向上させるために、遺伝子編集による細胞の振る舞いの変化に徐々に注意を向けています。この点で、クラスター化規則的な間隔を空けた短い回文反復(CRISPR)技術は、その比較的単純なアプリケーションにより、ゲノム編集の革新的なツールとして浮上しています1。CRISPR-Cas9システムは、Cas9ヌクレアーゼとシングルガイドRNA(sgRNA)で構成されており、標的DNA配列を特異的に認識して結合し、Cas9ヌクレアーゼをその場所で切断するように誘導し、ゲノムDNA2,3,4の二本鎖切断(DSB)を引き起こします。さらに、他の物質の導入は、ゲノムへの特定の挿入、欠失、または突然変異につながる可能性があり、標的遺伝子編集が可能になります。
機能ゲノミクス研究において、RNA干渉(RNAi)スクリーニングは、かつてがんにおける遺伝子の役割を調査するための大規模な機能喪失実験を行うための方法として広く利用されていました。RNAi技術は、標的遺伝子を特異的にサイレンシングすることにより遺伝子機能を研究し、研究者が重要な発がん因子を特定するのに役立ちます。しかし、オフターゲット効果や遺伝子ノックダウン効率が不完全であることには限界があります。オフターゲット効果は、他の非標的遺伝子のサイレンシングにつながり、それによって実験結果の精度と信頼性を損なう可能性がある1,2。さらに、RNAiは特定の遺伝子に対して低いノックダウン効率を示し、標的遺伝子の発現を完全に抑制できない可能性があります。従来のRNAiスクリーニングとは対照的に、CRISPRスクリーニングはより高い特異性と効率性を示します3。この技術により、特定の遺伝子を精密に編集できるだけでなく、ゲノムワイドな大規模スクリーニングも可能となり、遺伝子機能研究を強力にサポートします。CRISPR-Cas9システムに基づく強力な遺伝子編集ツールであるCRISPRスクリーニング技術は、細胞5、6、7、8の特定の遺伝子の未知の機能を効率的にスクリーニングし、明らかにするために使用されます。研究者は、特定の遺伝子または遺伝子領域に対してsgRNAをバッチで設計し、対応するsgRNAライブラリーを正確かつ厳密に調製し、その完全性と機能性を確保します9。その後、これらのsgRNAライブラリーはレンチウイルス粒子にカプセル化され、宿主細胞に効率的に感染するために使用されます。感染が成功した後、感染した細胞は、個別に定義されたスクリーニング条件下で培養されます。スクリーニング時には、スクリーニングされた細胞のゲノムDNAが抽出され、高水準の純度と量が維持されます。続いて、sgRNAの標的領域をPCR増幅にかけ、核酸の所望のセグメントを正確に複製するプロセス3,9を行う。最後に、増幅されたDNA断片に対してハイスループットシーケンシングが行われ、標的領域の包括的かつ効率的な解析が可能になり、研究対象の遺伝子の機能と挙動に関する貴重な洞察が得られます4。
がんは、複雑な病気として人間の健康にとって手ごわい脅威となっています。世界中の研究者と臨床医が、発がんの分子メカニズムを解明し、新しい治療戦略を開発するために協力して取り組んでいます。基礎研究の成果を臨床応用につなげるための国際協力が確立され、最終的には患者の転帰を改善することを目標としています。Sasmal et al.は、転移性がん細胞の正確な標的化のための合成ホスト-ゲストシステムに基づく生体直交アセンブリ戦略を提案し、数十人の科学者が医療技術を進歩させるのに大きく貢献しました。彼らの優れた研究活動は、高い革新性と独自の洞察力を持ち、科学界に有意義な貢献をしています10。がんは、DNA損傷応答の不規則な制御から生じるゲノム不安定性の激動状態を特徴としています11-14。DNA損傷には、一塩基欠損、一本鎖切断、およびDSBが含まれ、相同組換え(HR)および非相同末端結合(NHEJ)は、異なる段階でDSBの修復に関与する15,16,17。これに基づいて、放射線療法は、高エネルギー光線(X線やγ線など)を利用して腫瘍組織に放射線を照射し、腫瘍細胞にDNA損傷を引き起こし、それによって腫瘍細胞の成長と増殖を妨害する実行可能な治療選択肢として浮上しています18。しかし、放射線療法は、かなりの割合のがん患者において必ずしも望ましい効果をもたらすとは限らず、パラがん組織への損傷や、放射線療法に対する感度の低さなどの腫瘍の固有の特性によって課せられる制限に起因する可能性がある19,20,21。
理論的には、どの細胞タイプでもCRISPRスクリーニングに使用することができます。しかし、変異した集団で十分な代表性を維持するためには、多数の開始細胞が必要です。存在量が少ない細胞種は、ゲノムワイドスクリーニングには特に適していません。ライブラリーの選択に関しては、ほとんどのライブラリーは標的遺伝子ごとに3〜6個のgRNAを含み、集団内の各gRNAの分布を維持することは重要である18。特定のgRNAの濃縮または枯渇による表現の喪失は、結果の分布にばらつきをもたらす可能性があります。この問題に対処するためには、市場でテストされた市販のCRISPRライブラリーを選択することが好ましい選択であり得る20。 インビトロ 均質ながん細胞株を用いたCRISPRスクリーニングでは、 in vivo 腫瘍の遺伝的およびエピジェネティックな不均一性を完全に捉えられない可能性があります。 in vitro スクリーニングにより、DNA損傷修復と放射線誘発性オートクリンシグナル伝達に関与する主要な遺伝子が明らかになりましたが、低酸素誘導性放射線耐性(ROS、代謝適応、オートファジー経由 )、免疫介在性パラクリン効果、ECM依存性サイトカイン調節など、腫瘍微小環境を完全に再現することはできませんでした。放射線感受性または放射線耐性に関連する遺伝子を探索するためにCRISPRスクリーニングを使用する前に、これらの要因を慎重に検討する必要があります。現在の治療状況に照らして、放射線治療の有効性を効果的に高めるためには、放射線抵抗性と放射線感受性に関連する因子を特定し、深く研究することが急務です22。未知の遺伝子の機能を研究する上でのCRISPRスクリーニングの主な利点を考慮して、放射線感受性および放射線耐性遺伝子を効率的に同定するために、体系的に詳細な全ゲノムCRISPRスクリーニング技術が提供されます。
プロトコル
この研究で使用した試薬と機器は、 材料表に記載されています。
1. 適切な放射線量の選定
- 接着細胞の調製とプレーティング
- 継代および細胞増殖のために10% FBSを含むRPMI 1640完全細胞培養培地を使用して、細胞密度を5 x 105 cells/mLに調整します。細胞を3.5cmの培養皿に分配し、さまざまな線量で放射線を照射します。各ディッシュに2 mL(1 x 106 細胞)を加え、5% CO2で37°Cで一晩インキュベートします。
- 異なる放射線量の応用
- 3.5cmの培養皿に1から5までの番号を付けます。グループ #1 を対照群として利用し、残りの 4 つのグループを治療群として指定します。
- 6ウェルプレートの端を治療群用のシーリングメンブレンでシールし、それぞれ2、4、6、8Gyの放射線量を投与します。治療後、皿をインキュベーターに戻します。
- 放射線照射後の細胞株のプレーティング
- 6ウェルプレートと96ウェルプレートを調製します。3.5 cm培養皿中の細胞をPBSで1回洗浄し、対数的に増殖する細胞を0.25%トリプシンで消化し、10% FBSを含むRPMI 1640完全培地で細胞密度を1 x 105 細胞/mLに調整します。
- 10 μL/ウェル (1,000 細胞/100 μL) を 6 ウェルプレートに播種し、放射線量ごとに 3 回繰り返します。2 mLの完全培地を各ウェルに加え、14日後(各クローングループに約50個の細胞がある場合)にカウントします。
- 30 μL/ウェル (3,000 細胞/100 μL) を 96 ウェルプレートに播種し、放射線量ごとに 5 回繰り返します。70 μLの完全培地を各ウェルに加え、72時間後に細胞生存率を測定します。
- 細胞生存率の測定
注: クローン誘発阻害率 = 1 - (治療群のクローン数/対照群のクローン数) x 100%。CCK-8アッセイは、細胞デヒドロゲナーゼによって還元できる水溶性テトラゾリウム塩(WST-8)を利用して、水溶性の高い黄色のホルマザン生成物を生成します11。産生されるホルマザンの量は、生細胞の数に正比例し、その光学密度(波長450nmで測定)は、細胞の代謝活性と増殖状態を正確に反映している11,13。この確立された原理に基づき、CCK-8アッセイは、細胞増殖アッセイや腫瘍薬物感受性試験など、さまざまなアプリケーションに広く採用されています。このプロトコルでは、CCK-8アッセイを使用して、さまざまな放射線量下での細胞生存率を体系的に評価します。- CCK8試薬とFBSを含まないRPMI 1640培地を1:9の比率で混合します。96ウェルプレートの培地を捨て、CCK8含有培地100μLを各ウェルに加えます。
- 暗所で1時間インキュベートし、マイクロプレートリーダーを使用して450 nmのOD値を測定します。
注:細胞生存率= [(治療群のOD値-ブランクウェルのOD値)/(対照群のOD値-ブランクウェルのOD値)]×100%
- 放射線量の選択
- クローン原性阻害率と細胞生存率を統合し、研究ニーズに基づいて適切な放射線量を選択します。放射線耐性遺伝子と放射線感受性遺伝子の両方をスクリーニングするために、阻害率50%の放射線量を選択します。
2. 適切なMOIとピューロマイシン濃度の選択
- 接着細胞のめっき
- 細胞密度を3 x 105 cells/mLに調整し、1ウェルあたり1 mL(3 x 105 細胞)を12ウェルプレートに接種します。プレートを5%CO2で37°Cで一晩インキュベートします。
- レンチウイルス感染症
- ピペットを使用して12ウェルプレートから培地を吸引し、10% FBSを含む1 mLのRPMI 1640完全培地と交換します。品質チェック済みの全ゲノムCRISPRレンチウイルスライブラリー(市販のライブラリーを推奨)を調製し、対数濃度勾配(例:0-10-50-100-200-400-800)を設定します4,5,6,7,8。
- 対応する量のレンチウイルスをポリブレンの皿2μLに加え、室温で5分間平衡化します。レンチウイルスとポリブレンの混合物を各ウェルにゆっくりと滴下し、よく混合し、5%CO2で37°Cで一晩インキュベートします。
- 細胞死のための最小ピューロマイシン濃度の決定
- 親細胞密度を3 x 105 細胞/mLに調整し、ウェルあたり1 mL(3 x 105 細胞)を12ウェルプレートに接種します。プレートを5%CO2で37°Cで一晩インキュベートします。
- 12ウェルプレートの各ウェルにピューロマイシンを0-0.1-0.2-0.5-0.5-1-2-4-8 μMの濃度勾配で添加し、72時間後に細胞をカウントします。ウェル内のすべての細胞を死滅させる最小濃度は、細胞死滅のための最小ピューロマイシン濃度であり、ウイルス感染細胞のその後のスクリーニングに使用されます。
- 感染後のピューロマイシン選択
- 感染後2日目に、各ウェルから培地を吸引し、10%FBSを含むRPMI 1640完全培地1mLと交換します。48時間培養を続けます。
- 感染の72時間後、既存の培地を、細胞死滅のための最小ピューロマイシン濃度を含む完全な培地と交換します。12ウェルプレートに2ウェルをレンチウイルス感染のない状態で陰性および陽性対照として設置します。ネガティブコントロールをピューロマイシンで治療し、ポジティブコントロールは未治療のままにします。.72時間培養を続けます。
- ピューロマイシン選択の72時間後、ネガティブコントロールのすべての親細胞は死滅しますが、ポジティブコントロールの親細胞は最小限の死を示します。各ウェルのMOIを計算します。
注:MOI =(ウイルス感染群の細胞数/陽性対照群の細胞数)x 100%。その後のスクリーニングには、MOI が ~0.3 のウイルス濃度を使用します。
3. ゲノムワイドCRISPRレンチウイルスライブラリー感染
- 接着細胞株の播種
- 継代および細胞増殖のために、10% FBSを含むRPMI 1640完全培地で細胞密度を1 x 107 cells/mLに調整します。1 mLの細胞(1 x 107 細胞)を各15 cm培養皿に37°Cで5%CO2 と共に8時間接種します。細胞が接着し、70%〜80%のコンフルエンスに達すると、ウイルス感染の準備が整いました(コピー数は約500になります)。
- レンチウイルス感染症
- 15 cm培養皿から培地を吸引し、10% FBSを含む15 mLのRPMI 1640完全培地と交換します。品質チェック済みの全ゲノムCRISPRレンチウイルスライブラリーを調製します。
- MOI = 0.3〜30 μL/皿のポリブレンに対応する量のレンチウイルスを加え、室温で5分間平衡化し、レンチウイルスとポリブレンの混合物を15 cmの培養皿にゆっくりと滴下し、よく混合し、5%CO2 で37°Cで一晩インキュベートします。同時に、親細胞を対比させた15cmの培養皿を調製します。
- 感染後のピューロマイシン選択
- ウイルス感染後2日目に、15 cm培養皿から培地を吸引し、10% FBSを含む15 mLのRPMI 1640完全培地と交換します。48時間培養を続けます。
- 感染後72時間で、培地を最小致死濃度のピューロマイシンを含む完全な培地と交換します。.感染していない親細胞を同様にネガティブコントロールとして扱い、72時間培養を続けます。ピューロマイシン選択の72時間後、陰性対照群の親細胞はすべて死滅し、レンチウイルス感染から生き残った細胞は感染に成功したと見なされます。
- Day 0ゲノムの抽出
- 0.25%トリプシンを使用して15 cm培養皿1枚から細胞を消化し、10% FBSを含むRPMI 1640完全培地に再懸濁し、細胞数をカウントします。このサンプルに Day 0 のラベルを付けます。
- 300 x g で5分間(室温)遠心分離し、上清を捨てます。PBS1 mLに再懸濁し、300 x g で5分間遠心分離し、上清を捨てます。Day 0のゲノムDNAを抽出し、ナノ滴UV分光光度計を使用してDNA濃度と純度を測定します。
- sgRNA完全性検出のためには、アガロースゲル電気泳動を用いて増幅されたsgRNAライブラリーを分析し、バンドが明確で有意な分解がないことを確認し、それによりライブラリーの完全性を維持する23。レンチウイルスライブラリのカバレッジ評価では、ディープシーケンシング技術を使用して、ライブラリ内のsgRNAのシーケンシング解析を行います24,25。
- CRISPRスクリーニングでは、PCR増幅はsgRNAフラグメントの完全性とライブラリ品質の予備的な検証として機能します6。ライブラリーカバレッジとsgRNAの分布パターンをより包括的に解析し、スクリーニング中に各標的遺伝子のsgRNAが適切に表現されるようにするには、スクリーニング9の堅牢性について深く理解するためにNGSを実施します。
注:スクリーニング前およびスクリーニング後のsgRNA存在量の変化を検出し、潜在的なsgRNAのオフターゲット統合またはライブラリコンタミネーションを特定することにより、CRISPRスクリーニングの精度と信頼性をさらに向上させることができます。これらのDay 0サンプルは、重要なネガティブコントロールとして機能し、PCR増幅とNGSによる包括的な品質評価を受けて、(1)必須遺伝子の枯渇の確認(適切なライブラリ表現を示す)、および(2)非必須遺伝子の安定した発現の実証(実験ベースライン条件の確立)9の2つの主要な目的を達成します。 - カバレッジが期待されるレベルに達していることを確認して、ライブラリの多様性と代表性を確保し、スクリーニングプロセスでのバイアスを回避します。感染効率の測定には、蛍光イメージングを使用して感染効率を評価し、実験要件を満たしていることを確認します。
4. スクリーニング条件としての放射線の適用
- グルーピング
- CRISPR感染細胞の密度を1 x 107 細胞/mLに調整し、15 cmシャーレごとに1 mLを接種します。5% CO2 で 37 °C で一晩インキュベートします。
- 放射線治療
- 細胞を治療群と対照群の2つのグループにランダムに分け、グループごとに6つの皿を用意します。治療群の細胞に適切な量の放射線を投与し、対照群の細胞は未処理のままにして正常に増殖させます。放射線照射後、5% CO2 と共に 37 °C で 7 日間インキュベートします。
- 2週目には、治療群細胞に適切な線量の放射線を繰り返し投与し、対照群細胞を未処理のままにして、正常に増殖させます。放射線療法後、5% CO2 と共に 37 °C でさらに 7 日間インキュベートします。
5. ゲノムの抽出とシーケンシング
- 14日目の抽出
- 14日間の治療後、治療群と対照群の細胞を0.25%トリプシンで消化し、10%FBSを含む完全なRPMI 1640培地を使用して再懸濁し、14日目-RTまたは14日目-NCとして標識します。
- 細胞を300 x g で5分間遠心分離し、上清を捨てます。PBS1 mLに再懸濁し、300 x g で5分間遠心分離し、上清を再度廃棄します。14日目のゲノムDNAを抽出し、ナノドロップUV吸光度分光法を使用してDNAの濃度と純度を検出します。
- PCR増幅
- 配列の対応するプライマーを調製し( 材料表を参照)、10 μMに希釈します.試薬を滅菌マイクロ遠心チューブに添加して20 μLの反応システムをセットアップし、300 x gで5秒間遠心分離し、よく混合します。増幅条件に応じてPCR機器のパラメータを設定し、PCR産物を取得します。
- アガロースゲル電気泳動検出
- ゲルキャスティングプレートを作製し、型の端部をアガロースでシールし、コームを挿入し、サンプルDNAの長さに応じて適切な濃度のアガロースゲルを調製します。
- 一定量のアガロース粉末を正確に計量し、適量の電気泳動緩衝液を加えてよく混合し、電子レンジで加熱して溶かします。少し冷やした後、適量の核酸色素を加えて穏やかに混ぜ、ゲルキャスティング型にゆっくりと流し込みます。ゲルが30分間固まるのを待ちます。
- コームを取り外し、電気泳動タンクに適量の電気泳動バッファーをゲルが覆われるまで加えます。DNAサンプルに適量のローディングバッファーを添加し、よく混合し、ピペットを使用して混合物をサンプルウェルにゆっくりと加えます。
- DNA断片のサイズとアガロースゲルの濃度に基づいて適切な電圧を設定します。電気泳動後、ゲルを慎重に取り出し、ゲルイメージャーに入れて結果を観察し、PCR増幅が成功したかどうかを確認します。
- Illuminaシーケンシング
- 3つのグループ(Day 0グループ、コントロールグループ、治療グループ)からゲノムDNAを採取し、ライブラリ構築とIlluminaシーケンシングのために企業に送ります。バイオインフォマティクス解析とシーケンシング結果の可視化を行い、放射線治療感受性および放射線治療抵抗性の遺伝子を得る26。
- データ解析のために複数の反復実験を設定し、実験結果に対して統計解析を実施して、データの一貫性と再現性を確保する27,28。
- シーケンシングデータの品質評価
- 生データを厳格なフィルタリングと品質管理メカニズムにさらして、シーケンシング情報の精度を保証します5.指定された変換アルゴリズムと、ベースコール28中にエラーが発生する可能性を評価するモデルを使用して、シーケンシングエラー率に基づいて各ヌクレオチドのPhredスコア(Qphred)を計算する。
- 各塩基位置のシーケンシングエラー率を 1% 未満(Q30 しきい値に相当)に維持し、シーケンシングデータの少なくとも 80% がこの Q30 標準を達成して、その後の分析手順をサポートします。
- データ処理とサンプルレベルの品質管理
(注)詳細は、以前に公開されたレポート29,30を参照されたい。- シーケンシングデータからのフィルタリングされたリードをsgRNAライブラリ配列にアラインメントします。ライブラリ内のsgRNAの数、sgRNAの平均存在量、検出されなかったsgRNAの数、sgRNAライブラリに正常にマッピングされた個々のサンプルのリードの割合などの統計を報告します。マッピング率を高くすると、カバレッジが大きくなります。
- 各サンプル中の各遺伝子(異なるsgRNAによって標的化される)のリードの濃縮をカウントします。MAGeCKの「中央値」正規化法を使用して、サンプル間で各sgRNAの支持リードを正規化します。sgRNAリードカウントの分布の評価、箱ひげ図の生成、主成分分析(PCA)の実施、相関ヒートマップの作成により、サンプルレベルの品質管理を行います。
- sgRNAのリードカウントがポアソン分布に従うと仮定します。異なるグループからの正規化されたsgRNAカウントを箱ひげ図に示して、サンプル内の全体的なデータ分布を視覚化し、グループ間の分布を比較します。
- PCAを使用すると、主成分間の違いと各成分内の変動率を反映することでデータ分析が簡素化され、グループ内およびグループ間の変動の観察が容易になります。相関ヒートマップを使用して、サンプル間の関係を示します。
- 基本的な分析
- グループ間での鑑別sgRNA解析を実施し、品質管理と予備データ処理後に必須遺伝子を同定します。関連遺伝子の機能濃縮解析を実施する30.MAGeCKまたはMLEなどの他の統計的手法によって計算されたロバストランクアグリゲーション(RRA)スコアに基づいて必須遺伝子をランク付けし、RRAスコアが低いほど重要性が高いことを示します。
- R言語または他のプログラミング言語を使用して、ランク付けされた結果に対してバイオインフォマティクス分析を実行し、GOおよびKEGG分析図30を使用して結果を視覚化する。遺伝子またはタンパク質を、IDマッピングまたは配列アノテーションを通じて、対応するGO用語(分子機能、生物学的プロセス、および細胞成分)にリンクします。
- KEGGをデータベースとして利用することで、高水準の機能や生命システムを理解し、同定した遺伝子カタログを細胞、種、生態系レベルでのシステム機能につなげることができます。GOおよびKEGG解析から上位10または20の経路を選択すると、経路の方向を直感的に表示できます。
結果
肺がんは、死亡率が最も高く、非常に侵攻性が高く、蔓延している医学的疾患です。肺がん細胞株A549を例に、放射線をスクリーニング条件としてゲノムワイドなCRISPRスクリーニングを実施すると、図 1に概略的なワークフローが示されています。まず、クローン形成とCCK8実験を通じて、さまざまな放射線量に対するA549細胞の感度を調査します(図2)。クローン作法では、コロニー数は2Gyで140±5.35であったのに対し、0Gyでは303±31.63であったのに対し、CCK-8アッセイでは、光学密度(OD)値は2Gyで0.65±0.05であったのに対し、0Gyでは1.35±0.08であった。放射線感受性および放射線抵抗性の候補遺伝子を調べるには、その後の放射線量として2Gy(IC50)を選択します。A549細胞を異なる用量のピューロマイシンで処理し、72時間後にカウントしました。A549細胞に対するピューロマイシンの最小死滅濃度は1μMとして検出されました(図3)。A549細胞は、異なる勾配MOIでレンチウイルスに感染しました。感染の72時間後、感染効率を蛍光顕微鏡で観察し、ピューロマイシン治療の72時間の前後にカウントしました。感染効率が~0.3のMOIが得られました(表1)。ヒトゲノムには19,050個の遺伝子が含まれており、各遺伝子に対応する6つのsgRNAとコピー数500(19,050 x 6 x 500 = 5.7 x 107 細胞)です。MOI = 0.3のレンチウイルスを使用すると、5.7 x 107 A549細胞に感染します。72時間後、細胞を1μMピューロマイシンでさらに72時間処理しました。Day 0のゲノムを採取し、PCR増幅を行った。CRISPR全ゲノムライブラリー内のsgRNA配列は231塩基対の長さを示しており、これはアガロースゲル電気泳動の結果によって裏付けられています(図4)。
その後、PCR産物はNovogeneに送られ、データ品質管理とサンプルレベルの品質管理が行われました。マッピング比率により、約60%の包括的なカバー率が得られましたが、これは、全ゲノムの処理中に非常に複雑で避けられない消耗を考慮すると、全ゲノムスクリーニング手順に適していると見なされる指標です(図5)。sgRNAのリードカウントはポアソン分布に準拠しており、理論的な期待に合致しています。その後のPCAプロットと相関ヒートマッピングによる分析では、異なるグループ間の識別可能な格差が鮮明に描かれており、グループ間の変動はグループ内の不一致を著しく上回っていました。さらに、グループ化されたサンプル内の変動率は許容範囲内に収まっており、サンプルレベルの品質管理対策の成功を実証しています。その後、MAGeCKによって考案されたRRAランキングは、R言語を利用したランキング結果の基本的なバイオインフォマティクス評価に着手するために利用されます。特に、GO用語の上位15の経路は、DNA損傷に関連するメカニズムを目立つように特徴としており、これは実験の基本的なスクリーニング基準とシームレスに一致しています。
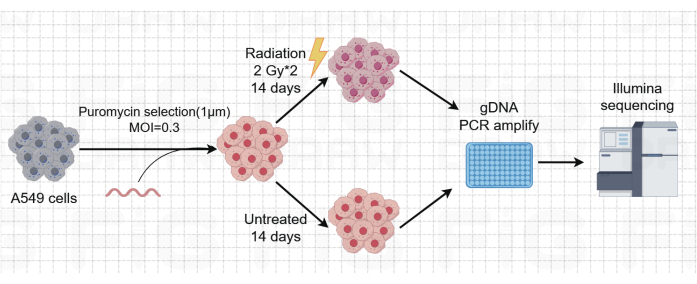
図1:A549肺がん細胞におけるゲノムワイド放射線治療CRISPR/Cas9スクリーニングの概略図。 ゲノムワイドCRISPR/Cas9と放射線療法を用いたA549細胞のスクリーニングワークフローの概略図。 この図の拡大版を表示するには、ここをクリックしてください。
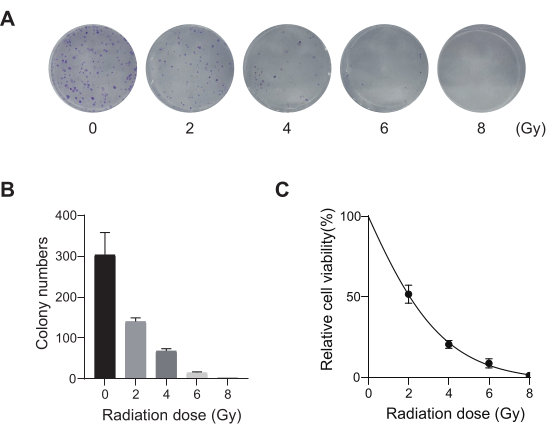
図2:A549細胞の放射線量の最適化 (A)A549細胞は、増加した放射線量(0 Gy、2 Gy、4 Gy、6 Gy、および8 Gy)に曝露され、14日間増殖させられました。コロニー形成アッセイの画像を示します。結果は、2つの独立した生物学的実験を代表するものです。(B)クローニングされたフォーメーションアッセイ画像の定量結果。エラーバーは標準偏差(n = 3)を表します。一元配置分散分析の結果は P < 0.01 でした。結果は、2つの独立した生物学的実験を代表するものです。(C)異なる放射線量への被ばく後のA549細胞の線量反応生存曲線。エラーバーは標準偏差(n = 3)を表します。一元配置分散分析の結果は 、P < 0.01でした。結果は、2つの独立した生物学的実験を代表するものです。 この図の拡大版を表示するには、ここをクリックしてください。
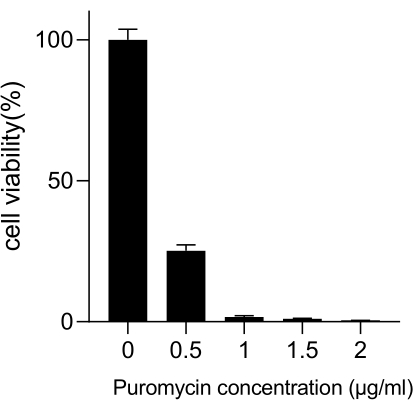
図3:A549細胞の選択に必要な最小ピューロマイシン濃度の決定。 A549細胞を高濃度のピューロマイシンで処理し、72時間インキュベートしました。エラーバーは標準偏差(n = 3)を表します。一元配置分散分析の結果は P < 0.01 でした。結果は、2つの独立した生物学的実験を代表するものです。 この図の拡大版を表示するには、ここをクリックしてください。
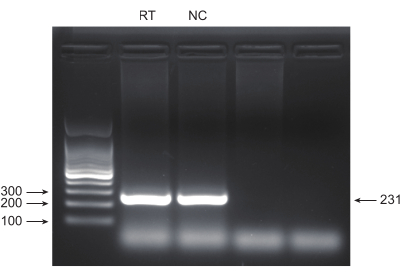
図4:アガロースゲル電気泳動によるPCR産物の検証。 アガロースゲル電気泳動を使用して、PCR産物バンドの存在と品質を調べました。 この図の拡大版を表示するには、ここをクリックしてください。
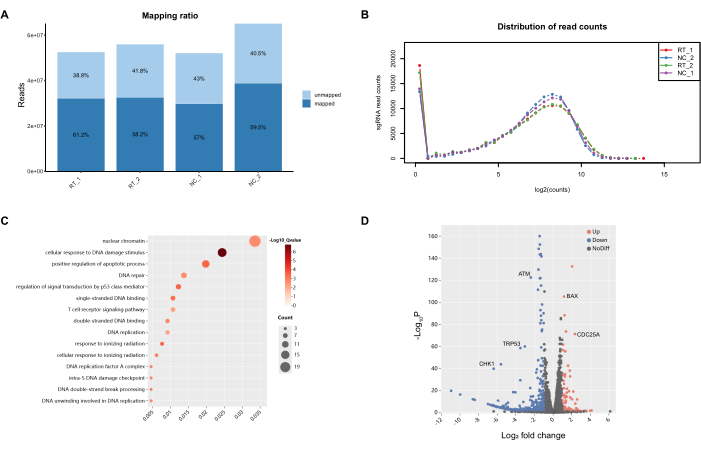
図5:PCR増幅製品の品質管理と基本分析。 (A)マッピング率は、参照ゲノムの約60%のカバレッジを示した。(B)sgRNAのリードカウント分布はポアソン分布に従った。(C)上位10のGene Ontology(GO)用語のほとんどがDNA損傷応答に関連していた。(D)遺伝子の濃縮パターンを示す火山プロットで、負の濃縮遺伝子は赤、正の濃縮遺伝子は青、有意でない遺伝子は灰色。 この図の拡大版を表示するには、ここをクリックしてください。
| ウイルス粒子 番号(VP) | カウント番号 (ピューロマイシン治療前) | カウント番号 (ピューロマイシン治療後) | 感染症 効率 |
| 1.23 × 104 | 1.23 × 105 | 1.04 × 104 | 0.084552846 |
| 3.72 × 104 | 1.24 × 106 | 3.61 × 104 | 0.291129032 |
| 6.15 × 104 | 1.23 × 107 | 5.4 × 104 | 0.43902439 |
表1:異なるウイルス粒子下でのピューロマイシン選択前後のA549細胞数。 さまざまなウイルス粒子下でのピューロマイシン処理の前後に記録されたA549細胞の数。
ディスカッション
最先端の遺伝子編集技術であるCRISPRスクリーニングは、科学研究の分野に大きな変化をもたらしました5。CRISPR-Cas9システムから生まれたこの技術は、その高い効率性と精度9により、遺伝子機能の研究に不可欠なツールとなっています9。CRISPR/Cas9の工学原理は、Cas9ヌクレアーゼが標的DNA配列を正確に特定して切断するように誘導するために、約20ヌクレオチドを持つ特定のsgRNAを設計および導入することを含み、ノックアウト、活性化、または抑制などの遺伝子編集を可能にします6。この原理は、ハイスループットでカスタマイズ可能なCRISPRスクリーニングの開発の根底にあります。膨大な数のsgRNAを含むライブラリを構築することで、研究者は複数の遺伝子を同時に編集およびスクリーニングし、特定の表現型や疾患状態に関連する遺伝子を迅速に特定できます。さらに、CRISPRスクリーニングでは、さまざまな細胞タイプ、組織、さらには生物全体での実験が可能となり、遺伝子の機能や作用機序をより包括的に理解することができます。
技術開発のスピードアップにより、CRISPRスクリーニングはさまざまな分野での応用に大きな可能性を示しています。例えば、CRISPRスクリーニングは、科学者が新規創薬標的を発見するのを支援し、それによって創薬と開発のプロセスを加速することができます。疾患研究では、疾患の発症メカニズムを解明し、診断や治療に新たな知見やアプローチを提供します。CRISPRスクリーニングは、従来のスクリーニング方法(RNAiや低分子阻害剤など)とはいくつかの重要な点で根本的に異なります:メカニズム的には、CRISPRは遺伝子編集を通じて恒久的な遺伝子改変を達成するのに対し、RNAiは遺伝子発現を一時的に抑制するだけで、低分子阻害剤はタンパク質機能を可逆的にブロックします28,29.特異性の面では、CRISPRは正確なDNAターゲティングを通じて優れた精度を示しますが、RNAiはオフターゲット効果を起こしやすい3,5。効果の持続期間に関しては、CRISPRは、従来の方法の一時的な介入と比較して、永続的な遺伝的変化を引き起こします。さらに、CRISPRはゲノムワイドなスクリーニング機能を可能にし、多様な細胞タイプで高い効率を維持しながら、複数の遺伝子/経路を同時に調査することができます9。従来のアプローチは通常、特定のパスウェイ解析に限定されており、特定の細胞株で有効性が低下することがよくあります3。これらの特徴により、CRISPRスクリーニングは機能ゲノミクス研究や標的探索、特に長期観察や正確な遺伝子調節を必要とする研究において特に価値があり、従来のスクリーニング手法に比べて独自の利点を発揮します。
しかし、このプロセスの特異性は絶対的なものではなく、Cas9タンパク質が、意図された標的配列5に類似しているが完全に同一ではないDNA領域を誤って認識し、切断する可能性がある。この現象は、一般にオフターゲット効果と呼ばれます。この意図しないDNA切断は、特に人間の治療に適用されると、ゲノムの不安定性と予測不可能な遺伝的結果につながる可能性があります。CRISPR技術は、複雑なゲノム編集タスクを処理する際にも苦労します。多数の遺伝子、遺伝子クラスター、または複雑な遺伝的設定を含む編集要求に直面した場合、CRISPRシステムは、編集プロセスの結果を正確に予測し、指示することに限界を感じる可能性があります1。
CRISPR技術を用いたゲノムワイドスクリーニングの実施における主な技術的ハードルは、数万の遺伝子を含むゲノムの非常に複雑なため、オフターゲット効果のリスクが大幅に増加することに起因しています9。その後のシーケンシング結果の完全性と信頼性を確保するためには、各細胞に単一のsgRNA23をトランスフェクションすることが不可欠です。そのためには、実験者がMOIを細心の注意を払って制御し、MOIが0.5未満に保たれるようにすると同時に、適切なコピー数の閾値(少なくとも500以上24)を維持する必要があります。全ゲノムの何万もの遺伝子のそれぞれが4〜6個のsgRNAの利用を必要とするという事実を考えると、全スクリーニングプロセスでは大量の細胞が必要となる25。実験の長期化、関与する細胞の数の多さ、およびリスクの上昇は、実験者がこれらの懸念を軽減するための戦略を考案することを必要とする26。潜在的な遺伝子は、RNA-seq、および機能獲得アッセイと機能喪失アッセイの両方でさらに検証する必要があります。以下の実験では、CRISPR編集を用いて同定された遺伝子の過剰発現およびノックアウト細胞を取得した後、ゲノムワイドゲノミクスおよびトランスクリプトミクスを実施して、特に非特異的Cas9切断を受ける可能性のある部分的なsgRNA相補性を有する遺伝子座における潜在的なオフターゲット変化にアクセスした。COSMIDのようなバイオインフォマティクス予測ツールは、高リスクのオフターゲット部位の特定に役立つ可能性があります。レスキューアッセイは、同定された遺伝子の関与の特異性を確保するためにも使用できます。
その後の解析では、差次的に発現するsgRNAの基準は、MAGeCKによって計算されたロバストランクアグリゲーション(RRA)スコアに依存しており、これには統計的有意性、フォールド変化、および複製の一貫性が含まれていた31,32。データセットの場合、各遺伝子は、遺伝子発現レベルと統計的有意性を組み合わせた包括的な基準に従ってランク付けされます。RRAでは、Spearmanのランク相関係数を用いて、すべてのデータセットにおける各遺伝子の網羅的なランクの状況を評価し、複数の遺伝子発現データセットのソーティング結果を統合します。ほとんどのデータセットで比較的高いランクの遺伝子は、真の差次的に発現する遺伝子と見なすことができる33。0.05未満のRRAのp値は、統計的に有意であると考えられた。少なくとも1.5倍の増加または33%の減少の倍率変化は、異なる表現と見なされました。さらに、3つの独立した反復の一貫性は、統計的に異なる表現と見なす必要がありました。必須遺伝子のRRAランキングの後、RRAスコアが低いほど重要性が高いことを意味します。
ゲノム編集の強力なツールとして、CRISPR-Cas9技術は、放射線耐性の領域を調査する上で比類のない可能性と精度を示しています26,27。ただし、そのアプリケーションにはまだ制限があります。オフターゲット効果のリスクは別として、放射線感受性に関連する複雑さは、スクリーニングプロセスに複雑さをもたらします27。この感受性は、単に孤立した遺伝子によって支配されているのではなく、むしろ複数の遺伝子とその調節ネットワークとの間の複雑な相互作用の結果として現れる26。したがって、放射線感受性に関連する特定の遺伝子の同定が成功したにもかかわらず、それらは全体像のほんの一部しか表していない可能性があり、根底にあるメカニズムを包括的に解明するための大きな課題を提起している34,35,36。ここでは、感染効率の低さ、sgRNAの分解、シーケンシングの失敗など、CRISPRスクリーニングの一般的な問題に対する解決策を示します:CRISPRスクリーニングではMOI値<0.5が必要なため、細胞の状態調整は、細胞が対数成長段階にあることを確認したり、蛍光アッセイを介して感染効率を評価したりするために重要です。sgRNAの分解の潜在的な原因には、ヌクレアーゼ汚染やベクター設計上の欠陥などがあるため、sgRNAライブラリの凍結融解サイクルの繰り返しを避け(アリコートを-80°Cで保存)、NGSシーケンシングを使用してsgRNAライブラリの完全性を検証する必要があります。シーケンシングの失敗は、シーケンシング深度が不十分であるか、またはsgRNAプライマー設計の問題に起因する可能性があるため、sgRNA配列の同定精度を向上させるためにペアエンドシーケンシングが推奨され、推奨シーケンシング深度は>500×カバレッジ37,38,39,40,41,42です。
放射線療法は依然としてがん治療の基礎ですが、異なる腫瘍タイプ間、さらには同じがん患者間でも治療効果に顕著なばらつきがあります9。この個人間の不均一性は、最適な腫瘍根絶を達成するために併用療法を頻繁に必要とします11,12。臨床診療では、放射線療法と免疫療法(特に免疫チェックポイント阻害剤および抗体薬物複合体)の統合が、顕著な治療パラダイムとして浮上しています19。このプロトコルは、比較的均質ながん細胞株を用いたin vitro CRISPRスクリーニングを提供しますが、これはin vivoでの腫瘍の遺伝的およびエピジェネティックな不均一性を完全に反映することはできませんでした。スクリーニングのための適切な放射線量を決定した後、その結果は潜在的な放射線感受性または放射線耐性遺伝子を提供しますが、これはin vitro細胞アッセイとin vivo動物モデルの両方でさらに確認する必要があります。さらに、in vivo CRISPRスクリーニングとシングルセルトランスクリプトミクスの組み合わせは、腫瘍の不均一性の複雑さを解読するのに役立つ可能性があります。in vitro CRISPRスクリーニングでは、細胞内のDNA損傷修復や放射線に応答した自己分泌メカニズムに重要な遺伝子が同定されましたが、実際にはTMEの不均一性を完全には再現しませんでした。例えば、低酸素症はROS産生を増加させ、代謝適応、抗酸化生成、オートファジー活性化を刺激するフィードバックループを引き起こし、最終的には放射線耐性を促進します18。放射線照射後、パラクリン機構で免疫細胞が活性化され、放射線感受性が誘導される可能性がある14。ECMは成長因子の貯蔵庫として機能し、サイトカインは放射線感受性にプラスとマイナスの両方の影響を及ぼします21。in vivo CRISPRスクリーニングは、TMEの不均一性を反映するために実施する必要があります。さらに、in vitro CRISPRスクリーニングは、細胞内のDNA損傷修復や放射線に応答する自己分泌メカニズムの重要な遺伝子のみを特定できましたが、腫瘍の血管系や全身への影響など、放射線感受性に対するin vivoの影響を完全には捉えていませんでした20,21。in vivoでの放射線感受性に関与する可能性のある因子を特定するために、in vivo CRISPRスクリーニングを実施する必要があります。
ここでは、CRISPRゲノムワイドスクリーニングの利用を目的とした包括的なプロトコルが考案され、放射線療法に対する感受性または耐性のいずれかに関与する遺伝子を特定します。この方法論は、新しい経路を解明することにより、腫瘍放射線治療研究の進歩への道を開くことを目指しています。このプロトコルは、化学療法における臨床的制約のメカニズムと代替治療薬の進行を調査するための参照としても役立つ可能性があります。要約すると、CRISPR技術は大きな可能性と価値を体現していますが、その固有の制約は、冷静な認識と賢明なアプローチの重要性を強調しています。将来の研究と応用が探求されるにつれて、CRISPR技術の制約を克服し、その可能性を最大限に引き出すために、CRISPR技術の探求と最適化に絶え間なく努力することが不可欠です。
開示事項
何一つ。
謝辞
本研究は、湖北省地域科学技術イノベーションプロジェクト(2024EIA001)および武漢大学中南病院医学科学技術イノベーションプラットフォーム構築支援プロジェクト(PTXM2025001)の支援を受けて行われました。 図 1 は Figdraw を使用して作成されました。
資料
| Name | Company | Catalog Number | Comments |
| 150 mm cell and tissue culture dish | Wuxi NEST Biotechnology Co., Ltd. | 715011 | Cell culture |
| 35 mm cell and tissue culture dish | Wuxi NEST Biotechnology Co., Ltd. | 706011 | Cell culture |
| A549 cell line | ATCC | - | - |
| Cell counting kit-8 | Shanghai Beyotime Biotech Inc | C0041 | For cell viability assay |
| CO2-independent medium | PHCbi | MCO-50AIC | Cell culture |
| Countess 3 FL automated cell counter | Themo Scientific | AMQAF2001 | For cell counting |
| EDTA | Gibco | 25200056 | Cell culture |
| Fetal bovine serum | Gibco | 10099141 | Cell culture |
| Fluorescence microscopy | LEICA | ebq 100-04 | For fluorescence microscope |
| Genome-wide CRISPR lentiviral library | Shanghai OBiO Technology Co., Ltd. | H5070 | For lentiviral infection |
| MiniAmp plus PCR | Themo Scientific | C37835 | For PCR amplification |
| NEST cell culture plates, 12-well | Wuxi NEST Biotechnology Co., Ltd. | 712002 | Cell culture |
| NEST cell culture plates, 6-well | Wuxi NEST Biotechnology Co., Ltd. | 703002 | Cell culture |
| NEST cell culture plates, 96-well | Wuxi NEST Biotechnology Co., Ltd. | 701001 | Cell culture |
| PBS | Gibco | C10010500BT | Cell culture |
| PCR forward primer (AATGGACTATCATATGCTTACCGTAACTTGAAAGTATTTCG) | Beijing AuGCT Biotech Co., Ltd. | - | For PCR amplification |
| PCR reverse primer (GATGTGCGCTCTGCCCACTGACGGGCA) | Beijing AuGCT Biotech Co., Ltd. | - | For PCR amplification |
| Polybrene | Shanghai Beyotime Biotech Inc | C0351 | For lentiviral infection |
| Puromycin | MCE | HY-K1057 | For seletion post lentiviral infection |
| RPMI 1640 cell culture medium | Gibco | 23400-021 | Cell culture |
| TIANGENamp genomic DNA kit | Beijing TIANGEN Biotech Co.,Ltd. | DP304 | For genomic DNA extraction |
| X-ray powder diffractometer | PerkinElmer | For radiotherapy |
参考文献
- Barrangou, R., Marraffini, L. A. CRISPR-Cas systems: Prokaryotes upgrade to adaptive immunity. Mol Cell. 54 (2), 234-244 (2014).
- Joung, J., et al. Genome-scale CRISPR-Cas9 knockout and transcriptional activation screening. Nat Protoc. 12 (4), 828-863 (2017).
- Sanjana, N. E., Shalem, O., Zhang, F. Improved vectors and genome-wide libraries for CRISPR screening. Nat Methods. 11 (8), 783-784 (2014).
- Arroyo, J. D., et al. A Genome-wide CRISPR death screen identifies genes essential for oxidative phosphorylation. Cell Metab. 24 (6), 875-885 (2016).
- Sidik, S. M., et al. A genome-wide CRISPR screen in toxoplasma identifies essential apicomplexan genes. Cell. 166 (6), 1423-1435 (2016).
- Jing, Y., et al. Genome-wide CRISPR activation screening in senescent cells reveals SOX5 as a driver and therapeutic target of rejuvenation. Cell Stem Cell. 30 (11), 1452-1471 (2023).
- Shifrut, E., et al. Genome-wide CRISPR screens in primary human t cells reveal key regulators of immune function. Cell. 175 (7), 1958-1971 (2018).
- Shalem, O., et al. Genome-scale CRISPR-Cas9 knockout screening in human cells. Science. 343 (6166), 84-87 (2014).
- Katti, A., Diaz, B. J., Caragine, C. M., Sanjana, N. E., Dow, L. E. CRISPR in cancer biology and therapy. Nat Rev Cancer. 22 (5), 259-279 (2022).
- Sasmal, R., et al. Synthetic host-guest assembly in cells and tissues: Fast, stable, and selective bioorthogonal imaging via molecular recognition. Anal Chem. 90 (19), 11305-11314 (2018).
- Roos, W. P., Thomas, A. D., Kaina, B. DNA damage and the balance between survival and death in cancer biology. Nat Rev Cancer. 16 (1), 20-33 (2016).
- Huang, R., Zhou, P. K. DNA damage repair: Historical perspectives, mechanistic pathways and clinical translation for targeted cancer therapy. Signal Transduct Target Ther. 6 (1), 254(2021).
- O'Connor, M. J. Targeting the DNA damage response in cancer. Mol Cell. 60 (4), 547-560 (2015).
- Lord, C. J., Ashworth, A. The DNA damage response and cancer therapy. Nature. 481 (7381), 287-294 (2012).
- Carusillo, A., Mussolino, C. DNA Damage: From threat to treatment. Cells. 9 (7), 1665(2020).
- Li, X., Heyer, W. D. Homologous recombination in DNA repair and DNA damage tolerance. Cell Res. 18 (1), 99-113 (2008).
- Chang, H., Pannunzio, N. R., Adachi, N., Lieber, M. R. Non-homologous DNA end joining and alternative pathways to double-strand break repair. Nat Rev Mol Cell Biol. 18 (8), 495-506 (2017).
- Andratschke, N., et al. European society for radiotherapy and oncology and European organization for research and treatment of cancer consensus on re-irradiation: Definition, reporting, and clinical decision making. Lancet Oncol. 23 (10), e469-e478 (2022).
- Olivares-Urbano, M. A., Grinan-Lison, C., Marchal, J. A., Nunez, M. I. CSC radioresistance: A therapeutic challenge to improve radiotherapy effectiveness in cancer. Cells. 9 (7), 1651(2020).
- Schaue, D., McBride, W. H. Opportunities and challenges of radiotherapy for treating cancer. Nat Rev Clin Oncol. 12 (9), 527-540 (2015).
- Allen, C., Her, S., Jaffray, D. A. Radiotherapy for cancer: Present and future. Adv Drug Deliv Rev. 109, 1-2 (2017).
- Xue, C., Greene, E. C. DNA repair pathway choices in CRISPR-Cas9-Mediated genome editing. Trends Genet. 37 (7), 639-656 (2021).
- Tzelepis, K., et al. A CRISPR dropout screen identifies genetic vulnerabilities and therapeutic targets in acute myeloid leukemia. Cell Rep. 17 (4), 1193-1205 (2016).
- Rottner, A. K., et al. A genome-wide CRISPR screen identifies CALCOCO2 as a regulator of beta cell function influencing type 2 diabetes risk. Nat Genet. 55 (1), 54-65 (2023).
- Chen, M., et al. CRISPR-Cas9 for cancer therapy: Opportunities and challenges. Cancer Lett. 447, 48-55 (2019).
- Gupta, D., et al. CRISPR-Cas9 system: A new-fangled dawn in gene editing. Life Sci. 232, 116636(2019).
- Li, B., et al. Genome-wide CRISPR screen identifies host dependency factors for influenza A virus infection. Nat Commun. 11 (1), 164(2020).
- Hung, K. L., et al. Targeted profiling of human extrachromosomal DNA by CRISPR-CATCH. Nat Genet. 54 (11), 1746-1754 (2022).
- Bhattacharya, D., Van Meir, E. G. A simple genotyping method to detect small CRISPR-Cas9 induced indels by agarose gel electrophoresis. Sci Rep. 9 (1), 4437(2019).
- Razavi, Z., Soltani, M., Souri, M., Van Wijnen, A. J. CRISPR innovations in tissue engineering and gene editing. Life Sci. 358, 123120(2024).
- Wang, W., et al. A genome-wide CRISPR-based screen identifies KAT7 as a driver of cellular senescence. Sci Transl Med. 13 (575), eabd2655(2021).
- Xu, X., et al. Engineered miniature CRISPR-Cas system for mammalian genome regulation and editing. Mol Cell. 81 (20), 4333-4345 (2021).
- Huang, M., et al. FACS-based genome-wide CRISPR screens define key regulators of DNA damage signaling pathways. Mol Cell. 83 (15), 2810-2828 (2023).
- Feng, J., et al. Genome-wide CRISPR screen identifies synthetic lethality between DOCK1 inhibition and metformin in liver cancer. Protein Cell. 13 (11), 825-841 (2022).
- Saurat, N., et al. Genome-wide CRISPR screen identifies neddylation as a regulator of neuronal aging and AD neurodegeneration. Cell Stem Cell. 31 (8), 1162-1174 (2024).
- Du, Y., Liu, Y., Hu, J., Peng, X., Liu, Z. CRISPR/Cas9 systems: Delivery technologies and biomedical applications. Asian J Pharm Sci. 18 (6), 100854(2023).
- Shi, J., et al. A genome-wide CRISPR screen identifies WDFY3 as a regulator of macrophage efferocytosis. Nat Commun. 13 (1), 7929(2022).
- Liu, N., Olson, E. N. CRISPR modeling and correction of cardiovascular disease. Circ Res. 130, (2022).
- Feng, X., et al. Genome-wide CRISPR screens using isogenic cells reveal vulnerabilities conferred by loss of tumor suppressors. Sci Adv. 8 (19), 6638(2022).
- Ghaemi, A., et al. CRISPR-Cas9 genome editing delivery systems for targeted cancer therapy. Life Sci. 267, 118969(2021).
- Derry, W. B. CRISPR: Development of a technology and its applications. FEBS J. 288 (2), 358-359 (2021).
- Sharma, G., Sharma, A. R., Bhattacharya, M., Lee, S. S., Chakraborty, C. CRISPR-Cas9: A preclinical and clinical perspective for the treatment of human diseases. Mol Ther. 29 (2), 571-586 (2021).
転載および許可
このJoVE論文のテキスト又は図を再利用するための許可を申請します
許可を申請さらに記事を探す
This article has been published
Video Coming Soon
Copyright © 2023 MyJoVE Corporation. All rights reserved