É necessária uma assinatura da JoVE para visualizar este conteúdo. Faça login ou comece sua avaliação gratuita.
Method Article
Gating cardíaco retrospectivo com um protótipo de tomógrafo computadorizado de raios-X de pequenos animais
Neste Artigo
Resumo
Fornecemos uma descrição abrangente do método de gating cardíaco retrospectivo intrínseco do CrumpCAT, um protótipo de tomografia computadorizada (TC) de raios-X de pequenos animais projetado e construído em nossa instituição de pesquisa.
Resumo
O CrumpCAT é um protótipo de tomografia computadorizada (TC) de raios-X de pequenos animais desenvolvido em nossa instituição de pesquisa. O detector CMOS com uma taxa de quadros máxima de 29 Hz e fontes semelhantes de raios-X de tungstênio com energias variando de 50 kVp a 80 kVp são amplamente utilizados em instrumentos de TC de raios-X pré-clínicos disponíveis comercialmente. Isso torna o trabalho descrito altamente relevante para outras instituições, apesar da sabedoria geralmente percebida de que esses detectores não são adequados para controlar as altas frequências cardíacas de camundongos (~ 600 batimentos / min). O scanner apresenta imagens de resolução média (200 μm) e alta (125 μm), fluoroscopia, gating respiratório retrospectivo e gating cardíaco retrospectivo, com reconstrução de imagem de projeção iterativa ou filtrada de volta. Entre essas características, o gating cardíaco é o recurso mais útil para estudar as funções cardíacas in vivo, pois elimina efetivamente o desfoque da imagem causado pelo movimento respiratório e cardíaco.
Aqui, descrevemos nosso método para imagens pré-clínicas de TC cardíaca retrospectiva intrínseca, com o objetivo de avançar na pesquisa sobre função cardíaca in vivo e análise de estrutura. O método de ativação cardíaca adquire um grande número de projeções no menor tempo de exposição prática (~ 20 ms) e, em seguida, extrai retrospectivamente sinais respiratórios e cardíacos de mudanças temporais nas sequências de projeção brutas. Esses sinais são usados para rejeitar projeções pertencentes à fase de inspiração de alta taxa de movimento do ciclo respiratório e para dividir as projeções restantes em 12 grupos, cada um correspondendo a uma fase do ciclo cardíaco. Cada grupo é reconstruído independentemente usando um método iterativo para produzir uma imagem volumétrica para cada fase cardíaca, resultando em um conjunto de dados quadridimensional (4D).
Essas imagens de fase podem ser analisadas coletivamente ou individualmente, permitindo uma avaliação detalhada da função cardíaca. Demonstramos a eficácia de ambas as abordagens do recurso de ativação cardíaca do protótipo do scanner por meio de resultados de imagem in vivo representativos.
Introdução
A pesquisa com pequenos animais geralmente emprega uma combinação de modalidades de imagem não invasivas, sendo a tomografia computadorizada (TC) de raios X, uma escolha de destaque devido à sua maturidade, custo-efetividade, velocidade 1,2 e capacidade de fornecer informações complementares juntamente com outras modalidades, como tomografia por emissão de pósitrons (PET)2,3 e tomografia computadorizada por emissão de fóton único (SPECT)2,4. No entanto, como outras técnicas de imagem, a TC é suscetível a artefatos de movimento fisiológico causados pelo coração batendo ou pela respiração, que introduzem o desfoque e limitam a precisão da pesquisa.
Para resolver essa limitação, o desfoque do movimento respiratório e cardíaco pode ser mitigado por meio de uma técnica conhecida como gating 5,6,7,8, onde a aquisição de dados é sincronizada com fases específicas do ciclo cardíaco ou respiratório (ou portões). Uma abordagem para conseguir isso, conhecida como gating prospectivo 3,6, envolve a conexão de sensores ao animal para fornecer sinais de gating em tempo real a um scanner compatível. Embora eficaz, esse método é trabalhoso e demorado, principalmente ao conectar sensores ao tórax e às patas de pequenos animais como camundongos, limitando assim a escala dos estudos. Alternativamente, o gating retrospectivo intrínseco 7,9,10,11 envolve a aquisição de dados de séries temporais sem o uso de sensores, mas identificando recursos nos dados que permitem a classificação retrospectiva dos resultados com base em sua fase no ciclo cardíaco ou respiratório. Essa abordagem oferece resultados comparáveis ao gating prospectivo, mas sem a necessidade de hardware adicional ou o esforço envolvido na conexão do sensor de pulso e, portanto, simplifica muito os protocolos experimentais.
Em nosso método de tomografia computadorizada cardíaca pré-clínica, utilizamos gating retrospectivo intrínseco para extrair ciclos respiratórios e cardíacos de variações de amplitude em regiões em projeções de raios-X que exibem as mudanças mais significativas entre quadros sucessivos. Para facilitar esse processo, um modelo de tórax de camundongo é co-registrado na primeira projeção posteroanterior usando Informações Mútuas12. Uma vez que o modelo está no lugar, as intensidades de pixel em uma janela próxima ao diafragma são somadas para gerar um sinal respiratório substituto, enquanto aquelas próximas ao miocárdio são somadas para derivar o sinal cardíaco substituto. Esses sinais são então filtrados passa-banda no domínio do tempo, e cada quadro no conjunto de dados recebe um número de fase fracionário (entre 0 e 1) com base em sua fase respiratória e cardíaca. Isso permite a seleção ou rejeição de projeções de acordo com seus valores de fase. Normalmente, os quadros correspondentes à fase final de expiração do ciclo respiratório (0,15 ≤ fase < 0,85) são retidos, enquanto os da fase de inspiração, onde o movimento é mais pronunciado, são descartados. Os quadros restantes são agrupados em 12 fases cardíacas, cada uma representando 1/12 (0,083) do ciclo cardíaco e são reconstruídos em imagens 3D usando um método iterativo (Ordered Subset Expectation Maximization [OSEM])13,14. Todo o processo está resumido na Figura 1.
Protocolo
Os protocolos experimentais em animais foram revisados e aprovados pelo Comitê Institucional de Cuidados e Uso de Animais da Universidade da Califórnia, Los Angeles (UCLA). Camundongos C57BL/6J (8 semanas de idade, machos, 24-26 g) foram usados neste protocolo. O tomógrafo utilizado neste estudo é o CrumpCAT (Figura 2), um protótipo desenvolvido em nossa instituição de pesquisa para pesquisa pré-clínica, que nos fornece o controle e a flexibilidade necessários para otimizar os protocolos de aquisição e reconstrução. O método pressupõe que os camundongos anestesiados terão uma frequência cardíaca não superior a 600 batimentos/min e uma frequência respiratória entre 20 e 180 respirações/min15.
1. Configurações do equipamento
- Opere a fonte de raios X em uma tensão de pico de 50 kVp com corrente contínua de 200 μA.
- Defina o binning de pixels da câmera de raios-X como 2 e o tempo de exposição para seu valor prático mais curto (tempo de exposição de 20 ms mais 14 ms de tempo de leitura para um tempo total de amostragem de 34 ms).
NOTA: A opção no-binning (binning 1) tem uma taxa de quadros mais lenta e, portanto, não pode ser usada para aquisições fechadas. A taxa de quadros máxima é de cerca de 30 quadros/s.
2. Preparação animal
- Anestesiar camundongos com uma mistura de oxigênio e gás isoflurano na concentração de 2,0%.
- Mantenha os animais sob anestesia por 10 minutos antes da tomografia computadorizada, para garantir que os sinais vitais estejam estáveis e que os animais estejam relaxados para evitar qualquer movimento corporal durante o exame. Julgue a profundidade da anestesia de um camundongo pela falta do reflexo de pinça do dedo do pé ao beliscar firmemente a correia entre os dedos dos pés com a unha.
- Para que a imagem cardíaca visualize as câmaras cardíacas, injete por via intravenosa 100 μL de agente de contraste de TC através da veia da cauda imediatamente antes da imagem de TC (Figura 3A).
- Coloque o camundongo anestesiado dentro da câmara de imagem da TC (Figura 3B).
3. Aquisição de dados
- Ligue o software CrumpCAT.
- Imagem de TC não fechada com alta resolução (125 μm, binning 1) (Figura 4A)
- Insira um ID de estudo na interface do usuário.
- Selecione Mouse Hi-Res no menu suspenso Protocolo .
- Clique no botão Digitalizar na interface do usuário para adquirir 720 projeções com um tempo de exposição de 80 ms/projeção.
- Imagem de TC não fechada com resolução média (200 μm, binning 2) (Figura 4B)
- Insira um ID de estudo na interface do usuário.
- Selecione Padrão do mouse no menu suspenso Protocolo .
- Clique no botão Digitalizar na interface do usuário para adquirir 720 projeções com um tempo de exposição de 100 ms/projeção.
- Imagem de TC fechada (200 μm, binning 2) (Figura 4C)
- Insira um ID de estudo na interface do usuário.
- Selecione Cardiac Gating no menu suspenso Protocolo .
- Clique no botão Digitalizar na interface do usuário e adquira 21.600 projeções com um tempo de exposição de 20 ms/projeção.
4. Pré-processamento de dados
NOTA: As etapas de pré-processamento são necessárias apenas para aquisições fechadas. Todas essas etapas são executadas automaticamente pelo software de reconstrução e nenhuma intervenção do operador é necessária.
- Extração de sinal
- Para o co-registro do modelo de tórax, deixe uma pequena imagem (modelo) representando grosseiramente um tórax de camundongo com as costelas, o coração, os pulmões e o fígado ser automaticamente co-registrada na primeira projeção de raios-X, maximizando a Informação Mútua12 entre a projeção e o modelo (Figura 5).
NOTA: Somente operações de conversão são executadas no modelo e o modelo co-registrado é usado para identificar regiões de interesse (ROIs) em todas as projeções. - Para extração de sinal respiratório, permita que uma ROI retangular (ROI-1) seja designada no modelo para representar o diafragma e o sinal respiratório a ser gerado somando as intensidades de pixel dentro da ROI-1 para cada projeção (Figura 5).
- Para extração de sinal cardíaco, deixe um segundo retângulo ROI (ROI-2) ser designado no modelo próximo ao coração, e o sinal cardíaco gerado pela soma das intensidades de pixel em ROI-2 para cada projeção (Figura 5).
NOTA: O modelo de tórax é co-registrado apenas na primeira projeção para identificar quais linhas e colunas de imagem (janela) devem ser usadas para extração de sinal. Essa janela é larga o suficiente para abranger todas as projeções, desde que o animal esteja razoavelmente centralizado, como é o caso ao usar nossa cama de animais16.
- Para o co-registro do modelo de tórax, deixe uma pequena imagem (modelo) representando grosseiramente um tórax de camundongo com as costelas, o coração, os pulmões e o fígado ser automaticamente co-registrada na primeira projeção de raios-X, maximizando a Informação Mútua12 entre a projeção e o modelo (Figura 5).
- Filtro passa-banda
- Permitir que um filtro passa-banda seja gerado combinando duas funções Sinc com diferentes frequências de corte no domínio temporal17, que correspondem a filtros passa-baixa no domínio da frequência, conforme descrito abaixo.
- Deixe o software aplicar o filtro passa-banda no sinal cardíaco usando uma banda de frequência de [300, 600] min-1 (Figura 6A).
NOTA: O sinal cardíaco não filtrado (Figura 6B) ainda contém uma contribuição significativa do movimento respiratório. - Da mesma forma, deixe o sinal respiratório ser filtrado usando uma banda de frequência de [20, 300] min-1. Os harmônicos mais altos são preservados para produzir um sinal que não é simplesmente senoidal e identifica mais claramente as duas fases principais: inspiração e expiração.
NOTA: Neste ponto, os sinais respiratórios e cardíacos podem ser facilmente interpolados e considerados funções contínuas de tempo para fins de cálculo do número de fases fracionárias.
- Atribuição de fase
- Deixe o software identificar o início de cada ciclo (respiratório ou cardíaco) encontrando os cruzamentos zero da primeira derivada do sinal. Cada cruzamento zero corresponde a um máximo no sinal e marca o início de um novo ciclo e o fim do anterior. Por exemplo, veja o sinal respiratório na Figura 7.
- Permitir que um valor de fase fracionário (entre 0 e 1) seja atribuído a cada projeção para cada um dos dois sinais (respiratório ou cardíaco).
NOTA: Por definição, o ponto inicial tem um valor de fase fracionário de zero (para o ciclo atual) ou um (para o ciclo anterior).
- Máscaras de seleção
- Que 12 máscaras binárias (uma por fase cardíaca) sejam criadas para selecionar projeções pertencentes a cada fase. Cada máscara contém 21.600 entradas (uma por projeção) que são 0 ou 1, o que significa rejeitar ou manter essa projeção, respectivamente.
- Para cada máscara (fase), observe que o software indica se uma projeção é mantida (1) ou descartada (0) de acordo com seu número de fase fracionário. Para a fase 0, as projeções com um valor de fase cardíaca no intervalo [0, 1/12] são mantidas. Para a fase 1, os valores da fase cardíaca no intervalo [1/12, 2/12] são mantidos e assim por diante.
- Para cada máscara (fase), observe que quaisquer projeções com fase respiratória menor que 0,15 ou maior que 0,85 são rejeitadas (0 na máscara) porque pertencem à fase de inspiração, que tem o maior movimento (Figura 8A). A Figura 8B mostra a atribuição da fase cardíaca às projeções para os primeiros 2 s.
NOTA: Nenhum viés significativo foi introduzido com este processo de seleção, uma vez que o número de projeções por fase foi relativamente constante em 1.800 ± 194.
5. Reconstrução de imagem
- Para tomografias computadorizadas não fechadas, reconstrua imagens em resolução média (200 μm) ou alta (125 μm) usando o algoritmo de retroprojeção filtrada (Feldkamp18) com um filtro Shepp-Logan.
- Para tomografias computadorizadas cardíacas, reconstrua cada imagem da fase cardíaca usando o algoritmo OSEM iterativo13, com 12 subconjuntos e 8 iterações, levando em consideração apenas as projeções selecionadas que contribuem para uma determinada fase (conforme indicado em uma máscara binária de seleção de fase).
- Para reduzir o ruído, aplique um filtro mediano pós-reconstrução na dimensão temporal (ou seja, através das imagens) e um filtro 3D-Gaussiano leve na dimensão espacial (σ = 38 μm).
NOTA: Para reconstrução de imagem com controle respiratório, use todas as fases cardíacas indistintamente. A correção de endurecimento do feixe (correção de água) é aplicada a cada reconstrução.
6. Avaliação por imagem e quantificação do volume do ventrículo esquerdo (VE)
- Abra a imagem da TC em um visualizador DICOM, como a Amida19 (Figura 9A).
- Melhore o contraste visível da imagem definindo o intervalo do valor CT (medido na Unidade Hounsfield [HU]) como [-500, 500] (Figura 9A).
- Avaliação da qualidade da imagem
- Desenhe um ROI para a região calcificada, que é definida como a região em torno do máximo com um valor de CT maior ou igual a 85% do valor máximo de HU (Figura 9B).
- Utilizar a relação contraste-ruído (CNR)20,21 como métrica para avaliar a qualidade da imagem e sua capacidade de identificar pequenas estruturas, como pequenas calcificações:
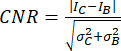 ,
,
onde I e σ representam a intensidade média e o desvio padrão de uma região: calcificação (subscrito C) ou background (subscrito B).
- Quantificação do volume do ventrículo esquerdo
- Desenhe um ROI 3D à mão livre para identificar o VE em cada fase (Figura 10A).
- Quantifique o volume do VE calculando voxels com um valor de CT limiar de 730 UH (Figura 10B). Um gráfico dos volumes do VE é mostrado na Figura 10C.
NOTA: O limite do valor CT não é absoluto; depende do agente de contraste usado, tamanho do animal, cepa, condição de saúde e tempo entre a injeção e a tomografia computadorizada. Amide é um software distribuído gratuitamente desenvolvido em nosso instituto, mas outros visualizadores de imagens mais sofisticados estão disponíveis (por exemplo, ORS Dragonfly22).
Resultados
Primeiro, comparamos imagens de TC não fechadas e fechadas para visualização de calcificação cardíaca em camundongos (machos, 30-32 g). O modelo murino de calcificação cardíaca foi criado pela indução de lesão cardíaca por congelamento e descongelamento rápido do tecido cardíaco (criolesão), conforme descrito anteriormente23. Com os protocolos de imagem de TC não fechados, as calcificações cardíacas foram mais claramente identificadas na imagem...
Discussão
A implementação de hardware específica descrita aqui é um sistema de TC de raios-X feito sob medida exclusivo para nosso instituto, mas o detector específico é amplamente utilizado em instrumentos de TC de raios-X pré-clínicos disponíveis comercialmente, tornando o trabalho descrito relevante para outras instituições. Este sistema é funcionalmente o protótipo de dois subsistemas microCT de raios-X disponíveis comercialmente e amplamente utilizados in vivo incorpora...
Divulgações
O Dr. Richard Taschereau é consultor da Sofie Biosciences e da Xodus Imaging. O Dr. Arion F. Chatziioannou é um dos fundadores da Sofie Biosciences.
Agradecimentos
Agradecemos a todos os membros do Centro de Tecnologia de Imagens Pré-clínicas Crump da UCLA por sua ajuda e apoio. Em particular, agradecemos a Mikayla Tamboline e Isabel Day por prepararem os animais para imagens de TC cardíaca e agradecemos a Sophie Shumilov por gerar algumas das ROIs do ventrículo esquerdo durante o estudo. Também agradecemos aos Drs. Arjun Deb e Yijie Wang (UCLA) por fornecerem os modelos murinos de lesão cardíaca isquêmica aguda para imagens de microCT de calcificação cardíaca. Este trabalho é apoiado pelo NIH Cancer Center Support Grant (2 P30 CA016042-44).
Materiais
| Name | Company | Catalog Number | Comments |
| C57BL/6J mice | Jackson Laboratory | 664 | Male, 8 weeks old, 24-26 g |
| Dexela camera | Varex | 1512 | Detector, 20 ms exposure, 74.8/149.6 µm pixel |
| VivoVist | Nanoprobes | 1301-5X0.25ML | CT Contrast agent |
| X-ray source | Moxtek | TUB00082 | 50 kV peak, 200 µA, 1.0 mm-thick Al filter |
Referências
- Schambach, S. J., Bag, S., Schilling, L., Groden, C., Brockmann, M. A. Application of micro-CT in small animal imaging. Methods. 50 (1), 2-13 (2010).
- Koba, W., Jelicks, L. A., Fine, E. J. MicroPET/SPECT/CT imaging of small animal models of disease. Am J Pathol. 182 (2), 319-324 (2013).
- Hutchins, G. D., Miller, M. A., Soon, V. C., Receveur, T. Small animal PET imaging. ILAR J. 49 (1), 54-65 (2008).
- Franc, B. L., Acton, P. D., Mari, C., Hasegawa, B. H. Small-animal SPECT and SPECT/CT: important tools for preclinical investigation. J Nucl Med. 49 (10), 1651-1663 (2008).
- Badea, C. T. H. L., Hedlund, L. W., Johnson, G. A. Micro-CT with respiratory and cardiac gating: Micro-CT with respiratory and cardiac gating. Med Phys. 31 (12), 3324-3329 (2004).
- Guo, X., Johnston, S. M., Qi, Y., Johnson, G. A., Badea, C. T. 4D micro-CT using fast prospective gating. Phys Med Biol. 57 (1), 257 (2011).
- Drangova, M., Ford, N. L., Detombe, S. A., Wheatley, A. R., Holdsworth, D. W. Fast retrospectively gated quantitative four-dimensional (4D) cardiac micro computed tomography imaging of free-breathing mice. Invest Radiol. 42 (2), 85-94 (2007).
- Blocker, S. J., Holbrook, M. D., Mowery, Y. M., Sullivan, D. C., Badea, C. T. The impact of respiratory gating on improving volume measurement of murine lung tumors in micro-CT imaging. PLoS One. 15 (2), e0225019 (2020).
- Bartling, S. H., et al. Retrospective motion gating in small animal CT of mice and rats. Invest Radiol. 42 (10), 704-714 (2007).
- Kuntz, J., et al. Fully automated intrinsic respiratory and cardiac gating for small animal CT. Phys Med Biol. 55 (7), 2069 (2010).
- Hahn, A., Sauppe, S., Lell, M., Kachelrieß, M. Automatic intrinsic cardiac and respiratory gating from cone-beam CT scans of the thorax region. SPIE Proc. Med Imaging. 9783, 200-205 (2016).
- Maes, F., Vandermeulen, D., Suetens, P. Medical image registration using mutual information. Proc IEEE. 91 (10), 1699-1722 (2003).
- Sheng, J., Chen, B., Ma, Y., Shi, Y. A novel reconstruction approach combining OSEM and split Bregman method for low dose CT. Biomedical Signal Processing and Control. 62, 102095 (2020).
- Romdhane, H., Cherni, M. A., Sallem, D. B. On the efficiency of OSEM algorithm for tomographic lung CT images reconstruction. 2016 Second International Image Processing, Applications and Systems (IPAS). , (2016).
- Ewald, A. J., Werb, Z., Egeblad, M. Monitoring of vital signs for long-term survival of mice under anesthesia. Cold Spring Harb Protoc. 2, 5563 (2011).
- Suckow, C., et al. Multimodality rodent imaging chambers for use under barrier conditions with gas anesthesia. Mol Imaging Biol. 11 (2), 100-106 (2009).
- Owen, M. . Practical signal processing. , 81 (2007).
- Feldkamp, L. A., Davis, L. C., Kress, J. W. Practical cone-beam algorithm. J Opt Soc Am. A-optics Image Sci Vision. 1 (6), 612-619 (1984).
- Loening, A. M., Gambhir, S. S. AMIDE: A free software tool for multimodality medical image analysis. Mol Imaging. 2 (3), 131-137 (2003).
- Rodriguez-Molares, A., et al. The generalized contrast-to-noise ratio: A formal definition for lesion detectability. IEEE Trans Ultrason Ferroelectr Freq Control. 67 (4), 745-759 (2020).
- Patterson, M. S., Foster, F. S. The improvement and quantitative assessment of B-mode images produced by an annular array/cone hybrid. Ultrason Imaging. 5 (3), 195-213 (1983).
- . Dragonfly 2022.2 Available from: https://www.theobjects.com/dragonfly (2022)
- Pillai, I. C. L., et al. Cardiac fibroblasts adopt osteogenic fates and can be targeted to attenuate pathological heart calcification. Cell Stem Cell. 20 (2), 218-232 (2017).
- Li, S., et al. A humanized monoclonal antibody targeting an ectonucleotidase rescues cardiac metabolism and heart function after myocardial infarction. Cell Rep Med. 5 (11), 101795 (2024).
- Nadkarni, R., Clark, D. P., Allphin, A. J., Badea, C. T. Investigating deep learning strategies for fast denoising of 5D cardiac photon-counting micro-CT images. Phys Med Biol. 69 (20), 205010 (2024).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoExplore Mais Artigos
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados