Un abonnement à JoVE est nécessaire pour voir ce contenu. Connectez-vous ou commencez votre essai gratuit.
Method Article
Déclenchement cardiaque rétrospectif à l’aide d’un prototype de tomographe à rayons X pour petits animaux
Dans cet article
Résumé
Nous fournissons une description complète de la méthode intrinsèque de déclenchement cardiaque rétrospectif du CrumpCAT, un prototype de scanner de tomodensitométrie (TDM) à rayons X pour petits animaux conçu et construit dans notre institut de recherche.
Résumé
Le CrumpCAT est un prototype de tomodensitomètre (TDM) pour petits animaux développé dans notre institut de recherche. Le détecteur CMOS avec une fréquence d’images maximale de 29 Hz et les sources de rayons X au tungstène similaires avec des énergies allant de 50 kVp à 80 kVp sont largement utilisés dans les instruments de tomodensitométrie à rayons X précliniques disponibles dans le commerce. Cela rend le travail décrit très pertinent pour d’autres institutions, malgré la sagesse généralement perçue selon laquelle ces détecteurs ne sont pas adaptés pour contrôler les fréquences cardiaques élevées des souris (~600 battements/min). Le scanner dispose d’une imagerie à résolution moyenne (200 μm) et haute (125 μm), d’une fluoroscopie, d’un déclenchement respiratoire rétrospectif et d’un déclenchement cardiaque rétrospectif, avec reconstruction itérative ou filtrée de l’image de projection. Parmi ces caractéristiques, le déclenchement cardiaque est la caractéristique la plus utile pour étudier les fonctions cardiaques in vivo, car il élimine efficacement le flou d’image causé par les mouvements respiratoires et cardiaques.
Nous décrivons ici notre méthode d’imagerie tomodensitométrie cardiaque, préclinique rétrospective et intrinsèque, visant à faire progresser la recherche sur l’analyse in vivo de la fonction et de la structure cardiaques. La méthode de déclenchement cardiaque acquiert un grand nombre de projections au temps d’exposition le plus court possible (~20 ms), puis extrait rétrospectivement des signaux respiratoires et cardiaques à partir de changements temporels dans des séquences de projection brutes. Ces signaux sont utilisés pour rejeter les projections appartenant à la phase d’inspiration à haut débit de mouvement du cycle respiratoire et pour diviser les projections restantes en 12 groupes, chacun correspondant à une phase du cycle cardiaque. Chaque groupe est reconstruit indépendamment à l’aide d’une méthode itérative pour produire une image volumétrique pour chaque phase cardiaque, ce qui donne un ensemble de données quadridimensionnelles (4D).
Ces images de phase peuvent être analysées collectivement ou individuellement, ce qui permet une évaluation détaillée de la fonction cardiaque. Nous avons démontré l’efficacité des deux approches de la fonction de contrôle cardiaque du prototype de scanner grâce à des résultats d’imagerie in vivo représentatifs.
Introduction
La recherche sur les petits animaux utilise souvent une combinaison de modalités d’imagerie non invasives, la tomodensitométrie à rayons X (TDM) étant un choix de premier plan en raison de sa maturité, de sa rentabilité, de sa vitesse 1,2 et de sa capacité à fournir des informations complémentaires avec d’autres modalités telles que la tomographie par émission de positons (TEP)2,3 et la tomographie par émission monophotonique (SPECT)2,4. Cependant, comme d’autres techniques d’imagerie, la TDM est sensible aux artefacts de mouvement physiologiques causés par les battements du cœur ou la respiration, ce qui introduit un flou et limite la précision de la recherche.
Pour remédier à cette limitation, le flou des mouvements respiratoires et cardiaques peut être atténué grâce à une technique connue sous le nom de gating 5,6,7,8, où l’acquisition de données est synchronisée avec des phases spécifiques du cycle cardiaque ou respiratoire (ou portes). Une approche pour y parvenir, connue sous le nom de porte prospective 3,6, consiste à fixer des capteurs à l’animal pour fournir des signaux de porte en temps réel à un scanner compatible. Bien qu’efficace, cette méthode demande beaucoup de main-d’œuvre et de temps, en particulier lorsqu’il s’agit de fixer des capteurs à la poitrine et aux pattes de petits animaux comme les souris, limitant ainsi l’échelle des études. Alternativement, le gate rétrospectif intrinsèque 7,9,10,11 consiste à acquérir des données de séries chronologiques sans utiliser de capteurs, mais en identifiant des caractéristiques dans les données qui permettent un tri rétrospectif des résultats en fonction de leur phase dans le cycle cardiaque ou respiratoire. Cette approche offre des résultats comparables à ceux du gate prospectif, mais sans avoir besoin de matériel supplémentaire ou d’efforts nécessaires à la fixation d’un capteur d’impulsions et, par conséquent, simplifie considérablement les protocoles expérimentaux.
Dans notre méthode d’imagerie préclinique par tomodensitométrie cardiaque, nous utilisons le déclenchement rétrospectif intrinsèque pour extraire les cycles respiratoires et cardiaques des variations d’amplitude dans les régions des projections de rayons X qui présentent les changements les plus significatifs entre les images successives. Pour faciliter ce processus, un modèle de thorax de souris est co-enregistré sur la première projection postéro-antérieure à l’aide de l’information mutuelle12. Une fois le modèle en place, les intensités des pixels dans une fenêtre près du diaphragme sont additionnées pour générer un signal respiratoire de substitution, tandis que celles près du myocarde sont additionnées pour dériver le signal cardiaque de substitution. Ces signaux sont ensuite filtrés par bande passante dans le domaine temporel, et chaque trame de l’ensemble de données se voit attribuer un numéro de phase fractionnaire (entre 0 et 1) en fonction de sa phase respiratoire et cardiaque. Cela permet de sélectionner ou de rejeter les projections en fonction de leurs valeurs de phase. En règle générale, les images correspondant à la phase de fin d’expiration du cycle respiratoire (phase 0,15 ≤ < 0,85) sont conservées, tandis que celles de la phase d’inspiration, où le mouvement est le plus prononcé, sont écartées. Les images restantes sont regroupées en 12 phases cardiaques, chacune représentant 1/12 (0,083) du cycle cardiaque et sont reconstruites en images 3D à l’aide d’une méthode itérative (Ordered Subset Expectations Maximization [OSEM])13,14. L’ensemble du processus est résumé dans la figure 1.
Protocole
Les protocoles d’expérimentation animale ont été examinés et approuvés par l’Institutional Animal Care and Use Committee de l’Université de Californie à Los Angeles (UCLA). Des souris C57BL/6J (âgées de 8 semaines, mâles, 24-26 g) ont été utilisées dans ce protocole. Le tomodensitomètre utilisé dans cette étude est le CrumpCAT (Figure 2), un prototype développé dans notre institut de recherche pour la recherche préclinique, nous offrant le contrôle et la flexibilité nécessaires pour optimiser les protocoles d’acquisition et de reconstruction. La méthode suppose que les souris anesthésiées auront une fréquence cardiaque ne dépassant pas 600 battements/min et une fréquence respiratoire comprise entre 20 et 180 respirations/min15.
1. Réglages de l’équipement
- Faites fonctionner la source de rayons X à une tension de crête de 50 kVp avec un courant continu de 200 μA.
- Réglez le regroupement des pixels de la caméra à rayons X sur 2 et le temps d’exposition sur sa valeur pratique la plus courte (temps d’exposition de 20 ms plus temps de lecture de 14 ms pour un temps d’échantillonnage total de 34 ms).
REMARQUE : L’option sans binning (binning 1) a une fréquence d’images plus lente et, par conséquent, ne peut pas être utilisée pour les acquisitions contrôlées. La fréquence d’images maximale est d’environ 30 images/s.
2. Préparation des animaux
- Anesthésie les souris avec un mélange d’oxygène et d’isoflurane à une concentration de 2,0 %.
- Gardez les animaux sous anesthésie pendant 10 minutes avant l’imagerie par tomodensitométrie, afin de s’assurer que les signes vitaux sont stables et que les animaux sont détendus pour éviter tout mouvement du corps pendant l’examen. Jugez de la profondeur de l’anesthésie d’une souris par l’absence de réflexe de pincement des orteils lorsque vous pincez fermement la sangle entre les orteils avec un ongle.
- Pour que l’imagerie cardiaque permette de visualiser les cavités cardiaques, injectez par voie intraveineuse 100 μL d’agent de contraste CT à travers la veine de la queue immédiatement avant l’imagerie CT (Figure 3A).
- Placez la souris anesthésiée à l’intérieur de la chambre d’imagerie CT (Figure 3B).
3. Acquisition de données
- Allumez le logiciel CrumpCAT.
- Imagerie TDM non fermée à haute résolution (125 μm, binning 1) (Figure 4A)
- Entrez un ID d’étude sur l’interface utilisateur.
- Sélectionnez Souris Hi-Res dans le menu déroulant Protocole .
- Cliquez sur le bouton Scanner de l’interface utilisateur pour acquérir 720 projections avec un temps d’exposition de 80 ms/projection.
- Imagerie TDM non fermée à résolution moyenne (200 μm, binning 2) (Figure 4B)
- Entrez un ID d’étude sur l’interface utilisateur.
- Sélectionnez Souris Standard dans le menu déroulant Protocole .
- Cliquez sur le bouton Scanner de l’interface utilisateur pour acquérir 720 projections avec un temps d’exposition de 100 ms/projection.
- Imagerie par tomodensitométrie à grille (200 μm, binning 2) (Figure 4C)
- Entrez un ID d’étude sur l’interface utilisateur.
- Sélectionnez Cardiac Gating dans le menu déroulant Protocole .
- Cliquez sur le bouton Scanner de l’interface utilisateur et acquérez 21 600 projections avec un temps d’exposition de 20 ms/projection.
4. Prétraitement des données
REMARQUE : Les étapes de prétraitement ne sont requises que pour les acquisitions contrôlées. Toutes ces étapes sont effectuées automatiquement par le logiciel de reconstruction et aucune intervention de l’opérateur n’est requise.
- Extraction du signal
- Pour le co-enregistrement du modèle de thorax, supposons qu’une petite image (modèle) représentant grossièrement le thorax d’une souris avec les côtes, le cœur, les poumons et le foie soit automatiquement co-enregistrée sur la première projection de rayons X, en maximisant l’information mutuelle12 entre la projection et le modèle (Figure 5).
REMARQUE : Seules les opérations de traduction sont effectuées sur le modèle et le modèle co-enregistré est utilisé pour identifier les régions d’intérêt (ROI) sur toutes les projections. - Pour l’extraction du signal respiratoire, permettez qu’un ROI rectangulaire (ROI-1) soit désigné dans le modèle pour représenter le diaphragme et le signal respiratoire à générer en additionnant les intensités de pixels à l’intérieur de ROI-1 pour chaque projection (Figure 5) .
- Pour l’extraction du signal cardiaque, un deuxième rectangle ROI (ROI-2) doit être désigné dans le modèle près du cœur, et le signal cardiaque est généré en additionnant les intensités de pixels en ROI-2 pour chaque projection (Figure 5).
REMARQUE : Le modèle de thorax n’est co-enregistré que sur la première projection afin d’identifier les lignes et les colonnes d’image (fenêtre) qui doivent être utilisées pour l’extraction du signal. Cette fenêtre est suffisamment large pour englober toutes les projections tant que l’animal est raisonnablement centré, comme c’est le cas lors de l’utilisation de notre lit pour animaux16.
- Pour le co-enregistrement du modèle de thorax, supposons qu’une petite image (modèle) représentant grossièrement le thorax d’une souris avec les côtes, le cœur, les poumons et le foie soit automatiquement co-enregistrée sur la première projection de rayons X, en maximisant l’information mutuelle12 entre la projection et le modèle (Figure 5).
- Filtre passe-bande
- Permet de générer un filtre passe-bande en combinant deux fonctions Sinc avec des fréquences de coupure différentes dans le domaine temporel17, qui correspondent à des filtres passe-bas dans le domaine fréquentiel comme décrit ci-dessous.
- Laissez le logiciel appliquer le filtre passe-bande sur le signal cardiaque en utilisant une bande de fréquence de [300, 600] min-1 (Figure 6A).
REMARQUE : Le signal cardiaque non filtré (Figure 6B) contient toujours une contribution significative du mouvement respiratoire. - De même, le signal respiratoire est filtré à l’aide d’une bande de fréquence de [20, 300] min-1. Les harmoniques supérieures sont préservées pour produire un signal qui n’est pas simplement sinusoïdal et identifie plus clairement les deux phases principales : l’inspiration et l’expiration.
REMARQUE : À ce stade, les signaux respiratoires et cardiaques peuvent facilement être interpolés et considérés comme des fonctions continues du temps dans le but de calculer le nombre de phase fractionnaire.
- Attribution de phase
- Laissez le logiciel identifier le début de chaque cycle (respiratoire ou cardiaque) en trouvant les passages à zéro de la dérivée première du signal. Chaque passage à zéro correspond à un maximum dans le signal et marque le début d’un nouveau cycle et la fin du précédent. Pour un exemple, voir le signal respiratoire dans la figure 7.
- Permettre d’attribuer une valeur de phase fractionnaire (entre 0 et 1) à chaque projection pour chacun des deux signaux (respiratoire ou cardiaque).
REMARQUE : Par définition, le point de départ a une valeur de phase fractionnaire de zéro (pour le cycle en cours) ou de un (pour le cycle précédent).
- Masques de sélection
- Créons 12 masques binaires (un par phase cardiaque) pour sélectionner les projections appartenant à chaque phase. Chaque masque contient 21 600 entrées (une par projection) qui sont soit un 0, soit un 1, ce qui signifie respectivement rejeter ou conserver cette projection.
- Pour chaque masque (phase), observez que le logiciel indique si une projection est conservée (1) ou écartée (0) en fonction de son numéro de phase fractionnaire. Pour la phase 0, les projections avec une valeur de phase cardiaque dans l’intervalle [0, 1/12] sont conservées. Pour la phase 1, les valeurs de phase cardiaque dans l’intervalle [1/12, 2/12] sont conservées, et ainsi de suite.
- Pour chaque masque (phase), notez que toutes les projections avec une phase respiratoire inférieure à 0,15 ou supérieure à 0,85 sont rejetées (0 dans le masque) car elles appartiennent à la phase d’inspiration, qui a le plus grand mouvement (Figure 8A). La figure 8B montre l’assignation de la phase cardiaque aux projections pour les 2 premières s.
REMARQUE : Aucun biais significatif n’a été introduit dans ce processus de sélection puisque le nombre de projections par phase était relativement constant à 1 800 ± 194.
5. Reconstruction de l’image
- Pour les tomodensitogrammes non contrôlés, reconstruisez les images à une résolution moyenne (200 μm) ou élevée (125 μm) à l’aide de l’algorithme de rétroprojection filtrée (Feldkamp18) avec un filtre Shepp-Logan.
- Pour les tomodensitogrammes cardiaques, reconstruisez chaque image de phase cardiaque à l’aide de l’algorithme itératif OSEM13, avec 12 sous-ensembles et 8 itérations, en ne prenant en compte que les projections sélectionnées contribuant à une phase donnée (comme indiqué dans un masque binaire de sélection de phase).
- Pour réduire le bruit, appliquez un filtre médian post-reconstruction dans la dimension temporelle (c’est-à-dire sur toutes les images) et un filtre 3D-Gaussien clair dans la dimension spatiale (σ = 38 μm).
REMARQUE : Pour la reconstruction d’images respiratoires, utilisez toutes les phases cardiaques indistinctement. La correction de durcissement des poutres (correction de l’eau) est appliquée à chaque reconstruction.
6. Évaluation de l’image et quantification du volume du ventricule gauche (VG)
- Ouvrez l’image CT dans une visionneuse DICOM telle qu’Amide19 (Figure 9A).
- Améliorez le contraste visible de l’image en définissant la plage de valeurs CT (mesurée en unité de Hounsfield [HU]) sur [-500, 500] (Figure 9A).
- Évaluation de la qualité de l’image
- Dessinez une zone d’intérêt pour la région calcifiée, qui est définie comme la région autour du maximum avec une valeur CT supérieure ou égale à 85 % de la valeur maximale de l’HU (Figure 9B).
- Utilisez le rapport contraste/bruit (CNR)20,21 comme mesure pour évaluer la qualité de l’image et sa capacité à identifier les petites structures, telles que les petites calcifications :
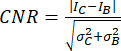 ,
,
où I et σ représentent l’intensité moyenne et l’écart-type d’une région : calcification (indice C) ou arrière-plan (indice B).
- Quantification du volume du ventricule gauche
- Dessinez un retour d’intérêt 3D à main levée pour identifier le VG dans chaque phase (Figure 10A).
- Quantifiez le volume VG en calculant les voxels avec une valeur CT seuil de 730 HU (Figure 10B). Un graphique des volumes VG est illustré à la figure 10C.
REMARQUE : Le seuil de valeur CT n’est pas absolu ; cela dépend de l’agent de contraste utilisé, de la taille de l’animal, de la souche, de l’état de santé et du temps entre l’injection et l’imagerie par tomodensitométrie. Amide est un logiciel distribué gratuitement développé par notre institut, mais d’autres visionneuses d’images plus sophistiquées sont disponibles (par exemple, ORS Dragonfly22).
Résultats
Nous avons d’abord comparé des images CT non fermées et fermées pour visualiser la calcification cardiaque chez la souris (mâle, 30-32 g). Le modèle murin de calcification cardiaque a été créé en induisant une lésion cardiaque par congélation-décongélation rapide du tissu cardiaque (cryo-lésion), comme décrit précédemment23. Avec les protocoles d’imagerie par tomodensitométrie sans porte, les calcifications cardiaques ont été plus clairemen...
Discussion
La mise en œuvre matérielle spécifique décrite ici est un système de tomodensitométrie à rayons X sur mesure, unique à notre institut, mais le détecteur spécifique est largement utilisé dans les instruments de tomodensitométrie à rayons X précliniques disponibles dans le commerce, ce qui rend le travail décrit pertinent pour d’autres institutions. Ce système est fonctionnellement le prototype de deux sous-systèmes microCT à rayons X in vivo disponibles dans l...
Déclarations de divulgation
Le Dr Richard Taschereau est consultant chez Sofie Biosciences et Xodus Imaging. Le Dr Arion F. Chatziioannou est l’un des fondateurs de Sofie Biosciences.
Remerciements
Nous remercions tous les membres du Crump Preclinical Imaging Technology Center de l’UCLA pour leur aide et leur soutien. En particulier, nous remercions Mikayla Tamboline et Isabel Day d’avoir préparé les animaux pour l’imagerie par tomodensitométrie cardiaque et nous remercions Sophie Shumilov d’avoir généré certains des retours d’intérêt du ventricule gauche au cours de l’étude. Nous remercions également les Drs Arjun Deb et Yijie Wang (UCLA) d’avoir fourni les modèles murins de lésions cardiaques ischémiques aiguës pour l’imagerie microCT de calcification cardiaque. Ce travail est soutenu par la subvention de soutien du NIH Cancer Center (2 P30 CA016042-44).
matériels
| Name | Company | Catalog Number | Comments |
| C57BL/6J mice | Jackson Laboratory | 664 | Male, 8 weeks old, 24-26 g |
| Dexela camera | Varex | 1512 | Detector, 20 ms exposure, 74.8/149.6 µm pixel |
| VivoVist | Nanoprobes | 1301-5X0.25ML | CT Contrast agent |
| X-ray source | Moxtek | TUB00082 | 50 kV peak, 200 µA, 1.0 mm-thick Al filter |
Références
- Schambach, S. J., Bag, S., Schilling, L., Groden, C., Brockmann, M. A. Application of micro-CT in small animal imaging. Methods. 50 (1), 2-13 (2010).
- Koba, W., Jelicks, L. A., Fine, E. J. MicroPET/SPECT/CT imaging of small animal models of disease. Am J Pathol. 182 (2), 319-324 (2013).
- Hutchins, G. D., Miller, M. A., Soon, V. C., Receveur, T. Small animal PET imaging. ILAR J. 49 (1), 54-65 (2008).
- Franc, B. L., Acton, P. D., Mari, C., Hasegawa, B. H. Small-animal SPECT and SPECT/CT: important tools for preclinical investigation. J Nucl Med. 49 (10), 1651-1663 (2008).
- Badea, C. T. H. L., Hedlund, L. W., Johnson, G. A. Micro-CT with respiratory and cardiac gating: Micro-CT with respiratory and cardiac gating. Med Phys. 31 (12), 3324-3329 (2004).
- Guo, X., Johnston, S. M., Qi, Y., Johnson, G. A., Badea, C. T. 4D micro-CT using fast prospective gating. Phys Med Biol. 57 (1), 257 (2011).
- Drangova, M., Ford, N. L., Detombe, S. A., Wheatley, A. R., Holdsworth, D. W. Fast retrospectively gated quantitative four-dimensional (4D) cardiac micro computed tomography imaging of free-breathing mice. Invest Radiol. 42 (2), 85-94 (2007).
- Blocker, S. J., Holbrook, M. D., Mowery, Y. M., Sullivan, D. C., Badea, C. T. The impact of respiratory gating on improving volume measurement of murine lung tumors in micro-CT imaging. PLoS One. 15 (2), e0225019 (2020).
- Bartling, S. H., et al. Retrospective motion gating in small animal CT of mice and rats. Invest Radiol. 42 (10), 704-714 (2007).
- Kuntz, J., et al. Fully automated intrinsic respiratory and cardiac gating for small animal CT. Phys Med Biol. 55 (7), 2069 (2010).
- Hahn, A., Sauppe, S., Lell, M., Kachelrieß, M. Automatic intrinsic cardiac and respiratory gating from cone-beam CT scans of the thorax region. SPIE Proc. Med Imaging. 9783, 200-205 (2016).
- Maes, F., Vandermeulen, D., Suetens, P. Medical image registration using mutual information. Proc IEEE. 91 (10), 1699-1722 (2003).
- Sheng, J., Chen, B., Ma, Y., Shi, Y. A novel reconstruction approach combining OSEM and split Bregman method for low dose CT. Biomedical Signal Processing and Control. 62, 102095 (2020).
- Romdhane, H., Cherni, M. A., Sallem, D. B. On the efficiency of OSEM algorithm for tomographic lung CT images reconstruction. 2016 Second International Image Processing, Applications and Systems (IPAS). , (2016).
- Ewald, A. J., Werb, Z., Egeblad, M. Monitoring of vital signs for long-term survival of mice under anesthesia. Cold Spring Harb Protoc. 2, 5563 (2011).
- Suckow, C., et al. Multimodality rodent imaging chambers for use under barrier conditions with gas anesthesia. Mol Imaging Biol. 11 (2), 100-106 (2009).
- Owen, M. . Practical signal processing. , 81 (2007).
- Feldkamp, L. A., Davis, L. C., Kress, J. W. Practical cone-beam algorithm. J Opt Soc Am. A-optics Image Sci Vision. 1 (6), 612-619 (1984).
- Loening, A. M., Gambhir, S. S. AMIDE: A free software tool for multimodality medical image analysis. Mol Imaging. 2 (3), 131-137 (2003).
- Rodriguez-Molares, A., et al. The generalized contrast-to-noise ratio: A formal definition for lesion detectability. IEEE Trans Ultrason Ferroelectr Freq Control. 67 (4), 745-759 (2020).
- Patterson, M. S., Foster, F. S. The improvement and quantitative assessment of B-mode images produced by an annular array/cone hybrid. Ultrason Imaging. 5 (3), 195-213 (1983).
- . Dragonfly 2022.2 Available from: https://www.theobjects.com/dragonfly (2022)
- Pillai, I. C. L., et al. Cardiac fibroblasts adopt osteogenic fates and can be targeted to attenuate pathological heart calcification. Cell Stem Cell. 20 (2), 218-232 (2017).
- Li, S., et al. A humanized monoclonal antibody targeting an ectonucleotidase rescues cardiac metabolism and heart function after myocardial infarction. Cell Rep Med. 5 (11), 101795 (2024).
- Nadkarni, R., Clark, D. P., Allphin, A. J., Badea, C. T. Investigating deep learning strategies for fast denoising of 5D cardiac photon-counting micro-CT images. Phys Med Biol. 69 (20), 205010 (2024).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationExplorer plus d’articles
This article has been published
Video Coming Soon