Zum Anzeigen dieser Inhalte ist ein JoVE-Abonnement erforderlich. Melden Sie sich an oder starten Sie Ihre kostenlose Testversion.
Method Article
Retrospektives Cardiac Gating mit einem Prototyp eines Röntgen-Computertomographen für Kleintiere
In diesem Artikel
Zusammenfassung
Wir bieten eine umfassende Beschreibung der intrinsischen retrospektiven kardialen Gating-Methode des CrumpCAT, einem Prototyp eines Röntgen-Computertomographen (CT) für Kleintiere, der an unserer Forschungseinrichtung entwickelt und gebaut wurde.
Zusammenfassung
Der CrumpCAT ist ein Prototyp eines an unserer Forschungseinrichtung entwickelten Prototyps eines Röntgen-Computertomographen (CT) für Kleintiere. Der CMOS-Detektor mit einer maximalen Bildrate von 29 Hz und ähnliche Wolfram-Röntgenquellen mit Energien im Bereich von 50 kVp bis 80 kVp werden häufig in kommerziell erhältlichen präklinischen Röntgen-CT-Instrumenten eingesetzt. Dies macht die beschriebene Arbeit für andere Institutionen von hoher Relevanz, trotz der allgemein angenommenen Meinung, dass diese Detektoren nicht geeignet sind, die hohen Herzfrequenzen von Mäusen (~600 Schläge/min) zu messen. Der Scanner verfügt über Bildgebung mit mittlerer (200 μm) und hoher (125 μm) Auflösung, Fluoroskopie, retrospektives respiratorisches Gating und retrospektives kardiales Gating mit iterativer oder gefilterter Projektionsbildrekonstruktion. Unter diesen Merkmalen ist das kardiale Gating die nützlichste Funktion für die Untersuchung von Herzfunktionen in vivo, da es Bildunschärfen, die durch Atem- und Herzbewegungen verursacht werden, effektiv eliminiert.
Hier beschreiben wir unsere Methode für die präklinische intrinsische retrospektive kardiogesteuerte CT-Bildgebung, die darauf abzielt, die Forschung zur in vivo kardialen Funktions- und Strukturanalyse voranzutreiben. Die Cardiac-Gating-Methode erfasst eine große Anzahl von Projektionen bei kürzester praktischer Belichtungszeit (~20 ms) und extrahiert dann retrospektiv respiratorische und kardiale Signale aus zeitlichen Veränderungen in rohen Projektionssequenzen. Diese Signale werden verwendet, um Projektionen, die zur Inspirationsphase mit hoher Bewegungsrate des Atemzyklus gehören, zu unterdrücken und die verbleibenden Projektionen in 12 Gruppen zu unterteilen, die jeweils einer Phase des Herzzyklus entsprechen. Jede Gruppe wird unabhängig voneinander mit einer iterativen Methode rekonstruiert, um für jede Herzphase ein volumetrisches Bild zu erstellen, was zu einem vierdimensionalen (4D) Datensatz führt.
Diese Phasenbilder können entweder einzeln oder einzeln analysiert werden, was eine detaillierte Beurteilung der Herzfunktion ermöglicht. Wir demonstrierten die Wirksamkeit beider Ansätze der Cardiac-Gating-Funktion des Prototyp-Scanners durch repräsentative in vivo Bildgebungsergebnisse.
Einleitung
In der Kleintierforschung wird häufig eine Kombination von nicht-invasiven Bildgebungsmodalitäten eingesetzt, wobei die Röntgen-Computertomographie (CT) aufgrund ihrer Ausgereiftheit, Kosteneffizienz, Geschwindigkeit 1,2 und ihrer Fähigkeit, ergänzende Informationen neben anderen Modalitäten wie der Positronen-Emissions-Tomographie (PET)2,3 und der Einzelphotonen-Emissions-Computertomographie (SPECT)2,4 zu liefern, eine herausragende Wahl ist . Wie andere bildgebende Verfahren ist die CT jedoch anfällig für physiologische Bewegungsartefakte, die durch das schlagende Herz oder die Atmung verursacht werden, was zu Unschärfen führt und die Genauigkeit der Forschung einschränkt.
Um diese Einschränkung zu beheben, kann die Bewegungsunschärfe der Atemwege und des Herzens durch eine Technik gemildert werden, die als Gating 5,6,7,8 bekannt ist und bei der die Datenerfassung mit bestimmten Phasen des Herz- oder Atemzyklus (oder Gates) synchronisiert wird. Ein Ansatz, um dies zu erreichen, bekannt als Prospective Gating 3,6, besteht darin, Sensoren am Tier anzubringen, um Echtzeit-Gating-Signale an einen kompatiblen Scanner zu liefern. Diese Methode ist zwar effektiv, aber arbeits- und zeitintensiv, insbesondere wenn Sensoren an der Brust und den Pfoten von Kleintieren wie Mäusen angebracht werden, wodurch der Umfang der Studien eingeschränkt wird. Alternativ beinhaltet das intrinsische retrospektive Gating 7,9,10,11 die Erfassung von Zeitreihendaten ohne den Einsatz von Sensoren, sondern durch die Identifizierung von Merkmalen in den Daten, die eine retrospektive Sortierung der Ergebnisse basierend auf ihrer Phase im Herz- oder Atemzyklus ermöglichen. Dieser Ansatz bietet Ergebnisse, die mit dem prospektiven Gating vergleichbar sind, jedoch ohne die Notwendigkeit zusätzlicher Hardware oder den Aufwand, der mit der Anbringung von Impulssensoren verbunden ist, und vereinfacht daher die experimentellen Protokolle erheblich.
In unserer Methode für die präklinische kardiale CT-Bildgebung verwenden wir ein intrinsisches retrospektives Gating, um respiratorische und kardiale Zyklen aus Amplitudenvariationen in Regionen in Röntgenprojektionen zu extrahieren, die die signifikantesten Veränderungen zwischen aufeinanderfolgenden Frames aufweisen. Um diesen Prozess zu erleichtern, wird eine Maus-Thoraxschablone mit Hilfe von Mutual Information12 auf der ersten posteroanterioren Projektion co-registriert. Sobald die Vorlage an Ort und Stelle ist, werden die Pixelintensitäten in einem Fenster in der Nähe des Zwerchfells summiert, um ein Surrogat-Atmungssignal zu erzeugen, während die Pixelintensitäten in der Nähe des Myokards summiert werden, um das Surrogat-Herzsignal abzuleiten. Diese Signale werden dann im Zeitbereich bandpassgefiltert, und jedem Frame im Datensatz wird eine fraktionierte Phasennummer (zwischen 0 und 1) zugewiesen, die auf seiner Atem- und Herzphase basiert. Dies ermöglicht die Auswahl oder Ablehnung von Projektionen entsprechend ihrer Phasenwerte. In der Regel werden Frames, die der End-Exspirationsphase des Atemzyklus entsprechen (0,15 ≤ Phase < 0,85), beibehalten, während die Frames aus der Inspirationsphase, in der die Bewegung am stärksten ausgeprägt ist, verworfen werden. Die restlichen Frames werden in 12 kardiale Phasen gruppiert, die jeweils 1/12 (0,083) des Herzzyklus repräsentieren, und mit einer iterativen Methode (Ordered Subset Expectation Maximization [OSEM])13,14 in 3D-Bilder rekonstruiert. Der gesamte Prozess ist in Abbildung 1 zusammengefasst.
Protokoll
Die Tierversuchsprotokolle wurden vom Institutional Animal Care and Use Committee der University of California, Los Angeles (UCLA) überprüft und genehmigt. In diesem Protokoll wurden C57BL/6J-Mäuse (8 Wochen alt, männlich, 24-26 g) verwendet. Bei dem in dieser Studie verwendeten CT-Scanner handelt es sich um den CrumpCAT (Abbildung 2), einen Prototyp, der an unserer Forschungseinrichtung für die präklinische Forschung entwickelt wurde und uns die Kontrolle und Flexibilität bietet, die wir benötigen, um die Erfassungs- und Rekonstruktionsprotokolle zu optimieren. Die Methode geht davon aus, dass anästhesierte Mäuse eine Herzfrequenz von nicht mehr als 600 Schlägen/min und eine Atemfrequenz zwischen 20 und 180 Atemzügen/min haben15.
1. Einstellungen der Ausrüstung
- Betreiben Sie die Röntgenquelle mit einer Spitzenspannung von 50 kVp und 200 μA Dauerstrom.
- Stellen Sie das Pixel-Binning der Röntgenkamera auf 2 und die Belichtungszeit auf den kürzesten praktischen Wert ein (20 ms Belichtungszeit plus 14 ms Lesezeit für eine Gesamtabtastzeit von 34 ms).
HINWEIS: Die Option ohne Binning (Binning 1) hat eine langsamere Bildrate und kann daher nicht für Gated Acquisitions verwendet werden. Die maximale Bildrate liegt bei etwa 30 Bildern/s.
2. Vorbereitung der Tiere
- Betäuben Sie Mäuse mit einem Gemisch aus Sauerstoff und Isoflurangas in einer Konzentration von 2,0 %.
- Halten Sie die Tiere vor der CT-Bildgebung 10 Minuten lang unter Narkose, um sicherzustellen, dass die Vitalparameter stabil sind und dass die Tiere entspannt sind, um Körperbewegungen während des Scans zu vermeiden. Beurteilen Sie die Narkosetiefe einer Maus anhand des fehlenden Zehenkneifreflexes, wenn das Gurtband zwischen den Zehen mit einem Fingernagel fest eingeklemmt wird.
- Für die kardiologische Bildgebung zur Visualisierung der Herzkammern injizieren Sie unmittelbar vor der CT-Bildgebung intravenös 100 μl CT-Kontrastmittel durch die Schwanzvene (Abbildung 3A).
- Platzieren Sie die anästhesierte Maus in der CT-Bildgebungskammer (Abbildung 3B).
3. Datenerfassung
- Schalten Sie die CrumpCAT-Software ein.
- CT-Bildgebung ohne Gated mit hoher Auflösung (125 μm, Binning 1) (Abbildung 4A)
- Geben Sie eine Studien-ID auf der Benutzeroberfläche ein.
- Wählen Sie Mouse Hi-Res im Dropdown-Menü Protokoll aus.
- Klicken Sie auf die Schaltfläche Scan auf der Benutzeroberfläche, um 720 Projektionen mit einer Belichtungszeit von 80 ms/Projektion zu erfassen.
- CT-Bildgebung ohne Gated mit mittlerer Auflösung (200 μm, Binning 2) (Abbildung 4B)
- Geben Sie eine Studien-ID auf der Benutzeroberfläche ein.
- Wählen Sie Mausstandard im Dropdown-Menü Protokoll aus.
- Klicken Sie auf die Schaltfläche Scan in der Benutzeroberfläche, um 720 Projektionen mit einer Belichtungszeit von 100 ms/Projektion zu erfassen.
- Gated CT-Bildgebung (200 μm, Binning 2) (Abbildung 4C)
- Geben Sie eine Studien-ID auf der Benutzeroberfläche ein.
- Wählen Sie Cardiac Gating im Dropdown-Menü Protokoll aus.
- Klicken Sie auf die Schaltfläche Scan auf der Benutzeroberfläche und erfassen Sie 21.600 Projektionen mit einer Belichtungszeit von 20 ms/Projektion.
4. Vorverarbeitung von Daten
HINWEIS: Vorverarbeitungsschritte sind nur für Gated Acquisitions erforderlich. Alle diese Schritte werden von der Rekonstruktionssoftware automatisch ausgeführt und es ist kein Bedienereingriff erforderlich.
- Signalextraktion
- Für die Koregistrierung von Thoraxschablonen wird ein kleines Bild (Schablone), das grob einen Mausthorax mit den Rippen, dem Herzen, der Lunge und der Leber darstellt, automatisch auf der ersten Röntgenprojektion mitregistriert, indem die gegenseitige Information12 zwischen der Projektion und der Schablone maximiert wird (Abbildung 5).
HINWEIS: Für die Vorlage werden nur Übersetzungsvorgänge durchgeführt, und die mitregistrierte Vorlage wird verwendet, um interessante Regionen (ROIs) für alle Prognosen zu identifizieren. - Lassen Sie für die Extraktion von Atemsignalen zu, dass in der Vorlage ein rechteckiger ROI (ROI-1) festgelegt wird, um das Zwerchfell und das zu erzeugende Atemsignal darzustellen, indem Sie die Pixelintensitäten innerhalb von ROI-1 für jede Projektion summieren (Abbildung 5).
- Für die Extraktion von Herzsignalen wird ein zweites Rechteck ROI (ROI-2) in der Vorlage in der Nähe des Herzens festgelegt und das Herzsignal durch Summieren der Pixelintensitäten in ROI-2 für jede Projektion erzeugt (Abbildung 5).
HINWEIS: Die Thoraxvorlage wird nur bei der ersten Projektion mitregistriert, um zu identifizieren, welche Bildzeilen und -spalten (Fenster) für die Signalextraktion verwendet werden sollen. Dieses Fenster ist breit genug, um alle Vorsprünge zu erfassen, solange das Tier vernünftig zentriert ist, wie es bei der Verwendung unseres Tierbettes16 der Fall ist.
- Für die Koregistrierung von Thoraxschablonen wird ein kleines Bild (Schablone), das grob einen Mausthorax mit den Rippen, dem Herzen, der Lunge und der Leber darstellt, automatisch auf der ersten Röntgenprojektion mitregistriert, indem die gegenseitige Information12 zwischen der Projektion und der Schablone maximiert wird (Abbildung 5).
- Bandpass-Filter
- Ermöglicht die Erzeugung eines Bandpassfilters durch Kombination von zwei Sinc-Funktionen mit unterschiedlichen Grenzfrequenzen im temporalen Bereich17, die Tiefpassfiltern im Frequenzbereich entsprechen, wie unten beschrieben.
- Lassen Sie die Software den Bandpassfilter auf das Herzsignal mit einem Frequenzband von [300, 600] min-1 anwenden (Abbildung 6A).
HINWEIS: Das ungefilterte Herzsignal (Abbildung 6B) enthält immer noch einen signifikanten Beitrag der Atembewegung. - In ähnlicher Weise lassen Sie das Atemsignal mit einem Frequenzband von [20, 300] min-1 filtern. Die höheren Oberschwingungen werden beibehalten, um ein Signal zu erzeugen, das nicht einfach sinusförmig ist und die beiden Hauptphasen Inspiration und Exspiration klarer identifiziert.
HINWEIS: Zu diesem Zeitpunkt können sowohl respiratorische als auch kardiale Signale leicht interpoliert und als kontinuierliche Zeitfunktionen für die Berechnung der fraktionierten Phasenzahl berücksichtigt werden.
- Phasenzuordnung
- Lassen Sie die Software den Beginn jedes Zyklus (respiratorisch oder kardial) identifizieren, indem sie die Nulldurchgänge der ersten Ableitung des Signals findet. Jeder Nulldurchgang entspricht einem Maximum im Signal und markiert den Beginn eines neuen Zyklus und das Ende des vorherigen. Ein Beispiel finden Sie unter dem Atemsignal in Abbildung 7.
- Lassen Sie zu, dass jeder Projektion für jedes der beiden Signale (respiratorisch oder kardial) ein fraktionierter Phasenwert (zwischen 0 und 1) zugewiesen wird.
HINWEIS: Per Definition hat der Anfangspunkt einen Phasenbruchwert von Null (für den aktuellen Zyklus) oder eins (für den vorherigen Zyklus).
- Auswahlmasken
- Lassen Sie 12 binäre Masken (eine pro Herzphase) erstellen, um Projektionen auszuwählen, die zu jeder Phase gehören. Jede Maske enthält 21.600 Einträge (einen pro Projektion), die entweder eine 0 oder eine 1 sind, was bedeutet, dass diese Projektion abgelehnt bzw. beibehalten wird.
- Beachten Sie, dass die Software für jede Maske (Phase) entsprechend ihrer Phasenbruchnummer anzeigt, ob eine Projektion beibehalten (1) oder verworfen (0) wird. Für Phase 0 werden Projektionen mit einem kardialen Phasenwert im Intervall [0, 1/12] beibehalten. Für Phase 1 werden die kardialen Phasenwerte im Intervall [1/12, 2/12] beibehalten usw.
- Beachten Sie für jede Maske (Phase), dass alle Projektionen mit einer Atemphase von weniger als 0,15 oder mehr als 0,85 zurückgewiesen werden (0 in der Maske), da sie zur Inspirationsphase gehören, die die größte Bewegung aufweist (Abbildung 8A). Abbildung 8B zeigt die Zuordnung der Herzphase zu den Projektionen für die ersten 2 s.
HINWEIS: Bei diesem Auswahlverfahren kam es zu keiner signifikanten Verzerrung, da die Anzahl der Projektionen pro Phase mit 1.800 ± 194 relativ konstant war.
5. Bildrekonstruktion
- Rekonstruieren Sie für CT-Scans ohne Gated Bilder mit mittlerer (200 μm) oder hoher (125 μm) Auflösung unter Verwendung des Algorithmus für gefilterte Rückprojektion (Feldkamp18) mit einem Shepp-Logan-Filter.
- Für herzgesteuerte CT-Scans rekonstruiert jedes Herzphasenbild unter Verwendung des iterativen OSEM-Algorithmus13 mit 12 Teilmengen und 8 Iterationen, wobei nur ausgewählte Projektionen berücksichtigt werden, die zu einer gegebenen Phase beitragen (wie in einer binären Phasenauswahlmaske angegeben).
- Um das Rauschen zu reduzieren, wenden Sie einen Medianfilter nach der Rekonstruktion in der zeitlichen Dimension (d. h. über Bilder hinweg) und einen leichten 3D-Gaußschen Filter in der räumlichen Dimension (σ = 38 μm) an.
HINWEIS: Verwenden Sie für die respiratorische Bildrekonstruktion alle Herzphasen undeutlich. Die Strahlhärtungskorrektur (Wasserkorrektur) wird bei jeder Rekonstruktion angewendet.
6. Bildbeurteilung und Quantifizierung des Volumens des linken Ventrikels (LV)
- Öffnen Sie das CT-Bild in einem DICOM-Viewer wie Amide19 (Abbildung 9A).
- Verbessern Sie den sichtbaren Bildkontrast, indem Sie den CT-Wert (gemessen in Hounsfield Unit [HU]) auf [-500, 500] einstellen (Abbildung 9A).
- Bewertung der Bildqualität
- Zeichnen Sie einen ROI für den verkalkten Bereich, der definiert ist als der Bereich um das Maximum mit einem CT-Wert von mindestens 85 % des maximalen HU-Werts (Abbildung 9B).
- Verwenden Sie das Kontrast-Rausch-Verhältnis (CNR)20,21 als Metrik zur Beurteilung der Bildqualität und ihrer Fähigkeit, kleine Strukturen, wie z. B. kleine Verkalkungen, zu identifizieren:
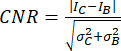 ,
,
wobei I und σ die mittlere Intensität und Standardabweichung einer Region darstellen: Verkalkung (tiefgestelltes C) oder Hintergrund (tiefgestelltes B).
- Quantifizierung des Volumens des linken Ventrikels
- Zeichnen Sie eine 3D-Freihand-ROI, um die LV in jeder Phase zu identifizieren (Abbildung 10A).
- Quantifizieren Sie das LV-Volumen durch Berechnung von Voxeln mit einem CT-Schwellenwert von 730 HU (Abbildung 10B). Ein Diagramm der LV-Volumina ist in Abbildung 10C dargestellt.
HINWEIS: Der Schwellenwert für den CT-Wert ist nicht absolut. Dies hängt vom verwendeten Kontrastmittel, der Größe des Tieres, dem Stamm, dem Gesundheitszustand und der Zeit zwischen der Injektion und der CT-Bildgebung ab. Amide ist eine frei vertriebene Software, die an unserem Institut entwickelt wurde, aber es stehen auch andere, ausgefeiltere Bildbetrachter zur Verfügung (z.B. ORS Dragonfly22).
Ergebnisse
Wir verglichen zunächst nicht-gated und gated CT-Bilder zur Visualisierung der Herzverkalkung bei Mäusen (männlich, 30-32 g). Das Mausmodell der Herzverkalkung wurde durch Induktion einer Herzschädigung durch schnelles Einfrieren und Auftauen des Herzgewebes (Kryo-Verletzung) erstellt, wie zuvor beschrieben23. Mit den nicht-gated CT-Bildgebungsprotokollen wurden Herzverkalkungen auf dem hochauflösenden Bild (125 μm, Binning 1) deutlicher identifiziert (
Diskussion
Bei der hier beschriebenen spezifischen Hardware-Implementierung handelt es sich um ein maßgeschneidertes Röntgen-CT-System, das nur an unserem Institut zu finden ist, aber der spezifische Detektor ist in kommerziell erhältlichen präklinischen Röntgen-CT-Instrumenten weit verbreitet, so dass die beschriebene Arbeit auch für andere Einrichtungen relevant ist. Dieses System ist funktional der Prototyp für zwei kommerziell erhältliche und weit verbreitete in vivo Röntgen-m...
Offenlegungen
Dr. Richard Taschereau ist Berater bei Sofie Biosciences und Xodus Imaging. Dr. Arion F. Chatziioannou ist einer der Gründer von Sofie Biosciences.
Danksagungen
Wir danken allen Mitgliedern des UCLA Crump Preclinical Imaging Technology Center für ihre Hilfe und Unterstützung. Insbesondere danken wir Mikayla Tamboline und Isabel Day für die Vorbereitung der Tiere auf die kardiale CT-Bildgebung und Sophie Shumilov für die Generierung einiger der ROIs des linken Ventrikels während der Studie. Wir danken auch Dr. Arjun Deb und Dr. Yijie Wang (UCLA) für die Bereitstellung der Mausmodelle der akuten ischämischen Herzschädigung für die mikroCT-Bildgebung der Herzverkalkung. Diese Arbeit wird durch den NIH Cancer Center Support Grant (2 P30 CA016042-44) unterstützt.
Materialien
| Name | Company | Catalog Number | Comments |
| C57BL/6J mice | Jackson Laboratory | 664 | Male, 8 weeks old, 24-26 g |
| Dexela camera | Varex | 1512 | Detector, 20 ms exposure, 74.8/149.6 µm pixel |
| VivoVist | Nanoprobes | 1301-5X0.25ML | CT Contrast agent |
| X-ray source | Moxtek | TUB00082 | 50 kV peak, 200 µA, 1.0 mm-thick Al filter |
Referenzen
- Schambach, S. J., Bag, S., Schilling, L., Groden, C., Brockmann, M. A. Application of micro-CT in small animal imaging. Methods. 50 (1), 2-13 (2010).
- Koba, W., Jelicks, L. A., Fine, E. J. MicroPET/SPECT/CT imaging of small animal models of disease. Am J Pathol. 182 (2), 319-324 (2013).
- Hutchins, G. D., Miller, M. A., Soon, V. C., Receveur, T. Small animal PET imaging. ILAR J. 49 (1), 54-65 (2008).
- Franc, B. L., Acton, P. D., Mari, C., Hasegawa, B. H. Small-animal SPECT and SPECT/CT: important tools for preclinical investigation. J Nucl Med. 49 (10), 1651-1663 (2008).
- Badea, C. T. H. L., Hedlund, L. W., Johnson, G. A. Micro-CT with respiratory and cardiac gating: Micro-CT with respiratory and cardiac gating. Med Phys. 31 (12), 3324-3329 (2004).
- Guo, X., Johnston, S. M., Qi, Y., Johnson, G. A., Badea, C. T. 4D micro-CT using fast prospective gating. Phys Med Biol. 57 (1), 257 (2011).
- Drangova, M., Ford, N. L., Detombe, S. A., Wheatley, A. R., Holdsworth, D. W. Fast retrospectively gated quantitative four-dimensional (4D) cardiac micro computed tomography imaging of free-breathing mice. Invest Radiol. 42 (2), 85-94 (2007).
- Blocker, S. J., Holbrook, M. D., Mowery, Y. M., Sullivan, D. C., Badea, C. T. The impact of respiratory gating on improving volume measurement of murine lung tumors in micro-CT imaging. PLoS One. 15 (2), e0225019 (2020).
- Bartling, S. H., et al. Retrospective motion gating in small animal CT of mice and rats. Invest Radiol. 42 (10), 704-714 (2007).
- Kuntz, J., et al. Fully automated intrinsic respiratory and cardiac gating for small animal CT. Phys Med Biol. 55 (7), 2069 (2010).
- Hahn, A., Sauppe, S., Lell, M., Kachelrieß, M. Automatic intrinsic cardiac and respiratory gating from cone-beam CT scans of the thorax region. SPIE Proc. Med Imaging. 9783, 200-205 (2016).
- Maes, F., Vandermeulen, D., Suetens, P. Medical image registration using mutual information. Proc IEEE. 91 (10), 1699-1722 (2003).
- Sheng, J., Chen, B., Ma, Y., Shi, Y. A novel reconstruction approach combining OSEM and split Bregman method for low dose CT. Biomedical Signal Processing and Control. 62, 102095 (2020).
- Romdhane, H., Cherni, M. A., Sallem, D. B. On the efficiency of OSEM algorithm for tomographic lung CT images reconstruction. 2016 Second International Image Processing, Applications and Systems (IPAS). , (2016).
- Ewald, A. J., Werb, Z., Egeblad, M. Monitoring of vital signs for long-term survival of mice under anesthesia. Cold Spring Harb Protoc. 2, 5563 (2011).
- Suckow, C., et al. Multimodality rodent imaging chambers for use under barrier conditions with gas anesthesia. Mol Imaging Biol. 11 (2), 100-106 (2009).
- Owen, M. . Practical signal processing. , 81 (2007).
- Feldkamp, L. A., Davis, L. C., Kress, J. W. Practical cone-beam algorithm. J Opt Soc Am. A-optics Image Sci Vision. 1 (6), 612-619 (1984).
- Loening, A. M., Gambhir, S. S. AMIDE: A free software tool for multimodality medical image analysis. Mol Imaging. 2 (3), 131-137 (2003).
- Rodriguez-Molares, A., et al. The generalized contrast-to-noise ratio: A formal definition for lesion detectability. IEEE Trans Ultrason Ferroelectr Freq Control. 67 (4), 745-759 (2020).
- Patterson, M. S., Foster, F. S. The improvement and quantitative assessment of B-mode images produced by an annular array/cone hybrid. Ultrason Imaging. 5 (3), 195-213 (1983).
- . Dragonfly 2022.2 Available from: https://www.theobjects.com/dragonfly (2022)
- Pillai, I. C. L., et al. Cardiac fibroblasts adopt osteogenic fates and can be targeted to attenuate pathological heart calcification. Cell Stem Cell. 20 (2), 218-232 (2017).
- Li, S., et al. A humanized monoclonal antibody targeting an ectonucleotidase rescues cardiac metabolism and heart function after myocardial infarction. Cell Rep Med. 5 (11), 101795 (2024).
- Nadkarni, R., Clark, D. P., Allphin, A. J., Badea, C. T. Investigating deep learning strategies for fast denoising of 5D cardiac photon-counting micro-CT images. Phys Med Biol. 69 (20), 205010 (2024).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten