È necessario avere un abbonamento a JoVE per visualizzare questo. Accedi o inizia la tua prova gratuita.
Method Article
Gating cardiaco retrospettivo con un prototipo di tomografo computerizzato a raggi X per piccoli animali
In questo articolo
Riepilogo
Forniamo una descrizione completa del metodo di gating cardiaco retrospettivo intrinseco del CrumpCAT, un prototipo di scanner per tomografia computerizzata (TC) a raggi X per piccoli animali progettato e costruito presso il nostro istituto di ricerca.
Abstract
Il CrumpCAT è un prototipo di scanner per tomografia computerizzata (CT) a raggi X per piccoli animali sviluppato presso il nostro istituto di ricerca. Il rivelatore CMOS con un frame rate massimo di 29 Hz e sorgenti di raggi X al tungsteno simili con energie comprese tra 50 kVp e 80 kVp sono ampiamente utilizzati negli strumenti CT a raggi X preclinici disponibili in commercio. Ciò rende il lavoro descritto molto rilevante per altre istituzioni, nonostante la saggezza generalmente percepita che questi rilevatori non siano adatti per controllare l'alta frequenza cardiaca dei topi (~600 battiti/min). Lo scanner è dotato di imaging a media (200 μm) e alta risoluzione (125 μm), fluoroscopia, gating respiratorio retrospettivo e gating cardiaco retrospettivo, con ricostruzione iterativa o filtrata dell'immagine a proiezione. Tra queste caratteristiche, il gating cardiaco è la funzione più utile per studiare le funzioni cardiache in vivo, in quanto elimina efficacemente la sfocatura dell'immagine causata dal movimento respiratorio e cardiaco.
Qui, descriviamo il nostro metodo per l'imaging TC cardioscopico retrospettivo intrinseco preclinico, volto a far progredire la ricerca sulla funzione cardiaca in vivo e sull'analisi della struttura. Il metodo del gating cardiaco acquisisce un gran numero di proiezioni al più breve tempo di esposizione pratico (~20 ms) e quindi estrae retrospettivamente i segnali respiratori e cardiaci dai cambiamenti temporali nelle sequenze di proiezione grezze. Questi segnali vengono utilizzati per respingere le proiezioni appartenenti alla fase di inspirazione ad alta velocità di movimento del ciclo respiratorio e per dividere le proiezioni rimanenti in 12 gruppi, ciascuno corrispondente a una fase del ciclo cardiaco. Ogni gruppo viene ricostruito in modo indipendente utilizzando un metodo iterativo per produrre un'immagine volumetrica per ogni fase cardiaca, ottenendo un set di dati quadridimensionale (4D).
Queste immagini di fase possono essere analizzate collettivamente o individualmente, consentendo una valutazione dettagliata della funzione cardiaca. Abbiamo dimostrato l'efficacia di entrambi gli approcci della funzione di gating cardiaco dello scanner prototipo attraverso risultati di imaging in vivo rappresentativi.
Introduzione
La ricerca sui piccoli animali impiega spesso una combinazione di modalità di imaging non invasive, con la tomografia computerizzata a raggi X (TC), che è una scelta importante grazie alla sua maturità, economicità, velocità 1,2 e capacità di fornire informazioni complementari insieme ad altre modalità come la tomografia a emissione di positroni (PET)2,3 e la tomografia computerizzata a emissione di fotoni singoli (SPECT)2,4. Tuttavia, come altre tecniche di imaging, la TC è suscettibile agli artefatti fisiologici del movimento causati dal battito cardiaco o dalla respirazione, che introducono sfocatura e limitano l'accuratezza della ricerca.
Per affrontare questa limitazione, l'offuscamento del movimento respiratorio e cardiaco può essere mitigato attraverso una tecnica nota come gating 5,6,7,8, in cui l'acquisizione dei dati è sincronizzata con fasi specifiche del ciclo cardiaco o respiratorio (o gates). Un approccio per raggiungere questo obiettivo, noto come gating prospettico 3,6, prevede il collegamento di sensori all'animale per fornire segnali di gating in tempo reale a uno scanner compatibile. Sebbene efficace, questo metodo è laborioso e richiede tempo, in particolare quando si collegano sensori al torace e alle zampe di piccoli animali come i topi, limitando così la scala degli studi. In alternativa, il gating retrospettivo intrinseco 7,9,10,11 prevede l'acquisizione di dati di serie temporali senza l'uso di sensori ma identificando le caratteristiche nei dati che consentono l'ordinamento retrospettivo dei risultati in base alla loro fase nel ciclo cardiaco o respiratorio. Questo approccio offre risultati paragonabili al gating prospettico, ma senza la necessità di hardware aggiuntivo o lo sforzo necessario per il collegamento del sensore di impulsi e, pertanto, semplifica notevolmente i protocolli sperimentali.
Nel nostro metodo per l'imaging TC cardiaco preclinico, utilizziamo il gating retrospettivo intrinseco per estrarre i cicli respiratori e cardiaci dalle variazioni di ampiezza nelle regioni nelle proiezioni a raggi X che mostrano i cambiamenti più significativi tra i fotogrammi successivi. Per facilitare questo processo, un modello toracico di topo viene co-registrato sulla prima proiezione posteroanteriore utilizzando l'informazione reciproca12. Una volta posizionato il modello, le intensità dei pixel in una finestra vicino al diaframma vengono sommate per generare un segnale respiratorio surrogato, mentre quelle vicino al miocardio vengono sommate per derivare il segnale cardiaco surrogato. Questi segnali vengono quindi filtrati passa-banda nel dominio del tempo e a ciascun fotogramma del set di dati viene assegnato un numero di fase frazionario (compreso tra 0 e 1) in base alla fase respiratoria e cardiaca. Ciò consente la selezione o la reiezione delle proiezioni in base ai loro valori di fase. Tipicamente, vengono mantenuti i fotogrammi corrispondenti alla fase di fine-espirazione del ciclo respiratorio (0,15 ≤ fase < 0,85), mentre quelli della fase di inspirazione, dove il movimento è più pronunciato, vengono scartati. I fotogrammi rimanenti sono raggruppati in 12 fasi cardiache, ciascuna delle quali rappresenta 1/12 (0,083) del ciclo cardiaco e sono ricostruiti in immagini 3D utilizzando un metodo iterativo (Ordered Subset Expectation Maximization [OSEM])13,14. L'intero processo è riassunto nella Figura 1.
Protocollo
I protocolli sperimentali sugli animali sono stati esaminati e approvati dall'Institutional Animal Care and Use Committee dell'Università della California, Los Angeles (UCLA). In questo protocollo sono stati utilizzati topi C57BL/6J (8 settimane di età, maschi, 24-26 g). Lo scanner TC utilizzato in questo studio è il CrumpCAT (Figura 2), un prototipo sviluppato presso il nostro istituto di ricerca per la ricerca preclinica, che ci fornisce il controllo e la flessibilità necessari per ottimizzare i protocolli di acquisizione e ricostruzione. Il metodo presuppone che i topi anestetizzati abbiano una frequenza cardiaca non superiore a 600 battiti/min e una frequenza respiratoria compresa tra 20 e 180 respiri/min15.
1. Impostazioni dell'attrezzatura
- Far funzionare la sorgente di raggi X a una tensione di picco di 50 kVp con corrente continua di 200 μA.
- Impostare il pixel binning della telecamera a raggi X su 2 e il tempo di esposizione sul valore pratico più breve (20 ms di tempo di esposizione più 14 ms di lettura per un tempo di campionamento totale di 34 ms).
NOTA: l'opzione no-binning (binning 1) ha un frame rate più lento e, pertanto, non può essere utilizzata per le acquisizioni gated. Il frame rate massimo è di circa 30 fotogrammi/s.
2. Preparazione degli animali
- Anestetizzare i topi con una miscela di ossigeno e gas isoflurano al 2,0% di concentrazione.
- Tenere gli animali sotto anestesia per 10 minuti prima della TAC, per garantire che i segni vitali siano stabili e che gli animali siano rilassati per evitare qualsiasi movimento del corpo durante la scansione. Giudica la profondità dell'anestesia di un topo dalla mancanza del riflesso di pizzicamento delle dita dei piedi quando pizzichi saldamente la fettuccia tra le dita dei piedi con un'unghia.
- Affinché l'imaging cardiaco dipendente possa visualizzare le camere cardiache, iniettare per via endovenosa 100 μL di mezzo di contrasto CT attraverso la vena caudale immediatamente prima dell'imaging TC (Figura 3A).
- Posizionare il topo anestetizzato all'interno della camera di imaging TC (Figura 3B).
3. Acquisizione dati
- Accendi il software CrumpCAT.
- Imaging TC non gated ad alta risoluzione (125 μm, binning 1) (Figura 4A)
- Immettere un ID studio nell'interfaccia utente.
- Selezionare Mouse Hi-Res nel menu a discesa Protocollo .
- Fare clic sul pulsante Scansione nell'interfaccia utente per acquisire 720 proiezioni con un tempo di esposizione di 80 ms/proiezione.
- Imaging TC non gated con risoluzione media (200 μm, binning 2) (Figura 4B)
- Immettere un ID studio nell'interfaccia utente.
- Selezionare Mouse Standard nel menu a discesa Protocollo .
- Fare clic sul pulsante Scansione sull'interfaccia utente per acquisire 720 proiezioni con un tempo di esposizione di 100 ms/proiezione.
- Imaging TC con gate (200 μm, binning 2) (Figura 4C)
- Immettere un ID studio nell'interfaccia utente.
- Selezionare Cardiac Gating nel menu a discesa Protocollo .
- Fare clic sul pulsante Scansione nell'interfaccia utente e acquisire 21.600 proiezioni con un tempo di esposizione di 20 ms/proiezione.
4. Pretrattamento dei dati
NOTA: le fasi di pre-elaborazione sono necessarie solo per le acquisizioni controllate. Tutti questi passaggi vengono eseguiti automaticamente dal software di ricostruzione e non è richiesto l'intervento dell'operatore.
- Estrazione del segnale
- Per la co-registrazione del modello del torace, lasciare che una piccola immagine (modello) che rappresenti grossolanamente un torace di topo con le costole, il cuore, i polmoni e il fegato venga automaticamente co-registrata alla prima proiezione a raggi X, massimizzando l'informazione reciproca12 tra la proiezione e il modello (Figura 5).
NOTA: sul modello vengono eseguite solo le operazioni di traduzione e il modello co-registrato viene utilizzato per identificare le regioni di interesse (ROI) su tutte le proiezioni. - Per l'estrazione del segnale respiratorio, lasciare che una ROI rettangolare (ROI-1) sia designata nel modello per rappresentare il diaframma e il segnale respiratorio da generare sommando le intensità dei pixel all'interno della ROI-1 per ciascuna proiezione (Figura 5).
- Per l'estrazione del segnale cardiaco, si lasci designare una seconda ROI rettangolare (ROI-2) nel modello vicino al cuore e il segnale cardiaco generato sommando le intensità dei pixel in ROI-2 per ciascuna proiezione (Figura 5).
NOTA: Il modello del torace viene co-registrato solo alla prima proiezione per identificare quali righe e colonne dell'immagine (finestra) devono essere utilizzate per l'estrazione del segnale. Quella finestra è abbastanza ampia da comprendere tutte le sporgenze purché l'animale sia ragionevolmente centrato, come nel caso di quando si utilizza la nostra cucciaper animali 16.
- Per la co-registrazione del modello del torace, lasciare che una piccola immagine (modello) che rappresenti grossolanamente un torace di topo con le costole, il cuore, i polmoni e il fegato venga automaticamente co-registrata alla prima proiezione a raggi X, massimizzando l'informazione reciproca12 tra la proiezione e il modello (Figura 5).
- Filtro passa-banda
- Consentire la generazione di un filtro passa-banda combinando due funzioni Sinc con diverse frequenze di taglio nel dominio temporale17, che corrispondono ai filtri passa-basso nel dominio della frequenza, come descritto di seguito.
- Lasciare che il software applichi il filtro passa-banda sul segnale cardiaco utilizzando una banda di frequenza di [300, 600] min-1 (Figura 6A).
NOTA: Il segnale cardiaco non filtrato (Figura 6B) contiene ancora un contributo significativo del movimento respiratorio. - Allo stesso modo, filtrare il segnale respiratorio utilizzando una banda di frequenza di [20, 300] min-1. Le armoniche più alte vengono preservate per produrre un segnale che non sia semplicemente sinusoidale e identifichi più chiaramente le due fasi principali: inspirazione ed espirazione.
NOTA: A questo punto, sia i segnali respiratori che quelli cardiaci possono essere facilmente interpolati e considerati funzioni continue del tempo ai fini del calcolo del numero di fase frazionario.
- Assegnazione delle fasi
- Lascia che il software identifichi l'inizio di ogni ciclo (respiratorio o cardiaco) trovando gli zero-crossing della derivata prima del segnale. Ogni passaggio per lo zero corrisponde a un massimo nel segnale e segna l'inizio di un nuovo ciclo e la fine di quello precedente. Per un esempio, vedere il segnale respiratorio nella Figura 7.
- Consentire l'assegnazione di un valore di fase frazionario (compreso tra 0 e 1) a ciascuna proiezione per ciascuno dei due segnali (respiratorio o cardiaco).
NOTA: Per definizione, il punto iniziale ha un valore di fase frazionario pari a zero (per il ciclo corrente) o uno (per il ciclo precedente).
- Maschere di selezione
- Lasciare che vengano create 12 maschere binarie (una per fase cardiaca) per selezionare le proiezioni appartenenti a ciascuna fase. Ogni maschera contiene 21.600 voci (una per proiezione) che sono rispettivamente uno 0 o un 1, ovvero rifiuta o mantiene tale proiezione.
- Per ogni maschera (fase), osservare che il software indichi se una proiezione viene mantenuta (1) o scartata (0) in base al suo numero di fase frazionario. Per la fase 0, vengono mantenute le proiezioni con un valore di fase cardiaca nell'intervallo [0, 1/12]. Per la fase 1, vengono mantenuti i valori della fase cardiaca nell'intervallo [1/12, 2/12] e così via.
- Per ogni maschera (fase), si noti che tutte le proiezioni con una fase respiratoria inferiore a 0,15 o superiore a 0,85 vengono scartate (0 nella maschera) perché appartengono alla fase di inspirazione, che ha il movimento più grande (Figura 8A). La Figura 8B mostra l'assegnazione della fase cardiaca alle proiezioni per i primi 2 s.
NOTA: Non è stata introdotta alcuna distorsione significativa con questo processo di selezione poiché il numero di proiezioni per fase era relativamente costante a 1.800 ± 194.
5. Ricostruzione dell'immagine
- Per le scansioni TC non gated, ricostruire le immagini a risoluzione media (200 μm) o alta (125 μm) utilizzando l'algoritmo di retroproiezione filtrata (Feldkamp18) con un filtro Shepp-Logan.
- Per le scansioni TC cardio-dipendenti, ricostruire ogni immagine della fase cardiaca utilizzando l'algoritmo iterativo OSEM13, con 12 sottogruppi e 8 iterazioni, tenendo conto solo delle proiezioni selezionate che contribuiscono a una data fase (come indicato in una maschera binaria di selezione di fase).
- Per ridurre il rumore, applicare un filtro mediano post-ricostruzione nella dimensione temporale (cioè attraverso le immagini) e un filtro gaussiano 3D leggero nella dimensione spaziale (σ = 38 μm).
NOTA: Per la ricostruzione dell'immagine respiratoria, utilizzare tutte le fasi cardiache indistintamente. La correzione dell'indurimento del fascio (correzione dell'acqua) viene applicata ad ogni ricostruzione.
6. Valutazione dell'immagine e quantificazione del volume del ventricolo sinistro (LV)
- Aprire l'immagine CT in un visualizzatore DICOM come Amide19 (Figura 9A).
- Migliorare il contrasto dell'immagine visibile impostando l'intervallo del valore CT (misurato in unità Hounsfield [HU]) su [-500, 500] (Figura 9A).
- Valutazione della qualità dell'immagine
- Disegnare un ROI per la regione calcificata, che è definita come la regione intorno al massimo con un valore CT maggiore o uguale all'85% del valore HU massimo (Figura 9B).
- Utilizzare il rapporto contrasto/rumore (CNR)20,21 come metrica per valutare la qualità dell'immagine e la sua capacità di identificare piccole strutture, come piccole calcificazioni:
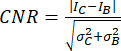 ,
,
dove I e σ rappresentano l'intensità media e la deviazione standard di una regione: calcificazione (pedice C) o fondo (pedice B).
- Quantificazione del volume del ventricolo sinistro
- Disegna un ROI 3D-Freehand per identificare il LV in ogni fase (Figura 10A).
- Quantificare il volume LV calcolando i voxel con un valore CT di soglia di 730 HU (Figura 10B). Un grafico dei volumi LV è mostrato nella Figura 10C.
NOTA: La soglia del valore CT non è assoluta; dipende dal mezzo di contrasto utilizzato, dalle dimensioni dell'animale, dal ceppo, dalle condizioni di salute e dal tempo tra l'iniezione e l'imaging TC. Amide è un software distribuito gratuitamente sviluppato presso il nostro istituto, ma sono disponibili altri visualizzatori di immagini più sofisticati (ad esempio, ORS Dragonfly22).
Risultati
Per prima cosa abbiamo confrontato le immagini TC non gated e gated per visualizzare la calcificazione cardiaca nei topi (maschi, 30-32 g). Il modello murino di calcificazione cardiaca è stato creato inducendo un danno cardiaco mediante rapido congelamento-scongelamento del tessuto cardiaco (criolesione), come descritto in precedenza23. Con i protocolli di imaging TC non gated, le calcificazioni cardiache sono state identificate più chiaramente sull'immagine ad ...
Discussione
L'implementazione hardware specifica qui descritta è un sistema CT a raggi X su misura unico per il nostro istituto, ma il rivelatore specifico è ampiamente utilizzato negli strumenti CT a raggi X preclinici disponibili in commercio, rendendo il lavoro descritto rilevante per altre istituzioni. Questo sistema è funzionalmente il prototipo di due sottosistemi microCT a raggi X in vivo disponibili in commercio e ampiamente utilizzati integrati
negli scanner PET/CT preclinici...
Divulgazioni
Il Dr. Richard Taschereau è consulente di Sofie Biosciences e Xodus Imaging. Il Dr. Arion F. Chatziioannou è uno dei fondatori di Sofie Biosciences.
Riconoscimenti
Ringraziamo tutti i membri dell'UCLA Crump Preclinical Imaging Technology Center per il loro aiuto e supporto. In particolare, ringraziamo Mikayla Tamboline e Isabel Day per aver preparato gli animali per l'imaging TC cardiaco e ringraziamo Sophie Shumilov per aver generato alcuni dei ROI del ventricolo sinistro durante lo studio. Ringraziamo anche i dottori Arjun Deb e Yijie Wang (UCLA) per aver fornito i modelli murini di danno cardiaco ischemico acuto per l'imaging microCT della calcificazione cardiaca. Questo lavoro è supportato dal NIH Cancer Center Support Grant (2 P30 CA016042-44).
Materiali
| Name | Company | Catalog Number | Comments |
| C57BL/6J mice | Jackson Laboratory | 664 | Male, 8 weeks old, 24-26 g |
| Dexela camera | Varex | 1512 | Detector, 20 ms exposure, 74.8/149.6 µm pixel |
| VivoVist | Nanoprobes | 1301-5X0.25ML | CT Contrast agent |
| X-ray source | Moxtek | TUB00082 | 50 kV peak, 200 µA, 1.0 mm-thick Al filter |
Riferimenti
- Schambach, S. J., Bag, S., Schilling, L., Groden, C., Brockmann, M. A. Application of micro-CT in small animal imaging. Methods. 50 (1), 2-13 (2010).
- Koba, W., Jelicks, L. A., Fine, E. J. MicroPET/SPECT/CT imaging of small animal models of disease. Am J Pathol. 182 (2), 319-324 (2013).
- Hutchins, G. D., Miller, M. A., Soon, V. C., Receveur, T. Small animal PET imaging. ILAR J. 49 (1), 54-65 (2008).
- Franc, B. L., Acton, P. D., Mari, C., Hasegawa, B. H. Small-animal SPECT and SPECT/CT: important tools for preclinical investigation. J Nucl Med. 49 (10), 1651-1663 (2008).
- Badea, C. T. H. L., Hedlund, L. W., Johnson, G. A. Micro-CT with respiratory and cardiac gating: Micro-CT with respiratory and cardiac gating. Med Phys. 31 (12), 3324-3329 (2004).
- Guo, X., Johnston, S. M., Qi, Y., Johnson, G. A., Badea, C. T. 4D micro-CT using fast prospective gating. Phys Med Biol. 57 (1), 257 (2011).
- Drangova, M., Ford, N. L., Detombe, S. A., Wheatley, A. R., Holdsworth, D. W. Fast retrospectively gated quantitative four-dimensional (4D) cardiac micro computed tomography imaging of free-breathing mice. Invest Radiol. 42 (2), 85-94 (2007).
- Blocker, S. J., Holbrook, M. D., Mowery, Y. M., Sullivan, D. C., Badea, C. T. The impact of respiratory gating on improving volume measurement of murine lung tumors in micro-CT imaging. PLoS One. 15 (2), e0225019 (2020).
- Bartling, S. H., et al. Retrospective motion gating in small animal CT of mice and rats. Invest Radiol. 42 (10), 704-714 (2007).
- Kuntz, J., et al. Fully automated intrinsic respiratory and cardiac gating for small animal CT. Phys Med Biol. 55 (7), 2069 (2010).
- Hahn, A., Sauppe, S., Lell, M., Kachelrieß, M. Automatic intrinsic cardiac and respiratory gating from cone-beam CT scans of the thorax region. SPIE Proc. Med Imaging. 9783, 200-205 (2016).
- Maes, F., Vandermeulen, D., Suetens, P. Medical image registration using mutual information. Proc IEEE. 91 (10), 1699-1722 (2003).
- Sheng, J., Chen, B., Ma, Y., Shi, Y. A novel reconstruction approach combining OSEM and split Bregman method for low dose CT. Biomedical Signal Processing and Control. 62, 102095 (2020).
- Romdhane, H., Cherni, M. A., Sallem, D. B. On the efficiency of OSEM algorithm for tomographic lung CT images reconstruction. 2016 Second International Image Processing, Applications and Systems (IPAS). , (2016).
- Ewald, A. J., Werb, Z., Egeblad, M. Monitoring of vital signs for long-term survival of mice under anesthesia. Cold Spring Harb Protoc. 2, 5563 (2011).
- Suckow, C., et al. Multimodality rodent imaging chambers for use under barrier conditions with gas anesthesia. Mol Imaging Biol. 11 (2), 100-106 (2009).
- Owen, M. . Practical signal processing. , 81 (2007).
- Feldkamp, L. A., Davis, L. C., Kress, J. W. Practical cone-beam algorithm. J Opt Soc Am. A-optics Image Sci Vision. 1 (6), 612-619 (1984).
- Loening, A. M., Gambhir, S. S. AMIDE: A free software tool for multimodality medical image analysis. Mol Imaging. 2 (3), 131-137 (2003).
- Rodriguez-Molares, A., et al. The generalized contrast-to-noise ratio: A formal definition for lesion detectability. IEEE Trans Ultrason Ferroelectr Freq Control. 67 (4), 745-759 (2020).
- Patterson, M. S., Foster, F. S. The improvement and quantitative assessment of B-mode images produced by an annular array/cone hybrid. Ultrason Imaging. 5 (3), 195-213 (1983).
- . Dragonfly 2022.2 Available from: https://www.theobjects.com/dragonfly (2022)
- Pillai, I. C. L., et al. Cardiac fibroblasts adopt osteogenic fates and can be targeted to attenuate pathological heart calcification. Cell Stem Cell. 20 (2), 218-232 (2017).
- Li, S., et al. A humanized monoclonal antibody targeting an ectonucleotidase rescues cardiac metabolism and heart function after myocardial infarction. Cell Rep Med. 5 (11), 101795 (2024).
- Nadkarni, R., Clark, D. P., Allphin, A. J., Badea, C. T. Investigating deep learning strategies for fast denoising of 5D cardiac photon-counting micro-CT images. Phys Med Biol. 69 (20), 205010 (2024).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneEsplora altri articoli
This article has been published
Video Coming Soon