Method Article
Otimização da reação de transcrição in vitro para produção de mRNA usando monitoramento cromatográfico em linha
* Estes autores contribuíram igualmente
Neste Artigo
Resumo
A otimização da transcrição in vitro (IVT) é fundamental para a produção econômica de mRNA. Este protocolo detalha um método cromatográfico analítico para análise em linha da reação IVT, monitorando a depleção de NTP e a produção de mRNA nos modos de batelada ou batelada alimentada, aplicável a várias modalidades de RNA, aumentando a produtividade e reduzindo custos.
Resumo
A reação de transcrição in vitro (IVT) é uma síntese enzimática complexa e multicomponente de mRNA a partir de um molde de DNA linear, catalisada por uma RNA polimerase, por exemplo, T7. Devido ao alto custo dos reagentes de IVT, a IVT é uma etapa crítica no processo de produção de substâncias medicamentosas de mRNA e tem sido o foco de intensa otimização no campo. Para diminuir o custo de produção de mRNA, os reagentes devem ser utilizados de forma otimizada. A otimização eficaz requer uma compreensão abrangente do impacto de reagentes individuais na cinética da reação, ou seja, o consumo de trifosfatos de nucleosídeos (NTPs) e a produção de mRNA. Tradicionalmente, técnicas analíticas de ponto final de baixo rendimento têm sido usadas para a análise de mRNA. Embora esses métodos forneçam informações valiosas sobre o conteúdo de mRNA, análises em linha e quase em tempo real são necessárias para entender completamente a reação de IVT. Demonstramos como um método analítico de cromatografia líquida que separa NTPs, bem como pDNA e mRNA, pode ser usado quase em tempo real para estudar a cinética da reação IVT. Com o conhecimento da influência de diferentes componentes de IVT na cinética e no rendimento, a análise cromatográfica rápida pode ser usada para converter a reação de IVT em lote em um modo de lote alimentado que aumenta ainda mais a produtividade e reduz o custo geral da reação.
Introdução
A pandemia de COVID-19 catalisou uma revolução sem precedentes na biomedicina, levando ao rápido desenvolvimento e autorização de vacinas baseadas em mRNA, como a Comirnaty da BioNTech/Pfizer e a Spikevax da Moderna, tanto na Europa quanto nos EUA 1,2,3. A impressionante eficácia e o rápido desenvolvimento dessas vacinas destacaram o imenso potencial terapêutico da tecnologia de mRNA, não apenas para doenças infecciosas, mas também para imunoterapias contra o câncer, terapias de reposição de proteínas, medicina regenerativa e reprogramação celular 4,5. Esta era, muitas vezes referida como a revolução do mRNA, ressalta a necessidade de processos de produção de mRNA eficientes e econômicos.
A produção de mRNA envolve múltiplas operações unitárias, sendo a reação de transcrição in vitro (IVT) uma operação unitária crítica e mais dispendiosa 6,7,8. A reação IVT sintetiza mRNA a partir de um molde de DNA usando RNA polimerase, tipicamente RNA polimerase T7 e trifosfatos de nucleosídeos (NTPs) na presença de íons de magnésio. O processo é relativamente simples e permite a produção de grandes quantidades de mRNA em um curto espaço de tempo, alcançando rendimentos de reação de 2-5 g/L em poucas horas e até 14 g/L em alguns relatórios 9,10,11. No entanto, a otimização do rendimento da reação IVT é crucial para reduzir os custos de produção e garantir a escalabilidade das vacinas e terapêuticas de mRNA12 e, devido às diferenças de sequência que influenciam o consumo de NTP, pode ser necessária para cada construto ou família de construtos.
A reação geralmente é realizada como um processo em lote, mas avanços recentes no processamento de lotes alimentados e análises em linha abriram novos caminhos para otimizar a produção de mRNA 7,13. As reações em batelada alimentada, que envolvem um bolus ou adição contínua de reagentes, podem potencialmente estender os tempos de reação e aumentar os rendimentos, evitando a inibição do substrato e a degradação do produto dependente de cofatores14,15.
O desenvolvimento de análises rápidas em linha tem sido um avanço significativo no campo da IVT, permitindo o monitoramento dos principais componentes da reação, como NTPs e mRNA, com atraso analítico mínimo 13,16, adicionando uma dimensão adicional ao monitoramento de IVT, que normalmente se concentra apenas na concentração de mRNA17. Tradicionalmente, os mRNAs têm sido analisados usando técnicas como eletroforese em gel de poliacrilamida (PAGE), eletroforese em gel de agarose ou eletroforese capilar. Esses são métodos de ponto final e, portanto, não podem ser usados para monitoramento de IVT em tempo real. Uma alternativa à análise descrita neste protocolo são os métodos que usam aptâmero de RNA iluminado e pares de corantes de fluorescência. Neste método, o aptâmero de RNA é marcado com RNA e incubado com um corante de fluorescência iluminado. A atividade de transcrição pode então ser visualizada pela intensidade de fluorescência17. Isso permite a análise em tempo real da quantidade e qualidade do mRNA transcrito durante o IVT 21, mas não permite o monitoramento simultâneo de outros componentes do IVT, por exemplo, NTPs. Outra alternativa que potencialmente permite a quantificação em tempo real de NTP e mRNA é a espectroscopia Raman18, mas até o momento, nenhum relatório mostrou sua utilidade para o monitoramento de IVT, sugerindo que ainda é necessária uma otimização adicional do método para obter a seletividade e sensibilidade necessárias.
O desenvolvimento contínuo de análises mais rápidas e seletivas suporta a otimização do IVT para maior rendimento nos modos de lote e lote alimentado. A reação permanece um processo complexo e multiparamétrico com múltiplos fatores de interação, para os quais os ótimos podem diferir entre os tipos de construção (por exemplo, mRNA vs saRNA). A concentração e a proporção de íons Mg2+ e NTPs são particularmente influentes, e seus níveis ideais devem ser cuidadosamente equilibrados para maximizar o rendimento do mRNA, minimizando a formação de RNA de fita dupla (dsRNA), um potente estimulante do sistema imunológico inato19,20. Recentemente, a alimentação de UTP em níveis de estado estacionário foi relatada como uma abordagem para reduzir a formação de dsRNA21. O monitoramento dos níveis de UTP quase em tempo real adicionaria um nível adicional de controle de processo.
Neste protocolo, demonstramos como o monitoramento em linha da reação IVT pode aumentar o rendimento da produção de mRNA no modo batelada e batelada alimentada.
Protocolo
1. Preparação do tampão
NOTA: Todos os tampões devem ser preparados sem RNase, o que significa que todos os produtos químicos e vidraria devem ser usados apenas para trabalhos sem RNase e manuseados com precaução. A água utilizada para a preparação do tampão e a limpeza dos artigos de vidro deve ser certificada como isenta de nuclease. Antes de realizar um experimento, todas as superfícies de trabalho e vidraria devem ser pulverizadas com um reagente de descontaminação que elimine as RNases. O reagente de descontaminação deve ser cuidadosamente lavado com água livre de RNase antes de usar vidraria/área de trabalho. Se possível, use consumíveis estéreis e de uso único.
- Preparação de tampões IVT: prepare 10x tampão IVT adicionando 400 mM Tris, 10 mM DTT, 20 mM espermidina, pH 7,9. Preparar um tampão de têmpera contendo 100 mM de EDTA, pH 8. Filtre cada tampão através de um filtro PES de 0,22 μm.
- Preparação de fases móveis: preparar a fase móvel A (MPA) adicionando 50 mM HEPES, pH 7,0, fase móvel B (MPB) adicionando 50 mM HEPES, 100 mM Na4P2O7, pH 8,3, fase móvel C (MPC) adicionando 0,1 M NaOH, 1 M NaCl, fase móvel D (MPD) adicionando 0,5 M HEPES, pH 7,0. Filtre cada tampão através de um filtro PES de 0,22 μm.
2. Preparação da reação de transcrição in vitro (IVT)
NOTA 1: A reação IVT pode ser uma reação em lote, onde todos os reagentes são adicionados no início, e a reação é interrompida depois que os NTPs são esgotados / a produção de mRNA atinge um platô, ou pode ser uma reação em lote alimentado, onde os NTPs esgotados são reabastecidos com NTPs adicionais e com isso a produção de mRNA aumenta adicionalmente. O molde linear do pDNA usado na reação de IVT pode ser obtido usando a enzima da limitação que cliva logo após a sequência poli(A) no ADN do plasmídeo ou pela reação de PCR. Em ambos os casos, a reação deve ser seguida de purificação, cromatográfica ou usando kits comerciais de purificação de DNA.
- Reação de IVT em batelada
- Defina o termobloco para 37 °C e 300 rpm. Descongelar todos os reagentes enumerados no quadro 1 , com excepção das enzimas, e pré-aquecê-los a 37 °C no termobloco. Prepare a mistura de NTP 25 mM usando a solução-mãe 100 mM de ATP, CTP, UTP e GTP.
- Enquanto os reagentes estão descongelando, prepare alíquotas de amostra para o experimento de curso de tempo. Pegue tubos estéreis de 0,5 mL e pipete 2 μL de EDTA 100 mM em cada tubo. Marque cada tubo com um número IVT designado (por exemplo, IVT001, IVT002, etc.) e ponto de tempo (por exemplo, 0 min, 15 min, 30 min, 60 min, etc.).
- Quando os reagentes forem descongelados e mantidos a 37 °C por aproximadamente 15 min, remova as enzimas (T7 RNA Polimerase, Pirofosfatase, Inibidor de Rnase) do freezer a -20 °C e mantenha-o em um refrigerador até que seja armazenado novamente no freezer.
- Para misturar reagentes IVT individuais (exceto enzimas), use um vórtice. Misture as enzimas pipetando suavemente para cima e para baixo.
- Misture os reagentes IVT em proporções definidas no projeto experimental (por exemplo, o protocolo IVT genérico é mostrado na Tabela 1) em um tubo de 1,5 mL. Ao realizar a otimização de IVT, o volume final de IVT pode variar entre 50-100 μL. Ao projetar o volume total de IVT, considere o número de amostras de 2 μL a serem retiradas da reação durante o experimento de curso de tempo. Certifique-se de que o volume total das amostras não exceda 50% do volume total de IVT (por exemplo, se a amostragem for de 2 μL a cada 15 min por 3 h, o volume total de alíquotas coletadas é de 30 μL, o que significa que o volume total de IVT deve ser de no mínimo 60 μL).
- Misture os IVTs com cuidado, adicione a RNA polimerase T7 como o último reagente. Quando todos os reagentes forem adicionados, misture bem o IVT pipetando para cima e para baixo, não use vórtice.
- Imediatamente após todos os reagentes IVT serem adicionados e homogeneizados, remova 2 μL de mistura IVT do tubo e pipete-o em tubos de 0,5 mL previamente preparados contendo 2 μL de EDTA 100 mM descritos na etapa 2.1.2 (ponto de tempo 0 min).
- Colocar tubos de reação contendo misturas de reação IVT em um termobloco e incubá-los a 37 °C.
- Defina o temporizador para os intervalos de tempo desejados (por exemplo, 15 min/30 min). Em cada momento, retirar 2 μl de uma amostra de IVT de cada mistura de reacção de IVT preparada e pipetá-la para um tubo previamente preparado (ver passo 2.1.2) contendo 2 μL de EDTA a 100 mM. Estas alíquotas serão utilizadas para a análise cromatográfica (ver passo 3).
- Depois que a análise cromatográfica confirmar a depleção completa dos NTPs, extinguir as reações de IVT a granel com EDTA até uma concentração final de 50 mM (por exemplo, 30 μL de IVT inativar com 30 μL de EDTA 100 mM).
- Reação de IVT em lote alimentado
NOTA: A produção de mRNA pode ser aumentada adicionalmente adicionando NTPs e íons Mg2+ após o esgotamento dos NTPs.- Misturar a reação de IVT em lote e obter amostras conforme descrito no passo 2.1. Prepare a ração NTP+MgCl2 para o lote alimentado. Para preparar, por exemplo, 500 μL de mistura NTP + MgCl2 , misture 106 μL de cada 200 mM NTP, 76,25 μL de 1 M MgCl2 e 5,5 μL de ddH2O. As concentrações finais na mistura são 42,4 mM cada NTP e 152,5 mM MgCl2.
- Monitore o consumo de NTP com análises on-line descritas na etapa 3. Quando a análise em linha confirmar que a concentração de NTP cai abaixo de 10% da concentração inicial, adicione a ração contendo NTP + Mg2+ ao IVT em massa.
- Meça/calcule o volume preciso da reação IVT em massa restante em um tubo de 1,5 mL depois que todas as alíquotas forem tomadas para análise. Adicione um volume apropriado de mistura NTP + MgCl2 a um IVT de modo que a concentração final seja de 4 mM de cada NTP e 12 mM de MgCl2. Por exemplo, para 80 μL de IVT a granel deixados em um tubo de 1,5 mL, adicione 16,8 μL de mistura NTP + MgCl2 .
- Continue com a amostragem conforme escrito na etapa 2.1. Depois que os NTPs se esgotarem novamente, repita a seção 2.2.2. Continue com a alimentação e amostragem até que a produção de mRNA desejada seja alcançada.
- Depois que a produção de mRNA desejada for alcançada, inative toda a reação como na etapa 2.1.10.
3. Preparação da análise cromatográfica
- Preparação para o teste de adequação do padrão e do sistema (SST)
- Prepare o SST combinando reagente de capeamento, NTPs, modelo de pDNA e mRNA. Certifique-se de que as concentrações finais sejam aproximadamente 3 μM para o reagente de capeamento, 5 μM para cada NTP, 4 ng/μL para o molde de pDNA e 10 ng/μL para o mRNA. Certifique-se de que o SST final contenha 0,1 M de NaCl.
- Prepare uma nova curva de calibração antes de cada análise. Crie o padrão de calibração diluindo uma amostra de mRNA purificada de concentração conhecida com MPA. Os pontos de calibração devem ser 0,5 ng/μL, 2 ng/μL, 5 ng/μL, 10 ng/μL, 15 ng/μL e 20 ng/μL, cada um com uma concentração final de 0,1 M NaCl.
- Se necessário, prepare uma curva de calibração para todos os NTPs. Certifique-se de que as concentrações finais de cada NTP nos padrões de calibração sejam 0.5 μM, 2 μM, 5 μM, 10 μM, 15 μM e 20 μM.
- Condicionamento de coluna e preparação para análise
- Equilibre 0,1 mL de coluna analítica à temperatura ambiente por 12 h antes da análise. Fixar a coluna ao sistema cromatográfico na direcção indicada no invólucro da coluna.
- Lave a coluna com uma vazão de 1 mL/min: primeiro com 50 volumes de coluna (CV) de ddH2O e depois com 50 CV de MPA.
- Execute pelo menos 3 amostras em branco (injetando apenas MPA) antes da análise para definir a linha de base. Utilizar o método cromatográfico descrito em Skok et al.15.
- Quando a linha de base estiver estável e duas medições consecutivas em branco forem comparáveis (linha de base reprodutível), prossiga para executar a amostra SST e as amostras da curva de calibração.
- Se os critérios SST forem cumpridos e a separação do reagente de capeamento e dos NTP for satisfatória, prosseguir com a análise das amostras IVT. Depois de analisar todas as amostras de IVT, injete um padrão SST novamente para confirmar que as condições do sistema permaneceram estáveis durante toda a análise.
- Preparação da amostra
NOTA: As amostras para análise cromatográfica são preparadas imediatamente antes da análise. Caso contrário, as amostras são mantidas num congelador a -20 °C (máximo 24 h) ou <-65 °C (mais de 24 h).- Antes da diluição para análise cromatográfica, vórtice e centrifugação da amostra IVT temperada. Analisar todas as amostras de IVT inativadas por EDTA (ver passo 2.1.9) com análise cromatográfica utilizando o método descrito em Skok et al.15.
- Antes da análise, diluir as amostras de IVT para obter uma concentração dentro da curva de calibração. Use o mesmo fator de diluição durante todo o experimento de curso de tempo para garantir que as áreas de NTP possam ser comparadas para determinar quando um NTP está esgotado.
- Para determinar a diluição mínima necessária para amostras de IVT, use a seguinte fórmula: Diluição necessária = (concentração final esperada de mRNA) / (diluição durante a inativação) / (padrão de calibração mais alto)
Por exemplo, se uma produção de mRNA de 8 mg/mL for esperada em IVT, a concentração na amostra temperada será de 4 mg/mL. Para atingir a concentração dentro da curva de calibração, dilua ainda mais a amostra em 200 vezes.
Diluição necessária = (8000 ng/μL]) / 2 / (20 ng/μL) = 200 - Pipetar o MPA e o NaCl 4 M para um frasco de vidro cónico. Certifique-se de que a concentração final de NaCl na amostra seja de 100 mM. Por fim, adicione a amostra de IVT temperada.
- O Vortex preparou a amostra e a inseriu no amostrador automático para análise. Defina a temperatura do amostrador automático para 4 °C.
- Injetar 100 μL de amostras diluídas na coluna analítica. Medir a absorvância A260 e A280 para cada amostra de IVT.
- Para analisar amostras com concentrações conhecidas, ou se não for necessário rastrear o consumo de NTP com base nas diferenças de área, dilua as amostras para atingir uma concentração alvo de 10 ng/μL.
4. Quantificação da amostra e análise dos dados
- Antes da integração de cada amostra de IVT, subtrair a amostra em branco do cromatograma da amostra.
- Integre todos os picos de NTP, pDNA e mRNA em A260 e use as áreas de pico para calcular as concentrações de NTPs e mRNA no IVT.
NOTA: Em alternativa, as áreas de pico podem ser utilizadas para comparar a percentagem de cada NTP remanescente na reação IVT, desde que o fator de diluição seja consistente em todos os momentos. - Com base nas concentrações de mRNA/NTP, determinadas por análise cromatográfica em linha, decida se o IVT pode ser inativado ou se NTPs adicionais devem ser inseridos na reação de IVT (consulte a etapa 2).
Resultados
A análise cromatográfica descrita neste protocolo pode ser usada para otimização de IVT ou para converter uma reação de IVT em lote em uma reação de batelada alimentada (Figura 1).
Para testar o efeito de diferentes composições de tampão na cinética da reação IVT, três reações diferentes de IVT foram misturadas de acordo com o protocolo escrito na Tabela 2. O tampão contendo Tris, onde o pH foi ajustado com HCl, foi comparado ao tampão Tris, onde o pH foi ajustado com ácido acético. Além disso, ambos os tampões Tris foram comparados com o tampão HEPES, onde o pH foi ajustado com NaOH. A composição de todo o tampão 1x IVT foi de 40 mM Tris/HEPES, 10 mM DTT, 2 mM de espermidina e pH 7,9.
A partir de 100 μL de mistura de reações IVT, 2 μL de amostra IVT foram retirados da reação IVT e temperados com 2 μL de EDTA 100 mM a cada 15 min na primeira hora de incubação e depois a cada 30 min até 180 min de incubação. O rendimento final esperado de mRNA para esta condição de reação foi de 15 mg / mL, o que significa que a diluição de 2 vezes devido à têmpera foi seguida por uma diluição de 400 vezes em MPA + NaCl para análise. A amostra foi diluída 800 vezes no total, o que significa que, mesmo com produção de 15 mg/mL, a concentração de mRNA carregada na coluna cromatográfica analítica ainda estava dentro das concentrações da curva de calibração.
As áreas de NTP e mRNA em A260 foram integradas para cada amostra individual em cada ponto de tempo individual (tx). As áreas foram então convertidas em concentrações para mRNA, usando a curva de calibração de mRNA e porcentagem de consumo para NTPs, usando a área A260 de NTPs em 0 min (t0) como 100% (veja a equação abaixo).
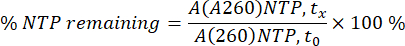
Os resultados podem ser mostrados como gráficos para cada reação IVT individual, onde o tempo é mostrado no eixo x e a concentração de mRNA e os NTPs restantes são mostrados no eixo y (Figura 2A). Todos os IVT, ou seja, as concentrações de mRNA na dependência do tempo, também podem ser plotados juntos em um gráfico, e a cinética de produção de mRNA pode ser estudada e as condições ideais de IVT selecionadas (Figura 2B).
Para o experimento do lote alimentado, o IVT foi misto conforme escrito na Tabela 3. De 300 μL de mistura de reação IVT, 2 μL de amostra IVT foram removidos da reação IVT e temperados com 2 μL de EDTA 100 mM a cada 30 min. Além disso, a amostra foi coletada imediatamente após cada adição de NTP + MgCl2 a granel. Uma solução de alimentação contendo 42,4 mM de cada NTP e 152,5 mM de MgCl2 foi preparada com antecedência, e 106 μL de cada NTP individual de 200 mM foram misturados com 76,2 μL de 1 M MgCl2. Um regime alimentar foi estabelecido, onde a alimentação em bolo foi adicionada a cada hora (aos 60 min, 120 min, 180 min e 240 min de incubação). O regime de alimentação é descrito na Tabela 3 As amostras foram analisadas quase em tempo real com análises conforme descrito para a reação IVT em massa. Após 300 minutos de monitoramento, a reação de IVT foi extinta.
Os resultados podem ser mostrados como produção restante de NTPs/mRNA ao longo do tempo de incubação. Como as adições de ração em bolus também diluem a reação de IVT, a concentração de mRNA na IVT cai a cada adição de ração ( Figura 3A ). O aumento na massa de mRNA também pode ser medido e apresentado por fatores de transcrição (definidos como mmRNA / mpDNA), mostrando aumento linear na produção de mRNA ao longo do tempo ( Figura 3B ).
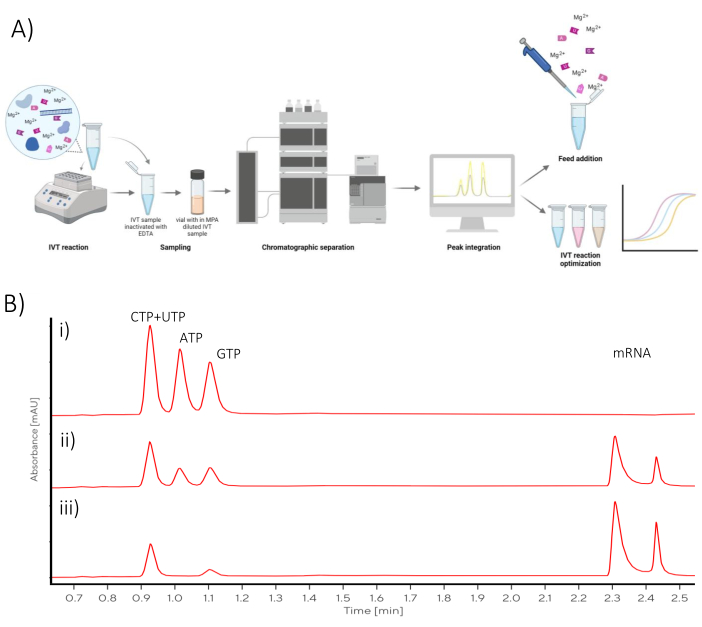
Figura 1: Visão geral esquemática (A) Representação do fluxo de trabalho de otimização de IVT com quantificação de análise cromatográfica em linha de NTPs e mRNA. B) Cromatogramas representativos de uma reação IVT descontínua, amostrados a (i) t0, (ii) ponto médio, (iii) pontos finais da reação. Clique aqui para ver uma versão maior desta figura.
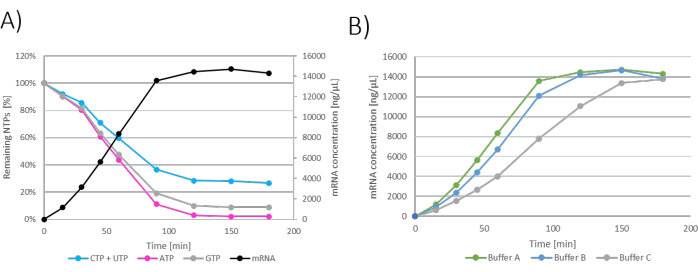
Figura 2: Gráficos de IVT em lote. (A) Gráfico representativo da reação IVT com produção de mRNA e consumo de NTP no eixo y. Platô de concentração de mRNA visível em 120 - 180 min de incubação que se correlaciona com o consumo de NTP como NTP limitante (ATP) é consumido em 120 min. (B) Gráfico IVT mostrando a influência do tampão IVT na cinética IVT. O IVT contendo o tampão A (Tris + ácido acético) mostrou a produção de mRNA mais rápida, seguido pelo tampão B (Tris + HCl) e o tampão C (HEPES + NaOH) como os mais lentos. Clique aqui para ver uma versão maior desta figura.

Figura 3: Gráfico IVT do lote federal. (A) Gráfico IVT onde alimentações NTP + MgCl2 foram adicionadas em 60, 120, 180 e 240 min. A concentração de mRNA na reação IVT cai a cada adição de ração devido à diluição com ração NTP + MgCl2 . (B) O aumento da massa de mRNA mostrado pelo fator de transcrição é linear durante toda a incubação da reação. Clique aqui para ver uma versão maior desta figura.
Tabela 1: Protocolo genérico de IVT. Clique aqui para baixar esta tabela.
Tabela 2: Protocolo de IVT em lote. Clique aqui para baixar esta tabela.
Tabela 3: Protocolo IVT em lote federal. Clique aqui para baixar esta tabela.
Discussão
Neste método, as amostras de IVT são analisadas em diferentes momentos durante a incubação usando uma coluna multimodal cromatográfica analítica que separa NTPs, pDNA e mRNA, permitindo assim um monitoramento rigoroso do consumo de NTP e da produção de mRNA. Este método mede a concentração de NTPs e mRNA de maneira quantitativa com base nas mudanças nas áreas A260 em pontos de tempo designados. Como o método fornece informações sobre a concentração de NTP e mRNA, ele é altamente adequado para otimização de IVT, onde o objetivo principal geralmente é maximizar o rendimento de mRNA e minimizar o tempo de reação; portanto, entender o efeito de diferentes reagentes IVT na cinética da produção de mRNA é fundamental22. Mostramos como o monitoramento em linha pode ser aplicado para otimizar a reação IVT nos modos de batelada e batelada alimentada.
A principal vantagem deste método é uma análise em linha que permite o monitoramento quase em tempo real da IVT, pois cada amostra é analisada em menos de 8 minutos. A preparação de amostras para análise é simples, pois apenas a diluição em MPA é necessária sem qualquer pré-tratamento prévio da amostra. O volume de amostra necessário para análise é muito baixo, por exemplo, 1 μL de IVT. Esse pequeno volume de pipetagem pode resultar em desvios analíticos devido a erros de pipetagem. No entanto, como o alto rendimento da abordagem analítica permite medições triplicadas, os outliers são facilmente identificáveis e os desvios da curva cinética esperada não são difíceis de detectar.
Uma das limitações do método é a coeluição cromatográfica de UTP e CTP. A quantificação precisa de UTP e CTP geralmente não é necessária, pois o NTP limitante em uma sequência de mRNA geralmente é ATP ou GTP. Se for necessária uma quantificação separada de UTP e CTP, a diferença na absorbância de UV a 260 nm e 280 nm para UTP e CTP pode ser aproveitada para derivar a abundância relativa de cada NTP no pico cromatográfico.
Este método analítico não separa espécies de RNA acima de 100 nucleotídeos de comprimento; portanto, não pode ser usado para detectar diferenças na qualidade do mRNA, por exemplo, não diferencia entre mRNA não poliadenilado e poliadenilado, entre dsRNA ou transcritos abortivos e ssRNA e não é adequado para estudos de estabilidade, pois não pode diferenciar entre mRNA degradado e não degradado. No entanto, o método pode ser utilizado para a quantificação de outras modalidades de RNA, como circRNA, tRNA e saRNA. Embora essas moléculas variem em tamanho e estrutura, o mesmo método analítico pode ser empregado para estudar os rendimentos de produção de cada uma em uma reação IVT.
Divulgações
Os autores não têm nada a divulgar.
Agradecimentos
Os autores gostariam de agradecer a Tomas Kostelec, Blaž Bakalar, Nejc Pavlin, Andreja Gramc Livk e Anže Martinčič Celjar pelas discussões úteis.
Materiais
| Name | Company | Catalog Number | Comments |
| 0.22 µm PES membrane filter, e. g. Sartolab BT 500 | Sartorius | 180E14---------E | |
| 0.5 mL plastic tubes; e. g. e. g. DNA LoBind, PCR clean | Eppendorf | 30108035 | |
| 1.5 mL plastic tubes; e. g. DNA LoBind, PCR clean | Eppendorf | 30108051 | |
| ATP Solution | MEBEP BIOSCIENCE | R1331T2 | |
| Benchtop cooler -20 °C | Brand | 114935 | |
| CIMac PrimaS 0.1 mL Analytical Column (2 µm) | Sartorius BIA Separations | 110.5118-2, 2 µm channels | |
| CTP Solution | MEBEP BIOSCIENCE | R3331T2 | |
| DTT | Sigma | 10197777001 | |
| EDTA-Na2 x 2H2O | Kemika | e.g. 11368 08 | |
| GTP Solution | MEBEP BIOSCIENCE | R2331T2 | |
| HEPES | Merck | 1.10110.1000 | |
| HPLC high recovery vial | Macherey-Nagel | 702860 | |
| MgCl2 | Invitrogen | AM 9530G | |
| Microvolume spectrophotometer | Thermo Scientic | Nanodrop | |
| mRNA standard, mFix4, 4000 nt | Sartorius BIA Separations | BIA-mFix4.1.1 | |
| Na4P2O7 + 10 H2O | Sigma | S6422-500G | |
| NaCl | Fluka | 31434-1KG-M | |
| NaOH | Merck | 1064691000 | |
| PATfix mRNA analytical platform | Sartorius BIA Separations | PAT0021 | |
| Pipette 100 – 1000 μL | Eppendorf | Reference 2 | |
| Pipette 2 – 10 μL | Eppendorf | Reference 2 | |
| Pipette 20 – 200 μL | Eppendorf | Reference 2 | |
| Pyrophosphatase | MEBEP BIOSCIENCE | M2403L | |
| Rnase Away Decontamination Reagent | Thermo Scientic | 10328011 | |
| RNAse inhibitor | MEBEP BIOSCIENCE | RNK3501 | |
| Spermidine | Sigma | 85558-5G | |
| T7 mRNA polymerase | MEBEP BIOSCIENCE | TR01 | |
| Thermoblock | Thermo Scientic | EPPE5382000.015 | |
| Trizma Base | Sigma | T6066-1KG | |
| UTP Solution | MEBEP BIOSCIENCE | R5331T2 | |
| Vial caps | Macherey-Nagel | 70245 | |
| Vortex | IKA | V1900 |
Referências
- Barbier, A. J., Jiang, A. Y., Zhang, P., Wooster, R., Anderson, D. G. The clinical progress of mRNA vaccines and immunotherapies. Nat Biotechnol. 40, 840-854 (2022).
- Baden, L. R., et al. Efficacy and safety of the mRNA-1273 SARS-CoV-2 vaccine. N Engl J Med. 384 (5), 403 (2021).
- Lopez Bernal, J., et al. Effectiveness of Covid-19 Vaccines against the B.1.617.2 (Delta) Variant. N Engl J Med. 385 (7), 585-594 (2021).
- Sahin, U., Karikó, K., Türeci, &. #. 2. 1. 4. ;. mRNA-based therapeutics - developing a new class of drugs. Nat Rev Drug Discov. 13, 759-780 (2014).
- Baptista, B. R., Carapito, N., Pichon, C., Sousa, F. mRNA, a revolution in Biomedicine. Pharmaceutics. 13, 2090 (2021).
- Rosa, S. S., Prazeres, D. M. F., Azevedo, A. M., Marques, M. P. C. mRNA vaccines manufacturing: Challenges and bottlenecks. Vaccine. 39, 2190 (2021).
- Whitley, J., et al. Development of mRNA manufacturing for vaccines and therapeutics: mRNA platform requirements and development of a scalable production process to support early phase clinical trials. Transl. Res. 242, 38-55 (2022).
- Kis, Z., Kontoravdi, C., Shattock, R., Shah, N. Resources, production scales and time required for producing RNA vaccines for the global pandemic demand. Vaccines. 9 (1), 3 (2021).
- Hengelbrock, A., Schmidt, A., Helgers, H., Vetter, F. L., Strube, J. Scalable mRNA machine for regulatory approval of variable scale between 1000 clinical doses to 10 million manufacturing scale doses. Processes. 11, 745 (2023).
- Rosa, S. S., et al. Maximizing mRNA vaccine production with Bayesian optimization. Biotechnol Bioeng. 119 (11), 3127-3139 (2022).
- Sari, Y., Sousa, S. S., Jeffries, J., Marques, M. P. C. Comprehensive evaluation of T7 promoter for enhanced yield and quality in mRNA production. Sci Rep. 14, 9655 (2024).
- Kumar, A., et al. The mRNA vaccine development landscape for infectious diseases. Nat Rev Drug Discov. 21 (5), 333-334 (2022).
- Pregeljc, D., et al. Increasing yield of in vitro transcription reaction with at-line high pressure liquid chromatography monitoring. Biotechnol Bioeng. 120 (3), 737-747 (2023).
- Kern, J. A., Davis, R. H. Application of a fed-batch system to produce RNA by in vitro transcription. Biotechnol Prog. 15 (2), 174-184 (1999).
- Skok, J., et al. Gram-Scale mRNA Production Using a 250-mL Single-Use Bioreactor. Chemie Ingenieur Technik. 94 (12), 1928-1935 (2022).
- Welbourne, E. N., et al. Anion exchange HPLC monitoring of mRNA in vitro transcription reactions to support mRNA manufacturing process development. Front Mol Biosci. 11, 1250833 (2024).
- Lee, K. H., Song, J., Kim, S., Han, S. R., Lee, S. W. Real-time monitoring strategies for optimization of in vitro transcription and quality control of RNA. Front Mol Biosci. 10, 1229246 (2023).
- Matuszczyk, J. C., et al. Raman spectroscopy provides valuable process insights for cell-derived and cellular products. Curr Opin Biotechnol. 81, 102937 (2023).
- Mu, X., Hur, S. Immunogenicity of in vitro-transcribed RNA. Acc Chem Res. 54 (21), 4012-4023 (2021).
- Cho, E., et al. Mesoporous silica particle as an RNA adsorbent for facile purification of in vitro-transcribed RNA. Int J Mol Sci. 24 (15), 12408 (2023).
- Ziegenhals, T., et al. Formation of dsRNA by-products during in vitro transcription can be reduced by using low steady-state levels of UTP. Front Mol Biosci. 10, 1291045 (2023).
- Höfer, K., Langejürgen, L. V., Jäschke, A. Universal aptamer-based real-time monitoring of enzymatic RNA synthesis. J Am Chem Soc. 135 (37), 13692-13694 (2013).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoExplore Mais Artigos
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados