Method Article
오염이 제거된 객담 샘플에 대한 표적 차세대 염기서열분석을 위한 매트릭스 기반 DNA 추출
* 이 저자들은 동등하게 기여했습니다
요약
Mycobacterium tuberculosis의 다운스트림 표적 차세대 염기서열분석을 위해 마그네틱 비드 정제와 결합된 매트릭스 기반 추출 방법을 사용하여 오염이 제거된 객담에서 DNA를 추출하기 위한 최적화된 방법을 제시합니다.
초록
차세대 염기서열분석(NGS)은 이제 적시에 정확한 약물 내성 결핵(DR-TB) 진단을 위한 강력한 도구로 인식되고 있습니다. 표적 NGS(tNGS)는 약물 내성과 관련된 특정 유전자에 초점을 맞춤으로써 간소화된 접근 방식을 제공하며, 몇 주에서 몇 달에 이르는 처리 시간이 걸리는 기존 배양 기반 방법의 필요성을 우회합니다. 세계보건기구(WHO)는 특히 자원이 제한된 환경에서 치료를 안내하고 치료 결과를 개선하기 위해 결핵(TB) 진단을 개선하기 위한 가치 있는 전략으로 tNGS를 권장했습니다. WHO에서 권장하는 tNGS 분석 중 신속하고 포괄적인 약물 감수성 검사, 계통 결정 및 균주 분류를 제공하는 방법을 선택했습니다. 표준화된 DNA 추출 방법을 사용할 수 있지만 시간이 많이 걸리고 노동 집약적일 수 있습니다. 이 문제를 해결하기 위해 당사는 마그네틱 비드 정제와 함께 간소화된 매트릭스 기반 DNA 추출 프로토콜을 최적화했습니다. 이 방법은 오염이 제거된 가래 침전물에서 직접 DNA를 추출하는 빠르고 효율적인 접근 방식을 제공하여 신속한 다운스트림 tNGS 분석을 가능하게 합니다. 이 프로토콜은 객담 침전물에서 DNA를 추출하는 과정을 간소화함으로써 일상적인 임상 환경에서 tNGS의 광범위한 채택을 촉진할 수 있으며, 궁극적으로 환자 결과를 개선하고 전 세계 결핵 통제 노력에 기여할 수 있습니다.
서문
2023년 전 세계적으로 약 370만 명의 결핵 환자가 진단과 치료를 받지 못한 것으로 추정되며, 이는 결핵이 전 세계 보건에 미치는 심각한 위협을 강조합니다1. WHO는 2023년에 약 400,000명이 리팜피신 내성(RR-TB) 또는 다제내성 결핵(MDR-TB)에 걸린 것으로 추정합니다1. 결핵 및 DR-TB를 신속하게 진단하고 치료하는 것은 결핵 발병률 및 사망률을 줄이기 위한 이정표와 목표를 달성하는 데 필수적입니다1.
기존 배양 방법과 표현형 약물 감수성 검사(pDST)에 의존하면 임상 분리 및 치료의 내성 프로필 결정이 지연되고 처리 시간이 6-8주가 소요되며 복잡한 생물 봉쇄 인프라가 필요합니다. DR-TB의 NGS와 함께 정기 진단 검사를 통해 포괄적인 약물 내성 프로파일을 제공하고 MDR-TB 요법의 개인화를 개선할 수 있으며, 효과적인 치료 기간을 몇 주 또는 몇 개월에서 며칠로 단축할 수 있습니다 2,3,4.
2023년과 2024년에 WHO는 1차 및 2차 항결핵제에 대한 감수성을 신속하게 결정하기 위해 tNGS를 새로운 진단 범주로 사용할 것을 권장했습니다5. 따라서 생물안전성 레벨 3(BSL-3) 실험실에서 Mycobacterium tuberculosis (MTB) 배양 없이 치료 결정을 안내하는 데 유용한 도구입니다. tNGS 접근법은 중합효소연쇄반응(PCR)을 사용하여 염기서열분석 전에 약물 내성을 부여하는 유전자 표적을 증폭하는 압축된 형태의 염기서열분석입니다. WHO에서 권장하는 tNGS 검사 중 리팜피신, 이소니아지드, 에탐부톨, 피라진아미드, 플루오로퀴놀론, 아미카신, 스트렙토마이신, 리네졸리드, 베다퀼린, 클로파지민에 대한 내성 검출을 위한 등급 기반 기준을 충족하는 것으로 보고된 Deeplex Myc-TB 분석을 선택했습니다. 당사는 이 분석을 사용하여 다운스트림 tNGS에 대한 이 프로토콜을 사용하여 추출한 DNA의 적합성을 평가합니다.
또한, WHO는 MTB의 약물 내성과 관련된 돌연변이 목록2판을 발행하여 약물 감수성을 예측하고 치료를 안내하기 위한 전체 게놈 염기서열 분석(WGS) 및 tNGS의 사용에 대한 로드맵을 제공했습니다6. 최근의 체계적 문헌고찰 및 메타분석에 따르면 tNGS는 mMTB 게놈의 다양한 내성 부여 영역에 있는 23개의 표적을 기준으로 pDST7에 비해 약물 내성 검출에 대한 민감도와 특이도가 각각 94.1%와 98.1%인 것으로 나타났습니다.
그러나 이러한 방법의 구현은 필요한 워크플로, 인프라 및 장비와 특히 관련된 복잡성과 비용으로 인해 여전히 어렵습니다. 중요한 과제는 오염이 제거된 가래의 침전물에서 직접 충분한 고품질 마이코박테리아 DNA를 분리하는 것이며, 이는 다운스트림 tNGS 응용 분야를 위한 중요한 단계입니다. 이를 해결하기 위해 tNGS에 맞춘 빠르고 간단한 DNA 추출 방법을 제시합니다.
선택한 MTB tNGS 분석에 대한 표준화된 DNA 추출 방법에는 사내 수동 및 자동 프로토콜8이 포함됩니다. 여기에서는 단순화된 매트릭스 기반 DNA 추출 프로토콜에 대해 설명합니다(그림 1). 이 방법은 금속과 단백질을 결합하여 오염이 제거된 가래 침전물에서 직접 고품질 핵산을 추출할 수 있는 InstaGene 매트릭스(IGM)를 사용합니다. 이 대안은 더 빠른 처리 시간과 다운스트림 tNGS를 위한 충분한 DNA 수율을 제공합니다. 이 프로토콜은 수동 및 자동 분석법의 복잡성을 극복하는 동시에 MTB에서 내성을 부여하는 변이체의 신속한 진단을 위해 고품질 tNGS를 보장합니다. 마이코박테리학 분야에서 tNGS 사용에 대한 관심이 높아짐에 따라 이 프로토콜은 일상적인 진단 워크플로우에 쉽게 채택할 수 있습니다.
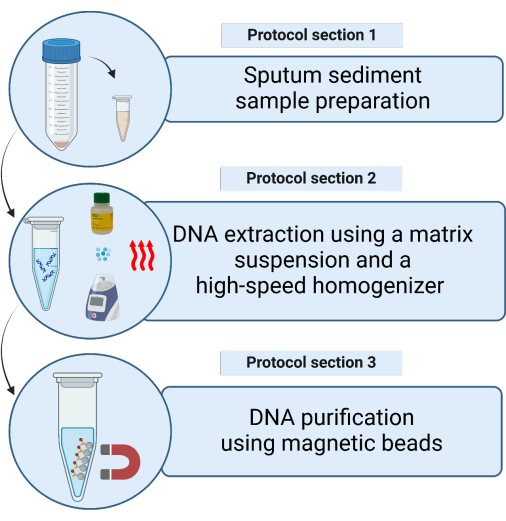
그림 1: 매트릭스 현탁액을 사용하여 오염이 제거된 가래 침전물 샘플에서 마이코박테리아 DNA를 추출하는 방법의 개략도.이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
프로토콜
이 연구는 스텔렌보스 대학교의 연구 윤리 위원회: 생물 및 환경 안전(REC: BES): BES-2024-25384 및 인간 연구 윤리 위원회: N21/09/093 및 N09/11/296의 승인을 받았습니다.
1. 가래 침전물 시료 준비
참고: 다음 단계는 DNA 추출 전에 생물 안전 레벨 3(BSL3) 실험실에서 수행해야 합니다.
- 500 μL-2 mL를 1.5 mL 또는 2 mL low-bind tube로 옮겨 N-acetyl-L-L-cysteine(NALC) 및 수산화나트륨(NaOH)(NALC/NaOH)을 사용하여 이전에 오염을 제거한 객담 침전물 샘플의 부분 표본을 준비합니다.
- 객담 침전물 샘플이 들어있는 튜브를 튜브 랙에 똑바로 세우고 샘플을 80 ° C의 오븐에 넣고 1 시간 동안 배양하여 열처리를 통해 마이코박테리아를 비활성화합니다.
- 1시간 후 샘플이 든 튜브 랙을 제거하고 튜브 표면을 닦은 다음 생물 안전 레벨 2(BSL2) 실험실로 운송하기 위해 적절한 용기에 넣습니다.
알림: 샘플을 허용하십시오.amp추가 취급 또는 보관하기 전에 실온으로 식히십시오.
2. 매트릭스 현탁액과 고속 균질기를 사용한 DNA 추출(그림 2)
- 시작하기 전에 2mm 직경의 유리 구슬 3개를 1.5 또는 2mL 나사 캡 튜브에 느슨하게 캡을 씌우고 오토클레이브를 추가합니다(이상적으로는 DNA 추출 하루 전).
- 냉장고에서 매트릭스 현탁액을 제거하고 자석 교반기(시약 병에 자석이 포함되어 있음)에 올려 시약을 실온으로 가져오고 시약 혼합을 시작합니다.
- 가열 블록을 켜고 56°C로 예열합니다(두 개의 가열 블록을 사용할 수 있는 경우 두 번째 가열 블록을 100°C로 예열합니다.
알림: 다음 단계는 BSL2 시설의 실험실 벤치에서 수행할 수 있습니다. - 열이 비활성화된 가래 침전물 샘플을 16,800 × g (또는 최대 속도)으로 15분 동안 원심분리합니다. 20-200 μL 피펫으로 상층액을 부드럽게 흡입하고 폐기하여 펠릿이 방해받지 않도록 합니다.
- 매트릭스 현탁액을 부드럽게 흔들어 혼합하고(와류가 발생하지 않음) 잘 혼합된 매트릭스 200μL를 추가하여 펠릿을 재현탁합니다.
- 직경 2mm의 사전 멸균된 유리 구슬 3개가 들어 있는 1.5mL 나사 캡 튜브에 전체 부피를 피펫으로 넣습니다.
- 샘플을 가열 블록에 넣고 56°C에서 15분 동안 배양합니다.
알림: s를 제거한 직후amp가열 블록에서 가열 블록에서 하나의 가열 블록만 사용할 수 있는 경우 100단계 후에 가열 블록을 2.7°C로 설정하십시오. - 세포를 분산시키기 위해 10초 동안 와류를 사용하여 샘플을 균질화합니다.
- 샘플을 가열 블록에 넣고 100°C에서 10분 동안 배양합니다.
- 다음 매개변수를 가진 고속 균질화기를 사용하여 샘플을 균질화합니다: 4.0m/s에서 60초의 1회 주기.
- 12,000 × g 에서 5분 동안 원심분리기. 130 μL의 DNA 함유 상등액을 펠릿(세포 파편이 있는 매트릭스)을 방해하지 않고 새로운 1.5 mL 저결합 튜브로 옮깁니다. 매트릭스와 세포 파편이 들어 있는 첫 번째 튜브를 버립니다.
참고: 안전한 정지 지점. 중단하는 경우 DNA 정제를 계속할 준비가 될 때까지 -20 °C에서 샘플을 동결합니다. - 마그네틱 비드를 이용한 DNA 정제를 진행합니다.
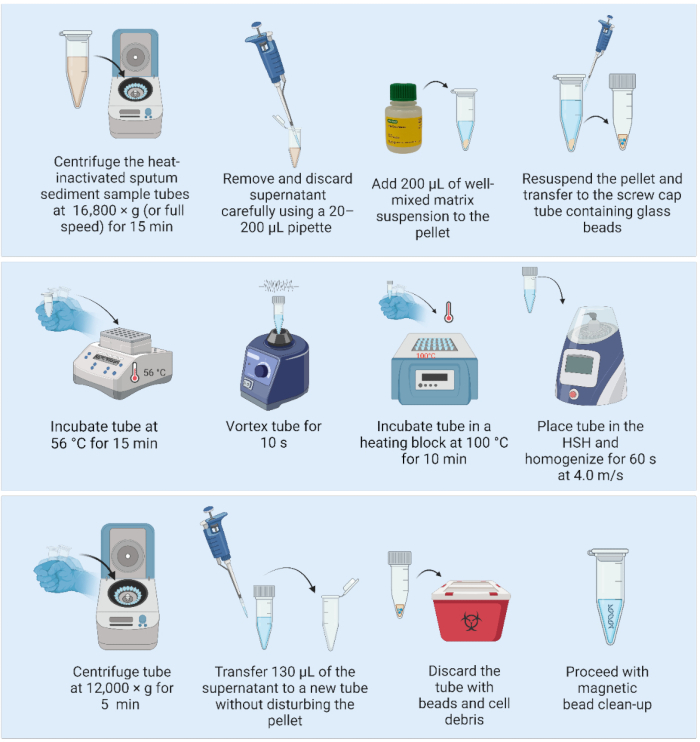
그림 2: 고속 균질화기와 결합된 매트릭스 기반 DNA 추출 방법. 약어: HSH = 고속 균질화기. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
3. 마그네틱 비드를 사용한 DNA 정제(그림 3)
참고: DNA 정제를 진행하기 전에 3.1 및 3.2 단계를 수행하십시오.
- 냉장고에서 마그네틱 비드를 제거하고 용기를 실온에 30분 동안 두어 평형을 이룹니다. 또는 마그네틱 비드 스톡 병을 소용돌이치게 하여 모든 비드를 재현탁시키고 1,280μL 분취량을 새 튜브(21개 시료 정제용)로 흡인하고 실온에서 평형을 이룹니다.
- 여과 및 멸균된 물 9mL에 순수 에틸 알코올 7.2mL를 첨가하여 80% 에탄올 9mL 배치를 준비합니다.
알림: 다음 단계는 BSL2 시설의 실험실 벤치에서 수행할 수 있습니다. - 마그네틱 비드 스톡 병 또는 준비된 부분 표본을 철저히 소용돌이쳐 사용하기 전에 비드가 재현탁되도록 합니다. 균질한 현탁액을 보장하기 위해 10개의 샘플마다 와류를 반복합니다.
- 추출된 DNA(130μL)에 마그네틱 비드의 1.2배(156μL)를 추가합니다.
참고: DNA(배양)가 더 많은 샘플의 경우 샘플의 더 작은 부분 표본을 처리하고(예: 50μL) 1.2배 부피의 마그네틱 비드로 정제할 수 있습니다. - 10회 피펫팅하여 샘플을 혼합합니다. 실온에서 5분 동안 샘플을 배양합니다.
- 샘플을 1.5mL 튜브 마그네틱 랙에 3분 동안 또는 액체가 투명해질 때까지 놓습니다. 조심스럽게 흡인하고 비드를 방해하지 않고 상등액을 버리십시오.
- 자석에 튜브를 끼우고 200μL의 80% 에탄올을 첨가하여 비드가 방해받지 않고 30초 동안 배양할 수 있도록 합니다.
- 조심스럽게 흡인하고 구슬을 방해하지 않고 에탄올을 버리십시오.
- 3.7단계를 반복하여 총 두 번 세탁합니다.
- 두 번째 세척 후 에탄올을 제거하고 버립니다. 1-10 μL 피펫으로 잔류 에탄올을 제거합니다.
- 튜브를 열어 두어 10분 동안 또는 비드가 무광택 모양이 될 때까지 비드를 자연 건조시킵니다.
알림: 구슬이 과도하게 건조되어 금이 가지 않도록 하십시오. - 비드가 무광택 모양이 되면 즉시 자석에서 튜브를 제거하고 각 샘플의 비드에 50μL의 뉴클레아제가 없는 물(NFW)을 직접 추가합니다. 모든 튜브에 NFW를 추가한 후 10회 피펫팅하여 각 개별 샘플을 혼합합니다. 튜브 내부에 붙어 있는 비드가 있는지 검사하고 필요한 경우 혼합 단계를 반복합니다. 실온에서 5분 동안 샘플을 배양합니다.
참고: 배양 시간은 5분에서 10분으로 늘릴 수 있지만 배양 시간을 5분 미만으로 줄이지는 마십시오. - 튜브를 마그네틱 랙에 3분 동안 또는 액체가 맑아질 때까지 다시 놓습니다. 마그네틱 랙에 있는 튜브를 사용하여 DNA가 함유된 상등액을 명확하게 표시된 멸균 저결합 튜브로 옮깁니다. 비드가 상층액으로 옮겨지지 않도록 하십시오.
참고: 이송 중 피펫 팁에 비드가 보이면 상층액을 마그네틱 랙의 튜브에 다시 피펫으로 넣어 3분 더 분리하십시오. 비드를 새 튜브에 옮기지 마십시오.
안전한 정지 지점. 중단하는 경우 NGS에 적합한 방법을 사용하여 정량화를 계속하고 MTB tNGS 분석을 사용하여 후속 PCR 증폭을 계속할 준비가 될 때까지 샘플을 -20°C에서 동결합니다.
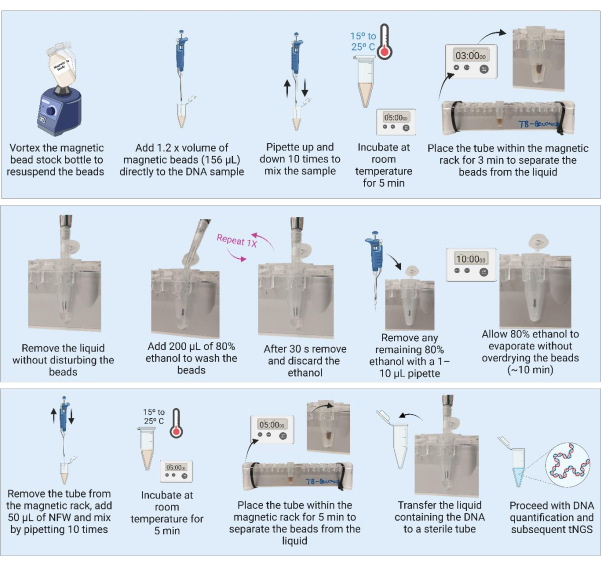
그림 3: 마그네틱 비드를 사용한 DNA 정제 및 농축. 약어: NFW = 뉴클레아제가 없는 물; tNGS = 표적 차세대 염기서열분석(targeted next-generation sequencing). 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
결과
샘플 설명
박테리아 부하가 1+ 이상인 현미경 검사에서 AFB(Acid-fast Bacilli)에 양성인 총 165개의 가래 침전물 샘플이 남아프리카 케이프타운의 NHLS(National Health Laboratory Services) Green Point에서 정기적으로 수집 및 처리되었습니다. 두 가지 다른 부피[약 2mL(n = 102) 및 500μL(n = 63)]를 사용하여 가래 침전물 샘플에서 DNA를 추출했습니다. 이 비교는 더 작은 퇴적물 샘플 부피가 다운스트림 tNGS에 충분한 DNA를 생성할 수 있는지 여부를 평가하기 위해 수행되었습니다.
그림 4는 다양한 객담 도말 등급(1+, 2+ 및 3+)과 객담 침전물 부피(2mL 및 500μL)에 걸쳐 총 DNA 농도(ng/μL)의 상자 플롯 비교를 보여줍니다. DNA 수율은 일반적으로 추출을 위해 500μL의 침전물을 사용할 때 전체적으로 감소하며 도말 등급에 따라 변동성이 있습니다. 따라서, 부피가 2mL 이상인 퇴적물 샘플에서 추출한 평균 DNA 농도는 샘플의 AFB에 의해 계층화된 500μL 퇴적물에서 추출한 DNA 농도보다 평균적으로 높았습니다.
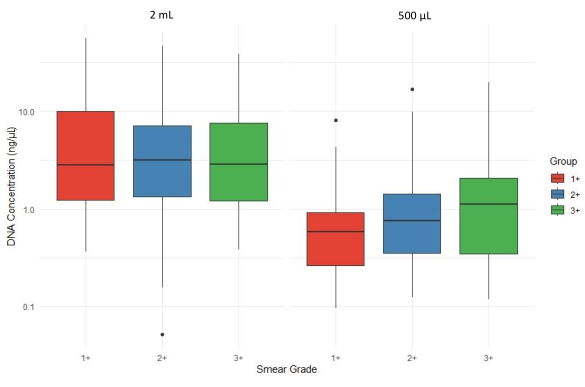
그림 4: 도말 등급에 걸쳐 DNA 수율에 대한 가래 침전물의 양이 미치는 영향.이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
그림 5는 두 가지 다른 주입량(2mL 및 500μL)에서 처리된 다양한 도말 등급(1+, 2+ 및 3+)을 가진 객담 침전물 샘플에 대한 모든 표적의 평균 염기서열분석 판독 수(평균 커버리지 깊이)를 보여주는 상자 그림을 보여줍니다. 이상값은 개별 점으로 표시되며, y축은 적용 범위의 변동을 수용하기 위해 로그 눈금으로 표시됩니다. 500μL 퇴적물에서 추출한 3+ 샘플의 높은 사분위수 범위는 더 높은 박테리아 부하에서 더 큰 변동성을 시사합니다.
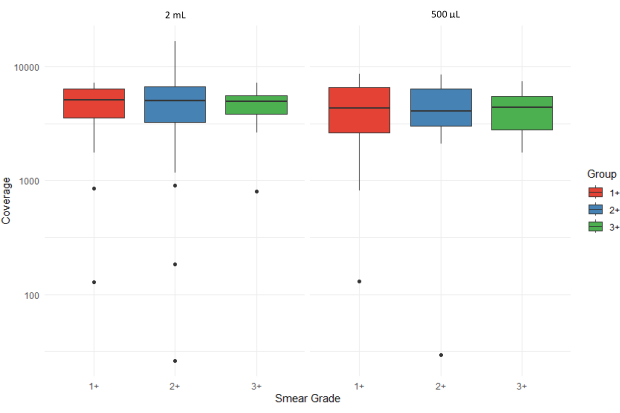
그림 5: 도말 등급과 시료 주입량에 걸쳐 모든 표적에 대한 평균 염기서열분석 판독 수(coverage depth) 비교. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
그림 6은 관련 웹 애플리케이션의 생물정보학 파이프라인을 사용하여 염기서열분석 결과의 수용 가능성을 평가한 것을 보여줍니다. 염기서열분석 품질 점수는 +++(매우 수용 가능), ++(수용 가능), +(약간 수용 가능), -(수용 불가) 및 ND(결정되지 않음)로 분류됩니다. 그래프에 막대가 없으면 해당 특정 범주에 대한 결과를 얻지 못했음을 나타냅니다. 도말 등급 1+는 500μL 투입량 시료 세트에 대한 도말 등급 2+ 및 3+에 비해 허용되지 않는 ND 결과의 비율이 더 높다는 것을 보여줍니다. 500μL의 가래 침전물에서 추출한 샘플의 평균 염기서열분석 범위 깊이는 4,316인 반면, 2mL 가래 침전물에서 추출한 샘플의 경우 4,810이었습니다. 이는 객담 퇴적물에서 추출한 DNA가 투입량에 관계없이 다운스트림 tNGS를 수행하기에 적합했음을 시사합니다.
염기서열분석 결과의 수용 가능성의 분포에 기초하여, 도말 등급이 3+인 샘플은 1+ 및 2+ 도말 등급 샘플에 비해 ++ 및 +++로 점수가 매겨진 대부분의 샘플 염기서열에서 가장 높은 성공률을 보였습니다. 500μL 입력 샘플의 경우 - 및 + 범주의 샘플 비율은 2mL 입력 샘플에 비해 모든 도말 등급에서 상대적으로 높습니다. 이는 더 많은 샘플이 500μL 입력에 대해 품질이 낮고 수용 수준이 낮은 범주에 속한다는 것을 시사합니다. 2mL 입력 샘플의 경우, 특히 도말 등급 3+의 경우 고품질 ++ 및 +++ 범주의 샘플의 비율이 더 높습니다. 이는 2mL 입력의 샘플이 500μL 입력의 샘플에 비해 더 나은 염기서열분석 품질 점수를 생성할 가능성이 더 높다는 것을 시사합니다.
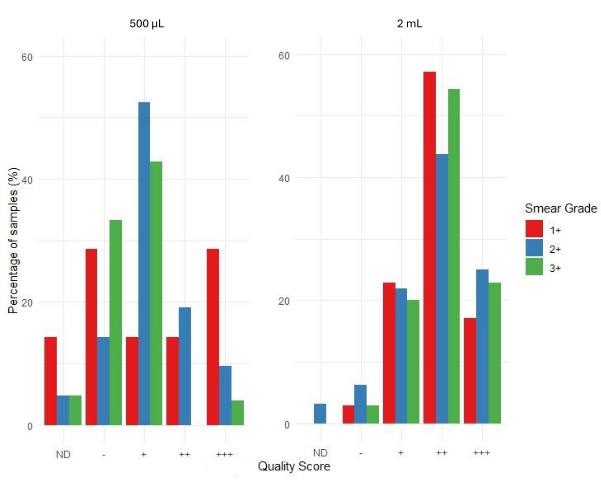
그림 6: 도말 등급 및 샘플 입력 볼륨에 따른 품질 점수 분포.이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
토론
고속 균질화기와 결합된 매트릭스 기반 추출 방법의 조합은 원래 Shea et al.9에 의해 발표되었습니다. 이 방법은 MTBC 배양 9,10에서 추출한 DNA의 WGS에 대해 개발 및 최적화되었습니다. MTB tNGS 분석과 함께 사용하기 위해 이 방법을 최적화했습니다. 21개 샘플의 DNA 추출 및 정제는 배양 및 원심분리 단계를 포함하여 총 4시간 및 40분의 처리 시간을 가졌습니다. 도말 등급이 1+에서 3+까지 다양한 샘플에서 DNA를 추출하기 위한 비드 클린업과 결합된 설명된 프로토콜의 신뢰성은 평균 수율(그림 4), 결합된 모든 표적의 전체 적용 깊이(그림 5) 및 관련 웹 애플리케이션에 의해 결정된 염기서열분석 결과 수용성 점수(그림 6)를 기반으로 입증되었습니다.
최근 연구에서는 도말 등급과 DNA 농도 또는 염기서열분석 판독 깊이 사이에 일관된 관계가 없음을 발견했습니다. 저자들은 시료 처리 및 세척 단계의 변동성이 특히 오염 물질이 효율적으로 제거되지 않는 경우 높은 도말 등급의 시료의 성능에 영향을 미칠 수 있다고 제안했습니다. 또한 비표적 DNA가 MTB DNA11과 경쟁할 수 있는 DNA 풀의 복잡성으로 인해 객담 퇴적물로부터의 염기서열분석은 여전히 어렵습니다. 입력 DNA 농도와 염기서열분석 깊이 사이의 명확한 상관관계는 관찰되지 않았지만, PCR 억제로 이어질 수 있으므로 표적 PCR 전 DNA 입력이 100ng를 초과해서는 안 된다는 점에 유의하는 것이 중요합니다. 또한 박테리아 부하가 높고 2+ 및 3+ 가래 침전물 샘플에는 PCR 증폭 및 라이브러리 준비를 방해할 수 있는 인간 DNA, 세포 파편 및 기타 억제제와 같은 더 많은 오염 물질이 포함될 수 있습니다.
정제 단계는 또한 농도 단계로 기능하여 tNGS 이전에 mycobacterial DNA의 수율을 향상시킵니다. 이 방법은 박테리아 부하가 낮은 시료를 처리하는 데 효과적이며 500μL의 작은 부피에서 신뢰할 수 있는 성능을 입증하여 제한된 시료 부피를 사용할 수 있는 일상적인 설정에 적합합니다. 이 응용 프로그램은 클리닉에 대한 후속 방문의 필요성을 줄여 후속 조치에 대한 환자 손실 위험을 최소화 할 수 있습니다. 또한 이 방법은 간단하고 고급 실험실 전문 지식이 필요하지 않으므로 리소스가 제한된 환경으로의 통합을 더욱 지원합니다.
그러나 프로토콜에 대한 몇 가지 사항에 유의해야 합니다. 샘플은 80 ° C에서 배양 직후 동결하거나 냉장 보관해서는 안됩니다. 열처리 직후 급속 냉각으로 인해 튜브 내부 표면에 응결이 형성되어 샘플이 희석되고 다운스트림 DNA 추출에 영향을 미칠 수 있습니다. 또한 갑작스러운 냉각은 열처리 중에 완전히 비활성화되지 않았을 수 있는 핵산 단편화 또는 효소 활성을 촉진하여 샘플 분해 위험을 증가시킬 수 있습니다. 이러한 고려 사항은 MTB 검출을 위해 가래 샘플을 처리할 때 특히 중요합니다. Ziehl-Neelsen 염색에 대한 WHO 권장 사항에 따르면 도말 등급 1+는 100개의 오일 침지 당 10-99 AFB에 해당하고, 2+는 최소 50개 필드에서 필드당 1-10 AFB를 나타내며, 3+는 최소 20개 필드에서 필드당 10 AFB 이상을 나타냅니다12.
이 방법의 한계는 고속 균질화 장치 또는 비드 박동 장치를 사용한 수동 균질화에 대한 요구 사항으로 인해 자동화가 부족하다는 것입니다. 이 문제를 해결하기 위해 더 짧은 배양 시간, 더 낮은 배양 온도 및 고속 균질화에 대한 대체 접근 방식을 테스트하여 프로토콜을 개선하는 데 지속적인 노력이 집중되고 있습니다.
따라서 고속 균질화기와 결합된 이 매트릭스 기반 DNA 추출 방법은 열 및 비드 박동을 사용하여 게놈 DNA를 방출하는 동시에 금속 및 단백질과 같은 PCR 억제제를 제거하는 신속한 DNA 추출 기술입니다.
공개
저자 T. R.은 UC 샌디에이고와의 서비스 계약을 통해 FIND로부터 자금 지원을 받습니다. 저자 T. R.은 약물 내성 결핵(R01AI176401)에 대한 tNGS 솔루션을 개발하고 평가하기 위해 NIH로부터 보조금을 받았습니다. 저자 T. R.은 Verus Diagnostics Inc.의 공동 창립자, 이사회 구성원 및 무보수 주주입니다.
감사의 말
저자는 기술 지원을 제공한 남아프리카 공화국 그린 포인트의 NHLS의 결핵 실험실 팀에 감사를 표합니다. 이 작업은 미국 국립보건원(NIH)과 혁신신진단재단(FIND), TS ELiOT 프로젝트 보조금 ID: R01AI153213, Unitaid 보조금 ID: 2019-32-FIND MDR의 지원을 받았습니다. 피규어는 BioRender.com 사용하여 생성되었습니다.
자료
| Name | Company | Catalog Number | Comments |
| Alternative: Benchmark BeadBlaster 24 | Benchmark Scientific | D2400 | |
| AMPure XP Bead-Based Reagent | Beckman Coulter | A63881 | |
| Autoclaved or sterile glass beads (2 mm) | Sigma-Aldrich | Z273627-1EA | |
| Ethyl alcohol, Pure | Sigma-Aldrich | E7023-500ML | |
| Filter pipette tips (10 µL) | Bio-Smart Scientific | FT-10-R | |
| Filter pipette tips (1000 µL) | Bio-Smart Scientific | FT1000-R | |
| Filter pipette tips (20 µl) | Bio-Smart Scientific | FT20-R | |
| Filter pipette tips (200 µL) | Bio-Smart Scientific | FT200-R | |
| Heating-block ranging from 37 °C to 100 °C | Eppendorf | 5382000031 | |
| High-speed homogenizer (HSM) as FastPrep-24 | MP Biomedicals | 116005500 | |
| Incubator 80 °C | Lasec Group | IBLPDHG-9030A | |
| InstaGene Matrix | Bio-Rad Laboratories Inc. | BBRD7326030 | |
| Low-binding microtubes (1.5 mL or 2 mL) | Eppendorf | EP0030108051-250EA | |
| Magnetic rack for 1.5 – 2 mL tubes | Thermo Fisher Scientific Inc. | 12321D | |
| Magnetic stirrer | Merck | Z671886 | |
| Microcentrifuge | Eppendorf | 5406000046 | |
| Milli-Q IX 7015 Pure Water Purification System | Merck | ZIX7015T0C | |
| Nuclease-free water (NFW), molecular grade | Thermo Fisher Scientific Inc. | AM9937 | |
| Pipettes (P10, P20, P200, and P1000) | Eppendorf | EP3123000918-1EA | |
| Qubit Assay Tubes | Thermo Fisher Scientific Inc. | Q32856 | |
| Qubit dsDNA HS Assay Kit | Thermo Fisher Scientific Inc. | Q33231 | |
| Qubit fluorometer instrument | Thermo Fisher Scientific Inc. | Q33226 | |
| Screw cap tubes (1.5 mL) | Scientific Specialties Inc. | P2TUB056C-0001.5ST | |
| Vortex | Lasec Group | WMBB0L0E0216 |
참고문헌
- Global Tuberculosis Report 2024. Geneva: WHO. , World Health Organization. https://www.who.int/teams/global-tuberculosis-programme/tb-reports/global-tuberculosis-report-2024 (2024).
- De Araujo, L., et al. Implementation of targeted next-generation sequencing for the diagnosis of drug-resistant tuberculosis in low-resource settings: a programmatic model, challenges, and initial outcomes. Front Public Health. 11, 1204064(2023).
- Murphy, S. G., et al. Direct detection of drug-resistant Mycobacterium tuberculosis using targeted next generation sequencing. Front Public Health. 11, 1206056(2023).
- Kambli, P., et al. Targeted next generation sequencing directly from sputum for comprehensive genetic information on drug resistant Mycobacterium tuberculosis. Tuberculosis. 127, 102051(2021).
- WHO Consolidated Guidelines on Tuberculosis.Module 3: Diagnosis. Rapid diagnostics for tuberculosis detection. Third edition. , World Health Organization. https://iris.who.int/bitstream/handle/10665/376221/9789240089488-eng.pdf?sequence=1 (2024).
- Catalogue of mutations in Mycobacterium tuberculosis. complex and their association with drug resistance. Second edition. , World Health Organization. https://iris.who.int/bitstream/handle/10665/374061/9789240082410-eng.pdf?sequence=1 (2023).
- Schwab, T. C., et al. Targeted next-generation sequencing to diagnose drug-resistant tuberculosis: a systematic review and meta-analysis. Lancet Infect Dis. 24 (10), 1162-1176 (2024).
- Deeplex Myc-TB manual version 5. , GenoScreen. https://acrobat.adobe.com/link/track?uri=urn%3Aaaid%3Ascds%3AUS%3A67c2b53f-c91d-3a73-a6ea-4769e7c8fe57 (2023).
- Shea, J., et al. Comprehensive whole-genome sequencing and reporting of drug resistance profiles on clinical cases of Mycobacterium tuberculosis in New York State. J Clin Microbiol. 55 (6), 1871-1882 (2017).
- Conceição, E. C., et al. Mycobacterium tuberculosis. DNA extraction using InstaGene Matrix and high-speed homogenizer from clinical primary culture. , https://www.protocols.io/view/mycobacterium-tuberculosis-dna-extraction-using-in-dhvs366e (2024).
- Nilgiriwala, K., et al. Genomic sequencing from sputum for tuberculosis disease diagnosis, lineage determination, and drug susceptibility prediction. J Clin Microbiol. 61 (3), e0157822(2023).
- Laboratory Services in Tuberculosis Control Part II: Microscopy. Geneva: WHO. , World Health Organization. https://iris.who.int/bitstream/handle/10665/65942/WHO_TB_98.258_(part2).pdf?sequence=2 (1998).
재인쇄 및 허가
JoVE'article의 텍스트 или 그림을 다시 사용하시려면 허가 살펴보기
허가 살펴보기더 많은 기사 탐색
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. 판권 소유