Method Article
基于基质的 DNA 提取,用于对去污痰液样本进行靶向下一代测序
* 这些作者具有相同的贡献
摘要
我们提出了一种优化的从去污痰液中提取 DNA 的方法,该方法使用基于基质的提取方法结合磁珠纯化,用于结 核分枝杆菌的下游靶向下一代测序。
摘要
新一代测序 (NGS) 现在被认为是及时准确诊断耐药结核病 (DR-TB) 的有力工具。靶向 NGS (tNGS) 提供了一种简化的方法,它专注于与耐药性相关的特定基因,无需传统的基于培养的方法,周转时间从数周到数月不等。世界卫生组织 (WHO) 建议将 tNGS 作为改善结核病 (TB) 诊断的宝贵策略,以指导治疗和改善治疗结果,尤其是在资源有限的环境中。在 WHO 推荐的 tNGS 检测中,我们选择了一种提供快速和全面的药敏试验、谱系测定和菌株分型的方法。虽然可以使用标准化的 DNA 提取方法,但它们可能非常耗时且耗费人力。为了应对这一挑战,我们优化了一种简化的、基于基质的 DNA 提取方案,并结合磁珠纯化。该方法提供了一种直接从去污痰沉积物中提取 DNA 的快速有效方法,可实现快速的下游 tNGS 分析。通过简化从痰液沉积物中提取 DNA 的过程,该方案可以促进 tNGS 在常规临床环境中的广泛采用,最终有助于改善患者预后并为全球结核病控制工作做出贡献。
引言
据估计,到 2023 年,全球有 370 万结核病患者未得到诊断和治疗,这凸显了结核病对全球健康构成的重大威胁1。WHO 估计,到 2023 年,约有 400,000 人患上了利福平耐药 (RR-TB) 或耐多药结核病 (MDR-TB)1。快速诊断和治疗结核病和 DR-TB 对于实现降低结核病发病率和死亡率的里程碑和目标至关重要1。
依靠传统的培养方法和表型药物敏感性测试 (pDST) 会延迟确定临床分离株的耐药性和治疗,周转时间为 6-8 周,并且需要复杂的生物防护基础设施。常规诊断检测与 DR-TB 的 NGS 相结合,可以提供全面的耐药性概况并改善耐多药结核病方案的个性化,同时还可以将有效治疗的时间从数周或数月缩短到第 2,3,4 天。
2023 年和 2024 年,WHO 建议使用 tNGS 作为一类新的诊断方法,以快速确定对一线和二线抗结核药物的敏感性5。这使其成为指导治疗决策的宝贵工具,无需在生物安全 3 级 (BSL-3) 实验室中进行 结核分枝杆菌 (MTB) 培养。tNGS 方法是一种浓缩的测序形式,它使用聚合酶链反应 (PCR) 在测序前扩增赋予耐药性的基因靶标。在 WHO 推荐的 tNGS 检测中,我们选择了 Deeplex Myc-TB 检测,据报道,该检测符合检测对利福平、异烟肼、乙胺丁醇、吡嗪酰胺、氟喹诺酮类药物、阿米卡星、链霉素、利奈唑胺、贝达喹啉和氯法齐明耐药性的分类标准。我们使用该测定来评估使用此方案提取的 DNA 对下游 tNGS 的适用性。
此外,WHO 发布了 MTB 中与耐药性相关的突变目录第2 版, 为使用全基因组测序 (WGS) 和 tNGS 预测药物敏感性和指导治疗提供了路线图6。最近的一项系统评价和荟萃分析表明,与 pDST7 相比,tNGS 检测耐药性的灵敏度和特异性分别为 94.1% 和 98.1%,基于 MTB 基因组中各个耐药性赋予区域的 23 个靶标。
然而,由于与所需工作流程、基础设施和设备特别相关的复杂性和成本,这些方法的实施仍然具有挑战性。一个关键的挑战是直接从去污痰液的沉淀物中分离出足够的高质量分枝杆菌 DNA,这是下游 tNGS 应用的关键步骤。为了解决这个问题,我们提出了一种为 tNGS 量身定制的快速简单的 DNA 提取方法。
所选 MTB tNGS 检测的标准化 DNA 提取方法包括内部手动和自动方案8。在这里,我们描述了一种简化的、基于基质的 DNA 提取方案(图 1)。该方法利用 InstaGene 基质 (IGM),它结合金属和蛋白质,允许直接从去污的痰液沉积物中提取高质量的核酸。这种替代方案为下游 tNGS 提供了更快的周转时间和足够的 DNA 产量。该方案克服了手动和自动方法的复杂性,同时确保高质量的 tNGS 用于快速诊断赋予 MTB 耐药性的变异。随着人们对在分枝杆菌学领域使用 tNGS 的兴趣日益浓厚,该方案可以促进其在常规诊断工作流程中的采用。
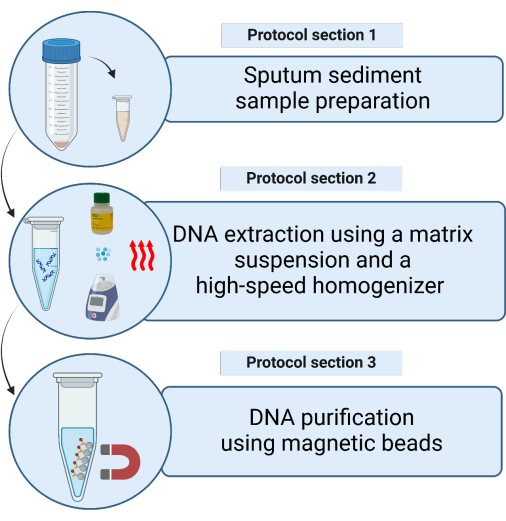
图 1:使用基质悬浮液从去污的痰液沉积物样品中提取分枝杆菌 DNA 的方法示意图。请单击此处查看此图的较大版本。
研究方案
这项研究已获得研究伦理委员会的批准:斯泰伦博斯大学生物与环境安全 (REC: BES):BES-2024-25384 和人类研究伦理委员会:N21/09/093 和 N09/11/296。
1. 痰液沉淀物样品制备
注:在 DNA 提取之前,应在生物安全 3 级 (BSL3) 实验室中执行以下步骤。
- 通过将 500 μL-2 mL 转移到 1.5 mL 或 2 mL 低结合管中,制备先前使用 N-乙酰-L-半胱氨酸 (NALC) 和氢氧化钠 (NaOH) (NALC/NaOH) 进行净化的痰液沉淀物样品的等分试样。
- 将含有痰液沉淀物样品的试管直立在试管架上,并将样品放入 80 °C 的烘箱中,孵育 1 小时以通过热处理灭活分枝杆菌。
- 1 小时后,取下装有样品的试管架,擦拭试管表面,然后将它们放入适当的容器中,以便运输到生物安全二级 (BSL2) 实验室。
注:在进一步处理或储存之前,让样品冷却至室温。
2. 使用基质悬液和高速匀浆器提取 DNA(图 2)
- 开始前,将三个直径为 2 mm 的玻璃珠加入 1.5 或 2 mL 螺口盖管中,松散盖上,然后高压灭菌(最好至少在 DNA 提取前一天)。
- 从冰箱中取出基质悬浮液,将其放在磁力搅拌器上(试剂瓶中附有磁铁),使试剂达到室温并开始混合试剂。
- 打开加热块并预热至 56 °C(如果有两个加热块可用,则将第二个预热至 100 °C。
注意:以下步骤可以在 BSL2 设施的实验室工作台上执行: - 将热灭活的痰液沉淀物样品以 16,800 × g (或全速)离心 15 分钟。用 20-200 μL 移液器轻轻吸出并丢弃上清液,确保沉淀不受干扰。
- 通过轻轻摇晃(不要涡旋)混合基质悬浮液,并加入 200 μL 充分混合的基质以重悬沉淀。
- 将全体积移液到包含三个直径为 2 mm 的预灭菌玻璃珠的 1.5 mL 螺旋盖管中。
- 将样品置于加热块中,并在 56 °C 下孵育 15 分钟。
注:从加热块中取出样品后,如果只有一个加热块可用,请在步骤 2.7 后立即将加热块设置为 100 °C。 - 使用涡旋将样品匀浆 10 秒以分散细胞。
- 将样品放入加热块中,并在 100 °C 下孵育 10 分钟。
- 使用具有以下参数的高速均质器对样品进行均质化:以 4.0 m/s 的速度进行一个 60 秒的循环。
- 以 12,000 × g 离心 5 分钟。将 130 μL 含 DNA 的上清液转移到新鲜的 1.5 mL 低结合管中,而不会干扰沉淀(含有细胞碎片的基质)。丢弃含有基质和细胞碎片的第一管。
注意:安全停止点。如果停止,则在 -20 °C 下冷冻样品,直到准备好继续 DNA 纯化。 - 使用磁珠进行 DNA 纯化。
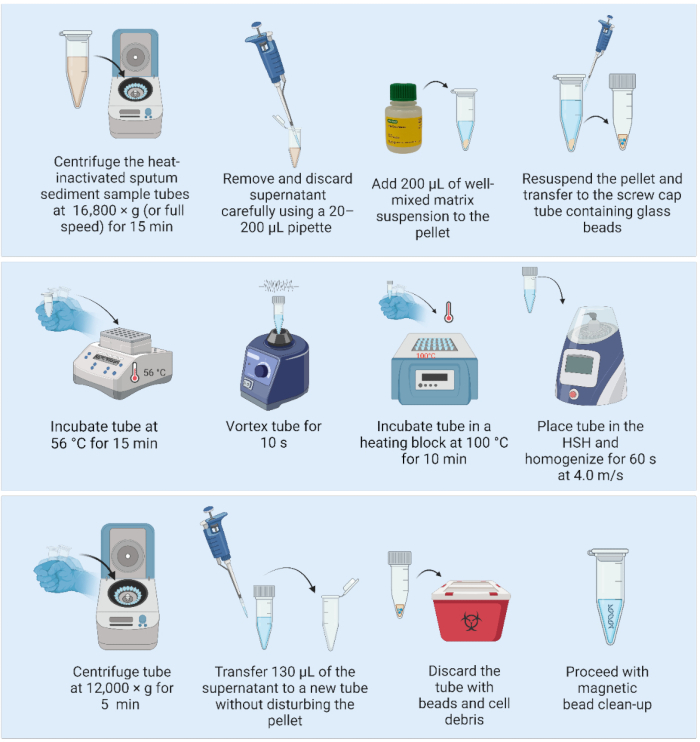
图 2:基于基质的 DNA 提取方法与高速匀浆器联用。 缩写:HSH = 高速均质机。 请单击此处查看此图的较大版本。
3. 使用磁珠纯化 DNA(图 3)
注:在进行 DNA 纯化之前,请执行步骤 3.1 和 3.2。
- 从冰箱中取出磁珠,将容器在室温下放置 30 分钟以平衡。或者,涡旋磁珠储备瓶以重悬所有磁珠,并将 1,280 μL 等分试样吸出到新试管中(用于纯化 21 个样品),并在室温下放置以平衡。
- 将 7.2 mL 纯乙醇添加到 1.8 mL 过滤和灭菌的水中,制备一批 9 mL 80% 乙醇。
注:以下步骤可以在 BSL2 设施的实验室工作台上进行。 - 彻底涡旋磁珠原液瓶或准备好的等分试样,以确保在使用前重新悬浮磁珠。每 10 个样品后重复涡旋,以确保悬浮液均匀。
- 向提取的 DNA (130 μL) 中加入 1.2 倍体积 (156 μL) 的磁珠。
注:对于含有大量 DNA(培养物)的样品,可以处理较小等分试样的样品(例如 50 μL)并用 1.2 倍体积的磁珠进行纯化。 - 通过移液 10 次混合样品。在室温下孵育样品 5 分钟。
- 将样品放在 1.5 mL 试管磁力架上 3 分钟或直到液体变得透明。小心吸出并丢弃上清液,不要干扰珠子。
- 将试管放在磁力架上,加入 200 μL 80% 乙醇,确保磁珠不受干扰并孵育 30 秒。
- 小心吸出并丢弃乙醇,不要干扰珠子。
- 重复步骤 3.7 总共洗涤两次。
- 第二次洗涤后,取出并丢弃乙醇。用 1-10 μL 移液器去除任何残留的乙醇。
- 将试管打开,将珠子风干 10 分钟,或直到珠子呈哑光外观。
注意:不要让珠子过度干燥和破裂。 - 一旦磁珠呈哑光外观,立即从磁珠中取出试管,并将 50 μL 无核酸酶水 (NFW) 直接添加到每个样品的磁珠上。将 NFW 添加到所有试管中后,通过移液 10 次混合每个单独的样品。检查试管中是否有卡在试管内侧的珠子,必要时重复混合步骤。在室温下孵育样品 5 分钟。
注意:孵育时间可以从 5 分钟增加到 10 分钟,但不要将孵育时间减少到 5 分钟以下。 - 将试管放回磁力架上 3 分钟或直到液体澄清。将试管放在磁力架上,将含 DNA 的上清液转移到标记清晰、无菌、低结合的试管中。确保珠子没有转移到上清液中。
注:如果在转移过程中移液器吸头中可以看到珠子,请将上清液移回磁力架上的试管中,再分离 3 分钟。不要将微珠转移到新管中。
安全停止点。如果停止,将样品在 -20 °C 下冷冻,直到准备好继续使用适合 NGS 的方法进行定量,并随后使用 MTB tNGS 检测进行 PCR 扩增。
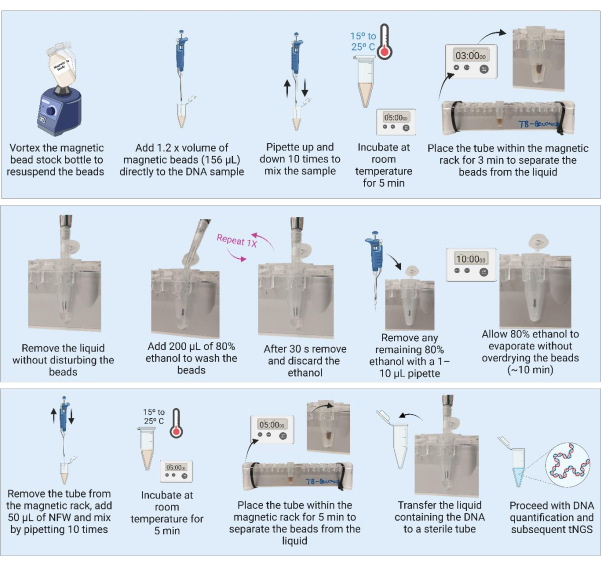
图 3:使用磁珠进行 DNA 纯化和浓缩。 缩写:NFW = 无核酸酶水;tNGS = 靶向下一代测序。 请单击此处查看此图的较大版本。
结果
样品描述
南非开普敦国家卫生实验室服务 (NHLS) 绿点定期收集和处理了总共 165 份痰沉积物样本,显微镜检查抗酸杆菌 (AFB) 阳性,细菌载量至少为 1+。使用两种不同体积 [约 2 mL (n = 102) 和 500 μL (n = 63)] 从痰液沉淀物样品中提取 DNA。进行这种比较是为了评估较小的沉积物样品体积是否可以为下游 tNGS 产生足够的 DNA。
图 4 显示了不同痰涂片等级(1+、2+ 和 3+)和痰液沉淀物体积(2 mL 和 500 μL)的总 DNA 浓度 (ng/μL) 的箱线图比较。使用 500 μL 沉淀物提取时,DNA 产量通常会总体下降,并且不同弥散条带等级之间存在差异。因此,从体积至少为 2 mL 的沉积物样品中提取的平均 DNA 浓度平均高于从 500 μL 沉积物中提取的 DNA 浓度,按样品的 AFB 分层。
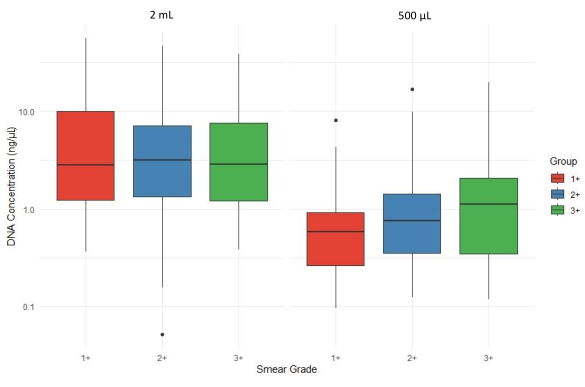
图 4:痰液沉淀物体积对不同弥散条带等级的 DNA 产量的影响。请点击此处查看此图的较大版本。
图 5 显示了箱线图,说明了从两个不同起始体积(2 mL 和 500 μL)处理的不同弥散条带等级(1+、2+ 和 3+)的痰沉积物样品的所有靶标的平均测序读数(平均覆盖深度)。异常值由单个点表示,y 轴以对数刻度显示,以适应覆盖范围的变化。从 500 μL 沉积物中提取的 3+ 样品的高四分位距表明,细菌载量较高时变异性更大。
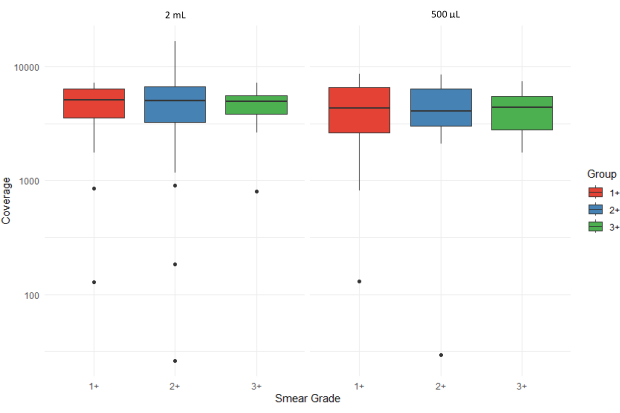
图 5:不同弥散条带等级和样品起始量的所有靶标的平均测序读长数(覆盖深度)的比较。 请单击此处查看此图的较大版本。
图 6 描述了使用相关 Web 应用程序的生物信息学管道对测序结果可接受性的评估。测序质量分数分为 +++(高度可接受)、++(可接受)、+(勉强可接受)、-(不可接受)和 ND(未确定)。图表中没有条形表示没有获得该特定类别的结果。对于 500 μL 起始体积样品组,与 2+ 和 3+ 级弥散条带相比,1+ 级弥散条带显示不可接受和 ND 结果的比例更高。从 500 μL 痰液沉淀物中提取的样品的平均测序覆盖深度为 4,316,而从 2 mL 痰液沉淀物中提取的样品为 4,810 个。这表明从痰液沉淀物中提取的 DNA 足以进行下游 tNGS,而与起始量无关。
根据测序结果可接受性的分布,弥散条带等级为 3+ 的样本成功率最高,与 1+ 和 2+ 弥散条带等级的样本相比,大多数样本序列的评分为 ++ 和 +++。对于 500 μL 起始样品,与 2 mL 起始样品相比,所有弥散条带等级中 - 和 + 类样品的比例相对较高。这表明 500 μL 起始量的样品属于质量较低、可接受性较差的类别。对于 2 mL 起始样品,高质量 ++ 和 +++ 类别中的样品比例较高,尤其是 3+ 级弥散条带。这表明,与起始量为 500 μL 的样品相比,起始量为 2 mL 的样品更有可能产生更好的测序质量评分。
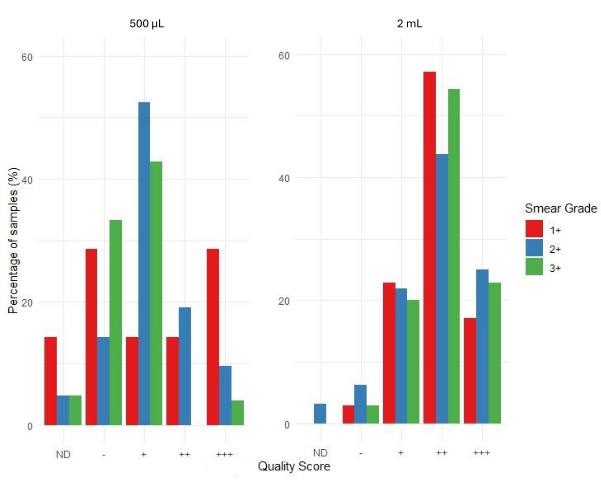
图 6:按弥散条带等级和样品起始量划分的质量评分分布。请点击此处查看此图的较大版本。
讨论
基于基质的萃取方法与高速均质器相结合的文章最初由 Shea 等人发表9。该方法针对从 MTBC 培养物中提取的 DNA 的 WGS 进行开发和优化 9,10。我们优化了该方法以用于 MTB tNGS 测定。21 份样品的 DNA 提取和纯化的综合周转时间为 4 h 和 40 min,包括孵育和离心步骤。根据平均产量(图 4)、所有靶标组合的总体覆盖深度(图 5)和由相关 Web 应用程序确定的测序结果可接受性评分(图 6),证明了所述方案与珠子纯化相结合的可靠性,用于从弥散条带等级为 1+ 到 3+ 的样品中提取 DNA。
最近的一项研究发现,弥散条带等级与 DNA 浓度或测序读长深度之间没有一致的关系。作者认为,样品处理和净化步骤的可变性可能会影响高弥散条带级样品的性能,尤其是在无法有效去除污染物的情况下。此外,由于 DNA 库的复杂性,从痰沉积物中测序仍然具有挑战性,其中非靶标 DNA 可以与 MTB DNA11 竞争。虽然我们没有观察到输入 DNA 浓度和测序深度之间有任何明显的相关性,但重要的是要注意,根据制造商的说明,在靶标 PCR 之前 DNA 输入不应超过 100 ng,因为这可能导致 PCR 抑制。此外,较高的细菌载量、2+ 和 3+ 痰沉积物样品可能含有更多的污染物,例如人类 DNA、细胞碎片和其他抑制剂,这些污染物会干扰 PCR 扩增和文库制备。
纯化步骤还用作浓缩步骤,在 tNGS 之前提高分枝杆菌 DNA 的产量。该方法可有效处理细菌载量低的样品,并且在体积低至 500 μL 的情况下表现出可靠的性能,使其适用于样品量有限的常规设置。它的应用可以减少对诊所的随访需求,从而最大限度地降低患者失访的风险。此外,该方法简单,不需要高级实验室专业知识,进一步支持其整合到资源有限的环境中。
但是,应注意有关该协议的一些要点。样品在 80 °C 下孵育后不应立即冷冻或冷藏。 热处理后立即快速冷却会导致试管内表面形成冷凝水,导致样品稀释并可能影响下游 DNA 提取。此外,突然冷却可能会促进核酸片段化或热处理过程中可能未完全灭活的酶活性,从而增加样品降解的风险。在处理痰液样本以进行 MTB 检测时,这些考虑因素尤其重要。根据 WHO 对 Ziehl-Neelsen 染色的建议,涂片等级 1+ 对应于每 100 个油浸场 10-99 个 AFB,2+ 表示至少 50 个油田中每个油田 1-10 个 AFB,3+ 表示至少 20 个油田中每个油田超过 10 个 AFB12。
这种方法的一个局限性是由于需要使用高速均质器或打珠装置进行手动均质化,因此缺乏自动化。为了解决这个问题,正在进行的工作重点是通过测试更短的孵育时间、更低的孵育温度和高速均质化的替代方法来完善方案。
因此,这种基于基质的 DNA 提取方法与高速匀浆器相结合,是一种快速 DNA 提取技术,它采用加热和磁珠跳动来释放基因组 DNA,同时去除 PCR 抑制剂,例如金属和蛋白质。
披露声明
作者 TR 通过与加州大学圣地亚哥分校的服务合同获得 FIND 的资金支持。作者 TR 获得了 NIH 的赠款,用于开发和评估耐药结核病 (R01AI176401) 的 tNGS 解决方案。作者 TR 是 Verus Diagnostics Inc. 的联合创始人、董事会成员和无薪股东。
致谢
作者要感谢南非 NHLS - Green Point 的结核病实验室团队提供的技术援助。这项工作得到了美国国立卫生研究院 (NIH) 和创新新诊断基金会 (FIND)、TS ELiOT 项目资助 ID:R01AI153213、国际药品采购机制资助 ID:2019-32-FIND MDR 的支持。图是使用 BioRender.com 创建的。
材料
| Name | Company | Catalog Number | Comments |
| Alternative: Benchmark BeadBlaster 24 | Benchmark Scientific | D2400 | |
| AMPure XP Bead-Based Reagent | Beckman Coulter | A63881 | |
| Autoclaved or sterile glass beads (2 mm) | Sigma-Aldrich | Z273627-1EA | |
| Ethyl alcohol, Pure | Sigma-Aldrich | E7023-500ML | |
| Filter pipette tips (10 µL) | Bio-Smart Scientific | FT-10-R | |
| Filter pipette tips (1000 µL) | Bio-Smart Scientific | FT1000-R | |
| Filter pipette tips (20 µl) | Bio-Smart Scientific | FT20-R | |
| Filter pipette tips (200 µL) | Bio-Smart Scientific | FT200-R | |
| Heating-block ranging from 37 °C to 100 °C | Eppendorf | 5382000031 | |
| High-speed homogenizer (HSM) as FastPrep-24 | MP Biomedicals | 116005500 | |
| Incubator 80 °C | Lasec Group | IBLPDHG-9030A | |
| InstaGene Matrix | Bio-Rad Laboratories Inc. | BBRD7326030 | |
| Low-binding microtubes (1.5 mL or 2 mL) | Eppendorf | EP0030108051-250EA | |
| Magnetic rack for 1.5 – 2 mL tubes | Thermo Fisher Scientific Inc. | 12321D | |
| Magnetic stirrer | Merck | Z671886 | |
| Microcentrifuge | Eppendorf | 5406000046 | |
| Milli-Q IX 7015 Pure Water Purification System | Merck | ZIX7015T0C | |
| Nuclease-free water (NFW), molecular grade | Thermo Fisher Scientific Inc. | AM9937 | |
| Pipettes (P10, P20, P200, and P1000) | Eppendorf | EP3123000918-1EA | |
| Qubit Assay Tubes | Thermo Fisher Scientific Inc. | Q32856 | |
| Qubit dsDNA HS Assay Kit | Thermo Fisher Scientific Inc. | Q33231 | |
| Qubit fluorometer instrument | Thermo Fisher Scientific Inc. | Q33226 | |
| Screw cap tubes (1.5 mL) | Scientific Specialties Inc. | P2TUB056C-0001.5ST | |
| Vortex | Lasec Group | WMBB0L0E0216 |
参考文献
- Global Tuberculosis Report 2024. Geneva: WHO. , World Health Organization. https://www.who.int/teams/global-tuberculosis-programme/tb-reports/global-tuberculosis-report-2024 (2024).
- De Araujo, L., et al. Implementation of targeted next-generation sequencing for the diagnosis of drug-resistant tuberculosis in low-resource settings: a programmatic model, challenges, and initial outcomes. Front Public Health. 11, 1204064(2023).
- Murphy, S. G., et al. Direct detection of drug-resistant Mycobacterium tuberculosis using targeted next generation sequencing. Front Public Health. 11, 1206056(2023).
- Kambli, P., et al. Targeted next generation sequencing directly from sputum for comprehensive genetic information on drug resistant Mycobacterium tuberculosis. Tuberculosis. 127, 102051(2021).
- WHO Consolidated Guidelines on Tuberculosis.Module 3: Diagnosis. Rapid diagnostics for tuberculosis detection. Third edition. , World Health Organization. https://iris.who.int/bitstream/handle/10665/376221/9789240089488-eng.pdf?sequence=1 (2024).
- Catalogue of mutations in Mycobacterium tuberculosis. complex and their association with drug resistance. Second edition. , World Health Organization. https://iris.who.int/bitstream/handle/10665/374061/9789240082410-eng.pdf?sequence=1 (2023).
- Schwab, T. C., et al. Targeted next-generation sequencing to diagnose drug-resistant tuberculosis: a systematic review and meta-analysis. Lancet Infect Dis. 24 (10), 1162-1176 (2024).
- Deeplex Myc-TB manual version 5. , GenoScreen. https://acrobat.adobe.com/link/track?uri=urn%3Aaaid%3Ascds%3AUS%3A67c2b53f-c91d-3a73-a6ea-4769e7c8fe57 (2023).
- Shea, J., et al. Comprehensive whole-genome sequencing and reporting of drug resistance profiles on clinical cases of Mycobacterium tuberculosis in New York State. J Clin Microbiol. 55 (6), 1871-1882 (2017).
- Conceição, E. C., et al. Mycobacterium tuberculosis. DNA extraction using InstaGene Matrix and high-speed homogenizer from clinical primary culture. , https://www.protocols.io/view/mycobacterium-tuberculosis-dna-extraction-using-in-dhvs366e (2024).
- Nilgiriwala, K., et al. Genomic sequencing from sputum for tuberculosis disease diagnosis, lineage determination, and drug susceptibility prediction. J Clin Microbiol. 61 (3), e0157822(2023).
- Laboratory Services in Tuberculosis Control Part II: Microscopy. Geneva: WHO. , World Health Organization. https://iris.who.int/bitstream/handle/10665/65942/WHO_TB_98.258_(part2).pdf?sequence=2 (1998).
转载和许可
请求许可使用此 JoVE 文章的文本或图形
请求许可探索更多文章
This article has been published
Video Coming Soon
版权所属 © 2025 MyJoVE 公司版权所有,本公司不涉及任何医疗业务和医疗服务。