Method Article
Estrazione del DNA basata su matrice per il sequenziamento mirato di nuova generazione su campioni di espettorato decontaminato
* Questi autori hanno contribuito in egual misura
In questo articolo
Riepilogo
Presentiamo un metodo ottimizzato per l'estrazione del DNA dall'espettorato decontaminato utilizzando un metodo di estrazione basato su matrice combinato con una purificazione a microsfere magnetiche per il sequenziamento mirato a valle di nuova generazione del Mycobacterium tuberculosis.
Abstract
Il sequenziamento di nuova generazione (NGS) è ora riconosciuto come un potente strumento per una diagnosi tempestiva e accurata della tubercolosi resistente ai farmaci (DR-TB). L'NGS mirato (tNGS) offre un approccio semplificato concentrandosi su geni specifici associati alla resistenza ai farmaci, bypassando la necessità di metodi tradizionali basati sulla coltura con tempi di risposta che vanno da settimane a mesi. L'Organizzazione Mondiale della Sanità (OMS) ha raccomandato il tNGS come strategia preziosa per migliorare la diagnosi della tubercolosi (TB) al fine di guidare il trattamento e migliorare i risultati del trattamento, in particolare in contesti con risorse limitate. Tra i test tNGS raccomandati dall'OMS, abbiamo selezionato un metodo che fornisce test rapidi e completi di suscettibilità ai farmaci, determinazione del lignaggio e tipizzazione del ceppo. Sebbene siano disponibili metodi standardizzati di estrazione del DNA, possono richiedere molto tempo e lavoro. Per affrontare questa sfida, abbiamo ottimizzato un protocollo di estrazione del DNA semplificato e basato su matrice, in combinazione con la purificazione con biglie magnetiche. Questo metodo offre un approccio rapido ed efficiente per l'estrazione del DNA direttamente dai sedimenti dell'espettorato decontaminati, consentendo una rapida analisi tNGS a valle. Semplificando il processo di estrazione del DNA dal sedimento dell'espettorato, questo protocollo potrebbe facilitare una più ampia adozione del tNGS in contesti clinici di routine, contribuendo in ultima analisi a migliorare gli esiti dei pazienti e contribuendo agli sforzi globali di controllo della tubercolosi.
Introduzione
Si stima che nel 2023 circa 3,7 milioni di persone affette da tubercolosi non siano state diagnosticate e trattate a livello globale, evidenziando la significativa minaccia rappresentata dalla tubercolosi per la salute globale1. L'OMS stima che nel 2023 circa 400.000 persone abbiano sviluppato una tubercolosi resistente alla rifampicina (RR-TB) o multiresistente ai farmaci (MDR-TB)1. La diagnosi e il trattamento rapido della TB e della DR-TB è essenziale per raggiungere i traguardi e gli obiettivi di riduzione dell'incidenza e della mortalità della TBC1.
L'affidamento sui metodi di coltura convenzionali e sui test di suscettibilità ai farmaci fenotipici (pDST) ritarda la determinazione del profilo di resistenza degli isolati clinici e del trattamento, con tempi di consegna di 6-8 settimane, e richiede una complessa infrastruttura di biocontenimento. I test diagnostici di routine, in combinazione con l'NGS della DR-TB, possono fornire un profilo completo di resistenza ai farmaci e migliorare la personalizzazione dei regimi di MDR-TB, riducendo al contempo il tempo per un trattamento efficace da settimane o mesi a giorni 2,3,4.
Nel 2023 e nel 2024, l'OMS ha raccomandato l'uso del tNGS come nuova classe di diagnostica per determinare rapidamente la suscettibilità ai farmaci antitubercolari di prima e seconda linea5. Questo lo rende uno strumento prezioso per guidare le decisioni terapeutiche senza la necessità di colture di Mycobacterium tuberculosis (MTB) nei laboratori di livello di biosicurezza 3 (BSL-3). Un approccio tNGS è una forma condensata di sequenziamento che utilizza la reazione a catena della polimerasi (PCR) per amplificare i bersagli genici che conferiscono resistenza ai farmaci prima del sequenziamento. Tra i test tNGS raccomandati dall'OMS, abbiamo selezionato il test Deeplex Myc-TB, che è stato segnalato per soddisfare i criteri basati sulla classe per rilevare la resistenza a rifampicina, isoniazide, etambutolo, pirazinamide, fluorochinoloni, amikacina, streptomicina, linezolid, bedaquilina e clofazimina. Utilizziamo questo test per valutare l'idoneità del DNA estratto utilizzando questo protocollo per il tNGS a valle.
Inoltre, l'OMS ha pubblicato laseconda edizione del catalogo delle mutazioni associate alla resistenza ai farmaci nella MTB, fornendo una tabella di marcia per l'uso del sequenziamento dell'intero genoma (WGS) e del tNGS per prevedere la suscettibilità ai farmaci e guidare il trattamento6. Una recente revisione sistematica e una meta-analisi hanno mostrato che il tNGS aveva una sensibilità e una specificità rispettivamente del 94,1% e del 98,1% per il rilevamento della resistenza ai farmaci, sulla base di 23 bersagli in varie regioni che conferiscono resistenza nel genoma MTB, rispetto a pDST7.
Tuttavia, l'implementazione di questi metodi rimane impegnativa a causa della complessità e dei costi specificamente associati ai flussi di lavoro, all'infrastruttura e alle attrezzature necessarie. Una sfida critica è isolare una quantità sufficiente di DNA micobatterico di alta qualità direttamente dal sedimento dell'espettorato decontaminato, un passaggio cruciale per le applicazioni tNGS a valle. Per risolvere questo problema, presentiamo un metodo di estrazione del DNA rapido e semplice su misura per tNGS.
Il metodo standardizzato di estrazione del DNA per il test MTB tNGS selezionato include un protocollo manuale e automatizzato interno8. Qui descriviamo un protocollo di estrazione del DNA semplificato e basato su matrice (Figura 1). Il metodo si avvale della matrice InstaGene (IGM), che lega metalli e proteine consentendo un'estrazione di qualità dell'acido nucleico direttamente dai sedimenti dell'espettorato decontaminati. Questa alternativa fornisce un tempo di risposta più rapido e una resa di DNA sufficiente per il tNGS a valle. Questo protocollo supera le complessità dei metodi manuali e automatizzati, garantendo al contempo un tNGS di qualità per la diagnosi rapida delle varianti che conferiscono resistenza in MTB. Con il crescente interesse per l'utilizzo del tNGS nel campo della micobatteriologia, questo protocollo potrebbe facilitarne l'adozione nei flussi di lavoro diagnostici di routine.
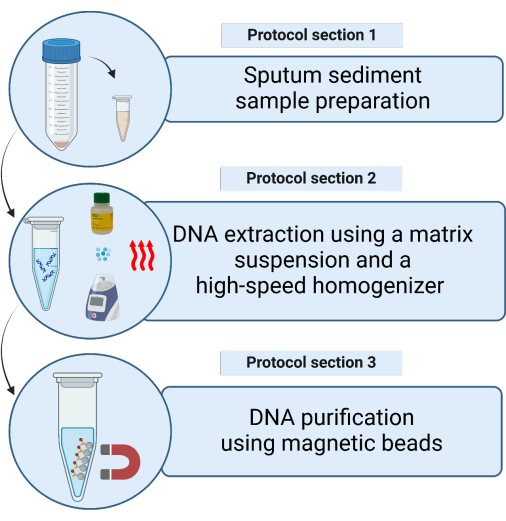
Figura 1: Rappresentazione schematica dei metodi per estrarre il DNA micobatterico da campioni di sedimenti di espettorato decontaminati utilizzando una sospensione di matrice. Fare clic qui per visualizzare una versione più grande di questa figura.
Protocollo
Questa ricerca è stata approvata dal Comitato Etico per la Ricerca: Sicurezza Biologica e Ambientale (REC: BES) dell'Università di Stellenbosch: BES-2024-25384 e dal Comitato Etico per la Ricerca Umana: N21/09/093 e N09/11/296.
1. Preparazione del campione di sedimento di espettorato
NOTA: I seguenti passaggi devono essere eseguiti in un laboratorio di biosicurezza di livello tre (BSL3) prima dell'estrazione del DNA.
- Preparare un'aliquota del campione di sedimento dell'espettorato precedentemente decontaminato utilizzando N-acetil-L-cisteina (NALC) e idrossido di sodio (NaOH) (NALC/NaOH) trasferendo 500 μL-2 mL in una provetta a basso legame da 1,5 mL o 2 mL.
- Collocare le provette contenenti i campioni di sedimento di espettorato in posizione verticale in un rack per provette e porre i campioni in un forno a 80 °C e incubare per 1 ora per inattivare i micobatteri attraverso il trattamento termico.
- Dopo 1 ora, rimuovere il rack per provette con i campioni, pulire le superfici delle provette e metterle in un contenitore appropriato per il trasporto in un laboratorio di livello di biosicurezza due (BSL2).
NOTA: Lasciare raffreddare i campioni a temperatura ambiente prima di continuare a maneggiarli o conservarli.
2. Estrazione del DNA mediante la sospensione della matrice e un omogeneizzatore ad alta velocità (Figura 2)
- Prima di iniziare, aggiungere tre perle di vetro di 2 mm di diametro a provette con tappo a vite da 1,5 o 2 mL, tappate senza stringere e sterilizzare in autoclave (idealmente almeno un giorno prima dell'estrazione del DNA).
- Rimuovere la sospensione della matrice dal frigorifero e posizionarla su un agitatore magnetico (un magnete è incluso nel flacone del reagente) per portare il reagente a temperatura ambiente e iniziare a miscelare il reagente.
- Accendere il blocco riscaldante e preriscaldare a 56 °C (se sono disponibili due blocchi riscaldanti, preriscaldare il secondo a 100 °C.
NOTA: I seguenti passaggi possono essere eseguiti su un banco da laboratorio in una struttura BSL2: - Centrifugare il campione di sedimento di espettorato inattivato termicamente a 16.800 × g (o alla massima velocità) per 15 minuti. Con una pipetta da 20-200 μl, aspirare delicatamente ed eliminare il surnatante, assicurandosi che il pellet rimanga indisturbato.
- Mescolare la sospensione della matrice agitando delicatamente (non agitare) e aggiungere 200 μl della matrice ben miscelata per risospendere il pellet.
- Pipettare l'intero volume in una provetta con tappo a vite da 1,5 mL contenente tre perle di vetro presterilizzate di 2 mm di diametro.
- Collocare i campioni in un blocco riscaldante e incubare a 56 °C per 15 minuti.
NOTA: Subito dopo aver rimosso il campione dal blocco riscaldante, impostare il blocco riscaldante a 100 °C dopo il passaggio 2.7, se è disponibile un solo blocco riscaldante. - Omogeneizzare i campioni utilizzando un vortice per 10 s per disperdere le cellule.
- Collocare i campioni in un blocco riscaldante e incubare a 100 °C per 10 minuti.
- Omogeneizzare i campioni utilizzando un omogeneizzatore ad alta velocità con i seguenti parametri: un ciclo di 60 s a 4,0 m/s.
- Centrifugare a 12.000 × g per 5 min. Trasferire 130 μl del surnatante contenente DNA in una provetta fresca da 1,5 mL a basso legame senza disturbare il pellet (matrice con detriti cellulari). Scartare la prima provetta contenente la matrice e i detriti cellulari.
NOTA: PUNTO DI ARRESTO SICURO. In caso di interruzione, congelare i campioni a -20 °C fino a quando non sono pronti per continuare con la purificazione del DNA. - Procedere con la purificazione del DNA utilizzando biglie magnetiche.
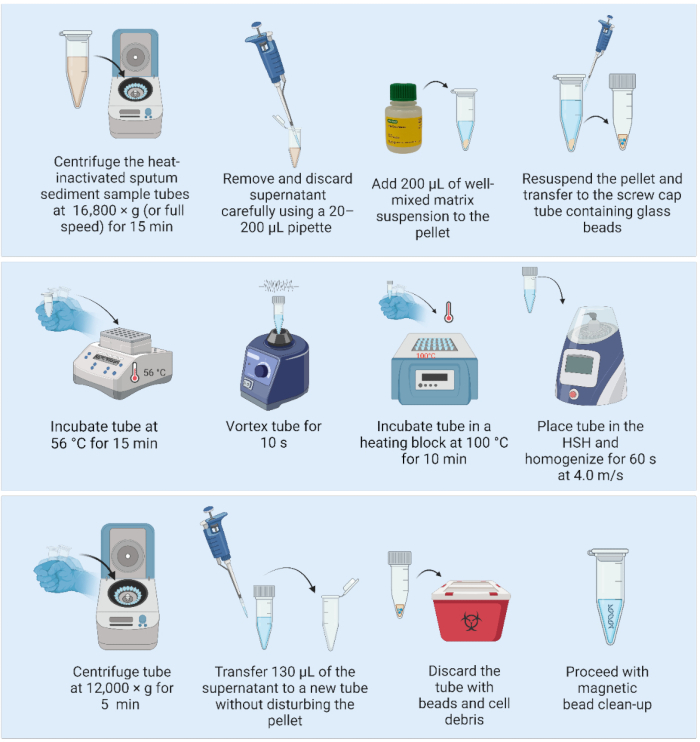
Figura 2: Metodo di estrazione del DNA basato su matrice accoppiato a un omogeneizzatore ad alta velocità. Abbreviazione: HSH = omogeneizzatore ad alta velocità. Clicca qui per visualizzare una versione più grande di questa figura.
3. Purificazione del DNA mediante biglie magnetiche (Figura 3)
NOTA: Prima di procedere con la purificazione del DNA, eseguire i passaggi 3.1 e 3.2.
- Togliete le perle magnetiche dal frigorifero e mettete il contenitore a temperatura ambiente per 30 minuti per equilibrare. In alternativa, agitare il flacone di supporto a microsfere magnetiche per risospendere tutte le perle e aspirare un'aliquota da 1.280 μl in una nuova provetta (per la purificazione di 21 campioni) e posizionarla a temperatura ambiente per equilibrarla.
- Preparare un lotto da 9 ml di etanolo all'80% aggiungendo 7,2 ml di alcol etilico puro a 1,8 ml di acqua filtrata e sterilizzata.
NOTA: I seguenti passaggi possono essere eseguiti su un banco da laboratorio in una struttura BSL2. - Agitare accuratamente il flacone di perline magnetiche o l'aliquota preparata per assicurarsi che le perle vengano risospese prima dell'uso. Ripetere il vortice ogni 10 campioni per garantire una sospensione omogenea.
- Aggiungere 1,2 volte il volume (156 μL) delle microsfere magnetiche al DNA estratto (130 μL).
NOTA: Per i campioni con quantità maggiori di DNA (coltura), è possibile elaborare un'aliquota più piccola del campione (ad es. 50 μL) e purificarla con 1,2 volte il volume di microsfere magnetiche. - Miscelare i campioni pipettando 10 volte. Incubare i campioni per 5 minuti a temperatura ambiente.
- Posizionare i campioni sul rack magnetico per provette da 1,5 mL per 3 minuti o fino a quando il liquido non diventa limpido. Aspirare e scartare con cura il surnatante senza disturbare le perline.
- Con le provette sul magnete, aggiungere 200 μl di etanolo all'80%, assicurandosi che le perle rimangano indisturbate e lasciate incubare per 30 s.
- Aspirare e scartare con cura l'etanolo senza disturbare le perline.
- Ripetere il passaggio 3.7 per un totale di due lavaggi.
- Dopo il secondo lavaggio, rimuovere e scartare l'etanolo. Rimuovere l'etanolo residuo con una pipetta da 1-10 μl.
- Lasciare i tubi aperti per asciugare le perline all'aria per 10 minuti, o fino a quando le perline non hanno un aspetto opaco.
NOTA: Non lasciare che le perline si asciughino eccessivamente e si incrino. - Una volta che le perle hanno un aspetto opaco, rimuovere immediatamente le provette dal magnete e aggiungere 50 μL di acqua priva di nucleasi (NFW) direttamente sulle perle in ciascun campione. Dopo aver aggiunto NFW a tutte le provette, miscelare ogni singolo campione pipettando 10 volte. Ispezionare il tubo per eventuali perline che sono bloccate all'interno del tubo e ripetere la fase di miscelazione se necessario. Incubare i campioni per 5 minuti a temperatura ambiente.
NOTA: L'incubazione può essere aumentata da 5 minuti a 10 minuti, ma non ridurre il tempo di incubazione a meno di 5 minuti. - Riposizionare le provette sulla griglia magnetica per 3 minuti o fino a quando il liquido non è limpido. Con le provette sul rack magnetico, trasferire il surnatante contenente DNA in una provetta chiaramente contrassegnata, sterile e a basso legame. Assicurarsi che le perle non vengano trasferite al surnatante.
NOTA: Se durante il trasferimento sono visibili delle perline nel puntale della pipetta, pipettare nuovamente il surnatante nella provetta sulla rastrelliera magnetica per separare per altri 3 minuti. Non trasferire le perline nel nuovo tubo.
PUNTO DI ARRESTO SICURO. In caso di interruzione, congelare i campioni a -20 °C fino al momento di continuare con la quantificazione utilizzando un metodo appropriato per NGS e la successiva amplificazione PCR utilizzando un test MTB tNGS.
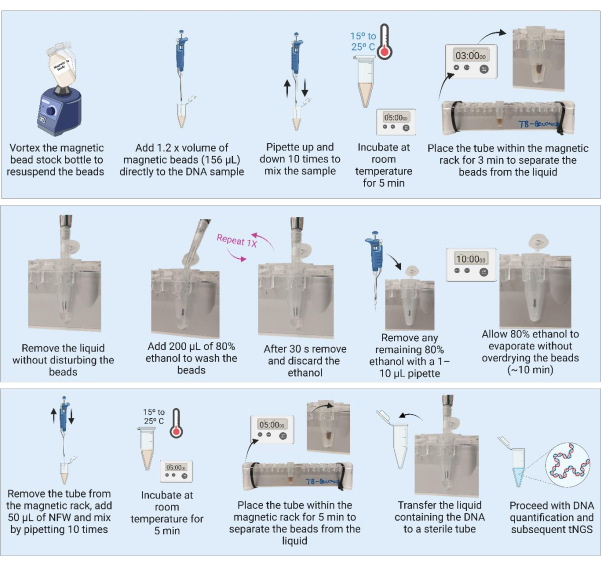
Figura 3: Purificazione e concentrazione del DNA mediante biglie magnetiche. Abbreviazioni: NFW = acqua priva di nucleasi; tNGS = sequenziamento mirato di nuova generazione. Clicca qui per visualizzare una versione più grande di questa figura.
Risultati
Descrizione del campione
Un totale di 165 campioni di sedimenti di espettorato, positivi per bacilli acido-resistenti (AFB) al microscopio con una carica batterica di almeno 1+, sono stati raccolti ed elaborati di routine dal National Health Laboratory Services (NHLS) Green Point, Città del Capo, Sud Africa. Il DNA è stato estratto da campioni di sedimenti di espettorato utilizzando due volumi diversi [circa 2 mL (n = 102) e 500 μL (n = 63)]. Questo confronto è stato condotto per valutare se il volume del campione di sedimento più piccolo potesse produrre DNA sufficiente per il tNGS a valle.
La Figura 4 mostra un confronto boxplot della concentrazione totale di DNA (ng/μL) in diversi gradi di striscio di espettorato (1+, 2+ e 3+) e volumi di sedimenti di espettorato (2 mL e 500 μL). La resa di DNA generalmente diminuisce complessivamente quando si utilizzano 500 μL di sedimento per l'estrazione, con variabilità tra i gradi di striscio. Pertanto, le concentrazioni medie di DNA estratte da campioni di sedimenti con un volume di almeno 2 mL erano in media superiori alla concentrazione di DNA estratta da sedimenti di 500 μL, stratificata per AFB del campione.
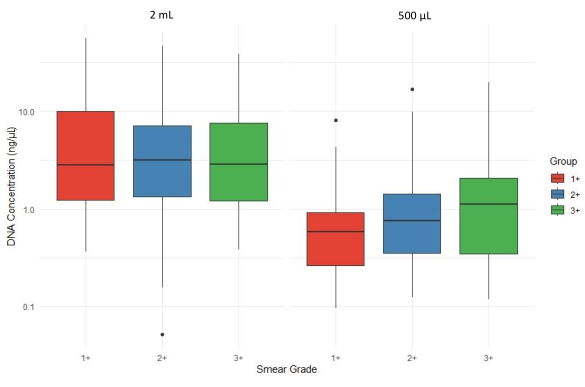
Figura 4: Impatto del volume del sedimento dell'espettorato sulla resa del DNA attraverso i gradi di striscio. Fare clic qui per visualizzare una versione più grande di questa figura.
La Figura 5 mostra i boxplot che illustrano il numero medio di letture di sequenziamento (profondità di copertura media) di tutti i target per campioni di sedimenti di espettorato con diversi gradi di striscio (1+, 2+ e 3+) elaborati da due diversi volumi di input (2 mL e 500 μL). I valori anomali sono rappresentati da singoli punti e l'asse y viene visualizzato su una scala logaritmica per adattarsi alle variazioni di copertura. L'elevato intervallo interquartile per i campioni 3+ estratti dai sedimenti da 500 μL suggerisce una maggiore variabilità con cariche batteriche più elevate.
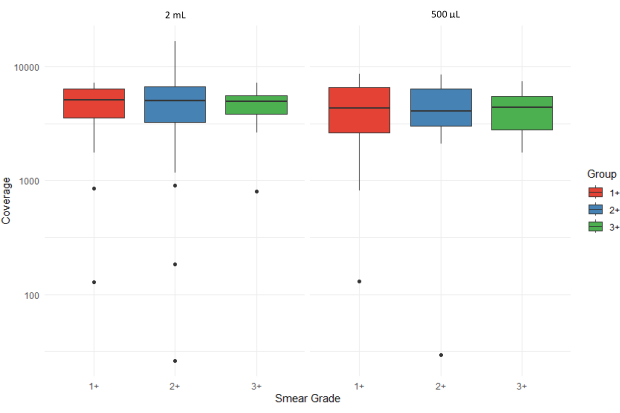
Figura 5: Confronto del numero medio di letture di sequenziamento (profondità di copertura) per tutti i target attraverso i gradi di striscio e i volumi di input del campione. Clicca qui per visualizzare una versione più grande di questa figura.
La Figura 6 illustra la valutazione dell'accettabilità dei risultati del sequenziamento utilizzando la pipeline bioinformatica dell'applicazione web associata. I punteggi di qualità del sequenziamento sono classificati come +++ (altamente accettabile), ++ (accettabile), + (marginalmente accettabile), - (inaccettabile) e ND (non determinato). L'assenza di una barra nel grafico indica che non sono stati ottenuti risultati per quella specifica categoria. Il grado di striscio 1+ mostra una percentuale più elevata di risultati inaccettabili e ND rispetto ai gradi di striscio 2+ e 3+ per il set di campioni con volume di input da 500 μl. I campioni estratti da 500 μL di sedimento di espettorato avevano una profondità media di copertura del sequenziamento di 4.316 rispetto a 4.810 per i campioni estratti da 2 mL di sedimento di espettorato. Ciò suggerisce che il DNA estratto dal sedimento dell'espettorato era adeguato per eseguire il tNGS a valle, indipendentemente dal volume di input.
Sulla base della distribuzione dell'accettabilità dei risultati del sequenziamento, i campioni con un grado di striscio di 3+ hanno avuto il più alto tasso di successo con la maggior parte delle sequenze di campioni valutate come ++ e +++ rispetto ai campioni di grado 1+ e 2+. Per i campioni di input da 500 μL, la percentuale di campioni nelle categorie - e + è relativamente più alta in tutti i gradi di striscio rispetto ai campioni di input da 2 mL. Ciò suggerisce che un maggior numero di campioni rientra nelle categorie di qualità inferiore, meno accettabili, per l'input da 500 μl. Per i campioni in ingresso da 2 mL, c'è una percentuale maggiore di campioni nelle categorie di qualità superiore, ++ e +++, in particolare per il grado di striscio 3+. Ciò suggerisce che i campioni con input di 2 mL hanno maggiori probabilità di produrre punteggi di qualità del sequenziamento migliori rispetto a quelli con input di 500 μL.
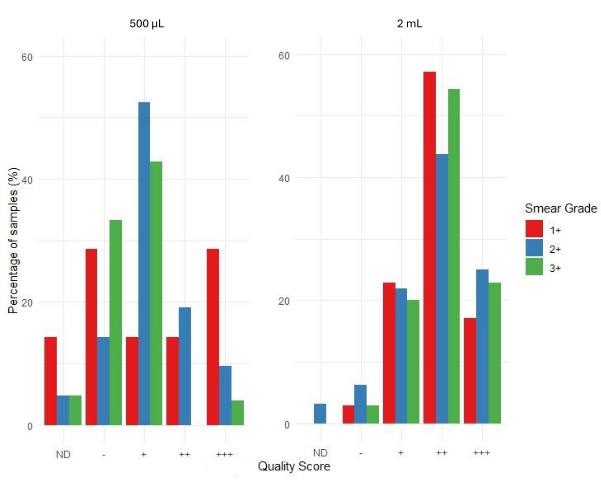
Figura 6: Distribuzione dei punteggi di qualità per grado di striscio e volumi di input del campione. Fare clic qui per visualizzare una versione più grande di questa figura.
Discussione
La combinazione del metodo di estrazione basato su matrice accoppiato con un omogeneizzatore ad alta velocità è stata originariamente pubblicata da Shea et al.9. Questo metodo è stato sviluppato e ottimizzato per la WGS del DNA estratto da colture MTBC 9,10. Abbiamo ottimizzato questo metodo per l'uso con un test MTB tNGS. L'estrazione e la purificazione del DNA di 21 campioni hanno avuto un tempo di risposta combinato di 4 ore e 40 minuti, comprese le fasi di incubazione e centrifugazione. L'affidabilità del protocollo descritto, unita a una pulizia delle microsfere per l'estrazione del DNA da campioni con gradi di striscio variabili da 1+ a 3+, è dimostrata in base alla resa media (Figura 4), alla profondità complessiva di copertura di tutti i target combinati (Figura 5) e al punteggio di accettabilità dei risultati del sequenziamento determinato dall'applicazione web associata (Figura 6).
Uno studio recente non ha trovato alcuna relazione coerente tra il grado dello striscio e la concentrazione del DNA o la profondità di lettura del sequenziamento. Gli autori hanno suggerito che la variabilità nell'elaborazione dei campioni e nelle fasi di pulizia può influire sulle prestazioni nei campioni ad alto grado di striscio, in particolare quando i contaminanti non vengono rimossi in modo efficiente. Inoltre, il sequenziamento dal sedimento dell'espettorato rimane impegnativo a causa della complessità del pool di DNA, dove il DNA non bersaglio può competere con MTB DNA11. Sebbene non sia stata osservata alcuna chiara correlazione tra la concentrazione di DNA in ingresso e la profondità di sequenziamento, è importante notare che l'input di DNA prima della PCR target non deve superare i 100 ng, secondo le istruzioni del produttore, in quanto ciò potrebbe portare all'inibizione della PCR. Inoltre, una carica batterica più elevata, i campioni di sedimenti di espettorato 2+ e 3+ potrebbero contenere più contaminanti, come DNA umano, detriti cellulari e altri inibitori, che possono interferire con l'amplificazione della PCR e la preparazione della libreria.
La fase di purificazione funziona anche come fase di concentrazione, migliorando la resa del DNA micobatterico prima del tNGS. Questo metodo è efficace per l'elaborazione di campioni con una bassa carica batterica e ha dimostrato prestazioni affidabili con volumi fino a 500 μl, rendendolo adatto per ambienti di routine in cui è disponibile un volume di campione limitato. La sua applicazione può ridurre la necessità di visite di follow-up presso le cliniche, riducendo così al minimo il rischio di perdita del paziente al follow-up. Inoltre, il metodo è semplice e non richiede competenze di laboratorio avanzate, supportando ulteriormente la sua integrazione in contesti con risorse limitate.
Tuttavia, è necessario notare alcuni punti relativi al protocollo. I campioni non devono essere congelati o refrigerati immediatamente dopo l'incubazione a 80 °C. Il rapido raffreddamento subito dopo il trattamento termico può causare la formazione di condensa sulle superfici interne della provetta, portando alla diluizione del campione e potenzialmente influenzando l'estrazione del DNA a valle. Inoltre, il raffreddamento improvviso può aumentare il rischio di degradazione del campione promuovendo la frammentazione dell'acido nucleico o l'attività enzimatica che potrebbe non essere stata completamente inattivata durante il trattamento termico. Queste considerazioni sono particolarmente rilevanti quando si elaborano campioni di espettorato per il rilevamento MTB. Secondo le raccomandazioni dell'OMS per la colorazione Ziehl-Neelsen, un grado di striscio di 1+ corrisponde a 10-99 AFB per 100 campi di immersione in olio, 2+ indica 1-10 AFB per campo in almeno 50 campi e 3+ rappresenta più di 10 AFB per campo in almeno 20 campi12.
Un limite di questo metodo è la sua mancanza di automazione a causa della necessità di omogeneizzazione manuale utilizzando un omogeneizzatore ad alta velocità o un dispositivo di battitura delle microlinee. Per risolvere questo problema, gli sforzi in corso si concentrano sul perfezionamento del protocollo testando tempi di incubazione più brevi, temperature di incubazione più basse e approcci alternativi all'omogeneizzazione ad alta velocità.
Pertanto, questo metodo di estrazione del DNA basato su matrice, combinato con un omogeneizzatore ad alta velocità, è una tecnica di estrazione rapida del DNA che impiega il calore e il bead beating per rilasciare il DNA genomico rimuovendo contemporaneamente gli inibitori della PCR, come metalli e proteine.
Divulgazioni
L'autore T. R. riceve il sostegno finanziario da FIND attraverso un contratto di servizio con l'UC San Diego. L'autore T. R. ha ricevuto finanziamenti dal NIH per sviluppare e valutare una soluzione tNGS per la tubercolosi resistente ai farmaci (R01AI176401). L'autore T. R. è co-fondatore, membro del consiglio di amministrazione e azionista non retribuito di Verus Diagnostics Inc.
Riconoscimenti
Gli autori desiderano ringraziare il team del laboratorio TB presso l'NHLS - Green Point, Sud Africa, per aver fornito assistenza tecnica. Questo lavoro è stato sostenuto dal National Institutes of Health (NIH) e dalla Foundation for Innovative New Diagnostics (FIND), dal progetto TS ELiOT Grant ID: R01AI153213, Unitaid Grant ID: 2019-32-FIND MDR. Le figure sono state create utilizzando BioRender.com.
Materiali
| Name | Company | Catalog Number | Comments |
| Alternative: Benchmark BeadBlaster 24 | Benchmark Scientific | D2400 | |
| AMPure XP Bead-Based Reagent | Beckman Coulter | A63881 | |
| Autoclaved or sterile glass beads (2 mm) | Sigma-Aldrich | Z273627-1EA | |
| Ethyl alcohol, Pure | Sigma-Aldrich | E7023-500ML | |
| Filter pipette tips (10 µL) | Bio-Smart Scientific | FT-10-R | |
| Filter pipette tips (1000 µL) | Bio-Smart Scientific | FT1000-R | |
| Filter pipette tips (20 µl) | Bio-Smart Scientific | FT20-R | |
| Filter pipette tips (200 µL) | Bio-Smart Scientific | FT200-R | |
| Heating-block ranging from 37 °C to 100 °C | Eppendorf | 5382000031 | |
| High-speed homogenizer (HSM) as FastPrep-24 | MP Biomedicals | 116005500 | |
| Incubator 80 °C | Lasec Group | IBLPDHG-9030A | |
| InstaGene Matrix | Bio-Rad Laboratories Inc. | BBRD7326030 | |
| Low-binding microtubes (1.5 mL or 2 mL) | Eppendorf | EP0030108051-250EA | |
| Magnetic rack for 1.5 – 2 mL tubes | Thermo Fisher Scientific Inc. | 12321D | |
| Magnetic stirrer | Merck | Z671886 | |
| Microcentrifuge | Eppendorf | 5406000046 | |
| Milli-Q IX 7015 Pure Water Purification System | Merck | ZIX7015T0C | |
| Nuclease-free water (NFW), molecular grade | Thermo Fisher Scientific Inc. | AM9937 | |
| Pipettes (P10, P20, P200, and P1000) | Eppendorf | EP3123000918-1EA | |
| Qubit Assay Tubes | Thermo Fisher Scientific Inc. | Q32856 | |
| Qubit dsDNA HS Assay Kit | Thermo Fisher Scientific Inc. | Q33231 | |
| Qubit fluorometer instrument | Thermo Fisher Scientific Inc. | Q33226 | |
| Screw cap tubes (1.5 mL) | Scientific Specialties Inc. | P2TUB056C-0001.5ST | |
| Vortex | Lasec Group | WMBB0L0E0216 |
Riferimenti
- Global Tuberculosis Report 2024. Geneva: WHO. , World Health Organization. https://www.who.int/teams/global-tuberculosis-programme/tb-reports/global-tuberculosis-report-2024 (2024).
- De Araujo, L., et al. Implementation of targeted next-generation sequencing for the diagnosis of drug-resistant tuberculosis in low-resource settings: a programmatic model, challenges, and initial outcomes. Front Public Health. 11, 1204064(2023).
- Murphy, S. G., et al. Direct detection of drug-resistant Mycobacterium tuberculosis using targeted next generation sequencing. Front Public Health. 11, 1206056(2023).
- Kambli, P., et al. Targeted next generation sequencing directly from sputum for comprehensive genetic information on drug resistant Mycobacterium tuberculosis. Tuberculosis. 127, 102051(2021).
- WHO Consolidated Guidelines on Tuberculosis.Module 3: Diagnosis. Rapid diagnostics for tuberculosis detection. Third edition. , World Health Organization. https://iris.who.int/bitstream/handle/10665/376221/9789240089488-eng.pdf?sequence=1 (2024).
- Catalogue of mutations in Mycobacterium tuberculosis. complex and their association with drug resistance. Second edition. , World Health Organization. https://iris.who.int/bitstream/handle/10665/374061/9789240082410-eng.pdf?sequence=1 (2023).
- Schwab, T. C., et al. Targeted next-generation sequencing to diagnose drug-resistant tuberculosis: a systematic review and meta-analysis. Lancet Infect Dis. 24 (10), 1162-1176 (2024).
- Deeplex Myc-TB manual version 5. , GenoScreen. https://acrobat.adobe.com/link/track?uri=urn%3Aaaid%3Ascds%3AUS%3A67c2b53f-c91d-3a73-a6ea-4769e7c8fe57 (2023).
- Shea, J., et al. Comprehensive whole-genome sequencing and reporting of drug resistance profiles on clinical cases of Mycobacterium tuberculosis in New York State. J Clin Microbiol. 55 (6), 1871-1882 (2017).
- Conceição, E. C., et al. Mycobacterium tuberculosis. DNA extraction using InstaGene Matrix and high-speed homogenizer from clinical primary culture. , https://www.protocols.io/view/mycobacterium-tuberculosis-dna-extraction-using-in-dhvs366e (2024).
- Nilgiriwala, K., et al. Genomic sequencing from sputum for tuberculosis disease diagnosis, lineage determination, and drug susceptibility prediction. J Clin Microbiol. 61 (3), e0157822(2023).
- Laboratory Services in Tuberculosis Control Part II: Microscopy. Geneva: WHO. , World Health Organization. https://iris.who.int/bitstream/handle/10665/65942/WHO_TB_98.258_(part2).pdf?sequence=2 (1998).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneEsplora altri articoli
This article has been published
Video Coming Soon