Method Article
酵母表面表示タンパク質の酵素修飾およびフローサイトメトリー評価
要約
ここでは、酵母表面表示基質を酵素修飾アッセイに使用するためのプロトコールを紹介します。このプラットフォームは、代表的な酵素修飾アッセイとして、チロシンホスファターゼSHP-2の基質の1つに対する脱リン酸化活性の分析を使用して実証されました。
要約
酵母表面ディスプレイは、タンパク質機能のハイスループットスクリーニングを可能にする遺伝子型-表現型連鎖戦略です。従来、酵母表面ディスプレイは、新しい結合タンパク質の進化に適用され、フローサイトメトリーを使用して結合強度のレベルを評価し、分類してきました。最近では、基質変異体の酵素修飾をスクリーニングするために酵母表面ディスプレイを適用することへの関心が高まっています。これには、フローサイトメトリーで読み取り可能な表現型を提供する相加的(リン酸化など)または減法的(タンパク質分解など)修飾があります。このような修飾は、細胞内共局在を用いて定期的に適用されますが、表示された基質の細胞外酵素修飾を達成する能力は、さらに多くの反応を研究に引き出す可能性があります。ここでは、酵母表面に表示された候補基質に対する細胞外酵素修飾のスクリーニングアッセイを設計および適用し、その後フローサイトメトリー分析を使用して評価するための技術について説明します。これらのプロトコルは、ホスファターゼ、脱リン酸化酵母、リン酸化チロシン残基を含むディスプレイ基質のコンテキストで提供し、この適用されたフレームワークを他の酵素-基質ペアのスクリーニングアッセイの開発にどのように適応できるかについてコメントします。
概要
酵素とその標的との間の相互作用の理解は、細胞の恒常性と疾患発症を制御する経路の生物学的特性評価に必要であるため、ますます興味深い研究分野になっています1,2。酵素は、生物学的生命を維持する多くの反応の触媒作用に関与しており、細胞代謝3,4、シグナル伝達5、さらにはゲノム修復6,7などの基本的なプロセスを制御する。これらのプロセスにおけるそれらの役割のために、それらの相互作用はまた、それらの活性の逸脱が細胞活動の深刻な調節不全を引き起こし、アポトーシスまたは有害な癌細胞の増殖を引き起こす可能性があるため、多くの疾患の発症にも役割を果たします2。酵素活性の研究は、新しい治療法8,9の開発において重要な応用があり、各特定の酵素-基質相互作用10に合わせたアッセイが必要とされている。これらの相互作用の評価と特性評価のための標準プロトコルとして、複数の酵素アッセイが確立されています。酵素的相互作用を解析するために開発されたアッセイは、活性化/阻害のための結合をモニターする検出アッセイ11または酵素による基質修飾をモニターするアッセイ12に分類される。
酵素の主要な役割の1つは、細胞の振る舞いを調節することです。シグナル伝達、すなわち細胞外トリガー13に対する細胞の細胞内応答は、細胞の生存と機能に関与している。細胞の増殖、分化、その他多くの機能プロセスはすべて、それらを支配する酵素的相互作用を伴うシグナル伝達経路に関与しています14,15。酵素は翻訳後修飾を触媒し、細胞外メッセージの正しい伝達に関与する大規模なシグナル伝達ネットワークをしばしば調節する16。タンパク質のリン酸化は、最も一般的な翻訳後修飾であり、細胞シグナル伝達や他の複数の細胞経路に遍在しています。その結果、プロテインキナーゼは、その重要な調節的役割により、潜在的な治療標的のかなりの割合として浮上しています17。ホスファターゼは、リン酸ベースの細胞シグナル伝達複合体18,19の天然調節分子であり、標的タンパク質20からリン酸残基を除去する能力を有する。過去10年間で、ホスファターゼは、複数の細胞型における下流のシグナル伝達経路の調節に関与していることから、がん治療21および炎症性疾患22の主要な治療標的となった。プロテインキナーゼとホスファターゼは、共に幅広い相互作用を提供し、特定の酵素アッセイプロトコルの開発を通じて研究することができます。
酵母表面ディスプレイは、酵素活性23,24の特性評価および評価のためのツールとして使用されてきた。これは、小胞体隔離戦略25,26と組み合わせた場合の翻訳後修飾プロセスのスクリーニングのためのハイスループットプラットフォームを提供する。これにより、キナーゼ-基質ペアは、KDEL受容体27への結合を通じて小胞体内で共局在し、保持することができ、キナーゼとその標的との間の近接性により、基質のリン酸化が増加する速度で起こり得る。KDEL受容体の結合は、C末端FEHDEL小胞体保持配列によって媒介され、他のHDEL配列25,28よりも強い保持能力を有することが示されている。次いで、リン酸化基質を酵母表面に固定し、その後のフローサイトメトリー29による評価を行う。現在、酵母表面上に示される基質の酵素修飾について確立された一般化可能なプロトコルはありません。私たちは、細胞外に発現するリン酸化基質変異体を利用し、既知のホスファターゼによる脱リン酸化によってそれらを修飾することにより、酵母表面ディスプレイの能力を拡大します。フローサイトメトリー解析は、既知のホスファターゼとのインキュベーションの結果としてのリン酸化中央値の変化を測定することにより、前述の基質の表現型評価のためのプラットフォームを提供します。これにより、表面ディスプレイタンパク質の翻訳後修飾に適応可能な方法を提供すると同時に、酵母表面ディスプレイプラットフォームを使用する際の相互作用の酵素修飾分析の方法も提供されます。
酵母表面ディスプレイプラットフォームへのキナーゼ-基質相互作用の導入、発現したリン酸化基質と組換えホスファターゼとの共インキュベーション、およびその後のフローサイトメトリーによる脱リン酸化活性の解析を説明する酵素修飾アッセイの開発と応用のための技術を紹介します。本報告では、酵母小胞体においてCD28の細胞質ドメインをリンパ球キナーゼ(LCK)と共局在化し、続いて酵母表面にリン酸化CD28を表示し、その後、Srcホモロジー領域2ドメイン含有ホスファターゼ-2(SHP-2)による脱リン酸化を行うことで、これを達成した。さまざまなペプチド配列中のリン酸化チロシン残基を検出するPAN抗ホスホチロシン抗体(本研究では4G10)を用いて、ホスファターゼ処理の関数としてのリン酸化レベルの定量化を行います。詳細なプロセスは、酵素と基質の相互作用を調査するための一般化可能なアプローチを提供します。精製された方法で酵素と基質を研究する将来の方法。
プロトコル
1. プラスミドを保有する酵母の細胞増殖とタンパク質発現誘導
- 表1に記載されているレシピに従って、非プラスミド含有酵母(YPD)、プラスミド含有酵母細胞増殖(SD-CAA)、タンパク質発現誘導(SRG-CAA)、およびSD-CAAプレートの増殖に必要な培地を調製します。
- キナーゼ-基質ペアを含む酵母ディスプレイプラスミドDNAを、リチウムカチオンベースの方法30,31を通じてEBY-100酵母細胞に形質転換します。これは、さまざまなメーカーの酵母形質転換キットで一般的に採用されています。
注:エレクトロポレーション32 または他の好ましい酵母プラスミド形質転換技術は、形質転換されるプラスミドコンストラクトに応じて使用することができる。 - 10 mLのYPD培地を含む14 mLの培養チューブを調製します。EBY-100細胞を接種し、30°C、300rpmの振とうインキュベーターで培養物が0.8-1.0(8 x 106- 1 x 107細胞/mL)の光学密度(OD600nm)に達するまで増殖させます。
注:OD600nm は、それぞれの培地に酵母培養物の1:10希釈液を含む3mLサンプルキュベットと、サンプル希釈に使用する培地を含む3mLブランクキュベットを調製することによって測定されます。分光光度計のOD600nm プログラムは、最初にブランクキュベットを測定し、次に各サンプルに対応する希釈を設定して各サンプルキュベットを測定するために使用されます。1 OD600nm は1 ×10 7 酵母/ mLに相当します。 - 培養物を1,000 x g で3分間遠心分離して細胞を回収し、酵母形質転換キットに付属の1回目の 洗浄液、またはTE(10 mM Tris-HClおよび1.0 mM EDTA)31で洗浄します。
- 細胞を再び1,000 x g で3分間ペレット化し、酵母形質転換キットに付属の形質転換バッファー1 mLまたは滅菌水に再懸濁します。細胞は50μLのアリコートに分注し、-80°Cで最大6ヶ月間保存することができます。
- プラスミド形質転換では、プラスミド1個につき1アリコートを調製し、氷上で解凍した後、酵母ディスプレイコンストラクトを含む0.5〜1.5μgのプラスミドDNAを細胞に直接添加します。酵母形質転換キットで提供される0.5mLの形質転換溶液、または滅菌50%ポリエチレングリコールおよび0.1MLiOAc溶液31の0.5mLを添加する。細胞、プラスミドDNA、形質転換溶液の混合物をピペッティングで完全に混合します。
- 形質転換混合物を30°Cで30〜60分間静的にインキュベートし、ボルテックス混合物を15分間隔でインキュベートします。1,000 g× gで3分間遠心分離して細胞を回収します。
- 4.5 mLのSD-CAA培地を含む14 mLの培養チューブを調製します。目的のプラスミドを含む細胞を500 μLのSD-CAAに再懸濁し、調製した4.5 mLを接種します。
- 寒天に穴を開けないように注意しながら、接種した培養液50 μLを5 mLをSD-CAAプレートに分配し、30°Cで48時間静的にインキュベートして、形質転換効率を決定します。
- 5 mLのSD-CAA細胞培養液を振盪インキュベーターで30°C、300 rpmで少なくとも18時間インキュベートします。16時間後および20時間後の光学密度(OD600nm)を監視します。サンプルが6を超えない十分な光学密度まで成長したら、培養物を2,500 × gで3分間遠心分離します。酵母ペレットを乱さずに上清を捨てます。
- SRG-CAA中の酵母ペレットを最終外径600nm (<1 ×10 7 酵母/mL)未満に再懸濁します。
- 酵母培養物を30°C、300rpmの振盪インキュベーターで少なくとも8時間、24時間以内でインキュベートします。
注:酵母細胞におけるタンパク質発現の誘導は、20-37°Cの範囲で変化させることができる。30°Cはキナーゼ/基質対29,33の合成に適しているが、研究対象の特定のタンパク質に必要と思われる場合は調整することができる。 - 外径600nm を測定して、細胞密度を決定します。
注:この時点で、酵母培養物を4°Cで保存することにより、プロトコールを停止することができます。
2. 4G10抗ホスホチロシン抗体のビオチン化
- PEG4-NHS-ビオチンの2 mgバイアルを、滅菌PBS680 μLを加えて最終濃度5 mMに再懸濁します。
注:PEG4-NHS-ビオチンの再懸濁は、ビオチン化反応が行われる直前に新たに行う必要があります。NHSは水溶液中で加水分解します。滅菌PBSおよびバイアルの使用は、長期保存用の試薬の調製や、試薬の生存率や実施される高感度アッセイに対する汚染物質の潜在的な影響を軽減するための細胞ベースのアッセイでの使用に重要です。 - 4G10抗体濃度に基づいて、100 μgの抗体を滅菌済みの1.7 mLバイアルに加えます。
- ステップ2.1で調製した5 mM PEG4-NHS-ビオチン100 μgを保持したバイアルに1 μLを添加し、ビオチンと抗体のモル比を7.5:1にします。混合物を穏やかにピペットで動かして、反応を均質化します。
- 反応を室温で少なくとも2時間一定回転させてインキュベートします。
- 0.5 mL スピン脱塩カラムの製造元のプロトコルに従って、ビオチン化 4G10(B-4G10)のバッファーを PBS に交換します。
注:分子量カットオフ(MWCO)が7 kDaの脱塩カラムは、抗体のビオチン化に一般的に使用され、未反応のビオチンやその他の低分子を除去しながら、より大きな抗体を保持することができます。 - B-4G10抗体をPBSA(PBSと1 g/Lウシ血清アルブミン)で最終濃度1 μMに希釈します。B-4G10抗体を少量に分注して、凍結融解サイクルの繰り返しを防ぎます。
注:ビオチン化抗体は、4°Cで保存し、最大3ヶ月間毎日使用できますが、効率を大幅に損なうことはありません。使用しない残りのアリコートは、-20°Cで最大2年間保管してください。
3. 酵母細胞表面に発現する基質の脱リン酸化
- 文献34に前述したように、2倍作業緩衝液を調製する。
注:必要な成分の測定を容易にするために、2倍をお勧めします。 - ステップ 3.1 1:2 で調製した 2x バッファー溶液を脱イオン水で希釈することにより、1.7 mL バイアル中のサンプル用のワーキングバッファーを調製します。
注:各サンプルの総反応量は20μLで、各サンプルには10μLから18μLの作業バッファーが必要です。すべてのサンプルまたはコントロールに十分な作業バッファーを準備します。 - 表2に記載されている推奨サンプル調製に従って、1.7 mLバイアルに対応するコントロール名またはサンプル名をラベル付けします。
- ステップ1.9で測定したOD600nm に基づいて、各サンプルの対応する酵母培養物から200万個(2 × 106)酵母細胞を回収するために必要な培養量を計算します。
- 前のステップで計算した酵母培養液の量を、各サンプルの1.7 mLバイアルに加えます。
- バイアルを4,500 × gで1分間遠心分離します。マイクロピペットで上澄みを丁寧に取り除き、バイオハザード廃棄物として捨ててください。
- ペレット化した細胞を1 mLのPBSAに再懸濁し、 ステップ3.6を繰り返します。
注:ペレット化された細胞を乱さずに、できるだけ多くの上清を除去することが重要です。 - そのストック濃度に基づいて、20 μLの総反応容量で最終濃度が1,000 nMになるために必要な組換えヒトSHP-2の容量を計算します。
注:組換えSHP-2は、各アッセイで使用されるタンパク質が2回以上の凍結融解サイクルを経ないように、少量で分注する必要があります。アッセイのためにすべてのサンプルが準備された後にアリコートから残ったSHP-2は廃棄する必要があります。
組換え酵素のストック濃度は、ロット番号によって異なります。組換えSHP-2は、通常、0.2〜0.4 mg / mLのストック濃度で調製されます。SHP-2のストック濃度が0.324 mg/mLの場合、これは4.696 μMのストック濃度に相当します(SHP-2の分子量は69 kDa)。20 μLの反応でSHP-2ストックを4.26 μLすると、最終反応濃度は1,000 nM SHP-2になります。 - 15 mLの円錐形に調製した10 mLの脱イオン水に7.7 mgのDTTを加えて、5 mM DTT溶液を調製します。装置の制限によりミリグラムの秤量が困難な場合は、10 mLの脱イオン水に0.77 gのDTTを加え、100倍希釈してアッセイに使用する5 mM DTT溶液を作成します。
注:DTT溶液は、利用可能な装置を使用してミリグラムスケールの量を測定することができない場合に、5 mMに希釈するために高濃度のストック溶液で調製することができます。DTT溶液は、加水分解の傾向があり、水で希釈すると長期間にわたって不安定になるため、ワーキングバッファーで細胞の再懸濁に先立って新鮮に調製する必要があります。 - ステップ3.2で調製したワーキングバッファーにペレット化した細胞を再懸濁し、各サンプルまたはコントロールの最終反応量が20 μLになるようにします。
注:添加するワーキングバッファーの量は、各サンプルに含まれるDTT(2 μL)およびSHP-2(ステップ3.8で計算)の量に基づいて計算する必要があります。 - ステップ3.9で調製した5 mM DTT溶液2 μLを各サンプルまたはコントロールに添加し、最終反応濃度を0.5 mM DTTにします。
- ステップ3.8で計算したSHP-2の容量を各サンプルに加えて、最終容量を20μLにし、マイクロピペットを使用して穏やかに混合します。
- サンプルバイアルの蓋をパラフィルムで包み、漏れや相互汚染を防ぎます。
- サンプルを37°Cで2時間、ローター上で一定速度でインキュベートします。
- ローターからサンプルを取り出し、各サンプルに1 mLのPBSAを加えて反応を停止します。
- 手順3.6を繰り返します。
4. 脱リン酸化基質の細胞標識とフローサイトメトリー解析
- ステップ 3.16 のサンプルを 、表 2 に記載されているように、対応する一次試薬の 20 μL 混合物に再懸濁します。サンプルを室温で20分間インキュベートします。
注:使用されるすべての試薬濃度は、酵母細胞の表面に発現するタンパク質の数に対して過剰であると計算されています。この計算は、サンプル35中のすべての2×106酵母から10,000タンパク質/細胞が発現すると仮定しているが、通常はそのうちの~50%しか発現していない。表3は、表2で表される各試薬の過剰標識試薬比率を次のように計算したものである。

- 4,500 × g でサンプルを1分間遠心分離し、上清をバイオハザード廃棄物として廃棄します。
- 1mLのPBSAに再懸濁して細胞を一度洗浄します。手順4.2を繰り返します。
- 表2に記載されているように、対応する二次試薬の混合物である20 μLにサンプルを再懸濁します。光が当たらない状態でサンプルを15分間インキュベートします。
- 手順4.2を繰り返します。
- 手順4.3を繰り返します。
- 洗浄したサンプルを300〜500 μLのPBSAに再懸濁し、5 mLのポリスチレンチューブに移して、適切なフローサイトメーターを使用してすぐに分析します。
注:サンプルを輸送する必要がある場合は、濡れた氷の上に置いてください。推奨されませんが、サンプルは湿ったペレットとして4°Cで最大2時間保存できます。 - 新しい実験に必要なサイトメーターの起動と準備を行った後、File(ファイル)メニュー内のNew Experiment(新規実験)ボタンをクリックし、実験に名前を付けてSave(保存)をクリックし、取得したデータが目的のファイルパスに保存されていることを確認します。
- 上部ツールバー内のドットプロットアイコンを選択して、実行するサンプルごとに2つ以上のドットプロットを作成します。ドットプロットの 1 つで、X 軸名を選択して FSC-A チャネルを表示し、Y 軸名を選択して SSC-A チャネルを表示します。このプロットは、側散乱光 - 面積と前方散乱 - 面積を示しており、さらなる分析のために酵母細胞をゲートするために使用されます。
- 別のドットプロットで、X軸の名前を選択して、一次抗エピトープタグ抗体を標的とする二次試薬が蛍光を発するチャネルを表示します。Y軸名を選択すると、ストレプトアビジン二次試薬が蛍光を発するチャンネルが表示されます。このプロットは、酵母細胞として横方散布図と前方散布図プロットからゲートされたイベントのみを表示し、Y軸にチロシンリン酸化、X軸に基質表面発現を表示するために使用されます。
注:この例では、一次抗エピトープタグ抗体を標的とする二次試薬はFITC(AF-488)チャネルで蛍光を発し、ストレプトアビジン二次試薬はAF-647チャネルで蛍光を発します。使用されるチャネルは、標識中に使用される一次試薬および二次試薬によって異なる場合があります。 - 各サンプルチューブをサイトメーターのチューブホルダーにセットし、サイトメーターの[Run]を選択して、サンプルのロードとデータの取得を開始します。必要に応じて、表示するイベント、記録するイベント、記録する時間、およびサンプル流量を調整します。
- ステップ4.9で作成したSSC-A対FSC-Aのプロットで健康な酵母細胞を囲むゲートを定義します。 図 1 は、適用するゲーティング戦略の説明的表現を示しています。
注:SSC-A対FSC-Aのプロットでは、定義された酵母ゲート内で100,000のイベントを表示し、50,000のイベントを記録することが、さらなる分析のために十分なデータを視覚化して収集するための適切なガイドラインです。一重項酵母に対して選択するためのより厳密なゲーティング戦略は、最近報告された36のように、前方散乱高さと前方散乱面積のプロットに基づいて適用することができる。 図1 に示すゲーティング戦略は、スキャッターゲーティングによる一重項および一部のダブレット酵母を含む、それほど厳密でないアプローチに対応しています。 - フローサイトメトリーアナライザーを使用して、すべてのコントロールサンプルの蛍光を記録します。コントロールサンプルは、以下のステップ4.14で説明するように、ゲーティング戦略を定義するために最初に定期的に収集されます。
- 処理したサンプルを分析する前に、ステップ4.10で作成したプロットのゲーティング戦略を定義します。 図 1 は、適用するゲーティング戦略の説明的表現を示しています。
- フローサイトメーターとステップ4.12および4.14で定義されたゲーティング戦略を使用して、脱リン酸化サンプルの蛍光を記録します。
- フローサイトメトリー解析ソフトウェアを使用して取得したフローサイトメトリーデータを解析します。
- 表面でタンパク質を発現する細胞のY軸中央値と、サンプルとコントロール間で非表示細胞によって提供されるベースラインリン酸化を測定および比較することにより、脱リン酸化を評価します。リン酸化差の中央値の割合を次のように計算します。
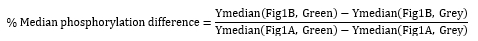
結果
1,000 nM SHP-2 なし(図 1A)および(図 1B)の 1,000 nM SHP-2 を 2 時間インキュベートしたモデルシステムの個々の複製からのフローサイトメトリー解析では、リン酸化差の中央値は 63.6% であり、これは、表面に表示されたすべてのイベントからの Y 軸中央値(リン酸化)から、処理されたサンプルと未処理のコントロールとの間の非表示イベントの Y 軸中央値として定義されるベースラインリン酸化シグナルを差し引いた比として定義されますプロトコルのステップ 4.12 で定義されています。中央値を分析する前に、サンプルはサイズ(前方散乱)と複雑さ(側散乱)に基づいてゲート化され、健康な細胞グループを包含していました。定義された条件下では、サンプルの脱リン酸化は一目瞭然で区別できるはずです(図1C)。
このアッセイは、4つの主要な方法からなる簡単な手順で定義されます(図2A)。我々が使用し、以前に発表した酵母表面ディスプレイシステムは、CD28の細胞質尾部とチロシンキナーゼLCK(図2B)29からなるキナーゼ-基質対の同時誘導性発現能力を持つ双方向プロモーターを含むプラスミドに基づいている。翻訳されたタンパク質の共局在は、シグナルペプチドを標的とする小胞体と、C末端ER保持配列に起因する滞留時間の増加によって強制される翻訳後修飾によって指示されます。Aga2pに融合したリン酸化基質の分泌は、表面発現につながります(図2C)。この基質は、2つのエピトープタグに挟まれるように設計されているため、翻訳の成功とその後の表面発現を細胞外に確認することができます。目的のホスファターゼ(この場合はチロシンホスファターゼSHP-2)と酵母細胞を表示する基質をインキュベートすると、基質結合リン酸の減少による酵素修飾の分析が可能になります(図2D)。
提示されたモデルシステムに最適な条件が定義されていますが、アッセイの一般化可能性により、分析するタンパク質の多様化が可能になります。アッセイの最適条件は、ホスファターゼの時間と濃度のさまざまな組み合わせを4重で評価する一連の滴定によって定義されました(図3)。解析したデータは、複製を伴う二元配置分散分析(p < 0.05)を通じて統計的有意性を示した。選択した条件である 2 時間および 1,000 nM (48.8% ± 10%) は、不等分散の t 検定分析に基づく 2 時間 (p < 0.05) での 750 nM の対応物と比較して、統計的有意性を維持しながら、約 50% の中央リン酸化差を提供しました。 また、t検定では、リン酸化差の割合の中央値がおおよそ(3時間で500 nM、750 nM、および1,000 nMでp > 0.05)得られた濃度のいずれかの濃度で時間を1時間増加させても、2時間および1,000 nMの結果と有意差は示されませんでした。
Tukey HSDの事後解析では、すべての濃度にわたるインキュベーション期間間のすべての平均比較が、1時間から2時間を除いて有意に異なることが明らかになりました。試験した複数の濃度を比較すると、250 nMを他のすべての濃度と比較すると、統計的に有意な平均差のみが観察され、250 nMを除くグループ内では同等のレベルのホスファターゼ活性が期待されることを示しています。サンプルを4時間および1,000 nM SHP-2で処理した場合(22.1%±5.5%)で20%の差が観察されたにもかかわらず、最適条件(t検定、p < 0.05)と比較して、DTTとの長期インキュベーションによる表面発現の低下と酵母の健康状態の低下により、この組み合わせを追求しないことを決定しました。これは、SHP-2ホスファターゼの適切な機能に必要な作業緩衝液の還元条件によって引き起こされると仮定しています。

図1:モデルシステムのフローサイトメトリー解析。 前方散乱光(X軸)と側方散乱光ゲーティング(Y軸)(左)を示す密度プロット、およびSHP-2なしで2時間インキュベートしたCD28細胞質ドメインの基質C末端エピトープタグ標識(X軸)と基質リン酸化(Y軸)(中央)(中央)による表面発現を示すドットプロット、およびSHP-2の最適濃度を1,000 nMと定義した(A)および(B)で2時間インキュベートしたCD28細胞質ドメイン。Y-Median は、相対的リン酸化測定としてのみ、表面発現細胞 (緑) を含む定義されたゲート内で測定されました。非表示の信号イベントは、背景の Y 中央値測定 (灰色) を定義するためにゲート化されました。(C)Y中央値の明白な視力の違いを示すサンプルのドットプロットオーバーレイ。 この図の拡大版を表示するには、ここをクリックしてください。
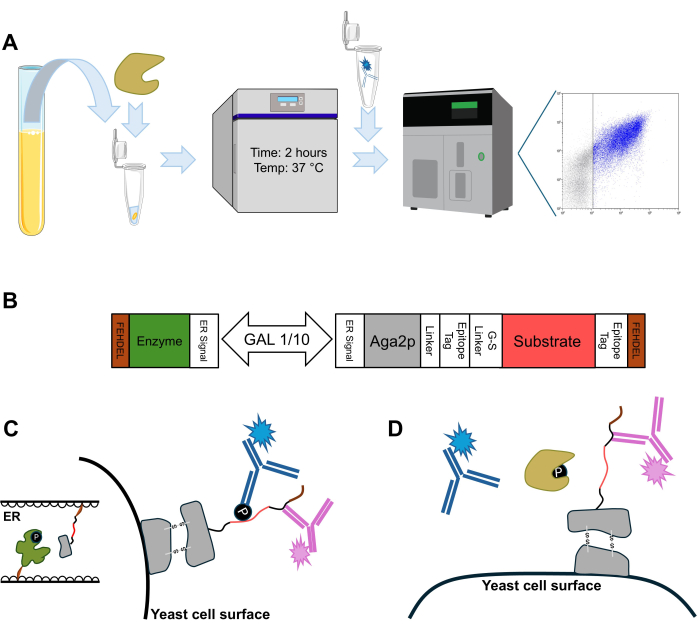
図2:酵母表面表示タンパク質の酵素修飾 (A)記載されている4つの方法における重要なステップを示すアッセイ概略図:酵素アッセイのためのホスファターゼ(tan)によるサンプルの調製、所望の酵素活性のためのインキュベーション、活性検出のための細胞洗浄と標識、フローサイトメトリー分析とデータ収集。(B)酵素-基質対の小胞体隔離および基質の表面発現に使用されるカセットの一般的な構造を表す遺伝子模式図。(C)キナーゼ-基質対の小胞体共局在をグラフ化(左)した後、酵母表面に示された翻訳後修飾基質の分泌を、抗ホスホチロシン抗体(青)と抗エピトープタグ抗体(ピンク)で標識して表面発現を確認(右)。(D)酵母細胞をホスファターゼ(tan)とインキュベートすると、表示された基質からリン酸基が除去され、抗ホスホチロシン抗体の標識が破壊され、フローサイトメトリーによる酵素修飾解析が容易になります。 この図の拡大版を表示するには、ここをクリックしてください。
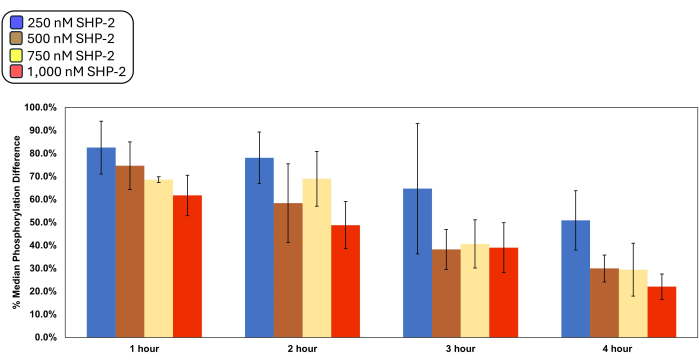
図3:ホスファターゼ活性と時間滴定。 酵母細胞を時間とホスファターゼ濃度の複数の組み合わせに曝露した後、フローサイトメトリー分析を行いました。処理したすべてのサンプルを、SHP-2を使用せずに同じ時間および緩衝条件でインキュベートしたコントロールと比較しました。リン酸化差の中央値の割合は、表面表示イベントからのY中央値の比率から、サンプルを含むSHP-2の非表示イベントにおけるY中央値によって提供されるベースラインノイズ信号を差し引いたものを、それぞれのコントロールで同じ関係で割ったものとして定義されました。帰納仮説は、二元配置分散分析 (p < 0.05) を使用して時間グループと濃度グループ間の変化を比較して有意差が観察された場合、棄却されます。リン酸化差の中央値に対して記述された変数の効果は、互いに独立しています(交互作用p > 0.05)。TukeyのHSD検定は、全体的なインキュベーション時間および濃度グループ間の差の有意性に関するさらなる情報を得るために事後分析のために実行され、不等分散を仮定した一連の t検定を使用して、指定された時間と濃度における個々のグループの統計的有意性を定義しました。データは、4回の反復の平均±標準偏差として表示されます。 この図の拡大版を表示するには、ここをクリックしてください。
表1:酵母増殖およびタンパク質誘導培地調製ガイドライン。 1 Lの選択的酵母増殖培地、選択的酵母タンパク質誘導培地、および酵母抽出物ペプトンデキストロース培地を調製するために各化学成分から必要な質量の表形式の説明。記載されている培地が適切に混合されたら、使用前にフィルター滅菌してください。選択的酵母増殖培地プレートの作成に関する追加の説明書が含まれています。 この表をダウンロードするには、ここをクリックしてください。
表2:基質の脱リン酸化および抗体標識戦略のための推奨サンプル調製。 酵母の表面に表示されるリン酸化基質の酵素修飾の測定に必要なサンプルの表形式の説明。サンプル調製とその後の抗体標識戦略は、必要なコントロールと分析する各サンプル(対応する希釈試薬の希釈を含む)の両方について指定されています。 この表をダウンロードするには、ここをクリックしてください。
表3:抗体とタンパク質の過剰比率の計算。 フローサイトメトリー用に標識されている間に酵母細胞の表面に発現されるタンパク質あたりに利用可能な抗体の理論数を表形式で示したものです。表された理論上の数値は、酵母細胞の100%が抗体の過剰を確保するためにその表面で10,000個のタンパク質を発現すると仮定しており、過剰率の計算は、表に表示され、プロバイダーから取得した試薬ストック濃度に基づいています。 この表をダウンロードするには、ここをクリックしてください。
補足表S1:コンストラクトカセットのアミノ酸配列。 Gal 1-10プロモーターの両側に位置するコンストラクトカセットのアミノ酸配列の表形式表現。強調表示されたシーケンスは、色分けされた説明に対応しています。プラスミドのGal-10側は、その理解を容易にするために逆アミノ酸配列翻訳として表されます。黒色で残されたすべての文字は、コンストラクトにモジュール性を提供するために使用される制限酵素消化部位のアミノ酸翻訳に対応しています。 このファイルをダウンロードするには、ここをクリックしてください。
ディスカッション
提示されたプロトコルは、酵母表面上のタンパク質の細胞外ディスプレイを使用して酵素相互作用の分析を可能にします。使用される表面表示プラスミドに小胞体隔離を組み込むと、細胞内相互作用が発生するように設計できるため、酵素と翻訳後修飾基質との間の特定の相互作用を細胞外で分析する能力が導入されます27,29。酵母表面ディスプレイを用いた以前に確立された酵素相互作用アッセイは、細胞内における目的タンパク質の発現を含み、表面ディスプレイは、標的タンパク質23,25,29間で起こる細胞内相互作用を検出するためのツールのみである。
このプロトコルは、標的となる酵素相互作用を細胞外環境に移動することにより、そのプラットフォーム上に構築されており、研究可能な酵素とその活性が監視される環境の両方に追加の柔軟性が導入されます。細胞外で起こる相互作用の研究は、研究者にインキュベーション環境を酵素活性にとってより最適になるように調整する機会を与え、酸化性が高い環境である酵母小胞体で活性が妨げられる研究可能な酵素を拡大する37。さらに、特定の基質に対する酵素の濃度を滴定する能力により、隔離中の反応速度飽和が仮定されているために細胞内では実施できなかった特定の酵素活性アッセイが可能になります。
プロトコル内では、望ましい結果が観察されることを保証するために注意すべきいくつかの重要なステップがあります。研究したコンストラクトを酵母にうまく形質転換することは、次のステップから最適な結果を得るために不可欠です。光学密度の正確なモニタリングは、培養物の健全な成長を追跡し、タンパク質の誘導やサンプル調製の前に培養物が過剰に増殖しないようにするために行う必要があります。酵母細胞増殖の対数期は、タンパク質産生が最も高くなる期間を含むが、固定期では、タンパク質産生に関与するメカニズムが停止する38。このことを念頭に置いて、光学密度測定は、酵母培養物が属する成長期を正確に測定し、タンパク質の誘導やアッセイの準備などのステップは、固定相の外側または培養物が生い茂ったときに行う必要があります(OD600nm < 6)。
酵素修飾アッセイについて、記載されたインキュベーション環境は、研究対象の酵素であるSHP-2および行われていた酵素活性である脱リン酸化に特異的であった。DTTは、SHP-234とのインキュベーション中に提供する還元環境に使用されました。したがって、酵素アッセイのインキュベーション環境を変更するために使用される化学物質の正確な濃度を測定して、サンプルと実験間で一貫した酵素活性を確保することが重要です。SHP-2は組換えタンパク質として使用され、酵素の取り扱いのさまざまなステップで温度を調節することが重要です。アッセイを成功させるには、酵素が2回以上の凍結融解サイクルを経ていない必要があり、各サンプルの調製中に氷上に存在する必要があります。次に、組換え酵素をアッセイ要件を満たすのに十分な量に分注することが不可欠です。実際のインキュベーションでは、インキュベーション混合物の均質性を確保するために、ローターから一定の動きをしながら、酵素に最適な温度(この場合は37°C)に温度を厳密に制御する必要があります。
組換え酵素を用いた分析の全体的な方法では、SHP-2と表面に表示されたリン酸化基質との間の相互作用に特異的な修飾が必要でした。このプロトコールを他の細胞外酵素-基質相互作用に適合させるには、使用する配列、活性緩衝環境、および検出に使用する試薬の変更が必要です。他のキナーゼ-基質-ホスファターゼ相互作用をアッセイするために、適応には、キナーゼ-基質ペアのタンパク質配列を 、補足表S1に記載されているコンストラクトカセット内のそれぞれの位置に置き換えることが含まれます。基質のタンパク質配列は、少なくとも目的のキナーゼのキナーゼドメインと共にプラスミドに含まれるべきであり、産生されたリン酸化基質を標的とするホスファターゼは、組換えタンパク質の形態であるべきである。LCK、CD28、およびSHP-2の間の代表的な相互作用は、コンストラクトカセット内の設計された小胞体隔離を、それらの標的酵素を用いて細胞外に調査すべき翻訳後修飾タンパク質を産生するためのツールとして使用する例を提供する。翻訳後修飾を経る必要のない目的の基質(例えば、添加キナーゼを使用して細胞外でリン酸化できる基質)は、コンストラクトカセット内に対になった酵素を介さずに酵母表面に発現させることができます。この場合、 補足表S1 に記載されているキナーゼのタンパク質配列は、コンストラクトカセットに含まれる基質の配列のみで除去される。以前の経験から、既知の基質を持つセリン-スレオニンキナーゼの共局在により、リン酸化されていない基質が示されたことに注意します(Ezagui and Stern、未発表のデータ)ため、細胞外ホスファターゼを適用する前に、成功した酵素修飾の厳密な試験を実施する必要があります。私たちは以前に、この適格性ステップ39に役立つ可能性のあるキナーゼ-基質共局在のプロトコールを発表しました。
キナーゼおよびホスファターゼは、しばしば不対システイン残基を含み、酸化されると、タンパク質内またはタンパク質間でジスルフィド結合を形成し、これは立体構造の変化によりタンパク質の触媒活性を混乱させる可能性がある40,41。このタンパク質生化学を理解することは、酵素修飾の適切な反応環境を決定するために不可欠です。その結果、使用する組換えタンパク質が活性を維持するように、還元剤をインキュベーション環境に補充する必要があります。DTTはこれらの目的で使用される一般的な還元剤ですが、アッセイ中の濃度を最適化する必要があります。DTT濃度を高くしすぎると、Aga1pアンカーとAga2pアンカーがジスルフィド結合を介して互いに保持され、DTT42の存在下で還元されるため、酵母表面への基質の表示が妨げられます。DTTの濃度は、基質42の表面ディスプレイに有害な影響を与えることなく、相対的に最大のホスファターゼ活性を可能にする最小濃度に調整された。アッセイ対象の酵素のインキュベーション環境は、このアッセイで使用するときに酵素活性の保持を確実にするために最適化する必要があります。酵素が、このアッセイで使用される0.5 mM DTTよりもかなり強力な還元環境を必要とする場合、プラットフォームは表面ディスプレイの減少によって制限され、目的の特定の酵素アッセイに最適ではない可能性があります。同様に、このプロトコルのインキュベーションステップ中に使用された2x緩衝液は、SHP−2活性を促進する許容可能な緩衝液に関する先行研究から含まれており、同様の研究は、このアッセイに使用されている任意の他の酵素のためのインキュベーション緩衝液を処方するために行われるべきである34。これらのバッファーを作成するための出発点には、目的の酵素のin vitroアプリケーションの成功に関する文献検索や、活性バッファーに対する酵素メーカーの推奨が含まれます。使用する組換え酵素は、データ収集前に標的酵素活性を発現させることができるように、許容可能な濃度とインキュベーション時間を特定するために、このアッセイ専用に滴定する必要があります。
このプロトコルを他のタイプの酵素-基質相互作用に適合させるには、新しい蛍光検出試薬を選択し、感度を滴定する必要があります。他の研究では、プロテアーゼ処理後のペプチド基質の有無を検出するためのエピトープタグ標的抗体の使用23,25や、ヒストンタンパク質の修飾を検出するためのアセチル化感受性抗体の使用など、この例が実証されている43。これらの試薬を適格性評価するには、ポジティブコントロール(対象化合物の修飾を検証可能に示すもの)とネガティブコントロール(対象化合物の修飾がないことを検証可能に示すもの)を確立する必要があります。これは、以前に目的の修飾を実証したコンストラクトの酵母表面表示を通じて行うことができ、または場合によっては、組換えタンパク質またはペプチドの固定化を通じて確立することができる。例えば、リン酸化(および他の多くの関心のある翻訳後修飾)の場合、既知の配列のペプチドは、ホスホチロシン(ポジティブコントロール)または非修飾チロシン(ネガティブコントロール)のいずれかと、ストレプトアビジン被覆ビーズ上でペプチドを固定化できるC末端ビオチンで合成できます。ペプチドでコーティングされたビーズを修飾特異的抗体で標識し、セクション4で説明したのと同様の方法でフローサイトメトリーを使用して検出の特異性と感度を評価することができます。ポジティブコントロールの最大シグナル、ネガティブコントロールの最小シグナルを提供し、修飾タンパク質あたりの抗体の十分な過剰倍率(ステップ4.1の注の式を使用)と、得られた抗体のアリコートごとに実施する実験数に対する経済的考慮とのバランスをとる濃度を見つけるために、抗体の異なる希釈を使用する必要があります。
酵母表面ディスプレイプラットフォームの容易さを細胞外酵素活性アッセイに適合させるためのプロトコルについて説明します。この方法は、インキュベーション中に組換えSHP-2によって脱リン酸化される酵母表面に示されるリン酸化CD28を使用して実証されていますが、使用するワーキングバッファーおよび酵素-基質ペアの修飾を通じて、多くのタイプの酵素修飾に一般化できます。
開示事項
著者は、この作品に関連する利益相反を開示していません。
謝辞
この研究は、L.A.S. (CBET - 2339172) への NSF CAREER 賞と南フロリダ大学からのスタートアップ資金によって支援されました。
図2Aでは、Servier https://smart.servier.com/ によるマイクロチューブオープン半透明アイコンは、CC-BY 3.0 Unported https://creativecommons.org/licenses/by/3.0/ の下でライセンスされています。修飾には、バッファーと酵母細胞の添加(左)と抗体の添加(中央右)が含まれます。
図2Aの試験管、インキュベーター、およびフローサイトメーターは、オープンアクセスで www.bioicons.com を通じて提供されました。
資料
| Name | Company | Catalog Number | Comments |
| 1 L Media Bottles | Corning | 06-414-1D | |
| 1.7/2.0 mL Microtubes | Axygen | MCT-175-C | |
| 10 µL SureOne Pipet Tips | Fisher Scientific | 02-707-438 | |
| 1000 µL SureOne Pipet Tips | Fisher Scientific | 02-707-408 | |
| 12 mL Polystyrene Round-Bottom Tubes | Greiner | 07-000-212 | |
| 3 mL platic Cuvettes | BRAND | 759076D | |
| 300 µL SureOne Pipet Tips | Fisher Scientific | 02-707-411 | |
| 5 mL Serological Pipettes | Fisher Scientific | 13-678-11D | |
| Acid Casein (Casamino Acids) | Fisher Scientific | BP-1424-500 | |
| Analytical Balance | Mettler Toledo | 30243397 | |
| Bacteriological Petri Dish | Corning | Falcon 351008 | |
| Biosafety Cabinets | Labconco | Logic Class II, Type A2 302310102 | |
| Biospectrometer | Eppendorf | Kinetic 6136000010 | |
| Bovine Serum Albumin | Fisher bioreagents | BP1600-100 | |
| Citric Acid | Fisher Scientific | A940-500 | |
| CytoFLEX Flow Cytometry Analyzer | Beckam Coulter | Cytoflex C09745 | CytExpert software |
| Dextrose | Fisher Scientific | D16-1 | |
| Dithiothreitol | Fisher bioreagents | BP172-5 | |
| Donkey anti-goat FITC | Invitrogen | A16000 | |
| EDTA | Alfa Aesar | H56165.30 | |
| Ez-Link PEG4-NHS-Biotin | Thermo Scientific | A39259 | |
| Frozen-EZ Yeast Transformation II Kit | Zymo Research | T2001 | |
| Galactose | Fisher Scientific | BP656-500 | |
| General Purpose Refrigerator | Marvel Scientific | MS24RAS4RW | |
| Goat anti-myc tag antibody | Bethyl | A190-104A | |
| Mictrotube Centrifuge | Eppendorf | 5425 R 5406000313 | |
| Mini Low Temperature Refrigerated Incubator | Fisher Scientific | 15-015-2632 | |
| Mouse anti-phosphotyrosine antibody 4G10 | BioXcell | BE0194 | |
| Parafilm M | Bemis | M PM999 | |
| Phosphate Buffered Saline | Fisher bioreagents | BP399-500 | |
| Pipette Controller | Eppendorf | easypet 3 4430000018 | |
| Raffinose | Thermo Scientific | J21060-36 | |
| Recombinant human Active SHP-2 Protein | R&D Systems | 1894-SH | |
| Refrigerated Centrifuge | Eppendorf | 5910 R | |
| Saccharomyces cerevisiae yeast surface display strain EBY 100 | ATCC | MYA-4941 | |
| Shaker Incubator | Eppendorf | M1335-0002 New Brunswick Innova 42 | |
| Single Channel Pipette Set | Eppendorf | 05-403-151 | |
| Sodium Chloride | Fisher Scientific | S671-500 | |
| Sodium Citrate Dihydrate | Fisher Scientific | S279-500 | |
| Sodium Phosphate Dibasic Heptahydrate | Fisher Scientific | S373-500 | |
| Sodium Phosphate Monobasic Monohydrate | Fisher Scientific | S468-500 | |
| Streptavidin Alexa Fluor 647 | Invitrogen | S32357 | |
| Top Loading Balance | Mettler Toledo | ||
| Tris hydrochloride | EMD Millipore | 648317-100GM | |
| Tube revolver rotator | Fisher Scientific | 11-676-341 | |
| Weighing Paper | Fisher Scientific | 09-898-12B | |
| Yeast Nitrogen Base | BD Difco | 291940 | |
| Zeba Spin Desalting Columns | Thermo Scientific | 89883 |
参考文献
- Lea, M. A., Weber, G. Role of enzymes in homeostasis: VIII. Inhibition of the activity of glycolytic enzymes by free fatty acids. J Biol Chem. 243 (6), 1096-1102 (1968).
- Mahé, M., Rios-Fuller, T. J., Karolin, A., Schneider, R. J. Genetics of enzymatic dysfunctions in metabolic disorders and cancer. Front Oncol. 13, 1230934(2023).
- Fernandez-de-Cossio-Diaz, J., Vazquez, A. A physical model of cell metabolism. Sci Rep. 8 (1), 8349(2018).
- Metallo, C. M., Vander Heiden, M. G. Understanding metabolic regulation and its influence on cell physiology. Mol Cell. 49 (3), 388-398 (2013).
- Mildvan, A. S. Mechanisms of signaling and related enzymes. Proteins. 29 (4), 401-416 (1997).
- Frosina, G. Overexpression of enzymes that repair endogenous damage to DNA. Eur J Biochem. 267 (8), 2135-2149 (2000).
- Schärer, O. D. Chemistry and biology of DNA repair. Angew Chem Int Ed. 42 (26), 2946-2974 (2003).
- de la Fuente, M., et al. Enzyme therapy: Current challenges and future perspectives. Int J Mol Sci. 22 (17), 9181(2021).
- Robertson, J. G. Enzymes as a special class of therapeutic target: clinical drugs and modes of action. Curr Opin Struct Biol. 17 (6), 674-679 (2007).
- Goddard, J. -P., Reymond, J. -L. Enzyme assays for high-throughput screening. Curr Opin Biotechnol. 15 (4), 314-322 (2004).
- Helm, J. S., Hu, Y., Chen, L., Gross, B., Walker, S. Identification of active-site inhibitors of MurG using a generalizable, high-throughput glycosyltransferase screen. J Am Chem Soc. 125 (37), 11168-11169 (2003).
- Veldhuyzen, W. F., Nguyen, Q., McMaster, G., Lawrence, D. S. A light-activated probe of intracellular protein kinase activity. J Am Chem Soc. 125 (44), 13358-13359 (2003).
- Torres, M., Forman, H. J. Encyclopedia of Respiratory. Laurent, G. J., Shapiro, S. D. , Academic Press. 10-18 (2006).
- Blume-Jensen, P., Hunter, T. Oncogenic kinase signalling. Nature. 411 (6835), 355-365 (2001).
- Martin, G. S. Cell signaling and cancer. Cancer Cell. 4 (3), 167-174 (2003).
- Lothrop, A. P., Torres, M. P., Fuchs, S. M. Deciphering post-translational modification codes. FEBS Lett. 587 (8), 1247-1257 (2013).
- Graves, J. D., Krebs, E. G. Protein phosphorylation and signal transduction. Pharmacol Ther. 82 (2), 111-121 (1999).
- Hafen, E. Kinases and phosphatases--A marriage is consummated. Science. 280 (5367), 1212-1213 (1998).
- Westphal, R. S., Anderson, K. A., Means, A. R., Wadzinski, B. E. A signaling complex of Ca2+-calmodulin-dependent protein kinase IV and protein phosphatase 2A. Science. 280 (5367), 1258-1261 (1998).
- Barford, D., Das, A. K., Egloff, M. -P. The structure and mechanism of protein phosphatases: Insights into catalysis and regulation. Annu Rev Biophys Biomol Struct. 27, 133-164 (1998).
- Liu, Q., Qu, J., Zhao, M., Xu, Q., Sun, Y. Targeting SHP2 as a promising strategy for cancer immunotherapy. Pharmacol Res. 152, 104595(2020).
- Pan, J., Zhou, L., Zhang, C., Xu, Q., Sun, Y. Targeting protein phosphatases for the treatment of inflammation-related diseases: From signaling to therapy. Signal Transduct Targeted Ther. 7 (1), 177(2022).
- Denard, C. A., et al. YESS 2.0, a tunable platform for enzyme evolution, yields highly active TEV protease variants. ACS Synth Biol. 10 (1), 63-71 (2021).
- Lim, S., Glasgow, J. E., Filsinger Interrante, M., Storm, E. M., Cochran, J. R. Dual display of proteins on the yeast cell surface simplifies quantification of binding interactions and enzymatic bioconjugation reactions. Biotechnol J. 12 (5), (2017).
- Yi, L., et al. Engineering of TEV protease variants by yeast ER sequestration screening (YESS) of combinatorial libraries. Proc Natl Acad Sci USA. 110 (18), 7229-7234 (2013).
- Yi, L., et al. Yeast endoplasmic reticulum sequestration screening for the engineering of proteases from libraries expressed in yeast. Methods Mol Biol. 1319, 81-93 (2015).
- Semenza, J. C., Hardwick, K. G., Dean, N., Pelham, H. R. ERD2, a yeast gene required for the receptor-mediated retrieval of luminal ER proteins from the secretory pathway. Cell. 61 (7), 1349-1357 (1990).
- Mei, M., et al. Characterization of aromatic residue-controlled protein retention in the endoplasmic reticulum of Saccharomyces cerevisiae. J Biol Chem. 292 (50), 20707-20719 (2017).
- Ezagui, J., Russell, B., Mairena, Y., Stern, L. A. Endoplasmic reticulum sequestration empowers phosphorylation profiling on the yeast surface. AIChE J. 68 (12), e17931(2022).
- Kawai, S., Murata, K. Genetic Transformation Systems in Fungi. van den Berg, M. A., Maruthachalam, K. 1, Springer International Publishing. 187-192 (2015).
- Kawai, S., Hashimoto, W., Murata, K. Transformation of Saccharomyces cerevisiae and other fungi: methods and possible underlying mechanism. Bioeng Bugs. 1 (6), 395-403 (2010).
- Loock, M., et al. High-efficiency transformation and expression of genomic libraries in yeast. Methods Protoc. 6 (5), 89(2023).
- Huang, D., Gore, P. R., Shusta, E. V. Increasing yeast secretion of heterologous proteins by regulating expression rates and post-secretory loss. Biotechnol Bioeng. 101 (6), 1264-1275 (2008).
- Yu, B., et al. Targeting protein tyrosine phosphatase SHP2 for the treatment of PTPN11-associated malignancies. Mol Cancer Ther. 12 (9), 1738-1748 (2013).
- Stern, L. A., et al. Geometry and expression enhance enrichment of functional yeast-displayed ligands via cell panning. Biotechnol Bioeng. 113 (11), 2328-2341 (2016).
- Pan, X., et al. Optimized single-cell gates for yeast display screening. Protein Eng Design Sel. 38, gzae018(2025).
- Margittai, É, et al. Production of H2O2 in the endoplasmic reticulum promotes in vivo disulfide bond formation. Antioxid Redox Signal. 16 (10), 1088-1099 (2012).
- Werner-Washburne, M., Braun, E., Johnston, G. C., Singer, R. A. Stationary phase in the yeast Saccharomyces cerevisiae. Microbiol Rev. 57 (2), 383-401 (1993).
- Ezagui, J., Stern, L. A. Tyrosine phosphorylation screening on the yeast surface by magnetic bead selection and FACS. Methods Mol Biol. 2681, 275-290 (2023).
- Yarnall, M. T. N., Kim, S. H., Korntner, S., Bishop, A. C. Destabilization of the SHP2 and SHP1 protein tyrosine phosphatase domains by a non-conserved “backdoor” cysteine. Biochem Biophys Rep. 32, 101370(2022).
- Dustin, C. M., Heppner, D. E., Lin, M. J., van der Vliet, A. Redox regulation of tyrosine kinase signalling: more than meets the eye. J Biochem. 167 (2), 151-163 (2020).
- Stern, L. A., Csizmar, C. M., Woldring, D. R., Wagner, C. R., Hackel, B. J. Titratable avidity reduction enhances affinity discrimination in mammalian cellular selections of yeast-displayed ligands. ACS Comb Sci. 19 (5), 315-323 (2017).
- Waldman, A. C., Rao, B. M., Keung, A. J. Mapping the residue specificities of epigenome enzymes by yeast surface display. Cell Chem Biol. 28 (12), 1772-1779.e4 (2021).
転載および許可
このJoVE論文のテキスト又は図を再利用するための許可を申請します
許可を申請さらに記事を探す
This article has been published
Video Coming Soon
Copyright © 2023 MyJoVE Corporation. All rights reserved