Method Article
Enzymatische Modifikation und Durchflusszytometrie-Bewertung von Proteinen, die auf der Hefeoberfläche angezeigt werden
* Diese Autoren haben gleichermaßen beigetragen
In diesem Artikel
Zusammenfassung
Hier stellen wir ein Protokoll für die Verwendung von oberflächenexponierten Substraten der Hefe für enzymatische Modifikationsassays vor. Die Plattform wurde anhand der Analyse der Dephosphorylierungsaktivität der Tyrosinphosphatase SHP-2 gegen eines ihrer Substrate als repräsentativer enzymatischer Modifikationsassay demonstriert.
Zusammenfassung
Die Anzeige der Hefeoberfläche ist eine Genotyp-Phänotyp-Kopplungsstrategie, die ein Hochdurchsatz-Screening der Proteinfunktion ermöglicht. Traditionell wird die Darstellung der Hefeoberfläche auf die Evolution neuer Bindungsproteine angewendet, wobei die Durchflusszytometrie verwendet wird, um die Bindungsstärke zu beurteilen und zu sortieren. In jüngster Zeit besteht ein wachsendes Interesse an der Anwendung von Hefeoberflächendisplays für das Screening enzymatischer Modifikationen von Substratvarianten, wobei additive (z. B. Phosphorylierung) oder subtraktive (z. B. Proteolyse) Modifikationen einen Phänotyp liefern, der durch Durchflusszytometrie ablesbar ist. Solche Modifikationen werden regelmäßig durch intrazelluläre Kolokalisierung angewendet, aber die Fähigkeit, eine extrazelluläre enzymatische Modifikation der angezeigten Substrate zu erreichen, könnte viele weitere Reaktionen für die Untersuchung eröffnen. Hier beschreiben wir Techniken für das Design und die Anwendung von Screening-Assays für die extrazelluläre enzymatische Modifikation auf Kandidatensubstrate, die auf der Hefeoberfläche angezeigt werden, und die anschließende Bewertung mittels Durchflusszytometrie-Analyse. Wir stellen diese Protokolle im Zusammenhang mit Phosphatasen zur Dephosphorylierung von Hefesubstraten zur Verfügung, die phosphorylierte Tyrosinreste enthalten, und kommentieren, wie dieses angewandte Framework für die Entwicklung von Screening-Assays für andere Enzym-Substrat-Paare angepasst werden kann.
Einleitung
Das Verständnis der Wechselwirkungen zwischen Enzymen und ihren Zielen ist aufgrund seiner Notwendigkeit für die biologische Charakterisierung der Signalwege, die die zelluläre Homöostase und die Krankheitsentwicklung steuern, zu einem zunehmend interessanten Forschungsgebiet geworden 1,2. Enzyme sind für die Katalyse vieler Reaktionen verantwortlich, die biologisches Leben aufrechterhalten, und steuern notwendige Signalwege wie den Zellstoffwechsel 3,4, die Signalübertragung5 und sogar grundlegende Prozesse wie die Genomreparatur 6,7. Aufgrund ihrer Rolle in diesen Prozessen spielen ihre Wechselwirkungen auch eine Rolle bei der Entstehung vieler Krankheiten, da Abweichungen in ihrer Aktivität zu schweren Dysregulationen der Zellaktivität führen können, die eine Apoptose oder Vermehrung schädlicher Krebszellen verursachen2. Die Untersuchung der enzymatischen Aktivität hat wichtige Anwendungen bei der Entwicklung neuer Therapeutika 8,9 gefunden, die Assays erfordern, die auf jede spezifische Enzym-Substrat-Wechselwirkungzugeschnitten sind 10. Mehrere enzymatische Assays haben sich als Standardprotokolle für die Bewertung und Charakterisierung dieser Wechselwirkungen etabliert. Assays, die zur Analyse enzymatischer Wechselwirkungen entwickelt wurden, werden in Detektionsassays eingeteilt, die die Bindung zur Aktivierung/Hemmungüberwachen 11, oder Assays, die die Substratmodifikation durch Enzymeüberwachen 12.
Eine wichtige Rolle von Enzymen ist die Regulierung des Zellverhaltens. Die Signaltransduktion, die intrazelluläre Reaktion einer Zelle auf einen extrazellulären Auslöser13, ist für das Überleben und die Funktionalität der Zellen verantwortlich. Zellproliferation, Differenzierung und viele andere funktionelle Prozesse beinhalten Signalwege mit enzymatischen Wechselwirkungen, die gesteuert werden14,15. Enzyme katalysieren posttranslationale Modifikationen, die häufig die massiven Signalnetzwerke modulieren, die für die korrekte Übertragung extrazellulärer Nachrichten verantwortlich sind16. Die Proteinphosphorylierung ist die häufigste posttranslationale Modifikation, die in der zellulären Signalübertragung und mehreren anderen zellulären Signalwegen allgegenwärtig ist. Folglich haben sich Proteinkinasen aufgrund ihrer entscheidenden regulatorischen Rolle als signifikanter Teil potenzieller therapeutischer Ziele herausgestellt17. Phosphatasen sind die natürlichen modulatorischen Moleküle für phosphatbasierte Zellsignalkomplexe18,19 und haben die Fähigkeit, Phosphatreste von ihren Zielproteinenzu entfernen 20. In den letzten zehn Jahren haben sich Phosphatasen zu einem wichtigen therapeutischen Ziel für die Behandlung von Krebs21 und entzündlichen Erkrankungen22 entwickelt, da sie an der Regulation nachgeschalteter Signalwege in mehreren Zelltypen beteiligt sind. Zusammen bieten Proteinkinasen und Phosphatasen eine breite Palette von Wechselwirkungen, die durch die Entwicklung spezifischer enzymatischer Assay-Protokolle untersucht werden können.
Das Display der Hefeoberfläche wurde als Werkzeug zur Charakterisierung und Bewertung der enzymatischen Aktivität verwendet23,24. Es bietet eine Hochdurchsatzplattform für das Screening posttranslationaler Modifikationsprozesse in Kombination mit endoplasmatischen Retikulum-Sequestrierungsstrategien25,26. Dies ermöglicht die Kolokalisierung und Beibehaltung von Kinase-Substrat-Paaren im endoplasmatischen Retikulum durch Bindung an KDEL-Rezeptoren27, wo die Phosphorylierung des Substrats aufgrund der Nähe zwischen Kinasen und ihren Zielen mit erhöhter Geschwindigkeit erfolgen kann. Die KDEL-Rezeptorbindung wird durch eine C-terminale FEHDEL-Retikulum-Retentionssequenz vermittelt, die nachweislich eine stärkere Retentionsfähigkeit aufweist als andere HDEL-Sequenzen25,28. Das phosphorylierte Substrat wird dann an der Hefeoberfläche verankert, wo es anschließend durch Durchflusszytometrieausgewertet werden kann 29. Derzeit gibt es keine verallgemeinerbaren Protokolle für die enzymatische Modifikation von Substraten, die auf der Hefeoberfläche angezeigt werden. Wir erweitern die Kapazitäten der Hefeoberflächendarstellung, indem wir die extrazellulär exprimierten phosphorylierten Substratvarianten nutzen und sie durch Dephosphorylierung durch ihre bekannte Phosphatase modifizieren. Die durchflusszytometrische Analyse bietet dann eine Plattform für die phänotypische Bewertung der oben genannten Substrate durch die Messung von Veränderungen des Phosphorylierungsmedians als Folge der Inkubation mit der bekannten Phosphatase. Dies bietet eine anpassungsfähige Methode zur posttranslationalen Modifikation von oberflächenexponierten Proteinen und gleichzeitig eine Methode zur enzymatischen Modifikationsanalyse von Wechselwirkungen bei der Verwendung der Hefeoberflächenanzeigeplattform.
Wir stellen Techniken zur Entwicklung und Anwendung eines enzymatischen Modifikationsassays vor, der die Einführung einer Kinase-Substrat-Interaktion in die Display-Plattform der Hefeoberfläche, die Co-Inkubation des exprimierten phosphorylierten Substrats mit einer rekombinanten Phosphatase und die anschließende Analyse der Dephosphorylierungsaktivität durch Durchflusszytometrie beschreibt. In diesem Bericht wird dies durch die Co-Lokalisierung der zytoplasmatischen Domäne von CD28 mit der Lymphozytenkinase (LCK) im endoplasmatischen Retikulum der Hefe erreicht, gefolgt von der Darstellung des phosphorylierten CD28 auf der Hefeoberfläche und der anschließenden Dephosphorylierung durch die domänenenthaltende Phosphatase-2 (SHP-2) der Src-Homologieregion 2. Ein pan-Anti-Phosphotyrosin-Antikörper (in dieser Studie 4G10), der phosphorylierte Tyrosinreste in einer Vielzahl von Peptidsequenzen nachweist, wird zur Quantifizierung des Phosphorylierungsgrades in Abhängigkeit von der Phosphatasebehandlung verwendet. Der detaillierte Prozess bietet einen verallgemeinerbaren Ansatz zur Untersuchung von Enzym-Substrat-Wechselwirkungen; Eine prospektive Methode zur Untersuchung von Enzymen und Substraten in gereinigter Form.
Protokoll
1. Zellwachstum von Hefe, die Plasmide beherbergt, und Induktion der Proteinexpression
- Bereiten Sie nach dem in Tabelle 1 beschriebenen Rezept die Medien vor, die für das Wachstum von nicht-plasmidhaltiger Hefe (YPD), plasmidhaltiger Hefezellwachstum (SD-CAA) und die Induktion der Proteinexpression (SRG-CAA) sowie SD-CAA-Platten erforderlich sind.
- Transformieren Sie Hefe-Display-Plasmid-DNA, die das Kinase-Substrat-Paar enthält, in EBY-100-Hefezellen durch die auf Lithiumkationen basierende Methode30,31, die üblicherweise für die Verwendung in Hefetransformationskits einer Vielzahl von Herstellern verwendet wird.
HINWEIS: Die Elektroporation32 oder andere bevorzugte Hefeplasmid-Transformationstechniken können je nach Plasmidkonstrukt, das transformiert wird, verwendet werden. - Bereiten Sie ein 14-ml-Kulturröhrchen mit 10 mL YPD-Medien vor. Beimpfen Sie EBY-100-Zellen und züchten Sie sie in einem Schüttelinkubator bei 30 °C und 300 U/min, bis die Kultur eine optische Dichte (OD600 nm) von 0,8-1,0 (8 x 106- 1 x 107 Zellen/ml) erreicht.
HINWEIS: OD600 nm wird gemessen, indem 3 ml-Probenküvetten mit 1:10-Verdünnungen von Hefekulturen in ihrem jeweiligen Medium und 3-ml-Blankoküvetten mit dem für die Probenverdünnung verwendeten Medium hergestellt werden. Das OD600-nm-Programm auf dem Spektralphotometer wird verwendet, um zuerst die leere Küvette und dann jede Probenküvette zu messen, indem die entsprechende Verdünnung für jede Probe eingestellt wird. 1 OD600nm entspricht 1 × 107 Hefe/ml. - Ernten Sie die Zellen, indem Sie die Kultur 3 Minuten lang bei 1.000 x g zentrifugieren und mit der 1. Waschlösung im Hefetransformationskit oder TE (10 mM Tris-HCl und 1,0 mM EDTA)31 waschen.
- Pelletieren Sie die Zellen erneut bei 1.000 x g für 3 Minuten und resuspendieren Sie in 1 ml des im Hefetransformationskit enthaltenen Transformationspuffers oder in sterilem Wasser. Die Zellen sollten in 50 μL Aliquots aliquotiert werden und können bis zu 6 Monate bei -80 °C gelagert werden.
- Für die Plasmidtransformation wird ein Aliquot pro Plasmid hergestellt und auf Eis aufgetaut, dann werden 0,5-1,5 μg Plasmid-DNA, die das Hefe-Display-Konstrukt enthält, direkt in die Zellen gegeben. 0,5 ml der im Hefetransformationskit bereitgestellten Transformationslösung werden zugegeben, oder 0,5 ml eines sterilen 50%igen Polyethylenglykols und 0,1 M LiOAc-Lösung31. Kombinieren Sie die Mischung aus Zellen, Plasmid-DNA und Transformationslösung gründlich durch Pipettieren.
- Transformationsgemisch statisch für 30-60 min bei 30 °C inkubieren, Wirbelgemisch in Abständen von 15 min. Ernte der Zellen durch Zentrifugieren bei 1.000 × g für 3 min.
- Bereiten Sie ein 14-ml-Kulturröhrchen mit 4,5 mL SD-CAA-Medien vor. Resuspendieren Sie die Zellen, die das gewünschte Plasmid in 500 μl SD-CAA enthalten, und inokulieren Sie die vorbereiteten 4,5 mL.
- Achten Sie darauf, den Agar nicht zu durchstechen, verteilen Sie 50 μl der 5 ml der inokulierten Kultur auf eine SD-CAA-Platte und inkubieren Sie statisch bei 30 °C für 48 Stunden, um die Transformationseffizienz zu bestimmen.
- Inkubieren Sie die 5 ml SD-CAA-Zellkultur in einem Schüttelinkubator bei 30 °C und 300 U/min für mindestens 18 Stunden. Überwachen Sie die optische Dichte (OD600nm) nach 16 h und 20 h. Sobald die Probe auf eine ausreichende optische Dichte von nicht mehr als 6 angewachsen ist, wird die Kultur 3 Minuten lang bei 2.500 × g zentrifugiert. Entsorgen Sie den Überstand, ohne das Hefepellet zu stören.
- Resuspendieren Sie das Hefepellet in SRG-CAA auf einen End-ODvon 600 nm weniger als 1 (<1 × 107 Hefe/ml).
- Inkubieren Sie die Hefekultur in einem Schüttelbrutschrank bei 30 °C und 300 U/min für mindestens 8 h, jedoch nicht länger als 24 h.
ANMERKUNG: Die Induktion der Proteinexpression in Hefezellen kann zwischen 20 und 37 °C variiert werden. 30 °C ist für die Synthese von Kinase/Substrat-Paarengeeignet 29,33, kann aber angepasst werden, wenn dies für die zu untersuchenden spezifischen Proteine als notwendig erachtet wird. - Messen Sie OD600 nm , um die Zelldichte zu bestimmen.
HINWEIS: Das Protokoll kann an dieser Stelle gestoppt werden, indem die Hefekulturen bei 4 °C gelagert werden.
2. Biotinylierung des 4G10-Anti-Phosphotyrosin-Antikörpers
- Eine Durchstechflasche mit 2 mg PEG4-NHS-Biotin wird durch Zugabe von 680 μl sterilem PBS auf eine Endkonzentration von 5 mM resuspendiert.
HINWEIS: Die Resuspension von PEG4-NHS-Biotin sollte unmittelbar vor der Biotinylierungsreaktion frisch erfolgen. NHS hydrolysiert in wässriger Lösung. Die Verwendung von sterilem PBS und Fläschchen ist wichtig für die Vorbereitung von Reagenzien für die Langzeitlagerung und für die Verwendung in zellbasierten Assays, um mögliche Auswirkungen von Kontaminanten auf die Lebensfähigkeit der Reagenzien oder die durchgeführten empfindlichen Assays zu mildern. - Basierend auf der 4G10-Antikörperkonzentration 100 μg Antikörper in ein steriles 1,7-ml-Fläschchen geben.
- 1 μl des vorbereiteten 5 mM PEG4-NHS-Biotin aus Schritt 2.1 in das Fläschchen mit den 100 μg 4G10-Antikörper geben, um ein molares Verhältnis von Biotin zu Antikörper von 7,5:1 zu erreichen. Pipettieren Sie die Mischung vorsichtig, um die Reaktion zu homogenisieren.
- Die Reaktion wird bei Raumtemperatur mit konstanter Rotation mindestens 2 h lang inkubiert.
- Befolgen Sie das Protokoll des Herstellers für 0,5-ml-Spin-Entsalzungssäulen, um den Puffer vom biotinylierten 4G10 (B-4G10) in PBS auszutauschen.
HINWEIS: Entsalzungssäulen mit einem Molekulargewichts-Cutoff (MWCO) von 7 kDa werden häufig für die Biotinylierung von Antikörpern verwendet, um die Entfernung von nicht umgesetztem Biotin und anderen kleinen Molekülen zu ermöglichen, während der größere Antikörper erhalten bleibt. - Verdünnen Sie den B-4G10-Antikörper auf eine Endkonzentration von 1 μM in PBSA (PBS mit 1 g/l Rinderserumalbumin). Aliquotieren Sie den B-4G10-Antikörper in kleinere Volumina, um wiederholte Gefrier-/Auftauzyklen zu verhindern.
HINWEIS: Biotinylierte Antikörper können bis zu 3 Monate lang bei 4 °C für den täglichen Gebrauch gelagert werden, ohne dass die Wirksamkeit wesentlich beeinträchtigt wird. Die restlichen Aliquote, die nicht verwendet werden, werden maximal 2 Jahre lang bei -20 °C gelagert.
3. Dephosphorylierung von Substraten, die auf der Oberfläche der Hefezellen exprimiert werden
- Bereiten Sie die 2x Arbeitspufferlösung vor, wie zuvor in der Literaturbeschrieben 34.
HINWEIS: 2x wird empfohlen, um die Messung der benötigten Inhaltsstoffe zu erleichtern. - Bereiten Sie den Arbeitspuffer für die Proben in einem 1,7-ml-Fläschchen vor, indem Sie die in Schritt 3.1 hergestellte 2x-Pufferlösung 1:2 in deionisiertem Wasser verdünnen.
HINWEIS: Das Gesamtreaktionsvolumen für jede Probe beträgt 20 μl, und jede Probe benötigt zwischen 10 μl und 18 μl Arbeitspuffer. Bereiten Sie genügend Arbeitspuffer für alle Proben oder Kontrollen vor. - Gemäß der in Tabelle 2 beschriebenen empfohlenen Probenvorbereitung beschriften Sie 1,7-ml-Fläschchen mit dem entsprechenden Kontroll- oder Probennamen.
- Auf der Grundlage des in Schritt 1.9 gemessenen OD600 nm ist das Kulturvolumen zu berechnen, das erforderlich ist, um für jede Probe zwei Millionen (2 × 106) Hefezellen aus der entsprechenden Hefekultur zu gewinnen.
- Geben Sie das im vorherigen Schritt berechnete Volumen der Hefekultur für jede Probe in das 1,7-ml-Fläschchen.
- Zentrifugieren Sie das Fläschchen 1 Minute lang bei 4.500 × g. Überstand vorsichtig mit einer Mikropipette entfernen und als biologisch gefährlichen Abfall entsorgen.
- Die pelletierten Zellen werden in 1 ml PBSA resuspendiert und Schritt 3.6 wiederholt.
HINWEIS: Es ist wichtig, so viel Überstand wie möglich zu entfernen, ohne die pelletierten Zellen zu stören. - Berechnen Sie auf der Grundlage seiner Stammkonzentration das Volumen des rekombinanten humanen SHP-2, das erforderlich ist, um eine Endkonzentration von 1.000 nM in einem Gesamtreaktionsvolumen von 20 μl zu erreichen.
HINWEIS: Rekombinantes SHP-2 sollte in kleinen Volumina aliquotiert werden, so dass kein Protein, das in jedem Assay verwendet wird, mehr als zwei Gefrier-Auftau-Zyklen durchlaufen hat. Alle SHP-2-Reste eines Aliquots, nachdem alle Proben für einen Assay vorbereitet wurden, sollten verworfen werden.
Die Stammkonzentrationen rekombinanter Enzyme können je nach Chargennummer variieren. Rekombinantes SHP-2 wird üblicherweise in einer Stammkonzentration von 0,2-0,4 mg/ml formuliert. Bei einer Stammkonzentration von 0,324 mg/mL SHP-2 entspricht dies einer Stammkonzentration von 4,696 μM (SHP-2 hat ein Molekulargewicht von 69 kDa). 4,26 μl des SHP-2-Stammes in einer 20-μl-Reaktion führen zu einer endgültigen Reaktionskonzentration von 1.000 nM SHP-2. - 7,7 mg DTT in 10 mL deionisiertes Wasser geben, das in einem konischen 15-ml-Wasser hergestellt wurde, um eine 5 mM DTT-Lösung herzustellen. Wenn das Wiegen von Milligramm aufgrund von Geräteeinschränkungen schwierig ist, fügen Sie 0,77 g DVB-T zu 10 ml deionisiertem Wasser hinzu und führen Sie dann eine 100-fache Verdünnung durch, um die für den Assay verwendete 5-mM-DVB-T-Lösung herzustellen.
HINWEIS: Die DVB-T-Lösung kann in Stammlösungen mit höherer Konzentration hergestellt werden, um auf 5 mM verdünnt zu werden, wenn es mit den verfügbaren Geräten nicht möglich ist, Mengen im Milligramm-Maßstab zu messen. Die DTT-Lösung muss frisch vor der Resuspension der Zellen in einem Arbeitspuffer hergestellt werden, da sie zur Hydrolyse neigt und bei Verdünnung in Wasser über lange Zeiträume instabil ist. - Die pelletierten Zellen werden in dem in Schritt 3.2 hergestellten Arbeitspuffer resuspendiert, so dass das endgültige Reaktionsvolumen in jeder Probe oder Kontrolle 20 μl beträgt.
HINWEIS: Die Menge des zugegebenen Arbeitspuffers sollte auf der Grundlage der Menge an DTT (2 μl) und SHP-2 (berechnet in Schritt 3.8) berechnet werden, die in jeder Probe enthalten ist. - 2 μl der in Schritt 3.9 hergestellten 5 mM DTT-Lösung werden zu jeder Probe oder Kontrolle für eine endgültige Reaktionskonzentration von 0,5 mM DTT gegeben.
- Das in Schritt 3.8 berechnete SHP-2-Volumen wird zu jeder Probe hinzugefügt, um ein Endvolumen von 20 μl zu erhalten, und mit einer Mikropipette vorsichtig gemischt.
- Wickeln Sie die Deckel der Probenfläschchen in Parafilm ein, um ein Auslaufen oder Kreuzkontamination zu verhindern.
- Die Proben werden bei 37 °C für 2 h auf einem Rotor bei konstanter Drehzahl inkubiert.
- Entnehmen Sie die Proben aus dem Rotor und stoppen Sie die Reaktion, indem Sie jeder Probe 1 ml PBSA hinzufügen.
- Wiederholen Sie Schritt 3.6.
4. Zellmarkierung und durchflusszytometrische Analyse von dephosphorylierten Substraten
- Proben aus Schritt 3.16 werden in einer 20-μl-Mischung der entsprechenden Primärreagenzien resuspendiert, wie in Tabelle 2 beschrieben. Inkubieren Sie die Proben 20 Minuten lang bei Raumtemperatur.
HINWEIS: Alle verwendeten Reagenzkonzentrationen wurden so berechnet, dass sie in Bezug auf die Anzahl der Proteine, die auf der Oberfläche von Hefezellen exprimiert werden, über dem Niveau liegen. Die Berechnung geht von der Expression von 10.000 Proteinen/Zelle aus allen 2 x 106 Hefen in einer Probe35 aus, während dies routinemäßig nur ~50% von ihnen der Fall ist. Tabelle 3 zeigt das überschüssige Markierungsreagenzienverhältnis für jedes der in Tabelle 2 ausgedrückten Reagenzien, berechnet wie folgt:

- Zentrifugieren Sie die Proben bei 4.500 × g für 1 min und entsorgen Sie den Überstand als biologisch gefährlichen Abfall.
- Waschen Sie die Zellen einmal, indem Sie sie in 1 ml PBSA resuspendieren. Wiederholen Sie Schritt 4.2.
- Die Proben werden in einer 20-μl-Mischung der entsprechenden Sekundärreagenzien resuspendiert, wie in Tabelle 2 beschrieben. Inkubieren Sie die Proben 15 Minuten lang unter Lichteinfall.
- Wiederholen Sie Schritt 4.2.
- Wiederholen Sie Schritt 4.3.
- Die gewaschenen Proben werden in 300-500 μl PBSA resuspendiert und in 5 mL Polystyrolröhrchen überführt, um sie sofort mit einem geeigneten Durchflusszytometer zu analysieren.
HINWEIS: Wenn Proben transportiert werden müssen, bewahren Sie sie auf nassem Eis auf. Es wird nicht empfohlen, aber die Proben können bei 4 °C für maximal 2 h als Nasspellets gelagert werden. - Nachdem Sie den erforderlichen Start und die Vorbereitung des Zytometers für ein neues Experiment durchgeführt haben, klicken Sie im Menü Datei auf die Schaltfläche Neues Experiment, benennen Sie das Experiment und klicken Sie auf Speichern, um sicherzustellen, dass die erfassten Daten im gewünschten Dateipfad gespeichert werden.
- Wählen Sie das Punktdiagramm-Symbol in der oberen Symbolleiste aus, um zwei oder mehr Punktdiagramme für jedes auszuführende Muster zu erstellen. Wählen Sie für eines der Punktdiagramme den Namen der X-Achse aus, um den FSC-A-Kanal anzuzeigen, und den Namen der Y-Achse, um den SSC-A-Kanal anzuzeigen. Dieses Diagramm zeigt die Side Scatter - Area im Vergleich zur Forward Scatter - Area und wird verwendet, um Hefezellen für die weitere Analyse zu gaten.
- Wählen Sie in einem anderen Punktdiagramm den Namen der X-Achse aus, um den Kanal anzuzeigen, in dem das sekundäre Reagenz, das auf den primären Anti-Epitop-Tag-Antikörper abzielt, fluoresziert. Wählen Sie den Namen der Y-Achse aus, um den Kanal anzuzeigen, in dem das Sekundärreagenz von Streptavidin fluoresziert. Dieses Diagramm zeigt nur die Ereignisse, die von der Seite nach vorne als Hefezellen gesteuert werden, und wird verwendet, um die Tyrosinphosphorylierung auf der Y-Achse und die Expression der Substratoberfläche auf der X-Achse anzuzeigen.
HINWEIS: In diesem Beispiel fluoresziert das Sekundärreagenz, das auf den primären Anti-Epitop-Tag-Antikörper abzielt, im FITC-Kanal (AF-488), und das Sekundärreagenz von Streptavidin fluoresziert im AF-647-Kanal. Die verwendeten Kanäle können je nach den primären und sekundären Reagenzien, die während der Markierung verwendet werden, variieren. - Setzen Sie jedes Probenröhrchen in den Röhrchenhalter des Zytometers ein und wählen Sie Ausführen für das Zytometer, um mit dem Laden der Probe und der Datenerfassung zu beginnen. Passen Sie die anzuzeigenden Ereignisse, die aufzuzeichnenden Ereignisse, die aufzuzeichnende Zeit und die Abtastdurchflussrate nach Bedarf an.
- Definieren Sie ein Tor, das die gesunden Hefezellen im Diagramm SSC-A vs. FSC-A umgibt, das in Schritt 4.9 erstellt wurde. Abbildung 1 veranschaulicht eine beschreibende Darstellung der anzuwendenden Gating-Strategie.
HINWEIS: Auf dem SSC-A- und FSC-A-Diagramm sind 100.000 anzuzeigende Ereignisse und 50.000 aufzuzeichnende Ereignisse innerhalb des definierten Hefegatters eine gute Richtlinie, um ausreichende Daten für die weitere Analyse zu visualisieren und zu sammeln. Strengere Gating-Strategien zur Selektion von Singulett-Hefe können auf der Grundlage eines Diagramms von Forward Scatter-Height versus Forward Scatter-Area angewendet werden, wie kürzlich berichtetwurde 36. Die in Abbildung 1 gezeigte Gating-Strategie entspricht einem weniger stringenten Ansatz, der Singulett- und etwas Dublett-Hefe durch Scatter-Gating umfasst. - Aufzeichnung der Fluoreszenz aller Kontrollproben mit einem Durchflusszytometrie-Analysator. Zuerst werden routinemäßig Kontrollproben entnommen, um eine Gating-Strategie zu definieren, wie unten in Schritt 4.14 beschrieben.
- Definieren Sie eine Gating-Strategie für Ihre in Schritt 4.10 erstellte Parzelle vor der Analyse der behandelten Proben. Abbildung 1 veranschaulicht eine beschreibende Darstellung der anzuwendenden Gating-Strategie.
- Aufzeichnung der Fluoreszenz von dephosphorylierten Proben mit dem Durchflusszytometer und der in den Schritten 4.12 und 4.14 definierten Gating-Strategie.
- Analysieren Sie Durchflusszytometriedaten, die mit einer Durchflusszytometrie-Analysesoftware erfasst wurden.
- Bewertung der Dephosphorylierung durch Messung und Vergleich des Medians der Y-Achse von Zellen, die Proteine auf ihrer Oberfläche exprimieren, und der Phosphorylierung zu Studienbeginn, die von nicht anzeigenden Zellen zwischen Proben und Kontrollen bereitgestellt wird. Berechnen Sie die prozentuale mediane Phosphorylierungsdifferenz wie folgt:
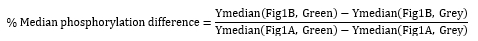
Ergebnisse
Die durchflusszytometrische Analyse eines einzelnen Replikats unseres Modellsystems, das 2 Stunden lang ohne (Abbildung 1A) und mit (Abbildung 1B) 1.000 nM SHP-2 inkubiert wurde, zeigt eine mediane Phosphorylierungsdifferenz von 63,6 %, die definiert ist als das Verhältnis des Medians der Y-Achse (Phosphorylierung) von allen an der Oberfläche angezeigten Ereignissen abzüglich des Phosphorylierungssignals zu Studienbeginn, definiert als der Median der Y-Achse der nicht angezeigten Ereignisse zwischen der behandelten Probe und der nicht behandelten Kontrolle, wie in der Gleichung beschrieben im Protokollschritt 4.12 definiert. Vor der Analyse der Mediane wurden die Proben basierend auf ihrer Größe (Vorwärtsstreuung) und Komplexität (Seitenstreuung) so begrenzt, dass sie eine gesunde Gruppe von Zellen umfasste. Unter den definierten Bedingungen sollte die Dephosphorylierung der Probe gut sichtbar erkennbar sein (Abbildung 1C).
Der Assay wird durch ein einfaches Verfahren definiert, das aus vier Hauptmethoden besteht (Abbildung 2A). Das von uns verwendete und bereits veröffentlichte Hefeoberflächenanzeigesystem basiert auf einem Plasmid, das einen bidirektionalen Promotor mit der Fähigkeit zur gleichzeitigen induzierbaren Expression eines Kinase-Substrat-Paares enthält, das aus dem zytoplasmatischen Schwanz von CD28 und der Tyrosinkinase LCK besteht (Abbildung 2B)29. Die Co-Lokalisierung der translatierten Proteine wird durch ein endoplasmatisches Retikulum gesteuert, das auf das Signalpeptid abzielt, und die posttranslationale Modifikation, die durch eine erhöhte Verweilzeit infolge einer C-terminalen ER-Retentionssequenz erzwungen wird. Die Sekretion des phosphorylierten Substrats, das mit Aga2p fusioniert ist, führt zur Oberflächenexpression (Abbildung 2C). Das Substrat ist so konzipiert, dass es von zwei Epitop-Tags flankiert wird, was die extrazelluläre Bestätigung der erfolgreichen Translation und die anschließende Oberflächenexpression ermöglicht. Die Inkubation des Substrats, das Hefezellen zeigt, mit der interessierenden Phosphatase (in diesem Fall Tyrosinphosphatase SHP-2) ermöglicht die Analyse der Enzymmodifikation durch die Abnahme des substratgebundenen Phosphats (Abbildung 2D).
Obwohl für das vorgestellte Modellsystem optimale Bedingungen definiert wurden, erlaubt die Generalisierbarkeit des Assays eine Diversifizierung der zu analysierenden Proteine. Die optimalen Bedingungen für den Assay wurden durch eine Reihe von Titrationen definiert, bei denen verschiedene Kombinationen von Zeit und Konzentration der Phosphatase in vierfacher Ausfertigung ausgewertet wurden (Abbildung 3). Die analysierten Daten zeigten eine statistische Signifikanz durch eine bidirektionale ANOVA mit Replikation (p < 0,05). Die gewählten Bedingungen von 2 h und 1.000 nM (48,8 % ± 10 %) boten einen mittleren Phosphorylierungsunterschied von etwa 50 % bei gleichzeitiger Beibehaltung der statistischen Signifikanz im Vergleich zu seinem Gegenstück von 750 nM nach 2 h (p < 0,05), basierend auf einer t-Test-Analyse mit ungleicher Varianz. Der t-Test zeigte auch keinen signifikanten Unterschied zum 2-Stunden- und 1.000-nM-Ergebnis, indem die Zeit unter einer der Konzentrationen, die einen ungefähren medianen Prozentsatz der Phosphorylierungsdifferenz ergaben, um 1 Stunde verlängert wurde (p > 0,05 für 500 nM, 750 nM und 1.000 nM nach 3 Stunden).
Die Tukey HSD-Post-hoc-Analyse zeigt, dass alle mittleren Vergleiche zwischen den Inkubationsperioden über alle Konzentrationen hinweg signifikant unterschiedlich sind, abgesehen von 1 h bis 2 h. Beim Vergleich der getesteten Mehrfachkonzentrationen beobachten wir nur statistisch signifikante mittlere Unterschiede beim Vergleich von 250 nM mit allen anderen Konzentrationen, was darauf hindeutet, dass innerhalb der Gruppen, mit Ausnahme von 250 nM, vergleichbare Niveaus der Phosphataseaktivität zu erwarten sind. Obwohl wir einen Unterschied von 20 % beobachteten, wenn die Proben bei 4 h und 1.000 nM SHP-2 behandelt wurden (22,1 % ± 5,5 %) im Vergleich zu den optimalen Bedingungen (t-Test, p < 0,05), entschieden wir uns, diese Kombination aufgrund der reduzierten Oberflächenexpression und der verminderten Hefegesundheit durch die lange Inkubation mit DTT nicht weiter zu verfolgen. Wir vermuten, dass dies durch die reduzierenden Bedingungen des Arbeitspuffers verursacht wird, der für die ordnungsgemäße Funktion der SHP-2-Phosphatase benötigt wird.

Abbildung 1: Durchflusszytometrische Analyse des Modellsystems. Dichtediagramme mit Vorwärtsstreuung (X-Achse) im Vergleich zu Seitenstreuung (Y-Achse) (links) und Punktdiagramme, die die Oberflächenexpression durch die C-terminale Epitop-Tag-Markierung des Substrats (X-Achse) im Vergleich zur Substratphosphorylierung (Y-Achse) (Mitte) der zytoplasmatischen CD28-Domäne anzeigen, die 2 h lang (A) ohne SHP-2 und (B) mit der optimalen Konzentration von SHP-2 definiert als 1.000 nM inkubiert wurde. Der Y-Median wurde innerhalb der definierten Gates, die oberflächenexprimierte Zellen (grün) umfassen, nur als relative Phosphorylierungsmessung gemessen. Nicht anzeigende Signalereignisse wurden gegated, um die Hintergrund-Y-Median-Messung (grau) zu definieren. (C) Punktdiagramm-Überlagerung von Proben, die einen Unterschied im Y-Median zeigen. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
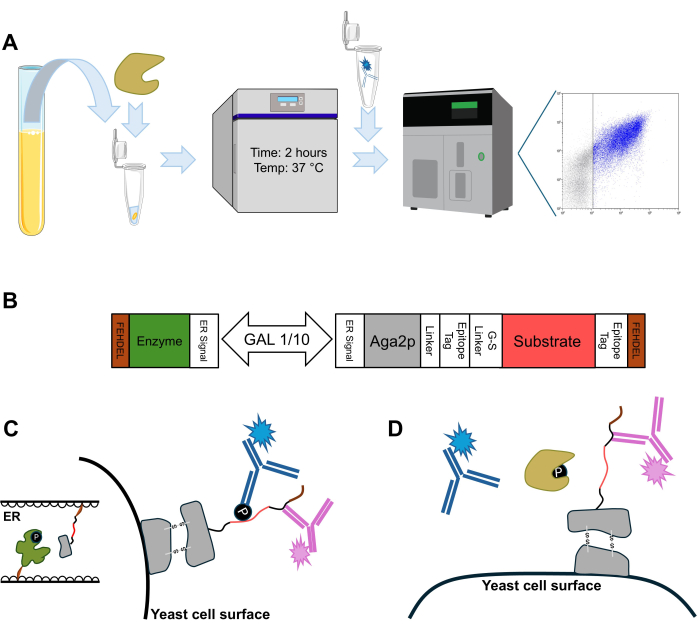
Abbildung 2: Enzymatische Modifikation von Proteinen, die auf der Hefeoberfläche angezeigt werden. (A) Assay-Schema, das die kritischen Schritte innerhalb der vier beschriebenen Methoden zeigt: Vorbereitung von Proben mit Phosphatase (tan) für den enzymatischen Assay, Inkubation für die gewünschte enzymatische Aktivität, Zellwaschung und Markierung zum Nachweis der Aktivität sowie Durchflusszytometrie-Analyse und Datenerfassung. (B) Genschema, das eine allgemeine Struktur der Kassette darstellt, die für die Sequestrierung eines Enzym-Substrat-Paares im endoplasmatischen Retikulum und die Oberflächenexpression des Substrats verwendet wird. (C) Grafische Darstellung der Kolokalisation eines endoplasmatischen Retikulums eines Kinase-Substrat-Paares (links), gefolgt von der Sekretion des posttranslational modifizierten Substrats, das auf der Hefeoberfläche angezeigt und mit einem Anti-Phosphotyrosin-Antikörper (blau) und einem Anti-Epitop-Tag-Antikörper (rosa) markiert wurde, um die Oberflächenexpression zu bestätigen (rechts). (D) Die Inkubation von Hefezellen mit Phosphatase (tan) entfernt die Phosphatgruppe aus dem angezeigten Substrat, stört die Markierung von Anti-Phosphotyrosin-Antikörpern und erleichtert die enzymatische Modifikationsanalyse durch Durchflusszytometrie. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
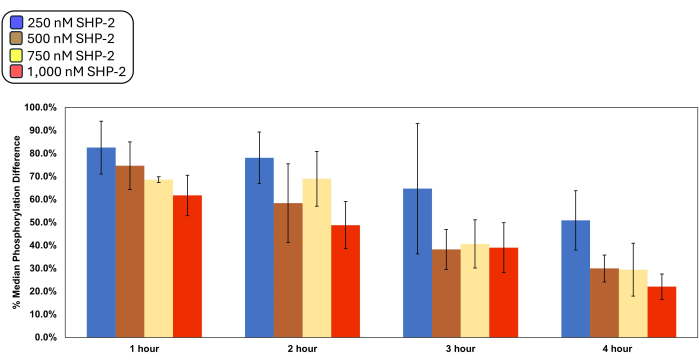
Abbildung 3: Phosphataseaktivität und Zeittitration. Hefezellen wurden mehreren Kombinationen von Zeit und Phosphatasekonzentration ausgesetzt, gefolgt von einer durchflusszytometrischen Analyse. Alle behandelten Proben wurden mit einer Kontrolle verglichen, die für die gleiche Zeitdauer und Pufferbedingungen ohne SHP-2 inkubiert wurde. Die prozentuale mediane Phosphorylierungsdifferenz wurde definiert als das Verhältnis des Y-Medians aus den an der Oberfläche angezeigten Ereignissen abzüglich des Ausgangsrauschsignals, das durch den Y-Median bei nicht anzeigenden Ereignissen in SHP-2-haltigen Proben geliefert wird, dividiert durch die gleiche Beziehung in ihrer jeweiligen Kontrolle. Die Nullhypothese wird verworfen, wenn signifikante Unterschiede beim Vergleich von Änderungen zwischen Zeit- und Konzentrationsgruppen unter Verwendung der bidirektionalen ANOVA beobachtet werden (p < 0,05). Der Effekt der beschriebenen Variablen über die prozentuale mediane Phosphorylierungsdifferenz ist unabhängig voneinander (Wechselwirkung p > 0,05). Der HSD-Test nach Tukey wurde für die Post-hoc-Analyse durchgeführt, um weitere Informationen über die Signifikanz der Differenz über die Gesamtinkubationszeit und die Konzentrationsgruppen zu erhalten, und eine Reihe von t-Tests, die von einer ungleichen Varianz ausgehen, wurden verwendet, um die statistische Signifikanz einzelner Gruppen zu einem bestimmten Zeitpunkt und einer bestimmten Konzentration zu definieren. Die Daten werden als Mittelwert ± Standardabweichung von vier Replikaten dargestellt. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
Tabelle 1: Richtlinien für das Hefewachstum und die Zubereitung von Proteininduktionsmedien. Tabellarische Beschreibung der Masse, die von jeder chemischen Komponente benötigt wird, um 1 l selektives Hefe-Wachstumsmedium, selektives Hefeprotein-Induktionsmedium und Hefeextrakt-Pepton-Dextrose-Medium zu formulieren. Sobald das beschriebene Medium richtig gemischt wurde, vor seiner Verwendung mit dem Filter sterilisieren. Zusätzliche Anweisungen sind für die Herstellung von selektiven Hefewachstumsmedienplatten enthalten. Bitte klicken Sie hier, um diese Tabelle herunterzuladen.
Tabelle 2: Empfohlene Probenvorbereitung für die Substratdephosphorylierung und Antikörpermarkierungsstrategie. Tabellarische Beschreibung der Proben, die für die Messung enzymatischer Modifikationen von phosphorylierten Substraten auf der Oberfläche der Hefe benötigt werden. Die Probenvorbereitung und die anschließende Antikörpermarkierungsstrategie werden sowohl für die erforderlichen Kontrollen als auch für jede zu analysierende Probe spezifiziert, einschließlich entsprechender Verdünnungen von Markierungsreagenzien. Bitte klicken Sie hier, um diese Tabelle herunterzuladen.
Tabelle 3: Berechnungen des Antikörper-Protein-Überschussverhältnisses. Tabellarische Beschreibung der theoretischen Anzahl der verfügbaren Antikörper pro Protein, die auf der Oberfläche einer Hefezelle exprimiert werden, während sie für die Durchflusszytometrie markiert werden. Die ausgedrückte theoretische Zahl geht davon aus, dass 100 % der Hefezellen 10.000 Proteine auf ihrer Oberfläche exprimieren, um einen Antikörperüberschuss zu gewährleisten, und die Berechnungen des Überschussverhältnisses basieren auf der in der Tabelle angezeigten und vom Anbieter erworbenen Reagenzienbestandskonzentration. Bitte klicken Sie hier, um diese Tabelle herunterzuladen.
Ergänzende Tabelle S1: Aminosäuresequenz von Konstruktkassetten. Tabellarische Darstellung der Aminosäuresequenz für die Konstruktkassetten, die sich auf beiden Seiten des Gal 1-10-Promotors befinden. Hervorgehobene Sequenzen entsprechen ihren farbcodierten Beschreibungen. Die Gal-10-Seite des Plasmids wird als umgekehrte Aminosäuresequenztranslation dargestellt, um das Verständnis zu erleichtern. Alle schwarz belassenen Zeichen entsprechen der Aminosäuretranslation von Restriktionsenzymverdaustellen, die verwendet werden, um dem Konstrukt Modularität zu verleihen. Bitte klicken Sie hier, um diese Datei herunterzuladen.
Diskussion
Das vorgestellte Protokoll ermöglicht die Analyse enzymatischer Wechselwirkungen unter Verwendung der extrazellulären Darstellung von Proteinen auf der Hefeoberfläche. Die Einbeziehung der Sequestrierung des endoplasmatischen Retikulums in das verwendete Oberflächen-Display-Plasmid ermöglicht es, spezifische Wechselwirkungen zwischen Enzymen und posttranslational modifizierten Substraten extrazellulär zu analysieren, da intrazelluläre Wechselwirkungen so gestaltet werden können, dass sie auftreten können27,29. Die zuvor etablierten enzymatischen Interaktionsassays unter Verwendung der Hefeoberflächenanzeige beinhalten die intrazelluläre Expression der interessierenden Proteine, wobei die Oberflächenanzeige lediglich ein Werkzeug zum Nachweis intrazellulärer Wechselwirkungen ist, die zwischen den Zielproteinen auftreten 23,25,29.
Dieses Protokoll baut auf dieser Plattform auf, indem es die gezielten enzymatischen Wechselwirkungen in die extrazelluläre Umgebung verlagert, was zusätzliche Flexibilität sowohl bei den Enzymen, die untersucht werden können, als auch bei der Umgebung, in der ihre Aktivität überwacht wird, bietet. Die untersuchten Wechselwirkungen, die extrazellulär stattfinden, geben den Forschern die Möglichkeit, die Inkubationsumgebung so anzupassen, dass sie für die enzymatische Aktivität optimaler ist, und die zu untersuchenden Enzyme erweitern, deren Aktivitäten im endoplasmatischen Retikulum der Hefe, einer stark oxidierenden Umgebung, behindert werden37. Darüber hinaus ermöglicht die Fähigkeit, die Konzentration von Enzymen in Bezug auf ein gegebenes Substrat zu titrieren, spezifische enzymatische Aktivitätsassays, die aufgrund der hypothetischen Reaktionsgeschwindigkeitssättigung während der Sequestrierung nicht intrazellulär durchgeführt werden konnten.
Innerhalb des Protokolls gibt es mehrere kritische Schritte, die zu beachten sind, um sicherzustellen, dass das gewünschte Ergebnis eingehalten wird. Die erfolgreiche Umwandlung der untersuchten Konstrukte in Hefe ist essentiell für optimale Ergebnisse aus den folgenden Schritten. Eine genaue Überwachung der optischen Dichte sollte durchgeführt werden, um das gesunde Wachstum der Kulturen zu verfolgen und sicherzustellen, dass sie vor der Proteininduktion oder Probenvorbereitung nicht überwachsen. Die logarithmische Phase des Wachstums von Hefezellen umfasst den Zeitraum, in dem die Proteinproduktion am höchsten ist, während in der stationären Phase die für die Proteinproduktion verantwortlichen Mechanismen blockiert werden38. Vor diesem Hintergrund ermöglichen optische Dichtemessungen eine genaue Messung der Wachstumsphasen, in denen sich die Hefekulturen befinden, und Schritte wie die Proteininduktion und die Vorbereitung für Assays sollten außerhalb der stationären Phase oder bei Überwucherung der Kulturen durchgeführt werden (OD600 nm < 6).
Für den enzymatischen Modifikationsassay war die beschriebene Inkubationsumgebung spezifisch für das untersuchte Enzym SHP-2 und die durchgeführte enzymatische Aktivität, die Dephosphorylierung. DTT wurde für die reduzierende Umgebung verwendet, die es während der Inkubation mit SHP-234 bietet. Daher ist es wichtig, genaue Konzentrationen der Chemikalien zu messen, die zur Modifizierung der Inkubationsumgebung in den enzymatischen Assays verwendet werden, um eine konsistente enzymatische Aktivität zwischen Proben und Experimenten zu gewährleisten. SHP-2 wurde als rekombinantes Protein verwendet, und es ist entscheidend, die Temperatur während der verschiedenen Schritte des Umgangs mit dem Enzym zu regulieren. Für einen erfolgreichen Assay sollte das Enzym nicht mehr als zwei Gefrier-Auftau-Zyklen durchlaufen haben und sich während der Vorbereitung jeder Probe auf Eis befinden. Es ist dann zwingend erforderlich, das rekombinante Enzym in ein ausreichendes Volumen zu aliquotieren, um die Anforderungen des Assays zu erfüllen. Während der eigentlichen Inkubation muss die Temperatur streng auf die optimale Temperatur für das Enzym kontrolliert werden, in diesem Fall 37 °C, mit ständiger Bewegung von einem Rotor, um die Homogenität der Inkubationsmischung zu gewährleisten.
Die Gesamtmethode für die Analyse mit rekombinanten Enzymen erforderte Modifikationen, die spezifisch für die Wechselwirkung zwischen SHP-2 und dem oberflächendargestellten phosphorylierten Substrat sind. Die Anpassung des Protokolls an andere extrazelluläre Enzym-Substrat-Wechselwirkungen beinhaltet die Modifikation der verwendeten Sequenzen, der Aktivitätspufferumgebung und des für den Nachweis verwendeten Reagenzes. Für die Untersuchung anderer Kinase-Substrat-Phosphatase-Wechselwirkungen beinhaltet die Adaption das Ersetzen der Proteinsequenzen für ein Kinase-Substrat-Paar an ihren jeweiligen Positionen in der Konstruktkassette, die in der ergänzenden Tabelle S1 beschrieben ist. Die Proteinsequenz des Substrats sollte zusammen mit mindestens der Kinasedomäne der interessierenden Kinase in das Plasmid eingeschlossen sein, und die Phosphatase, die auf das hergestellte phosphorylierte Substrat abzielt, sollte in Form eines rekombinanten Proteins vorliegen. Die repräsentative Wechselwirkung zwischen LCK, CD28 und SHP-2 ist ein Beispiel für die Verwendung der designten endoplasmatischen Retikulum-Sequestrierung in der Konstruktkassette als Werkzeug zur Herstellung posttranslational modifizierter Proteine, die extrazellulär mit ihrem Zielenzym untersucht werden sollen. Substrate von Interesse, die keine posttranslationalen Modifikationen durchlaufen müssen (z. B. Substrate, die extrazellulär mit zugesetzter Kinase phosphoryliert werden könnten), können auf der Hefeoberfläche exprimiert werden, ohne dass ein gepaartes Enzym innerhalb der Konstruktkassette vorhanden ist. In diesem Fall würde die Proteinsequenz für die Kinase, die in der Ergänzungstabelle S1 beschrieben ist, entfernt werden, wobei nur die Sequenz des Substrats in die Konstruktkassette aufgenommen wird. Wir stellen aus unseren früheren Erfahrungen fest, dass die Co-Lokalisierung einer Serin-Threonin-Kinase mit einem bekannten Substrat zu einem Substrat führte, das nicht phosphoryliert war (Ezagui und Stern, unveröffentlichte Daten), so dass vor der Anwendung der extrazellulären Phosphatase ein rigoroser Test der erfolgreichen enzymatischen Modifikation durchgeführt werden muss. Wir haben bereits ein Protokoll für die Kinase-Substrat-Co-Lokalisierung veröffentlicht, das für diesen qualifizierenden Schritt hilfreich sein kann39.
Kinasen und Phosphatasen enthalten häufig ungepaarte Cysteinreste, die bei Oxidation Disulfidbindungen innerhalb des Proteins oder zwischen Proteinen bilden, die die katalytische Aktivität des Proteins aufgrund von Konformationsänderungen stören können40,41. Das Verständnis dieser Proteinbiochemie ist essentiell für die Bestimmung der richtigen Reaktionsumgebung für die enzymatische Modifikation. Infolgedessen muss der Inkubationsumgebung ein Reduktionsmittel zugeführt werden, um sicherzustellen, dass das verwendete rekombinante Protein aktiv bleibt. DTT ist ein gängiges Reduktionsmittel, das für diese Zwecke verwendet wird, aber die Konzentration im Assay muss optimiert werden. Die Verwendung einer zu hohen DVB-T-Konzentration behindert die Darstellung des Substrats auf der Hefeoberfläche, da die Aga1p- und Aga2p-Anker durch Disulfidbindungen aneinander gehalten werden, die in Gegenwart von DTT42 reduziert werden. Die Konzentration von DTT wurde auf die minimale Konzentration eingestellt, die eine relative maximale Phosphataseaktivität ermöglicht, ohne nachteilige Auswirkungen auf die Oberflächendarstellung der Substratezu haben 42. Die Inkubationsumgebung für das zu testende Enzym sollte optimiert werden, um die Beibehaltung der enzymatischen Aktivität bei Verwendung in diesem Assay zu gewährleisten. Wenn ein Enzym eine reduzierende Umgebung benötigt, die erheblich stärker ist als die in diesem Assay verwendeten 0,5 mM DTT, ist die Plattform durch die Verringerung der Oberflächendarstellung begrenzt und möglicherweise nicht optimal für den gewünschten spezifischen enzymatischen Assay. In ähnlicher Weise wurde der während des Inkubationsschritts in diesem Protokoll verwendete 2x-Puffer aus früheren Forschungen zu akzeptablen Puffern zur Förderung der SHP-2-Aktivität eingeschlossen, und ähnliche Untersuchungen sollten zur Formulierung eines Inkubationspuffers für jedes andere Enzym durchgeführt werden, das für diesen Assay verwendet wird34. Ausgangspunkte für die Herstellung dieser Puffer können die Literaturrecherche nach erfolgreichen In-vitro-Anwendungen des interessierenden Enzyms oder die Empfehlung des Enzymherstellers für einen Aktivitätspuffer sein. Das verwendete rekombinante Enzym sollte speziell für diesen Assay titriert werden, um eine akzeptable Konzentration und Inkubationszeit zu ermitteln, die es ermöglicht, dass die angestrebte enzymatische Aktivität vor der Datenerfassung auftritt.
Für die Anpassung dieses Protokolls an andere Arten von Enzym-Substrat-Wechselwirkungen müssen neue Fluoreszenzdetektionsreagenzien ausgewählt und hinsichtlich der Sensitivität titriert werden. Andere Studien haben Beispiele dafür gezeigt, darunter die Verwendung von Antikörpern, die auf Epitop-Tags abzielen, um das Vorhandensein oder Fehlen von Peptidsubstraten nach einer Proteasebehandlungnachzuweisen23,25 und acetylierungsempfindliche Antikörper zum Nachweis von Modifikationen an Histonproteinen43. Zur Qualifizierung dieser Reagenzien müssen eine Positivkontrolle (eine Kontrolle, die die Änderung des Interesses nachweislich nachweist) und eine Negativkontrolle (eine Kontrolle, die nachweislich das Fehlen der Änderung des Interesses nachweist) eingerichtet werden. Dies könnte durch die Darstellung der Hefeoberfläche eines Konstrukts erfolgen, das zuvor die interessierende Modifikation gezeigt hat, oder in einigen Fällen könnte es durch Immobilisierung von rekombinanten Proteinen oder Peptiden etabliert werden. Zum Beispiel könnten im Falle der Phosphorylierung (und vieler anderer posttranslationaler Modifikationen von Interesse) Peptide bekannter Sequenz entweder mit Phosphotyrosin (Positivkontrolle) oder unmodifiziertem Tyrosin (Negativkontrolle) und einem C-terminalen Biotin synthetisiert werden, das eine Immobilisierung der Peptide auf Streptavidin-beschichteten Kügelchen ermöglichen würde. Die peptidbeschichteten Kügelchen könnten mit dem modifikationsspezifischen Antikörper markiert und mittels Durchflusszytometrie mit ähnlichen Methoden wie den in Abschnitt 4 beschriebenen auf Spezifität und Sensitivität des Nachweises untersucht werden. Unterschiedliche Verdünnungen von Antikörpern sollten verwendet werden, um eine Konzentration zu finden, die ein maximales Signal der Positivkontrolle, ein minimales Signal der Negativkontrolle liefert und ein ausreichendes Faltungsüberschuss des Antikörpers pro modifiziertem Protein (unter Verwendung der Gleichung in der Anmerkung aus Schritt 4.1) mit wirtschaftlichen Überlegungen für die Anzahl der durchzuführenden Experimente pro Aliquot des erhaltenen Antikörpers ausgleicht.
Wir beschreiben ein Protokoll zur Anpassung der Leichtigkeit der Hefeoberflächenanzeigeplattform für extrazelluläre enzymatische Aktivitätsassays. Das Verfahren wird unter Verwendung von phosphoryliertem CD28 demonstriert, das auf der Hefeoberfläche angezeigt wird, um während der Inkubation durch rekombinantes SHP-2 dephosphoryliert zu werden, ist aber für viele Arten der enzymatischen Modifikation durch die Modifikation des verwendeten Arbeitspuffers und des verwendeten Enzym-Substrat-Paares verallgemeinerbar.
Offenlegungen
Die Autoren haben keine Interessenkonflikte im Zusammenhang mit dieser Arbeit offenzulegen.
Danksagungen
Diese Arbeit wurde durch einen NSF CAREER Award an L.A.S. (CBET - 2339172) und Startup-Fonds der University of South Florida unterstützt.
In Abbildung 2A ist das microtube-open-translucent icon von Servier https://smart.servier.com/ unter CC-BY 3.0 Unported https://creativecommons.org/licenses/by/3.0/ lizenziert. Zu den Modifikationen gehören die Zugabe von Puffer und einer Hefezelle (links) sowie die Zugabe von Antikörpern (Mitte-rechts).
Das Reagenzglas, der Inkubator und das Durchflusszytometer in Abbildung 2A wurden über www.bioicons.com unter freiem Zugang zur Verfügung gestellt.
Materialien
| Name | Company | Catalog Number | Comments |
| 1 L Media Bottles | Corning | 06-414-1D | |
| 1.7/2.0 mL Microtubes | Axygen | MCT-175-C | |
| 10 µL SureOne Pipet Tips | Fisher Scientific | 02-707-438 | |
| 1000 µL SureOne Pipet Tips | Fisher Scientific | 02-707-408 | |
| 12 mL Polystyrene Round-Bottom Tubes | Greiner | 07-000-212 | |
| 3 mL platic Cuvettes | BRAND | 759076D | |
| 300 µL SureOne Pipet Tips | Fisher Scientific | 02-707-411 | |
| 5 mL Serological Pipettes | Fisher Scientific | 13-678-11D | |
| Acid Casein (Casamino Acids) | Fisher Scientific | BP-1424-500 | |
| Analytical Balance | Mettler Toledo | 30243397 | |
| Bacteriological Petri Dish | Corning | Falcon 351008 | |
| Biosafety Cabinets | Labconco | Logic Class II, Type A2 302310102 | |
| Biospectrometer | Eppendorf | Kinetic 6136000010 | |
| Bovine Serum Albumin | Fisher bioreagents | BP1600-100 | |
| Citric Acid | Fisher Scientific | A940-500 | |
| CytoFLEX Flow Cytometry Analyzer | Beckam Coulter | Cytoflex C09745 | CytExpert software |
| Dextrose | Fisher Scientific | D16-1 | |
| Dithiothreitol | Fisher bioreagents | BP172-5 | |
| Donkey anti-goat FITC | Invitrogen | A16000 | |
| EDTA | Alfa Aesar | H56165.30 | |
| Ez-Link PEG4-NHS-Biotin | Thermo Scientific | A39259 | |
| Frozen-EZ Yeast Transformation II Kit | Zymo Research | T2001 | |
| Galactose | Fisher Scientific | BP656-500 | |
| General Purpose Refrigerator | Marvel Scientific | MS24RAS4RW | |
| Goat anti-myc tag antibody | Bethyl | A190-104A | |
| Mictrotube Centrifuge | Eppendorf | 5425 R 5406000313 | |
| Mini Low Temperature Refrigerated Incubator | Fisher Scientific | 15-015-2632 | |
| Mouse anti-phosphotyrosine antibody 4G10 | BioXcell | BE0194 | |
| Parafilm M | Bemis | M PM999 | |
| Phosphate Buffered Saline | Fisher bioreagents | BP399-500 | |
| Pipette Controller | Eppendorf | easypet 3 4430000018 | |
| Raffinose | Thermo Scientific | J21060-36 | |
| Recombinant human Active SHP-2 Protein | R&D Systems | 1894-SH | |
| Refrigerated Centrifuge | Eppendorf | 5910 R | |
| Saccharomyces cerevisiae yeast surface display strain EBY 100 | ATCC | MYA-4941 | |
| Shaker Incubator | Eppendorf | M1335-0002 New Brunswick Innova 42 | |
| Single Channel Pipette Set | Eppendorf | 05-403-151 | |
| Sodium Chloride | Fisher Scientific | S671-500 | |
| Sodium Citrate Dihydrate | Fisher Scientific | S279-500 | |
| Sodium Phosphate Dibasic Heptahydrate | Fisher Scientific | S373-500 | |
| Sodium Phosphate Monobasic Monohydrate | Fisher Scientific | S468-500 | |
| Streptavidin Alexa Fluor 647 | Invitrogen | S32357 | |
| Top Loading Balance | Mettler Toledo | ||
| Tris hydrochloride | EMD Millipore | 648317-100GM | |
| Tube revolver rotator | Fisher Scientific | 11-676-341 | |
| Weighing Paper | Fisher Scientific | 09-898-12B | |
| Yeast Nitrogen Base | BD Difco | 291940 | |
| Zeba Spin Desalting Columns | Thermo Scientific | 89883 |
Referenzen
- Lea, M. A., Weber, G. Role of enzymes in homeostasis: VIII. Inhibition of the activity of glycolytic enzymes by free fatty acids. J Biol Chem. 243 (6), 1096-1102 (1968).
- Mahé, M., Rios-Fuller, T. J., Karolin, A., Schneider, R. J. Genetics of enzymatic dysfunctions in metabolic disorders and cancer. Front Oncol. 13, 1230934(2023).
- Fernandez-de-Cossio-Diaz, J., Vazquez, A. A physical model of cell metabolism. Sci Rep. 8 (1), 8349(2018).
- Metallo, C. M., Vander Heiden, M. G. Understanding metabolic regulation and its influence on cell physiology. Mol Cell. 49 (3), 388-398 (2013).
- Mildvan, A. S. Mechanisms of signaling and related enzymes. Proteins. 29 (4), 401-416 (1997).
- Frosina, G. Overexpression of enzymes that repair endogenous damage to DNA. Eur J Biochem. 267 (8), 2135-2149 (2000).
- Schärer, O. D. Chemistry and biology of DNA repair. Angew Chem Int Ed. 42 (26), 2946-2974 (2003).
- de la Fuente, M., et al. Enzyme therapy: Current challenges and future perspectives. Int J Mol Sci. 22 (17), 9181(2021).
- Robertson, J. G. Enzymes as a special class of therapeutic target: clinical drugs and modes of action. Curr Opin Struct Biol. 17 (6), 674-679 (2007).
- Goddard, J. -P., Reymond, J. -L. Enzyme assays for high-throughput screening. Curr Opin Biotechnol. 15 (4), 314-322 (2004).
- Helm, J. S., Hu, Y., Chen, L., Gross, B., Walker, S. Identification of active-site inhibitors of MurG using a generalizable, high-throughput glycosyltransferase screen. J Am Chem Soc. 125 (37), 11168-11169 (2003).
- Veldhuyzen, W. F., Nguyen, Q., McMaster, G., Lawrence, D. S. A light-activated probe of intracellular protein kinase activity. J Am Chem Soc. 125 (44), 13358-13359 (2003).
- Torres, M., Forman, H. J. Encyclopedia of Respiratory. Laurent, G. J., Shapiro, S. D. , Academic Press. 10-18 (2006).
- Blume-Jensen, P., Hunter, T. Oncogenic kinase signalling. Nature. 411 (6835), 355-365 (2001).
- Martin, G. S. Cell signaling and cancer. Cancer Cell. 4 (3), 167-174 (2003).
- Lothrop, A. P., Torres, M. P., Fuchs, S. M. Deciphering post-translational modification codes. FEBS Lett. 587 (8), 1247-1257 (2013).
- Graves, J. D., Krebs, E. G. Protein phosphorylation and signal transduction. Pharmacol Ther. 82 (2), 111-121 (1999).
- Hafen, E. Kinases and phosphatases--A marriage is consummated. Science. 280 (5367), 1212-1213 (1998).
- Westphal, R. S., Anderson, K. A., Means, A. R., Wadzinski, B. E. A signaling complex of Ca2+-calmodulin-dependent protein kinase IV and protein phosphatase 2A. Science. 280 (5367), 1258-1261 (1998).
- Barford, D., Das, A. K., Egloff, M. -P. The structure and mechanism of protein phosphatases: Insights into catalysis and regulation. Annu Rev Biophys Biomol Struct. 27, 133-164 (1998).
- Liu, Q., Qu, J., Zhao, M., Xu, Q., Sun, Y. Targeting SHP2 as a promising strategy for cancer immunotherapy. Pharmacol Res. 152, 104595(2020).
- Pan, J., Zhou, L., Zhang, C., Xu, Q., Sun, Y. Targeting protein phosphatases for the treatment of inflammation-related diseases: From signaling to therapy. Signal Transduct Targeted Ther. 7 (1), 177(2022).
- Denard, C. A., et al. YESS 2.0, a tunable platform for enzyme evolution, yields highly active TEV protease variants. ACS Synth Biol. 10 (1), 63-71 (2021).
- Lim, S., Glasgow, J. E., Filsinger Interrante, M., Storm, E. M., Cochran, J. R. Dual display of proteins on the yeast cell surface simplifies quantification of binding interactions and enzymatic bioconjugation reactions. Biotechnol J. 12 (5), (2017).
- Yi, L., et al. Engineering of TEV protease variants by yeast ER sequestration screening (YESS) of combinatorial libraries. Proc Natl Acad Sci USA. 110 (18), 7229-7234 (2013).
- Yi, L., et al. Yeast endoplasmic reticulum sequestration screening for the engineering of proteases from libraries expressed in yeast. Methods Mol Biol. 1319, 81-93 (2015).
- Semenza, J. C., Hardwick, K. G., Dean, N., Pelham, H. R. ERD2, a yeast gene required for the receptor-mediated retrieval of luminal ER proteins from the secretory pathway. Cell. 61 (7), 1349-1357 (1990).
- Mei, M., et al. Characterization of aromatic residue-controlled protein retention in the endoplasmic reticulum of Saccharomyces cerevisiae. J Biol Chem. 292 (50), 20707-20719 (2017).
- Ezagui, J., Russell, B., Mairena, Y., Stern, L. A. Endoplasmic reticulum sequestration empowers phosphorylation profiling on the yeast surface. AIChE J. 68 (12), e17931(2022).
- Kawai, S., Murata, K. Genetic Transformation Systems in Fungi. van den Berg, M. A., Maruthachalam, K. 1, Springer International Publishing. 187-192 (2015).
- Kawai, S., Hashimoto, W., Murata, K. Transformation of Saccharomyces cerevisiae and other fungi: methods and possible underlying mechanism. Bioeng Bugs. 1 (6), 395-403 (2010).
- Loock, M., et al. High-efficiency transformation and expression of genomic libraries in yeast. Methods Protoc. 6 (5), 89(2023).
- Huang, D., Gore, P. R., Shusta, E. V. Increasing yeast secretion of heterologous proteins by regulating expression rates and post-secretory loss. Biotechnol Bioeng. 101 (6), 1264-1275 (2008).
- Yu, B., et al. Targeting protein tyrosine phosphatase SHP2 for the treatment of PTPN11-associated malignancies. Mol Cancer Ther. 12 (9), 1738-1748 (2013).
- Stern, L. A., et al. Geometry and expression enhance enrichment of functional yeast-displayed ligands via cell panning. Biotechnol Bioeng. 113 (11), 2328-2341 (2016).
- Pan, X., et al. Optimized single-cell gates for yeast display screening. Protein Eng Design Sel. 38, gzae018(2025).
- Margittai, É, et al. Production of H2O2 in the endoplasmic reticulum promotes in vivo disulfide bond formation. Antioxid Redox Signal. 16 (10), 1088-1099 (2012).
- Werner-Washburne, M., Braun, E., Johnston, G. C., Singer, R. A. Stationary phase in the yeast Saccharomyces cerevisiae. Microbiol Rev. 57 (2), 383-401 (1993).
- Ezagui, J., Stern, L. A. Tyrosine phosphorylation screening on the yeast surface by magnetic bead selection and FACS. Methods Mol Biol. 2681, 275-290 (2023).
- Yarnall, M. T. N., Kim, S. H., Korntner, S., Bishop, A. C. Destabilization of the SHP2 and SHP1 protein tyrosine phosphatase domains by a non-conserved “backdoor” cysteine. Biochem Biophys Rep. 32, 101370(2022).
- Dustin, C. M., Heppner, D. E., Lin, M. J., van der Vliet, A. Redox regulation of tyrosine kinase signalling: more than meets the eye. J Biochem. 167 (2), 151-163 (2020).
- Stern, L. A., Csizmar, C. M., Woldring, D. R., Wagner, C. R., Hackel, B. J. Titratable avidity reduction enhances affinity discrimination in mammalian cellular selections of yeast-displayed ligands. ACS Comb Sci. 19 (5), 315-323 (2017).
- Waldman, A. C., Rao, B. M., Keung, A. J. Mapping the residue specificities of epigenome enzymes by yeast surface display. Cell Chem Biol. 28 (12), 1772-1779.e4 (2021).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenWeitere Artikel entdecken
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten