Method Article
Modification enzymatique et évaluation par cytométrie en flux des protéines affichées à la surface de la levure
* Ces auteurs ont contribué à parts égales
Dans cet article
Résumé
Ici, nous introduisons un protocole pour l’utilisation de substrats affichés à la surface de levure pour des tests de modification enzymatique. La plateforme a été démontrée en utilisant l’analyse de l’activité de déphosphorylation de la tyrosine phosphatase SHP-2 contre l’un de ses substrats en tant que test de modification enzymatique représentatif.
Résumé
L’affichage de la surface de la levure est une stratégie de liaison génotype-phénotype qui permet un criblage à haut débit de la fonction protéique. Traditionnellement, l’affichage de la surface de la levure a été appliqué à l’évolution de nouvelles protéines de liaison, la cytométrie en flux étant utilisée pour évaluer et trier par niveaux de force de liaison. Récemment, on s’est intéressé de plus en plus à l’application de l’affichage de surface de levure pour le criblage de modifications enzymatiques de variantes de substrat, avec des modifications additives (par exemple, la phosphorylation) ou soustractives (par exemple, la protéolyse) fournissant un phénotype lisible par cytométrie en flux. De telles modifications sont régulièrement appliquées en utilisant la co-localisation intracellulaire, mais la capacité de réaliser une modification enzymatique extracellulaire des substrats affichés pourrait ouvrir beaucoup plus de réactions à l’étude. Ici, nous décrivons des techniques de conception et d’application de tests de criblage pour la modification enzymatique extracellulaire à des substrats candidats affichés à la surface de la levure et une évaluation ultérieure à l’aide d’une analyse par cytométrie en flux. Nous fournissons ces protocoles dans le contexte des phosphatases déphosphorylant des substrats de levure contenant des résidus de tyrosine phosphorylés et commentons comment ce cadre appliqué peut être adapté au développement de tests de criblage pour d’autres paires enzyme-substrat.
Introduction
La compréhension des interactions entre les enzymes et leurs cibles est devenue un domaine de recherche de plus en plus intéressant en raison de sa nécessité dans la caractérisation biologique des voies contrôlant l’homéostasie cellulaire et le développement de la maladie 1,2. Les enzymes sont responsables de la catalyse de nombreuses réactions qui maintiennent la vie biologique, contrôlant les voies nécessaires telles que le métabolisme cellulaire 3,4, la signalisation5 et même des processus fondamentaux comme la réparation du génome 6,7. En raison de leur rôle dans ces processus, leurs interactions jouent également un rôle dans le développement de nombreuses maladies, car des déviations dans leur activité peuvent provoquer une dérégulation sévère de l’activité cellulaire, provoquant l’apoptose ou la prolifération de cellules cancéreuses nocives2. L’étude de l’activité enzymatique a eu des applications importantes dans le développement de nouvelles thérapies 8,9, nécessitant des dosages adaptés à chaque interaction enzyme-substratspécifique 10. De multiples essais enzymatiques ont été établis comme protocoles standard pour l’évaluation et la caractérisation de ces interactions. Les essais mis au point pour analyser les interactions enzymatiques sont classés en essais de détection qui surveillent la liaison pour l’activation/inhibition11 ou en essais qui surveillent la modification du substrat par les enzymes12.
L’un des rôles majeurs des enzymes est la régulation du comportement cellulaire. La transduction du signal, c’est-à-dire la réponse intracellulaire d’une cellule à un déclencheur extracellulaire13, est responsable de la survie et de la fonctionnalité cellulaires. La prolifération cellulaire, la différenciation et de nombreux autres processus fonctionnels impliquent tous des voies de signalisation avec des interactions enzymatiques qui les régissent14,15. Les enzymes catalysent les modifications post-traductionnelles, qui modulent souvent les réseaux de signalisation massifs responsables de la transmission correcte des messages extracellulaires16. La phosphorylation des protéines est la modification post-traductionnelle la plus courante, omniprésente dans la signalisation cellulaire et de multiples autres voies cellulaires. Par conséquent, les protéines kinases sont apparues comme une proportion importante des cibles thérapeutiques potentielles en raison de leur rôle régulateur critique17. Les phosphatases sont les molécules modulatrices naturelles des complexes de signalisation cellulaire à base de phosphate18,19, ayant la capacité d’éliminer les résidus de phosphate de leurs protéines cibles20. Au cours de la dernière décennie, les phosphatases sont devenues une cible thérapeutique majeure pour le traitement du cancer21 et des maladies inflammatoires22 en raison de leur implication dans la régulation des voies de signalisation en aval dans plusieurs types de cellules. Ensemble, les protéines kinases et les phosphatases fournissent un large éventail d’interactions, qui peuvent être étudiées grâce au développement de protocoles de dosage enzymatique spécifiques.
L’affichage de la surface de la levure a été utilisé comme outil pour la caractérisation et l’évaluation de l’activité enzymatique23,24. Il fournit une plate-forme à haut débit pour le criblage des processus de modification post-traductionnelle lorsqu’il est combiné à des stratégies de séquestration du réticulum endoplasmique25,26. Cela permet aux paires kinase-substrat d’être co-localisées et conservées dans le réticulum endoplasmique en se liant aux récepteurs KDEL27, où la phosphorylation du substrat peut se produire à des taux accrus en raison de la proximité entre les kinases et leurs cibles. La liaison du récepteur KDEL est médiée par une séquence de rétention du réticulum endoplasmique Fehdel C-terminale qui s’est avérée avoir une capacité de rétention plus forte que les autres séquences HDEL25,28. Le substrat phosphorylé est ensuite ancré à la surface de la levure pour son évaluation ultérieure par cytométrie en flux29. À l’heure actuelle, il n’existe aucun protocole généralisable établi pour la modification enzymatique des substrats affichés à la surface de la levure. Nous élargissons les capacités d’affichage de la surface de la levure en tirant parti des variantes de substrat phosphorylées exprimées extracellulairement et en les modifiant par déphosphorylation par leur phosphatase connue. L’analyse par cytométrie en flux fournit ensuite une plate-forme pour l’évaluation phénotypique des substrats susmentionnés par la mesure des altérations de la médiane de phosphorylation à la suite de l’incubation avec la phosphatase connue. Il s’agit d’une méthode adaptable pour la modification post-traductionnelle des protéines affichées en surface, tout en fournissant une méthode pour l’analyse de la modification enzymatique des interactions lors de l’utilisation de la plate-forme d’affichage de surface de levure.
Nous présentons des techniques pour le développement et l’application d’un test de modification enzymatique qui décrit l’introduction d’une interaction kinase-substrat dans la plate-forme d’affichage de surface de la levure, la co-incubation du substrat phosphorylé exprimé avec une phosphatase recombinante, et l’analyse ultérieure de l’activité de déphosphorylation par cytométrie en flux. Dans ce rapport, cela est accompli en co-localisant le domaine cytoplasmique de CD28 avec la kinase lymphocytaire (LCK) dans le réticulum endoplasmique de la levure, suivi de l’affichage du CD28 phosphorylé à la surface de la levure et de la déphosphorylation ultérieure par la phosphatase-2 contenant le domaine d’homologie Src 2 (SHP-2). Un anticorps anti-phosphotyrosine (4G10 dans cette étude), qui détecte les résidus de tyrosine phosphorylés dans une grande variété de séquences peptidiques, est utilisé pour quantifier le niveau de phosphorylation en fonction du traitement à la phosphatase. Le processus détaillé fournit une approche généralisable pour étudier les interactions enzyme-substrat ; Une manière prospective d’étudier les enzymes et les substrats de manière purifiée.
Protocole
1. Croissance cellulaire de la levure hébergeant le plasmide et induction de l’expression des protéines
- En suivant la recette décrite dans le tableau 1, préparer les milieux nécessaires à la croissance des levures non plasmidiques (YPD), à la croissance des cellules de levure contenant des plasmides (SD-CAA) et à l’induction de l’expression des protéines (SRG-CAA) ainsi que les plaques SD-CAA.
- Transformez l’ADN plasmidique d’affichage de levure contenant la paire kinase-substrat en cellules de levure EBY-100 grâce à la méthode à base de cations lithium30,31, couramment adoptée pour une utilisation dans les kits de transformation de levure de divers fabricants.
REMARQUE : L’électroporation32 ou d’autres techniques de transformation de plasmide de levure préférées peuvent être utilisées en fonction de la construction du plasmide transformée. - Préparez un tube de culture de 14 mL avec 10 mL de milieu YPD. Inoculer les cellules EBY-100 et les faire croître dans un incubateur à 30 °C, 300 tr/min jusqu’à ce que la culture atteigne une densité optique (DO600 nm) de 0,8-1,0 (8 x 106- 1 x 107 cellules/mL).
REMARQUE : Le diamètre extérieurde 600 nm est mesuré en préparant des cuvettes d’échantillon de 3 mL contenant des dilutions 1:10 de cultures de levure dans leurs milieux respectifs et des cuvettes vierges de 3 mL contenant les milieux utilisés pour les dilutions des échantillons. Le programme OD600nm sur le spectrophotomètre permet de mesurer d’abord la cuvette à blanc, puis chaque cuvette d’échantillon en réglant la dilution correspondante préparée pour chaque échantillon. 1 DO600nm correspond à 1 ×10 7 levure/mL. - Récolter les cellules en centrifugeant la culture à 1 000 x g pendant 3 min, et laver avec la1ère solution de lavage fournie dans le kit de transformation de levure, ou TE (10 mM Tris-HCl et 1,0 mM EDTA)31.
- Granulez à nouveau les cellules à 1 000 x g pendant 3 min et mettez-les en suspension dans 1 mL du tampon de transformation fourni dans le kit de transformation de levure, ou de l’eau stérile. Les cellules doivent être aliquotes en aliquotes de 50 μL et peuvent être stockées à -80 °C jusqu’à 6 mois.
- Pour la transformation du plasmide, une aliquote par plasmide est préparée et décongelée sur de la glace, puis 0,5 à 1,5 μg d’ADN plasmidique contenant la construction d’affichage de la levure est ajouté directement aux cellules. On ajoute 0,5 mL de la solution de transformation fournie dans le kit de transformation de levure, ou 0,5 mL d’une solution stérile de polyéthylène glycol à 50 % et de solution de LiOAc31 à 0,1 M. Combinez soigneusement le mélange de cellules, d’ADN plasmidique et de solution de transformation par pipetage.
- Incuber le mélange de transformation de manière statique pendant 30 à 60 min à 30 °C, le mélange vortex à 15 minutes d’intervalle. Récoltez les cellules par centrifugation à 1 000 × g pendant 3 min.
- Préparez un tube de culture de 14 mL avec 4,5 mL de milieu SD-CAA. Remettre en suspension les cellules contenant le plasmide désiré dans 500 μL de SD-CAA et inoculer les 4,5 mL préparés.
- En prenant soin de ne pas percer la gélose, répartir 50 μL des 5 mL de culture inoculée sur une plaque SD-CAA et incuber statiquement à 30 °C pendant 48 h pour déterminer l’efficacité de la transformation.
- Incuber les 5 mL de culture cellulaire SD-CAA dans un incubateur à agitation à 30 °C, 300 tr/min pendant au moins 18 h. Surveiller la densité optique (OD600nm) après 16 h et 20 h. Une fois que l’échantillon a atteint une densité optique suffisante ne dépassant pas 6, centrifuger la culture pendant 3 min à 2 500 × g. Jetez le surnageant sans déranger la pastille de levure.
- Remettre en suspension la pastille de levure dans le SRG-CAA jusqu’à une DO finale de600 nm inférieure à 1 (<1 × 107 levure/mL).
- Incuber la culture de levure dans un incubateur à agitation à 30 °C, 300 tr/min pendant au moins 8 h mais pas plus de 24 h.
REMARQUE : L’induction de l’expression des protéines dans les cellules de levure peut varier n’importe où de 20 à 37 °C. 30 °C convient à la synthèse des paires kinase/substrat29,33 mais peut être ajustée si nécessaire pour les protéines spécifiques étudiées. - Mesurez la DO600 nm pour déterminer la densité cellulaire.
REMARQUE : Le protocole peut être arrêté à ce stade en stockant les cultures de levure à 4 °C.
2. Biotinylation de l’anticorps anti-phosphotyrosine 4G10
- Remettre en suspension un flacon de 2 mg de PEG4-NHS-Biotine à une concentration finale de 5 mM en ajoutant 680 μL de PBS stérile.
REMARQUE : La remise en suspension de PEG4-NHS-Biotine doit être effectuée immédiatement avant que la réaction de biotinylation ne soit effectuée. Le NHS s’hydrolyse en solution aqueuse. L’utilisation de PBS et de flacons stériles est importante pour la préparation des réactifs destinés à l’entreposage à long terme et pour l’utilisation dans les essais cellulaires afin d’atténuer les effets potentiels des contaminants sur la viabilité des réactifs ou les essais sensibles effectués. - En fonction de la concentration d’anticorps 4G10, ajouter 100 μg d’anticorps dans un flacon stérile de 1,7 mL.
- Ajouter 1 μL de 5 mM de PEG4-NHS-Biotine préparée à partir de l’étape 2.1 dans le flacon contenant les 100 μg d’anticorps 4G10 pour obtenir un rapport molaire biotine/anticorps de 7,5:1. Pipeter doucement le mélange pour homogénéiser la réaction.
- Incuber la réaction à température ambiante avec une rotation constante pendant au moins 2 h.
- Suivre le protocole du fabricant pour les colonnes de dessalage par centrifugation de 0,5 mL afin d’échanger le tampon du 4G10 biotinylé (B-4G10) dans du PBS.
REMARQUE : Les colonnes de dessalage avec une coupure de poids moléculaire (MWCO) de 7 kDa sont couramment utilisées pour la biotinylation des anticorps afin de permettre l’élimination de la biotine et d’autres petites molécules n’ayant pas réagi tout en conservant l’anticorps plus gros. - Diluer l’anticorps B-4G10 jusqu’à une concentration finale de 1 μM dans du PBSA (PBS avec 1 g/L d’albumine sérique bovine). Aliquote l’anticorps B-4G10 en plus petits volumes pour éviter les cycles répétés de congélation/décongélation.
REMARQUE : Les anticorps biotinylés peuvent être conservés à 4 °C pour une utilisation quotidienne jusqu’à 3 mois sans perte d’efficacité significative. Conservez le reste des aliquotes non utilisées à -20 °C pendant un maximum de 2 ans.
3. Déphosphorylation des substrats exprimés à la surface des cellules de levure
- Préparez la solution tampon 2x de travail comme décrit précédemment dans la littérature34.
REMARQUE : 2x est recommandé pour faciliter la mesure des ingrédients nécessaires. - Préparez le tampon de travail pour les échantillons dans un flacon de 1,7 mL en diluant la solution tampon 2x préparée à l’étape 3.1 1:2 dans de l’eau désionisée.
REMARQUE : Le volume total de réaction pour chaque échantillon sera de 20 μL, et chaque échantillon nécessitera entre 10 μL et 18 μL de tampon de travail. Préparez suffisamment de tampon de travail pour tous les échantillons ou commandes. - Après la préparation recommandée de l’échantillon décrite au tableau 2, étiqueter les flacons de 1,7 mL avec le témoin ou le nom de l’échantillon correspondant.
- Sur la base de la DOde 600 nm mesurée à l’étape 1.9, calculez le volume de culture nécessaire pour récupérer deux millions (2 × 106) de cellules de levure à partir de la culture de levure correspondante pour chaque échantillon.
- Ajouter le volume de culture de levure calculé à l’étape précédente dans le flacon de 1,7 mL pour chaque échantillon.
- Centrifugez le flacon pendant 1 min à 4 500 × g. Retirer délicatement le surnageant à l’aide d’une micropipette et le jeter comme déchet biologique.
- Remettre les cellules granulées en suspension dans 1 mL de PBSA et répéter l’étape 3.6.
REMARQUE : Il est important d’éliminer autant de surnageant que possible sans déranger les cellules granulées. - D’après sa concentration de stock, calculer le volume de SHP-2 humain recombinant requis pour avoir une concentration finale de 1 000 nM dans un volume de réaction total de 20 μL.
REMARQUE : Le SHP-2 recombinant doit être aliquote en petites quantités de sorte que toute protéine utilisée dans chaque essai n’ait pas subi plus de deux cycles de congélation-décongélation. Tout résidu de SHP-2 provenant d’une aliquote après que tous les échantillons ont été préparés pour un essai doit être jeté.
Les concentrations de stock d’enzymes recombinantes peuvent varier en fonction du numéro de lot. Le SHP-2 recombinant est couramment formulé à une concentration de base de 0,2 à 0,4 mg/mL. Pour une concentration mère de 0,324 mg/mL SHP-2, cela correspond à une concentration mère de 4,696 μM (SHP-2 a un poids moléculaire de 69 kDa). 4,26 μL de la matière première de SHP-2 dans une réaction de 20 μL donnent une concentration de réaction finale de 1 000 nM de SHP-2. - Ajouter 7,7 mg de DTT dans 10 mL d’eau déminéralisée préparée dans un cône de 15 mL pour créer une solution de DTT de 5 mM. Si les limites de l’équipement rendent difficile la pesée des milligrammes, ajoutez 0,77 g de DTT à 10 ml d’eau désionisée, puis effectuez une dilution 100x pour créer la solution de DTT de 5 mM utilisée pour le dosage.
REMARQUE : La solution DTT peut être préparée dans des solutions mères à concentration plus élevée pour être diluée à 5 mM s’il n’est pas possible de mesurer des quantités à l’échelle du milligramme à l’aide de l’équipement disponible. La solution de DTT doit être préparée fraîche avant la remise en suspension de la cellule dans le tampon de travail en raison de sa tendance à l’hydrolyse, ce qui la rend instable sur de longues périodes lorsqu’elle est diluée dans l’eau. - Remettre les cellules granulées en suspension dans le tampon de travail préparé à l’étape 3.2 de sorte que le volume de réaction final dans chaque échantillon ou témoin soit de 20 μL.
REMARQUE : La quantité de tampon de travail ajoutée doit être calculée en fonction de la quantité de DTT (2 μL) et de SHP-2 (calculée à l’étape 3.8) qui se trouvera dans chaque échantillon. - Ajouter 2 μL de la solution de DTT 5 mM préparée à l’étape 3.9 à chaque échantillon ou témoin pour une concentration de réaction finale de 0,5 mM de DTT.
- Ajouter le volume de SHP-2 calculé à l’étape 3.8 à chaque échantillon pour un volume final de 20 μL et mélanger délicatement à l’aide d’une micropipette.
- Enveloppez les couvercles des flacons d’échantillon dans un parafilm pour éviter les fuites ou la contamination croisée.
- Incuber les échantillons à 37 °C pendant 2 h sur un rotor à vitesse constante.
- Retirer les échantillons du rotor et arrêter la réaction en ajoutant 1 mL de PBSA à chaque échantillon.
- Répétez l’étape 3.6.
4. Marquage cellulaire et analyse par cytométrie en flux de substrats déphosphorylés
- Mettre en suspension les échantillons de l’étape 3.16 dans un mélange de 20 μL de leurs réactifs primaires correspondants, comme décrit dans le tableau 2. Incuber les échantillons pendant 20 min à température ambiante.
REMARQUE : Il a été calculé que toutes les concentrations de réactifs utilisées étaient supérieures au nombre de protéines exprimées à la surface des cellules de levure. Le calcul suppose l’expression de 10 000 protéines/cellule à partir de toutes les levures 2 x 106 dans un échantillon35, alors que seulement ~50 % d’entre elles le font habituellement. Le tableau 3 montre le rapport de réactif de marquage en excès pour chacun des réactifs exprimé dans le tableau 2, calculé comme suit :

- Centrifugez les échantillons à 4 500 × g pendant 1 min et jetez le surnageant en tant que déchet biologique dangereux.
- Laver les cellules une fois en les mettant en suspension dans 1 mL de PBSA. Répétez l’étape 4.2.
- Remettre en suspension les échantillons dans un mélange de 20 μL de leurs réactifs secondaires correspondants, comme décrit dans le tableau 2. Incuber les échantillons pendant 15 min en l’absence de lumière.
- Répétez l’étape 4.2.
- Répétez l’étape 4.3.
- Remettre en suspension les échantillons lavés dans 300 à 500 μL de PBSA et les transférer dans des tubes de polystyrène de 5 mL pour les analyser immédiatement à l’aide d’un cytomètre en flux approprié.
REMARQUE : Si des échantillons doivent être transportés, conservez-les sur de la glace humide. Ce n’est pas recommandé, mais les échantillons peuvent être conservés à 4 °C pendant un maximum de 2 h sous forme de granulés humides. - Après avoir effectué le démarrage et la préparation nécessaires du cytomètre pour une nouvelle expérience, cliquez sur le bouton Nouvelle expérience dans le menu Fichier , nommez l’expérience, puis cliquez sur Enregistrer pour vous assurer que les données acquises sont enregistrées dans le chemin d’accès au fichier souhaité.
- Sélectionnez l’icône de pointage dans la barre d’outils supérieure pour créer deux point-plots ou plus pour chaque échantillon à exécuter. Pour l’un des diagrammes à points, sélectionnez le nom de l’axe X pour afficher le canal FSC-A et le nom de l’axe Y pour afficher le canal SSC-A. Ce graphique montre l’aire de dispersion latérale par rapport à l’aire de diffusion directe et est utilisé pour bloquer les cellules de levure pour une analyse plus approfondie.
- Sur un autre graphique à points, sélectionnez le nom de l’axe X pour afficher le canal dans lequel le réactif secondaire ciblant l’anticorps anti-épitope primaire devient fluorescent. Sélectionnez le nom de l’axe Y pour afficher le canal dans lequel le réactif secondaire de la streptavidine devient fluorescent. Ce graphique ne montrera que les événements contrôlés par le biais du graphique de dispersion latéral par rapport au graphique de dispersion vers l’avant en tant que cellules de levure et est utilisé pour afficher la phosphorylation de la tyrosine sur l’axe Y et l’expression de surface du substrat sur l’axe X.
REMARQUE : Dans cet exemple, le réactif secondaire ciblant l’anticorps anti-épitope primaire est fluorescent dans le canal FITC (AF-488) et le réactif secondaire de la streptavidine dans le canal AF-647. Les canaux utilisés peuvent varier en fonction des réactifs primaires et secondaires utilisés lors de l’étiquetage. - Placez chaque tube d’échantillon dans le support de tube du cytomètre et sélectionnez Exécuter pour que le cytomètre commence à charger l’échantillon et à acquérir des données. Ajustez les événements à afficher, les événements à enregistrer, le temps d’enregistrement et le débit d’échantillon si nécessaire.
- Définissez une porte autour des cellules de levure saines dans le graphique SSC-A vs FSC-A créé à l’étape 4.9. La figure 1 illustre une représentation descriptive de la stratégie de contrôle à appliquer.
REMARQUE : Sur le graphique SSC-A par rapport au graphique FSC-A, 100 000 événements à afficher et 50 000 événements à enregistrer dans la porte de levure définie constituent une bonne ligne directrice pour visualiser et collecter suffisamment de données pour une analyse plus approfondie. Des stratégies de contrôle plus strictes pour sélectionner la levure singulet peuvent être appliquées sur la base d’un graphique de la hauteur de dispersion vers l’avant par rapport à la zone de diffusion vers l’avant, comme récemment signalé36. La stratégie de déclenchement illustrée à la figure 1 correspond à une approche moins stricte, incluant la levure singulet et un peu de levure doublet par le biais d’un désaccord de dispersion. - Enregistrez la fluorescence de tous les échantillons de contrôle à l’aide d’un analyseur de cytométrie en flux. Des échantillons de contrôle sont systématiquement prélevés en premier pour aider à définir une stratégie de contrôle, comme décrit ci-dessous à l’étape 4.14.
- Définissez une stratégie de contrôle pour votre parcelle créée à l’étape 4.10 avant d’analyser les échantillons traités. La figure 1 illustre une représentation descriptive de la stratégie de contrôle à appliquer.
- Enregistrer la fluorescence des échantillons déphosphorylés à l’aide du cytomètre en flux et de la stratégie de déclenchement définie aux étapes 4.12 et 4.14.
- Analysez les données de cytométrie en flux acquises à l’aide d’un logiciel d’analyse de cytométrie en flux.
- Évaluez la déphosphorylation en mesurant et en comparant la médiane de l’axe Y des cellules exprimant des protéines à leur surface et la phosphorylation de base fournie par les cellules non affichées entre les échantillons et les témoins. Calculez le pourcentage de différence médiane de phosphorylation comme suit :
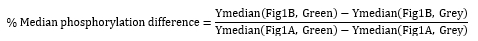
Résultats
L’analyse par cytométrie en flux d’une réplique individuelle de notre système modèle incubée pendant 2 h sans (Figure 1A) et avec (Figure 1B) 1 000 nM SHP-2 révèle une différence de phosphorylation médiane de 63,6 %, qui est définie comme le rapport de la médiane de l’axe Y (phosphorylation) de tous les événements affichés en surface moins le signal de phosphorylation de base défini comme la médiane de l’axe Y des événements non affichés entre l’échantillon traité et le témoin non traité, comme décrit dans l’équation défini à l’étape 4.12 du protocole. Avant l’analyse des médianes, les échantillons ont été classés en fonction de leur taille (Forward Scatter) et de leur complexité (Side Scatter) afin d’englober un groupe sain de cellules. Dans les conditions définies, la déphosphorylation de l’échantillon doit être visible à la vue de tous (figure 1C).
Le dosage est défini par une procédure simple composée de quatre méthodes principales (Figure 2A). Le système d’affichage de surface de levure que nous utilisons et que nous avons publié précédemment est basé sur un plasmide contenant un promoteur bidirectionnel avec la capacité d’expression inductible simultanée d’une paire kinase-substrat composée de la queue cytoplasmique de CD28 et de la tyrosine kinase LCK (Figure 2B)29. La co-localisation des protéines traduites est dirigée par un réticulum endoplasmique ciblant le peptide signal et la modification post-traductionnelle est forcée par l’augmentation du temps de séjour résultant d’une séquence de rétention C-terminale du RE. La sécrétion du substrat phosphorylé fusionné à Aga2p conduit à l’expression en surface (Figure 2C). Le substrat est conçu pour être flanqué de deux étiquettes d’épitopes, ce qui permet la confirmation extracellulaire de la traduction réussie et de l’expression de surface ultérieure. L’incubation du substrat présentant des cellules de levure avec la phosphatase d’intérêt (dans ce cas, la tyrosine phosphatase SHP-2) permet d’analyser la modification enzymatique par la diminution du phosphate lié au substrat (Figure 2D).
Même si des conditions optimales ont été définies pour le système modèle présenté, la généralisabilité du test permet de diversifier les protéines à analyser. Les conditions optimales pour le dosage ont été définies à l’aide d’une série de titrages où différentes combinaisons de temps et de concentration de phosphatase ont été évaluées en quatre exemplaires (figure 3). Les données analysées ont démontré une signification statistique grâce à une ANOVA à deux facteurs avec réplication (p < 0,05). Les conditions choisies de 2 h et 1 000 nM (48,8 % ± 10 %) offraient une différence de phosphorylation médiane d’environ 50 % tout en maintenant une signification statistique par rapport à son homologue de 750 nM à 2 h (p < 0,05) sur la base d’une analyse de test t avec une variance inégale. L’essai t n’a pas non plus révélé de différence significative par rapport aux résultats de 2 h et de 1 000 nM en augmentant le temps de 1 h sous l’une des concentrations qui ont fourni un pourcentage approximatif de différence médiane de phosphorylation (p > 0,05 pour 500 nM, 750 nM et 1 000 nM à 3 h).
L’analyse post-hoc de la HSD de Tukey révèle que toutes les comparaisons moyennes entre les périodes d’incubation pour toutes les concentrations sont significativement différentes, à l’exception de 1 h à 2 h. Lorsque l’on compare les multiples concentrations testées, on observe seulement des différences moyennes statistiquement significatives lorsque l’on compare 250 nM à toutes les autres concentrations, ce qui indique que des niveaux comparables d’activité de la phosphatase sont attendus dans les groupes, à l’exception de 250 nM. Malgré l’observation d’une différence de 20 % lorsque les échantillons ont été traités à 4 h et 1 000 nM SHP-2 (22,1 % ± 5,5 %), par rapport aux conditions optimales (test t, p < 0,05), nous avons décidé de ne pas poursuivre cette combinaison en raison de l’expression de surface réduite et de la santé des levures diminuée de la longue incubation avec DTT. Nous émettons l’hypothèse que cela est causé par les conditions réductrices du tampon de travail, qui est nécessaire au bon fonctionnement de la phosphatase SHP-2.

Figure 1 : Analyse par cytométrie en flux du système modèle. Graphiques de densité montrant la diffusion directe (axe X) par rapport à la porte de diffusion latérale (axe Y) (à gauche) et graphiques à points montrant l’expression de surface à travers le marquage de l’épitope C-terminal du substrat (axe X) par rapport à la phosphorylation du substrat (axe Y) (au centre) du domaine cytoplasmique CD28 incubé pendant 2 h (A) sans SHP-2 et (B) avec la concentration optimale de SHP-2 définie à 1 000 nM. La médiane Y a été mesurée à l’intérieur des portes définies englobant les cellules exprimées en surface (vert) uniquement en tant que mesure de phosphorylation relative. Les événements de signal non affichés ont été contrôlés pour définir la mesure Y-Médiane de fond (gris). (C) Superposition d’échantillons montrant une différence de visibilité en médiane Y. Veuillez cliquer ici pour voir une version agrandie de cette figure.
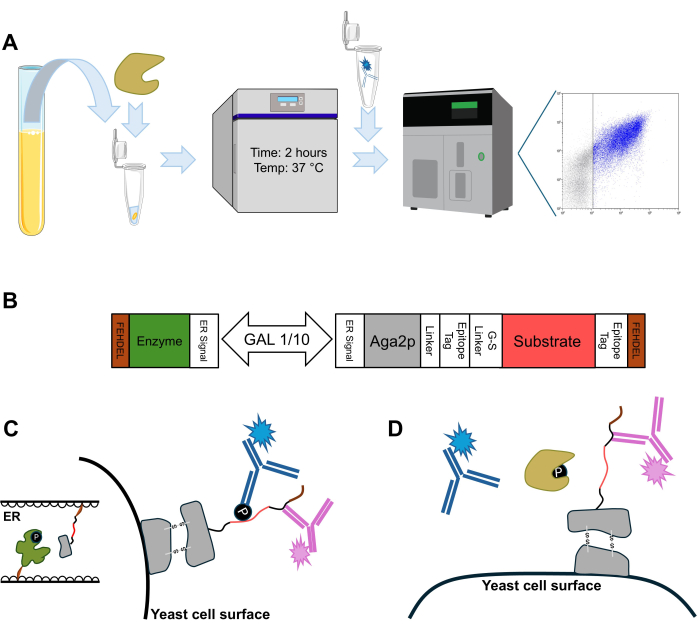
Figure 2 : Modification enzymatique des protéines affichées à la surface de la levure. (A) Schéma de dosage présentant les étapes critiques des quatre méthodes décrites : préparation d’échantillons avec de la phosphatase (tan) pour le dosage enzymatique, incubation pour l’activité enzymatique souhaitée, lavage cellulaire et marquage pour la détection de l’activité, et analyse par cytométrie en flux et collecte de données. (B) Schéma de gène représentant une structure générale de la cassette utilisée pour la séquestration du réticulum endoplasmique d’une paire enzyme-substrat et l’expression de surface du substrat. (C) Représentation graphique de la co-localisation du réticulum endoplasmique d’une paire kinase-substrat (à gauche) suivie de la sécrétion du substrat modifié post-traductionnellement affiché à la surface de la levure et marqué avec un anticorps anti-phosphotyrosine (en bleu) et un anticorps anti-épitope (en rose) pour confirmer l’expression de surface (à droite). (D) L’incubation de cellules de levure avec de la phosphatase (tan) élimine le groupe phosphate du substrat affiché, perturbant le marquage des anticorps anti-phosphotyrosine, facilitant l’analyse de la modification enzymatique par cytométrie en flux. Veuillez cliquer ici pour voir une version agrandie de cette figure.
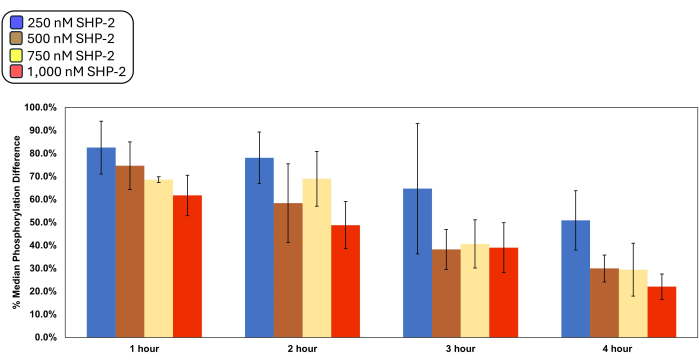
Figure 3 : Activité de la phosphatase et titrage temporel. Les cellules de levure ont été exposées à de multiples combinaisons de temps et de concentration de phosphatase, suivies d’une analyse par cytométrie en flux. Tous les échantillons traités ont été comparés à un témoin incubé pendant la même durée et dans les mêmes conditions tampons sans SHP-2. Le pourcentage de différence médiane de phosphorylation a été défini comme le rapport entre la médiane Y et les événements affichés en surface moins le signal de bruit de référence fourni par la médiane Y dans les événements non affichés dans les échantillons contenant SHP-2, divisé par la même relation dans leur contrôle respectif. L’hypothèse nulle est rejetée lorsque des différences significatives sont observées en comparant les changements entre les groupes de temps et de concentration à l’aide de l’ANOVA à deux facteurs (p < 0,05). L’effet des variables décrites sur le pourcentage de différence médiane de phosphorylation est indépendant les unes des autres (Interaction p > 0,05). Le test HSD de Tukey a été effectué pour une analyse a posteriori afin d’obtenir de plus amples renseignements sur la signification de la différence entre les groupes de temps d’incubation et de concentration globaux, et une série de tests t supposant une variance inégale a été utilisée pour définir la signification statistique de groupes individuels à un moment et à une concentration spécifiés. Les données sont présentées sous forme d’écart type moyen ± de quatre répétitions. Veuillez cliquer ici pour voir une version agrandie de cette figure.
Tableau 1 : Lignes directrices pour la préparation des milieux de croissance de la levure et d’induction des protéines. Description tabulaire de la masse requise pour chaque composant chimique afin de formuler 1 L de milieu de croissance sélectif de levure, de milieu d’induction sélectif de protéines de levure et de milieu de peptone dextrose d’extrait de levure. Une fois que le milieu décrit a été correctement mélangé, filtrez-le avant de l’utiliser. Des instructions supplémentaires sont incluses pour la fabrication de plaques de milieu de croissance de levure sélective. Veuillez cliquer ici pour télécharger ce tableau.
Tableau 2 : Préparation recommandée de l’échantillon pour la déphosphorylation du substrat et la stratégie de marquage des anticorps. Description tabulaire des échantillons nécessaires à la mesure des modifications enzymatiques des substrats phosphorylés affichés à la surface de la levure. La préparation de l’échantillon et la stratégie de marquage des anticorps qui suit sont spécifiées pour les contrôles requis et pour chaque échantillon à analyser, y compris les dilutions correspondantes des réactifs de marquage. Veuillez cliquer ici pour télécharger ce tableau.
Tableau 3 : Calculs du rapport d’excès d’anticorps par rapport à l’excès de protéines. Description tabulaire du nombre théorique d’anticorps disponibles par protéine exprimée à la surface d’une cellule de levure lors du marquage pour la cytométrie en flux. Le nombre théorique exprimé suppose que 100 % des cellules de levure expriment 10 000 protéines à leur surface pour garantir l’excès d’anticorps et les calculs du rapport d’excès sont basés sur la concentration du stock de réactifs affichée dans le tableau et acquise auprès du fournisseur. Veuillez cliquer ici pour télécharger ce tableau.
Tableau supplémentaire S1 : Séquence d’acides aminés des cassettes de construction. Représentation tabulaire de la séquence d’acides aminés pour les cassettes de construction situées des deux côtés du promoteur Gal 1-10. Les séquences en surbrillance correspondent à leurs descriptions codées par couleur. Le côté Gal-10 du plasmide est représenté par une traduction inverse de la séquence d’acides aminés pour faciliter sa compréhension. Tous les caractères laissés en noir correspondent à la traduction en acides aminés des sites de digestion des enzymes de restriction utilisés pour fournir de la modularité à la construction. Veuillez cliquer ici pour télécharger ce fichier.
Discussion
Le protocole présenté permet d’analyser les interactions enzymatiques en utilisant l’affichage extracellulaire des protéines à la surface de la levure. L’incorporation de la séquestration du réticulum endoplasmique dans le plasmide de surface utilisé introduit la capacité d’analyser des interactions spécifiques entre les enzymes et les substrats modifiés post-traductionnellement en raison des interactions intracellulaires qui peuvent être conçues pour se produire27,29. Les tests d’interaction enzymatique précédemment établis utilisant l’affichage de surface de la levure impliquent l’expression des protéines d’intérêt intracellulaire, l’affichage de surface étant uniquement un outil de détection des interactions intracellulaires qui se produisent entre les protéines cibles 23,25,29.
Ce protocole s’appuie sur cette plate-forme en déplaçant les interactions enzymatiques ciblées vers l’environnement extracellulaire, ce qui introduit une flexibilité supplémentaire à la fois dans les enzymes qui peuvent être étudiées et dans l’environnement dans lequel leur activité est surveillée. Les interactions étudiées qui se produisent extracellulairement donnent aux chercheurs la possibilité d’adapter l’environnement d’incubation pour qu’il soit plus optimal pour l’activité enzymatique, en élargissant les enzymes qui pourraient être étudiées dont les activités sont entravées dans le réticulum endoplasmique de la levure, qui est un environnement fortement oxydant37. De plus, la capacité de titrer la concentration des enzymes par rapport à un substrat donné permet des tests d’activité enzymatique spécifiques qui ne pourraient pas être effectués intracellulaires en raison de la saturation hypothétique du taux de réaction pendant la séquestration.
Dans le protocole, il y a plusieurs étapes critiques à noter pour s’assurer que le résultat souhaité sera respecté. La transformation réussie des constructions étudiées en levure est essentielle pour obtenir des résultats optimaux à partir des étapes suivantes. Une surveillance précise de la densité optique doit être effectuée pour suivre la croissance saine des cultures et s’assurer qu’elles ne prolifèrent pas avant l’induction des protéines ou la préparation de l’échantillon. La phase logarithmique de la croissance des cellules de levure englobe la période où la production de protéines est la plus élevée, tandis que dans la phase stationnaire, les mécanismes responsables de la production de protéines s’arrêtent38. En gardant cela à l’esprit, les mesures de densité optique fournissent une mesure précise des phases de croissance dans lesquelles se trouvent les cultures de levure, et des étapes telles que l’induction des protéines et la préparation des tests doivent être effectuées en dehors de la phase stationnaire ou lorsque les cultures deviennent envahies par la végétation (OD600nm < 6).
Pour l’essai de modification enzymatique, l’environnement d’incubation décrit était spécifique à l’enzyme étudiée, SHP-2, et à l’activité enzymatique réalisée, la déphosphorylation. Le DTT a été utilisé pour l’environnement réducteur qu’il fournit pendant l’incubation avec SHP-234. Par conséquent, il est important de mesurer avec précision les concentrations des produits chimiques utilisés pour modifier l’environnement d’incubation dans les essais enzymatiques afin d’assurer une activité enzymatique cohérente entre les échantillons et les expériences. SHP-2 a été utilisé comme protéine recombinante, et il est essentiel de réguler la température pendant les différentes étapes de manipulation de l’enzyme. Pour un test réussi, l’enzyme ne doit pas avoir subi plus de deux cycles de gel-dégel et doit être sur de la glace lors de la préparation de chaque échantillon. Il est alors impératif d’aliquoter l’enzyme recombinante dans un volume suffisant pour satisfaire aux exigences du dosage. Pendant l’incubation proprement dite, la température doit être strictement contrôlée à la température optimale pour l’enzyme, 37 °C dans ce cas, avec un mouvement constant d’un rotor pour assurer l’homogénéité du mélange d’incubation.
La méthode globale d’analyse avec des enzymes recombinantes a nécessité des modifications spécifiques à l’interaction entre SHP-2 et le substrat phosphorylé affiché en surface. L’adaptation du protocole à d’autres interactions enzyme-substrat extracellulaire implique la modification des séquences utilisées, de l’environnement du tampon d’activité et du réactif utilisé pour la détection. Pour tester d’autres interactions kinase-substrat-phosphatase, l’adaptation implique le remplacement des séquences protéiques d’une paire kinase-substrat dans leurs positions respectives dans la cassette de construction décrite dans le tableau supplémentaire S1. La séquence protéique du substrat ainsi qu’au moins le domaine kinase de la kinase d’intérêt doivent être incluses dans le plasmide, et la phosphatase ciblant le substrat phosphorylé produit doit se présenter sous la forme d’une protéine recombinante. L’interaction représentative entre LCK, CD28 et SHP-2 fournit un exemple d’utilisation de la séquestration du réticulum endoplasmique conçue dans la cassette de construction comme outil de production de protéines modifiées post-traductionnellement à étudier extracellulairement avec leur enzyme cible. Les substrats d’intérêt qui n’ont pas besoin de subir de modifications post-traductionnelles (par exemple, les substrats qui pourraient être phosphorylés extracellulairement à l’aide de kinase ajoutée) peuvent être exprimés à la surface de la levure sans enzyme appariée dans la cassette de construction. Dans ce cas, la séquence protéique de la kinase décrite dans le tableau supplémentaire S1 serait supprimée avec seulement la séquence du substrat incluse dans la cassette de construction. Nous notons de notre expérience antérieure que la co-localisation d’une sérine-thréonine kinase avec un substrat connu a entraîné l’affichage d’un substrat qui n’était pas phosphorylé (Ezagui et Stern, données non publiées), de sorte que des tests rigoureux de modification enzymatique réussie doivent être effectués avant l’application de la phosphatase extracellulaire. Nous avons précédemment publié un protocole de co-localisation kinase-substrat qui pourrait être utile pour cette étape de qualification39.
Les kinases et les phosphatases contiennent souvent des résidus de cystéine non appariés qui, lorsqu’ils sont oxydés, forment des liaisons disulfure à l’intérieur de la protéine ou à travers les protéines, ce qui peut perturber l’activité catalytique de la protéine en raison d’un changement de conformation40,41. La compréhension de la biochimie de cette protéine est essentielle pour déterminer l’environnement réactionnel approprié pour la modification enzymatique. Par conséquent, un agent réducteur doit être ajouté à l’environnement d’incubation pour s’assurer que la protéine recombinante utilisée reste active. Le DTT est un agent réducteur couramment utilisé à ces fins, mais la concentration dans le test doit être optimisée. L’utilisation d’une concentration trop élevée de DTT entrave l’affichage du substrat à la surface de la levure, car les ancres Aga1p et Aga2p sont maintenues l’une à l’autre par des liaisons disulfure, qui sont réduites en présence de DTT42. La concentration de DTT a été ajustée à la concentration minimale qui permettrait une activité maximale relative de la phosphatase sans avoir d’effets néfastes sur l’affichage en surface des substrats42. L’environnement d’incubation de l’enzyme testée doit être optimisé pour assurer la rétention de l’activité enzymatique lorsqu’elle est utilisée dans cet essai. Si une enzyme nécessite un environnement réducteur considérablement plus fort que le DTT de 0,5 mM utilisé dans ce test, la plate-forme est limitée par la réduction de l’affichage de surface et peut ne pas être optimale pour le test enzymatique spécifique souhaité. De même, le tampon 2x utilisé pendant l’étape d’incubation dans ce protocole a été inclus à partir de recherches antérieures sur les tampons acceptables favorisant l’activité de SHP-2, et des recherches similaires devraient être effectuées pour formuler un tampon d’incubation pour toute autre enzyme utilisée pour cet essai34. Les points de départ de la fabrication de ces tampons peuvent inclure la recherche documentaire pour des applications in vitro réussies de l’enzyme d’intérêt ou la recommandation du fabricant de l’enzyme pour un tampon d’activité. L’enzyme recombinante utilisée doit être titrée spécifiquement pour cet essai afin d’identifier une concentration et un temps d’incubation acceptables, ce qui permet à l’activité enzymatique ciblée de se produire avant la collecte des données.
Pour l’adaptation de ce protocole à d’autres types d’interaction enzyme-substrat, de nouveaux réactifs de détection fluorescente devront être sélectionnés et titrés en fonction de leur sensibilité. D’autres études en ont donné des exemples, notamment l’utilisation d’anticorps ciblant l’épitope pour détecter la présence ou l’absence de substrats peptidiques après un traitement à la protéase23,25 et d’anticorps sensibles à l’acétylation pour détecter les modifications des protéines d’histones43. Pour qualifier ces réactifs, un contrôle positif (qui démontre de manière vérifiable la modification de l’intérêt) et un contrôle négatif (qui démontre de manière vérifiable une absence de modification de l’intérêt) doivent être établis. Cela pourrait se faire par l’affichage à la surface de la levure d’une construction qui a déjà démontré la modification de l’intérêt, ou dans certains cas, pourrait être établi par l’immobilisation de protéines recombinantes ou de peptides. Par exemple, dans le cas de la phosphorylation (et de nombreuses autres modifications post-traductionnelles d’intérêt), les peptides de séquence connue pourraient être synthétisés avec soit de la phosphotyrosine (contrôle positif), soit de la tyrosine non modifiée (contrôle négatif) et une biotine C-terminale qui permettrait l’immobilisation des peptides sur des billes enrobées de streptavidine. Les billes recouvertes de peptides ont pu être marquées avec l’anticorps spécifique de la modification et évaluées pour leur spécificité et leur sensibilité de détection à l’aide de la cytométrie en flux avec des méthodes similaires à celles décrites à la section 4. Différentes dilutions d’anticorps doivent être utilisées pour trouver une concentration qui fournit le signal maximal du témoin positif, un signal minimal du témoin négatif et qui équilibre l’excès de repliement suffisant de l’anticorps par protéine modifiée (en utilisant l’équation de la note de l’étape 4.1) avec des considérations économiques pour le nombre d’expériences à mener par aliquote d’anticorps obtenue.
Nous décrivons un protocole permettant d’adapter la facilité de la plate-forme d’affichage de la surface de la levure pour les tests d’activité enzymatique extracellulaire. La méthode est démontrée en utilisant du CD28 phosphorylé affiché à la surface de la levure pour être déphosphorylé par SHP-2 recombinant pendant l’incubation, mais il est généralisable à de nombreux types de modification enzymatique par la modification du tampon de travail et de la paire enzyme-substrat utilisée.
Déclarations de divulgation
Les auteurs n’ont aucun conflit d’intérêts lié à ce travail à divulguer.
Remerciements
Ce travail a été soutenu par un prix NSF CAREER à L.A.S. (CBET - 2339172) et des fonds de démarrage de l’Université de Floride du Sud.
Dans la figure 2A, l’icône microtube-open-translucide de Servier https://smart.servier.com/ est sous licence CC-BY 3.0 Unported https://creativecommons.org/licenses/by/3.0/. Les modifications comprennent l’ajout d’un tampon et d’une cellule de levure (à gauche) et l’ajout d’anticorps (au centre à droite).
Le tube à essai, l’incubateur et le cytomètre en flux de la figure 2A ont été fournis par www.bioicons.com en libre accès.
matériels
| Name | Company | Catalog Number | Comments |
| 1 L Media Bottles | Corning | 06-414-1D | |
| 1.7/2.0 mL Microtubes | Axygen | MCT-175-C | |
| 10 µL SureOne Pipet Tips | Fisher Scientific | 02-707-438 | |
| 1000 µL SureOne Pipet Tips | Fisher Scientific | 02-707-408 | |
| 12 mL Polystyrene Round-Bottom Tubes | Greiner | 07-000-212 | |
| 3 mL platic Cuvettes | BRAND | 759076D | |
| 300 µL SureOne Pipet Tips | Fisher Scientific | 02-707-411 | |
| 5 mL Serological Pipettes | Fisher Scientific | 13-678-11D | |
| Acid Casein (Casamino Acids) | Fisher Scientific | BP-1424-500 | |
| Analytical Balance | Mettler Toledo | 30243397 | |
| Bacteriological Petri Dish | Corning | Falcon 351008 | |
| Biosafety Cabinets | Labconco | Logic Class II, Type A2 302310102 | |
| Biospectrometer | Eppendorf | Kinetic 6136000010 | |
| Bovine Serum Albumin | Fisher bioreagents | BP1600-100 | |
| Citric Acid | Fisher Scientific | A940-500 | |
| CytoFLEX Flow Cytometry Analyzer | Beckam Coulter | Cytoflex C09745 | CytExpert software |
| Dextrose | Fisher Scientific | D16-1 | |
| Dithiothreitol | Fisher bioreagents | BP172-5 | |
| Donkey anti-goat FITC | Invitrogen | A16000 | |
| EDTA | Alfa Aesar | H56165.30 | |
| Ez-Link PEG4-NHS-Biotin | Thermo Scientific | A39259 | |
| Frozen-EZ Yeast Transformation II Kit | Zymo Research | T2001 | |
| Galactose | Fisher Scientific | BP656-500 | |
| General Purpose Refrigerator | Marvel Scientific | MS24RAS4RW | |
| Goat anti-myc tag antibody | Bethyl | A190-104A | |
| Mictrotube Centrifuge | Eppendorf | 5425 R 5406000313 | |
| Mini Low Temperature Refrigerated Incubator | Fisher Scientific | 15-015-2632 | |
| Mouse anti-phosphotyrosine antibody 4G10 | BioXcell | BE0194 | |
| Parafilm M | Bemis | M PM999 | |
| Phosphate Buffered Saline | Fisher bioreagents | BP399-500 | |
| Pipette Controller | Eppendorf | easypet 3 4430000018 | |
| Raffinose | Thermo Scientific | J21060-36 | |
| Recombinant human Active SHP-2 Protein | R&D Systems | 1894-SH | |
| Refrigerated Centrifuge | Eppendorf | 5910 R | |
| Saccharomyces cerevisiae yeast surface display strain EBY 100 | ATCC | MYA-4941 | |
| Shaker Incubator | Eppendorf | M1335-0002 New Brunswick Innova 42 | |
| Single Channel Pipette Set | Eppendorf | 05-403-151 | |
| Sodium Chloride | Fisher Scientific | S671-500 | |
| Sodium Citrate Dihydrate | Fisher Scientific | S279-500 | |
| Sodium Phosphate Dibasic Heptahydrate | Fisher Scientific | S373-500 | |
| Sodium Phosphate Monobasic Monohydrate | Fisher Scientific | S468-500 | |
| Streptavidin Alexa Fluor 647 | Invitrogen | S32357 | |
| Top Loading Balance | Mettler Toledo | ||
| Tris hydrochloride | EMD Millipore | 648317-100GM | |
| Tube revolver rotator | Fisher Scientific | 11-676-341 | |
| Weighing Paper | Fisher Scientific | 09-898-12B | |
| Yeast Nitrogen Base | BD Difco | 291940 | |
| Zeba Spin Desalting Columns | Thermo Scientific | 89883 |
Références
- Lea, M. A., Weber, G. Role of enzymes in homeostasis: VIII. Inhibition of the activity of glycolytic enzymes by free fatty acids. J Biol Chem. 243 (6), 1096-1102 (1968).
- Mahé, M., Rios-Fuller, T. J., Karolin, A., Schneider, R. J. Genetics of enzymatic dysfunctions in metabolic disorders and cancer. Front Oncol. 13, 1230934(2023).
- Fernandez-de-Cossio-Diaz, J., Vazquez, A. A physical model of cell metabolism. Sci Rep. 8 (1), 8349(2018).
- Metallo, C. M., Vander Heiden, M. G. Understanding metabolic regulation and its influence on cell physiology. Mol Cell. 49 (3), 388-398 (2013).
- Mildvan, A. S. Mechanisms of signaling and related enzymes. Proteins. 29 (4), 401-416 (1997).
- Frosina, G. Overexpression of enzymes that repair endogenous damage to DNA. Eur J Biochem. 267 (8), 2135-2149 (2000).
- Schärer, O. D. Chemistry and biology of DNA repair. Angew Chem Int Ed. 42 (26), 2946-2974 (2003).
- de la Fuente, M., et al. Enzyme therapy: Current challenges and future perspectives. Int J Mol Sci. 22 (17), 9181(2021).
- Robertson, J. G. Enzymes as a special class of therapeutic target: clinical drugs and modes of action. Curr Opin Struct Biol. 17 (6), 674-679 (2007).
- Goddard, J. -P., Reymond, J. -L. Enzyme assays for high-throughput screening. Curr Opin Biotechnol. 15 (4), 314-322 (2004).
- Helm, J. S., Hu, Y., Chen, L., Gross, B., Walker, S. Identification of active-site inhibitors of MurG using a generalizable, high-throughput glycosyltransferase screen. J Am Chem Soc. 125 (37), 11168-11169 (2003).
- Veldhuyzen, W. F., Nguyen, Q., McMaster, G., Lawrence, D. S. A light-activated probe of intracellular protein kinase activity. J Am Chem Soc. 125 (44), 13358-13359 (2003).
- Torres, M., Forman, H. J. Encyclopedia of Respiratory. Laurent, G. J., Shapiro, S. D. , Academic Press. 10-18 (2006).
- Blume-Jensen, P., Hunter, T. Oncogenic kinase signalling. Nature. 411 (6835), 355-365 (2001).
- Martin, G. S. Cell signaling and cancer. Cancer Cell. 4 (3), 167-174 (2003).
- Lothrop, A. P., Torres, M. P., Fuchs, S. M. Deciphering post-translational modification codes. FEBS Lett. 587 (8), 1247-1257 (2013).
- Graves, J. D., Krebs, E. G. Protein phosphorylation and signal transduction. Pharmacol Ther. 82 (2), 111-121 (1999).
- Hafen, E. Kinases and phosphatases--A marriage is consummated. Science. 280 (5367), 1212-1213 (1998).
- Westphal, R. S., Anderson, K. A., Means, A. R., Wadzinski, B. E. A signaling complex of Ca2+-calmodulin-dependent protein kinase IV and protein phosphatase 2A. Science. 280 (5367), 1258-1261 (1998).
- Barford, D., Das, A. K., Egloff, M. -P. The structure and mechanism of protein phosphatases: Insights into catalysis and regulation. Annu Rev Biophys Biomol Struct. 27, 133-164 (1998).
- Liu, Q., Qu, J., Zhao, M., Xu, Q., Sun, Y. Targeting SHP2 as a promising strategy for cancer immunotherapy. Pharmacol Res. 152, 104595(2020).
- Pan, J., Zhou, L., Zhang, C., Xu, Q., Sun, Y. Targeting protein phosphatases for the treatment of inflammation-related diseases: From signaling to therapy. Signal Transduct Targeted Ther. 7 (1), 177(2022).
- Denard, C. A., et al. YESS 2.0, a tunable platform for enzyme evolution, yields highly active TEV protease variants. ACS Synth Biol. 10 (1), 63-71 (2021).
- Lim, S., Glasgow, J. E., Filsinger Interrante, M., Storm, E. M., Cochran, J. R. Dual display of proteins on the yeast cell surface simplifies quantification of binding interactions and enzymatic bioconjugation reactions. Biotechnol J. 12 (5), (2017).
- Yi, L., et al. Engineering of TEV protease variants by yeast ER sequestration screening (YESS) of combinatorial libraries. Proc Natl Acad Sci USA. 110 (18), 7229-7234 (2013).
- Yi, L., et al. Yeast endoplasmic reticulum sequestration screening for the engineering of proteases from libraries expressed in yeast. Methods Mol Biol. 1319, 81-93 (2015).
- Semenza, J. C., Hardwick, K. G., Dean, N., Pelham, H. R. ERD2, a yeast gene required for the receptor-mediated retrieval of luminal ER proteins from the secretory pathway. Cell. 61 (7), 1349-1357 (1990).
- Mei, M., et al. Characterization of aromatic residue-controlled protein retention in the endoplasmic reticulum of Saccharomyces cerevisiae. J Biol Chem. 292 (50), 20707-20719 (2017).
- Ezagui, J., Russell, B., Mairena, Y., Stern, L. A. Endoplasmic reticulum sequestration empowers phosphorylation profiling on the yeast surface. AIChE J. 68 (12), e17931(2022).
- Kawai, S., Murata, K. Genetic Transformation Systems in Fungi. van den Berg, M. A., Maruthachalam, K. 1, Springer International Publishing. 187-192 (2015).
- Kawai, S., Hashimoto, W., Murata, K. Transformation of Saccharomyces cerevisiae and other fungi: methods and possible underlying mechanism. Bioeng Bugs. 1 (6), 395-403 (2010).
- Loock, M., et al. High-efficiency transformation and expression of genomic libraries in yeast. Methods Protoc. 6 (5), 89(2023).
- Huang, D., Gore, P. R., Shusta, E. V. Increasing yeast secretion of heterologous proteins by regulating expression rates and post-secretory loss. Biotechnol Bioeng. 101 (6), 1264-1275 (2008).
- Yu, B., et al. Targeting protein tyrosine phosphatase SHP2 for the treatment of PTPN11-associated malignancies. Mol Cancer Ther. 12 (9), 1738-1748 (2013).
- Stern, L. A., et al. Geometry and expression enhance enrichment of functional yeast-displayed ligands via cell panning. Biotechnol Bioeng. 113 (11), 2328-2341 (2016).
- Pan, X., et al. Optimized single-cell gates for yeast display screening. Protein Eng Design Sel. 38, gzae018(2025).
- Margittai, É, et al. Production of H2O2 in the endoplasmic reticulum promotes in vivo disulfide bond formation. Antioxid Redox Signal. 16 (10), 1088-1099 (2012).
- Werner-Washburne, M., Braun, E., Johnston, G. C., Singer, R. A. Stationary phase in the yeast Saccharomyces cerevisiae. Microbiol Rev. 57 (2), 383-401 (1993).
- Ezagui, J., Stern, L. A. Tyrosine phosphorylation screening on the yeast surface by magnetic bead selection and FACS. Methods Mol Biol. 2681, 275-290 (2023).
- Yarnall, M. T. N., Kim, S. H., Korntner, S., Bishop, A. C. Destabilization of the SHP2 and SHP1 protein tyrosine phosphatase domains by a non-conserved “backdoor” cysteine. Biochem Biophys Rep. 32, 101370(2022).
- Dustin, C. M., Heppner, D. E., Lin, M. J., van der Vliet, A. Redox regulation of tyrosine kinase signalling: more than meets the eye. J Biochem. 167 (2), 151-163 (2020).
- Stern, L. A., Csizmar, C. M., Woldring, D. R., Wagner, C. R., Hackel, B. J. Titratable avidity reduction enhances affinity discrimination in mammalian cellular selections of yeast-displayed ligands. ACS Comb Sci. 19 (5), 315-323 (2017).
- Waldman, A. C., Rao, B. M., Keung, A. J. Mapping the residue specificities of epigenome enzymes by yeast surface display. Cell Chem Biol. 28 (12), 1772-1779.e4 (2021).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationExplorer plus d’articles
This article has been published
Video Coming Soon