Method Article
酵母表面显示蛋白的酶修饰和流式细胞术评估
摘要
在这里,我们介绍了一种使用酵母表面显示的底物进行酶促修饰测定的方案。使用酪氨酸磷酸酶 SHP-2 对其底物之一的去磷酸化活性分析作为代表性酶修饰测定法来证明该平台。
摘要
酵母表面显示是一种基因型-表型连锁策略,能够对蛋白质功能进行高通量筛选。传统上,酵母表面显示已应用于新结合蛋白的进化,流式细胞术用于按结合强度水平进行评估和排序。最近,人们越来越关注应用酵母表面显示来筛选底物变体的酶修饰,加性修饰(例如磷酸化)或减化修饰(例如蛋白水解)可提供流式细胞术可读的表型。这种修饰通常使用细胞内共定位进行应用,但实现所展示底物的细胞外酶促修饰的能力可能会开启更多的研究反应。在这里,我们描述了设计和应用细胞外酶修饰筛选测定到酵母表面显示的候选底物的技术,以及随后使用流式细胞术分析进行的评估。我们在磷酸酶去磷酸化酵母展示的含有磷酸化酪氨酸残基的底物的背景下提供了这些方案,并评论了该应用框架如何适应开发其他酶-底物对的筛选测定。
引言
了解酶与其靶标之间的相互作用已成为一个越来越有趣的研究领域,因为它在控制细胞稳态和疾病发展的途径的生物学表征中的必要性 1,2。酶负责催化维持生物生命的许多反应,控制必要的途径,如细胞代谢 3,4、信号传导5,甚至基因组修复 6,7 等基本过程。由于它们在这些过程中的作用,它们的相互作用也在许多疾病的发展中发挥作用,因为它们活性的偏差会导致细胞活性严重失调,从而导致有害癌细胞凋亡或增殖2。酶活性的研究在新疗法的开发中具有重要应用8,9,需要针对每种特定酶-底物相互作用量身定制的检测10。多种酶测定已被确立为评估和表征这些相互作用的标准方案。为分析酶相互作用而开发的检测分为监测激活/抑制结合的检测检测11 或监测酶底物修饰的检测12。
酶的一个主要作用是调节细胞行为。信号转导是细胞对细胞外触发因素13 的细胞内反应,负责细胞存活和功能。细胞增殖、分化和许多其他功能过程都涉及信号转导通路,其中酶相互作用控制它们14,15。酶催化翻译后修饰,这通常调节负责正确传递细胞外信息的大型信号网络16。蛋白磷酸化是最常见的翻译后修饰,在细胞信号转导和多种其他细胞通路中普遍存在。因此,由于其关键的调节作用,蛋白激酶已成为潜在治疗靶标的重要比例17。磷酸酶是基于磷酸盐的细胞信号转导复合物的天然调节分子18,19,具有从其靶蛋白中去除磷酸盐残基的能力20。在过去十年中,磷酸酶已成为癌症治疗21 和炎症性疾病22 的主要治疗靶点,因为它们参与多种细胞类型中下游信号通路的调节。蛋白激酶和磷酸酶共同提供了广泛的相互作用,可以通过开发特定的酶检测方案来研究。
酵母表面显示已被用作表征和评估酶活性的工具23,24。当与内质网隔离策略结合使用时,它为筛选翻译后修饰过程提供了一个高通量平台25,26。这允许激酶-底物对通过与 KDEL 受体27 结合共定位并保留在内质网中,由于激酶与其靶标之间的接近,底物的磷酸化速率可能增加。KDEL 受体结合由 C 端 FEHDEL 内质网保留序列介导,显示比其他 HDEL 序列具有更强的保留能力25,28。然后将磷酸化底物锚定到酵母表面,以便随后通过流式细胞术进行评估29。目前,还没有为酵母表面显示的底物的酶促修饰建立可推广的方案。我们通过利用细胞外表达的磷酸化底物变体并通过其已知磷酸酶的去磷酸化对其进行修饰来扩展酵母表面显示的能力。然后,流式细胞术分析提供了一个平台,通过测量由于与已知磷酸酶孵育而导致的磷酸化中位数的变化,为上述底物的表型评估提供了一个平台。这为表面显示蛋白的翻译后修饰提供了一种适应性强的方法,同时也提供了一种在使用酵母表面显示平台时对相互作用进行酶促修饰分析的方法。
我们提出了酶修饰测定的开发和应用技术,该技术描述了将激酶-底物相互作用引入酵母表面展示平台,表达的磷酸化底物与重组磷酸酶的共孵育,以及随后通过流式细胞术分析去磷酸化活性。在本报告中,这是通过将 CD28 的细胞质结构域与淋巴细胞激酶 (LCK) 共定位在酵母内质网中,然后在酵母表面显示磷酸化的 CD28,随后通过含有 Src 同源区域 2 结构域的磷酸酶-2 (SHP-2) 去磷酸化来实现的。泛抗磷酸酪氨酸抗体(在本研究中为 4G10)可检测多种肽序列中的磷酸化酪氨酸残基,用于定量磷酸化水平作为磷酸酶处理的函数。详细的过程为研究酶-底物相互作用提供了一种通用的方法;一种以纯化方式研究酶和底物的前瞻性方法。
研究方案
1. 携带质粒的酵母的细胞生长和蛋白质表达的诱导
- 按照 表 1 中描述的配方,准备不含质粒的酵母 (YPD)、含质粒的酵母细胞生长 (SD-CAA) 和诱导蛋白质表达 (SRG-CAA) 以及 SD-CAA 板生长所需的培养基。
- 通过基于锂阳离子的方法30,31 将含有激酶-底物对的酵母展示质粒 DNA 转化到 EBY-100 酵母细胞中,该方法通常用于各种制造商的酵母转化试剂盒。
注:根据被转化的质粒构建体,可以使用电穿孔32 或其他优选的酵母质粒转化技术。 - 制备含有 10 mL YPD 培养基的 14 mL 培养管。接种 EBY-100 细胞,并在 30 °C、300 rpm 的振荡培养箱中生长,直到培养物达到 0.8-1.0(8 x 106- 1 x 107 个细胞/mL)的光密度 (OD600nm)。
注:OD600nm 的测量方法是制备 3 mL 样品比色皿(在各自的培养基中含有 1:10 稀释的酵母培养物)和 3 mL 含有用于样品稀释的培养基的空白比色皿。分光光度计上的 OD600nm 程序用于首先测量空白比色皿,然后通过设置为每个样品准备的相应稀释度来测量每个样品比色皿。1 OD600nm 对应于 1 × 107 个酵母/mL。 - 通过以 1,000 x g 离心培养物 3 分钟来收获细胞,并使用酵母转化试剂盒中提供的第 1 种 洗涤溶液或 TE(10 mM Tris-HCl 和 1.0 mM EDTA)洗涤31。
- 再次以 1,000 x g 的浓度沉淀细胞 3 分钟,然后重悬于酵母转化试剂盒中提供的 1 mL 转化缓冲液或无菌水中。细胞应分装成 50 μL 等分试样,可在 -80 °C 下储存长达 6 个月。
- 对于质粒转化,制备每个质粒一个等分试样并在冰上解冻,然后将 0.5-1.5 μg 含有酵母展示构建体的质粒 DNA 直接加入到细胞中。加入 0.5 mL 酵母转化试剂盒中提供的转化溶液,或 0.5 mL 无菌 50% 聚乙二醇和 0.1 M LiOAc 溶液31。通过移液将细胞、质粒 DNA 和转化溶液的混合物彻底混合。
- 在 30 °C 下静态孵育转化混合物 30-60 分钟,每隔 15 分钟涡旋混合物。通过在 1,000 × g 离心 3 分钟来收获细胞。
- 准备一个装有 4.5 mL SD-CAA 培养基的 14 mL 培养管。将含有所需质粒的细胞重悬于 500 μL SD-CAA 中,并接种制备的 4.5 mL。
- 注意不要刺穿琼脂,将 50 μL 的 5 mL 接种培养物分配到 SD-CAA 板上,并在 30 °C 下静态孵育 48 小时以确定转化效率。
- 将 5 mL 的 SD-CAA 细胞培养物在 30 °C、300 rpm 的振荡培养箱中孵育至少 18 小时。16 小时和 20 小时后监测光密度 (OD600nm)。一旦样品生长到不超过 6 的足够光密度,将培养物以 2,500 × g 离心 3 分钟。丢弃上清液,不要干扰酵母沉淀。
- 将酵母沉淀重悬于 SRG-CAA 中,至最终 OD600nm ,小于 1(<1 × 107 酵母/mL)。
- 将酵母培养物在 30 °C、300 rpm 的振荡培养箱中孵育至少 8 小时但不超过 24 小时。
注:酵母细胞中蛋白质表达的诱导可以在 20-37 °C 之间变化。30 °C 适用于合成激酶/底物对29,33,但如果认为有必要,可以调整正在研究的特定蛋白质。 - 测量 OD600nm 以确定细胞密度。
注意:此时可以通过将酵母培养物储存在 4 °C 来终止该方案。
2. 4G10 抗磷酸酪氨酸抗体的生物素化
- 通过添加 680 μL 无菌 PBS,重悬 2 mg 小瓶 PEG4-NHS-生物素至终浓度为 5 mM。
注:PEG4-NHS-生物素的重悬应在进行生物素化反应之前立即新鲜进行。NHS 在水溶液中水解。使用无菌 PBS 和样品瓶对于制备用于长期储存的试剂和用于基于细胞的分析非常重要,以减轻污染物对试剂活力或正在进行的敏感分析的任何潜在影响。 - 根据 4G10 抗体浓度,将 100 μg 抗体加入无菌 1.7 mL 样品瓶中。
- 将步骤 2.1 中制备的 5 mM PEG4-NHS-生物素 1 μL 添加到装有 100 μg 4G10 抗体的小瓶中,以达到生物素与抗体的摩尔比为 7.5:1。轻轻吸取混合物以使反应匀浆。
- 在室温下以恒定旋转孵育反应至少 2 小时。
- 按照制造商的 0.5 mL 离心脱盐柱方案,将缓冲液从生物素化的 4G10 (B-4G10) 交换成 PBS。
注:截留分子量 (MWCO) 为 7 kDa 的脱盐柱通常用于抗体的生物素化,以去除未反应的生物素和其他小分子,同时保留较大的抗体。 - 在 PBSA(含 1 g/L 牛血清白蛋白的 PBS)中将 B-4G10 抗体稀释至终浓度为 1 μM。将 B-4G10 抗体分装成更小的体积,以防止重复冻融循环。
注:生物素化抗体可在 4 °C 下储存以供日常使用长达 3 个月,而不会显著降低效率。将其余未使用的等分试样在 -20 °C 下储存最长 2 年。
3. 酵母细胞表面表达的底物的去磷酸化
- 如文献34 中先前所述,准备 2x 工作缓冲液。
注意:建议使用 2 倍,以便于测量所需的成分。 - 通过在去离子水中以 1:2 的比例稀释步骤 3.1 中制备的 2x 缓冲溶液,在 1.7 mL 小瓶中制备样品,制备工作缓冲液。
注:每个样品的总反应体积为 20 μL,每个样品需要 10 μL 至 18 μL 的工作缓冲液。为所有样品或对照准备足够的工作缓冲液。 - 按照 表 2 中描述的推荐样品制备方法,在 1.7 mL 样品瓶上标记相应的对照品或样品名称。
- 根据步骤 1.9 中测量的 OD600nm ,计算从每个样品的相应酵母培养物中回收两百万 (2 × 106) 个酵母细胞所需的培养物体积。
- 将上一步中计算的酵母培养物体积添加到每个样品的 1.7 mL 样品瓶中。
- 将样品瓶以 4,500 × g 离心 1 分钟。使用微量移液器小心去除上清液,并作为生物危害性废物丢弃。
- 将沉淀的细胞重悬于 1 mL PBSA 中,然后重复 步骤 3.6。
注:在不干扰沉淀细胞的情况下去除尽可能多的上清液非常重要。 - 根据其储备液浓度,计算在 20 μL 总反应体积中最终浓度为 1,000 nM 所需的重组人 SHP-2 体积。
注:重组 SHP-2 应以小体积等分,以便每次测定中使用的任何蛋白质不超过两次冻融循环。在制备完所有样品进行检测后,等分试样中残留的任何 SHP-2 都应丢弃。
重组酶的储备液浓度可能因批号而异。重组 SHP-2 的储备液浓度通常为 0.2-0.4 mg/mL。对于 0.324 mg/mL SHP-2 的储备液浓度,这对应于 4.696 μM 的储备液浓度(SHP-2 的分子量为 69 kDa)。在 20 μL 反应中加入 4.26 μL SHP-2 储备液,得到 1,000 nM SHP-2 的最终反应浓度。 - 将 7.7 mg DTT 加入 10 mL 去离子水中,溶于 15 mL 锥形中,制成 5 mM DTT 溶液。如果设备限制难以称量毫克级,则向 10 mL 去离子水中加入 0.77 g DTT,然后进行 100 倍稀释,制成用于检测的 5 mM DTT 溶液。
注:如果无法使用现有设备测量毫克级量,则可以在更高浓度的储备液中制备 DTT 溶液,将其稀释至 5 mM。DTT 溶液需要制备新鲜的细胞,然后再在工作缓冲液中重悬细胞,因为它容易水解,使其在水中稀释时长时间不稳定。 - 在步骤 3.2 中制备的工作缓冲液中重悬沉淀的细胞,使每个样品或对照中的最终反应体积为 20 μL。
注:添加的工作缓冲液的量应根据每个样品中的 DTT (2 μL) 和 SHP-2(在步骤 3.8 中计算)的量计算。 - 向每个样品或对照中加入 2 μL 步骤 3.9 中制备的 5 mM DTT 溶液,最终反应浓度为 0.5 mM DTT。
- 将步骤 3.8 中计算的 SHP-2 体积添加到每个样品中,最终体积为 20 μL,并使用微量移液器轻轻混合。
- 用封口膜包裹样品瓶盖,以防止泄漏或交叉污染。
- 将样品在 37 °C 下在转子上以恒定速度孵育 2 小时。
- 从转子中取出样品,并向每个样品中加入 1 mL PBSA 终止反应。
- 重复步骤 3.6。
4. 去磷酸化底物的细胞标记和流式细胞术分析
- 如 表 2 所述,将步骤 3.16 中的样品重悬于相应主要试剂的 20 μL 混合物中。在室温下孵育样品 20 分钟。
注:根据酵母细胞表面表达的蛋白质数量,所有使用的试剂浓度均已计算过量。该计算假设样品35 中所有 2 x 106 酵母的表达量为 10,000 种蛋白质/细胞,而通常只有 ~50% 的酵母表达。表 3 显示了表 2 中表示的每种试剂的过量标记试剂比率,计算如下:

- 将样品以 4,500 × g 离心 1 分钟,并将上清液作为生物危害性废物丢弃。
- 通过在 1 mL PBSA 中重悬来洗涤细胞一次。重复步骤 4.2。
- 如 表 2 所述,将样品重悬于相应二级试剂的 20 μL 混合物中。在没有光线的情况下孵育样品 15 分钟。
- 重复步骤 4.2。
- 重复步骤 4.3。
- 将洗涤后的样品重悬于 300-500 μL PBSA 中,并转移到 5 mL 聚苯乙烯管中,立即使用合适的流式细胞仪进行分析。
注:如果需要运输样品,请放在湿冰上。不建议这样做,但样品可以作为湿颗粒在 4 °C 下储存最多 2 小时。 - 为新实验执行必要的细胞仪启动和准备后,单击 File(文件)菜单中的 New Experiment(新建实验)按钮,为实验命名,然后单击 Save(保存)以确保采集的数据保存在所需的文件路径中。
- 选择上方工具栏中的点图图标,为每个要运行的样品创建两个或多个点图。对于其中一个点图,选择 X 轴名称以显示 FSC-A 通道,选择 Y 轴名称以显示 SSC-A 通道。此图显示了侧向散射 - 面积与前向散射 - 面积,用于对酵母细胞进行门控以进行进一步分析。
- 在另一个点图上,选择 X 轴名称以显示靶向一抗表位标签抗体的二级试剂发出荧光的通道。选择 Y 轴名称可显示链霉亲和素二抗试剂发出荧光的通道。该图将仅显示从侧面与前向散点图门控的事件为酵母细胞,并用于在 Y 轴上显示酪氨酸磷酸化,在 X 轴上显示底物表面表达。
注:在本例中,靶向一抗表位标签抗体的二抗试剂在 FITC (AF-488) 通道中发出荧光,而链霉亲和素二抗试剂在 AF-647 通道中发出荧光。使用的通道可能因标记过程中使用的一级和二级试剂而异。 - 将每个样品管放入细胞仪的试管架中,然后为细胞仪选择“运行”以开始加载样品并采集数据。根据需要调整要显示的事件、要记录的事件、要记录的时间和样品流速。
- 在步骤 4.9 中创建的 SSC-A 与 FSC-A 图中定义围绕健康酵母细胞的门。 图 1 说明了要应用的门控策略的描述性表示。
注:在 SSC-A 与 FSC-A 图中,要显示 100,000 个事件,并在定义的酵母门内记录 50,000 个事件是可视化和收集足够数据以供进一步分析的良好指南。可以根据最近报道的前向散射高度与前向散射面积图36 来应用更严格的单线态酵母选择门控策略。 图 1 所示的门控策略对应于一种不太严格的方法,包括通过分散门控的单重态和一些双联酵母。 - 使用流式细胞术分析仪记录所有对照样品的荧光。首先定期收集对照样品以帮助定义门控策略,如下文步骤 4.14 中所述。
- 在分析处理样品之前,为步骤 4.10 中创建的图定义设门策略。 图 1 说明了要应用的门控策略的描述性表示。
- 使用流式细胞仪和步骤 4.12 和 4.14 中定义的门控策略记录去磷酸化样品的荧光。
- 分析使用流式细胞术分析软件采集的流式细胞术数据。
- 通过测量和比较在其表面表达蛋白质的细胞的 Y 轴中位数与样品和对照之间非显示细胞提供的基线磷酸化来评估去磷酸化。按如下方式计算磷酸化中位数百分比差异:
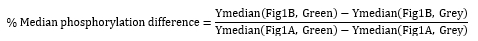
结果
来自我们的模型系统的单个复制品的流式细胞术分析在没有(图 1A)和(图 1B)1,000 nM SHP-2 的情况下孵育 2 小时,显示中位磷酸化差异为 63.6%,其定义为来自所有表面显示事件的 Y 轴中位数(磷酸化)减去基线磷酸化信号的比率,定义为处理样品和未处理对照之间未显示事件的 Y 轴中位数,如方程中所述在协议步骤 4.12 中定义。在分析中位数之前,根据样本的大小(前向散射)和复杂性(侧向散射)对样本进行门控,以包含一组健康的细胞。在规定的条件下,样品去磷酸化应该是在显而易见的地方可以区分的(图 1C)。
该测定由一个简单的程序定义,该程序由四种主要方法组成(图 2A)。我们使用和之前发布的酵母表面展示系统基于一种质粒,该质粒含有双向启动子,能够同时诱导表达由 CD28 的细胞质尾部和酪氨酸激酶 LCK 组成的激酶-底物对(图 2B)29。翻译蛋白的共定位由靶向信号肽的内质网和由 C 端 ER 保留序列导致的停留时间增加所迫使的翻译后修饰指导。与 Aga2p 融合的磷酸化底物的分泌导致表面表达(图 2C)。底物设计为两侧有两个表位标签,这允许细胞外确认成功翻译和随后的表面表达。将显示酵母细胞的底物与感兴趣的磷酸酶(在本例中为酪氨酸磷酸酶 SHP-2)一起孵育,允许通过减少底物结合的磷酸盐来分析酶修饰(图 2D)。
尽管已经为所提出的模型系统定义了最佳条件,但该测定的普遍性允许分析蛋白质的多样化。通过一系列滴定确定测定的最佳条件,其中磷酸酶的时间和浓度的不同组合一式四份进行评估(图 3)。分析的数据通过具有复制的双向方差分析显示统计学意义 (p < 0.05)。所选的 2 小时和 1,000 nM 条件 (48.8% ± 10%) 与 2 小时时的 750 nM 对应物相比,提供了大约 50% 的中位磷酸化差异,同时保持了统计学显着性 (p < 0.05) 基于不等方差的 t 检验分析。 t 检验还显示,在任何提供近似中位磷酸化差异百分比的浓度下,将时间增加 1 小时,与 2 小时和 1,000 nM 结果没有显着差异(3 小时时 500 nM、750 nM 和 1,000 nM 的 p > 0.05)。
Tukey HSD 事后分析显示,除 1 小时至 2 小时外,所有浓度的孵育期之间的所有平均比较均存在显著差异。当比较测试的多种浓度时,我们只观察到在将 250 nM 与所有其他浓度进行比较时具有统计学意义的平均差异,表明除 250 nM 外,组内预计磷酸酶活性水平相当。尽管在样品处理 4 小时和 1,000 nM SHP-2 时观察到 20% 的差异(22.1% ± 5.5%),但与最佳条件相比(t 检验,p < 0.05),我们决定不追求这种组合,因为表面表达降低和酵母健康降低与 DTT 长期孵育。我们假设这是由工作缓冲液的还原条件引起的,这是 SHP-2 磷酸酶正常功能所必需的。

图 1:模型系统的流式细胞术分析。 密度图显示前向散射(X 轴)与侧向散射门控(Y 轴)(左)和点图显示通过底物 C 末端表位标签标记(X 轴)与底物磷酸化(Y 轴)(中)孵育 2 小时的 CD28 细胞质结构域的表面表达(A),无 SHP-2 和 (B),SHP-2 的最佳浓度定义为 1,000 nM。在包含表面表达细胞(绿色)的定义门内测量 Y 中位数仅作为相对磷酸化测量。对非显示信号事件进行选通,以定义背景 Y 中位数测量值(灰色)。(C) 样品的点图叠加显示 Y 中位数的普通视力差异。 请单击此处查看此图的较大版本。
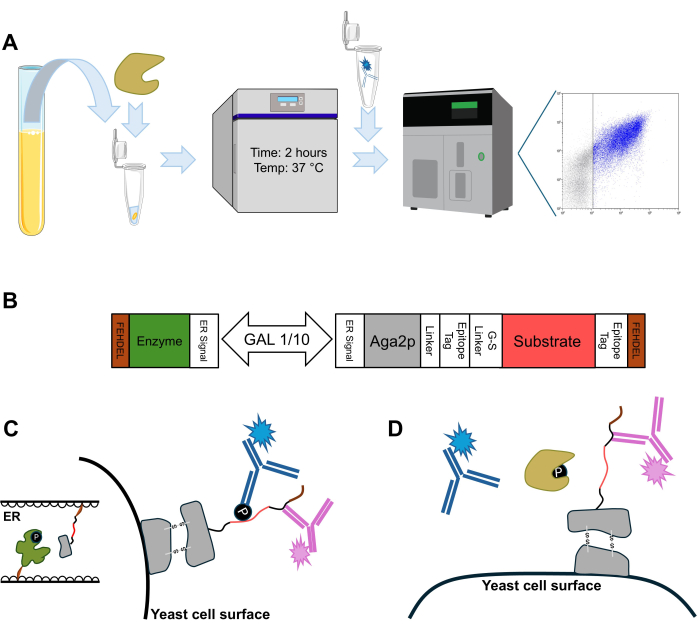
图 2:酵母表面显示蛋白质的酶促修饰。 (A) 测定示意图展示了所述四种方法中的关键步骤:用磷酸酶 (tan) 制备样品以进行酶促测定,孵育以获得所需的酶活性,细胞洗涤和标记以检测活性,以及流式细胞术分析和数据收集。(B) 基因示意图,代表用于酶-底物对的内质网隔离和底物表面表达的盒的一般结构。(C) 激酶-底物对的内质网共定位(左图)的图形表示,然后分泌翻译后修饰的底物,显示在酵母表面,并用抗磷酸酪氨酸抗体(蓝色)和抗表位标签抗体(粉红色)标记以确认表面表达(右图)。(D) 酵母细胞与磷酸酶 (tan) 一起孵育,从显示的底物中去除磷酸基团,破坏抗磷酸酪氨酸抗体标记,促进通过流式细胞术进行酶修饰分析。 请单击此处查看此图的较大版本。
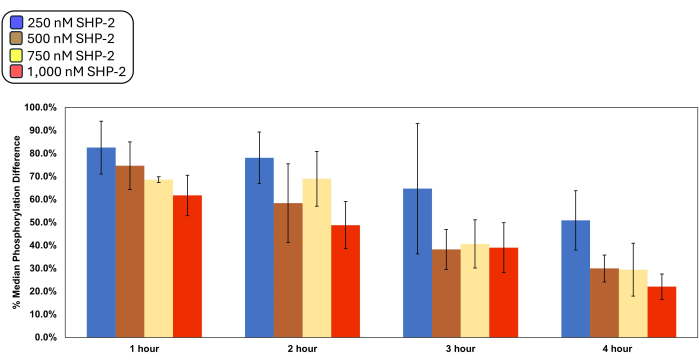
图 3:磷酸酶活性和时间滴定。 酵母细胞暴露于时间和磷酸酶浓度的多种组合中,然后进行流式细胞术分析。将所有处理过的样品与在相同时间和缓冲液条件下孵育的对照进行比较,不含 SHP-2。磷酸化中位数百分比差异定义为表面显示事件的 Y 中值减去含 SHP-2 的样品中非显示事件中 Y 中值提供的基线噪声信号的比率,除以各自对照中的相同关系。当使用双向方差分析比较时间组和浓度组之间的变化时观察到显著差异时,原假设被拒绝 (p < 0.05)。所描述的变量对中位磷酸化百分比差异的影响彼此独立(交互作用 p > 0.05)。进行 Tukey 的 HSD 检验进行事后分析,以进一步了解总孵育时间和浓度组之间差异的显著性,并使用一系列假设不等方差的 t 检验来定义各个组在指定时间和浓度下的统计显着性。数据以 4 次重复的平均值±标准差表示。 请单击此处查看此图的较大版本。
表 1:酵母生长和蛋白质诱导培养基制备指南。 以表格形式描述了配制 1 L 选择性酵母生长培养基、选择性酵母蛋白诱导培养基和酵母提取物蛋白胨葡萄糖培养基所需的每种化学成分所需的质量。一旦所述培养基正确混合,请在使用前进行过滤灭菌。包括制作选择性酵母生长培养基板的其他说明。 请点击此处下载此表格。
表 2:底物去磷酸化和抗体标记策略的推荐样品制备。 测量酵母表面显示的磷酸化底物的酶促修饰所需样品的表格描述。为所需的对照和要分析的每个样品指定了样品制备和后续抗体标记策略,包括标记试剂的相应稀释度。 请点击此处下载此表格。
表 3:抗体与蛋白质过量比率计算。 在标记用于流式细胞术时,在酵母细胞表面表达的每种蛋白质的理论可用抗体数量的表格描述。表达的理论数字假设 100% 的酵母细胞在其表面表达 10,000 个蛋白质,以确保抗体过量,并且过量比率的计算基于表中显示的试剂储备浓度,并从供应商处获得。 请点击此处下载此表格。
补充表 S1:构建盒的氨基酸序列。 位于 Gal 1-10 启动子两侧的构建盒的氨基酸序列的表格表示。突出显示的序列对应于其颜色编码的描述。质粒的 Gal-10 侧表示为反向氨基酸序列翻译,以促进其理解。所有以黑色表示的字符都对应于用于为构建体提供模块化的限制性内切酶消化位点的氨基酸翻译。 请点击此处下载此文件。
讨论
提出的方案允许使用酵母表面蛋白质的细胞外显示来分析酶相互作用。将内质网隔离液螯合到所使用的表面展示质粒中,引入了分析酶和翻译后修饰底物之间的细胞外特异性相互作用的能力,因为细胞内相互作用可以设计为发生27,29。先前建立的使用酵母表面显示的酶相互作用测定涉及目标蛋白质在细胞内的表达,表面显示仅作为检测靶蛋白之间发生的细胞内相互作用的工具 23,25,29。
该方案通过将靶向酶相互作用转移到细胞外环境来构建在该平台上,这在可以研究的酶和监测其活性的环境中引入了额外的灵活性。研究的细胞外发生的相互作用使研究人员有机会调整孵育环境,使其更适合酶活性,从而扩展可以研究的酶,这些酶的活性在酵母内质网(一个高度氧化的环境)中受到阻碍37。此外,相对于给定底物滴定酶浓度的能力允许进行特定的酶活性测定,由于螯合过程中假设的反应速率饱和,这些测定无法在细胞内进行。
在该协议中,需要注意几个关键步骤,以确保观察到预期的结果。将研究的构建体成功转化到酵母中对于以下步骤的最佳结果至关重要。应准确监测光密度,以跟踪培养物的健康生长,并确保它们在蛋白质诱导或样品制备之前不会过度生长。酵母细胞生长的对数期包括蛋白质产生量最高的时期,而在固定期,负责蛋白质产生的机制停滞不前38。牢记这一点,光密度测量可以准确测量酵母培养物所处的生长期,蛋白质诱导和检测准备等步骤应在固定相之外或培养物过度生长时进行(OD600nm < 6)。
对于酶修饰测定,所描述的孵育环境特定于所研究的酶 SHP-2 和正在进行的酶活性,即去磷酸化。DTT 用于它在与 SHP-234 孵育期间提供的还原环境。因此,在酶法测定中测量用于改变孵育环境的化学品的准确浓度非常重要,以确保样品和实验之间的酶活性一致。SHP-2 用作重组蛋白,在处理酶的不同步骤中调节温度至关重要。为了成功进行检测,酶不应经过超过两次冻融循环,并且在制备每个样品期间应置于冰上。然后,必须将重组酶分装成足够的体积以满足检测要求。在实际孵育过程中,需要将温度严格控制在酶的最佳温度,在本例中为 37 °C,转子不断移动,以确保孵育混合物的均匀性。
使用重组酶进行分析的总体方法需要对 SHP-2 与表面显示的磷酸化底物之间的相互作用进行特异性修饰。使方案适应其他细胞外酶 - 底物相互作用涉及修改使用的序列、活性缓冲液环境和用于检测的试剂。为了检测其他激酶-底物-磷酸酶相互作用,驯化包括将激酶-底物对的蛋白质序列替换到 补充表 S1 中描述的构建盒中的相应位置。底物的蛋白质序列以及目标激酶的至少激酶结构域应包含在质粒中,并且靶向产生的磷酸化底物的磷酸酶应为重组蛋白的形式。LCK、CD28 和 SHP-2 之间的代表性相互作用提供了一个示例,说明在构建盒中使用设计的内质网隔离作为生产翻译后修饰蛋白的工具,以便用它们的靶向酶在细胞外进行研究。不需要经过翻译后修饰的目标底物(例如,可以使用添加的激酶在细胞外磷酸化的底物)可以在酵母表面表达,而无需在构建盒内使用配对酶。在这种情况下, 补充表 S1 中描述的激酶的蛋白质序列将被去除,仅底物序列包含在构建盒中。我们确实从我们之前的经验中注意到,丝氨酸-苏氨酸激酶与已知底物的共定位导致底物未被磷酸化(Ezagui 和 Stern,未发表的数据),因此在应用细胞外磷酸酶之前,必须对成功的酶修饰进行严格的测试。我们之前已经发表了一种激酶 - 底物共定位的方案,这可能对这个合格的步骤39 有帮助。
激酶和磷酸酶通常包含不成对的半胱氨酸残基,这些残基被氧化时,在蛋白质内或跨蛋白质形成二硫键,这会由于构象变化而破坏蛋白质的催化活性40,41。了解这种蛋白质生物化学对于确定酶促修饰的适当反应环境至关重要。因此,需要在孵育环境中补充还原剂,以确保所用重组蛋白保持活性。DTT 是用于这些目的的常见还原剂,但必须优化测定中的浓度。使用过高的 DTT 浓度会阻碍底物在酵母表面的显示,因为 Aga1p 和 Aga2p 锚通过二硫键相互保持,在 DTT42 存在下二硫键会减少。将 DTT 的浓度调整为允许相对最大磷酸酶活性而不会对底物表面显示产生不利影响的最小浓度42。应优化所检测的任何酶的孵育环境,以确保在该检测中使用时保留酶活性。如果酶需要比该测定中使用的 0.5 mM DTT 强得多的还原环境,则该平台会受到表面显示减少的限制,并且可能不是所需的特定酶测定的最佳选择。同样,该方案中孵育步骤中使用的 2x 缓冲液来自先前的研究中,用于促进 SHP-2 活性的可接受缓冲液,并且应进行类似的研究来为用于该测定的任何其他酶配制孵育缓冲液34。制备这些缓冲液的起点可以包括文献搜索,以成功获得目标酶的体外应用,或酶制造商对活性缓冲液的推荐。所使用的重组酶应专门针对该测定进行滴定,以确定可接受的浓度和孵育时间,从而允许在数据收集之前进行靶向酶活性。
为了使该方案适应其他类型的酶-底物相互作用,需要选择新的荧光检测试剂并滴定以提高灵敏度。其他研究已经证明了这一点,包括使用表位标签靶向抗体检测蛋白酶处理后肽底物的存在与否23,25,以及使用乙酰化敏感抗体检测组蛋白修饰43。为了鉴定这些试剂,必须建立阳性对照(可验证地证明感兴趣的修饰的对照)和阴性对照(可验证地证明缺乏感兴趣的修饰的对照)。这可以通过先前已证明感兴趣的修饰的构建体的酵母表面展示来完成,或者在某些情况下可以通过固定重组蛋白或肽来实现。例如,在磷酸化(以及许多其他感兴趣的翻译后修饰)的情况下,已知序列的肽可以用磷酸酪氨酸(阳性对照)或未修饰的酪氨酸(阴性对照)和 C 端生物素合成,这将使肽能够固定在链霉亲和素包被的珠子上。肽包被的微珠可以用修饰特异性抗体标记,并使用流式细胞术使用类似于第 4 节中描述的方法评估检测的特异性和灵敏度。应使用不同的抗体稀释液来找到一种浓度,该浓度可提供阳性对照的最大信号、阴性对照的最小信号,并平衡每种修饰蛋白的抗体的足够倍数(使用步骤 4.1 中注释中的方程)与经济考虑每等分试样获得的抗体进行实验的数量。
我们描述了一种方案,用于使酵母表面显示平台的易用性用于细胞外酶活性测定。该方法使用酵母表面显示的磷酸化 CD28 在孵育过程中被重组 SHP-2 去磷酸化来证明,但通过修饰所使用的工作缓冲液和酶-底物对,该方法可推广用于多种类型的酶修饰。
披露声明
作者没有与本作品相关的利益冲突需要披露。
致谢
这项工作得到了 L.A.S. (CBET - 2339172) 的 NSF CAREER 奖和南佛罗里达大学的启动基金的支持。
在 图 2A 中,Servier https://smart.servier.com/ 的 microtube-open-translucent 图标根据 CC-BY 3.0 Unported https://creativecommons.org/licenses/by/3.0/ 获得许可。修饰包括添加缓冲液和酵母细胞(左)和添加抗体(中右)。
图 2A 中的试管、培养箱和流式细胞仪是通过 www.bioicons.com 在开放获取下提供的。
材料
| Name | Company | Catalog Number | Comments |
| 1 L Media Bottles | Corning | 06-414-1D | |
| 1.7/2.0 mL Microtubes | Axygen | MCT-175-C | |
| 10 µL SureOne Pipet Tips | Fisher Scientific | 02-707-438 | |
| 1000 µL SureOne Pipet Tips | Fisher Scientific | 02-707-408 | |
| 12 mL Polystyrene Round-Bottom Tubes | Greiner | 07-000-212 | |
| 3 mL platic Cuvettes | BRAND | 759076D | |
| 300 µL SureOne Pipet Tips | Fisher Scientific | 02-707-411 | |
| 5 mL Serological Pipettes | Fisher Scientific | 13-678-11D | |
| Acid Casein (Casamino Acids) | Fisher Scientific | BP-1424-500 | |
| Analytical Balance | Mettler Toledo | 30243397 | |
| Bacteriological Petri Dish | Corning | Falcon 351008 | |
| Biosafety Cabinets | Labconco | Logic Class II, Type A2 302310102 | |
| Biospectrometer | Eppendorf | Kinetic 6136000010 | |
| Bovine Serum Albumin | Fisher bioreagents | BP1600-100 | |
| Citric Acid | Fisher Scientific | A940-500 | |
| CytoFLEX Flow Cytometry Analyzer | Beckam Coulter | Cytoflex C09745 | CytExpert software |
| Dextrose | Fisher Scientific | D16-1 | |
| Dithiothreitol | Fisher bioreagents | BP172-5 | |
| Donkey anti-goat FITC | Invitrogen | A16000 | |
| EDTA | Alfa Aesar | H56165.30 | |
| Ez-Link PEG4-NHS-Biotin | Thermo Scientific | A39259 | |
| Frozen-EZ Yeast Transformation II Kit | Zymo Research | T2001 | |
| Galactose | Fisher Scientific | BP656-500 | |
| General Purpose Refrigerator | Marvel Scientific | MS24RAS4RW | |
| Goat anti-myc tag antibody | Bethyl | A190-104A | |
| Mictrotube Centrifuge | Eppendorf | 5425 R 5406000313 | |
| Mini Low Temperature Refrigerated Incubator | Fisher Scientific | 15-015-2632 | |
| Mouse anti-phosphotyrosine antibody 4G10 | BioXcell | BE0194 | |
| Parafilm M | Bemis | M PM999 | |
| Phosphate Buffered Saline | Fisher bioreagents | BP399-500 | |
| Pipette Controller | Eppendorf | easypet 3 4430000018 | |
| Raffinose | Thermo Scientific | J21060-36 | |
| Recombinant human Active SHP-2 Protein | R&D Systems | 1894-SH | |
| Refrigerated Centrifuge | Eppendorf | 5910 R | |
| Saccharomyces cerevisiae yeast surface display strain EBY 100 | ATCC | MYA-4941 | |
| Shaker Incubator | Eppendorf | M1335-0002 New Brunswick Innova 42 | |
| Single Channel Pipette Set | Eppendorf | 05-403-151 | |
| Sodium Chloride | Fisher Scientific | S671-500 | |
| Sodium Citrate Dihydrate | Fisher Scientific | S279-500 | |
| Sodium Phosphate Dibasic Heptahydrate | Fisher Scientific | S373-500 | |
| Sodium Phosphate Monobasic Monohydrate | Fisher Scientific | S468-500 | |
| Streptavidin Alexa Fluor 647 | Invitrogen | S32357 | |
| Top Loading Balance | Mettler Toledo | ||
| Tris hydrochloride | EMD Millipore | 648317-100GM | |
| Tube revolver rotator | Fisher Scientific | 11-676-341 | |
| Weighing Paper | Fisher Scientific | 09-898-12B | |
| Yeast Nitrogen Base | BD Difco | 291940 | |
| Zeba Spin Desalting Columns | Thermo Scientific | 89883 |
参考文献
- Lea, M. A., Weber, G. Role of enzymes in homeostasis: VIII. Inhibition of the activity of glycolytic enzymes by free fatty acids. J Biol Chem. 243 (6), 1096-1102 (1968).
- Mahé, M., Rios-Fuller, T. J., Karolin, A., Schneider, R. J. Genetics of enzymatic dysfunctions in metabolic disorders and cancer. Front Oncol. 13, 1230934(2023).
- Fernandez-de-Cossio-Diaz, J., Vazquez, A. A physical model of cell metabolism. Sci Rep. 8 (1), 8349(2018).
- Metallo, C. M., Vander Heiden, M. G. Understanding metabolic regulation and its influence on cell physiology. Mol Cell. 49 (3), 388-398 (2013).
- Mildvan, A. S. Mechanisms of signaling and related enzymes. Proteins. 29 (4), 401-416 (1997).
- Frosina, G. Overexpression of enzymes that repair endogenous damage to DNA. Eur J Biochem. 267 (8), 2135-2149 (2000).
- Schärer, O. D. Chemistry and biology of DNA repair. Angew Chem Int Ed. 42 (26), 2946-2974 (2003).
- de la Fuente, M., et al. Enzyme therapy: Current challenges and future perspectives. Int J Mol Sci. 22 (17), 9181(2021).
- Robertson, J. G. Enzymes as a special class of therapeutic target: clinical drugs and modes of action. Curr Opin Struct Biol. 17 (6), 674-679 (2007).
- Goddard, J. -P., Reymond, J. -L. Enzyme assays for high-throughput screening. Curr Opin Biotechnol. 15 (4), 314-322 (2004).
- Helm, J. S., Hu, Y., Chen, L., Gross, B., Walker, S. Identification of active-site inhibitors of MurG using a generalizable, high-throughput glycosyltransferase screen. J Am Chem Soc. 125 (37), 11168-11169 (2003).
- Veldhuyzen, W. F., Nguyen, Q., McMaster, G., Lawrence, D. S. A light-activated probe of intracellular protein kinase activity. J Am Chem Soc. 125 (44), 13358-13359 (2003).
- Torres, M., Forman, H. J. Encyclopedia of Respiratory. Laurent, G. J., Shapiro, S. D. , Academic Press. 10-18 (2006).
- Blume-Jensen, P., Hunter, T. Oncogenic kinase signalling. Nature. 411 (6835), 355-365 (2001).
- Martin, G. S. Cell signaling and cancer. Cancer Cell. 4 (3), 167-174 (2003).
- Lothrop, A. P., Torres, M. P., Fuchs, S. M. Deciphering post-translational modification codes. FEBS Lett. 587 (8), 1247-1257 (2013).
- Graves, J. D., Krebs, E. G. Protein phosphorylation and signal transduction. Pharmacol Ther. 82 (2), 111-121 (1999).
- Hafen, E. Kinases and phosphatases--A marriage is consummated. Science. 280 (5367), 1212-1213 (1998).
- Westphal, R. S., Anderson, K. A., Means, A. R., Wadzinski, B. E. A signaling complex of Ca2+-calmodulin-dependent protein kinase IV and protein phosphatase 2A. Science. 280 (5367), 1258-1261 (1998).
- Barford, D., Das, A. K., Egloff, M. -P. The structure and mechanism of protein phosphatases: Insights into catalysis and regulation. Annu Rev Biophys Biomol Struct. 27, 133-164 (1998).
- Liu, Q., Qu, J., Zhao, M., Xu, Q., Sun, Y. Targeting SHP2 as a promising strategy for cancer immunotherapy. Pharmacol Res. 152, 104595(2020).
- Pan, J., Zhou, L., Zhang, C., Xu, Q., Sun, Y. Targeting protein phosphatases for the treatment of inflammation-related diseases: From signaling to therapy. Signal Transduct Targeted Ther. 7 (1), 177(2022).
- Denard, C. A., et al. YESS 2.0, a tunable platform for enzyme evolution, yields highly active TEV protease variants. ACS Synth Biol. 10 (1), 63-71 (2021).
- Lim, S., Glasgow, J. E., Filsinger Interrante, M., Storm, E. M., Cochran, J. R. Dual display of proteins on the yeast cell surface simplifies quantification of binding interactions and enzymatic bioconjugation reactions. Biotechnol J. 12 (5), (2017).
- Yi, L., et al. Engineering of TEV protease variants by yeast ER sequestration screening (YESS) of combinatorial libraries. Proc Natl Acad Sci USA. 110 (18), 7229-7234 (2013).
- Yi, L., et al. Yeast endoplasmic reticulum sequestration screening for the engineering of proteases from libraries expressed in yeast. Methods Mol Biol. 1319, 81-93 (2015).
- Semenza, J. C., Hardwick, K. G., Dean, N., Pelham, H. R. ERD2, a yeast gene required for the receptor-mediated retrieval of luminal ER proteins from the secretory pathway. Cell. 61 (7), 1349-1357 (1990).
- Mei, M., et al. Characterization of aromatic residue-controlled protein retention in the endoplasmic reticulum of Saccharomyces cerevisiae. J Biol Chem. 292 (50), 20707-20719 (2017).
- Ezagui, J., Russell, B., Mairena, Y., Stern, L. A. Endoplasmic reticulum sequestration empowers phosphorylation profiling on the yeast surface. AIChE J. 68 (12), e17931(2022).
- Kawai, S., Murata, K. Genetic Transformation Systems in Fungi. van den Berg, M. A., Maruthachalam, K. 1, Springer International Publishing. 187-192 (2015).
- Kawai, S., Hashimoto, W., Murata, K. Transformation of Saccharomyces cerevisiae and other fungi: methods and possible underlying mechanism. Bioeng Bugs. 1 (6), 395-403 (2010).
- Loock, M., et al. High-efficiency transformation and expression of genomic libraries in yeast. Methods Protoc. 6 (5), 89(2023).
- Huang, D., Gore, P. R., Shusta, E. V. Increasing yeast secretion of heterologous proteins by regulating expression rates and post-secretory loss. Biotechnol Bioeng. 101 (6), 1264-1275 (2008).
- Yu, B., et al. Targeting protein tyrosine phosphatase SHP2 for the treatment of PTPN11-associated malignancies. Mol Cancer Ther. 12 (9), 1738-1748 (2013).
- Stern, L. A., et al. Geometry and expression enhance enrichment of functional yeast-displayed ligands via cell panning. Biotechnol Bioeng. 113 (11), 2328-2341 (2016).
- Pan, X., et al. Optimized single-cell gates for yeast display screening. Protein Eng Design Sel. 38, gzae018(2025).
- Margittai, É, et al. Production of H2O2 in the endoplasmic reticulum promotes in vivo disulfide bond formation. Antioxid Redox Signal. 16 (10), 1088-1099 (2012).
- Werner-Washburne, M., Braun, E., Johnston, G. C., Singer, R. A. Stationary phase in the yeast Saccharomyces cerevisiae. Microbiol Rev. 57 (2), 383-401 (1993).
- Ezagui, J., Stern, L. A. Tyrosine phosphorylation screening on the yeast surface by magnetic bead selection and FACS. Methods Mol Biol. 2681, 275-290 (2023).
- Yarnall, M. T. N., Kim, S. H., Korntner, S., Bishop, A. C. Destabilization of the SHP2 and SHP1 protein tyrosine phosphatase domains by a non-conserved “backdoor” cysteine. Biochem Biophys Rep. 32, 101370(2022).
- Dustin, C. M., Heppner, D. E., Lin, M. J., van der Vliet, A. Redox regulation of tyrosine kinase signalling: more than meets the eye. J Biochem. 167 (2), 151-163 (2020).
- Stern, L. A., Csizmar, C. M., Woldring, D. R., Wagner, C. R., Hackel, B. J. Titratable avidity reduction enhances affinity discrimination in mammalian cellular selections of yeast-displayed ligands. ACS Comb Sci. 19 (5), 315-323 (2017).
- Waldman, A. C., Rao, B. M., Keung, A. J. Mapping the residue specificities of epigenome enzymes by yeast surface display. Cell Chem Biol. 28 (12), 1772-1779.e4 (2021).
转载和许可
请求许可使用此 JoVE 文章的文本或图形
请求许可探索更多文章
This article has been published
Video Coming Soon
版权所属 © 2025 MyJoVE 公司版权所有,本公司不涉及任何医疗业务和医疗服务。