Method Article
固形腫瘍の予知免疫モデリング
要約
RNAベースのアプローチを使用して、固形腫瘍組織の定量的免疫プロファイルを決定し、免疫腫瘍学バイオマーカー探索のための臨床コホートを活用することは、分子および情報学的プロトコルを介して記述されています。
要約
免疫療法は腫瘍学患者の治療において約束を示すが、腫瘍微小環境の複雑な不均一性は治療応答の予測を困難にする。腫瘍組織とその周辺に存在する免疫細胞の相対的集団を解決する能力は、臨床的に理解応答に関連することが示されているが、フローサイトメトリーおよび免疫組織化学などの伝統的な技術によって制限されている(IHC)は、必要な組織の大量性、正確な細胞型マーカーの欠如、および多くの技術的および物流上のハードルのために。1つのアッセイ(例えば、免疫プリズムプロファイリングアッセイ)は、少量のRNAと高度に分解されたRNAの両方に対応することによってこれらの課題を克服し、臨床的にアーカイブされた固形腫瘍組織から抽出されたRNAの共通の特徴である。このアッセイは、試薬キットとクラウドベースのインフォマティクスを介してアクセスされ、Illuminaシーケンシングプラットフォーム向けのエンドツーエンドの定量的、ハイスループットな免疫プロファイリングソリューションを提供します。研究者は、ホルマリン固定パラフィン埋め込み(FFPE)組織の2つのセクションまたは(サンプルの品質に応じて)総RNAの20〜40ngから始まり、プロトコルは8つの免疫細胞タイプと10の免疫脱出を定量化する免疫プロファイルレポートを生成します遺伝子は、腫瘍微小環境の完全なビューを捕捉する。得られたデータを利用するために追加のバイオインフォマティクス分析は必要ありません。適切なサンプルコホートを使用すると、プロトコルはまた、関心のある患者集団内の統計的に有意なバイオマーカーを識別するために使用され得る。
概要
ホルマリン固定およびパラフィン埋込み(FFPE)のヒト組織サンプルにおける腫瘍浸潤リンパ球(TILs)および他の免疫関連分子の定量化は、臨床研究1、2、3において示された値を示している。フローサイトメトリーや単細胞リボ核酸(RNA)シーケンシングなどの一般的な技術は、新鮮な組織および血液4に有用であるが、生存可能な細胞懸濁液を作ることができないため、FFPE材料の分析には適さない。FFPE組織においてこれらの細胞を定量化するために使用されている現在の方法は、大きな課題に苦しんでいます。免疫組織化学(IHC)および他の同様のイメージングワークフローは、細胞表面タンパク質を検出するための特異的抗体を必要とし、再現性定量を可能にするために実験室間で標準化することは困難である可能性があります 5。nCounterシステムのようなプラットホームは、検出の感度および特異性を制限する主要な免疫細胞6を定義するために単一の遺伝子の発現に依存する。スタンドアロンのソフトウェアツールと組み合わせたより一般的なRNAシーケンシング方法は、利用可能ですが、7、8、9、10、11、12を使用する前に、大幅な最適化と検証が必要です。レーザー捕捉マイクロ解剖(LCM)とFFPE組織のRNAシーケンシングを組み合わせた最近の進歩は、約束を示しています。しかし、より高いスループット、ターンキーソリューションは、堅牢なバイオマーカー13、14を同定することを目的としたトランスレーショナル研究に必要とされる。多次元バイオマーカーを生成する方法は、予測免疫モデリング、治療応答者、癌サブタイプ、または高い予測精度および統計的有意性を有する生存結果を含む患者コホートを定義する15、16の精密医療の時代においてますます重要になってきている。
このニーズに対応するために、標準化されたRNAシーケンシング試薬および雲ベースのインフォマティクスを使用して、固形腫瘍FFPE組織における免疫細胞の感受性および特異的定量を可能にする免疫プロファイリングアッセイが開発されました。FFPE組織から分解されたRNAを収容することに加えて、プロトコルは、コア針生検、針吸引、およびミクロまたはマクロ解剖組織などの制限組織サンプルから得られるRNAを収容することができる。各サンプルからのRNAデータは、免疫細胞の遺伝子発現モデルのデータベースと比較され、免疫健康発現モデルと呼ばれ、免疫細胞をサンプル中に存在する全細胞の割合として定量する。簡単に言えば、これらのモデルは、機械学習法を用いて構築され、精製免疫細胞集団(正規細胞表面マーカーを用いて単離された)から生成された全トランスクリプトームデータからユニークな多原性発現パターンを同定した(正規細胞表面マーカーを用いて分離された)17,18。この技術の基礎となる多次元健康発現モデルにより、アッセイは異種混合物中に存在する全細胞のパーセントとして各免疫細胞を定量化することができます。これにより、研究者は、臨床値19、20を有することが示されているインターおよびイントラサンプル免疫細胞比較を生成することができます。その他の用途としては、代表的な結果に記載されているように、治療前および後の免疫応答の定量が挙げられる。アッセイは、8種類の免疫細胞型(遺伝子発現モデル由来)の絶対割合を含む腫瘍および腫瘍微小環境の免疫コンテクスチャの複数の特徴について報告する:CD4+ T細胞、CD8+ T細胞、CD56+ナチュラルキラー細胞、CD19+B細胞、CD14+単球、Tregs、M1マクロファージ、およびM2マクロファージ。 さらに、アッセイは、10個の免疫脱出遺伝子(PD-1、PD-L1、CTLA4、OX40、TIM-3、BTLA、ICOS、CD47、IDO1、およびARG1)の発現(100万回当たりのトランスクリプト、またはTPM)を報告する。
この試薬キットは、図 1に示すように、ハイブリッド キャプチャ ベースのライブラリ準備方法に従って、高品質のライブラリを、イユミナ プラットフォームでシーケンス処理の準備を整えるために使用します。研究者が実験室にIlluminaシーケンシングプラットフォームを持っていない場合、彼らはシーケンシングのためにコアラボにサンプルを提出することができます。生成されると、シーケンスデータは、自動化された分析のためにPrism Portalにアップロードされ、個々のサンプルに対する包括的で定量的なプロファイルを、免疫レポート(図2A)の形でユーザに返されます。ユーザーは、プリズムポータルでサンプルグループを定義してバイオマーカーレポート(図2B)を生成し、2つの患者コホートを区別する統計的に有意なバイオマーカーを強調することもできる。重要なことに、試薬キットによって生成されたデータは研究用のみであり、診断目的で使用することはできません。

図1:ワークフローの概要このプロトコルでは、RNAはまずcDNAに変換される。シーケンシングアダプタは、リゲーションされ、アダプター-リゲーテッドcDNAを増幅し、PCRによってバーコード化してプリキャプチャライブラリを作成します。ビオチン化プローブは、特定のcDNA標的にハイブリダイズされ、ストレプトアビジンビーズを使用して捕捉されます。非結合、非標的cDNAは洗浄によって除去される。最終的な PCR エンリッチメントにより、キャプチャ後のライブラリをシーケンス処理の準備ができます。*総RNAはヒトサンプルからのものでなければなりません。無傷または分解(FFPE)RNAである可能性があります。この図の大きなバージョンを表示するには、ここをクリックしてください。

図2:代表免疫報告ワークフローは、処理された各サンプルに対する個別の免疫レポート(A)と、定義された患者コホートのバイオマーカーレポート(B)の2つのレポートを生成します。この図の大きなバージョンを表示するには、ここをクリックしてください。
このプロトコルは、約16時間の準備時間(全RNAからシーケンシングの準備ができたライブラリまで)を必要とします。ただし、プロトコルに記載されているように、いくつかのオプションの停止点があります。このアッセイは、トランスクリプトミクスの豊かでダイナミックな性質を利用して、従来の単一分析体バイオマーカーを超えて多次元遺伝子発現モデルに移行し、標準化された組織サンプルの包括的な生物学的特徴付けを可能にします。試薬および使いやすいソフトウェア用具。機械学習と健康発現モデルのデータベースを活用して、貴重な臨床サンプルのより正確で定量的な免疫プロファイルを導き出し、発見することで、研究者は自分の研究室で現代的な技術を活用することができます。完全な統計解析を用いて多次元RNAバイオマーカー。
プロトコル
ここに示す代表的な結果で利用されたヒト組織サンプルは、評判の良いエンティティ(TriStarテクノロジーグループ)から購入され、学術的および商業的研究を可能にするドナーの同意を得ただけでなく、有能な倫理からの承認を得ました委員 会。
パート I: プリキャプチャ ライブラリの準備
1. RNAの定量と資格
- フルオロメトリックアッセイを用いてRNAを定量し、アッセイへの適切な入力を決定する。電気泳動を使用して入力 RNA の品質を評価し、RNA インテグリティ数 (RIN) とフラグメントの割合を決定 > 200 ヌクレオチド (DV200)値。
- インタクト(RIN > 7)または部分的に分解されたRNAサンプル(RIN = 2〜7)の場合は、ステップ2.1から始めて、高品質/インタクトRNAのライブラリ調製手順に従います。RNAの品質は、サーマルサイクラープログラム#1(補足表2)で正しい断片化時間を選択するために重要です。
- 非常に劣化したサンプル(例えば、RIN = 1〜2またはFFPE)の場合、DV200値を決定します。これらのサンプルは断片化を必要とせず、ステップ 2.2 から始まる分解 RNA の指示に従います。
- 20 ng の RNA (高品質/インタクト RNA と RIN > 2) または 40 ng の RNA (DV 200 で分解/FFPE RNA を20%で) を希釈して、各サンプルのサンプルの適切な量の RNA を準備します。ヌクレアーゼフリー水で5μLに。DV200 < 20% でサンプルを処理することはお勧めしません。キットに付属のコントロールRNAサンプルの場合、適切なRNAの1μLをヌクレアーゼフリー水の4μLで希釈します。コントロールサンプルは、標識された高品質(Intact)または分解(FFPE)RNA材料に関して記述されたのと同じ処理に従います。キットに含まれるすべての試薬については、補足表 1を参照してください。
2. RNAの断片化とプライミング
- RIN > 2 で高品質/インタクト RNA を使用する場合は、ステップ 2.1.1 に従ってください。
- 高品質なRNAの場合、表1に従って、ヌクレアーゼフリーPCRチューブで氷上で断片化およびプライミング反応を組み立てます。
- 数回上下にピペットで混ぜ合わせます。その後、マイクロ遠心分離機でサンプルを短時間スピンダウンします。
注: プロトコルのすべての遠心回転では、最低 3 s の場合は 1,000 x g以上の速度が推奨されます。 - サンプルをサーマルサイクラーに入れ、プログラム#1 (補足表 2)を使用します。
- すぐにチューブを氷に移し、高品質RNA用の第1鎖cDNA合成に進みます(ステップ3.1)。高品質と FFPE RNA の両方の同時準備のために、断片化インキュベーション中に FFPE RNA (ステップ 2.2) の準備を開始します。
- 数回上下にピペットで混ぜ合わせます。その後、マイクロ遠心分離機でサンプルを短時間スピンダウンします。
- 高品質なRNAの場合、表1に従って、ヌクレアーゼフリーPCRチューブで氷上で断片化およびプライミング反応を組み立てます。
| フラグメント化とプライミングミックス | ボリューム(μL) |
| 無傷または部分的に分解された RNA (20 ng) | 5 |
| 第1鎖合成反応バッファー | 4 |
| ランダムプライマー | 1 |
| 総体積 | 10 |
表1:高品質RNAの断片化とプライミング反応高品質RNAの断片化およびプライミング反応の成分は、示されている量に応じて氷上で組み立てられ、混合されるべきである。第一鎖合成反応バッファーとランダムプライマーのマスターミックスを作り、RNAサンプルに加えることができる。
- DV 200 で分解/FFPE RNA を20%と分解するには、ステップ 2.2.1 に従ってください。
- 断片化を必要としない高度に分解された(FFPE)RNAの場合、表2に記載されているようにプライミング反応を組み立てます。インタクト RNA の場合は、必ずステップ 2.1 に従ってください。
- 数回上下にピペットで混ぜ合わせます。その後、マイクロ遠心分離機でサンプルを短時間スピンダウンします。
- サンプルをサーマルサイクラーに入れ、プログラム#2 (補足表 2)を使用します。
- チューブを氷に移し、分解性の高い(FFPE)RNAの第1鎖cDNA合成に進みます(ステップ3.2)。
- 断片化を必要としない高度に分解された(FFPE)RNAの場合、表2に記載されているようにプライミング反応を組み立てます。インタクト RNA の場合は、必ずステップ 2.1 に従ってください。
| プライミング反応 | ボリューム(μL) |
| FFPE RNA (40 ng) | 5 |
| ランダムプライマー | 1 |
| 総体積 | 6 |
表2:高分解RNAに対するランダムプライミング反応高度に分解されたRNAのためのプライミング反応の成分は、ヌクレアーゼフリーPCRチューブで氷上に組み立てる必要があります。
3. 第1鎖cDNA合成
- RIN > 2 で高品質/インタクト RNA を使用する場合は、ステップ 3.1.1 に従ってください。
- インタクトRNA(高品質)の場合、表3に従って、ヌクレアーゼフリーPCRチューブで氷上で第1の鎖合成反応を組み立てます。
- 氷の上で反応を維持し、数回上下にピペットで完全に混ぜます。サンプルを微小遠心分離機で簡単にスピンダウンし、直接第1鎖合成インキュベーションに進みます(ステップ4)。
- インタクトRNA(高品質)の場合、表3に従って、ヌクレアーゼフリーPCRチューブで氷上で第1の鎖合成反応を組み立てます。
| 第1のストランド合成 | ボリューム(μL) |
| フラグメント化およびプライミング RNA (ステップ 2.1.3) | 10 |
| 第1鎖合成特異性試薬 | 8 |
| ファーストストランド合成酵素ミックス | 2 |
| 総体積 | 20 |
表3:高品質RNAに対する第1の鎖合成反応高品質RNAの断片化およびプライミング反応の成分は、与えられた量に応じて氷の上に組み立てられ、混合されるべきである。第1鎖合成特異性試薬と第1鎖合成酵素ミックスのマスターミックスを作り、断片化した、プライミングされたRNAサンプルに添加することができます。
- DV 200 で分解/FFPE RNA を20%と分解するには、ステップ 3.2.1 に従ってください。
- 高分解RNA(FFPE)の場合、表4に従って、ヌクレアーゼフリーPCRチューブで氷上で第1の鎖合成反応を組み立てる。
- 氷の上で反応を維持し、数回上下にピペットで完全に混ぜます。サンプルを微小遠心分離機で簡単にスピンダウンし、直接第1鎖合成インキュベーションに進みます(ステップ4)。
- 高分解RNA(FFPE)の場合、表4に従って、ヌクレアーゼフリーPCRチューブで氷上で第1の鎖合成反応を組み立てる。
| 第1のストランド合成 | ボリューム(μL) |
| プライミング RNA (ステップ 2.2.3) | 6 |
| 第1鎖合成反応バッファー | 4 |
| 第1鎖特異性試薬 | 8 |
| ファーストストランド合成酵素ミックス | 2 |
| 総体積 | 20 |
表4:高分解RNAに対する第1の鎖合成反応。高度に分解されたRNAの断片化およびプライミング反応の成分は、示された体積に応じて氷上に組み立てられ、混合されるべきである。第1鎖合成反応バッファー、第1鎖合成特異性試薬、第1鎖合成酵素ミックスのマスターミックスを作り、プライミングRNAサンプルに添加することができます。
4. 第一鎖合成インキュベーション
- チューブを氷の上に置いておき、数回上下にピペットで混ぜ合わせます。マイクロ遠心分離機でサンプルを簡単にスピンダウンします。プログラム#3(補足表2)に続いて、予熱熱サイクラーでサンプルをインキュベートします。
5. 第二鎖cDNA合成
- 第2の鎖cDNA合成反応を、表5に記載された成分を組み立てて、ステップ4.1から第1の鎖反応生成物を含めて調製する。
| 第二鎖合成反応 | ボリューム(μL) |
| 第1ストランド合成製品(ステップ4.1) | 20 |
| 第二鎖合成反応バッファー | 8 |
| 第二鎖合成酵素ミックス | 4 |
| ヌクレアーゼフリーの水 | 48 |
| 総体積 | 80 |
表5:第2鎖合成反応。第2鎖cDNA合成反応の成分は、図に示す体積に応じて、氷上で組み立てられ、混合されるべきである。第2鎖合成反応バッファー、第2鎖合成酵素ミックス、ヌクレアーゼフリー水のマスターミックスを作り、第1鎖合成産物に添加することができます。
- チューブを氷の上に置いておき、数回上下にピペットで混ぜ合わせます。プログラム#4(補足表2)に続いてサーマルサイクラーでインキュベートします。
6. SPRI(固相可逆固定固定)ビーズを用いたcDNAクリーンアップ
- SPRIビーズを使用前に少なくとも30分間室温まで温め、約30sの渦SPRIビーズを再サスペンドします。
- 第2鎖合成反応に144μLの再懸濁ビーズを加える(約80μL)。少なくとも10回上下にピペットで混ぜ、室温で5分間インキュベートします。
- マイクロ遠心分離機でチューブを短く回転させ、チューブを磁気ラックに置き、ビーズと上清を分離します。溶液が明確になってから、慎重に除去し、上清を廃棄します。DNAを含むビーズを邪魔しないように注意してください。
- 磁気ラックの上で、作製した80%エタノールをチューブに180μL加えます。室温で30sをインキュベートし、慎重に除去し、上清を廃棄します。
- 合計 2 つの洗浄手順に対して、手順 6.4 を 1 回繰り返します。
- 残留エタノールを完全に除去します。磁気ラックにチューブを残し、蓋を開けた状態で約3分間、または目に見えて乾燥するまでビーズを乾燥させます。これはDNAの回収量が低下する可能性があるため、ビーズを乾燥させないでください。
- 磁石からチューブを取り出し、ビーズに53 μL 0.1x TEバッファ(試薬キットに含まれるもの、補足表1を参照)を加えます。ピペットを少なくとも10回上下に混ぜ、十分に混ぜます。室温で2分間インキュベートします。
- チューブを磁気ラックに置き、ビーズを上清から完全に分離できるようにします。清浄ヌクレアーゼフリーPCRチューブに上清の50 μLを移します。ビーズを邪魔しないように注意してください。これはプロトコル中の任意の停止点であり、cDNAサンプルは-20°Cで保存され得る。
7. cDNAライブラリの修復の終了
- 表6に記載されている成分をステップ6.8から第2の鎖合成産物に組み立てることにより、氷上での終期修復反応を組み立てます。
| 終了修復反応 | ボリューム(μL) |
| 第二鎖合成製品(ステップ6.8) | 50 |
| 終了修復リアクション バッファ | 7 |
| エンドリペア酵素ミックス | 3 |
| 総体積 | 60 |
表6:修復反応を終了する。エンド修復反応の成分は、示されているボリュームに応じて、組み立て、氷上に混合する必要があります。エンドリペア反応バッファーとエンドリペア酵素ミックスのマスターミックスを作り、第二鎖合成製品に添加することができます。
- ピペットを50μLにセットし、ボリューム全体を少なくとも10回上下にピペットにして十分に混ぜます。チューブの側面からすべての液体を収集するために簡単に遠心分離します。よく混ぜることが大切です。少量の気泡の存在は、パフォーマンスを妨げない。
- プログラム#5(補足表2)に続いて、サーマルサイクラーでサンプルをインキュベートします。
8. アダプターのライゲーション
- ライゲーション反応を設定する前に、表7に示すように、氷冷アダプター希釈バッファーでアダプターを希釈し、必要なサンプル数を掛け、さらに10%余分に加えた。希釈されたアダプターを氷の上に置いておく。
| ライゲーション希釈 | ボリューム(μL) |
| アダプター | 0.5 |
| アダプター希釈バッファー | 2 |
| 総体積 | 2.5 |
表 7: アダプター希釈.アダプタは、示されているボリュームに従って、アダプタ希釈バッファーを使用して氷上で希釈する必要があります。
- 表8に記載されている成分を、記載されている順序で、ステップ7.3から終末プレップ反応産物に添加して氷上でライゲーション反応を組み立てる。なお、ライゲーションマスターミックスとライゲーションエンハンサーは、事前に混合することができます。この混合物は、4°Cで少なくとも8時間安定である。アダプターのライゲーションステップで使用する前に、ライゲーションマスターミックス、ライゲーションエンハンサーおよびアダプターをプリミックスしないでください。
| ライゲーション反応 | ボリューム(μL) |
| エンドプレップDNA(ステップ7.3) | 60 |
| 希釈アダプター (ステップ 8.1) | 2.5 |
| ライゲーションエンハンサー | 1 |
| ライゲーションマスターミックス | 30 |
| 総体積 | 93.5 |
表8:ライゲーション反応。アダプターライゲーション反応の成分は、示されている順序で示された体積に従って氷上で組み立てる必要があります。ライゲーションエンハンサーとライゲーションマスターミックスのマスターミックスを作り、希釈されたアダプターでエンドプレップDNAに追加することができます。希釈されたアダプターとライゲーションマスターミックスまたはライゲーションエンハンサーを混合してからエンドプレップDNAと混合しないでください。
- ピペットを80μLにセットし、ボリューム全体を少なくとも10回上下にピペットにして十分に混ぜます。チューブの側面からすべての液体を収集するために迅速なスピンを実行します。ライゲーションマスターミックスは非常に粘性があります。不完全な混合はライゲーション効率の低下をもたらすので、ライゲーション反応の適切な混合を確保するように注意してください。少量の気泡の存在は、パフォーマンスを妨げない。
- プログラム#6 (補足表 2)をインキュベートし、サーマルサイクラーからライゲーション混合物を取り出し、3 μL のアダプター処理酵素を加え、合計容量 96.5 μL を得ました。
- ピペットを数回上下に何度か混合し、プログラム#7(補足表2)に従ってインキュベートしてから直ちにライゲーション反応の精製に進みます。
9. SPRIネズを用いたライゲーション反応の精製
- SPRIビーズを使用前に少なくとも30分間室温まで温め、約30分間渦SPRIビーズを再サスペンドします。
- 87 μLの再懸濁SPRIビーズを加え、少なくとも10回上下にピペットで混ぜます。室温で10分間インキュベートします。
- マイクロ遠心分離機でチューブを短く回転させ、チューブを磁気ラックに置き、ビーズと上清を分離します。溶液が明らかになった後(〜5分)、慎重に除去し、上清を廃棄します。ビーズを捨てないでください。
- 磁気ラックの上で、作製した80%エタノールをチューブに180μL加えます。室温で30sをインキュベートし、慎重に除去し、上清を廃棄します。合計 2 つの洗浄手順に対して、ステップ 9.4 を 1 回繰り返します。
- 残留エタノールを完全に除去します。磁気ラックにチューブを残し、蓋を開けた状態で約3分間、または目に見えて乾燥するまでビーズを乾燥させます。これはDNAの回収量が低下する可能性があるため、ビーズを乾燥させないでください。
- 磁石からチューブを取り出し、17 μLの0.1x TEバッファーをビーズに加えます。ピペットを少なくとも10回上下に混ぜ、十分に混ぜます。室温で2分間インキュベートし、チューブを磁気ラックに置き、ビーズを上清から完全に分離させます。
- 上清の 15 μL をクリーンヌクレアーゼフリー PCR チューブに移します。ビーズを邪魔しないように注意してください。これはプロトコル中の任意の停止点であり、アダプター-リゲーテッドDNAは-20°Cで保存され得る。
10. アダプターのリゲーテッド DNA の PCR 濃縮
- 表 9に記載されている PCR 反応をセットアップします。プリキャプチャPCRマスターミックスとユニバーサルプライマーを含むマスターミックスを作り、アダプター合わせDNAに加えることができます。多重化シーケンスの場合は、各反応に固有のインデックスプライマーを使用し、各サンプルに個別に追加します。
| PCR エンリッチメント | ボリューム(μL) |
| アダプター合わせDNA (ステップ 10.1) | 15 |
| プリキャプチャPCRマスターミックス | 25 |
| ユニバーサル PCR プライマー | 5 |
| インデックス (X) プライマー | 5 |
| 総体積 | 50 |
表9:アダプター合わせDNAのPCR濃縮。アダプターのリゲーテッド DNA 反応の PCR 濃縮のコンポーネントは、図に示されているボリュームに従って、組み立てて氷上に混合する必要があります。プリキャプチャPCRマスターミックスとユニバーサルPCRプライマーのマスターミックスを作り、アダプター合わせDNAに加えることができます。多重化シーケンスの場合、各サンプルには一意のインデックス プライマーを指定する必要があります。
- 10回軽くピペットで混ぜます。マイクロ遠心分離機でチューブを短く回転させ、サーマルサイクラーに入れ、プログラム#8(補足表2)を使用してPCR増幅を行います。
11. SPRIビーズを用いたPCR反応の精製
- SPRIビーズを使用前に少なくとも30分間室温まで温め、約30分間渦SPRIビーズを再サスペンドします。
- 各PCR反応に45μLの再懸濁ビーズを加えます(約50μL)。室温で5分間インキュベートする前に、少なくとも10回上下にピペットでよく混ぜます。
- マイクロ遠心分離機でチューブを短く回転させ、チューブを磁気ラックに置き、ビーズと上清を分離します。溶液が明らかになった後(〜5分)、慎重に除去し、上清を廃棄します。DNAを含むビーズを邪魔しないように注意してください。
- 磁気ラックの中で、作製した80%エタノールを180μLのチューブに加えます。室温で30sをインキュベートし、慎重に除去し、上清を廃棄します。合計 2 つの洗浄手順に対して、ステップ 11.4 を 1 回繰り返します。
- 残留エタノールを完全に除去します。磁気ラックにチューブを残し、蓋を開けた状態で約3分間、または目に見えて乾燥するまでビーズを乾燥させます。これはDNAの回収量が低下する可能性があるため、ビーズを乾燥させないでください。
- 磁石からチューブを取り出し、ビーズに23 μL 0.1x TEバッファーを追加します。ピペットを少なくとも10回上下に混ぜ、十分に混ぜます。室温で2分間インキュベートします。
- チューブを磁気ラックに置き、ビーズを上清から完全に分離できるようにします。上清子の20 μLをクリーンヌクレアーゼフリーPCRチューブに移します。ビーズを邪魔しないように注意してください。これはプロトコルの任意の停止点であり、プリキャプチャーライブラリは-20°Cに格納されてもよい。
12. プリキャプチャーライブラリの検証と定量化
- フルオロメーターと高感度アッセイキットを使用して、プリキャプチャーライブラリの濃度を測定します。第2部:ハイブリダイゼーションとキャプチャに進むには、最低歩留まり200ngが必要です。
- デジタル電気泳動システムで1μLのライブラリを実行します。必要に応じて、高感度チップの過負荷を避けるためにサンプルを希釈します。
- エレクトロフェログラムが、ピークサイズが約 250~400 bp の狭い分布を示していることを確認します(代表的な結果、図3および図4を参照)。
- 128 bp のピーク(アダプターダイマー)がバイオアナライザのトレースに表示され、信号の強度が 250-400 bp ライブラリ信号の強度 ≥(代表的な結果を参照)の場合(例えば、図5を参照)、サンプル容量(ステップ 11.7 から 50 μL)を 0.1x TE バッファーで起動し、SPRI Bead 浄化処理を繰り返します(ステップ 11)。これはプロトコルの任意の停止点であり、プリキャプチャーライブラリは、パートIIに移る前に-20°Cで保存され得る:ImmunoPrismハイブリダイゼーションおよびキャプチャー。
第二部:ハイブリダイゼーションとキャプチャー
13. ブロッキングオリゴ、Cot-1 DNA、プリキャプチャーライブラリーDNA、ドライを組み合わせる
- ステップ11で作成したバーコードライブラリとステップ12で定量化し、ヌクレアーゼフリーPCRチューブまたは1.5mLマイクロチューブにCot-1 DNAとブロッキングオリゴを混ぜ合わせる。.
| 試薬 | 数量/数量 |
| ステップ 10.10 のバーコードライブラリ | 200 ng |
| コット1 DNA | 2 μg |
| ブロッキングオリゴ | 2 μL |
表10:ハイブリダイゼーション調製および乾燥ハイブリダイゼーションの準備でライブラリを乾燥させるために組み合わせる成分は、示されている量に応じて組み立てる必要があります。
- 30~45°Cに設定した真空濃縮器を使用してチューブの内容物を乾燥させます。これは、プロトコル内のオプションの停止点です。乾燥後、チューブは、室温(15〜25°C)で一晩または-20°Cで長く保管され得る。
14. DNA捕捉プローブをライブラリとハイブリダイズする
- 2xビーズ洗浄バッファーおよびハイブリダイゼーションバッファー、ハイブリダイゼーションバッファーエンハンサー、イムノプリズムプローブパネル、10xウォッシュバッファー1、10xウォッシュバッファ2、10xウォッシュバッファ3、および10xストリンジェントウォッシュバッファを室温で解凍します。使用前に、塩の結晶化のためのハイブリダイゼーションバッファーを検査します。結晶が存在する場合は、65°Cでチューブを加熱し、緩衝液が完全に可溶化されるまで断続的に振る。
- 室温で、チューブ内にハイブリダイゼーションマスターミックスを作成します。サンプル数でボリュームを乗算し、表 11に続いて 10% 余分に追加します。
| ハイブリダイゼーションマスターミックス | ボリューム(μL) |
| ハイブリダイゼーションバッファ | 8.5 |
| ハイブリダイゼーションバッファエンハンサー | 2.7 |
| イムノプリズムプローブパネル | 5 |
| ヌクレアーゼフリー水 | 0.8 |
| 総体積 | 17 |
表 11: ハイブリダイゼーションマスターミックスハイブリダイゼーションマスターミックスのコンポーネントは、図に示されているボリュームに応じて、室温で組み立てられ、混合する必要があります。
- ボルテックスまたはピペットを上下に混ぜ合わせます。次いで、乾燥DNAを含む各チューブにハイブリダイゼーションマスターミックスの17μLを加える。チューブを密封し、室温で5分間インキュベートします。
- サンプルをボルテックスし、完全に混合されていることを確認し、マイクロ遠心分離機でサンプルを短時間スピンダウンします。該当する場合は、各サンプルを1.5 mLマイクロチューブからヌクレアーゼフリーPCRチューブに移します。
- サンプルをサーマルサイクラーに入れ、プログラム#9を実行します (補足表 2)。
- インキュベーション中に、洗浄バッファー(ステップ15)およびストレプトアビジンビーズ(ステップ16)を準備し、緩衝液を予熱し、ストレプトアビジンビーズを平衡化するのに十分な時間を確保する。
15. 洗浄バッファーの準備
注:洗浄バッファーは、2x (ビーズウォッシュバッファー) または 10x (他のすべての洗浄バッファー) 濃縮溶液として供給されます。
- ハイブリダイゼーションインキュベーションの間に、2xビーズウォッシュバッファと10xウォッシュバッファを希釈して1xの作業溶液を作成し、必要なサンプル数を掛け、10%余分なサンプルを追加します。10xウォッシュバッファー1が曇っている場合は、65°Cの水浴または加熱ブロックでボトルを加熱し、微粒子を再懸濁させる。凍結した1xウォッシュバッファーは、解凍後に混合する必要があります。
| 洗浄バッファー | 濃縮バッファー (μL) | ヌクレアーゼフリー水(μL) | 合計(μL) |
| ビーズウォッシュバッファー | 150 | 150 | 300 |
| 洗浄バッファー 1 | 25 | 225 | 250 |
| 洗浄バッファー 2 | 15 | 135 | 150 |
| 洗浄バッファー 3 | 15 | 135 | 150 |
| ストリンジェントウォッシュバッファー | 30 | 270 | 300 |
表12:洗浄バッファー希釈。濃度洗浄バッファーは、示されている体積に従って、室温でヌクレアーゼを含まない水で希釈する必要があります。
- 1xウォッシュバッファーをヌクレアーゼフリーPCRチューブにアリコートし、表13に示すように適切な温度に配置する。ピペッティングに十分な過大を含むようにしてください。加熱バッファの場合は、蓋を70°Cに設定して65°Cに設定したサーマルサイクラーを使用します。
| 洗浄バッファー | 保持温度 | ボリューム/チューブ(μL) | チューブ/サンプルの数 |
| ビーズウォッシュバッファー | RT (15-25 °C) | 100 | 3 |
| 洗浄バッファー 1 | 65°C | 100 | 1 |
| 洗浄バッファー 1 | RT (15-25 °C) | 150 | 1 |
| 洗浄バッファー 2 | RT (15-25 °C) | 150 | 1 |
| 洗浄バッファー 3 | RT (15-25 °C) | 150 | 1 |
| ストリンジェントウォッシュバッファー | 65°C | 150 | 2 |
表 13: 希釈洗浄バッファー.希釈された洗浄バッファーは、示されているサンプルあたりの体積およびチューブ数に応じて、別々のチューブに分ける必要があります。洗浄バッファーは、使用前に指定温度で保持する必要があります。
- 表14に示すように、ビーズ再懸濁ミックスを室温で準備し、必要なサンプル数を掛け、10%余分に加えます。
| ビーズリサスペンションミックス | ボリューム(μL) |
| ハイブリダイゼーションバッファ | 8.5 |
| ハイブリダイゼーションバッファエンハンサー | 2.7 |
| ヌクレアーゼフリー水 | 5.8 |
| 総体積 | 17 |
表14:ビーズリサスペンションミックス。ビーズ再懸濁ミックスのコンポーネントは、図に示されているボリュームに応じて、室温で組み立てられ、混合する必要があります。
16. ストレプトアビジンビーズを準備する
- 使用前に少なくとも30分間室温でストレプトアビジンビーズを平衡化します。15 s、アリコート 50 μL のビーズを捕捉毎ヌクレアーゼフリー PCR チューブにボルテックスして、ビーズを完全に混合します。
- 1xビーズウォッシュバッファ(ステップ15.1で作成)の100 μLを各チューブに加えます。ピペットを10回軽く上下に混ぜます。チューブを磁気ラックに置き、ビーズを上清から完全に分離できるようにします。
- 透明な上清を取り除き、廃棄します。ビーズを邪魔しないように注意してください。
- 以下の洗浄を行う。
- 磁気ラックから取り外します。ビーズを含む各チューブに100μLのビーズウォッシュバッファを加え、ピペットを上下に10回加え、混ぜ合わせます。
- チューブを磁気ラックに置き、ビーズを上清から完全に分離できるようにします。
- 透明な上清を慎重に取り外して捨てます。
- 合計 2 回の処理について、手順 16.4 を 1 回繰り返します。
- 磁気ラックから取り外します。ステップ15.3から17 μLのビーズリサスペンションミックスを各チューブに加えます。ピペットを何度か上下に繰り上げて、徹底的に混ぜます。ビーズがチューブの側面に貼り付けられていないことを確認します。必要に応じて、チューブを短く回転して下部のビーズを集めます。
17. ハイブリッド化ターゲットをストレプトアビジンビーズにバインドする
- 4時間のハイブリダイゼーションインキュベーションが完了したら、サーマルサイクラーからサンプルを取り出し、加熱した蓋を70°Cにセットして65°Cでインキュベートするようにサーマルサイクラーをセットします。
- マルチチャンネルピペットを使用して、完全に均質化されたビーズの17 μLをサンプルに移します。10回上下にピペットで十分に混ぜます。
- プログラム#10(補足表2)の後に熱サイクラーにチューブを入れることで、DNAをビーズに結合する。インキュベーション中に、ストリップチューブを10〜12分ごとに短く取り外し、3sの渦を少し外してビーズが懸濁液中に残るようにします。または、数回上下にピペットで混ぜます。ストレプトアビジンビーズを洗い流す(ステップ18)。
18. ストレプトアビジンビーズを洗浄してバインドされていないDNAを除去する
- ステップ15.2の1xウォッシュバッファを使用し、洗浄中に熱いバッファをサーマルサイクラーに保存します。
- ステップ 17.3 からチューブに 100 μL の予熱 1x ウォッシュバッファー 1 を加えます。10回上下にピペットで十分に混ぜます。チューブを磁気ラックに置き、ビーズを上清から完全に分離できるようにします。
- ピペットと非結合DNAを含む上清を廃棄します。磁気ラックから取り外します。
- 以下の65°C洗浄を行う。
- 150 μLの予熱1xストリンジェントウォッシュバッファーを追加します。
- 少なくとも10回上下にピペットで混ぜます。ピペッティング中に泡を避けてください。すべてのチューブにビーズが完全に再懸濁されていることを確認してください。
- 65°Cで5分間サーマルサイクラーにインキュベートします。
- チューブを磁気ラックに置き、ビーズを上清から完全に分離できるようにします。ピペットと非結合DNAを含む上清を廃棄します。磁気ラックから取り外します。
- 2 つのストリンジェントなワッシュの合計について、ステップ 18.4 を繰り返します。
- 最初の室温洗浄を行います。
- 室温1xウォッシュバッファー1の150μLを加えます。
- ピペットを10~20回上下にしてビーズを完全に再サスペンずします。
- チューブを密封し、2分間インキュベートし、30秒間穏やかに渦を起き、30秒間休みます。すべての井戸のビーズは、インキュベーション全体を通してすべてのチューブに完全に再懸濁されたままであることを確認してください。
- チューブを簡単に遠心分離します。
- チューブを磁気ラックに置き、ビーズを上清から完全に分離できるようにします。ピペットと上清を捨てます。
- チューブを密封し、一時的に遠心分離機を押します。磁気ラックに戻し、10 μl ピペットを使用して、残留洗浄バッファーを取り除きます。
- 2回目の室温洗浄を行います。
- 室温1xウォッシュバッファ2の150 μLを加えます。
- ピペットを10~20回上下にしてビーズを完全に再サスペンずします。
- チューブを密封し、2分間インキュベートし、30秒間穏やかに渦を起き、30秒間休みます。すべての井戸のビーズは、インキュベーション全体を通してすべてのチューブに完全に再懸濁されたままであることを確認してください。
- チューブを簡単に遠心分離します。
- 洗浄バッファー 2 に再懸濁したビーズの全容積を、ヌクレアーゼフリー PCR チューブに移します。重要:ビーズを新鮮なチューブに移すことによって、ターゲット外の汚染を避ける必要があります。
- チューブを磁気ラックに置き、ビーズを上清から完全に分離できるようにします。ピペットと上清を捨てます。
- チューブを密封し、一時的に遠心分離機を押します。磁気ラックに戻し、10 μl ピペットを使用して、残留洗浄バッファーを取り除きます。
- 第3の室温洗浄を行う。
- 室温1xウォッシュバッファー3の150μLを加えます。
- ピペットを10~20回上下にしてビーズを完全に再サスペンずします。
- チューブを密封し、2分間インキュベートし、30秒間穏やかに渦を起き、30秒間休みます。すべての井戸のビーズは、インキュベーション全体を通してすべてのチューブに完全に再懸濁されたままであることを確認してください。
- チューブを簡単に遠心分離します。
- チューブを磁気ラックに置き、ビーズを上清から完全に分離できるようにします。ピペットと上清を捨てます。
- チューブを密封し、一時的に遠心分離機を押します。磁気ラックに戻り、10 μL ピペットを使用して、残留洗浄バッファーを取り除きます。
- 磁気ラックから取り出し、20 μLのヌクレアーゼを含まない水をビーズに加えます。
- ピペットを10回上下にして、チューブの側面に貼り付けたビーズが再懸濁されていることを確認します。
- 重要: ビーズを捨てないでください。ステップ19で捕捉されたDNAを持つ再懸濁ビーズの20 μL全体を使用してください。
19. 最終、ポストキャプチャPCRエンリッチメントを実行
- 表 15に従って、ポストキャプチャ PCR マスター ミックスを次の表に従って準備し、必要なサンプル数を乗算し、10% 余分に追加します。
| ポストキャプチャPCRマスターミックスコンポーネント | ボリューム(μL) |
| ポストキャプチャPCRマスターミックス | 25 |
| ポストキャプチャPCRプライマーミックス | 1.25 |
| ヌクレアーゼフリー水 | 3.75 |
| 総体積 | 30 |
表 15: ポストキャプチャ PCR マスター ミックスポストキャプチャPCRマスターミックスのコンポーネントは、図に示されている量に応じて、氷上で組み立てられ、混合する必要があります。
- 各サンプルにポストキャプチャPCRマスターミックスの30 μLを加え、最終反応量50μLを得るために、10回のピペットで完全に混合します。
- PCR チューブをサーマルサイクラーに入れ、プログラム #11 (補足表 2)に従ってインキュベートします。
20. ポストキャプチャPCRフラグメントを精製する
- SPRIビーズを使用前に少なくとも30分間室温まで温め、約30分間渦SPRIビーズを再サスペンドします。
- 各PCR濃縮キャプチャに75 μLの再懸濁ビーズを加えます(50 μL)。少なくとも10回上下にピペットで混ぜます。ストレプトアビジンビーズは、SPRIビーズ精製を妨げません。室温で5分間インキュベートします。
- マイクロ遠心分離機でチューブを短く回転させ、チューブを磁気ラックに置き、ビーズと上清を分離します。溶液が明確になってから、慎重に除去し、上清を廃棄します。DNAを含むビーズを邪魔しないように注意してください。
- 磁気ラックの中で、作製した80%エタノールを180μLのチューブに加えます。室温で30sをインキュベートし、慎重に除去し、上清を廃棄します。
- 合計 2 つの洗浄手順に対して、ステップ 20.4 を 1 回繰り返します。
- 残留エタノールを完全に除去します。チューブを磁気ラックに置いたまま、蓋を開けた状態で3分、または目に見えて乾燥するまで空気を乾かします。ビーズを過度に乾燥させないでください。これは、DNAの回復が低下する可能性があります。
- 磁石からチューブを取り外します。22 μLの0.1x TEバッファーを加えてビーズからDNAを溶出します。数回上下にピペットでうまく混ぜます。室温で2分間インキュベートします。溶液がはっきりするまで、磁気ラックの上にチューブを置きます。
- 上清の20μLを取り除き、きれいなヌクレアーゼフリーPCRチューブに移し、ビーズを邪魔しないように注意してください。これはプロトコル内の任意の停止点であり、ライブラリは-20°Cで保存されてもよい。
21. ライブラリの検証と定量化
- フルオロメーターと高感度アッセイキットを使用して、キャプチャされたライブラリの濃度を測定します。
- デジタル電気泳動高感度DNAチップを用いて、キャプチャしたライブラリの平均フラグメント長を測定し、システムソフトウェアを使用して各ライブラリの平均フラグメントサイズを計算します。平均フラグメントサイズは約 250~400 bp である必要があります(代表的な結果、図6および図7を参照)。これはプロトコル内の任意の停止点であり、完成したライブラリは-20°Cで保存されてもよい。
22. シーケンスプラットフォームでのシーケンス
- シーケンシングの場合は、ライブラリを2nMに希釈し、シーケンサーのロードと操作に関するメーカーのガイドラインに従ってください。長さが 50 bp 以上の最小深さ 1,500 万のシングルエンド読み取りまで、シーケンス ライブラリ。
23. 免疫プロファイルを生成し、クラウドベースのインフォマティクスツールであるプリズムポータルでバイオマーカーを発見するためのシーケンシングデータの分析
- https://prism.cofactorgenomics.com/にアクセスしてプリズム アカウントを作成する
- ログインしたら、プリズムの任意のページから上部ツールバーの[新しいプロジェクトを送信]をクリックして、非多重化されたFASTQシーケンスファイルをアップロードするか、またはPrismアカウントでBaseSpaceに保存されたファイルをアップロードします。
- プロジェクト名を含む新しいプロジェクト フォームに入力し、グループまたはコホート別のサンプルを入力します。サンプルのグループ化と対応するグループ名は、バイオマーカー発見レポートを生成するために必要です。バイオマーカー検出レポートを生成するには、グループごとに最低 3 つのサンプルが必要です。[アプリケーションの起動] ボタンをクリックしてフォームを送信します。成功した場合は確認ページが表示されます。
- ログイン中に、上部のツールバーまたは Prism の任意のページで [結果を表示] をクリックします。Prism を使用すると、ユーザーは、提出されたプロジェクトの状態を確認し、プロジェクトごとにサンプルレポートとバイオマーカー レポートを表示できます。ユーザーが Prism で作成したプロジェクトのテーブルが表示されます。テーブルには、ステータス、名前、および提出日の 3 つの列があります。
注: 各プロジェクトのステータスは、次のとおりです。
• プロジェクト分析が現在実行されている「実行中」、または
• プロジェクト分析が完了し、レポートが利用可能な「成功」。 - プロジェクトが解析を完了した場合(「成功」ステータスで示される)、個別サンプルレポートとバイオマーカー検出レポートを表示します。バイオマーカー検出レポートは、プロジェクトにグループごとに必要なサンプルが最低 3 つ含まれている場合にのみ使用できます。
- これらのレポートにアクセスするには、プロジェクトのテーブルに戻り、プロジェクトの名前をクリックします。このプロジェクト ページには、プロジェクトのサンプルごとに行が含まれるテーブルがあります。各サンプルの個別レポートにアクセスするには、[レポート] 列の各行のリンクをクリックします。表のすぐ下にあるバイオマーカー検出レポートのリンクをクリックします。このページにリンクがない場合、プロジェクトは解析を完了していません。
結果
プロトコル全体を通して、生成された材料の品質と量を評価するためのチェックポイントが多数あります。プロトコルに記載されたステップ12に続いて、図3に示すように、インタクトRNAサンプルの典型的なプリキャプチャーライブラリの代表であるエレクトロフェログラムが生成される(RIN = 7.8)。

図 3: インタクト RNA サンプルの典型的なプリキャプチャー ライブラリ バイオアナライザー トレースプリキャプチャー・ライブラリは、250~400塩基対(bp)の大きさの大きなピークとして表示されます。この図の大きなバージョンを表示するには、ここをクリックしてください。
図4に示す1,000bp前後の第2のピークで示されるように、過剰増幅を避けるために注意を払う必要があり、FFPE RNAサンプルから生成されたプリキャプチャーライブラリの代表的なエレクトロフェログラム(DV200= 46)。.このピークがメインピークに対して小さい場合(図に示すように、約 250 -400 塩基対 (bp) )、下流のステップまたは解析に干渉しません。2 番目のピークが 250-400 bp ピークに対して大きい場合、過剰増幅を減らすために、プリキャプチャー・ライブラリーを PCR サイクルを少なくしてリメイクできます。
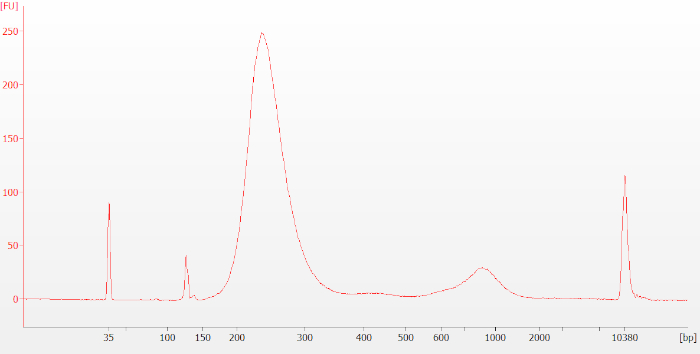
図4:FFPE RNAサンプルの典型的なプリキャプチャーライブラリバイオアナライザートレース2番目のピークは約1,000 bpで、過剰増幅を示しています。このピークが250〜400bp前後の主ピークに対して小さい場合、下流の工程や解析に干渉しません。2 番目のピークが 250-400 bp ピークに対して大きい場合、過剰増幅を減らすために、プリキャプチャー・ライブラリーを PCR サイクルを少なくしてリメイクできます。この図の大きなバージョンを表示するには、ここをクリックしてください。
ステップ 12.1.3 で説明したように、追加のクリーンアップが必要かどうかを判断するために、アダプター・ダイマーの存在を評価する必要があります。図5に示す電気泳動グラムは、受け入れられない(図5A、DV200=33)および許容可能な(図5B、DV200=46)レベルのアダプターダイマーを表すものであり、128bp前後のシャープピークとして現れる。

図5:プリキャプチャライブラリバイオアナライザのトレース。アダプタダイマーは128bpの周りのシャープなピークとして現れる。(B) 受容可能なアダプター・ダイマー・レベルは、このトレースに示されています。両方の痕跡は軽度の過剰増幅の証拠を示すが、これは免疫Prismアッセイを妨げるべきではない。この図の大きなバージョンを表示するには、ここをクリックしてください。
プロトコルの完了時に、シーケンシングの前に、最終的なライブラリは再びデジタル電気泳動を使用して評価されます。FFPE RNA から作られたライブラリは、無傷の RNA から作られたライブラリよりも平均サイズ分布が小さい傾向があります。インタクト RNA サンプルの場合、結果のトレースは図 6 (RIN = 9.5) のようになります。分解またはFFPE RNAの場合、得られるトレースは図7(DV 200 = 36)のようになります。

図 6: インタクト RNA サンプルの典型的な最終ライブラリ バイオアナライザー トレース最終的なライブラリは、サイズが250-400ベースペア(bp)の周りに広いピークとして表示されます。この図の大きなバージョンを表示するには、ここをクリックしてください。

図 7: FFPE RNA サンプルの典型的な最終ライブラリ バイオアナライザー トレースFFPE RNA から作られたライブラリは、無傷の RNA から作られたライブラリよりも平均サイズ分布が小さい傾向があります。この図の大きなバージョンを表示するには、ここをクリックしてください。
説明したように、このプロトコルで生成された結果は、図 8に示すように、2 つの主要な方法で適用できます。
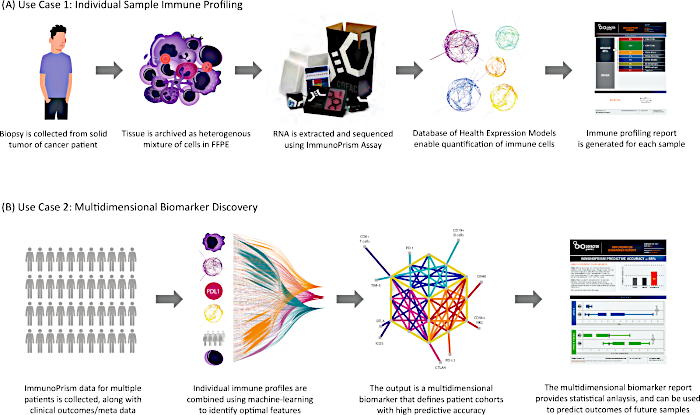
図 8: プロトコルの 2 つのユース ケースこの免疫プロファイリングアッセイによって生成された結果は、2つの主要な翻訳アプリケーションで適用されます。(A)最初の使用例は、ヒト固形腫瘍組織(FFPEアーカイブを含む)から始まり、サンプルに対して個々の免疫プロファイルを生成する。(B) ヒトサンプルのコホートに対して生成されたデータは、プリズムポータルを使用して多次元バイオマーカーとそれに対応するバイオマーカーレポートを生成する。この図の大きなバージョンを表示するには、ここをクリックしてください。
これらの各使用例を示すために、小さな翻訳研究の代表データが21.この研究で使用されるサンプルは、非小細胞肺癌(NSCLC)について診断され治療された7人の患者からの検体のセットである。サンプルは、前処置および後治療生検からの患者一致した固形腫瘍組織である。まず、個々のサンプルを分析して、図9に示す例のような免疫プロファイルを生成する。

図 9: NSCLC サンプルに対する個々の免疫レポートの例Prism Portal パイプラインは、処理された各サンプルのグラフィカル レポートを生成し、ここに示す NSCLC 固形腫瘍サンプルに対して生成された代表的なレポートを示します。(A) FFPE組織から抽出したRNAサンプル中に存在する免疫細胞の内訳を図表して表したレポートの正面。(B) 本報告の裏側には、免疫細胞の表(絶対パーセント)およびエスケープ遺伝子発現(100万分の100の転写産物における、またはTPM)、ならびにアッセイの性能の記述が含まれる。この図の大きなバージョンを表示するには、ここをクリックしてください。
治療前および治療後の免疫プロファイルは、治療(化学療法または放射線、この研究では)が腫瘍微小環境をどのように改変したかを理解するために使用することができる。図10に例を示し、各免疫細胞および総免疫内容のパーセンテージの変化が、単一の患者に対して化学療法前後に示される。
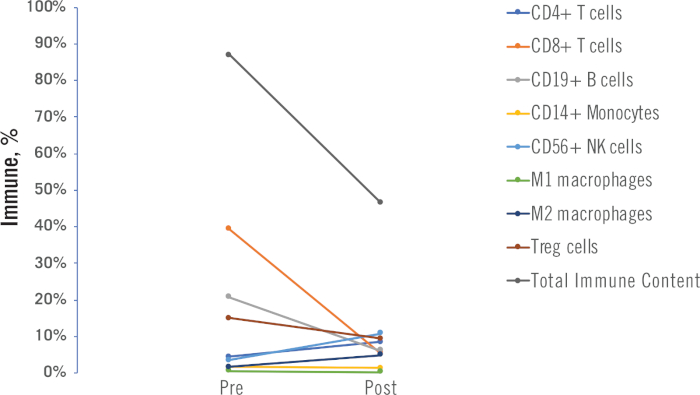
図10:前処置および後処理結果の例単一のNSCLC患者から治療前および後のサンプルから生成された個々の免疫細胞および総免疫内容データが示される。この例では、患者は治療として化学療法レジメンを受けた。この図の大きなバージョンを表示するには、ここをクリックしてください。
患者は、比較のために臨床転帰または表現型などの基準によってグループ化され得る。例えば、図11において、NSCLC試験におけるサンプルは、治療後の疾患進行までの時間に従って比較した。患者のサブセットは、18ヶ月で疾患再発を示し、別のサブセットは≤18ヶ月で、より速く進行した。中央値のデルタ値(前処置値と後処置値の差)を各サンプルについて比較し、疾患進行の推定バイオマーカーを同定する。
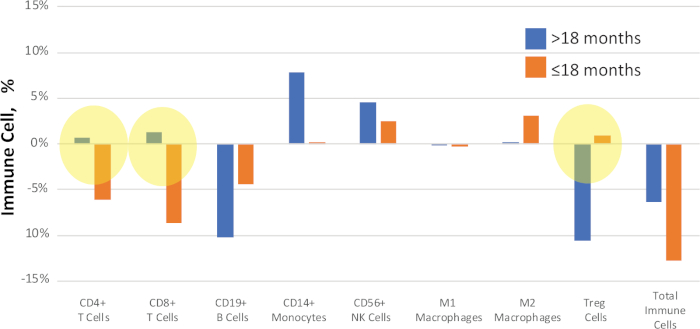
図 11: 臨床結果比較の例一致した前処置および治療後のNSCLCサンプルにおける免疫細胞の割合の間の定量的変化を「デルタ」値として計算および報告した。黄色で強調表示されているものは、生存状態間の明確な信号の変化を示しています。青いバーは、病気が進行するまでの18ヶ月間の>18ヶ月のデルタ値の中央値を表し、オレンジ色の棒は病気が進行するまでの18ヶ月間のデルタ値の中央値を表します。この図の大きなバージョンを表示するには、ここをクリックしてください。
最後に、同様のサンプルグループを使用して、プリトリートメントサンプルを特異的に調し、プリズムポータルを使用してバイオマーカーレポートを生成することによって予測バイオマーカーを同定することができます。図12に示す、上述の臨床表現型(疾患進行)と同じものが、サンプルグループを定義する。この例では、2つの免疫脱出遺伝子が、サンプルグループの統計的に有意な分化因子として同定された(CD47およびOX40、図12Aの下パネルに示す)。この例では、個々の遺伝子バイオマーカーが明確な統計的有意性を有する堅牢であるため、多次元バイオマーカーは有意な予測値を加えない(ImmunoPrism、図12Bの右上の棒グラフに標識されている)。アッセイの18個の分析結果すべてを含むデータの完全なテーブルは、統計分析と簡単な方法の要約を含むレポートの裏側に要約されています。

図 12: NSCLC サンプルのバイオマーカー レポートの例バイオマーカーディスカバリーパイプラインは、個々のバイオマーカーの視覚的なレポートと機械学習多次元バイオマーカーを詳細な統計で提供します。(A)この研究では、パイプラインは、18ヶ月の閾値を有する疾患進行を定義するための統計的に有意な2つの個別バイオマーカー(CD47およびOX40)を同定した。(B) レポートの裏側に、メソッドと完全な結果の詳細が含まれています。この図の大きなバージョンを表示するには、ここをクリックしてください。
補足表1:試薬キット材料ImmunoPrismキットに用意されている材料のリストと、メーカーのプロトコルで参照されている部品番号が記載されています。その他必要な機器および材料はすべて、材料表に記載されています。安全データシート(SDS)のhttps://cofactorgenomics.com/product/immunoprism-kit/を参照してください。このファイルを表示するには、ここをクリックしてください (右クリックしてダウンロードしてください)。
補足表 2: サーマルサイクラー プログラムプロトコル全体で参照される推奨サイクラー プログラムは、プログラミングを容易にするためにまとめられています。このファイルを表示するには、ここをクリックしてください (右クリックしてダウンロードしてください)。
補足表 3: 索引の順序付けガイド試薬キットに用意されているインデックスプライマーがリストされています。シーケンス後の非多重化の各反応に、固有のプライマーが追加されます。推奨される低レベル多重化の組み合わせも提供されます。このファイルを表示するには、ここをクリックしてください (右クリックしてダウンロードしてください)。
ディスカッション
このプロトコルには、20 ng の無傷または 40 ng の高度に分解された RNA (FFPE) が必要です。RNAサンプルは、DNA、塩(例えば、Mg2+、またはグアニジン塩)、二価カチオンキレート剤(例えば、EDTA、EGTA、クエン酸塩)、または有機物(例えば、フェノールおよびエタノール)を含まないべきである。DV200 <20%を持つ RNA サンプルを続行することはお勧めしません。これらの制御は、ライブラリの準備から分析まで、プロトコル全体を通してパフォーマンスを評価する手段を提供するので、キット内コントロールRNAの使用が強く推奨されます。
プロトコルは0.2 mL PCRストリップ管を使用して行われるように設計されています。好ましい場合、プロトコルは96ウェルPCRプレートのウェルを用いて行うこともできます。PCRチューブまたはストリップチューブのすべてのリファレンスの代わりに、96ウェルPCRプレートのウェルを使用してください。ビーズの精製や洗浄工程の間にビーズの完全な再懸濁を視覚的に確認することが重要であるため、透明な井戸のみを使用してPCRプレートを使用してください。
プロトコル全体を通して、特に指定がない限り、試薬を凍結または氷上に保管してください。完全に解凍されるまで試薬を使用しないでください。使用前に、すべての試薬を完全に混ぜるようにしてください。
使用後は、すぐに-20°Cに戻るまで、酵素を-20°Cに保ちます。分子グレードのヌクレアーゼフリー水のみを使用します。DEPC 処理水を使用することは推奨されません。ピペットを混ぜるときは、溶液がよく混ざるまで、穏やかに吸引し、全体の体積の少なくとも50%を分配する。ピペットは酵素を含むすべてのマスターミックスを混合します。渦を使用して酵素を混合すると、変性を引き起こし、その性能を損なう可能性があります。ビーズ精製の際には、分子グレードのエタノールから作ったての80%エタノール溶液を使用します。新鮮ではないエタノール溶液を使用すると、収率が低下する可能性があります。これは溶出効率を低下させることができるので、ビーズを過度に乾燥しないでください(過乾燥するとビーズはひび割れたように見えます)。
ステップ10で説明したように、一意のインデックスプライマーが各反応に追加されます。これらのインデックスのシーケンスに基づいて、低レベル多重化のために、特定のインデックスの組み合わせが最適です。これらのインデックスのシーケンスは、データのシーケンス後の変換を解除するために必要です。シーケンスと推奨される多重化の組み合わせは、補足表 3に記載されています。この同じステップでは、推奨されるPCRサイクルの数は、使用されるRNAの品質によって異なり、PCRの過剰増幅を防ぐためにいくつかの最適化が必要な場合があることに注意することが重要です。免疫プリズムインタクトコントロールRNAおよびその他の高品質RNAについては、10 PCRサイクルで最適化を開始します。免疫プリズム FFPE コントロール RNA およびその他の高度に分解/FFPE RNA の場合、15 PCR サイクルで最適化を開始します。PCRサイクルを最適化するために、解析する材料を代表するRNAを用いた試験ライブラリの作成をお勧めします。プリキャプチャー・ライブラリーの収量が一貫して得られる PCR サイクルの最小数 (>200 ng) を使用する必要があります。バイオアナライザのトレースで1000bp前後の二次ピークは、過剰増幅を示しています(図4)。過剰増幅は最小限に抑えるべきであるが、小さい二次ピークの存在はアッセイ結果を妨げない。
サンプル損失を最小限に抑え、チューブのスイッチングを回避するために、真空濃縮器で可能な場合、ステップ13はPCRチューブ、ストリップチューブ、または1.5 mLマイクロチューブの代わりに96ウェルPCRプレートで実行できます。ローターは多くのコンセントレーターで取り外すことができる。これにより、ストリップチューブまたはプレートが真空中に収まります。真空濃縮は、遠心分離無しの水性乾燥を使用して実行することができます。手順については、真空濃縮器のマニュアルを参照してください。サンプルをストリップチューブまたは96ウェルプレートで乾燥させた場合、ハイブリダイゼーション工程は同じ容器内で行うことができる。
ステップ17の間、ビード捕獲効率を高めるために10-12分ごとにボルテックスするようにしてください。チューブが開くのを防ぐために混合するときは、慎重に暖かいストリップチューブのキャップを保持します。
ステップ18で説明したワッシュは、高い非特異的汚染を避けるために重要であり、密接に従わなければなりません。洗浄のたびにビーズを完全に再懸濁し、洗浄バッファを完全に取り除き、洗浄バッファ2洗浄中にサンプルを新鮮なストリップチューブに移します(ステップ18.6.5)。ストレプトアビジンビーズが完全に再懸濁され、全体のインキュベーション中にサスペンションに残っていることを確認してください。チューブキャップに飛び散ってキャプチャに悪影響を与えるわけではありません。室温の洗い上がりの間、マイクロプレート渦ミキサーは、より容易な再懸濁のために2分間のインキュベーション期間全体のサンプルをボルテックスするために使用され得る。ストレプトアビジンビーズを乾燥させないでください。必要に応じて、ビーズの乾燥を避けるためにバッファー内のインキュベーションを拡張します。複数のストリップチューブを使用する場合は、一度に1つのストリップチューブを使用して、他のストリップチューブがサーモサイクラーに座っている間、各洗浄のために一度に作業します。これは、ビーズの乾燥や急いで過ぎないようにし、再懸濁液の不良や他の最適でない技術を生じさせるのに役立ちます。初めてのユーザーの場合は、一度に 8 つ以上のライブラリの反応を処理することはお勧めしません。
現在の免疫プロファイリング技術は、重要な解釈(RNAシーケンシング)を必要とする何千ものデータポイントから個々の離散データポイント(単一プレックスIHC)まで、連続した情報を提供します。ここで説明するプロトコルは、高感度を可能にする焦点を当てたスコープを持つ真ん中のどこかにあるアプローチを表しますが、臨床的に関連する転写データのサブセットのみをキャプチャします。バルクRNA抽出の性質上、このプロトコルは免疫細胞と腫瘍微小環境との間の空間的関係に関する情報を提供しないが、結果はこの情報を追加するイメージング技術と補完され得る。このプロトコルによって生成されたデータには、がんの生物学を病気として学び、治療を行うための治療法について学ぶ必要があります。代表的な結果に示すように、個々の免疫報告は、患者の免疫プロファイルが疾患の進行または治療などの事象に応答してどのように変化するかを理解するのに有用である。ここで示した結果はいくつかのユースケースを提供しますが、治療の作用機序を調査し、進行フリーおよび全体的な生存などの臨床結果の推定バイオマーカーを特定するなど、他のアプリケーションも同様です実用的。バイオマーカー発見用途にこのプロトコルを使用する場合、均質な集団が分析され、統計的な力のために十分なサンプルが含まれ、偏りの源が考慮されるように、良い研究設計を実践することが重要です。アッセイの焦点を絞った合理化された性質のために、一度発見されたこれらのバイオマーカーの臨床検証および下流適用への道を想像することは可能である。
開示事項
すべての著者は、この記事で使用される免疫Prism試薬キットおよびインフォマティクスツールを開発し、生産するCofactor Genomics, Inc.に採用されています。免疫プリズムアッセイは研究用のみであり、診断手順で使用するためのものではありません。
謝辞
著者らは、TriStarテクノロジーグループが代表的な結果に生物学的標本を提供し、コファクターゲノミクスの分子、分析、製品、商業チーム全体を技術的専門知識のために提供し、かつサポート。
資料
| Name | Company | Catalog Number | Comments |
| 0.2 mL PCR 8 tube strip | USA Scientific | 1402-2700 | USA Scientific 0.2 mL PCR 8-tube strip |
| 200 Proof Ethanol | MilliporeSigma | EX0276-1 | Prepare 80% by mixing with nuclease-free water on the day of the experiment |
| 96-well thermal cyclers | BioRad | 1861096 | |
| Solid-phase Reversible Immobilization (SPRI) Beads | Beckman-Coulter | A63882 | Agencourt AMPure XP – PCR Purification beads |
| Digital electrophoresis chips and kit | Agilent Technologies | 5067-4626 | Agilent High Sensitivity DNA chips and kit |
| Digital electrophoresis system | Agilent Technologies | G2939AA | Agilent 2100 Electrophoresis Bioanalyzer |
| Streptavidin Beads | ThermoFisher Scientific | 65306 | Dynabeads M-270 Streptavidin |
| ImmunoPrism Kit – 24 reaction | Cofactor Genomics | CFGK-302 | Cofactor ImmunoPrism Immune Profiling Kit – 24 reactions |
| Human Cot-1 DNA | ThermoFisher Scientific | 15279011 | Invitrogen brand |
| Magnetic separation rack | Alpaqua/Invitrogen | A001322/12331D | 96-well Magnetic Ring Stand |
| Microcentrifuge | Eppendorf | 22620701 | |
| Microcentrifuge tubes | USA Scientific | 1415-2600 | USA Scientific 1.5 mL low-adhesion microcentrifuge tube |
| NextSeq550 | Illumina | SY-415-1002 | Any Illumina sequencer may be used for this protocol |
| Nuclease-free water | ThermoFisher Scientific | AM9937 | |
| Prism Extraction Kit | Cofactor Genomics | CFGK-401 | Cofactor Prism FFPE Extraction Kit – 24 samples |
| Purified RNA | - | - | Purified from human tissue samples |
| Fluorometer | ThermoFisher Scientific | Q33226 | Qubit 4 System |
| Fluorometric Assay Tubes | Axygen | PCR-05-C | 0.5mL Thin Wall PCR Tubes with Flat Caps |
| High Sensitivity Fluorometric Reagent Kit | Life Technologies | Q32854 | Qubit dsDNA HS Assay Kit |
| Vacuum concentrator | Eppendorf | 22820001 | VacufugePlus |
| Vortex mixer | VWR | 10153-838 | |
| Water bath or heating block | VWR/USA Scientific | NA/2510-1102 | VWR water bath/USA Scientific heating block |
参考文献
- Brambilla, E., et al. Prognostic Effect of Tumor Lymphocytic Infiltration in Resectable Non-Small-Cell Lung Cancer. Journal of Clinical Oncology. 34 (11), 1223-1230 (2016).
- Iacono, D., et al. Tumour-infiltrating lymphocytes, programmed death ligand 1 and cyclooxygenase-2 expression in skin melanoma of elderly patients: clinicopathological correlations. Melanoma Research. 28 (6), 547-554 (2018).
- Fridman, W. H., Zitvogel, L., Sautes-Fridman, C., Kroemer, G. The immune contexture in cancer prognosis and treatment. Nature Reviews Clinical Oncology. 14 (12), 717-734 (2017).
- Sierant, M. C., Choi, J. Single-Cell Sequencing in Cancer: Recent Applications to Immunogenomics and Multi-omics Tools. Genomics Inform. 16, (2018).
- Klauschen, F., et al. Scoring of tumor-infiltrating lymphocytes: From visual estimation to machine learning. Seminars in Cancer Biology. 52 (Pt 2), 151-157 (2018).
- Danaher, P., et al. Gene expression markers of Tumor Infiltrating Leukocytes. Journal for ImmunoTherapy of Cancer. 5, 18 (2017).
- Aran, D., Hu, Z., Butte, A. J. xCell: digitally portraying the tissue cellular heterogeneity landscape. Genome Biology. 18 (1), 220 (2017).
- Newman, A. M., et al. Robust enumeration of cell subsets from tissue expression profiles. Nature Methods. 12 (5), 453-457 (2015).
- Becht, E., et al. Estimating the population abundance of tissue-infiltrating immune and stromal cell populations using gene expression. Genome Biology. 17 (1), 218 (2016).
- Newman, A. M., Gentles, A. J., Liu, C. L., Diehn, M., Alizadeh, A. A. Data normalization considerations for digital tumor dissection. Genome Biology. 18 (1), 128 (2017).
- Chen, S. H., et al. A gene profiling deconvolution approach to estimating immune cell composition from complex tissues. BMC Bioinformatics. 19 (Suppl 4), 154 (2018).
- Yoshihara, K., et al. Inferring tumour purity and stromal and immune cell admixture from expression data. Nature Communications. 4, 2612 (2013).
- Foley, J. W., et al. Gene-expression profiling of single cells from archival tissue with laser-capture microdissection and Smart-3SEQ. Genome Research. , (2019).
- Civita, P., et al. Laser Capture Microdissection and RNA-Seq Analysis: High Sensitivity Approaches to Explain Histopathological Heterogeneity in Human Glioblastoma FFPE Archived Tissues. Front Oncol. 9, 482 (2019).
- . PD-L1 in cancer: ESMO Biomarker Factsheet | OncologyPRO Available from: https://oncologypro.esmo.org/Education-Library/Factsheets-on-Biomarkers/PD-L1-in-Cancer (2019)
- Haslam, A., Prasad, V. Estimation of the Percentage of US Patients With Cancer Who Are Eligible for and Respond to Checkpoint Inhibitor Immunotherapy Drugs. JAMA Network Open. 2 (5), e192535 (2019).
- Maecker, H. T., McCoy, J. P., Nussenblatt, R. Standardizing immunophenotyping for the Human Immunology Project. Nature Reviews Immunology. 12 (3), 191-200 (2012).
- Schillebeeckx, I., et al. Analytical Performance of an Immunoprofiling Assay Based on RNA Models. Association for Molecular Pathology 2019 Annual Meeting. Journal of Molecular Diagnostics. 21, (2019).
- Uryvaev, A., Passhak, M., Hershkovits, D., Sabo, E., Bar-Sela, G. The role of tumor-infiltrating lymphocytes (TILs) as a predictive biomarker of response to anti-PD1 therapy in patients with metastatic non-small cell lung cancer or metastatic melanoma. Medical Oncology. 35 (3), 25 (2018).
- Wang, K., Shen, T., Siegal, G. P., Wei, S. The CD4/CD8 ratio of tumor-infiltrating lymphocytes at the tumor-host interface has prognostic value in triple-negative breast cancer. Human Pathology. 69, 110-117 (2017).
- Carney, W. P., Bhagat, M., LaFranzo, N. Multidimensional gene expression models for characterizing response and metastasis in solid tumor samples [abstract]. American Association for Cancer Research Annual Meeting. Cancer Research. 79 (13 Suppl), (2019).
転載および許可
このJoVE論文のテキスト又は図を再利用するための許可を申請します
許可を申請This article has been published
Video Coming Soon
Copyright © 2023 MyJoVE Corporation. All rights reserved