Method Article
Modelado inmune predictivo de tumores sólidos
En este artículo
Resumen
El uso de un enfoque basado en ARN para determinar los perfiles inmunitarios cuantitativos de los tejidos tumorales sólidos y aprovechar las cohortes clínicas para el descubrimiento de biomarcadores de oncitología se describe a través de protocolos moleculares e informáticos.
Resumen
Las inmunoterapias son prometedoras en el tratamiento de pacientes oncológicos, pero la heterogeneidad compleja del microambiente tumoral hace que la predicción de la respuesta al tratamiento sea un reto. Se ha demostrado que la capacidad de resolver las poblaciones relativas de células inmunitarias presentes en el tejido tumoral y sus alrededores es clínicamente relevante para entender la respuesta, pero está limitada por técnicas tradicionales como la citometría de flujo y la inmunohistoquímica ( IHC), debido a la gran cantidad de tejido requerido, la falta de marcadores de tipo celular precisos, y muchos obstáculos técnicos y logísticos. Un ensayo (por ejemplo, el ensayo de perfiles inmunes de ImmunoPrism) supera estos desafíos al acomodar tanto pequeñas cantidades de ARN como ARN altamente degradado, características comunes del ARN extraído del tejido tumoral sólido clínicamente archivado. Se accede al ensayo a través de un kit de reactivos y una informática basada en la nube que proporciona una solución de inmunoperfiles cuantitativa de extremo a extremo y de alto rendimiento para plataformas de secuenciación de Illumina. Los investigadores comienzan con tan sólo dos secciones de tejido de parafina incrustada en parafina fija (FFPE) o 20-40 ng de ARN total (dependiendo de la calidad de la muestra), y el protocolo genera un informe de perfil inmune que cuantifica ocho tipos de células inmunitarias y diez de escape inmune genes, capturando una visión completa del microambiente tumoral. No se requiere ningún análisis bioinformático adicional para hacer uso de los datos resultantes. Con las cohortes de muestra apropiadas, el protocolo también puede utilizarse para identificar biomarcadores estadísticamente significativos dentro de una población de pacientes de interés.
Introducción
La cuantificación de linfocitos tumorales infiltrantes (TIL) y otras moléculas relacionadas con el sistema inmunitario en muestras de tejido humano sólido con incrustación de formalina y parafina (FFPE) ha demostrado valor en la investigación clínica1,2,3. Las técnicas comunes como la citometría de flujo y la secuenciación de ácido ribonucleico de una sola célula (ARN) son útiles para el tejido fresco y la sangre4,pero no son adecuadas para el análisis de materiales FFPE debido a la incapacidad para crear suspensiones celulares viables. Los métodos actuales que se han utilizado para cuantificar estas células en el tejido FFPE sufren de grandes desafíos. La inmunohistoquímica (IHC) y otros flujos de trabajo de imágenes similares requieren anticuerpos específicos para detectar proteínas de superficie celular, que pueden ser difíciles de estandarizar entre laboratorios para permitir la cuantificación reproducible5. Plataformas como el sistema nCounter se basan en la expresión de genes únicos para definir células inmunitarias clave6,limitando la sensibilidad y la especificidad de la detección. Los métodos de secuenciación de ARN más genéricos, junto con las herramientas de software independientes, están disponibles, pero requieren una optimización y validación significativas antes de utilizar7,8,9,10,11,12. Los avances recientes en la combinación de microdisección de captura láser (LCM) con secuenciación de ARN para el tejido FFPE han demostrado ser prometedores; sin embargo, se requiere una solución llave en mano más de alto rendimiento para los estudios traslacionales destinados a identificar biomarcadores robustos13,14. Los métodos para generar biomarcadores multidimensionales, como el modelado inmune predictivo, que definen las cohortes de pacientes, incluidos los respondedores de terapia, los subtipos de cáncer o los resultados de supervivencia con alta precisión predictiva y significación estadística, son cada vez más importantes en la era de la medicina de precisión y la inmunoterapia15,16.
Para hacer frente a esta necesidad, se desarrolló un ensayo de perfiles inmunes para permitir la cuantificación sensible y específica de las células inmunitarias en el tejido FFPE tumoral sólido utilizando reactivos estandarizados de secuenciación de ARN e informática basada en la nube. Además de acomodar el ARN degradado del tejido FFPE, el protocolo es capaz de acomodar el ARN derivado de muestras de tejido limitantes, como biopsias de agujas de núcleo, aspiradores de agujas y tejido micro diseccionado o macrodiseccionado. Los datos de ARN de cada muestra se comparan con una base de datos de modelos de expresión génica de células inmunitarias, llamados modelos de expresión de salud inmune, para cuantificar las células inmunitarias como un porcentaje del total de células presentes en la muestra. En resumen, estos modelos se construyeron utilizando métodos de aprendizaje automático para identificar patrones de expresión multigénicos únicos a partir de datos de transcriptoma completo generados a partir de poblaciones de células inmunitarias purificadas (aisladas mediante marcadores canónicos de superficie celular)17,18. Los modelos multidimensionales de expresión de la salud subyacentes a la tecnología permiten al ensayo cuantificar cada célula inmune como un porcentaje del total de células presentes en la mezcla heterogénea. Esto permite al investigador generar comparaciones entre células intravenales e intramuestras, que han demostrado tener un valor clínico19,20. Otras aplicaciones incluyen la cuantificación de la respuesta inmune antes y después del tratamiento, como se describe en los resultados representativos. El ensayo informa sobre múltiples características de la contextura inmune del tumor y el microambiente tumoral, incluidos los porcentajes absolutos de ocho tipos de células inmunitarias (derivados de modelos de expresión génica): células CD4+ T, células CD8+ T, CÉLULAs CD56+ Natural Killer, células CD19+ B, CD14+ monocitos, Tregs, macrófagos M1 y macrófagos M2. Además, el ensayo informa de la expresión (en transcripciones por millón, o TPM) de diez genes de escape inmune: PD-1, PD-L1, CTLA4, OX40, TIM-3, BTLA, ICOS, CD47, IDO1 y ARG1.
El kit de reactivos se utiliza para preparar las bibliotecas de alta calidad para la secuenciación en una plataforma Illumina siguiendo un método híbrido de preparación de bibliotecas basada en captura, como se muestra en la Figura 1. Si un investigador no tiene una plataforma de secuenciación de Illumina en su laboratorio, puede enviar sus muestras a un laboratorio central para la secuenciación. Una vez generados, los datos de secuenciación se cargan en Prism Portal para el análisis automatizado, y se devuelve al usuario un perfil completo y cuantitativo para cada muestra individual, en forma de Informe Inmune(Figura 2A). Los usuarios también pueden definir agrupaciones de muestras en el Portal Prism para generar un Informe de Biomarcador(Figura 2B),destacando biomarcadores estadísticamente significativos que distinguen a dos cohortes de pacientes. Es importante destacar que los datos generados por el kit de reactivos son solo para uso de investigación y no se pueden utilizar con fines de diagnóstico.

Figura 1: Información general sobre el flujo de trabajo. En este protocolo, el ARN se convierte primero en ADNr. Los adaptadores de secuenciación están ligados, y el ADNc con ligadura de adaptadores es amplificado y con código de barras por PCR para crear una biblioteca de captura previa. Las sondas biotinylated se hibridan a objetivos específicos de ADNc que luego se capturan usando cuentas de estreptavidina. El lavado no está enlazado y no objetivo. Un enriquecimiento final de PCR produce una biblioteca posterior a la captura lista para la secuenciación. *El ARN total debe ser de muestras humanas; puede estar intacto o degradado (FFPE) ARN. Haga clic aquí para ver una versión más grande de esta figura.

Figura 2: Informes inmunes representativos. El flujo de trabajo genera dos informes, un informe inmune individual (A) para cada muestra procesada y un informe de biomarcador (B) para cohortes de pacientes definidas. Haga clic aquí para ver una versión más grande de esta figura.
El protocolo requiere aproximadamente 16 horas de tiempo de preparación (desde el ARN total hasta las bibliotecas listas para la secuenciación); sin embargo, hay una serie de puntos de parada opcionales, como se indica en el protocolo. El ensayo hace uso de la naturaleza rica y dinámica de la transcriptomica para ir más allá de biomarcadores de analito único heredados a modelos de expresión génica multidimensional, permitiendo así una caracterización biológica integral de muestras de tejidos con estandarizado reactivos y herramientas de software fáciles de usar. Permite a los investigadores utilizar una tecnología contemporánea en su propio laboratorio, aprovechando el aprendizaje automático y una base de datos de modelos de expresión sanitaria para obtener perfiles inmunes más precisos y cuantitativos de muestras clínicas preciosas, y descubrir biomarcadores de ARN multidimensionales con análisis estadístico completo.
Protocolo
Las muestras de tejido humano utilizadas en los resultados representativos que se muestran aquí se compraron a una entidad de buena reputación (TriStar Technology Group) y han informado el consentimiento del donante que permite la investigación académica y comercial, así como la aprobación de un Comité.
Parte I: Preparación previa a la captura de la biblioteca
1. Cuantificación y Calificación del ARN
- Cuantifique el ARN utilizando un ensayo fluorométrico para determinar la entrada adecuada para el ensayo. Evaluar la calidad del ARN de entrada utilizando la electroforesis para determinar el número de integridad del ARN (RIN) y el porcentaje de fragmentos >200 valores de nucleótidos (DV200).
- Para muestras de ARN intactas (RIN > 7) o parcialmente degradadas (RIN 2 a 7) siga los pasos de preparación de la biblioteca para el ARN de alta calidad/intacto, comenzando con el Paso 2.1. La calidad del ARN es importante para seleccionar el tiempo de fragmentación correcto en el Programa ciclodes térmicos #1 (Tabla Suplementaria 2).
- Para muestras altamente degradadas (p. ej., RIN 1 a 2 o FFPE), determine el valor DV200. Estas muestras no requieren fragmentación y seguirán las instrucciones para el ARN degradado, comenzando con el Paso 2.2.
- Preparar la cantidad apropiada de ARN total para cada muestra diluyendo 20 ng de ARN (ARN de alta calidad/intacto con RIN > 2) o 40 ng de ARN (ARN degradado/FFPE con DV200 > 20%) a 5 l en agua libre de nucleasas. No se recomienda procesar muestras con DV200 < 20%. Para las muestras de ARN de control provistas con el kit, diluir 1 l del ARN apropiado en 4 ml de agua libre de nucleasas. Las muestras de control seguirán el mismo procesamiento que se describe para los materiales de ARN de alta calidad (intacto) o degradado (FFPE), tal como se etiquetan. Consulte la Tabla Suplementaria 1 para todos los reactivos incluidos en el kit.
2. Fragmentación y priming del ARN
- Siga el paso 2.1.1 para EL ARN de alta calidad/intacto con RIN > 2.
- Para ARN de alta calidad, ensamble la fragmentación y la reacción de cebado sobre hielo en un tubo PCR libre de nucleasas de acuerdo con la Tabla 1.
- Mezcle bien pipeteando hacia arriba y hacia abajo varias veces. Luego, gire brevemente las muestras en una microcentrífuga
NOTA: Para todos los giros centrífugos en el protocolo, se recomienda una velocidad de 1.000 x g durante al menos 3 s. - Coloque las muestras en un ciclor térmico y utilice el programa #1 (Tabla Suplementaria 2).
- Transfiera inmediatamente los tubos al hielo y proceda a First Strand cDNA Synthesis for High Quality RNA (Paso 3.1). Para la preparación simultánea de ARN de alta calidad y FFPE, comience la preparación del ARN FFPE (Paso 2.2) durante la incubación de fragmentación.
- Mezcle bien pipeteando hacia arriba y hacia abajo varias veces. Luego, gire brevemente las muestras en una microcentrífuga
- Para ARN de alta calidad, ensamble la fragmentación y la reacción de cebado sobre hielo en un tubo PCR libre de nucleasas de acuerdo con la Tabla 1.
| Fragmentación y Mezcla de Priming | Volumen (L) |
| ARN intacto o parcialmente degradado (20 ng) | 5 |
| Búfer de reacción de síntesis de primer hilo | 4 |
| Primers aleatorios | 1 |
| Volumen total | 10 |
Tabla 1: Fragmentación y reacción de cebado para ARN de alta calidad. Los componentes de la fragmentación y la reacción de cebado para EL ARN de alta calidad deben ensamblarse y mezclarse sobre hielo de acuerdo con los volúmenes mostrados. Se puede hacer una mezcla maestra de búfer de reacción de síntesis de primera hebra y imprimaciones aleatorias que se pueden añadir a las muestras de ARN.
- Siga el paso 2.2.1 para ARN degradado/FFPE con DV200 > 20%.
- Para arns altamente degradados (FFPE) que no requieran fragmentación, ensamble la reacción de cebado como se describe en el Cuadro 2. Para el ARN intacto, recuerde seguir el Paso 2.1.
- Mezcle bien pipeteando hacia arriba y hacia abajo varias veces. A continuación, gire brevemente las muestras en una microcentrífuga.
- Coloque las muestras en un ciclor térmico y utilice el programa #2 (Tabla Suplementaria 2).
- Transfiera los tubos al hielo y proceda a First Strand cDNA Synthesis for Highly Degraded (FFPE) RNA (Paso 3.2).
- Para arns altamente degradados (FFPE) que no requieran fragmentación, ensamble la reacción de cebado como se describe en el Cuadro 2. Para el ARN intacto, recuerde seguir el Paso 2.1.
| Reacción de priming | Volumen (L) |
| ARN FFPE (40 ng) | 5 |
| Primers aleatorios | 1 |
| Volumen total | 6 |
Tabla 2: Reacción de cebado aleatorio para ARN altamente degradado. Los componentes de la reacción de cebado para ARN altamente degradado deben ensamblarse sobre hielo en un tubo de PCR libre de nucleasas.
3. Primera síntesis de ADNc Dehebrado
- Siga el paso 3.1.1 para EL ARN de alta calidad/intacto con RIN > 2.
- Para el ARN intacto (alta calidad), ensamble la reacción de síntesis de primera hebra sobre hielo en un tubo pcR libre de nucleasas según la Tabla 3.
- Manteniendo las reacciones sobre hielo, mezcle completamente pipeteando hacia arriba y hacia abajo varias veces. Gire brevemente las muestras en una microcentrífuga y proceda directamente a la incubación de la primera síntesis de hebras (Paso 4).
- Para el ARN intacto (alta calidad), ensamble la reacción de síntesis de primera hebra sobre hielo en un tubo pcR libre de nucleasas según la Tabla 3.
| Síntesis del primer hilo | Volumen (L) |
| ARN fragmentado y cebado (paso 2.1.3) | 10 |
| Reactivo de especificidad de síntesis de primer hilo | 8 |
| Mezcla de enzimas de síntesis de hebras | 2 |
| Volumen total | 20 |
Tabla 3: Reacción de la primera síntesis de hebras para ARN de alta calidad. Los componentes de la fragmentación y la reacción de cebado para ARN de alta calidad deben ensamblarse y mezclarse en hielo de acuerdo con los volúmenes indicados. Se puede hacer y añadir una mezcla maestra de First Strand Synthesis Eagent y First Strand Synthesis Enzyme Mix a las muestras de ARN fragmentadas y cebadas.
- Siga el paso 3.2.1 para ARN degradado/FFPE con DV200 > 20%.
- Para el ARN altamente degradado (FFPE), ensamble la reacción de síntesis de primera hebra sobre hielo en un tubo pcR libre de nucleasas según la Tabla 4.
- Manteniendo las reacciones sobre hielo, mezcle completamente pipeteando hacia arriba y hacia abajo varias veces. Gire brevemente las muestras en una microcentrífuga y proceda directamente a la incubación de la primera síntesis de hebras (Paso 4).
- Para el ARN altamente degradado (FFPE), ensamble la reacción de síntesis de primera hebra sobre hielo en un tubo pcR libre de nucleasas según la Tabla 4.
| Síntesis del primer hilo | Volumen (L) |
| ARN cebado (paso 2.2.3) | 6 |
| Búfer de reacción de síntesis de primer hilo | 4 |
| Reactivo de especificidad del primer hilo | 8 |
| Mezcla de enzimas de síntesis de hebras | 2 |
| Volumen total | 20 |
Tabla 4: Reacción de la primera síntesis de hebras para ARN altamente degradado. Los componentes de la fragmentación y la reacción de cebado para el ARN altamente degradado deben ensamblarse y mezclarse sobre hielo de acuerdo con los volúmenes mostrados. Se puede hacer y añadir una mezcla maestra de búfer de reacción de síntesis de primer hilo, reactivo de especificidad de síntesis de primera hebra y mezcla de enzimas de síntesis de primera hebra a las muestras de ARN cebado.
4. Incubación de síntesis de primera hebra
- Manteniendo los tubos sobre hielo, mezcle bien pipeteando hacia arriba y hacia abajo varias veces. Gire brevemente las muestras en una microcentrífuga. Incubar las muestras en un ciclor térmico precalentado siguiendo el programa #3 (Tabla Suplementaria 2).
5. Síntesis de ADNc de segundo hilo
- Preparar la segunda reacción de síntesis de ADNc de la segunda hebra en hielo ensamblando los componentes enumerados en la Tabla 5,incluido el primer producto de reacción de hebra del Paso 4.1.
| Reacción de síntesis de segundo hilo | Volumen (L) |
| Primer producto de síntesis de hebras (paso 4.1) | 20 |
| Búfer de reacción de síntesis de segundo hilo | 8 |
| Mezcla de enzimas de síntesis de segunda hebra | 4 |
| Agua libre de nucleasas | 48 |
| Volumen total | 80 |
Tabla 5: Reacción de la segunda síntesis de hebras. Los componentes de la segunda hebra de reacción de síntesis de ADNc deben ensamblarse y mezclarse en hielo de acuerdo con los volúmenes mostrados. Se puede hacer y añadir una mezcla maestra del búfer de reacción de síntesis de segundo hilo, la mezcla de enzimas de síntesis de segunda hebra y el agua libre de nucleasa al producto de síntesis de la primera hebra.
- Manteniendo los tubos sobre hielo, mezcle bien pipeteando hacia arriba y hacia abajo varias veces. Incubar en un ciclor térmico siguiendo el programa #4 (Tabla Suplementaria 2).
6. Limpieza de CDNA usando SPRI (inmovilización reversible de fase sólida) Perlas
- Deje que las perlas SPRI se calienten a temperatura ambiente durante al menos 30 minutos antes de su uso, y luego vórtice SPRI Abalorios durante aproximadamente 30 s para resuspender.
- Añadir 144 l de perlas resuspendidas a la reacción de síntesis de la segunda hebra (80 l). Mezcle bien pipeteando hacia arriba y hacia abajo al menos 10 veces e incubar durante 5 minutos a temperatura ambiente.
- Gire brevemente los tubos en una microcentrífuga y coloque los tubos en un estante magnético para separar las cuentas del sobrenadante. Una vez que la solución esté clara, retire y deseche cuidadosamente el sobrenadante. Tenga cuidado de no molestar a las cuentas, que contienen ADN.
- Añadir 180 s de etanol recién preparado 80% a los tubos mientras está en el bastidor magnético. Incubar a temperatura ambiente durante 30 s, y luego retirar cuidadosamente y desechar el sobrenadante.
- Repita el paso 6.4 una vez para un total de 2 pasos de lavado.
- Retire completamente el etanol residual. Deje los tubos en el estante magnético y seque al aire las perlas durante aproximadamente 3 minutos con la tapa abierta, o hasta que esté visiblemente seca. No seque en exceso las cuentas, ya que esto puede resultar en una menor recuperación del ADN.
- Retire los tubos del imán y añada 53 l 0,1x TE Tampón (incluido en el kit de reactivos, consulte Tabla suplementaria 1) a las perlas. Pipetear hacia arriba y hacia abajo al menos 10 veces para mezclar a fondo. Incubar durante 2 min a temperatura ambiente.
- Coloque los tubos en un bastidor magnético, permitiendo que las perlas se separen completamente del sobrenadante. Transfiera 50 l del sobrenadante a tubos PCR limpios sin nucleasa. Tenga cuidado de no molestar a las cuentas. Este es un punto de parada opcional en el protocolo, las muestras de ADNc pueden almacenarse a -20 oC.
7. Finalizar la reparación de la biblioteca cDNA
- Montar la reacción de reparación final sobre hielo ensamblando los componentes enumerados en la Tabla 6 al segundo producto de síntesis de hebras del Paso 6.8.
| Reacción de reparación final | Volumen (L) |
| Producto de síntesis de segunda hebra (paso 6.8) | 50 |
| Búfer de reacción de reparación final | 7 |
| Mezcla de enzimas de reparación final | 3 |
| Volumen total | 60 |
Tabla 6: Fin de la reacción de reparación. Los componentes de la reacción de reparación final deben montarse y mezclarse sobre hielo de acuerdo con los volúmenes mostrados. Se puede hacer y añadir una mezcla maestra del búfer de reacción de reparación final y la mezcla de enzimas de reparación final al segundo producto de síntesis de hebras.
- Ajuste una pipeta a 50 l y luego pipetee todo el volumen hacia arriba y hacia abajo al menos 10 veces para mezclar a fondo. Centrifugar brevemente para recoger todo el líquido de los lados de los tubos. Es importante mezclar bien. La presencia de una pequeña cantidad de burbujas no interferirá con el rendimiento.
- Incubar las muestras en un ciclor térmico siguiendo el programa #5 (Tabla Suplementaria 2).
8. Ligadura del adaptador
- Antes de configurar la reacción de ligadura, diluir el adaptador en el búfer de dilución del adaptador de hielo frío como se muestra en la Tabla 7,multiplicándose por el número requerido de muestras, más un 10% adicional. Mantenga el adaptador diluido sobre hielo.
| Dilución de ligadura | Volumen (L) |
| Adaptador | 0.5 |
| Tampón de dilución del adaptador | 2 |
| Volumen total | 2.5 |
Tabla 7: Dilución del adaptador. El adaptador debe diluirse sobre hielo con tampón de dilución del adaptador de acuerdo con los volúmenes mostrados.
- Ensamble la reacción de ligadura sobre hielo añadiendo los componentes descritos en la Tabla 8, en el orden indicado, al producto de reacción de preparación final del Paso 7.3. Tenga en cuenta que el Ligation Master Mix y Ligation Enhancer se pueden mezclar con anticipación. Esta mezcla es estable durante al menos 8 h a 4 oC. No premezcle el Ligation Master Mix, el Potenciador de Ligation y el Adaptador antes de usarlo en el Paso de Ligadura del Adaptador.
| Reacción de ligadura | Volumen (L) |
| ADN precortado extremo (paso 7.3) | 60 |
| Adaptador diluido (paso 8.1) | 2.5 |
| Potenciador de la ligadura | 1 |
| Ligation Master Mix | 30 |
| Volumen total | 93.5 |
Tabla 8: Reacción de ligadura. Los componentes de la reacción de ligadura del adaptador deben ensamblarse sobre hielo de acuerdo con los volúmenes mostrados en el orden indicado. Se puede hacer una mezcla maestra de Ligation Enhancer y Ligation Master Mix y añadirla al ADN end Prepped con adaptador diluido. No mezcle el adaptador diluido y el Ligation Master Mix o Ligation Enhancer antes de mezclar el con el ADN preparado al final.
- Ajuste una pipeta a 80 l y luego pipetee todo el volumen hacia arriba y hacia abajo al menos 10 veces para mezclar a fondo. Realice un giro rápido para recoger todo el líquido de los lados de los tubos. El Ligation Master Mix es muy viscoso. Tenga cuidado de asegurar una mezcla adecuada de la reacción de ligadura, ya que la mezcla incompleta resultará en una menor eficiencia de la ligadura. La presencia de una pequeña cantidad de burbujas no interferirá con el rendimiento.
- Incubar después de la #6 del programa(Tabla Suplementaria 2) y, a continuación, eliminar la mezcla de ligadura del ciclor térmico y añadir 3 l de enzima de procesamiento del adaptador, lo que resulta en un volumen total de 96,5 l.
- Pipetear hacia arriba y hacia abajo varias veces para mezclar bien, y luego incubar después de la #7 del Programa(Tabla Suplementaria 2) antes de proceder inmediatamente a la Purificación de la Reacción de Ligación.
9. Purificación de la reacción de ligación utilizando SPRI Neads
- Deje que las perlas SPRI se calienten a temperatura ambiente durante al menos 30 minutos antes de su uso, y luego vórtice SPRI Abalorios durante aproximadamente 30 s para resuspender.
- Añadir 87 s de perlas SPRI resuspendidas y mezclar bien pipeteando hacia arriba y hacia abajo al menos 10 veces. Incubar durante 10 min a temperatura ambiente.
- Gire brevemente los tubos en una microcentrífuga y coloque los tubos en un estante magnético para separar las cuentas del sobrenadante. Después de que la solución esté clara (5 min), retire y deseche cuidadosamente el sobrenadante. No deseche las perlas.
- Añadir 180 s de etanol recién preparado 80% a los tubos mientras está en el bastidor magnético. Incubar a temperatura ambiente durante 30 s, y luego retirar cuidadosamente y desechar el sobrenadante. Repita el paso 9.4 una vez para un total de 2 pasos de lavado.
- Retire completamente el etanol residual. Deje los tubos en el estante magnético y seque al aire las perlas durante aproximadamente 3 minutos con la tapa abierta, o hasta que esté visiblemente seca. No seque en exceso las cuentas, ya que esto puede resultar en una menor recuperación del ADN.
- Retire los tubos del imán y agregue 17 sl de tampón TE de 0,1x a las perlas. Pipetear hacia arriba y hacia abajo al menos 10 veces para mezclar a fondo. Incubar durante 2 minutos a temperatura ambiente, y luego colocar los tubos en un estante magnético, permitiendo que las cuentas se separen completamente del sobrenadante.
- Transfiera 15 l del sobrenadante a tubos PCR limpios sin nucleasa. Tenga cuidado de no molestar a las cuentas. Este es un punto de parada opcional en el protocolo, el ADN ligado por el adaptador puede almacenarse a -20 oC.
10. Enriquecimiento de PCR del ADN ligado del adaptador
- Configure la reacción PCR como se describe en la Tabla 9. Se puede hacer y añadir una mezcla maestra que contenga la mezcla maestra de PCR pre-captura y la Imprimación Universal al ADN ligado del adaptador. Para la secuenciación multiplexada, utilice imprimaciones de índice únicas para cada reacción y agréguelas a cada muestra individualmente.
| Enriquecimiento de PCR | Volumen (L) |
| ADN ligado del adaptador (Paso 10.1) | 15 |
| Pre-Captura PCR Master Mix | 25 |
| Universal PCR Primer | 5 |
| Primer índice (X) | 5 |
| Volumen total | 50 |
Tabla 9: Enriquecimiento de PCR del ADN ligado del adaptador. Los componentes del enriquecimiento de PCR de la reacción de ADN ligada del adaptador deben ensamblarse y mezclarse sobre hielo de acuerdo con los volúmenes mostrados. Se puede hacer y añadir al ADN ligado al adaptador la mezcla maestra de PCR pre-captura y la Universal PCR Primer. Para la secuenciación multiplexada, cada muestra debe tener un Primer índice único.
- Mezcle bien pipeteando suavemente hacia arriba y hacia abajo 10 veces. Girar brevemente los tubos en una microcentrífuga y colocar en un ciclor térmico y realizar amplificación de PCR utilizando #8 del programa (Tabla Suplementaria 2).
11. Purificación de la reacción pcR utilizando perlas SPRI
- Deje que las perlas SPRI se calienten a temperatura ambiente durante al menos 30 minutos antes de su uso, y luego vórtice SPRI Abalorios durante aproximadamente 30 s para resuspender.
- Añadir 45 l de perlas resuspendidas a cada reacción de PCR (50 l). Mezclar bien pipeteando hacia arriba y hacia abajo al menos 10 veces, antes de incubar durante 5 minutos a temperatura ambiente.
- Gire brevemente los tubos en una microcentrífuga y coloque los tubos en un estante magnético para separar las cuentas del sobrenadante. Después de que la solución esté clara (5 min), retire y deseche cuidadosamente el sobrenadante. Tenga cuidado de no perturbar las cuentas que contienen ADN.
- Añadir 180 s de etanol recién preparado 80% a los tubos mientras está en el bastidor magnético. Incubar a temperatura ambiente durante 30 s, y luego retirar cuidadosamente y desechar el sobrenadante. Repita el paso 11.4 una vez para un total de 2 pasos de lavado.
- Retire completamente el etanol residual. Deje los tubos en el estante magnético y seque al aire las perlas durante aproximadamente 3 minutos con la tapa abierta, o hasta que esté visiblemente seca. No seque en exceso las cuentas, ya que esto puede resultar en una menor recuperación del ADN.
- Retire los tubos del imán y añada un tampón TE de 23 l 0,1x a las perlas. Pipetear hacia arriba y hacia abajo al menos 10 veces para mezclar a fondo. Incubar durante 2 min a temperatura ambiente.
- Coloque los tubos en un bastidor magnético, permitiendo que las perlas se separen completamente del sobrenadante. Transfiera 20 l del sobrenadante a tubos pcR limpios sin nucleasa. Tenga cuidado de no molestar a las cuentas. Este es un punto de parada opcional en el protocolo, las bibliotecas de pre-captura se pueden almacenar a -20 oC.
12. Validar y cuantificar la biblioteca de precaptura
- Mida la concentración de la biblioteca de captura previa utilizando un fluorómetro y un kit de ensayo de alta sensibilidad. Se requiere un rendimiento mínimo de 200 ng para proceder a la Parte II: Hibridación y Captura.
- Ejecutar 1 l de biblioteca en un sistema de electroforesis digital. Si es necesario, diluir la muestra para evitar sobrecargar el chip de alta sensibilidad, de acuerdo con las recomendaciones del protocolo del fabricante.
- Compruebe que el electroferograma muestra una distribución estrecha con un tamaño máximo aproximado de 250-400 bp (consulte Resultados representativos, Figura 3 y Figura 4).
- Si un pico de 128 bp (adaptor-dimer) es visible en los rastros del Bioanalizador, y la intensidad de la señal es de la intensidad de la señal de la biblioteca de 250-400 bp (ver Resultados Representativos, Figura 5), y luego subir el volumen de la muestra (del paso 11.7) a 50 oL con 0.1x TE Buffer y repetir la purificación de SPRI Bead (Paso 11). Este es un punto de parada opcional en el protocolo, las bibliotecas de precaptura se pueden almacenar a -20 oC antes de pasar a la Parte II: Hibridación y captura de ImmunoPrism.
Parte II: Hibridación y Captura
13. Combinar Oligos de Bloqueo, ADN de Cot-1, ADN de la biblioteca de captura previa y seco
- Mezclar la biblioteca de códigos de barras preparada en el paso 11 y cuantificada en el paso 12, con ADN de cuna-1 y oligos de bloqueo en un tubo de PCR libre de nucleasas o microtubo de 1,5 ml, como se muestra en la Tabla 10.
| Reactivo | Cantidad/Volumen |
| Biblioteca de códigos de barras del paso 10.10 | 200 ng |
| ADN de cot-1 | 2 g |
| Bloqueo de Oligos | 2 l |
Tabla 10: Preparación y secado por hibridación. Los componentes que se combinarán para el secado de las bibliotecas en preparación de la hibridación deben ensamblarse de acuerdo con las cantidades mostradas.
- Seque el contenido del tubo utilizando un concentrador de vacío ajustado a 30-45 oC. Este es un punto de detención opcional en el protocolo. Después del secado, los tubos pueden almacenarse durante la noche a temperatura ambiente (15-25 oC) o durante más tiempo a -20 oC.
14. Hibridar sondas de captura de ADN con la biblioteca
- Descongelación 2x Tampón de lavado de cuentas y búfer de hibridación, potenciador de búfer de hibridación, panel de sonda InmunoPrism, 10x búfer de lavado 1, 10x búfer de lavado 2, búfer de lavado 10x 3 y búfer de lavado estricto de 10x a temperatura ambiente. Antes de su uso, inspeccione el búfer de hibridación para la cristalización de sales. Si hay cristales, caliente el tubo a 65 oC, agitando intermitentemente, hasta que el tampón esté completamente solubilizado.
- A temperatura ambiente, cree la Hybridization Master Mix en un tubo. Multiplique los volúmenes por el número de muestras y añada un 10% adicional, siguiendo el Cuadro 11.
| Hybridization Master Mix | Volumen (L) |
| Búfer de hibridación | 8.5 |
| Hybridization Buffer Enhancer | 2.7 |
| Panel de sonda ImmunoPrism | 5 |
| Agua libre de nucleasas | 0.8 |
| Volumen total | 17 |
Tabla 11: Mezcla maestra de hibridación. Los componentes de Hybridization Master Mix deben montarse y mezclarse a temperatura ambiente de acuerdo con los volúmenes mostrados.
- Vórtice o pipeta arriba y abajo para mezclar bien. A continuación, añada 17 s de la mezcla maestra de hibridación a cada tubo que contenga ADN seco. Sellar los tubos e incubar durante 5 min a temperatura ambiente.
- Vortex las muestras, asegurándose de que están completamente mezcladas, y espírelas brevemente en una microcentrífuga. Si procede, transfiera cada muestra de un microtubo de 1,5 ml a un tubo PCR sin nucleasas.
- Coloque las muestras en un ciclor térmico y ejecute program#9 (Tabla suplementaria 2).
- Durante la incubación, prepare los tampones de lavado (Paso 15) y las cuentas de estreptavidina (Paso 16), lo que permite tiempo suficiente para precalentar los tampones y equilibrar las cuentas de estreptavidina.
15. Preparar los búferes de lavado
NOTA: Los tampones de lavado se suministran como soluciones concentradas 2x (Bead Wash Buffer) o 10x (todos los demás tampones de lavado).
- Durante la incubación de hibridación, diluir el búfer de lavado de cuentas 2x y los búferes de lavado 10x para crear 1x soluciones de trabajo, multiplicándose por el número requerido de muestras y añadiendo un 10% adicional, siguiendo la Tabla 12. Si 10x Wash Buffer 1 está turbio, caliente la botella en un baño de agua o bloque de calentamiento de 65 oC para resuspender las partículas. Los búferes de lavado congelados 1 veces deben mezclarse después de la descongelación.
| Búferes de lavado | Zona de influencia concentrada (L) | Agua libre de nucleasas (L) | Total (L) |
| Búfer de lavado de cuentas | 150 | 150 | 300 |
| Búfer de lavado 1 | 25 | 225 | 250 |
| Búfer de lavado 2 | 15 | 135 | 150 |
| Búfer de lavado 3 | 15 | 135 | 150 |
| Búfer de lavado estricto | 30 | 270 | 300 |
Tabla 12: Dilución del tampón de lavado. Los tampones de lavado de concentración deben diluirse con agua libre de nucleasas a temperatura ambiente de acuerdo con los volúmenes mostrados.
- Alícuota los 1x tampones de lavado en tubos PCR sin nucleasa y colóquelos a las temperaturas adecuadas como se indica en la Tabla 13. Asegúrese de incluir suficiente exceso de edad para pipetear. Para los buffers calentados, utilice un ciclor térmico ajustado a 65 oC con la tapa establecida en 70 oC.
| Búferes de lavado | Temperatura de retención | Volumen/Tubo (L) | Número de tubos/muestra |
| Búfer de lavado de cuentas | RT (15-25 oC) | 100 | 3 |
| Búfer de lavado 1 | 65 oC | 100 | 1 |
| Búfer de lavado 1 | RT (15-25 oC) | 150 | 1 |
| Búfer de lavado 2 | RT (15-25 oC) | 150 | 1 |
| Búfer de lavado 3 | RT (15-25 oC) | 150 | 1 |
| Búfer de lavado estricto | 65 oC | 150 | 2 |
Tabla 13: Búferes de lavado diluidos. Los tampones de lavado diluidos deben alícitarse en tubos separados de acuerdo con los volúmenes y el número de tubos por muestra mostrados. Los tampones de lavado deben mantenerse a la temperatura indicada antes de su uso.
- Preparar la mezcla de resuspensión de perlas a temperatura ambiente como se muestra en la Tabla 14,multiplicándose por el número requerido de muestras y añadiendo un 10% adicional.
| Mezcla de resuspensión de perlas | Volumen (L) |
| Búfer de hibridación | 8.5 |
| Hybridization Buffer Enhancer | 2.7 |
| Agua libre de nucleasas | 5.8 |
| Volumen total | 17 |
Tabla 14: Mezcla de resuspensión de perlas. Los componentes de Bead Resuspension Mix deben montarse y mezclarse a temperatura ambiente de acuerdo con los volúmenes mostrados.
16. Preparar las cuentas de Streptavidin
- Equilibrar las perlas de estreptavidina a temperatura ambiente durante al menos 30 minutos antes de su uso. Mezcle bien las perlas vórtinas durante 15 s y la alícuota de 50 ml de perlas por captura en un tubo PCR libre de nucleasas.
- Añadir 100 l de 1 x tampón de lavado de cuentas (preparado en el paso 15.1) a cada tubo. Pipetear suavemente 10 veces para mezclar. Coloque el tubo en un bastidor magnético, permitiendo que las perlas se separen completamente del sobrenadante.
- Retire y deseche el sobrenadante transparente. Tenga cuidado de no molestar a las cuentas.
- Realice el siguiente lavado.
- Retirar del bastidor magnético. Agregue 100 s de 1 x Tampón de lavado de cuentas a cada tubo que contenga perlas, y luego pipetee hacia arriba y hacia abajo 10 veces para mezclar.
- Coloque el tubo en el bastidor magnético, permitiendo que las perlas se separen completamente del sobrenadante.
- Retire y deseche con cuidado el sobrenadante transparente.
- Repita el paso 16.4 una vez para un total de dos lavados.
- Retirar del bastidor magnético. Añadir 17 s de mezcla de resuspensión de perlas desde el paso 15.3 a cada tubo. Pipetear hacia arriba y hacia abajo varias veces para mezclar a fondo. Asegúrese de que las perlas no estén pegadas a los lados de los tubos. Si es necesario, gire brevemente los tubos para recoger las cuentas en la parte inferior.
17. Enlazar objetivo hibridado a las perlas de Streptavidin
- Una vez completada la incubación de 4 horas de hibridación, retire las muestras del ciclor térmico y ajuste el ciclor térmico para que incubaa a 65 oC con la tapa calentada a 70 oC.
- Con una pipeta multicanal, transfiera 17 ml de perlas totalmente homogeneizadas a las muestras. Mezcle bien pipeteando hacia arriba y hacia abajo 10 veces.
- Enlazar el ADN a las cuentas colocando los tubos en el ciclor térmico después del programa #10 (Tabla Suplementaria 2). Durante la incubación, retire brevemente los tubos de tiras cada 10-12 minutos y suavemente el vórtice durante 3 s para asegurarse de que las perlas permanezcan en suspensión. Alternativamente, mezcle pipeteando hacia arriba y hacia abajo varias veces. Proceda inmediatamente a Lavar las perlas de Streptavidin (Paso 18).
18. Lavar las perlas de streptavidina para eliminar el ADN sin ataduras
- Utilice los 1o búferes de lavado del paso 15.2 y almacene los tampónes calentados en el ciclor térmico durante los lavados.
- Añadir 100 l precalentados 1x Wash Buffer 1 a los tubos del paso 17.3. Mezcle bien pipeteando hacia arriba y hacia abajo 10 veces. Coloque los tubos en un bastidor magnético, permitiendo que las perlas se separen completamente del sobrenadante.
- Pipetear y desechar el sobrenadante, que contiene ADN sin enlazar. Retirar del bastidor magnético.
- Realice el siguiente lavado de 65 oC.
- Añadir 150 l de tampón de lavado precalentado 1x estricto.
- Mezcle bien pipeteando hacia arriba y hacia abajo al menos 10 veces. Evite las burbujas durante el pipeteo. Asegúrese de que las perlas estén completamente resuspendidas en todos los tubos.
- Incubar en el ciclor térmico a 65oC durante 5 min.
- Coloque los tubos en un bastidor magnético, permitiendo que las perlas se separen completamente del sobrenadante. Pipetear y desechar el sobrenadante, que contiene ADN sin enlazar. Retirar del bastidor magnético.
- Repita el paso 18.4 para un total de dos lavados estrictos.
- Realice el primer lavado a temperatura ambiente.
- Añadir 150 éL de temperatura ambiente 1x Wash Buffer 1.
- Pipetear hacia arriba y hacia abajo de 10 a 20 veces para resuspender completamente las cuentas.
- Sellar los tubos e incubar durante 2 min, alternando entre vórtice suavemente durante 30 s y descansando durante 30 segundos. Asegúrese de que las cuentas en todos los pozos permanezcan completamente resuspendidas en todos los tubos durante toda la incubación.
- Centrifugar brevemente los tubos.
- Coloque los tubos en un bastidor magnético, permitiendo que las perlas se separen completamente del sobrenadante. Pipetear y desechar el sobrenadante.
- Selle los tubos y centrifugar brevemente. Vuelva al bastidor magnético y utilice una pipeta de 10 l para eliminar cualquier tampón de lavado residual.
- Realice el segundo lavado a temperatura ambiente.
- Añadir 150 l de temperatura ambiente 1x Wash Buffer 2.
- Pipetear hacia arriba y hacia abajo de 10 a 20 veces para resuspender completamente las cuentas.
- Sellar los tubos e incubar durante 2 min, alternando entre vórtice suavemente durante 30 s y descansando durante 30 segundos. Asegúrese de que las cuentas en todos los pozos permanezcan completamente resuspendidas en todos los tubos durante toda la incubación.
- Centrifugar brevemente los tubos.
- Transfiera todo el volumen de perlas resuspendidas en Wash Buffer 2 a tubos PCR limpios sin nucleasas. Importante: La transferencia de las perlas a tubos nuevos es importante para evitar la contaminación fuera del objetivo.
- Coloque los tubos en un bastidor magnético, permitiendo que las perlas se separen completamente del sobrenadante. Pipetear y desechar el sobrenadante.
- Selle los tubos y centrifugar brevemente. Vuelva al bastidor magnético y utilice una pipeta de 10 l para eliminar cualquier tampón de lavado residual.
- Realice el tercer lavado a temperatura ambiente.
- Añadir 150 l de temperatura ambiente 1x Wash Buffer 3.
- Pipetear hacia arriba y hacia abajo de 10 a 20 veces para resuspender completamente las cuentas.
- Sellar los tubos e incubar durante 2 min, alternando entre vórtice suavemente durante 30 s y descansando durante 30 segundos. Asegúrese de que las cuentas en todos los pozos permanezcan completamente resuspendidas en todos los tubos durante toda la incubación.
- Centrifugar brevemente los tubos.
- Coloque los tubos en un bastidor magnético, permitiendo que las perlas se separen completamente del sobrenadante. Pipetear y desechar el sobrenadante.
- Selle los tubos y centrifugar brevemente. Vuelva al bastidor magnético y utilice una pipeta de 10 ml para eliminar cualquier tampón de lavado residual.
- Retirar del bastidor magnético y añadir 20 ml de agua libre de nucleasas a las cuentas.
- Pipetear hacia arriba y hacia abajo 10 veces para asegurarse de que las perlas pegadas al lado de los tubos han sido resuspendidas.
- Importante: No deseche las perlas. Utilice los 20 s de perlas resuspendidas con ADN capturado en el paso 19.
19. Realizar el enriquecimiento final, de PCR posterior a la captura
- Preparar la mezcla maestra de PCR posterior a la captura de acuerdo con la siguiente tabla, multiplicando por el número requerido de muestras y añadiendo un 10% adicional, según la Tabla 15.
| Componente de mezcla maestra PCR posterior a la captura | Volumen (L) |
| MasterMix PCR post-captura | 25 |
| Post-Captura PCR Primer Mix | 1.25 |
| Agua libre de nucleasas | 3.75 |
| Volumen total | 30 |
Tabla 15: Mezcla maestra PCR posterior a la captura. Los componentes de Post-Capture PCR Master Mix deben ensamblarse y mezclarse sobre hielo de acuerdo con los volúmenes mostrados.
- Añadir 30 l de la mezcla maestra de PCR posterior a la captura a cada muestra para un volumen de reacción final de 50 l. Mezclar a fondo pipeteando hacia arriba y hacia abajo 10 veces.
- Coloque los tubos PCR en el ciclor térmico e incubar después de la #11 del programa(Tabla Suplementaria 2).
20. Purificar fragmentos de PCR posteriores a la captura
- Deje que las perlas SPRI se calienten a temperatura ambiente durante al menos 30 minutos antes de su uso, y luego vórtice SPRI Abalorios durante aproximadamente 30 s para resuspender.
- Agregue 75 l de perlas resuspendidas a cada captura enriquecida con PCR (50 l). Mezcle bien pipeteando hacia arriba y hacia abajo al menos 10 veces. Las perlas de estreptavidina no interferirán con la purificación del cordón SPRI. Incubar durante 5 min a temperatura ambiente.
- Gire brevemente los tubos en una microcentrífuga y coloque los tubos en un estante magnético para separar las cuentas del sobrenadante. Una vez que la solución esté clara, retire y deseche cuidadosamente el sobrenadante. Tenga cuidado de no molestar a las cuentas, que contienen ADN.
- Añadir 180 s de etanol recién preparado 80% al tubo mientras está en el bastidor magnético. Incubar a temperatura ambiente durante 30 s, y luego retirar cuidadosamente y desechar el sobrenadante.
- Repita el paso 20.4 una vez para un total de 2 pasos de lavado.
- Retire completamente el etanol residual. Deje el tubo en el estante magnético y seque al aire 3 minutos con la tapa abierta, o hasta que se seque visiblemente. No seque demasiado las perlas. Esto puede resultar en una menor recuperación del ADN.
- Retire el tubo del imán. Eluye el ADN de las perlas añadiendo 22 sL de tampón te 0,1x. Mezcle bien pipeteando hacia arriba y hacia abajo varias veces. Incubar durante 2 min a temperatura ambiente. Coloque el tubo en el bastidor magnético hasta que la solución esté clara.
- Retire 20 l del sobrenadante y transfiera a un tubo de PCR limpio libre de nucleasas, teniendo cuidado de no molestar a las perlas. Este es un punto de detención opcional en el protocolo, las bibliotecas se pueden almacenar a -20 oC.
21. Validar y cuantificar la biblioteca
- Mida la concentración de la biblioteca capturada utilizando un fluorómetro y un kit de ensayo de alta sensibilidad.
- Mida la longitud media del fragmento de la biblioteca capturada utilizando un chip de ADN de alta sensibilidad de electroforesis digital y calcule el tamaño medio del fragmento para cada biblioteca utilizando el software del sistema. El tamaño medio del fragmento debe ser de aproximadamente 250-400 bp (consulte Resultados representativos, Figura 6 y Figura 7). Este es un punto de parada opcional en el protocolo, las bibliotecas completadas se pueden almacenar a -20 oC.
22. Secuenciación en una plataforma de secuenciación
- Para la secuenciación, diluir las bibliotecas a 2 nM y seguir las directrices del fabricante para cargar y operar el secuenciador. Secuencialas a una profundidad mínima de 15 millones de lecturas de un solo extremo de al menos 50 bp de longitud.
23. Análisis de los datos de secuenciación para generar perfiles inmunes y descubrir biomarcadores con Prism Portal, una herramienta informática basada en la nube
- Crea una cuenta de Prism visitando https://prism.cofactorgenomics.com/
- Una vez que haya iniciado sesión, haga clic en Enviar nuevo proyecto en la barra de herramientas superior desde cualquier página de Prism para cargar los archivos de secuenciación FASTQ demultiplexados o cargar archivos almacenados en BaseSpace con la cuenta de Prism.
- Complete el formulario Nuevo proyecto, incluido el nombre del proyecto y las muestras por grupo o cohorte. La agrupación de muestras y los nombres de agrupación correspondientes son necesarios para generar el Informe de detección de biomarcadores. Tenga en cuenta que se requiere un mínimo de 3 muestras por grupo para generar el informe de detección de biomarcadores. Haga clic en el botón Iniciar aplicación para enviar el formulario; aparecerá una página de confirmación si se realiza correctamente.
- Mientras ha iniciado sesión, haga clic en Ver resultados en la barra de herramientas superior o en cualquier página de Prism. Prism permite a un usuario ver el estado de los proyectos enviados y ver informes de muestras y biomarcadores por proyecto. Habrá una tabla de proyectos que el usuario ha creado en Prism. La tabla tiene tres columnas para el estado, el nombre y la fecha de envío.
NOTA: El estado de cada proyecto puede ser:
• "En ejecución", donde se está ejecutando el análisis del proyecto, o,
• "Success", donde el análisis del proyecto está completo y los informes están disponibles. - Si un proyecto ha finalizado el análisis (indicado por un estado "Éxito"), consulte los Informes de muestra individuales y un Informe de detección de biomarcadores. Tenga en cuenta que el informe de detección de biomarcadores solo estará disponible si el proyecto incluye el mínimo necesario de tres muestras por grupo.
- Para acceder a estos informes, vuelva a la tabla de proyectos y haga clic en el nombre del proyecto. En esta página del proyecto, habrá una tabla con una fila para cada ejemplo del proyecto. Haga clic en el vínculo de cada fila, en la columna Informe, para acceder al informe individual de cada ejemplo. Inmediatamente debajo de la tabla, haga clic en el vínculo del Informe de detección de biomarcadores. Si no hay vínculos en esta página, el proyecto no ha completado el análisis.
Resultados
Hay una serie de puntos de control a lo largo del protocolo que permiten al usuario evaluar la calidad y la cantidad de materiales generados. Siguiendo el paso 12 descrito en el protocolo, se genera un electroferograma como se muestra en la Figura 3,representativa de una biblioteca de precaptura típica para una muestra de ARN intacta (RIN n.o 7.8).

Figura 3: Seguimiento típico del bioanalizador de la biblioteca de precaptura para una muestra de ARN intacta. Las bibliotecas de captura previa aparecen como un pico amplio alrededor de 250-400 pares de bases (bp) de tamaño. Haga clic aquí para ver una versión más grande de esta figura.
Se debe tener cuidado de evitar la sobreamplificación, como se indica en el segundo pico alrededor de 1.000 bp que se muestra en la Figura 4, un electroferograma representativo de una biblioteca de precaptura generada a partir de una muestra de ARN FFPE (DV200 a 46). Si este pico es pequeño en relación con el pico principal (alrededor de 250-400 pares base (bp), como se muestra), no interferirá con los pasos o análisis descendentes. Si el segundo pico es grande en relación con el pico de 250-400 bp, la biblioteca de pre-captura se puede rehacer con menos ciclos de PCR para reducir la sobreamplificación.
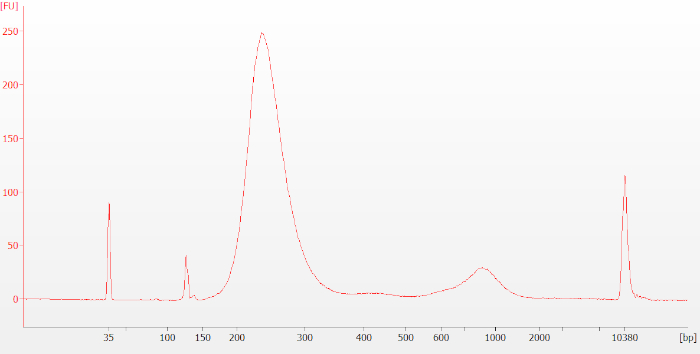
Figura 4: Seguimiento típico del bioanalizador de la biblioteca de captura previa para una muestra de ARN FFPE. El segundo pico alrededor de 1.000 bp es indicativo de sobreamplificación. Si este pico es pequeño en relación con el pico principal alrededor de 250-400 bp (como se muestra), no interferirá con los pasos o análisis aguas abajo. Si el segundo pico es grande en relación con el pico de 250-400 bp, la biblioteca de pre-captura se puede rehacer con menos ciclos de PCR para reducir la sobreamplificación. Haga clic aquí para ver una versión más grande de esta figura.
Como se describe en el paso 12.1.3, se debe evaluar la presencia de atenuadores de adaptador para determinar si es necesaria una limpieza adicional. Los electroferogramas que se muestran en la Figura 5 son representativos de los niveles inaceptables(Figura 5A, DV200 a 33) y aceptables(Figura 5B, DV200 x 46) de dimer adaptador, que aparecen como el pico agudo alrededor de 128 bp.

Figura 5: Seguimientos de bioanalyzer de la biblioteca de captura previa. El dimer del adaptador aparece como un pico agudo alrededor de 128 bp. (A) Los atenuadores de adaptador excesivo están presentes en este electroferograma. (B) Los niveles aceptables del dimer del adaptador se representan en este seguimiento. Ambas trazas muestran evidencia de sobreamplificación leve, pero esto no debe interferir con el ensayo ImmunoPrism. Haga clic aquí para ver una versión más grande de esta figura.
Al finalizar el protocolo, antes de la secuenciación, las bibliotecas finales se evalúan de nuevo utilizando electroforesis digital. Las bibliotecas hechas de ARN FFPE tienden a tener una distribución de tamaño promedio más pequeña que las bibliotecas hechas de ARN intacto. Para las muestras de ARN intactas, el seguimiento resultante debe ser similar a la Figura 6 (RIN 9.5). Para el ARN degradado o FFPE, el seguimiento resultante debe ser similar a la Figura 7 (DV200 a 36).

Figura 6: Seguimiento típico del bioanalizador de la biblioteca final para una muestra de ARN intacta. Las bibliotecas finales aparecen como un pico amplio alrededor de 250-400 pares base (bp) de tamaño. Haga clic aquí para ver una versión más grande de esta figura.

Figura 7: Seguimiento típico del bioanalizador de la biblioteca final para una muestra de ARN FFPE. Las bibliotecas hechas de ARN FFPE tienden a tener una distribución de tamaño promedio más pequeña que las bibliotecas hechas de ARN intacto. Haga clic aquí para ver una versión más grande de esta figura.
Como se describe, los resultados generados con este protocolo se pueden aplicar de dos maneras clave, como se muestra en la Figura 8.
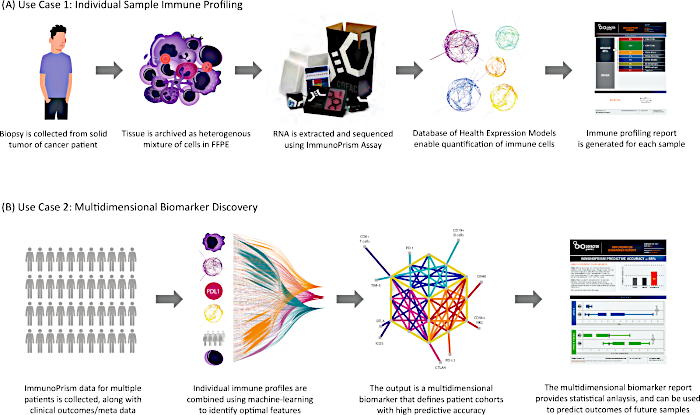
Figura 8: Dos casos de uso del protocolo. Los resultados generados por este ensayo de perfiles inmunes se aplican en dos aplicaciones traslacionales clave. (A) El primer caso de uso comienza a partir del tejido tumoral sólido humano (incluidos los archivos FFPE) y genera un perfil inmune individual para la muestra. (B) Una vez generados para una cohorte de muestras humanas, los datos se combinan utilizando el Portal Prism para generar un biomarcador multidimensional y el correspondiente Informe de Biomarcador. Haga clic aquí para ver una versión más grande de esta figura.
Para demostrar cada uno de estos casos de uso, se incluyen datos representativos de un pequeño estudio traslacional21. Las muestras utilizadas en este estudio son un conjunto de muestras de 7 pacientes diagnosticados y tratados para el cáncer de pulmón de células no pequeñas (NSCLC). Las muestras son tejido tumoral sólido emparejado con el paciente de biopsias previas y posteriores al tratamiento. En primer lugar, se analizaron muestras individuales para generar un perfil inmune, como el informe de ejemplo que se muestra en la Figura 9.

Figura 9: Ejemplo de informe inmune individual para una muestra de NSCLC. La canalización de Prism Portal genera un informe gráfico para cada muestra procesada, con un informe representativo generado para una muestra de tumor sólido NSCLC que se muestra aquí. (A) La parte frontal del informe representa gráficamente la descomposición de las células inmunitarias presentes en la muestra de ARN extraída del tejido FFPE. (B) El reverso del informe incluye una tabla de células inmunitarias (en porcentajes absolutos) y una expresión génica de escape (en transcripciones por millón, o TPM), así como una declaración de rendimiento para el ensayo. Haga clic aquí para ver una versión más grande de esta figura.
Los perfiles inmunológicos pre y post-tratamiento se pueden utilizar para entender cómo una terapia (quimioterapia o radiación, en este estudio) ha modificado el microambiente tumoral. Un ejemplo se muestra en la Figura 10,donde los cambios en el porcentaje para cada célula inmune y el contenido inmune total se muestran antes y después de la quimioterapia, para un solo paciente.
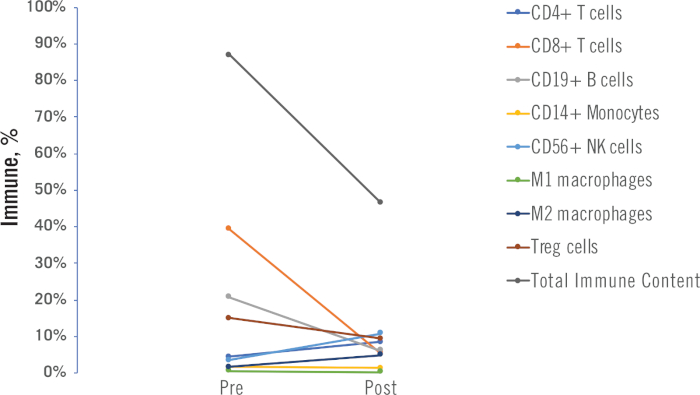
Figura 10: Ejemplo de resultados previos y posteriores al tratamiento. Se muestran datos individuales de células inmunitarias y contenido inmune total generados a partir de muestras previas y posteriores al tratamiento de un solo paciente de CPN. En este ejemplo, el paciente recibió un régimen de quimioterapia como tratamiento. Haga clic aquí para ver una versión más grande de esta figura.
Los pacientes pueden agruparse por criterios como resultados clínicos o fenotipos para la comparación. Por ejemplo, en la Figura 11,las muestras del estudio NSCLC se compararon según el tiempo con la progresión de la enfermedad después del tratamiento. Un subconjunto de los pacientes mostró recurrencia de la enfermedad en >18 meses, y otro subconjunto progresó más rápido, en 18 meses. El valor delta medio (diferencia entre los valores previos y posteriores al tratamiento) se compara para cada muestra para identificar biomarcadores putativos de progresión de la enfermedad.
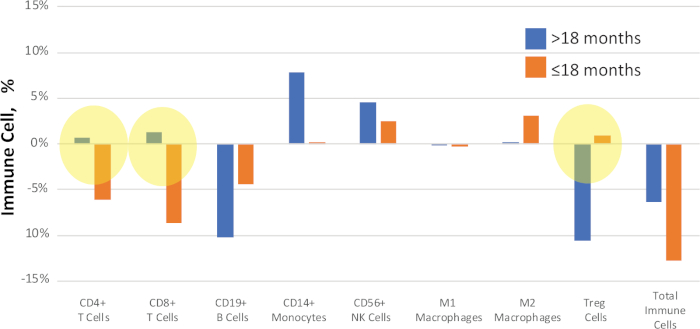
Figura 11: Ejemplo de comparación de resultados clínicos. Los cambios cuantitativos entre los porcentajes de células inmunitarias en las muestras de NSCLC coincidentes antes y después del tratamiento se calcularon y notificaron como el valor "delta". Los resaltados en amarillo muestran cambios claros en la señal entre el estado de supervivencia. Las barras azules representan valores delta medios durante >18 meses hasta la progresión de la enfermedad, las barras naranjas representan valores delta medios durante 18 meses hasta la progresión de la enfermedad. Haga clic aquí para ver una versión más grande de esta figura.
Por último, se pueden utilizar agrupaciones de muestras similares para examinar específicamente las muestras de pretratamiento para identificar biomarcadores predictivos mediante el uso del Portal Prism para generar un informe de biomarcadores. Se muestra en la Figura 12, el mismo fenotipo clínico (progresión de la enfermedad) descrito anteriormente define las agrupaciones de muestras. En este ejemplo, dos genes de escape inmune fueron identificados como diferenciadores estadísticamente significativos de las agrupaciones de muestras (CD47 y OX40, que se muestran en el panel inferior de la Figura 12A). En este ejemplo, dado que los biomarcadores de genes individuales son robustos con una importancia estadística clara, el biomarcador multidimensional no agrega un valor predictivo significativo (ImmunoPrism, tal como se etiqueta en el gráfico de barras superior derecho de la Figura 12B). La tabla completa de datos, incluidos los resultados de los 18 analitos para el ensayo, se resume en el reverso del informe, incluido el análisis estadístico y un breve resumen de los métodos.

Figura 12: Ejemplo de informe de biomarcadorpara muestras NSCLC. La canalización Biomarker Discovery ofrece un informe visual de biomarcadores individuales y un biomarcador multidimensional de aprendizaje automático, con estadísticas detalladas. (A) Para este estudio, la canalización identificó dos biomarcadores individuales (CD47 y OX40) como estadísticamente significativos para definir la progresión de la enfermedad con un umbral de 18 meses. (B) Los detalles sobre el método y los resultados completos se incluyen en el reverso del informe. Haga clic aquí para ver una versión más grande de esta figura.
Tabla suplementaria 1: Materiales del kit de reactivos. Se enumeran una lista de los materiales proporcionados en el kit ImmunoPrism, junto con los números de pieza a los que se hace referencia en el protocolo del fabricante. Todos los demás equipos y materiales requeridos se enumeran en la Tabla de Materiales. Visite https://cofactorgenomics.com/product/immunoprism-kit/ para obtener hojas de datos de seguridad (SDS). Haga clic aquí para ver este archivo (haga clic con el botón derecho para descargar).
Tabla Suplementaria 2: Programas de Ciclor Térmico. Los programas de ciclones recomendados a los que se hace referencia en todo el protocolo se resumen para facilitar la programación. Haga clic aquí para ver este archivo (haga clic con el botón derecho para descargar).
Tabla complementaria 3: Guía del índice de secuenciación. Se enumeran las imprimaciones de índice proporcionadas en el kit de reactivos; se añade una imprimación única a cada reacción para la desmultiplexación posterior a la secuenciación. También se proporcionan combinaciones recomendadas de multiplexación de bajo nivel. Haga clic aquí para ver este archivo (haga clic con el botón derecho para descargar).
Discusión
El protocolo requiere 20 ng intacto o 40 ng de ARN altamente degradado (FFPE). La muestra de ARN debe estar libre de ADN, sales (por ejemplo, Mg2+o sales de guanidinio), agentes quelantes de cationes divalentes (por ejemplo, EDTA, EGTA, citrato) o orgánicos (por ejemplo, fenol y etanol). No se recomienda proceder con muestras de ARN que tengan un DV200 <20%. Se recomienda encarecidamente el uso del ARN de control en el kit, ya que estos controles proporcionan un medio para evaluar el rendimiento a lo largo de todo el protocolo, desde la preparación de la biblioteca hasta el análisis.
El protocolo está diseñado para realizarse utilizando tubos de tira PCR de 0,2 ml. Si se prefiere, el protocolo también se puede realizar utilizando los pozos en una placa PCR de 96 pocillos. Simplemente utilice los pozos de una placa PCR de 96 pocillos en lugar de todas las referencias a tubos PCR o tubos de tiras. Utilice placas PCR con pozos claros solamente, ya que es fundamental confirmar visualmente la resuspensión completa de perlas durante las purificaciones de perlas y los pasos de lavado.
A lo largo del protocolo, mantenga los reactivos congelados o en hielo a menos que se especifique lo contrario. No utilice reactivos hasta que estén completamente desconlos. Asegúrese de mezclar a fondo todos los reactivos antes de su uso.
Mantener las enzimas a -20 oC hasta que estén listas para su uso y vuelva a -20 oC inmediatamente después de su uso. Utilice únicamente agua libre de nucleasas de grado molecular; no se recomienda el uso de agua tratada con DEPC. Cuando pipetee para mezclar, aspire suavemente y dispensar al menos el 50% del volumen total hasta que las soluciones estén bien mezcladas. Mezcla de pipetas todas las mezclas maestras que contienen enzimas. El uso de vórtice para mezclar las enzimas podría conducir a la desnaturalización y comprometer su rendimiento. Durante las purificaciones de cuentas, utilice soluciones de etanol al 80% recién hechas a partir de etanol de grado molecular. El uso de soluciones de etanol que no son frescas puede resultar en rendimientos más bajos. Evite secar en exceso las perlas, ya que esto puede reducir la eficiencia de la elución (las perlas se ven agrietadas si se secan en exceso).
Como se describe en el paso 10, se agregan imprimaciones de índice únicas a cada reacción. Según las secuencias de estos índices, para la multiplexación de bajo nivel, ciertas combinaciones de índices son óptimas. Las secuencias de estos índices son necesarias para desmultiplexar los datos posteriores a la secuenciación. Las secuencias y las combinaciones de multiplexación recomendadas se proporcionan en la Tabla Suplementaria 3. En este mismo paso, es importante tener en cuenta que el número de ciclos de PCR recomendados varía dependiendo de la calidad del ARN utilizado, y, alguna optimización puede ser necesaria para prevenir la sobreamplificación de PCR. Para el ARN de control intacto de InmunoPrism y otro ARN de alta calidad, inicie la optimización con 10 ciclos de PCR. Para el ARN de control de InmunoPrism FFPE y otro ARN altamente degradado/FFPE, inicie la optimización con 15 ciclos de PCR. Se recomienda producir una biblioteca de pruebas utilizando ARN representativo del material a analizar para optimizar los ciclos de PCR. Se debe utilizar el número mínimo de ciclos de PCR que producen consistentemente suficientes rendimientos de la biblioteca de captura previa (>200 ng). Un pico secundario alrededor de 1000 bp en la traza del bioanalizador es indicativo de sobreamplificación(Figura 4). Se debe minimizar la sobreamplificación, pero la presencia de un pequeño pico secundario no interferirá con los resultados del ensayo.
Para minimizar la pérdida de muestray evitar la conmutación de tubos, el paso 13 se puede realizar en tubos pcR, tubos de tiras o una placa PCR de 96 pocillos en lugar de microtubos de 1,5 ml, si el concentrador de vacío lo permite. El rotor se puede quitar en muchos concentradores. Esto permite que los tubos o placas de tiras quepan en el vacío. La concentración de vacío se puede ejecutar utilizando el ajuste de desecación acuosa sin centrifugación. Consulte el manual de su concentrador de vacío para obtener instrucciones. Si las muestras se secan en tubos de tiras o en una placa de 96 pocillos, el paso de hibridación se puede realizar en el mismo recipiente.
Durante el paso 17, asegúrese de vórtice cada 10-12 minutos para aumentar la eficiencia de captura de cuentas. Sujete cuidadosamente las tapas de los tubos de tiras calientes cuando mezcle para evitar que los tubos se abran.
Los lavados descritos en el paso 18 son críticos para evitar una alta contaminación inespecífica y deben seguirse de cerca. Asegúrese de resuspender completamente las perlas en cada lavado, retire completamente los tampón de lavado y, durante el lavado Wash Buffer 2, transfiera las muestras a un tubo de tira nuevo (Paso 18.6.5). Asegúrese de que las perlas de estreptavidina estén completamente resuspendidas y permanezcan en suspensión durante toda la incubación. Salpicaduras en las tapas del tubo no afectará negativamente a la captura. Durante los lavados a temperatura ambiente, se puede utilizar un mezclador de vórtice de microplaca para vórtice las muestras durante la totalidad del período de incubación de dos minutos para facilitar la resuspensión. No deje que las cuentas de estreptavidina se sequen. Si es necesario, extienda las incubaciones en los tampones para evitar el secado de las cuentas. Si utiliza más de un tubo de tira, trabaje con un tubo de tira a la vez para cada lavado mientras los otros tubos de tiras se sentan en el termociclador. Esto puede ayudar a evitar el secado excesivo de las perlas o la prisa, lo que resulta en una resuspensión deficiente u otras técnicas subóptimas. Para los usuarios por primera vez, no se recomienda procesar más de 8 reacciones de biblioteca a la vez.
Las técnicas actuales de perfiles inmunes proporcionan un continuo de información, desde miles de puntos de datos que requieren una interpretación significativa (secuenciación de ARN) hasta un punto de datos discreto individual (IHC de un solo plex). El protocolo descrito aquí representa un enfoque que está en algún lugar en el medio, con un alcance enfocado que permite una alta sensibilidad, pero capturando sólo un subconjunto de datos transcriptomicos clínicamente relevantes. Debido a la naturaleza de la extracción masiva de ARN, este protocolo no proporciona información sobre las relaciones espaciales entre las células inmunitarias y el microambiente tumoral, sin embargo, los resultados pueden complementarse con tecnologías de imágenes para agregar esta información. Hay un sinfín de aplicaciones para los datos generados por este protocolo, ya que hay mucho que aprender sobre la biología del cáncer como enfermedad, y las terapias que se están desarrollando para tratarlo. Como se muestra en los resultados representativos, el informe inmune individual es útil para entender cómo el perfil inmune de un paciente puede cambiar en respuesta a eventos como la progresión de la enfermedad o el tratamiento. Si bien los resultados presentados aquí proporcionan algunos casos de uso de ejemplo, otras aplicaciones como investigar el mecanismo de acción de una terapia y la identificación de biomarcadores putativos de resultados clínicos como la progresión libre y la supervivencia global también son Práctico. Cuando se utiliza este protocolo para aplicaciones de descubrimiento de biomarcadores, es importante practicar un buen diseño de estudio para garantizar que se analicen las poblaciones homogéneas, se incluyan muestras suficientes para la potencia estadística y se consideren fuentes de sesgo. Debido a la naturaleza centrada y aerodinámica del ensayo, es factible imaginar un camino hacia la validación clínica y la aplicación posterior de estos biomarcadores una vez descubiertos.
Divulgaciones
Todos los autores son empleados por Cofactor Genomics, Inc., la empresa que desarrolló y produce el kit de reactivos ImmunoPrism y las herramientas informáticas utilizadas en este artículo. El ensayo ImmunoPrism es solo para uso de investigación y no se utiliza en procedimientos de diagnóstico.
Agradecimientos
Los autores desean reconocer a TriStar Technology Group por proporcionar los especímenes biológicos para los resultados representativos, así como a todos los equipos moleculares, de análisis, productos y comerciales de Cofactor Genomics por su experiencia técnica y su experiencia técnica y su experiencia técnica y su experiencia técnica y su experiencia técnica y su experiencia técnica y su experiencia técnica y su experiencia técnica y su experiencia técnica y su experiencia técnica y su experiencia técnica y su experiencia técnica y su experiencia técnica y su experiencia técnica y su experiencia técnica y su experiencia técnica y su experiencia técnica y su experiencia técnica y su experiencia técnica y su experiencia técnica y su experiencia técnica y su experiencia técnica y su experiencia Apoyo.
Materiales
| Name | Company | Catalog Number | Comments |
| 0.2 mL PCR 8 tube strip | USA Scientific | 1402-2700 | USA Scientific 0.2 mL PCR 8-tube strip |
| 200 Proof Ethanol | MilliporeSigma | EX0276-1 | Prepare 80% by mixing with nuclease-free water on the day of the experiment |
| 96-well thermal cyclers | BioRad | 1861096 | |
| Solid-phase Reversible Immobilization (SPRI) Beads | Beckman-Coulter | A63882 | Agencourt AMPure XP – PCR Purification beads |
| Digital electrophoresis chips and kit | Agilent Technologies | 5067-4626 | Agilent High Sensitivity DNA chips and kit |
| Digital electrophoresis system | Agilent Technologies | G2939AA | Agilent 2100 Electrophoresis Bioanalyzer |
| Streptavidin Beads | ThermoFisher Scientific | 65306 | Dynabeads M-270 Streptavidin |
| ImmunoPrism Kit – 24 reaction | Cofactor Genomics | CFGK-302 | Cofactor ImmunoPrism Immune Profiling Kit – 24 reactions |
| Human Cot-1 DNA | ThermoFisher Scientific | 15279011 | Invitrogen brand |
| Magnetic separation rack | Alpaqua/Invitrogen | A001322/12331D | 96-well Magnetic Ring Stand |
| Microcentrifuge | Eppendorf | 22620701 | |
| Microcentrifuge tubes | USA Scientific | 1415-2600 | USA Scientific 1.5 mL low-adhesion microcentrifuge tube |
| NextSeq550 | Illumina | SY-415-1002 | Any Illumina sequencer may be used for this protocol |
| Nuclease-free water | ThermoFisher Scientific | AM9937 | |
| Prism Extraction Kit | Cofactor Genomics | CFGK-401 | Cofactor Prism FFPE Extraction Kit – 24 samples |
| Purified RNA | - | - | Purified from human tissue samples |
| Fluorometer | ThermoFisher Scientific | Q33226 | Qubit 4 System |
| Fluorometric Assay Tubes | Axygen | PCR-05-C | 0.5mL Thin Wall PCR Tubes with Flat Caps |
| High Sensitivity Fluorometric Reagent Kit | Life Technologies | Q32854 | Qubit dsDNA HS Assay Kit |
| Vacuum concentrator | Eppendorf | 22820001 | VacufugePlus |
| Vortex mixer | VWR | 10153-838 | |
| Water bath or heating block | VWR/USA Scientific | NA/2510-1102 | VWR water bath/USA Scientific heating block |
Referencias
- Brambilla, E., et al. Prognostic Effect of Tumor Lymphocytic Infiltration in Resectable Non-Small-Cell Lung Cancer. Journal of Clinical Oncology. 34 (11), 1223-1230 (2016).
- Iacono, D., et al. Tumour-infiltrating lymphocytes, programmed death ligand 1 and cyclooxygenase-2 expression in skin melanoma of elderly patients: clinicopathological correlations. Melanoma Research. 28 (6), 547-554 (2018).
- Fridman, W. H., Zitvogel, L., Sautes-Fridman, C., Kroemer, G. The immune contexture in cancer prognosis and treatment. Nature Reviews Clinical Oncology. 14 (12), 717-734 (2017).
- Sierant, M. C., Choi, J. Single-Cell Sequencing in Cancer: Recent Applications to Immunogenomics and Multi-omics Tools. Genomics Inform. 16, (2018).
- Klauschen, F., et al. Scoring of tumor-infiltrating lymphocytes: From visual estimation to machine learning. Seminars in Cancer Biology. 52 (Pt 2), 151-157 (2018).
- Danaher, P., et al. Gene expression markers of Tumor Infiltrating Leukocytes. Journal for ImmunoTherapy of Cancer. 5, 18 (2017).
- Aran, D., Hu, Z., Butte, A. J. xCell: digitally portraying the tissue cellular heterogeneity landscape. Genome Biology. 18 (1), 220 (2017).
- Newman, A. M., et al. Robust enumeration of cell subsets from tissue expression profiles. Nature Methods. 12 (5), 453-457 (2015).
- Becht, E., et al. Estimating the population abundance of tissue-infiltrating immune and stromal cell populations using gene expression. Genome Biology. 17 (1), 218 (2016).
- Newman, A. M., Gentles, A. J., Liu, C. L., Diehn, M., Alizadeh, A. A. Data normalization considerations for digital tumor dissection. Genome Biology. 18 (1), 128 (2017).
- Chen, S. H., et al. A gene profiling deconvolution approach to estimating immune cell composition from complex tissues. BMC Bioinformatics. 19 (Suppl 4), 154 (2018).
- Yoshihara, K., et al. Inferring tumour purity and stromal and immune cell admixture from expression data. Nature Communications. 4, 2612 (2013).
- Foley, J. W., et al. Gene-expression profiling of single cells from archival tissue with laser-capture microdissection and Smart-3SEQ. Genome Research. , (2019).
- Civita, P., et al. Laser Capture Microdissection and RNA-Seq Analysis: High Sensitivity Approaches to Explain Histopathological Heterogeneity in Human Glioblastoma FFPE Archived Tissues. Front Oncol. 9, 482 (2019).
- . PD-L1 in cancer: ESMO Biomarker Factsheet | OncologyPRO Available from: https://oncologypro.esmo.org/Education-Library/Factsheets-on-Biomarkers/PD-L1-in-Cancer (2019)
- Haslam, A., Prasad, V. Estimation of the Percentage of US Patients With Cancer Who Are Eligible for and Respond to Checkpoint Inhibitor Immunotherapy Drugs. JAMA Network Open. 2 (5), e192535 (2019).
- Maecker, H. T., McCoy, J. P., Nussenblatt, R. Standardizing immunophenotyping for the Human Immunology Project. Nature Reviews Immunology. 12 (3), 191-200 (2012).
- Schillebeeckx, I., et al. Analytical Performance of an Immunoprofiling Assay Based on RNA Models. Association for Molecular Pathology 2019 Annual Meeting. Journal of Molecular Diagnostics. 21, (2019).
- Uryvaev, A., Passhak, M., Hershkovits, D., Sabo, E., Bar-Sela, G. The role of tumor-infiltrating lymphocytes (TILs) as a predictive biomarker of response to anti-PD1 therapy in patients with metastatic non-small cell lung cancer or metastatic melanoma. Medical Oncology. 35 (3), 25 (2018).
- Wang, K., Shen, T., Siegal, G. P., Wei, S. The CD4/CD8 ratio of tumor-infiltrating lymphocytes at the tumor-host interface has prognostic value in triple-negative breast cancer. Human Pathology. 69, 110-117 (2017).
- Carney, W. P., Bhagat, M., LaFranzo, N. Multidimensional gene expression models for characterizing response and metastasis in solid tumor samples [abstract]. American Association for Cancer Research Annual Meeting. Cancer Research. 79 (13 Suppl), (2019).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoThis article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados