Method Article
הדמיה פלואורסצנטית של מולקולה אחת של דינמיקת DNA פולימראז ב-G-quadruplexes
In This Article
Summary
פרוטוקול זה מתאר בדיקת שכפול DNA חד-מולקולה מבוססת מיקרוסקופיה פלואורסצנטית, המאפשרת הדמיה בזמן אמת של אינטראקציות בין פולימראזות DNA ומכשולים כגון מבני G-quadruplex.
Abstract
היכולת של חלבונים המעורבים בשכפול DNA אוקריוטי להתגבר על מכשולים - כגון 'מחסומים' של חלבונים ו- DNA - היא קריטית להבטחת שכפול גנום נאמן. G-quadruplexes הם מבני חומצות גרעין מסדר גבוה יותר הנוצרים באזורים עשירים בגואנין של DNA והוכחו כמכשולים, המפריעים למסלולי התחזוקה הגנומיים. מחקר זה מציג שיטה מבוססת מיקרוסקופיה פלואורסצנטית בזמן אמת לצפייה באינטראקציות DNA פולימראז עם מבני G-quadruplex. אוליגונוקלאוטידים קצרים ומוקדמים של DNA המכילים G-quadruplex היו משותקים על כיסויי זכוכית פונקציונליים בתוך תא זרימה מיקרופלואידי. הוכנסו פולימראזות DNA עם תווית פלואורסצנטית, מה שאפשר לנטר את התנהגותם ואת הסטוכיומטריה שלהם לאורך זמן. גישה זו אפשרה התבוננות בהתנהגות פולימראז מכיוון שהיא נעצרה על ידי G-quadruplex. באופן ספציפי, באמצעות δ פולימראז שמרים עם תווית פלואורסצנטית, נמצא כי במפגש עם G-quadruplex, הפולימראז עובר מחזור רציף של קשירה וניתוק. ניתן להתאים את הבדיקה הזו של מולקולה בודדת לחקר אינטראקציות בין חלבונים שונים לתחזוקת DNA ומכשולים על מצע ה-DNA.
Introduction
DNA פולימראז הם אנזימים המזרזים שילוב של טריפוספטים נוקלאוזידים כדי לשכפל DNA 1,2,3,4,5. ככאלה, הם ממלאים תפקיד מפתח בתהליכי תחזוקת DNA חיוניים, כולל שכפול DNA 6,7,8 ותיקון 9,10,11,12. DNA פולימראז חייב לשכפל את הגנום בצורה מדויקת ויעילה כדי להבטיח שלמות גנומית, ולמנוע הצטברות של מוטציות בגנום. במהלך הסינתזה, פולימראז נתקל לעתים קרובות ב"מחסומים" כגון חלבונים הקשורים ל-DNA או מבני DNA משניים13. מחסומים אלה יכולים להאט או אפילו לחסום את התקדמות הפולימראז14. התגברות על מחסומים אלה חשובה כדי להבטיח שכפול גנומי נאמן, שכן כישלון לעשות זאת עלול להוביל לחוסר יציבות גנומית15,16.
סוג עיקרי אחד של מחסומים הוא G-quadruplexes (G4), מבני DNA משניים לא קנוניים שהוכחו כנוצרים ברצפים עשירים בגואנין בתוך הגנום האנושי17. ישנם למעלה מ-700,000 רצפים שונים בגנום האנושי המסוגלים ליצור G4, כולל אזורים בתוך טלומרים ומקדמי אונקוגנים18. מבני DNA אלה מאמצים קונפורמציות שונות בהתאם לרצף הנוקלאוטיד, האורך וקטיון המתכת הקשור 19,20,21. המשמעות של גיוון זה היא שפולימראז חייב להתגבר על מגוון טופולוגיות G4 שונות, פוטנציאלית בדרגות שונות של יעילות. הוכח כי כישלון של פולימראזים להתגבר או לעקוף מבנה G4 מעכב את התקדמות מזלג השכפול in vivo, מה שמוביל לחוסר יציבות גנומית22. מחקרי מבחנה הראו כי מבני G4 יכולים לעכב או לחסום לחלוטין פולימראזות שמרים 23,24,25,26,27. היכולת של מבני G4 לעכב או לחסום DNA פולימראז תלויה לחלוטין ביציבות הקינטית והתרמודינמית שלהם, כאשר חלק מהפולימראזים מסוגלים לפרוש28 של G4 מסוימים. בעוד שמחקרים אלה מספקים תובנה לגבי יכולתם של הפולימראזים להתגבר על מחסומי G-quadruplex, הם חסרים את היכולת לדמיין ישירות את התנהגות הפולימראז כאשר הם נתקלים ב-G4. גורלם של הפולימראזים - בין אם הם נשארים קשורים, נושרים או מתחלפים באופן דינמי - קובע אילו תהליכים במורד הזרם נגישים כדי לפתור את ה-G4.
במחקר זה, פותחה בדיקת מיקרוסקופיה חד-מולקולה מבוססת פלואורסצנטיות כדי לדמיין ולנטר ישירות את קשירת ה-DNA פולימראז למבני G4 בזמן אמת. בדיקה זו כוללת קשירת תבניות DNA יוצרות G4 לכיסוי ביוטיני בתא זרימה מיקרופלואידי, שבו ניתן להכניס פולימראזות DNA עם תווית פלואורסצנטית כדי ליזום סינתזת DNA. על ידי מדידת הקרינה של הפולימראזות לאורך זמן, ניתן לצפות ישירות בהתנהגותם בעת מפגש עם מבנה G4. מבנה ה-G4 שנמצא באונקוגן הסרטן c-MYC נבחר לבדיקה זו בשל רמת היציבות הגבוהה שלו. כעת ניתן להתאים פרוטוקול זה לקטלוג ההתנהגות של מגוון פולימראזים בכל תחומי החיים הקשורים לטופולוגיות ויציבות G4 שונות. בדיקה זו מציעה גישה חדשנית ובעלת תפוקה גבוהה להבהרת המנגנונים שבאמצעותם DNA פולימראז מנווט במכשולי DNA, ומספקת כלי רב עוצמה לקידום ההבנה של דינמיקת פולימראז.
Protocol
פרטי הריאגנטים והציוד המשמש מפורטים בטבלת החומרים.
1. ספקטרוסקופיה של דיכרואיזם מעגלי
הערה: לפני פיתוח הבדיקה, היה צורך לבצע ספקטרוסקופיה של דיכרואיזם מעגלי (CD) על רצף G4 שנבחר כדי להבטיח קיפול נכון. רצף 22 nt (5′-TGAGGGTGGGGGGGGGGGGAA-3′) יוצר את מבנה G4 הנגזר מהאונקוגן של סרטן c-MYC . ספקטרוסקופיה של CD בוצעה גם על רצף בקרה (5′- TGAGTGTGAAGACGATAGAA -3) שיש בו נוקלאוטידים מרכזיים ששונו המונעים היווצרות G4.
- כדי להכין את תבניות ה-DNA לספקטרוסקופיה של CD, הוסף 40 מיקרוליטר של 100 מיקרומטר של תבניות 22 nt ל-360 מיקרוליטר של Tris-HCl (pH 8.0), חומצה אתילנדיאמינטטראצטית (EDTA) (TE) בתוספת 200 מ"מ של אשלגן כלורי (KCl). רצוי 10 מיקרומטר של ריכוז DNA סופי לניסויי ספקטרוסקופיה CD.
הערה: נוכחותם של יוני K+ חיונית לייצוב מבנה G4 בתבנית. - מחממים את תמיסות ה-DNA ל-95 מעלות צלזיוס באמבט יבש דיגיטלי למשך 15 דקות. לאחר השלמתו, יש לקרר את התמיסות לאט למשך הלילה על ידי הנחת גוש החימום לפוליסטירן. תהליך קירור איטי זה אחראי לקיפול ה- G4.
- העבר 400 מיקרוליטר של תמיסות רצף היוצרים והבקרה G4 לקובט קוורץ באורך מסלול של 0.1 ס"מ באמצעות מזרק אטום לגז. מלאו את הקובט לאט כדי למנוע היווצרות בועות אוויר.
- מדוד את ספקטרום ה-CD בין 200 ננומטר ל-400 ננומטר באמצעות ספקטרופולארימטר ב-25 מעלות צלזיוס. הפרמטרים והתנאים למדידה זו תוארו בעבר29. ניתן למצוא את התוצאות באיור משלים 1.
הערה: הקפד להקליט את ספקטרום התקליטורים כדי שהמאגר לבדו יפעל כריק.
2. הכנת תבניות DNA
הערה: התבניות הניתנות לשכפול הן תבניות ליניאריות קצרות של 100 nt שהוכנו באמצעות טכניקות ביולוגיה מולקולרית סטנדרטיות. התבנית היוצרת G4 (5′-GAATTACATTTAAATTTTACACAGATACAGTCAATGAGAA
CTTCCAGGCGTAACGAGAGCACGGGGGGGGGGGGGGGGT
GGGACCTTAGCTTCGAGTTCCGAT-3′) מכיל את הרצף המסוגל ליצור G-quadruplex שמקורו ב-MYC (משלב 1) במרכזו. תבנית הבקרה (5′-GAATTACATTTAAATTTTACACAGATACAGTCAATGAGA
ACTTCCAGGCGTAACGAGAGCACGATGTAGCAGAACT
ל-GTGACCTTAGCTTCGAGTTCCGAT-3′) יש את אותו אזור בקרה (משלב 1) גם במרכזו.
- כדי לחשל את הפריימר לתבניות ה-DNA היוצרות G4 ולשלוט בהן, מערבבים 20 מיקרומטר של הפריימר (5′-Biotin-AF647-TCTTAATGTAAAATTTAAATGTC-3′) ל-20 מיקרומטר של תבניות 100 nt במאגר TE בתוספת 200 מ"מ KCl.
- מחממים את תמיסות תבנית ה-DNA ל-95 מעלות צלזיוס באמבט יבש דיגיטלי למשך 15 דקות. לאחר השלמתו, יש לקרר את התמיסות לאט למשך הלילה על ידי הנחת גוש החימום לתוך פוליסטירן כדי לאפשר קיפול של ה-G4 וחישול של שני הגדילים.
3. תיוג DNA פולימראז עם AF647
- תיוג הפולימראז עם הצבע
הערה: השמרים Pol δ באו לידי ביטוי וטוהרו כפי שתואר קודם לכן30. לאחר מכן היה צורך לתייג את הפולימראז בצבע מונו-ריאקטיבי של Alexa Fluor NHS ester (AF647) כדי לאפשר הדמיה ברמת המולקולה הבודדת. זה הושלם כפי שתואר קודם לכן5.- כדי לסמן את מסוף ה-N, דגרו עודף פי 3 של צבע AF647 עם 320 מיקרוליטר של 58.5 מיקרומטר פול δ במאגר המכיל 30 מ"מ של Tris-HCl (pH 7.6), 2 מ"מ של דיתיותרייטול (DTT), 300 מ"מ של נתרן כלורי (NaCl), 50 מ"מ של אשלגן גלוטמט ו-10% (v/v) גליצרול למשך 10 דקות ב-4 מעלות צלזיוס. סובב בעדינות את התמיסה כדי להבטיח ערבוב יסודי ולשפר את יעילות התיוג.
הערה: רצף החלבונים עשוי להכיל אמינים ראשוניים נוספים בחומצות אמינו ליזין. על ידי התאמת תנאי התגובה, כגון ה-pH, ניתן להצמיד את התווית באופן מועדף לקצה ה-N בניגוד לשאריות הליזין. זה עשוי לדרוש אופטימיזציה נוספת כדי להגביר את יעילות התיוג. - כדי להכין את עמודות התפלת הספין של 0.5 מ"ל (משקל מולקולרי של 7 K חתוך), צנטריפוגה תחילה את העמודות ב-1500 x גרם למשך דקה אחת כדי להסיר את תמיסת האחסון. השלך את התמיסות המופקדות בצינור האיסוף לאחר הצנטריפוגה.
הערה: הקפד לשחרר את מכסה עמודי הסיבוב במהלך שלבי הצנטריפוגה. זה יבטיח שהעמודות יהיו מסודרות כראוי. - אזנו את העמודות במאגר המכיל 30 מ"מ של Tris-HCl (pH 7.6), 300 מ"מ של NaCl, 3 מ"מ של DTT, 50 מ"מ של אשלגן גלוטמט ו-5% (v/v) גליצרול על ידי הוספת 300 מיקרוליטר של מאגר לראש העמודה. צנטריפוגה של העמודה ב-1500 x g למשך דקה אחת והשליכו את הזרימה. חזור על תהליך זה פעמיים נוספות.
- החל 200 מיקרוליטר מכל דגימת Pol δ על העמודה. צנטריפוגה של העמודים ב-1500 x גרם למשך 2 דקות ואוספים את δ הפול המטוהר בצינור מיקרו-צנטריפוגה של 1.5 מ"ל.
- הקפיאו את השברים המטוהרים בנוזל N2 ואחסנו את המינונים בטמפרטורה של -80 מעלות צלזיוס כדי למנוע פירוק חלבון לאורך זמן.
- כדי לסמן את מסוף ה-N, דגרו עודף פי 3 של צבע AF647 עם 320 מיקרוליטר של 58.5 מיקרומטר פול δ במאגר המכיל 30 מ"מ של Tris-HCl (pH 7.6), 2 מ"מ של דיתיותרייטול (DTT), 300 מ"מ של נתרן כלורי (NaCl), 50 מ"מ של אשלגן גלוטמט ו-10% (v/v) גליצרול למשך 10 דקות ב-4 מעלות צלזיוס. סובב בעדינות את התמיסה כדי להבטיח ערבוב יסודי ולשפר את יעילות התיוג.
- קביעת יעילות התיוג
- טען 2 מיקרוליטר של דגימת Pol δ המסומנת כעת AF647 על ספקטרופוטומטר UV-vis31.
הערה: ודא שריקון של מאגר Pol δ נמדד תחילה. - מדוד את אורכי גל העירור של 650 ננומטר עבור צבע AF647 ו-280 ננומטר עבור ה-Pol δ באמצעות הספקטרופוטומטר.
- באמצעות חוק באר-למברט ומקדמי ההכחדה המולרית של 270,000 ס"מ-1 M-1 עבור צבע AF647 ו-195,960 ס"מ-1 M-1 עבור הפולימראז, חישבו את יעילות התיוג.
הערה: יעילות התיוג שנקבעה עבור השמרים המסומנים AF647 Pol δ המשמשים בבדיקה זו חושבה כ-67%.
- טען 2 מיקרוליטר של דגימת Pol δ המסומנת כעת AF647 על ספקטרופוטומטר UV-vis31.
4. בדיקת הארכת פריימר אנסמבל
הערה: לפני ביצוע ניסויי שכפול של מולקולה אחת, יש צורך לאשר שה-DNA פולימראז נחסם על ידי ה-G-quadruplex באמצעות מבחני שכפול בתפזורת.
- הגדר את הטמפרטורה של בלוק חימום בתוך אמבטיה יבשה דיגיטלית ל-30 מעלות צלזיוס. זה יבטיח את הפעילות המקסימלית של השמרים פול δ.
- הכן "תערובות מאסטר" עבור תבניות יצירת ובקרה של G4 במאגר שכפול (25 מ"מ של Tris-HCl (pH 7.6), 10 מ"מ של מגנזיום אצטט, 50 מ"מ של אשלגן גלוטמט, 40 מיקרוגרם/מ"ל אלבומין בסרום בקר (BSA), 0.1 מ"מ של EDTA, 5 מ"מ של DTT ו-0.0025% (v/v) Tween-20). השלימו כל תערובת עם 1 מ"מ של DTT, 250 מיקרומטר כל אחד של dTTP, dCTP, dATP, dGTP (dNTPs), ו-10 ננומטר של תבנית ה-DNA המתאימה (יצירת G4 או בקרה). הניחו אותם בבלוק חימום כדי להבטיח שהם יגיעו ל-30 מעלות צלזיוס.
- הרוו 12 מיקרוליטר מכל תערובת מאסטר עם 12 מיקרוליטר של מאגר טעינת פורממיד (80% (w/v) פורממיד, 10 מ"מ של EDTA) כדי לשמש כבקרת T = 0 דקות.
- כדי להתחיל סינתזת DNA, הוסף שמרים Pol δ עם תווית AF647 לכל תערובת מאסטר (ריכוז סופי של 20 ננומטר). מערבבים היטב כדי להבטיח שה-DNA פולימראז משכפל את התבניות ביעילות מרבית.
הערה: יש לשמור את השמרים δ על קרח במהלך השימוש כדי לשמר את פעילות האנזימים. - לאחר 30 שניות, 60 שניות ו-180 שניות, הסר 12 מיקרוליטר מכל תערובת מאסטר והרוה עם 12 מיקרוליטר של מאגר טעינת פורממיד. היחס של 1:1 בין תערובת המאסטר למאגר טעינת הפורממיד מבטיח שתגובת השכפול לא יכולה להימשך לאחר הכיבוי.
- הסר את התמיסות המרוות כעת מבלוק החימום של 30 מעלות צלזיוס והנח אותן בבלוק חימום של 98 מעלות צלזיוס במקום זאת לדנטורציה של ה-dsDNA ל-ssDNA. לאחר שחלפו 10 דקות, הביאו את כל גוש החימום למיכל הג'ל להעמסת התמיסות לג'ל 15% tris-borate-EDTA (TBE)-urea polyacrylamide (PAGE)32.
הערה: יש לחמם את ג'ל ה-PAGE לפני טעינת הדגימות על ידי הפעלת אלקטרופורזה של הג'ל ב-180 וולט למשך 30 דקות במאגר TBE 1x. לאחר השלמתו, שטפו כל באר כדי להסיר אוריאה. - טען 15 מיקרוליטר מכל דגימה על הג'ל לצד סולם המכיל 0.02 מיקרומטר של אוליגונוקלאוטידים מסומנים 20 nt, 60 nt ו-100 nt במאגר טעינת פורממיד.
- הפעל את הג'ל למשך 60 דקות ב-180 וולט במאגר TBE 1x כדי להבטיח הפרדה מלאה.
- דמיין את הג'ל בערוץ Cy5 של הדמיה ביו-מולקולרית. זה יאפשר למדוד את גדיל ה-DNA המשוכפל המכיל את תווית ה-AF-647.
5. מיקרוסקופיה פלואורסצנטית של מולקולה אחת
- פונקציונליזציה של Coverslip
הערה: כדי לחבר את תבניות ה-DNA לכיסוי הזכוכית, תחילה יש להפעיל אותו עם אמינוסילן, ולאחר מכן קשירה של מולקולות PEG ביוטיניליות. תהליך זה ממזער את האינטראקציות הלא ספציפיות בין ה-DNA ו/או החלבונים לבין פני השטח.- נקה את הכיסויים (24 x 24 מ"מ) על ידי הכנסתם לצנצנות מכתימות ושפיכת אתנול. סוניקציה של הצנצנות למשך 30 דקות לפני שטיפת הכיסויים במים טהורים. חזור על תהליך זה עם 1 M אשלגן הידרוקסיד (KOH) לפני השטיפה שוב. חזור על שלבי ניקוי אלה פעם נוספת.
- שוטפים וממלאים צנצנת חדשה באצטון ומניחים את הכיסויים בפנים. פזרו 3-aminopropyltriethoxysilane לתוך הצנצנת ליצירת תמיסה של 2% v/v. מערבבים את הצנצנת למשך 3 דקות לפני שמרווה את התגובה עם עודף גדול של מים.
- יבש את הכיסויים עם N2 והנח אותם בנפרד על קופסה לחה מלאה במים. זה ימנע מהכיסויים להתייבש.
- הכן אסטר ביוטין-PEG-SVA:mPEG-SVA 1:25 ב-100 מ"מ של NaHCO3 (pH 8.2). מערבל את התערובת הזו במשך 20 שניות כדי להבטיח ערבוב יסודי.
- פיפטה 40 מיקרוליטר של תמיסת ה-PEG על כיסוי יבש. הנח כיסוי נוסף מעל כדי "לסנדוויץ'" את התמיסה בין שני הכיסויים. זה יפעיל את פני הכיסוי מבפנים. חזור על הפעולה עבור כל החלקות הכיסוי.
- דגרו את הכיסויים בחושך למשך 3 שעות. לאחר הדגירה יש להפריד את זוגות הכיסוי, לשטוף אותם בעודפי מים ולייבש אותם בגז N2 דחוס.
- חזור על שלבים 4.1.4-4.1.5 כדי למרוח שכבה שנייה של PEG על החלקות הכיסוי. הקפד לסנדוויץ' את הצדדים שפעלו בעבר יחד, אז היזהר לא להפוך אותם בטעות במהלך שלבי הכביסה. דגרו על התמיסות בחושך למשך הלילה לפני שטיפה וייבוש.
- אחסן את הכיסויים בוואקום כדי לשמור על הפונקציונליות שלהם. כיסויים פונקציונליים יציבים למשך חודש כאשר הם מאוחסנים כהלכה.
- הכנת תאי זרימה מיקרופלואידיים
הערה: תא זרימה מיקרופלואידי33 בנוי לניסויים של מולקולה בודדת (ראה איור משלים 2). זה יאפשר למאגרים, תבניות DNA וחלבונים לבוא במגע עם כיסוי פונקציונלי (ראה שלב 5.1).- הסר כיסוי פונקציונלי מוואקום והנח אותו על מתלה מיקרו-צינור מלא חלקית במים (קופסה לחה). מערבבים 100 מיקרוליטר של מאגר חוסם (50 מ"מ של Tris-HCl (pH 7.6), 50 מ"מ של KCl, 2% (v/v) Tween-20) עם 25 מיקרוליטר של תמיסת NeutrAvidin של 1 מ"ג/מ"ל (10% PBS) ומורחים זאת על פני הכיסוי. תן לזה לדגור במשך 15 דקות בטמפרטורת החדר בקופסה הלחה.
- שטפו את הכיסוי במים וייבשו ב-N2. זכור שרק צד אחד של הכיסוי מתפקד, אז זכור באיזה צד מדובר.
- הנח בלוק פולידימתילסילוקסן (PDMS) בהתאמה אישית34 על הכיסוי בתוך מחזיק תא הזרימה. זה ייצור תעלת זרימה בגובה 100 מיקרומטר ורוחב 1 מ"מ. לאחר מכן ניתן להכניס צינורות פוליאתילן (PE-60: קוטר כניסה של 0.76 מ"מ וקוטר חיצוני של 1.22 מ"מ) לתוך החורים בתא הזרימה כדי לספק גישה למאגרים ומצעים.
- הדמיה פלואורסצנטית של מולקולה אחת
הערה: ניסויים אלה אמורים להתבצע הן עם תבניות יצירת G4 והן עם תבניות הבקרה. זה יאפשר לדמיין את שני התנאים (חסימת δ הפול על ידי מבנה G4 וללא חסימה) ברמת המולקולה הבודדת באמצעות מיקרוסקופ השתקפות פנימית כוללת (TIRF).- יש לחמם 1 מ"ל של מאגר חוסם טווין (50 מ"מ של Tris-HCl (pH 7.6), 50 מ"מ של KCl, 2% (v/v) Tween-20) ו-500 מיקרוליטר של מאגר כביסה (25 מ"מ של Tris-HCl (pH 7.6), 10 מ"מ של מגנזיום אצטט, 250 מ"מ של אשלגן גלוטמט, 40 מיקרוגרם/מ"ל BSA, 0.1 מ"מ של EDTA, 5 מ"מ של DTT, 0.0025% (אנכי/אנכי) Tween-20) ומאגר שכפול ל-40 מעלות צלזיוס למשך 15 דקות. זה ישחרר את הגזים מהתמיסות. הסר גז מהתמיסות בתא ואקום למשך 15 דקות נוספות ב-800 mbar מתחת ללחץ האטמוספרי.
- קח את תא הזרימה הבנוי (ראה שלב 4.2) והנח אותו על הבמה של המיקרוסקופ. לאחר הנחת טיפת שמן על המטרה, הרם את המטרה כדי לעמוד בכיסוי.
הערה: ודא שלמטרה ולתא הזרימה יש זמן להגיע ל-31 מעלות צלזיוס. זה יגביר את הפעילות של השמרים Pol δ ויעזור להבטיח נתונים הניתנים לשחזור. - הכנס את צינור הכניסה למאגר חסימת Tween נטול הגז וחבר את השקע למשאבת המזרק. משוך לאחור את המזרק כדי למשוך את המאגר החוסם Tween דרך הצינור לתוך התעלה. אפשר למאגר זה לדגור למשך 30 דקות לפחות כדי למזער אינטראקציות לא ספציפיות.
- זרימה במאגר כביסה נטול גז של 200 מיקרוליטר לתוך התעלה בקצב של 100 מיקרוליטר לדקה. זה ישטוף את מאגר החסימה של Tween.
- לדלל את פתרונות תבנית ה-DNA ל-0.5 pM במאגר שכפול של μL 500. הזרימו 150 מיקרוליטר לתעלה בקצב של 10 מיקרוליטר לדקה. האירו את הדגימה באמצעות לייזר 647 ננומטר בכ-900 mWcm-2 במישור הדגימה כדי לדמיין תבניות DNA בודדות.
הערה: אם שדה הראייה (FOV) עם הדגימה אינו ממוקד לחלוטין, ייתכן שהחלקת הכיסוי אינה יושבת צמודה לתחתית תא הזרימה הבנוי. בנה תא זרימה חדש באמצעות כיסוי טרי אם זה קורה. - ברגע שצפיפות מספקת של כתמים (כנקודה אחת לכל 10 מיקרומטר2) נראית לעין, זרמו בתמיסה טרייה של מאגר שכפול (בתוספת 1 מ"מ של DTT) לתוך התעלה כדי לשטוף עודפי DNA. נפח של 250 מיקרוליטר מספיק בכדי להסיר לחלוטין את כל ה- DNA הלא מאוגד.
- עבור ל-FOV חדש וצלם תמונה של ה-DNA כדי לקבוע את מידת הקו-לוקליזציה בין הפולימראז המסומן למצע ה-DNA. לאחר השלמתו, הגדל את עוצמת הלייזר כדי להלבין את הכתמים הנותרים.
- הכן תמיסת פולימראז המכילה 1 מ"מ של DTT, 250 מיקרומטר של dNTPs ו-20 ננומטר של δ Pol עם תווית AF647 ב-200 מיקרוליטר של מאגר שכפול. זרימה ב-100 מיקרוליטר של תמיסת הפולימראז בקצב של 5 מיקרוליטר לדקה לתוך התעלה.
- לאחר שהדגימה נמצאת במיקוד וזווית ה-TIRF הותאמה, הגדר את עוצמת הלייזר של לייזר 647 ננומטר לכ-900 mWcm-2 במישור הדגימה. לאחר מכן, התחל לדמות את ה-FOV למשך הזמן הרצוי. רכוש בין 1-5 פריימים לשנייה למשך 10-20 דקות כדי ללכוד את כל אירועי השכפול.
- ניתוח נתונים
הערה: כל הניתוחים בוצעו באמצעות Python (גרסה 3.11.7). הקוד המותאם אישית המשמש לניתוח הנתונים זמין כאן: https://github.com/Single-molecule-Biophysics-UOW/G-quadruplex_binding_analysis.- ראשית, יש למקם את כתמי תבנית ה-DNA עם הכתמים הפלואורסצנטיים של השמרים המסומנים Pol δ. שתי פסגות נקבעו כמשותפות אם המרחק שלהן לאחר תיקון הסחיפה היה ≤ 3 פיקסלים. רק נקודות Pol δ המבצעות קולוקליזציה של תבניות DNA מנותחות עבור הקינטיקה המחייבת שלהן ומגמות ההתנהגות הכלליות.
- כדי לקבוע נתוני אירועי קשירה, מדוד את העוצמה לאורך זמן של כל נקודה מכיוון שהם מציגים התנהגות "הפעלה/כיבוי" לאורך הסרט. זה מאפשר לקבוע כמה פעמים קשירת Pol δ מתרחשת בתבנית DNA אחת.
הערה: ניתוח זה נעשה הן עבור ניסויי יצירת G4 והן עבור ניסויי תבנית הבקרה כדי להשוות את דינמיקת הכריכה.
הערה: בוצעו ניסויי בקרה, המבטיחים אירועי קשירה לא ספציפיים ל-G-quadruplex או לתבנית עצמה (23.2 שניות ±-4.9 שניות) אינם נכללים בניתוח אירועי הקשירה.
תוצאות
עבור בדיקה זו, תוכננו שני מצעי דנ"א עם פריימר 20-nt עם תווית פלואורסצנטית: אחד המכיל רצף יוצר G4 (איור 1A, משמאל) ואחד חסר רצף זה (איור 1A, מימין). כדי לאשר שה-G-quadruplex הוא מחסום יעיל לפעילות פולימראז, סינתזת ה-DNA על ידי Pol δ נוטרה על ג'ל PAGE לדנטורציה. הפעילות של δ ה-Pol המסומן המטוהר על מצעי ה-DNA נבדקה על ידי אלקטרופורזה של ג'ל. איור 1B (משמאל) מראה כי ה-Pol המסומן פלואורסצנטי δ אינו מסוגל לסנתז מעבר ל-G-quadruplex. לפני תחילת הסינתזה (t = 0 דקות), קיימת פס המתאים ל-20 nt, המייצג את הפריימר המסומן שעבר דנטורציה מגדיל התבנית. לאחר 3 דקות, פס 20 nt זה הוסב לפס 60-nt, מה שמצביע על סינתזה שהתרחשה על כל ה-DNA ומאשר שהפולימראז נחסם לחלוטין על ידי מבנה ה-G-quadruplex. חסימה זו מרמזת על כך שהפולימראז לא יכול היה להתפתח או לעקוף את המבנה. לעומת זאת, הסינתזה של תבנית בקרה שאינה מכילה את הרצף היוצר G-quadruplex (איור 1A, מימין) ייצרה פס של 100-nt לאחר 3 דקות (איור 1B, מימין).
לאחר אישור הסינתזה על התבניות וחסימה יעילה על ידי ה-G-quadruplex, מדידות אלו חזרו על עצמן במיקרוסקופ פלואורסצנטי של מולקולה אחת כדי לנטר את התנהגות הפולימראז. תבניות ה-DNA נקשרו לכיסויים פונקציונליים (איור 2A) בתא זרימה מיקרופלואידי, והמיקום של כל מצע נקבע על ידי הדמיית הפריימר המסומן פלואורסצנטי. לאחר מכן, δ שכותרתו Pol נטען בנוכחות dNTPs כדי להתחיל סינתזה. בתוך FOV טיפוסי, נקודות בודדות עוקבות כדי לכמת באיזו תדירות Pol δ נקשר לדנ"א ומתנתק ממנו (איור 2B,C). על ידי מדידת העוצמה כפונקציה של זמן בכל מצע דנ"א, ניתן ליצור מסלולים של מולקולה בודדת (איור 2C, איור משלים 3). ניתן למדוד את "זמן השהייה" האופייני, או משך הזמן הממוצע שבו Pol δ נשאר קשור לתבנית. עבור מצע G4, זמן השהות נקבע כ-6 שניות ±-2 שניות, בעוד שעבור מצע הביקורת, הוא היה 10 שניות ±-3 שניות (איור 2D, איור משלים 4). בנוסף, עבור כל מסלול, ניתן לכמת את מספר הפעמים ש-Pol δ נקשר לתבנית. מספר אירועי הקישור למצע G4 גבוה בהרבה בהשוואה לתבנית הבקרה (איור 2E). בעוד שישנם מקרים של יותר מאירוע כריכה אחד בתבנית הבקרה עקב קשירה וביטול כריכה לימודית, יש עלייה ברורה במספר אירועי הקישור בממוצע עבור התבנית היוצרת G4. זה מצביע על כך שלאחר שהסינתזה נעצרת על ידי ה-G-quadruplex, הפולימראז הקשור מתנתק מה-DNA לפני שפולימראזים חדשים מהתמיסה עוסקים במחזור רציף של קשירה וניתוק. לפיכך, מבחן מולקולה בודדת זה מספק תובנה שאין שני לה לגבי האופן שבו פולימראזות DNA מגיבות למחסומי DNA.

איור 1: בדיקת הארכת פריימר אנסמבל. (A) ייצוג סכמטי של מצעי הדנ"א. מצע G4 (משמאל) מכיל רצף יוצר G-quadruplex, בעוד שמצע הבקרה (מימין) לא. (B) בדיקת הרחבת DNA ראשונית של ה-G4 הדרוך ומצעי ה-DNA הבקרה המראים כי שמרים בעלי תווית פלואורסצנטית Pol δ חסומים לחלוטין על ידי ה-G-quadruplex. ג'ל ה-PAGE מראה את השכפול של תבניות ה-DNA לאורך זמן, וכתוצאה מכך הסטה של הפריימר המסומן 20 nt למוצר 60-nt עבור המצע G4 (משמאל), והמוצר 100-nt עבור תבנית הבקרה (מימין). M מייצג סולם המכיל 20 nt, 60 nt ו-100 nt אוליגוס. אנא לחץ כאן לצפייה בגרסה גדולה יותר של איור זה.
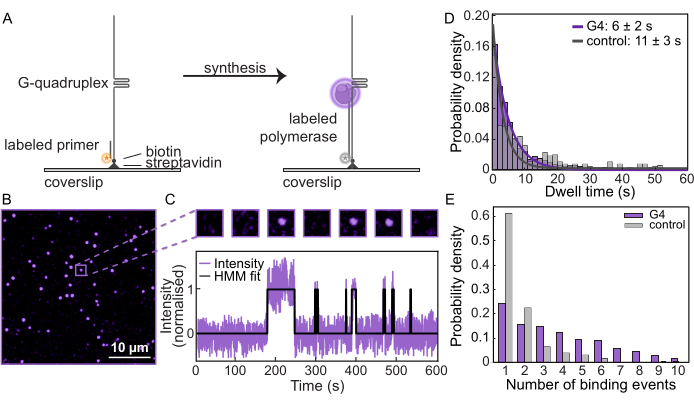
איור 2: מיקרוסקופיה פלואורסצנטית של מולקולה אחת. (A) ייצוג סכמטי של בדיקת הרחבת פריימר של מולקולה אחת באמצעות שמרים מסומנים AF647 Pol δ. (B) FOV טיפוסי המתקבל מבדיקת מולקולה בודדת. כל נקודה מייצגת פול עם תווית פלואורסצנטית δ הנקשרת לתבנית DNA. סרגל קנה מידה: 10 מיקרומטר. (C) (למעלה) מולקולה לדוגמה מבדיקת המולקולה הבודדת המציגה קשירת הפעלה/כיבוי כאשר δ Pol מחליף. (למטה) מסלול מולקולה בודדת של אותה מולקולה המראה את העוצמה לאורך זמן הניסוי. הקו השחור מייצג את התאמת מודל מרקוב הנסתר (HMM) לנתונים. (D) זמן השהייה של Pol δ על מצע G4 (סגול) ועל מצע הבקרה (אפור). הקווים מייצגים התאמות אקספוננציאליות, ומעניקים אורך חיים של 6 שניות ±-2 שניות על מצע G4 ו-10 שניות ±-3 שניות על מצע הבקרה. (E) מספר אירועי הקישור של Pol δ למצעי DNA בודדים. המספר החציוני של אירועי הקישור על מצע G4 (סגול) הוא 3.5, בהשוואה למצע הבקרה 1 (אפור). אנא לחץ כאן לצפייה בגרסה גדולה יותר של איור זה.
איור משלים 1: ספקטרוסקופיה CD. ספקטרום CD של רצף יוצר G4 (סגול) ובקרה (אפור) במאגר TE המכיל 200 מ"מ של KCl ב-25 מעלות צלזיוס. השיא האופייני ב-260 ננומטר, ואחריו השיא השלילי ב-240 ננומטר, אופייני ל-GQ תוך-מולקולרי מקביל. אנא לחץ כאן להורדת קובץ זה.
איור משלים 2: בניית תאי זרימה. (A) סכמטי של החלקים הבודדים של תא הזרימה. בלוק ה-PDMS המרובע יושב על גבי כיסוי המיקרוסקופ. לאחר מכן, חלקים אלה מותאמים יחד בתוך המכסה והתחתון של מחזיק תא הזרימה. (ב) תא הזרימה השלם. ניתן להכניס צינורות לכל צד של בלוק ה-PDMS כדי ליצור את התעלות שדרכן ניתן להזרים חוצצים דרך ההתקן באמצעות מיקרופלואידיקה. אנא לחץ כאן להורדת קובץ זה.
איור משלים 3: מסלולים של מולקולה בודדת. (A) מסלול לדוגמה של מולקולה שעוברת אירוע קישור יחיד במהלך מסגרת הזמן של 10 הדקות של הניסוי. (B) מסלול לדוגמה של מולקולה שעוברת אפס אירועי קשירה במהלך מסגרת הזמן של 10 הדקות של הניסוי. הקו השחור ב-(A) ו-(B) מייצג את התאמת מודל מרקוב הנסתר (HMM) לנתונים. אנא לחץ כאן להורדת קובץ זה.
איור משלים 4: זמני שהייה. (A) זמן השהייה של Pol δ על מצע G4. הקו מייצג את ההתאמה האקספוננציאלית, ומעניק אורך חיים של 6 שניות ± 2 שניות. (B) זמן השהייה של Pol δ על מצע הבקרה. ההתאמה האקספוננציאלית מעניקה אורך חיים של 10 שניות ± 3 שניות. אנא לחץ כאן להורדת קובץ זה.
איור משלים 5: בקרת פוטו-הלבנה. התרשים מציג את הזמן הממוצע עבור אנזים שמרים בודד המסומן ב-AF647 Pol δ לעבור פוטו-הלבנה על ידי לייזר 647 ננומטר. הקו מייצג את ההתאמה האקספוננציאלית, ומעניק אורך חיים של 39 ± 6 שניות.
Discussion
כאן, תוארה בדיקה מבוססת פלואורסצנציה של מולקולה אחת המספקת תובנה לגבי התנהגותו של DNA פולימראז כאשר הוא נתקל ב-G-quadruplex. בעוד שהפרוטוקולים של יצירת תבניות DNA, תיוג δ Pol ובדיקות שכפול DNA בתפזורת הם כולם פשוטים, ביצוע בדיקות מיקרוסקופיה של מולקולה אחת הוא מאתגר יותר מבחינה טכנית. בשל אופיין של טכניקות מולקולה בודדת, יש לנקוט בזהירות רבה כדי להימנע מהכנסת אבק, זיהום או בועות אוויר, שכן אלה יטשטשו את ה-FOV ויעכבו את איסוף הנתונים.
מגבלה של ניסויי מיקרוסקופיה TIRF של מולקולה אחת היא ההלבנה הפוטו-הלבנה של הפלואורופורים המחוברים קוולנטית לביו-מולקולות המעניינות. פוטוהלבנה היא תהליך בלתי הפיך המוביל לאובדן פלואורסצנטי קבוע35. כדי להפחית זאת במהלך ניסויים, חיוני להגביל את משך החשיפה ללייזר, להתאים את עוצמת הלייזר ולייעל את תזמון ההדמיה. אסטרטגיות אלו עוזרות לשמר אותות פלואורסצנטיים, ומבטיחות תקופות תצפית אמינות וממושכות יותר. על ידי כוונון עדין של פרמטרים אלה, אות ה-Pol δ נשאר למשך המדידה. כדי לייעל את עוצמת הלייזר עבור מבחני הסינתזה של מיקרוסקופיה פלואורסצנטית של מולקולה אחת, רצוי למדוד את קצב ההלבנה על ידי הדמיית הפולימראז המסומן על כיסוי זכוכית נקייה. על ידי שינוי שיטתי של עוצמת הלייזר והערכת קצב הפוטו-הלבנה על פני שדה הראייה, ניתן לזהות את עוצמת הלייזר האופטימלית המאזנת את עוצמת אות הפלואורופור עם ההתנגדות לפוטו-הלבנה (ראה איור משלים 5).
היתרון העיקרי של גישה זו של מולקולה בודדת על פני שיטות מסורתיות מבוססות אנסמבל הוא היכולת שלה לדמיין ישירות כאשר DNA פולימראז בודד נתקל במבנה G4 ומקיים אינטראקציה איתו. שיטות מסורתיות מבוססות אנסמבל (כגון אלקטרופורזה של ג'ל) הדגימו את היכולת של מבני G4 לחסום DNA פולימראזות 23,36,37. עם זאת, טכניקות אלו אינן מספקות את המידע הקינטי והמכניסטי בזמן אמת של אינטראקציה זו, הדרוש כדי לפרק שלבים ותוצאות קינטיות מולקולריות שונות. טכניקות של מולקולה בודדת מציעות תובנה שאין שני לה לגבי הקינטיקה, המנגנונים וההתנהגויות של ביומולקולות שלעתים קרובות מוסתרות על ידי אנסמבל ממוצעשל 38. כעת ניתן לראות כיצד פולימראזות DNA פועלות בזמן אמת - בין אם הן מחליפות, מתעכבות, מתנתקות או עוקפות מחסומי DNA39. עם קביעת פרוטוקול זה, ניתן לשנות בקלות את זהות ה-G4 ממבנה ה-c-MYC המקביל שנבחר לכל טופולוגיה מקבילה, אנטי-מקבילית או היברידית. יישום מבחן מולקולה בודדת זה יגלה אם אותם פולימראזות DNA מתנהגים אחרת כאשר הם נתקלים בטופולוגיות G4 חלופיות. ככאלה, שיטות של מולקולה בודדת חיוניות למענה על שאלות ספציפיות הנוגעות לאופן האינטראקציה בין חלבוני הגוף ל-DNA.
באמצעות הדמיה ישירה של אינטראקציות DNA פולימראז עם G-quadruplexes, זוהה מסלול חילופי שמרים שלא אופיין בעבר עבור שמרים Pol δ. תגלית זו מצביעה על כך שהפולימראז מתנתק כאשר הוא נתקל ב-G-quadruplex, וממתין להתערבות של חלבון אחר כדי לפתור את המבנה לפני התחלת סינתזת ה-DNA. ניתן להתאים פרוטוקול זה כדי לחקור אינטראקציות בין חלבונים שונים לתחזוקת הגנום ומכשולי DNA, ולהציע תובנות שאין שני להן לגבי האופן שבו אנזימים תאיים מנווטים במכשולים גנומיים. לדוגמה, ניתן לשנות את המחסום של בדיקה זו ממבנה G4 לקישור צולב חלבון-DNA, סוג של נגע DNA שבו חלבון נקשר באופן קוולנטי באופן בלתי הפיך ל-DNA, ומשמש כמכשול לשכפול DNA40. בדיקות כאלה חיוניות להבנת התהליכים הבסיסיים של שכפול, תיקון ורקומבינציה של DNA. על ידי מתן אפשרות לחקר דינמיקת DNA-חלבון ברמה המולקולרית, בדיקה זו מספקת כלי רב עוצמה להבהרת המנגנונים העומדים בבסיס השלמות הגנומית.
Disclosures
המחברים מצהירים שאין להם אינטרסים פיננסיים מתחרים.
Acknowledgements
נ.ק.-א. מכיר במימון שניתן על ידי מלגת תוכנית ההכשרה למחקר של ממשלת אוסטרליה. L.M.S. אסירת תודה על המימון שקיבלה מהמועצה הלאומית לבריאות ומחקר רפואי (מענק חוקר 2007778). J.S.L אסירת תודה על זכייתה בפרס Discovery Early Career Award (DE240100780) וב-NHMRC Investigator EL1 (2025412) במימון ממשלת אוסטרליה. S.H.M אסירת תודה על זכייתה במלגת הקריירה של ברוס וורן Molecular Horizons Ealy.
Materials
| Name | Company | Catalog Number | Comments |
| 100 nt control DNA template | Integrated DNA Technologies | Primary control template in primer extension assays | |
| 100 nt G4-forming DNA template | Integrated DNA Technologies | Primary G4-forming template in primer extension assays | |
| 15% Mini-PROTEAN TBE-Urea Gel, 12 well, 20 µL | Bio-Rad | 4566055 | Gel electrophoresis |
| 20 nt labelled/biotinylated primer oligonucleotide | Integrated DNA Technologies | Required for primer extension of the 100 nt templates | |
| 22 nt control DNA template | Integrated DNA Technologies | Primary control template in circular dichroism spectroscopy experiments | |
| 22 nt G4-forming DNA template | Integrated DNA Technologies | Primary G4-forming template in circular dichroism spectroscopy experiments | |
| 24 x 24 mm coverslip | Marienfeld Superior | 100062 | Required for TIRF microscopy |
| 3-aminopropyltriethoxysilane | Thermo Fisher Scientific | A10668.22 | Coverslip functionalization |
| 647 nm laser | Coherent | 1196627 | Select wavelength to correspond to stain/dye of choice |
| 670 nm emission filter | Chroma Technology Corp | ET655lp | Ensures only the relevant wavelengths are let through to the detector |
| Amersham Typhoon 5 biomolecular imager | Cytiva | 29187191 | Gel analysis |
| Biotin-PEG-SVA (MW 5000) | Laysan Bio | BIO-PEG-SVA-5K-100MG | Coverslip functionalization |
| Deoxynucleotides triphosphate solution mix | Jena Bioscience | NU-1005L | Replication building blocks |
| Digital dry bath (115V) | Bio-Rad | 1660571 | Heating solutions |
| Dithiothreitol | Sigma-Aldrich | 646563 | Disulfide bond reduction |
| EM-CCD camera | Andor | iXon 897 | Capturing single-molecule movies |
| Formamide | Sigma-Aldrich | F9037 | DNA denaturing during ensemble DNA replication assays |
| Gas tight syringe, 1 mL | SGE | 21964 | Filling cuvette for circular dichroism spectroscopy |
| Inverted microscope with CFI Apo TIRF 100x oil-immersion objective | Nikon | Eclipse Ti-E | Facilitates single-molecule TIRF microscopy |
| J-810 Spectrophotometer | Jasco | J-810 | Measuring circular dichroism spectroscopy |
| mPEG-SVA (MW 5000) | Laysan Bio | MPEG-SVA-5K-100MG | Coverslip functionalization |
| NanoDrop One Spectrophotometer | Thermofisher Scientific | ND-ONE | Measuring protein and label concentrations |
| NeutrAvidin | Thermo Fisher Scientific | 31000 | Tethering DNA templates to the functionalized coverslips during single-molecule experiments |
| Polyethylene tubing | Instech | BTPE-60 | Needed for flow cell construction |
| Quartz cuvette, 10 mm | Starna Scientific | 1/Q/10/CD | Holds sample for circular dichroism spectroscopy |
| Syringe pump | Adelab Scientific | NE-1002X | Used to flow buffers and substrates through a microfluidic flow cell |
| Tris-EDTA buffer | Thermofisher Scientific | 93283 | DNA solution buffer |
| Yeast polymerase δ | Michael O'Donnell Lab | Replicating the DNA templates | |
| Zeba spin desalting column, 7 K MWCO, 0.5 mL | Thermo Fisher Scientific | 89882 | Polymerase purification |
References
- Sutton, M. D. Walker, G. C. Managing DNA polymerases: Coordinating DNA replication, DNA repair, and DNA recombination. Proc Natl Acad Sci U. S. A. 98 (15), 8342-8349 (2001).
- Garg, P. Burgers, P. M. J. DNA polymerases that propagate the eukaryotic DNA replication fork. Crit Rev Biochem Mol Biol. 40 (2), 115-128 (2005).
- Burgers, P. M. J. Eukaryotic DNA polymerases in DNA replication and DNA repair. Chromosoma. 107 (4), 218-227 (1998).
- Tsai, M.-D. How DNA polymerases catalyze DNA replication, repair, and mutation. Biochem. 53 (17), 2749-2751 (2014).
- Lewis, J. S. et al. Tunability of DNA polymerase stability during eukaryotic DNA replication. Mol Cell. 77 (1), 17-25 (2020).
- Chilkova, O. et al. The eukaryotic leading and lagging strand DNA polymerases are loaded onto primer-ends via separate mechanisms but have comparable processivity in the presence of PCNA. Nucleic Acids Res. 35 (19), 6588-6597 (2007).
- Kawasaki, Y. Sugino, A. Yeast replicative DNA polymerases and their role at the replication fork. Mol Cells. 12 (3), 277-285 (2001).
- Stillman, B. DNA polymerases at the replication fork in eukaryotes. Mol Cell. 30 (3), 259-260 (2008).
- Wood, R. D. Shivji, M. K. Which DNA polymerases are used for DNA repair in eukaryotes? Carcinogenesis. 18 (4), 605-610 (1997).
- Budd, M. E. Campbell, J. L. The roles of the eukaryotic DNA polymerases in DNA repair synthesis. Mutat Res DNA Repair. 384 (3), 157-167 (1997).
- Yang, W. Gao, Y. Translesion and repair DNA polymerases: Diverse structure and mechanism. Annu Rev Biochem. 87, 239-261 (2018).
- Bridges, B. A. DNA repair: Polymerases for passing lesions. Curr Biol. 9 (13), 475-477 (1999).
- Wilkinson, E. M., Spenkelink, L. M., Van Oijen, A. M. Observing protein dynamics during DNA-lesion bypass by the replisome. Front Mol Biosci. 9, 968424 (2022).
- Mueller, S. H., Spenkelink, L. M., Van Oijen, A. M. When proteins play tag: The dynamic nature of the replisome. Biophys Rev. 11 (4), 641-651 (2019).
- Zybailov, B. L., Sherpa, M. D., Glazko, G. V., Raney, K. D., Glazko, V. I. G4-quadruplexes, and genome instability. Mol Biol. 47 (2), 197-204 (2013).
- Loeb, L. A. Monnat, R. J. DNA polymerases and human disease. Nat Rev Genet. 9 (8), 594-604 (2008).
- Oganesian, L. Bryan, T. M. Physiological relevance of telomeric G-quadruplex formation: A potential drug target. Bioessays. 29 (2), 155-165 (2007).
- Chambers, V. S. et al. High-throughput sequencing of DNA G-quadruplex structures in the human genome. Nat Biotechnol. 33 (8), 877-881 (2015).
- Cheng, Y., Zhang, Y., You, H. Characterization of G-quadruplexes folding/unfolding dynamics and interactions with proteins from single-molecule force spectroscopy. Biomol. 11 (11), 1579 (2021).
- Tippana, R., Xiao, W., Myong, S. G-quadruplex conformation and dynamics are determined by loop length and sequence. Nucleic Acids Res. 42 (12), 8106-8114 (2014).
- Kusi-Appauh, N., Ralph, S. F., Van Oijen, A. M., Spenkelink, L. M. Understanding G-quadruplex biology and stability using single-molecule techniques. J Phys Chem B. 127 (25), 5521-5540 (2023).
- Obi, I. et al. Stabilization of G-quadruplex DNA structures in Schizosaccharomyces pombe causes single-strand DNA lesions and impedes DNA replication. Nucleic Acids Res. 48 (19), 10998-11015 (2020).
- Kamath-Loeb, A. S., Loeb, L. A., Johansson, E., Burgers, P. M., Fry, M. Interactions between the Werner syndrome helicase and DNA polymerase delta specifically facilitate copying of tetraplex and hairpin structures of the d(CGG)n trinucleotide repeat sequence. J Biol Chem. 276 (19), 16439-16446 (2001).
- Kaguni, L. S. Clayton, D. A. Template-directed pausing in in vitro DNA synthesis by DNA polymerase a from Drosophila melanogaster embryos. Proc Natl Acad Sci. 79 (4), 983-987 (1982).
- Sato, K., Martin-Pintado, N., Post, H., Altelaar, M., Knipscheer, P. Multistep mechanism of G-quadruplex resolution during DNA replication. Sci Adv. 7 (39), eabf8653 (2021).
- Castillo Bosch, P. et al. FANCJ promotes DNA synthesis through G-quadruplex structures. EMBO J. 33 (21), 2521-2533 (2014).
- Paeschke, K., Capra, John A., Zakian, Virginia A. DNA replication through G-quadruplex motifs is promoted by the Saccharomyces cerevisiae Pif1 DNA helicase. Cell. 145 (5), 678-691 (2011).
- Guo, L. et al. Joint efforts of replicative helicase and SSB ensure inherent replicative tolerance of G-quadruplex. Adv Sci. (Weinh.). 11 (9), e2307696 (2024).
- Pham, S. Q. T. et al. A new class of quadruplex DNA-binding nickel Schiff base complexes. Dalton Trans. 49 (15), 4843-4860 (2020).
- Georgescu, R. E. et al. Mechanism of asymmetric polymerase assembly at the eukaryotic replication fork. Nat Struct Mol Biol. 21 (8), 664-670 (2014).
- Au - Desjardins, P., Au - Hansen, J. B., Au - Allen, M. Microvolume protein concentration determination using the NanoDrop 2000c spectrophotometer. J Vis Exp. 33, e1610 (2009).
- Summer, H., Grämer, R., Dröge, P. Denaturing urea polyacrylamide gel electrophoresis (Urea PAGE). J Vis Exp. 32, e1485 (2009).
- Geertsema, H. J., Duderstadt, K. E., Van Oijen, A. M. in DNA Replication: Methods and Protocols., eds Vengrova, S., Dalgaard, J.) 219-238, Springer New York, New York, NY (2015).
- Lewis, J. S. et al. Single-molecule visualization of fast polymerase turnover in the bacterial replisome. eLife. 6, e23932 (2017).
- Fish, K. N. Total internal reflection fluorescence (TIRF) microscopy. Curr Protoc Cytom. 12, 12-18 (2009).
- Sommers, J. A., Estep, K. N., Maul, R. W., Brosh, R. M. Biochemical analysis of DNA synthesis blockage by G-quadruplex structure and bypass facilitated by a G4-resolving helicase. Methods. 204, 207-214 (2022).
- Edwards, D. N., Machwe, A., Wang, Z., Orren, D. K. Intramolecular telomeric G-quadruplexes dramatically inhibit DNA synthesis by replicative and translesion polymerases, revealing their potential to lead to genetic change. PLoS One. 9 (1), 80664 (2014).
- Mueller, S. H. et al. Rapid single-molecule characterization of enzymes involved in nucleic-acid metabolism. Nucleic Acids Res. 51 (1), 5 (2022).
- Lewis, J. S., Van Oijen, A. M., Spenkelink, L. M. Embracing heterogeneity: Challenging the paradigm of replisomes as deterministic machines. Chem Rev. 123 (23), 13419-13440 (2023).
- Stingele, J., Bellelli, R., Boulton, S. J. Mechanisms of DNA-protein crosslink repair. Nat Rev Mol Cell Biol. 18 (9), 563-573 (2017).
Reprints and Permissions
Request permission to reuse the text or figures of this JoVE article
Request PermissionThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. All rights reserved