Method Article
Visualización de fluorescencia de una sola molécula de la dinámica de la ADN polimerasa en G-Quadruplex
En este artículo
Resumen
Este protocolo describe un ensayo de replicación de ADN de una sola molécula basado en microscopía de fluorescencia, que permite la visualización en tiempo real de las interacciones entre las ADN polimerasas y obstáculos como las estructuras G-cuádruples.
Resumen
La capacidad de las proteínas implicadas en la replicación del ADN eucariota para superar obstáculos, como los "obstáculos" de proteínas y ADN, es fundamental para garantizar una duplicación fiel del genoma. Los G-cuádruples son estructuras de ácidos nucleicos de orden superior que se forman en regiones del ADN ricas en guanina y se ha demostrado que actúan como obstáculos, interfiriendo con las vías de mantenimiento genómico. Este estudio presenta un método basado en microscopía de fluorescencia en tiempo real para observar las interacciones de la ADN polimerasa con estructuras G-cuádruples. Los oligonucleótidos de ADN cortos y cebados que contenían un G-cuádruple se inmovilizaron en cubreobjetos de vidrio funcionalizados dentro de una celda de flujo microfluídico. Se introdujeron ADN polimerasas marcadas con fluorescencia, lo que permitió monitorizar su comportamiento y estequiometría a lo largo del tiempo. Este enfoque permitió observar el comportamiento de la polimerasa cuando se detuvo por un G-cuádruple. Específicamente, utilizando δ de levadura polimerasa marcados con fluorescencia, se encontró que al encontrarse con un G-cuádruple, la polimerasa se somete a un ciclo continuo de unión y desunión. Este ensayo de una sola molécula se puede adaptar para estudiar las interacciones entre varias proteínas de mantenimiento del ADN y los obstáculos en el sustrato del ADN.
Introducción
Las ADN polimerasas son enzimas que catalizan la incorporación de nucleósidos trifosfatos para duplicar el ADN 1,2,3,4,5. Como tales, desempeñan un papel clave en los procesos esenciales de mantenimiento del ADN, incluida la replicación del ADN 6,7,8 y la reparación 9,10,11,12. Las ADN polimerasas deben replicar el genoma de manera precisa y eficiente para garantizar la integridad genómica, evitando la acumulación de mutaciones en el genoma. Durante la síntesis, las polimerasas a menudo encuentran "obstáculos" como proteínas unidas al ADN o estructuras secundarias de ADN13. Estos obstáculos pueden retrasar o incluso bloquear la progresión de la polimerasa14. Superar estos obstáculos es importante para garantizar una duplicación fiel del genoma, ya que de lo contrario se puede provocar inestabilidad genómica 15,16.
Una de las principales clases de obstáculos son los G-cuádruples (G4), estructuras secundarias de ADN no canónicas que se han demostrado que se forman en secuencias ricas en guanina dentrodel genoma humano. Hay más de 700.000 secuencias diferentes en el genoma humano capaces de formar un G4, incluyendo regiones dentro de los telómeros y promotores de oncogenes18. Estas estructuras de ADN adoptan diversas conformaciones en función de la secuencia de nucleótidos, la longitud y el catión metálico unido 19,20,21. Esta diversidad significa que las polimerasas deben superar una gama de topologías G4 diferentes, potencialmente con diversos grados de eficiencia. Se ha demostrado que el fracaso de las polimerasas para superar o eludir una estructura G4 impide la progresión de la horquilla de replicación in vivo, lo que conduce a la inestabilidad genómica22. Los estudios in vitro han demostrado que las estructuras G4 pueden detener o bloquear completamente las polimerasasde levadura 23,24,25,26,27. La capacidad de las estructuras G4 para detener o bloquear las ADN polimerasas depende completamente de su estabilidad cinética y termodinámica, y algunas polimerasas pueden desplegar ciertas28 G4. Si bien estos estudios proporcionan información sobre la capacidad de las polimerasas para superar los obstáculos del G-cuádruple, carecen de la capacidad de visualizar directamente el comportamiento de la polimerasa al encontrarse con un G4. El destino de las polimerasas, ya sea que permanezcan unidas, se caigan o se intercambien dinámicamente, determina qué procesos posteriores son accesibles para resolver el G4.
En este estudio, se desarrolló un ensayo de microscopía de molécula única basado en fluorescencia para visualizar y monitorear directamente la unión de la ADN polimerasa con las estructuras G4 en tiempo real. Este ensayo consiste en atar plantillas de ADN formadoras de G4 a un cubreobjetos biotinilado en una celda de flujo microfluídico, donde se pueden introducir ADN polimerasas marcadas con fluorescencia para iniciar la síntesis de ADN. Al medir la fluorescencia de las polimerasas a lo largo del tiempo, se puede observar directamente su comportamiento al encontrarse con una estructura G4. La estructura G4 encontrada en el oncogén del cáncer c-MYC fue elegida para este ensayo debido a su alto nivel de estabilidad. Este protocolo ahora se puede adaptar para catalogar el comportamiento de una variedad de polimerasas en todos los dominios de la vida asociados con diferentes topologías y estabilidades G4. Este ensayo ofrece un enfoque innovador y de alto rendimiento para dilucidar los mecanismos por los cuales las ADN polimerasas sortean los obstáculos del ADN, proporcionando una poderosa herramienta para avanzar en la comprensión de la dinámica de la polimerasa.
Protocolo
Los detalles de los reactivos y el equipo utilizado se enumeran en la Tabla de Materiales.
1. Espectroscopía de dicroísmo circular
NOTA: Antes de desarrollar el ensayo, fue necesario realizar una espectroscopia de dicroísmo circular (CD) en la secuencia G4 seleccionada para garantizar un plegamiento correcto. La secuencia 22 nt (5′-TGAGGGTGGGGGGGGGGAA-3′) forma la estructura G4 derivada del oncogén canceroso c-MYC . La espectroscopia CD también se realizó en una secuencia de control (5'- TGAGTGTGAAGACGATGTAGAA -3) que tiene nucleótidos clave alterados que impiden la formación de G4.
- Para preparar las plantillas de ADN para la espectroscopia de CD, agregue 40 μL de 100 μM de las plantillas de 22 nt a 360 μL de tampón de Tris-HCl (pH 8.0), ácido etilendiaminotetraacético (EDTA) (TE) suplementado con 200 mM de cloruro de potasio (KCl). Se desea una concentración final de ADN de 10 μM para los experimentos de espectroscopia de CD.
NOTA: La presencia de iones K+ es esencial para estabilizar la estructura G4 en la plantilla. - Calentar las soluciones de ADN a 95 °C en un baño seco digital durante 15 min. Una vez completado, enfríe las soluciones lentamente durante la noche colocando el bloque calefactor en poliestireno. Este lento proceso de enfriamiento es responsable del plegado del G4.
- Transfiera 400 μL de las soluciones de secuencia de control y formación de G4 a una cubeta de cuarzo de 0,1 cm de longitud utilizando una jeringa hermética al gas. Llene la cubeta lentamente para evitar la formación de burbujas de aire.
- Mida el espectro de CD entre 200 nm y 400 nm utilizando un espectropolarímetro a 25 °C. Los parámetros y condiciones para esta medición han sido descritos previamente29. Los resultados se pueden encontrar en la Figura Suplementaria 1.
NOTA: Asegúrese de registrar el espectro del CD solo para que el búfer actúe como un espacio en blanco.
2. Preparación de plantillas de ADN
NOTA: Las plantillas que se pueden replicar son plantillas lineales cortas, cebadas de 100 nt, preparadas utilizando técnicas estándar de biología molecular. La plantilla de formación G4 (5′-GAATTACATTTAAATTTTACACAGATACAGTCAATGAGAA
CTTCCAGGCGTAACGAGAGCACGGGGTGGGGGGGG
GGGACCTTAGCTTCGAGTTCCGAT-3′) contiene la secuencia capaz de formar un G-cuádruple derivado de MYC (desde el paso 1) en su centro. La plantilla de control (5′-GAATTACATTTAAATTTTACACAGATACAGTCAATGAGA
ACTTCCAGGCGTAACGAGAGCACGATGTAGCAGAACT
GTGACCTTAGCTTCGAGTTCCGAT-3′) tiene la misma región de control (del paso 1) también en su centro.
- Para recocer el cebador en las plantillas de ADN de control y formación de G4, mezcle 20 μM del cebador (5′-Biotina-AF647-TCTTAATGTAAATTTAAAATGTGTC-3′) con 20 μM de las plantillas de 100 nt en tampón TE complementado con 200 mM de KCl.
- Calentar las soluciones de molde de ADN a 95 °C en un baño seco digital durante 15 min. Una vez completado, enfríe las soluciones lentamente durante la noche colocando el bloque calefactor en poliestireno para permitir el plegado del G4 y el recocido de las dos hebras.
3. Marcaje de la ADN polimerasa con AF647
- Marcaje de la polimerasa con el colorante
NOTA: La levadura Pol δ se exprimió y purificó como se describió anteriormente30. A continuación, la polimerasa debía marcarse con el colorante monorreactivo Alexa Fluor NHS éster (AF647) para permitir la visualización a nivel de una sola molécula. Esto se completó como se describió anteriormente5.- Para marcar el extremo N-terminal, incube un exceso de 3 veces del colorante AF647 con 320 μL de 58,5 μM de Pol δ en un tampón que contenga 30 mM de Tris-HCl (pH 7,6), 2 mM de ditiotreitol (DTT), 300 mM de cloruro de sodio (NaCl), 50 mM de glutamato de potasio y glicerol al 10% (v/v) durante 10 min a 4 °C. Gire suavemente la solución para garantizar una mezcla completa y mejorar la eficiencia del etiquetado.
NOTA: La secuencia de proteínas puede contener aminas primarias adicionales en los aminoácidos de lisina. Al ajustar las condiciones de reacción, como el pH, la etiqueta se puede adherir preferentemente al N-terminal en lugar de a los residuos de lisina. Esto puede requerir una mayor optimización para aumentar la eficiencia del etiquetado. - Para preparar las columnas de desalinización por centrifugación de 0,5 mL (corte de peso molecular de 7 K), primero centrifugar las columnas a 1500 x g durante 1 min para eliminar la solución de almacenamiento. Deseche las soluciones depositadas en el tubo de recolección después de la centrifugación.
NOTA: Asegúrese de aflojar la tapa de las columnas de centrifugación durante los pasos de centrifugación. Esto asegurará que las columnas estén correctamente eluidas. - Equilibre las columnas en un tampón que contenga 30 mM de Tris-HCl (pH 7,6), 300 mM de NaCl, 3 mM de DTT, 50 mM de glutamato de potasio y glicerol al 5% (v/v) añadiendo 300 μL de tampón a la parte superior de la columna. Centrifugar la columna a 1500 x g durante 1 min y desechar el flujo. Repita este proceso dos veces más.
- Aplique 200 μL de cada muestra de Pol δ a la columna. Centrifugar las columnas a 1500 x g durante 2 min y recoger el Pol δ purificado en un tubo de microcentrífuga de 1,5 mL.
- Congele las fracciones purificadas en N2 líquido y almacene las alícuotas a -80 °C para evitar la degradación de las proteínas con el tiempo.
- Para marcar el extremo N-terminal, incube un exceso de 3 veces del colorante AF647 con 320 μL de 58,5 μM de Pol δ en un tampón que contenga 30 mM de Tris-HCl (pH 7,6), 2 mM de ditiotreitol (DTT), 300 mM de cloruro de sodio (NaCl), 50 mM de glutamato de potasio y glicerol al 10% (v/v) durante 10 min a 4 °C. Gire suavemente la solución para garantizar una mezcla completa y mejorar la eficiencia del etiquetado.
- Determinación de la eficiencia del etiquetado
- Cargue 2 μL de la muestra de Pol δ ahora etiquetada con AF647 en un espectrofotómetro UV-vis31.
NOTA: Asegúrese de que primero se mida un blanco del búfer Pol δ. - Mida las longitudes de onda de excitación de 650 nm para el colorante AF647 y de 280 nm para el δ Pol utilizando el espectrofotómetro.
- Utilizando la ley de Beer-Lambert y los coeficientes de extinción molar de 270.000 cm-1 M-1 para el colorante AF647 y 195.960 cm-1 M-1 para la polimerasa, calcule la eficiencia del etiquetado.
NOTA: La eficiencia de etiquetado determinada para la levadura Pol marcada con AF647 δ utilizada en este ensayo se calculó en aproximadamente el 67%.
- Cargue 2 μL de la muestra de Pol δ ahora etiquetada con AF647 en un espectrofotómetro UV-vis31.
4. Ensayo de extensión de cebador conjunto
NOTA: Antes de realizar experimentos de replicación de una sola molécula, es necesario confirmar que la ADN polimerasa está bloqueada por el G-cuádruple mediante ensayos de replicación masiva.
- Ajuste la temperatura de un bloque calefactor dentro de un baño seco digital a 30 °C. Esto asegurará la máxima actividad de la levadura Pol δ.
- Prepare "mezclas maestras" para las plantillas de control y formación de G4 en tampón de replicación (25 mM de Tris-HCl (pH 7,6), 10 mM de acetato de magnesio, 50 mM de glutamato de potasio, 40 μg/mL de albúmina sérica bovina (BSA), 0,1 mM de EDTA, 5 mM de DTT y 0,0025% (v/v) Tween-20). Complemente cada mezcla con 1 mM de DTT, 250 μM de dTTP, dCTP, dATP, dGTP (dNTP) y 10 nM de la plantilla de ADN respectiva (formadora de G4 o control). Colóquelos en un bloque calefactor para asegurarse de que alcancen los 30 °C.
- Enfríe 12 μL de cada mezcla maestra con 12 μL de tampón de carga de formamida (80% (p/v) de formamida, 10 mM de EDTA) para que actúe como control T = 0 min.
- Para iniciar la síntesis de ADN, agregue δ de levadura Pol marcada con AF647 a cada mezcla maestra (concentración final de 20 nM). Mezcle bien para asegurarse de que las ADN polimerasas repliquen las plantillas con la máxima eficiencia.
NOTA: Mantenga la levadura Pol δ en hielo durante su uso para preservar la actividad enzimática. - Después de 30 s, 60 s y 180 s, retire 12 μL de cada mezcla maestra y enfríe con 12 μL de tampón de carga de formamida. La relación 1:1 entre la mezcla maestra y el tampón de carga de formamida garantiza que la reacción de replicación no pueda continuar una vez enfriada.
- Retire las soluciones ahora apagadas del bloque calefactor a 30 °C y colóquelas en un bloque calefactor a 98 °C para desnaturalizar el dsDNA a ssDNA. Una vez transcurridos 10 minutos, lleve todo el bloque calefactor al tanque de gel para cargar las soluciones en un gel desnaturalizante de tris-borato-EDTA (TBE)-urea (PAGE)32 desnaturalizante.
NOTA: El gel PAGE debe calentarse antes de la carga de las muestras ejecutando la electroforesis en gel a 180 V durante 30 min en 1x tampón TBE. Una vez completado, enjuague cada pocillo para eliminar la urea. - Cargue 15 μL de cada muestra en el gel junto a una escalera que contenga 0,02 μM de oligonucleótidos marcados con 20 nt, 60 nt y 100 nt en tampón de carga de formamida.
- Haga funcionar el gel durante 60 minutos a 180 V en 1x tampón TBE para garantizar una separación completa.
- Imagen del gel en el canal Cy5 de un generador de imágenes biomoleculares. Esto permitirá medir la cadena de ADN replicada que contiene la etiqueta AF-647.
5. Microscopía de fluorescencia de molécula única
- Funcionalización del cubreobjetos
NOTA: Para unir las plantillas de ADN al cubreobjetos de vidrio, primero se debe funcionalizar con aminosilano, seguido de la unión de moléculas de PEG biotiniladas. Este proceso minimiza las interacciones inespecíficas entre el ADN y/o las proteínas y la superficie.- Limpie los cubreobjetos (24 x 24 mm) insertándolos en frascos de tinte y vertiéndolos en etanol. Sonicar los frascos durante 30 minutos antes de enjuagar los cubreobjetos con agua pura. Repita este proceso con hidróxido de potasio (KOH) 1 M antes de enjuagar nuevamente. Repita estos pasos de limpieza una vez más.
- Enjuague y llene un frasco nuevo con acetona y coloque los cubreobjetos adentro. Diseminar 3-aminopropiltrietoxisilano en el frasco para formar una solución v/v al 2%. Agite el frasco durante 3 minutos antes de apagar la reacción con un gran exceso de agua.
- Seque los cubreobjetos con N2 y colóquelos individualmente en una caja húmeda llena de agua. Esto evitará que los cubreobjetos se sequen.
- Prepare un éster de biotina-PEG-SVA a escala 1:25:mPEG-SVA en 100 mM de NaHCO3 (pH 8,2). Agite esta mezcla durante 20 s para asegurar una mezcla completa.
- Pipetear 40 μL de la solución de PEG en un cubreobjetos seco. Coloque otro cubreobjetos en la parte superior para "intercalar" la solución entre los dos cubreobjetos. Esto funcionalizará las caras del cubreobjetos en el interior. Repita el procedimiento para todos los cubreobjetos.
- Incubar los cubreobjetos en la oscuridad durante 3 h. Después de la incubación, separe los pares de cubreobjetos, enjuáguelos con el exceso de agua y séquelos con gas N2 comprimido.
- Repita los pasos 4.1.4-4.1.5 para aplicar una segunda capa de PEG a los cubreobjetos. Asegúrese de intercalar los lados previamente funcionalizados, así que tenga cuidado de no voltearlos accidentalmente durante los pasos de lavado. Incuba las soluciones en la oscuridad durante la noche antes de enjuagarlas y secarlas.
- Guarde los cubreobjetos en el vacío para preservar su funcionalidad. Los cubreobjetos funcionalizados son estables durante un mes cuando se almacenan correctamente.
- Preparación de celdas de flujo microfluídicas
NOTA: Se construye una celda de flujo microfluídica33 para experimentos de una sola molécula (ver Figura suplementaria 2). Esto permitirá que los tampones, las plantillas de ADN y las proteínas entren en contacto con un cubreobjetos funcionalizado (ver paso 5.1).- Retire un cubreobjetos funcionalizado de una aspiradora y colóquelo en una gradilla de microtubos parcialmente llena de agua (caja húmeda). Mezclar 100 μL de un tampón de bloqueo (50 mM de Tris-HCl (pH 7,6), 50 mM de KCl, 2% (v/v) Tween-20) con 25 μL de 1 mg/mL de solución de NeutrAvidin (10% PBS) y extenderlo sobre la superficie del cubreobjetos. Deje incubar durante 15 minutos a temperatura ambiente en la caja húmeda.
- Lave el cubreobjetos con agua y séquelo en N2. Recuerde que solo un lado del cubreobjetos está funcionalizado, así que recuerde de qué lado es.
- Coloque el bloque34 de polidimetilsiloxano (PDMS) hecho a medida en el cubreobjetos dentro del soporte de la celda de flujo. Esto generará un canal de flujo de 100 μm de alto y 1 mm de ancho. A continuación, se pueden insertar tubos de polietileno (PE-60: 0,76 mm de diámetro de entrada y 1,22 mm de diámetro exterior) en los orificios de la celda de flujo para proporcionar acceso a tampones y sustratos.
- Imágenes de fluorescencia de una sola molécula
NOTA: Estos experimentos deben realizarse con las plantillas de formación G4 y de control. Esto permitirá visualizar ambas condiciones (bloqueo de la δ Pol por una estructura G4 y ausencia de bloqueo) a nivel de una sola molécula utilizando microscopía de reflexión interna total (TIRF).- Calentar 1 mL de alícuotas de un tampón de bloqueo Tween (50 mM de Tris-HCl (pH 7,6), 50 mM de KCl, 2% (v/v) Tween-20) y 500 μL de alícuotas de un tampón de lavado (25 mM de Tris-HCl (pH 7,6), 10 mM de acetato de magnesio, 250 mM de glutamato de potasio, 40 μg/mL de BSA, 0,1 mM de EDTA, 5 mM de DTT, 0,0025% (v/v) Tween-20) y búfer de replicación a 40 °C durante 15 min. Esto liberará los gases de las soluciones. Desgasificar las soluciones en una cámara de vacío durante 15 minutos más a 800 mbar por debajo de la presión atmosférica.
- Tome la celda de flujo construida (consulte el paso 4.2) y colóquela en la platina del microscopio. Después de colocar una gota de aceite en el objetivo, levante el objetivo para encontrarse con el cubreobjetos.
NOTA: Asegúrese de que el objetivo y la celda de flujo tengan tiempo de alcanzar los 31 °C. Esto aumentará la actividad de la levadura Pol δ y ayudará a garantizar la reproducción de los datos. - Inserte el tubo de entrada en el tampón de bloqueo Tween desgasificado y conecte la salida a la bomba de jeringa. Tire hacia atrás de la jeringa para extraer el tampón de bloqueo Tween a través del tubo hacia el canal. Deje que este tampón se incube durante al menos 30 minutos para minimizar las interacciones inespecíficas.
- Fluya en 200 μL de tampón de lavado desgasificado en el canal a una velocidad de 100 μL/min. Esto vaciará el búfer de bloqueo de interpolación.
- Diluir las soluciones molde de ADN a 0,5 pM en tampón de replicación de 500 μL. Caudal 150 μL en el canal a una velocidad de 10 μL/min. Ilumine la muestra con un láser de 647 nm a aproximadamente 900 mWcm-2 en el plano de la muestra para visualizar plantillas de ADN individuales.
NOTA: Si el campo de visión (FOV) de la muestra no está completamente enfocado, es posible que el cubreobjetos no esté al ras de la parte inferior de la celda de flujo construida. Construya una nueva celda de flujo utilizando un cubreobjetos nuevo si esto ocurre. - Una vez que se vea una densidad suficiente de manchas (aproximadamente 1 mancha por 10 μm2), fluya una solución fresca de tampón de replicación (suplementada con 1 mM de DTT) en el canal para lavar el exceso de ADN. Un volumen de 250 μL es suficiente para eliminar completamente todo el ADN no unido.
- Pase a un nuevo campo de visión y capture una imagen del ADN para determinar el grado de colocalización entre la polimerasa marcada y el sustrato de ADN. Una vez completado, aumente la potencia del láser para fotoblanquear las manchas restantes.
- Prepare una solución de polimerasa que contenga 1 mM de DTT, 250 μM de dNTPs y 20 nM de Pol δ marcado con AF647 en 200 μL de tampón de replicación. Fluya en 100 μL de la solución de polimerasa a una velocidad de 5 μL/min en el canal.
- Una vez que la muestra esté enfocada y se haya ajustado el ángulo TIRF, ajuste la potencia del láser de un láser de 647 nm a aproximadamente 900 mWcm-2 en el plano de la muestra. A continuación, comience a obtener imágenes del campo de visión durante el tiempo deseado. Adquiera entre 1 y 5 fotogramas por segundo durante 10 a 20 minutos para capturar todos los eventos de replicación.
- Análisis de datos
NOTA: Todos los análisis se realizaron utilizando Python (v. 3.11.7). El código personalizado utilizado para el análisis de datos está disponible aquí: https://github.com/Single-molecule-Biophysics-UOW/G-quadruplex_binding_analysis.- En primer lugar, colocalice las manchas molde de ADN con las manchas fluorescentes de la levadura Pol δ marcada. Se determinó que dos picos estaban colocalizados si su distancia después de la corrección de la deriva era ≤ 3 píxeles. Solo las manchas Pol δ que colocalizan las plantillas de ADN se analizan para determinar su cinética de unión y sus tendencias generales de comportamiento.
- Para determinar los datos de eventos vinculantes, mida la intensidad a lo largo del tiempo de cada punto a medida que muestran un comportamiento de "encendido/apagado" a lo largo de la película. Esto permite determinar cuántas veces se produce la unión de Pol δ en una sola plantilla de ADN.
NOTA: Este análisis se realiza tanto para los experimentos de formación de G4 como para los de plantilla de control para comparar la dinámica de unión.
NOTA: Se han llevado a cabo experimentos de control, que garantizan que los eventos de unión no específicos al G-cuádruple o a la propia plantilla (23,2 s ± 4,9 s) no se incluyan en el análisis de los eventos de unión.
Resultados
Para este ensayo, se diseñaron dos sustratos de ADN con un cebador de 20 nt marcado con fluorescencia: uno que contenía una secuencia formadora de G4 (Figura 1A, izquierda) y otro que carecía de esta secuencia (Figura 1A, derecha). Para confirmar que el G-quadruplex es un obstáculo eficaz para la actividad de la polimerasa, la síntesis de ADN por Pol δ se monitoreó en un gel desnaturalizante PAGE. La actividad del Pol δ marcado purificado en los sustratos de ADN se examinó mediante electroforesis en gel. La Figura 1B (izquierda) muestra que el Pol δ marcado con fluorescencia es incapaz de sintetizar más allá del G-cuádruple. Antes del inicio de la síntesis (t = 0 min), está presente una banda correspondiente a 20 nt, que representa el cebador marcado que se ha desnaturalizado de la hebra moldura. Después de 3 minutos, esta banda de 20 nt se convirtió en una banda de 60 nt, lo que indica que se produjo la síntesis en todo el ADN y confirmó que la polimerasa estaba completamente bloqueada por la estructura G-quadruplex. Este bloqueo implica que la polimerasa no podría desplegarse ni eludir la estructura. Por el contrario, la síntesis de una plantilla de control que no contiene la secuencia de formación de G-quadruplex (Figura 1A, derecha) produjo una banda de 100 nt después de 3 minutos (Figura 1B, derecha).
Después de confirmar la síntesis en las plantillas y el bloqueo eficiente por el G-quadruplex, estas mediciones se repitieron en un microscopio de fluorescencia de una sola molécula para monitorear el comportamiento de la polimerasa. Las plantillas de ADN se unieron a cubreobjetos funcionalizados (Figura 2A) en una celda de flujo microfluídica, y la posición de cada sustrato se determinó visualizando el cebador marcado con fluorescencia. A continuación, se cargó el δ Pol etiquetado en presencia de dNTP para iniciar la síntesis. Dentro de un campo de visión típico, se rastrean puntos individuales para cuantificar la frecuencia con la que Pol δ se une y se disocia del ADN (Figura 2B, C). Al medir la intensidad en función del tiempo en cada sustrato de ADN, se pueden generar trayectorias de una sola molécula (Figura 2C, Figura 3 suplementaria). Se puede medir la característica "tiempo de permanencia", es decir, la duración media en la que Pol δ permanece ligada a la plantilla. Para el sustrato G4, el tiempo de permanencia se determinó en 6 s ± 2 s, mientras que para el sustrato control fue de 10 s ± 3 s (Figura 2D, Figura 4 suplementaria). Además, para cada trayectoria, se puede cuantificar el número de veces que Pol δ se une a la plantilla. El número de eventos de unión al sustrato G4 es mucho mayor en comparación con la plantilla de control (Figura 2E). Aunque hay instancias de más de un evento de enlace en la plantilla de control debido al enlace y desenlace escolástico, hay un claro aumento en el número de eventos de enlace en promedio para la plantilla de formación G4. Esto sugiere que, después de que la síntesis es detenida por el G-cuádruple, la polimerasa unida se disocia del ADN antes de que las nuevas polimerasas de la solución inicien un ciclo continuo de unión y desunión. Por lo tanto, este ensayo de molécula única proporciona una visión sin precedentes de cómo las ADN polimerasas responden a los obstáculos del ADN.

Figura 1: Ensayo de extensión de cebador conjunto. (A) Representación esquemática de los sustratos de ADN. El sustrato G4 (izquierda) contiene una secuencia de formación de G-cuádruple, mientras que el sustrato de control (derecha) no la contiene. (B) Ensayo conjunto de extensión de la primasa del ADN G4 cebado y sustratos de ADN de control que muestra que la levadura Pol δ marcada con fluorescencia está completamente bloqueada por el G-cuádruple. El gel PAGE muestra la replicación de las plantillas de ADN a lo largo del tiempo, lo que resulta en un cambio del cebador de 20 nt marcado al producto de 60 nt para el sustrato G4 (izquierda) y al producto de 100 nt para la plantilla de control (derecha). M representa una escalera que contiene oligonucleótidos marcados de 20 nt, 60 nt y 100 nt. Haga clic aquí para ver una versión más grande de esta figura.
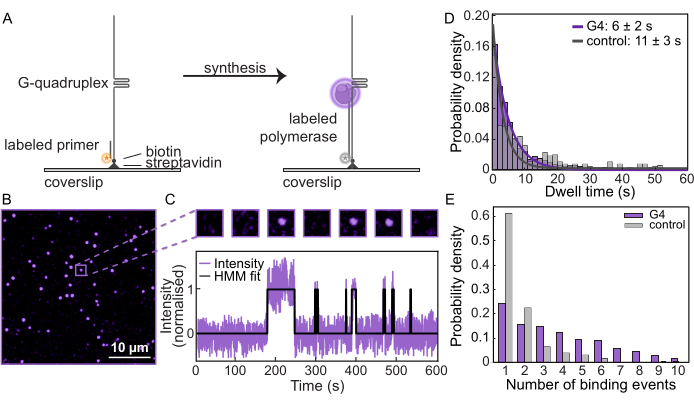
Figura 2: Microscopía de fluorescencia de una sola molécula. (A) Representación esquemática del ensayo de extensión del cebador de una sola molécula utilizando levadura Pol δ marcada con AF647. (B) Campo de visión típico obtenido a partir de un ensayo de una sola molécula. Cada mancha representa una δ Pol marcada con fluorescencia que se une a una plantilla de ADN. Barra de escala: 10 μm. (C) (Arriba) Molécula de ejemplo del ensayo de una sola molécula que muestra la unión on/off a medida que se intercambia el δ Pol. (Abajo) Trayectoria de una sola molécula de la misma molécula que muestra la intensidad a lo largo del tiempo del experimento. La línea negra representa el ajuste del modelo de Markov oculto (HMM) a los datos. (D) Tiempo de permanencia de Pol δ en el sustrato G4 (púrpura) y el sustrato control (gris). Las líneas representan ajustes exponenciales, lo que proporciona una vida útil de 6 s ± 2 s en el sustrato G4 y de 10 s ± 3 s en el sustrato de control. (E) Número de eventos de unión de Pol δ a sustratos individuales de ADN. La mediana del número de eventos de unión en el sustrato G4 (púrpura) es 3,5, en comparación con el sustrato de control 1 (gris). Haga clic aquí para ver una versión más grande de esta figura.
Figura complementaria 1: Espectroscopía CD. Espectros CD de la secuencia de formación de G4 (púrpura) y de control (gris) en un tampón TE que contiene 200 mM de KCl a 25 °C. El pico característico a 260 nm, seguido por el pico negativo a 240 nm, es característico de un GQ intramolecular paralelo. Haga clic aquí para descargar este archivo.
Figura complementaria 2: Construcción de la celda de flujo. (A) Esquema de las piezas individuales de la celda de flujo. El bloque cuadrado de PDMS se coloca en la parte superior del cubreobjetos del microscopio. A continuación, estas piezas se encajan dentro de la tapa y la parte inferior del soporte de la celda de flujo. (B) La celda de flujo completa. Se pueden insertar tubos en cada lado del bloque PDMS para formar los canales a través de los cuales los tampones pueden fluir a través del dispositivo a través de la microfluídica. Haga clic aquí para descargar este archivo.
Figura complementaria 3: Trayectorias de una sola molécula. (A) Ejemplo de trayectoria de una molécula que experimenta un solo evento de unión durante el período de tiempo de 10 minutos del experimento. (B) Ejemplo de trayectoria de una molécula que experimenta eventos de unión a cero durante el período de tiempo de 10 minutos del experimento. La línea negra en (A) y (B) representa el ajuste del modelo de Markov oculto (HMM) a los datos. Haga clic aquí para descargar este archivo.
Figura complementaria 4: Tiempos de permanencia. (A) Tiempo de permanencia de Pol δ en el sustrato G4. La línea representa el ajuste exponencial, dando un tiempo de vida de 6 s ± 2 s. (B) Tiempo de permanencia de Pol δ en el sustrato de control. El ajuste exponencial proporciona una vida útil de 10 s ± 3 s. Haga clic aquí para descargar este archivo.
Figura complementaria 5: Control del fotoblanqueo. El gráfico muestra el tiempo medio que tarda una enzima Pol δ de levadura individual marcada con AF647 en ser fotoblanqueada por el láser de 647 nm. La línea representa el ajuste exponencial, dando una vida útil de 39 ± 6 s. Haga clic aquí para descargar este archivo.
Discusión
Aquí, se ha descrito un ensayo basado en fluorescencia de una sola molécula que proporciona información sobre el comportamiento de una ADN polimerasa cuando se encuentra con un G-cuádrupplex. Si bien los protocolos de generación de plantillas de ADN, etiquetado de δ Pol y ensayos de replicación masiva de ADN son sencillos, la ejecución de ensayos de microscopía de una sola molécula es más desafiante desde el punto de vista técnico. Debido a la naturaleza de las técnicas de una sola molécula, se debe tener mucho cuidado para evitar la introducción de polvo, contaminación o burbujas de aire, ya que oscurecerán el campo de visión y dificultarán la recopilación de datos.
Una limitación de los experimentos de microscopía TIRF de una sola molécula es el fotoblanqueo de los fluoróforos que están acoplados covalentemente a las biomoléculas de interés. El fotoblanqueo es un proceso irreversible que conduce a la pérdida permanente de fluorescencia35. Para mitigar esto durante los experimentos, es esencial limitar la duración de la exposición al láser, ajustar la intensidad del láser y optimizar el tiempo de obtención de imágenes. Estas estrategias ayudan a preservar las señales de fluorescencia, lo que garantiza períodos de observación más fiables y prolongados. Al ajustar estos parámetros, la señal de Pol δ permanece durante toda la medición. Para optimizar la potencia del láser para los ensayos de síntesis de microscopía de fluorescencia de molécula única, es aconsejable medir la tasa de fotoblanqueo mediante la obtención de imágenes de la polimerasa marcada en un cubreobjetos de vidrio limpio. Al variar sistemáticamente la potencia del láser y evaluar la tasa de fotoblanqueo en todo el campo de visión, se puede identificar la intensidad óptima del láser que equilibra la intensidad de la señal del fluoróforo con la resistencia al fotoblanqueo (consulte la Figura complementaria 5).
La ventaja clave de este enfoque de una sola molécula sobre los métodos tradicionales basados en conjuntos es su capacidad para visualizar directamente cuándo una ADN polimerasa individual se encuentra e interactúa con una estructura G4. Los métodos tradicionales basados en conjuntos (como la electroforesis en gel) han demostrado la capacidad de las estructuras G4 para bloquear las ADN polimerasas 23,36,37. Estas técnicas, sin embargo, no proporcionan la información cinética y mecanicista en tiempo real de esta interacción, que es necesaria para desentrañar los diferentes pasos y resultados cinéticos moleculares. Las técnicas de una sola molécula ofrecen una visión sin precedentes de la cinética, los mecanismos y los comportamientos de las biomoléculas, a menudo ocultas por un promedio de38 conjuntos. Ahora es posible ver cómo actúan las ADN polimerasas en tiempo real, ya sea que intercambien, se detengan, se disocien o evitenlos obstáculos del ADN. Con este protocolo establecido, la identidad del G4 se puede cambiar fácilmente de la estructura c-MYC paralela elegida a cualquier topología paralela, antiparalela o híbrida. La aplicación de este ensayo de una sola molécula revelará si las mismas ADN polimerasas se comportan de manera diferente cuando se encuentran con topologías G4 alternativas. Como tal, los métodos de una sola molécula son vitales para responder preguntas específicas sobre cómo interactúan las proteínas del cuerpo y el ADN.
A través de la visualización directa de las interacciones de la ADN polimerasa con los G-cuádruples, se ha identificado una vía de intercambio previamente no caracterizada para la levadura Pol δ. Este descubrimiento sugiere que la polimerasa se desacopla al encontrarse con un G-cuádruple, a la espera de la intervención de otra proteína para resolver la estructura antes de reiniciar la síntesis de ADN. Este protocolo se puede adaptar para investigar las interacciones entre varias proteínas de mantenimiento del genoma y los obstáculos del ADN, lo que ofrece información sin precedentes sobre cómo las enzimas celulares navegan por los impedimentos genómicos. Por ejemplo, el obstáculo de este ensayo puede modificarse de una estructura G4 a un enlace cruzado proteína-ADN, un tipo de lesión del ADN en la que una proteína se une irreversiblemente de forma covalente al ADN, actuando como un obstáculo para la replicación del ADN40. Tales exámenes son cruciales para comprender los procesos fundamentales de la replicación, reparación y recombinación del ADN. Al permitir el estudio de la dinámica de las proteínas de ADN a nivel molecular, este ensayo proporciona una poderosa herramienta para dilucidar los mecanismos subyacentes a la integridad genómica.
Divulgaciones
Los autores declaran que no tienen intereses financieros contrapuestos.
Agradecimientos
N.K.-A. reconoce la financiación otorgada por la Beca del Programa de Capacitación en Investigación del Gobierno de Australia. L.M.S. está agradecida por la financiación que ha recibido del Consejo Nacional de Investigación Médica y de Salud (Researcher Grant 2007778). J.S.L. está agradecido de haber recibido un premio Discovery Early Career Award (DE240100780) y NHMRC Investigator EL1 (2025412) financiado por el gobierno australiano. S.H.M está agradecido de haber recibido la beca de carrera Bruce Warren Molecular Horizons Ealy.
Materiales
| Name | Company | Catalog Number | Comments |
| 100 nt control DNA template | Integrated DNA Technologies | Primary control template in primer extension assays | |
| 100 nt G4-forming DNA template | Integrated DNA Technologies | Primary G4-forming template in primer extension assays | |
| 15% Mini-PROTEAN TBE-Urea Gel, 12 well, 20 µL | Bio-Rad | 4566055 | Gel electrophoresis |
| 20 nt labelled/biotinylated primer oligonucleotide | Integrated DNA Technologies | Required for primer extension of the 100 nt templates | |
| 22 nt control DNA template | Integrated DNA Technologies | Primary control template in circular dichroism spectroscopy experiments | |
| 22 nt G4-forming DNA template | Integrated DNA Technologies | Primary G4-forming template in circular dichroism spectroscopy experiments | |
| 24 x 24 mm coverslip | Marienfeld Superior | 100062 | Required for TIRF microscopy |
| 3-aminopropyltriethoxysilane | Thermo Fisher Scientific | A10668.22 | Coverslip functionalization |
| 647 nm laser | Coherent | 1196627 | Select wavelength to correspond to stain/dye of choice |
| 670 nm emission filter | Chroma Technology Corp | ET655lp | Ensures only the relevant wavelengths are let through to the detector |
| Amersham Typhoon 5 biomolecular imager | Cytiva | 29187191 | Gel analysis |
| Biotin-PEG-SVA (MW 5000) | Laysan Bio | BIO-PEG-SVA-5K-100MG | Coverslip functionalization |
| Deoxynucleotides triphosphate solution mix | Jena Bioscience | NU-1005L | Replication building blocks |
| Digital dry bath (115V) | Bio-Rad | 1660571 | Heating solutions |
| Dithiothreitol | Sigma-Aldrich | 646563 | Disulfide bond reduction |
| EM-CCD camera | Andor | iXon 897 | Capturing single-molecule movies |
| Formamide | Sigma-Aldrich | F9037 | DNA denaturing during ensemble DNA replication assays |
| Gas tight syringe, 1 mL | SGE | 21964 | Filling cuvette for circular dichroism spectroscopy |
| Inverted microscope with CFI Apo TIRF 100x oil-immersion objective | Nikon | Eclipse Ti-E | Facilitates single-molecule TIRF microscopy |
| J-810 Spectrophotometer | Jasco | J-810 | Measuring circular dichroism spectroscopy |
| mPEG-SVA (MW 5000) | Laysan Bio | MPEG-SVA-5K-100MG | Coverslip functionalization |
| NanoDrop One Spectrophotometer | Thermofisher Scientific | ND-ONE | Measuring protein and label concentrations |
| NeutrAvidin | Thermo Fisher Scientific | 31000 | Tethering DNA templates to the functionalized coverslips during single-molecule experiments |
| Polyethylene tubing | Instech | BTPE-60 | Needed for flow cell construction |
| Quartz cuvette, 10 mm | Starna Scientific | 1/Q/10/CD | Holds sample for circular dichroism spectroscopy |
| Syringe pump | Adelab Scientific | NE-1002X | Used to flow buffers and substrates through a microfluidic flow cell |
| Tris-EDTA buffer | Thermofisher Scientific | 93283 | DNA solution buffer |
| Yeast polymerase δ | Michael O'Donnell Lab | Replicating the DNA templates | |
| Zeba spin desalting column, 7 K MWCO, 0.5 mL | Thermo Fisher Scientific | 89882 | Polymerase purification |
Referencias
- Sutton, M. D. Walker, G. C. Managing DNA polymerases: Coordinating DNA replication, DNA repair, and DNA recombination. Proc Natl Acad Sci U. S. A. 98 (15), 8342-8349 (2001).
- Garg, P. Burgers, P. M. J. DNA polymerases that propagate the eukaryotic DNA replication fork. Crit Rev Biochem Mol Biol. 40 (2), 115-128 (2005).
- Burgers, P. M. J. Eukaryotic DNA polymerases in DNA replication and DNA repair. Chromosoma. 107 (4), 218-227 (1998).
- Tsai, M.-D. How DNA polymerases catalyze DNA replication, repair, and mutation. Biochem. 53 (17), 2749-2751 (2014).
- Lewis, J. S. et al. Tunability of DNA polymerase stability during eukaryotic DNA replication. Mol Cell. 77 (1), 17-25 (2020).
- Chilkova, O. et al. The eukaryotic leading and lagging strand DNA polymerases are loaded onto primer-ends via separate mechanisms but have comparable processivity in the presence of PCNA. Nucleic Acids Res. 35 (19), 6588-6597 (2007).
- Kawasaki, Y. Sugino, A. Yeast replicative DNA polymerases and their role at the replication fork. Mol Cells. 12 (3), 277-285 (2001).
- Stillman, B. DNA polymerases at the replication fork in eukaryotes. Mol Cell. 30 (3), 259-260 (2008).
- Wood, R. D. Shivji, M. K. Which DNA polymerases are used for DNA repair in eukaryotes? Carcinogenesis. 18 (4), 605-610 (1997).
- Budd, M. E. Campbell, J. L. The roles of the eukaryotic DNA polymerases in DNA repair synthesis. Mutat Res DNA Repair. 384 (3), 157-167 (1997).
- Yang, W. Gao, Y. Translesion and repair DNA polymerases: Diverse structure and mechanism. Annu Rev Biochem. 87, 239-261 (2018).
- Bridges, B. A. DNA repair: Polymerases for passing lesions. Curr Biol. 9 (13), 475-477 (1999).
- Wilkinson, E. M., Spenkelink, L. M., Van Oijen, A. M. Observing protein dynamics during DNA-lesion bypass by the replisome. Front Mol Biosci. 9, 968424 (2022).
- Mueller, S. H., Spenkelink, L. M., Van Oijen, A. M. When proteins play tag: The dynamic nature of the replisome. Biophys Rev. 11 (4), 641-651 (2019).
- Zybailov, B. L., Sherpa, M. D., Glazko, G. V., Raney, K. D., Glazko, V. I. G4-quadruplexes, and genome instability. Mol Biol. 47 (2), 197-204 (2013).
- Loeb, L. A. Monnat, R. J. DNA polymerases and human disease. Nat Rev Genet. 9 (8), 594-604 (2008).
- Oganesian, L. Bryan, T. M. Physiological relevance of telomeric G-quadruplex formation: A potential drug target. Bioessays. 29 (2), 155-165 (2007).
- Chambers, V. S. et al. High-throughput sequencing of DNA G-quadruplex structures in the human genome. Nat Biotechnol. 33 (8), 877-881 (2015).
- Cheng, Y., Zhang, Y., You, H. Characterization of G-quadruplexes folding/unfolding dynamics and interactions with proteins from single-molecule force spectroscopy. Biomol. 11 (11), 1579 (2021).
- Tippana, R., Xiao, W., Myong, S. G-quadruplex conformation and dynamics are determined by loop length and sequence. Nucleic Acids Res. 42 (12), 8106-8114 (2014).
- Kusi-Appauh, N., Ralph, S. F., Van Oijen, A. M., Spenkelink, L. M. Understanding G-quadruplex biology and stability using single-molecule techniques. J Phys Chem B. 127 (25), 5521-5540 (2023).
- Obi, I. et al. Stabilization of G-quadruplex DNA structures in Schizosaccharomyces pombe causes single-strand DNA lesions and impedes DNA replication. Nucleic Acids Res. 48 (19), 10998-11015 (2020).
- Kamath-Loeb, A. S., Loeb, L. A., Johansson, E., Burgers, P. M., Fry, M. Interactions between the Werner syndrome helicase and DNA polymerase delta specifically facilitate copying of tetraplex and hairpin structures of the d(CGG)n trinucleotide repeat sequence. J Biol Chem. 276 (19), 16439-16446 (2001).
- Kaguni, L. S. Clayton, D. A. Template-directed pausing in in vitro DNA synthesis by DNA polymerase a from Drosophila melanogaster embryos. Proc Natl Acad Sci. 79 (4), 983-987 (1982).
- Sato, K., Martin-Pintado, N., Post, H., Altelaar, M., Knipscheer, P. Multistep mechanism of G-quadruplex resolution during DNA replication. Sci Adv. 7 (39), eabf8653 (2021).
- Castillo Bosch, P. et al. FANCJ promotes DNA synthesis through G-quadruplex structures. EMBO J. 33 (21), 2521-2533 (2014).
- Paeschke, K., Capra, John A., Zakian, Virginia A. DNA replication through G-quadruplex motifs is promoted by the Saccharomyces cerevisiae Pif1 DNA helicase. Cell. 145 (5), 678-691 (2011).
- Guo, L. et al. Joint efforts of replicative helicase and SSB ensure inherent replicative tolerance of G-quadruplex. Adv Sci. (Weinh.). 11 (9), e2307696 (2024).
- Pham, S. Q. T. et al. A new class of quadruplex DNA-binding nickel Schiff base complexes. Dalton Trans. 49 (15), 4843-4860 (2020).
- Georgescu, R. E. et al. Mechanism of asymmetric polymerase assembly at the eukaryotic replication fork. Nat Struct Mol Biol. 21 (8), 664-670 (2014).
- Au - Desjardins, P., Au - Hansen, J. B., Au - Allen, M. Microvolume protein concentration determination using the NanoDrop 2000c spectrophotometer. J Vis Exp. 33, e1610 (2009).
- Summer, H., Grämer, R., Dröge, P. Denaturing urea polyacrylamide gel electrophoresis (Urea PAGE). J Vis Exp. 32, e1485 (2009).
- Geertsema, H. J., Duderstadt, K. E., Van Oijen, A. M. in DNA Replication: Methods and Protocols., eds Vengrova, S., Dalgaard, J.) 219-238, Springer New York, New York, NY (2015).
- Lewis, J. S. et al. Single-molecule visualization of fast polymerase turnover in the bacterial replisome. eLife. 6, e23932 (2017).
- Fish, K. N. Total internal reflection fluorescence (TIRF) microscopy. Curr Protoc Cytom. 12, 12-18 (2009).
- Sommers, J. A., Estep, K. N., Maul, R. W., Brosh, R. M. Biochemical analysis of DNA synthesis blockage by G-quadruplex structure and bypass facilitated by a G4-resolving helicase. Methods. 204, 207-214 (2022).
- Edwards, D. N., Machwe, A., Wang, Z., Orren, D. K. Intramolecular telomeric G-quadruplexes dramatically inhibit DNA synthesis by replicative and translesion polymerases, revealing their potential to lead to genetic change. PLoS One. 9 (1), 80664 (2014).
- Mueller, S. H. et al. Rapid single-molecule characterization of enzymes involved in nucleic-acid metabolism. Nucleic Acids Res. 51 (1), 5 (2022).
- Lewis, J. S., Van Oijen, A. M., Spenkelink, L. M. Embracing heterogeneity: Challenging the paradigm of replisomes as deterministic machines. Chem Rev. 123 (23), 13419-13440 (2023).
- Stingele, J., Bellelli, R., Boulton, S. J. Mechanisms of DNA-protein crosslink repair. Nat Rev Mol Cell Biol. 18 (9), 563-573 (2017).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoExplorar más artículos
This article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados