Method Article
G-四链体 DNA 聚合酶动力学的单分子荧光可视化
摘要
该方案概述了一种基于荧光显微镜的单分子 DNA 复制测定,能够实时可视化 DNA 聚合酶与障碍物(如 G 四链体结构)之间的相互作用。
摘要
参与真核 DNA 复制的蛋白质克服障碍(例如蛋白质和 DNA“路障”)的能力对于确保忠实的基因组复制至关重要。G-四链体是在 DNA 中富含鸟嘌呤的区域形成的高阶核酸结构,已被证明是干扰基因组维持途径的障碍。本研究介绍了一种基于荧光显微镜的实时方法来观察 DNA 聚合酶与 G-四链体结构的相互作用。将含有 G-四链体的短引物 DNA 寡核苷酸固定在微流体流动池内的功能化玻璃盖玻片上。引入荧光标记的 DNA 聚合酶,可以随着时间的推移监测它们的行为和化学计量。这种方法能够观察聚合酶的行为,因为它被 G 四链体停滞。具体来说,使用荧光标记的酵母聚合酶 δ,发现在遇到 G-四链体时,聚合酶会经历连续的结合和解结合循环。这种单分子检测可用于研究各种 DNA 维持蛋白与 DNA 底物上障碍物之间的相互作用。
引言
DNA 聚合酶是催化核苷三磷酸掺入复制 DNA的酶 1,2,3,4,5。因此,它们在基本的 DNA 维持过程中起着关键作用,包括 DNA 复制 6,7,8 和修复 9,10,11,12。DNA 聚合酶必须准确有效地复制基因组,以确保基因组完整性,防止突变在基因组中积累。在合成过程中,聚合酶经常遇到“障碍”,例如 DNA 结合蛋白或二级 DNA 结构13。这些障碍可以减缓甚至阻止聚合酶进程14。克服这些障碍对于确保忠实的基因组复制非常重要,因为如果不这样做,会导致基因组不稳定15,16。
一类主要的障碍是 G-四链体 (G4),这是一种非经典二级 DNA 结构,已被证明在人类基因组中以富含鸟嘌呤的序列形成17。人类基因组中有超过 700,000 个不同的序列能够形成 G4,包括端粒和癌基因启动子内的区域18。这些 DNA 结构根据核苷酸序列、长度和结合的金属阳离子采用不同的构象 19,20,21。这种多样性意味着聚合酶必须克服一系列不同的 G4 拓扑结构,并且可能具有不同程度的效率。聚合酶无法克服或绕过 G4 结构已被证明会阻碍体内复制叉的进程,从而导致基因组不稳定22。体外研究表明,G4 结构可以阻止或完全阻断酵母聚合酶 23,24,25,26,27。G4 结构阻止或阻断 DNA 聚合酶的能力完全取决于它们的动力学和热力学稳定性,一些聚合酶能够展开某些 G4的 28。虽然这些研究提供了对聚合酶克服 G-四链体障碍的能力的见解,但它们缺乏在遇到 G4 时直接可视化聚合酶行为的能力。聚合酶的命运 - 它们是保持结合、脱落还是动态交换 - 决定了哪些下游工艺可用于解析 G4。
在这项研究中,开发了一种基于荧光的单分子显微镜测定法,可以直接实时观察和监测 DNA 聚合酶与 G4 结构的结合。该测定涉及将形成 G4 的 DNA 模板拴在微流体流动池中的生物素化盖玻片上,其中可以引入荧光标记的 DNA 聚合酶以启动 DNA 合成。通过测量聚合酶随时间变化的荧光,可以直接观察到它们在遇到 G4 结构时的行为。在 c-MYC 癌症癌基因中发现的 G4 结构因其高水平的稳定性而被选择用于该检测。该协议现在可以适应对与不同 G4 拓扑和稳定性相关的所有生命领域的各种聚合酶的行为进行分类。该检测提供了一种创新的高通量方法来阐明 DNA 聚合酶穿越 DNA 障碍的机制,为促进对聚合酶动力学的理解提供了强大的工具。
研究方案
材料 表中列出了所用试剂和所用设备的详细信息。
1. 圆二色谱光谱
注:在开发测定法之前,有必要对选定的 G4 序列进行圆二色谱 (CD) 光谱分析,以确保正确折叠。22 nt 序列 (5'-TGAGGGTGGGGAGGGGGGGGAA-3') 形成源自 c-MYC 癌症癌基因的 G4 结构。还对对照序列 (5'-TGAGTGTGAAGACGATGTAGAA -3) 进行了 CD 光谱分析,该序列的关键核苷酸发生了改变,阻止了 G4 的形成。
- 要制备用于 CD 光谱分析的 DNA 模板,将 40 μL 的 100 μM 的 22 nt 模板添加到 360 μL 的 Tris-HCl (pH 8.0)、乙二胺四乙酸 (EDTA) (TE) 缓冲液中,并补充有 200 mM 氯化钾 (KCl)。CD 光谱实验需要 10 μM 的最终 DNA 浓度。
注:K+ 离子的存在对于稳定模板中的 G4 结构至关重要。 - 在数字干浴中将 DNA 溶液加热至 95 °C 15 分钟。完成后,将加热块放入聚苯乙烯中,将溶液缓慢冷却过夜。这种缓慢的冷却过程是导致 G4 折叠的原因。
- 使用气密注射器将 400 μL G4 形成和控制序列溶液转移到 0.1 cm 光程的石英比色皿中。缓慢填充比色皿以避免形成气泡。
- 使用 25 °C 的光谱偏振仪测量 200 nm 和 400 nm 之间的 CD 光谱。 此测量的参数和条件已在前面29 中描述。结果可以在 补充图 1 中找到。
注:确保记录缓冲液的 CD 光谱,使其单独充当空白。
2. DNA 模板的制备
注:能够复制的模板是使用标准分子生物学技术制备的短的 100 nt 引物线性模板。G4 形成模板 (5′-GAATTACATTTAAATTTTACACAGATACAGTCAATGAGAA
CTTCCAGGCGTAACGAGAGCACGGGGGGT
GGGACCTTAGCTTCGAGTTCCGAT-3′)包含能够在其中心形成 MYC 衍生的 G 四链体(来自步骤 1)的序列。对照模板 (5′-GAATTACATTTAAATTTTACACAGATACAGTCAATGAGA
ACTTCCAGGCGTAACGAGAGCACGATGTAGCAGAACT
GTGACCTTAGCTTCGAGTTCCGAT-3′)在其中心也具有相同的控制区域(来自步骤 1)。
- 要将引物退火到 G4 形成和对照 DNA 模板,将 20 μM 引物(5'-生物素-AF647-TCTTAATGTAAATTAAAATGTGTC-3')混合到 20 μM 的 100 nt 模板中,溶于补充有 200 mM KCl 的 TE 缓冲液中。
- 在数字干浴中将 DNA 模板溶液加热至 95 °C 15 分钟。完成后,将加热块放入聚苯乙烯中,将溶液缓慢冷却过夜,以允许 G4 折叠和两根股线退火。
3. 用 AF647 标记 DNA 聚合酶
- 用染料标记聚合酶
注:酵母 Pol δ 如前所述表达和纯化30.然后需要用 Alexa Fluor NHS 酯单反应性染料 (AF647) 标记聚合酶,以便在单分子水平上可视化。这是如前所述完成的5.- 为了标记 N 端,将 3 倍过量的 AF647 染料与 320 μL 58.5 μM Pol δ 在含有 30 mM Tris-HCl(pH 7.6)、2 mM 二硫苏糖醇 (DTT)、300 mM 氯化钠 (NaCl)、50 mM 谷氨酸钾和 10% (v/v) 甘油的缓冲液中在 4 °C 下孵育 10 分钟。 轻轻旋转溶液以确保充分混合并提高标记效率。
注:蛋白质序列可能在赖氨酸氨基酸中含有额外的伯胺。通过调整反应条件(如 pH 值),标记可以优先贴在 N 端,而不是赖氨酸残基上。这可能需要进一步优化以提高贴标效率。 - 要制备 0.5 mL 离心脱盐柱(截留分子量为 7 K),首先将色谱柱以 1500 x g 离心 1 分钟以去除储存溶液。离心后丢弃沉积在收集管中的溶液。
注:确保在离心步骤中松开离心柱的盖子。这将确保色谱柱得到正确洗脱。 - 通过在柱顶部添加 300 μL 缓冲液,在含有 30 mM Tris-HCl (pH 7.6)、300 mM NaCl、3 mM DTT、50 mM 谷氨酸钾和 5% (v/v) 甘油的缓冲液中平衡色谱柱。将色谱柱以 1500 x g 离心 1 分钟,然后弃去流出液。再重复此过程两次。
- 将 200 μL 的每个 Pol δ 样品加入色谱柱中。将色谱柱以 1500 x g 离心 2 分钟,并将纯化的 Pol δ 收集在 1.5 mL 微量离心管中。
- 将纯化的级分冷冻在液体 N2 中,并将等分试样储存在 -80 °C 以防止蛋白质随时间降解。
- 为了标记 N 端,将 3 倍过量的 AF647 染料与 320 μL 58.5 μM Pol δ 在含有 30 mM Tris-HCl(pH 7.6)、2 mM 二硫苏糖醇 (DTT)、300 mM 氯化钠 (NaCl)、50 mM 谷氨酸钾和 10% (v/v) 甘油的缓冲液中在 4 °C 下孵育 10 分钟。 轻轻旋转溶液以确保充分混合并提高标记效率。
- 确定贴标效率
- 将 2 μL 现在 AF647 标记的 Pol δ 样品加载到紫外-可见分光光度计31 上。
注意:确保首先测量 Pol δ 缓冲液的空白。 - 使用分光光度计测量 AF647 染料的 650 nm 激发波长和 Pol δ 的 280 nm 激发波长。
- 使用 Beer-Lambert 定律和 AF647 染料的 270,000 cm-1 M-1 和聚合酶的 195,960 cm-1 M-1 的摩尔消光系数,计算标记效率。
注:计算出该测定中使用的 AF647 标记的酵母 Pol δ 的标记效率约为 67%。
- 将 2 μL 现在 AF647 标记的 Pol δ 样品加载到紫外-可见分光光度计31 上。
4. 集成引物延伸测定
注:在进行单分子复制实验之前,有必要 通过 批量复制测定确认 DNA 聚合酶被 G-四链体封闭。
- 将数字干浴中加热块的温度设置为 30 °C。 这将确保酵母 Pol δ 的最大活性。
- 在复制缓冲液(25 mM Tris-HCl (pH 7.6)、10 mM 乙酸镁、50 mM 谷氨酸钾、40 μg/mL 牛血清白蛋白 (BSA)、0.1 mM EDTA、5 mM DTT 和 0.0025% (v/v) 吐温-20)中制备 G4 形成和对照模板的“预混液”。用 1 mM DTT、250 μM dTTP、dCTP、dATP、dGTP (dNTP) 和 10 nM 的相应 DNA 模板(G4 形成或对照)补充每种混合物。将它们放入加热块中,以确保它们达到 30 °C。
- 用 12 μL 甲酰胺上样缓冲液(80% (w/v) 甲酰胺,10 mM EDTA)淬灭 12 μL 每种预混液,作为 T = 0 min 对照。
- 为了开始 DNA 合成,将 AF647 标记的酵母 Pol δ添加到每个预混液中(终浓度为 20 nM)。充分混合以确保 DNA 聚合酶以最高效率复制模板。
注意:使用过程中将酵母 Pol δ 放在冰上以保持酶活性。 - 30 秒、60 秒和 180 秒后,去除 12 μL 每种预混液,并用 12 μL 甲酰胺上样缓冲液淬灭。预混液与甲酰胺上样缓冲液的比例为 1:1,可确保复制反应在淬灭后无法继续。
- 从 30 °C 加热块中取出现已淬灭的溶液,并将其放入 98 °C 加热块中,以将 dsDNA 变性为 ssDNA。10 分钟后,将整个加热块带到凝胶槽中,将溶液加载到变性的 15% 三硼酸盐-EDTA (TBE)-尿素聚丙烯酰胺 (PAGE) 凝胶32 中。
注意:在加载样品之前,需要在 1x TBE 缓冲液中以 180 V 运行凝胶电泳 30 分钟,从而加热 PAGE 凝胶。完成后,冲洗每个孔以去除尿素。 - 将每个样品 15 μL 加载到凝胶上,旁边是含有 0.02 μM 20 nt、60 nt 和 100 nt 标记寡核苷酸的甲酰胺上样缓冲液的分子量标准。
- 在 1x TBE 缓冲液中以 180 V 电压运行凝胶 60 分钟,以确保完全分离。
- 在生物分子成像仪的 Cy5 通道中对凝胶进行成像。这将允许测量含有 AF-647 标记的复制 DNA 链。
5. 单分子荧光显微镜
- 盖玻片功能化
注:要将 DNA 模板连接到玻璃盖玻片上,必须首先用氨基硅烷功能化,然后用生物素化的 PEG 分子栓系。这个过程最大限度地减少了 DNA 和/或蛋白质与表面之间的非特异性相互作用。- 将盖玻片 (24 x 24 mm) 插入染色罐中并倒入乙醇,以清洁盖玻片 (24 x 24 mm)。对罐子进行超声处理 30 分钟,然后用纯水冲洗盖玻片。用 1 M 氢氧化钾 (KOH) 重复此过程,然后再次冲洗。再次重复这些清洁步骤。
- 冲洗并用丙酮填充一个新罐子,然后将盖玻片放入其中。将 3-氨基丙基三乙氧基硅烷扩散到罐中,形成 2% v/v 溶液。搅拌罐子 3 分钟,然后用大量水淬灭反应。
- 用 N2 干燥盖玻片,然后将它们单独放在装满水的潮湿箱中。这将防止盖玻片变干。
- 在 100 mM NaHCO3 (pH 8.2) 中制备 1:25 生物素-PEG-SVA:mPEG-SVA 酯。将这种混合物涡旋 20 秒以确保充分混合。
- 将 40 μL 的 PEG 溶液移液到干燥的盖玻片上。将另一个盖玻片放在顶部,将溶液“夹”在两个盖玻片之间。这将使内侧盖玻片的表面功能化。对所有盖玻片重复上述步骤。
- 将盖玻片在黑暗中孵育 3 小时。孵育后,将盖玻片对分开,用多余的水冲洗,然后用压缩的 N2 气体干燥。
- 重复步骤 4.1.4-4.1.5 将第二层 PEG 涂在盖玻片上。确保将先前功能化的侧面夹在一起,因此在洗涤步骤中要小心不要意外翻转它们。将溶液在黑暗中孵育过夜,然后冲洗和干燥。
- 将盖玻片存放在真空中以保持其功能。正确储存后,功能化的盖玻片可稳定保存一个月。
- 微流体流动池的制备
注:构建微流体流通池33 用于单分子实验(参见 补充图 2)。这将允许缓冲液、DNA 模板和蛋白质与功能化的盖玻片接触(参见步骤 5.1)。- 从真空中取出功能化的盖玻片,并将其放在部分装满水的微管架(潮湿箱)上。将 100 μL 封闭缓冲液(50 mM Tris-HCl (pH 7.6)、50 mM KCl、2% (v/v) Tween-20)与 25 μL 1 mg/mL 中性亲和素溶液(10% PBS)混合,并将其涂抹在盖玻片表面。让它在室温下在潮湿箱中孵育 15 分钟。
- 用水清洗盖玻片并在 N2 中干燥。请记住,盖玻片只有一侧是功能化的,因此请记住它是哪一侧。
- 将定制的聚二甲基硅氧烷 (PDMS) 模块34 放在流通池支架内的盖玻片上。这将产生一个 100 μm 高和 1 mm 宽的流道。然后,可以将聚乙烯管(PE-60:入口直径为 0.76 mm,外径为 1.22 mm)插入流通池的孔中,以提供缓冲液和基质的通道。
- 单分子荧光成像
注意:这些实验将使用 G4 形成和对照模板进行。这将允许使用全内反射 (TIRF) 显微镜在单分子水平上可视化这两种情况(G4 结构阻断 Pol δ且无阻断)。- 加热 1 mL 等分试样的吐温封闭缓冲液(50 mM Tris-HCl (pH 7.6)、50 mM KCl、2% (v/v) Tween-20)和 500 μL 等分试样的洗涤缓冲液(25 mM Tris-HCl (pH 7.6)、10 mM 乙酸镁、250 mM 谷氨酸钾、40 μg/mL BSA、0.1 mM EDTA、5 mM DTT、 0.0025% (v/v) 吐温-20)和复制缓冲液至 40 °C 15 分钟。这将从溶液中释放气体。在真空室中以低于大气压 800 mbar 的压力再脱气 15 分钟。
- 取出构建的流通池(参见步骤 4.2)并将其放在显微镜的载物台上。在物镜上滴一滴油后,抬起物镜以符合盖玻片。
注意:确保物镜和流通池有时间达到 31 °C。 这将增加酵母 Pol δ 的活性,并有助于确保数据的可重复性。 - 将入口管插入脱气的 Tween 封闭缓冲液中,并将出口连接到注射泵。向后拉注射器,将 Tween 封闭缓冲液通过管道吸入通道。让该缓冲液孵育至少 30 分钟,以尽量减少非特异性相互作用。
- 以 100 μL/min 的速率将 200 μL 脱气洗涤缓冲液流入通道。这将刷新 Tween 阻塞缓冲区。
- 在 500 μL 复制缓冲液中将 DNA 模板溶液稀释至 0.5 pM。以 10 μL/min 的速率将 150 μL 流入通道。使用大约 900 mWcm-2 的 647nm 激光在样品平面上照射样品,以可视化单个 DNA 模板。
注:如果样品的视野 (FOV) 未完全聚焦,则盖玻片可能未与构建的流通池底部齐平。如果发生这种情况,请使用新的盖玻片构建新的流通池。 - 一旦可见足够密度的斑点(每 10 μm2 约 1 个斑点),即可将新鲜的复制缓冲液溶液(补充有 1 mM DTT)流入通道以洗出多余的 DNA。250 μL 的体积足以完全去除所有未结合的 DNA。
- 移动到新的 FOV 并捕获 DNA 图像,以确定标记的聚合酶和 DNA 底物之间的共定位程度。完成后,增加激光功率以光漂白剩余的斑点。
- 在 200 μL 复制缓冲液中制备含有 1 mM DTT、250 μM dNTP 和 20 nM AF647 标记的 Pol δ 的聚合酶溶液。以 5 μL/min 的速率将 100 μL 聚合酶溶液流入通道。
- 一旦样品聚焦并调整了 TIRF 角度,在样品平面上将 647 nm 激光器的激光功率设置为大约 900 mWcm-2 。然后,开始对 FOV 进行所需时间长度的成像。采集 1-5 帧/秒 10-20 分钟以捕获所有复制事件。
- 数据分析
注意:所有分析均使用 Python (v. 3.11.7) 进行。用于数据分析的自定义代码可在此处获得:https://github.com/Single-molecule-Biophysics-UOW/G-quadruplex_binding_analysis。- 首先,将 DNA 模板点与标记的酵母 Pol δ 的荧光点共定位。如果两个峰值在漂移校正后的距离≤ 3 像素,则确定它们被共定位。仅分析共定位 DNA 模板的 Pol δ 点的结合动力学和整体行为趋势。
- 要确定绑定事件数据,请测量每个点随时间变化的强度,因为它们在整个影片中表现出“开/关”行为。这能够确定 Pol δ 结合在单个 DNA 模板上发生的次数。
注意:此分析针对 G4 形成和对照模板实验进行,以比较结合动力学。
注:已经进行了对照实验,以确保结合事件的分析中不包括与 G-四链体或模板本身(23.2 秒± 4.9 秒)的非特异性结合事件。
结果
对于该测定,设计了两种带有荧光标记的 20-nt 引物的 DNA 底物:一种含有 G4 形成序列(图 1A,左),另一种缺乏该序列(图 1A,右)。为了确认 G-四链体是聚合酶活性的有效障碍,在变性 PAGE 凝胶上监测了 Pol δ 的 DNA 合成。通过凝胶电泳检测纯化的标记 Pol δ 在 DNA 底物上的活性。 图 1B (左)显示荧光标记的 Pol δ 无法合成超过 G 四链体。在合成开始之前 (t = 0 min),存在对应于 20 nt 的条带,代表已从模板链变性的标记引物。3 分钟后,该 20 nt 条带转化为 60 nt 条带,表明所有 DNA 均发生合成,并确认聚合酶被 G-四链体结构完全封闭。这种阻断意味着聚合酶既不能展开也不能绕过结构。相比之下,不包含 G 四链体形成序列的对照模板的合成(图 1A,右)在 3 分钟后产生 100 nt 条带(图 1B,右)。
在确认模板上的合成并被 G-四链体有效封闭后,在单分子荧光显微镜上重复这些测量以监测聚合酶的行为。将 DNA 模板拴在微流体流动池中的功能化盖玻片(图 2A)上,并通过可视化荧光标记的引物来确定每种底物的位置。然后,在 dNTP 存在下加载标记的 Pol δ 以启动合成。在典型的 FOV 中,跟踪各个点以量化 Pol δ 与 DNA 结合和解离的频率(图 2B、C)。通过测量每个 DNA 底物的强度随时间的变化,可以生成单分子轨迹(图 2C, 补充图 3)。可以测量特征的“停留时间”,或 Pol δ 保持绑定到模板的平均持续时间。对于 G4 基材,停留时间确定为 6 秒± 2 秒,而对于对照基材,为 10 秒± 3 秒(图 2D, 补充图 4)。此外,对于每个轨迹,可以量化 Pol δ 与模板结合的次数。与对照模板相比,与 G4 底物的结合事件数量要高得多(图 2E)。虽然由于学术绑定和取消绑定,控件模板中存在多个绑定事件的实例,但 G4 格式模板的绑定事件数平均明显增加。这表明,在合成被 G-四链体终止后,结合的聚合酶在溶液中的新聚合酶进行连续的结合和解结合循环之前与 DNA 解离。因此,这种单分子检测为 DNA 聚合酶如何响应 DNA 障碍提供了无与伦比的见解。

图 1:集成引物延伸测定。 (A) DNA 底物的示意图。G4 底物(左)包含 G-四链体形成序列,而对照底物(右)则不包含。(B) 引物 G4 和对照 DNA 底物的集合 DNA 引物酶延伸测定显示荧光标记的酵母 Pol δ 被 G-四链体完全阻断。PAGE 凝胶显示 DNA 模板随时间推移的复制,导致标记的 20 nt 引物转变为 G4 底物的 60 nt 产物(左图)和对照模板的 100 nt 产物(右图)。M 代表含有标记的 20 nt、60 nt 和 100 nt 寡核苷酸的分子量标准。 请单击此处查看此图的较大版本。
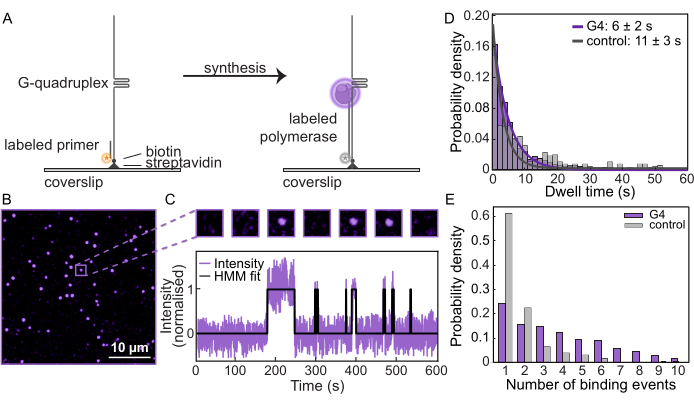
图 2:单分子荧光显微镜。 (A) 使用 AF647 标记的酵母 Pol δ 的单分子引物延伸测定的示意图。(B) 从单分子测定中获得的典型 FOV。每个位点代表一个与 DNA 模板结合δ荧光标记的 Pol。比例尺:10 μm。(C) (顶部)来自单分子测定的示例分子,显示 Pol δ 交换时的开/关结合。(下)同一分子的单分子轨迹,显示实验过程中的强度。黑线表示隐马尔可夫模型 (HMM) 与数据的拟合。(D) Pol δ 在 G4 衬底(紫色)和对照衬底(灰色)上的停留时间。这些线表示指数拟合,在 G4 衬底上的寿命为 6 秒± 2 秒,在对照衬底上的寿命为 10 秒± 3 秒。(E) Pol δ 与单个 DNA 底物结合事件的数量。与对照底物 1(灰色)相比,G4 底物(紫色)上的结合事件中位数为 3.5。 请单击此处查看此图的较大版本。
补充图 1:CD 光谱。 在 25 °C 下含有 200 mM KCl 的 TE 缓冲液中形成 G4 的 CD 光谱(紫色)和对照序列(灰色)。 260 nm 处的特征峰和 240 nm 处的负峰是平行分子内 GQ 的特征。 请点击此处下载此文件。
补充图 2:流通池构建。(A) 流通池各个部分的示意图。方形 PDMS 块位于显微镜盖玻片的顶部。然后将这些部件组装在一起,放在流通池支架的盖子和底部。(B) 完整的流通池。可以将管子插入 PDMS 模块的每一侧,以形成通道,缓冲液 可以通过微流体 流过设备。 请点击此处下载此文件。
补充图 3:单分子轨迹。 (A) 在实验的 10 分钟时间范围内经历单个结合事件的分子的示例轨迹。(B) 在实验的 10 分钟时间范围内经历零结合事件的分子的示例轨迹。(A) 和 (B) 中的黑线表示隐马尔可夫模型 (HMM) 与数据的拟合。 请点击此处下载此文件。
补充图 4:停留时间。 (A) Pol δ 在 G4 衬底上的停留时间。该线表示指数拟合,寿命为 6 秒± 2 秒。(B) Pol δ 在对照衬底上的停留时间。指数拟合的寿命为 10 秒± 3 秒。 请单击此处下载此文件。
补充图 5:光漂白控制。 该图显示了单个 AF647 标记的酵母 Pol δ 酶被 647 nm 激光光漂白的平均时间。该线代表指数拟合,寿命为 39 ± 6 秒。 请单击此处下载此文件。
讨论
在这里,描述了一种基于单分子荧光的分析,它提供了对 DNA 聚合酶遇到 G-四链体时的行为的见解。虽然 DNA 模板生成、Pol δ标记和大量 DNA 复制检测的方案都很简单,但执行单分子显微镜检测在技术上更具挑战性。由于单分子技术的性质,必须非常小心地避免引入灰尘、污染或气泡,因为这些会掩盖 FOV 并妨碍数据收集。
单分子 TIRF 显微镜实验的一个局限性是与目标生物分子共价偶联的荧光团的光漂白。光漂白是一个不可逆的过程,会导致永久性荧光损失35。为了在实验过程中缓解这种情况,必须限制激光曝光持续时间、调整激光强度并优化成像时间。这些策略有助于保留荧光信号,确保更可靠和更长的观察时间。通过微调这些参数,Pol δ 信号在测量期间保持不变。为了优化单分子荧光显微镜合成测定的激光功率,建议通过在干净的玻璃盖玻片上对标记的聚合酶进行成像来测量光漂白速率。通过系统地改变激光功率并评估整个视场的光漂白速率,可以确定平衡荧光团信号强度和抗光漂白性的最佳激光强度(参见 补充图 5)。
与传统的基于集合的方法相比,这种单分子方法的主要优势在于,它能够直接可视化单个 DNA 聚合酶何时遇到 G4 结构并与之相互作用。传统的基于集合的方法(如凝胶电泳)已经证明了 G4 结构能够阻断 DNA 聚合酶 23,36,37。然而,这些技术未能提供这种相互作用的实时动力学和机理信息,而这些信息对于理清不同的分子动力学步骤和结果是必要的。单分子技术为生物分子的动力学、机制和行为提供了无与伦比的洞察力,这些生物分子通常被集合平均38 所隐藏。现在可以实时查看 DNA 聚合酶如何发挥作用 - 它们是否交换、停滞、解离或绕过 DNA 障碍39。建立此协议后,可以轻松地将 G4 的身份从选定的并行 c-MYC 结构更改为任何并行、反并行或混合拓扑。应用这种单分子检测将揭示相同的 DNA 聚合酶在遇到替代 G4 拓扑结构时是否表现不同。因此,单分子方法对于回答有关人体蛋白质和 DNA 如何相互作用的特定问题至关重要。
通过直接观察 DNA 聚合酶与 G-四链体的相互作用,已经确定了酵母 Pol δ 以前未表征的交换途径。这一发现表明,聚合酶在遇到 G-四链体时脱离,等待另一种蛋白质的干预以解析结构,然后再重新开始 DNA 合成。该方案可用于研究各种基因组维持蛋白和 DNA 障碍之间的相互作用,为细胞酶如何导航基因组障碍提供无与伦比的见解。例如,该测定的障碍可以从 G4 结构转变为蛋白质-DNA 交联,这是一种 DNA 损伤,其中蛋白质与 DNA 不可逆地共价结合,成为 DNA 复制的障碍40。此类检查对于了解 DNA 复制、修复和重组的基本过程至关重要。通过在分子水平上研究 DNA-蛋白质动力学,该测定为阐明基因组完整性的机制提供了强大的工具。
披露声明
作者声明他们没有相互竞争的经济利益。
致谢
N.K.-A.感谢澳大利亚政府研究培训计划奖学金提供的资金。LMS 感谢她从国家健康与医学研究委员会(研究员 Grant 2007778)获得的资助。JSL 很高兴成为澳大利亚政府资助的 Discovery Early Career Award (DE240100780) 和 NHMRC Investigator EL1 (2025412) 的获得者。S.H.M 很高兴成为 Bruce Warren Molecular Horizons Ealy 职业奖学金的获得者。
材料
| Name | Company | Catalog Number | Comments |
| 100 nt control DNA template | Integrated DNA Technologies | Primary control template in primer extension assays | |
| 100 nt G4-forming DNA template | Integrated DNA Technologies | Primary G4-forming template in primer extension assays | |
| 15% Mini-PROTEAN TBE-Urea Gel, 12 well, 20 µL | Bio-Rad | 4566055 | Gel electrophoresis |
| 20 nt labelled/biotinylated primer oligonucleotide | Integrated DNA Technologies | Required for primer extension of the 100 nt templates | |
| 22 nt control DNA template | Integrated DNA Technologies | Primary control template in circular dichroism spectroscopy experiments | |
| 22 nt G4-forming DNA template | Integrated DNA Technologies | Primary G4-forming template in circular dichroism spectroscopy experiments | |
| 24 x 24 mm coverslip | Marienfeld Superior | 100062 | Required for TIRF microscopy |
| 3-aminopropyltriethoxysilane | Thermo Fisher Scientific | A10668.22 | Coverslip functionalization |
| 647 nm laser | Coherent | 1196627 | Select wavelength to correspond to stain/dye of choice |
| 670 nm emission filter | Chroma Technology Corp | ET655lp | Ensures only the relevant wavelengths are let through to the detector |
| Amersham Typhoon 5 biomolecular imager | Cytiva | 29187191 | Gel analysis |
| Biotin-PEG-SVA (MW 5000) | Laysan Bio | BIO-PEG-SVA-5K-100MG | Coverslip functionalization |
| Deoxynucleotides triphosphate solution mix | Jena Bioscience | NU-1005L | Replication building blocks |
| Digital dry bath (115V) | Bio-Rad | 1660571 | Heating solutions |
| Dithiothreitol | Sigma-Aldrich | 646563 | Disulfide bond reduction |
| EM-CCD camera | Andor | iXon 897 | Capturing single-molecule movies |
| Formamide | Sigma-Aldrich | F9037 | DNA denaturing during ensemble DNA replication assays |
| Gas tight syringe, 1 mL | SGE | 21964 | Filling cuvette for circular dichroism spectroscopy |
| Inverted microscope with CFI Apo TIRF 100x oil-immersion objective | Nikon | Eclipse Ti-E | Facilitates single-molecule TIRF microscopy |
| J-810 Spectrophotometer | Jasco | J-810 | Measuring circular dichroism spectroscopy |
| mPEG-SVA (MW 5000) | Laysan Bio | MPEG-SVA-5K-100MG | Coverslip functionalization |
| NanoDrop One Spectrophotometer | Thermofisher Scientific | ND-ONE | Measuring protein and label concentrations |
| NeutrAvidin | Thermo Fisher Scientific | 31000 | Tethering DNA templates to the functionalized coverslips during single-molecule experiments |
| Polyethylene tubing | Instech | BTPE-60 | Needed for flow cell construction |
| Quartz cuvette, 10 mm | Starna Scientific | 1/Q/10/CD | Holds sample for circular dichroism spectroscopy |
| Syringe pump | Adelab Scientific | NE-1002X | Used to flow buffers and substrates through a microfluidic flow cell |
| Tris-EDTA buffer | Thermofisher Scientific | 93283 | DNA solution buffer |
| Yeast polymerase δ | Michael O'Donnell Lab | Replicating the DNA templates | |
| Zeba spin desalting column, 7 K MWCO, 0.5 mL | Thermo Fisher Scientific | 89882 | Polymerase purification |
参考文献
- Sutton, M. D. Walker, G. C. Managing DNA polymerases: Coordinating DNA replication, DNA repair, and DNA recombination. Proc Natl Acad Sci U. S. A. 98 (15), 8342-8349 (2001).
- Garg, P. Burgers, P. M. J. DNA polymerases that propagate the eukaryotic DNA replication fork. Crit Rev Biochem Mol Biol. 40 (2), 115-128 (2005).
- Burgers, P. M. J. Eukaryotic DNA polymerases in DNA replication and DNA repair. Chromosoma. 107 (4), 218-227 (1998).
- Tsai, M.-D. How DNA polymerases catalyze DNA replication, repair, and mutation. Biochem. 53 (17), 2749-2751 (2014).
- Lewis, J. S. et al. Tunability of DNA polymerase stability during eukaryotic DNA replication. Mol Cell. 77 (1), 17-25 (2020).
- Chilkova, O. et al. The eukaryotic leading and lagging strand DNA polymerases are loaded onto primer-ends via separate mechanisms but have comparable processivity in the presence of PCNA. Nucleic Acids Res. 35 (19), 6588-6597 (2007).
- Kawasaki, Y. Sugino, A. Yeast replicative DNA polymerases and their role at the replication fork. Mol Cells. 12 (3), 277-285 (2001).
- Stillman, B. DNA polymerases at the replication fork in eukaryotes. Mol Cell. 30 (3), 259-260 (2008).
- Wood, R. D. Shivji, M. K. Which DNA polymerases are used for DNA repair in eukaryotes? Carcinogenesis. 18 (4), 605-610 (1997).
- Budd, M. E. Campbell, J. L. The roles of the eukaryotic DNA polymerases in DNA repair synthesis. Mutat Res DNA Repair. 384 (3), 157-167 (1997).
- Yang, W. Gao, Y. Translesion and repair DNA polymerases: Diverse structure and mechanism. Annu Rev Biochem. 87, 239-261 (2018).
- Bridges, B. A. DNA repair: Polymerases for passing lesions. Curr Biol. 9 (13), 475-477 (1999).
- Wilkinson, E. M., Spenkelink, L. M., Van Oijen, A. M. Observing protein dynamics during DNA-lesion bypass by the replisome. Front Mol Biosci. 9, 968424 (2022).
- Mueller, S. H., Spenkelink, L. M., Van Oijen, A. M. When proteins play tag: The dynamic nature of the replisome. Biophys Rev. 11 (4), 641-651 (2019).
- Zybailov, B. L., Sherpa, M. D., Glazko, G. V., Raney, K. D., Glazko, V. I. G4-quadruplexes, and genome instability. Mol Biol. 47 (2), 197-204 (2013).
- Loeb, L. A. Monnat, R. J. DNA polymerases and human disease. Nat Rev Genet. 9 (8), 594-604 (2008).
- Oganesian, L. Bryan, T. M. Physiological relevance of telomeric G-quadruplex formation: A potential drug target. Bioessays. 29 (2), 155-165 (2007).
- Chambers, V. S. et al. High-throughput sequencing of DNA G-quadruplex structures in the human genome. Nat Biotechnol. 33 (8), 877-881 (2015).
- Cheng, Y., Zhang, Y., You, H. Characterization of G-quadruplexes folding/unfolding dynamics and interactions with proteins from single-molecule force spectroscopy. Biomol. 11 (11), 1579 (2021).
- Tippana, R., Xiao, W., Myong, S. G-quadruplex conformation and dynamics are determined by loop length and sequence. Nucleic Acids Res. 42 (12), 8106-8114 (2014).
- Kusi-Appauh, N., Ralph, S. F., Van Oijen, A. M., Spenkelink, L. M. Understanding G-quadruplex biology and stability using single-molecule techniques. J Phys Chem B. 127 (25), 5521-5540 (2023).
- Obi, I. et al. Stabilization of G-quadruplex DNA structures in Schizosaccharomyces pombe causes single-strand DNA lesions and impedes DNA replication. Nucleic Acids Res. 48 (19), 10998-11015 (2020).
- Kamath-Loeb, A. S., Loeb, L. A., Johansson, E., Burgers, P. M., Fry, M. Interactions between the Werner syndrome helicase and DNA polymerase delta specifically facilitate copying of tetraplex and hairpin structures of the d(CGG)n trinucleotide repeat sequence. J Biol Chem. 276 (19), 16439-16446 (2001).
- Kaguni, L. S. Clayton, D. A. Template-directed pausing in in vitro DNA synthesis by DNA polymerase a from Drosophila melanogaster embryos. Proc Natl Acad Sci. 79 (4), 983-987 (1982).
- Sato, K., Martin-Pintado, N., Post, H., Altelaar, M., Knipscheer, P. Multistep mechanism of G-quadruplex resolution during DNA replication. Sci Adv. 7 (39), eabf8653 (2021).
- Castillo Bosch, P. et al. FANCJ promotes DNA synthesis through G-quadruplex structures. EMBO J. 33 (21), 2521-2533 (2014).
- Paeschke, K., Capra, John A., Zakian, Virginia A. DNA replication through G-quadruplex motifs is promoted by the Saccharomyces cerevisiae Pif1 DNA helicase. Cell. 145 (5), 678-691 (2011).
- Guo, L. et al. Joint efforts of replicative helicase and SSB ensure inherent replicative tolerance of G-quadruplex. Adv Sci. (Weinh.). 11 (9), e2307696 (2024).
- Pham, S. Q. T. et al. A new class of quadruplex DNA-binding nickel Schiff base complexes. Dalton Trans. 49 (15), 4843-4860 (2020).
- Georgescu, R. E. et al. Mechanism of asymmetric polymerase assembly at the eukaryotic replication fork. Nat Struct Mol Biol. 21 (8), 664-670 (2014).
- Au - Desjardins, P., Au - Hansen, J. B., Au - Allen, M. Microvolume protein concentration determination using the NanoDrop 2000c spectrophotometer. J Vis Exp. 33, e1610 (2009).
- Summer, H., Grämer, R., Dröge, P. Denaturing urea polyacrylamide gel electrophoresis (Urea PAGE). J Vis Exp. 32, e1485 (2009).
- Geertsema, H. J., Duderstadt, K. E., Van Oijen, A. M. in DNA Replication: Methods and Protocols., eds Vengrova, S., Dalgaard, J.) 219-238, Springer New York, New York, NY (2015).
- Lewis, J. S. et al. Single-molecule visualization of fast polymerase turnover in the bacterial replisome. eLife. 6, e23932 (2017).
- Fish, K. N. Total internal reflection fluorescence (TIRF) microscopy. Curr Protoc Cytom. 12, 12-18 (2009).
- Sommers, J. A., Estep, K. N., Maul, R. W., Brosh, R. M. Biochemical analysis of DNA synthesis blockage by G-quadruplex structure and bypass facilitated by a G4-resolving helicase. Methods. 204, 207-214 (2022).
- Edwards, D. N., Machwe, A., Wang, Z., Orren, D. K. Intramolecular telomeric G-quadruplexes dramatically inhibit DNA synthesis by replicative and translesion polymerases, revealing their potential to lead to genetic change. PLoS One. 9 (1), 80664 (2014).
- Mueller, S. H. et al. Rapid single-molecule characterization of enzymes involved in nucleic-acid metabolism. Nucleic Acids Res. 51 (1), 5 (2022).
- Lewis, J. S., Van Oijen, A. M., Spenkelink, L. M. Embracing heterogeneity: Challenging the paradigm of replisomes as deterministic machines. Chem Rev. 123 (23), 13419-13440 (2023).
- Stingele, J., Bellelli, R., Boulton, S. J. Mechanisms of DNA-protein crosslink repair. Nat Rev Mol Cell Biol. 18 (9), 563-573 (2017).
转载和许可
请求许可使用此 JoVE 文章的文本或图形
请求许可探索更多文章
This article has been published
Video Coming Soon
版权所属 © 2025 MyJoVE 公司版权所有,本公司不涉及任何医疗业务和医疗服务。