Un abonnement à JoVE est nécessaire pour voir ce contenu. Connectez-vous ou commencez votre essai gratuit.
Method Article
Purification du réticulum sarco-endoplasmique Ca2+-ATPase du muscle de lapin
Dans cet article
Résumé
Ce protocole décrit une méthode de purification SERCA améliorée, qui inclut le disaccharide tréhalose dans l’étape finale de centrifugation. Ce glucide stabilise les protéines dans des conditions difficiles. Le SERCA purifié était catalytiquement actif et présentait une grande pureté, ce qui le rendait adapté aux études structurelles et fonctionnelles.
Résumé
Certaines ATPases de type P, telles que le réticulum sarco/endoplasmique Ca2+-ATPase (SERCA), sont des protéines membranaires intrinsèquement labiles qui nécessitent des conditions physicochimiques spécifiques lors de la purification pour les obtenir avec une pureté et une qualité structurelle élevées et sous une forme catalytiquement active. Le disaccharide tréhalose est un soluté compatible qui est synthétisé et accumulé en fortes concentrations dans le cytoplasme de la levure pour stabiliser les membranes et les protéines. L’utilisation du tréhalose comme additif dans le protocole de purification de la membrane plasmique H+-ATPase permet d’obtenir une préparation de haute qualité, dont la structure hexamérique est mise en évidence par les méthodes d’analyse biochimique. Le tréhalose peut donc être utilisé comme additif stabilisant pour la purification des protéines membranaires (P-ATPases). Ce protocole décrit la modification du protocole classique de purification du SERCA en soumettant le SERCA à une centrifugation sur un gradient de concentration de tréhalose. L’inclusion de ce glucide a conduit à la purification de SERCA sous une forme catalytiquement active de haute pureté et, surtout, sous une forme stable. La caractérisation biochimique partielle du SERCA PURIFIÉ (SDS-PAGE, cinétique enzymatique, marquage FITC, spectroscopie de dichroïsme circulaire) a montré que l’enzyme est adaptée aux études fonctionnelles et structurales. L’utilisation du tréhalose dans le protocole de purification des ATPases de type P et d’autres protéines membranaires labiles (et cytosoliques) est suggérée.
Introduction
Les protéines/enzymes membranaires sont des composants biologiques essentiels des cellules car elles jouent un rôle essentiel dans divers processus 1,2,3. Certaines des fonctions peuvent inclure le transport d’ions et de molécules à l’intérieur et à l’extérieur des compartiments cellulaires/internes (actifs/passifs), la reconnaissance cellule-cellule, la liaison intercellulaire, l’ancrage/l’attachement, et la détection de l’environnement externe par intégration avec la machinerie de transduction du signal dans des conditions physiques et chimiques normales et difficiles (sel élevé, eau faible, température élevée, résistance aux médicaments, etc.)3 Par conséquent, la détermination de la structure tridimensionnelle (3D) des protéines et/ou des enzymes membranaires est devenue d’une grande importance pour la recherche fondamentale et appliquée 4,7,6. Il est important de noter que les protéines/enzymes membranaires ont été largement utilisées comme cibles pour la découverte de médicaments (qu’elles soient naturelles ou intentionnelles)7,8,9. C’est-à-dire que les protéines membranaires ont une importance inhérente à la santé 9,10,11.
Le caractère hydrophobe des protéines/enzymes membranaires est la propriété physicochimique la plus difficile sur le plan technique pour le laboratoire expérimental 12,13,14, encore plus lorsqu’il s’agit de travailler avec des protéines membranaires intégrales oligomères et/ou hautement labiles 15,16. Il est hautement souhaitable d’isoler des quantités appropriées de la protéine/enzyme membranaire avec la meilleure qualité possible pour les essais expérimentaux fonctionnels et les études structurales17. Les protéines membranaires, étant intrinsèquement hydrophobes, sont très difficiles à purifier, et le choix du détergent est généralement l’une des questions les plus importantes à prendre en compte 17,18,19,20. À cet égard, les méthodes de laboratoire utilisées pour isoler les protéines membranaires causent généralement un certain degré de dommages à l’arrangement structurel 3D de la protéine21. Certaines de ces méthodes comprennent l’utilisation (a) de sonications (ondes ultrasonores à haute fréquence/énergie), (b) de détergents solubilisants de protéines (durs, moyens ou doux)22, (c) de pressions relativement élevées dans la chromatographie sur colonne et l’ultracentrifugation à grande vitesse23, (d) de molécules précipitantes, (e) d’enzymes digestives, et d’autres21. Tous ces processus peuvent contribuer ou être la principale cause de déstabilisation des protéines lors de la purification21. À cet égard, certains protocoles semblent fonctionner relativement bien pour une protéine membranaire donnée. Cependant, l’optimisation est toujours la bienvenue lors de l’utilisation de nouveaux tests modernes ou de méthodes qui nécessitent une préparation de protéines membranaires de meilleure qualité pour obtenir des résultats satisfaisants24. Les étapes d’optimisation peuvent inclure, sans s’y limiter, l’amélioration de la conception des constructions, la recherche de conditions optimales pour l’expression des protéines membranaires, l’établissement de meilleures conditions de manipulation (pH, température, etc.), la recherche du meilleur détergent compatible, l’ajustement des étapes de purification telles que le temps de sonication, la vitesse de centrifugation, la reformulation des solutions tampons par l’ajout d’agents stabilisants, etc.25,26,27. Par conséquent, tout changement dans la méthodologie de purification qui conduit à une augmentation de la qualité (pureté) et de l’activité de la protéine/enzyme membranaire purifiée est important.
Dans la famille des ATPases de type P, la membrane plasmique de levure H+-ATPase semble être l’un des membres les plus labiles28,29. La H+-ATPase perd son activité d’hydrolyse de l’ATP lors de la déshydratation (lyophilisation), du choc thermique, etc.28,29. L’utilisation du tréhalose comme stabilisateur protéique a été testée dans l’isolement de la membrane plasmique H+-ATPase de la levure K. lactis30,31 ; la préparation de H+-ATPase obtenue était de grande pureté et catalytiquement active. Il est important de noter que cela a permis de résoudre l’état oligomérique de l’enzyme par des méthodes biochimiques, révélant qu’il s’agit d’un hexamère et confirmé plus tard par cryo-microscopie électronique 31,32,33,34,35. Par conséquent, il semble probable que l’arrangement tridimensionnel délicat (3D) des protéines membranaires puisse être perdu dans des conditions relativement difficiles lors de la purification36. Une cinétique d’inactivation biphasique est observée pour la H+-ATPase lors de l’inactivation induite par la chaleur29. Dans les cellules de levure, comme dans de nombreux autres organismes, le disaccharide tréhalose s’accumule à des concentrations élevées dans des conditions de stress environnemental 37,38,39. Le tréhalose maintient l’intégrité de la membrane et la fonction de transport en stabilisant les protéines (à la fois intégrées dans la membrane et cytosoliques) et les membranes cellulaires40,41. Le mécanisme de stabilisation du tréhalose a été étudié de manière approfondie par plusieurs groupes et par notre laboratoire 42,43,44,45. Des expériences avec la H+-ATPase et d’autres enzymes ont montré que le tréhalose est le stabilisateur protéique le plus efficace parmi les mono- et disaccharides28,29. Cela a conduit à son inclusion dans le protocole de purification H+-ATPase30. Récemment, le tréhalose a également été utilisé dans la purification du réticulum sarcoplasmique Ca2+-ATPase (SERCA) du muscle à contraction rapide du lapin avec de bons résultats en termes de pureté et d’activitédes protéines 46. Par conséquent, le tréhalose semble être un bon additif approprié pour la purification des ATPases de type P et probablement d’autres protéines membranaires et cytosoliques.
Pour les P-ATPases, l’existence de domaines cytoplasmiques structurels est un avantage expérimental, en particulier pour les études d’interaction substrat/ligand47 ; La liaison à l’ATP a été étudiée dans les domaines N recombinants hautement purs 47,48,49, éliminant ainsi les considérations techniques pour la purification des enzymes membranaires entières 47,48,50, entre autres 51. Malheureusement, certaines études fonctionnelles (conversion catalytique/énergétique) et structurelles (arrangement des sous-unités et interaction avec d’autres protéines) nécessitent encore l’intégralité de la P-ATPase52,53. À cet égard, la purification de SERCA a été réalisée par plusieurs groupes de recherche 54,55,56,57,58. Cependant, des améliorations peuvent encore être mises en œuvre46, par exemple, augmenter l’intégrité de l’ATPase59 purifiée, éviter la dénaturation/perturbation des protéines des complexes protéiques25, augmenter la solubilisation sans dénaturation de la protéine membranaire (c’est-à-dire éviter la formation d’agrégats macromoléculaires)60, conduire à une meilleure compatibilité avec les dosages et autres méthodes analytiques en aval61 De plus, au fur et à mesure que de nouvelles stratégies expérimentales, additifs, inhibiteurs d’enzymes, etc. apparaissent dans la littérature scientifique 61,62,63,64,65,66, ils doivent parfois être testés avec l’ensemble de la P-ATPase. Ce travail décrit le protocole de purification de SERCA et l’utilisation du tréhalose comme additif pour stabiliser la structure protéique et l’activité de l’ATPase ; C’est-à-dire qu’en plus d’augmenter la qualité enzymatique (structurelle), le tréhalose aide à prévenir la perte de structure et d’activité enzymatiques pendant l’isolement et le stockage des enzymes, ce qui permet d’économiser du matériel biologique et donc de réduire le nombre de purifications enzymatiques.
Protocole
Toutes les procédures sur les animaux ont été effectuées conformément aux directives internationales et locales (NORMA Oficial Mexicana NOM-062-ZOO-1999) pour la manipulation des animaux dans les laboratoires expérimentaux 51,67,68. Du tissu musculaire a été obtenu à partir d’Oryctolagus cuniculus de type sauvage provenant d’une unité locale de manipulation d’animaux (INE/CITES/DGVS-ZOO-E0055-SLP-98)46. Un vétérinaire spécialisé dans la gestion des animaux de laboratoire a effectué la dissection et le traitement musculaires initiaux. Les détails des réactifs et de l’équipement utilisés dans cette étude sont énumérés dans la table des matériaux.
1. Isolement du réticulum sarcoplasmique à partir d’un muscle de lapin à contraction rapide (Durée, 7 - 9 h)
REMARQUE : Pour plus de détails sur la procédure, voir Champeil et al.69.
- Obtenez du tissu musculaire à contraction rapide (~70 g des deux pattes arrière par animal) à partir d’Oryctolagus cuniculus de type sauvage.
REMARQUE : L’identification visuelle préalable des muscles à disséquer est requise70,71. - Faites macérer le muscle à contraction rapide dans un mélangeur en le suspendant en trois volumes de 100 mM de KCl, avec 1 min de marche et 1 min de repos glacé, répétez trois fois72.
- Retirer les débris tissulaires par centrifugation (1500 × g) à 4 °C pendant 5 min ; Une seconde centrifugation (4200 × g) est effectuée si nécessaire.
- Prélever le surnageant et l’homogénéiser à l’aide d’un broyeur à mouchoirs (10 coups à basse vitesse, c’est-à-dire 500-1 500 tr/min)72,73. Centrifuger l’homogénat (10 000 × g) à 4 °C pendant 15 min.
REMARQUE : Des précautions doivent être prises lors de l’homogénéisation, car l’utilisation d’une vitesse relativement élevée peut entraîner des dommages au système et à l’opérateur. - Centrifuger le surnageant (contenant le réticulum sarcoplasmique) (33 000 × g) à 4 °C pendant 120 min.
- Mettre les pastilles en suspension dans 40 mL de 0,5 M de saccharose et centrifuger (12 000 × g) à 4 °C pendant 15 min. Diluer le surnageant (0,6 M de KCl et 0,15 M de saccharose) et centrifuger la suspension (34 000 × g) à 4 °C pendant 165 min.
- Mettre en suspension la pastille contenant les feuilles de vésicules du réticulum sarcoplasmique (SRV) dans 0,3 M de saccharose, 0,1 M de KCl et 5 mM de Tris-HCl, pH 7,0. Déterminez la concentration en protéines en utilisant le test de Lowry74 et l’albumine sérique humaine comme étalon protéique.
- Aliquote les SRV (30-32 mg/mL) dans un volume de 1 mL. Stockez les échantillons SRV à -72 °C pour un traitement ultérieur.
REMARQUE : Le rendement en protéines est de 100 à 110 mg de SRV par animal.
2. Purification SERCA (Durée, 18 - 20 h)
REMARQUE : Pour plus de détails sur la procédure, veuillez consulter Rivera-Morán et al.46.
- Diluer la suspension de SRV à 2 mg de protéines/mL en utilisant 75 mM de Tris-HCl, pH 7,2, 0,6 M de KCl, 6 mM d’EDTA, 1 mM d’EGTA et 0,1 % (p/v) de désoxycholate (COD). Incuber la suspension SRV sur de la glace pendant 10 min et agiter doucement. Centrifuger la suspension SRV (100 000 × g) à 4 °C pendant 60 min.
REMARQUE : Le DOC doit être ajouté goutte à goutte. - Mettre les pastilles en suspension dans <3 mL de 25 mM de Tris-HCl, pH 7,5, 0,3 M de KCl, 45 % de glycérol (v/v) et 2 mM d’EDTA. Homogénéiser la suspension et l’ajuster à un volume final de 10 mL. Déterminez la concentration en protéines, puis ajustez-la à 6,5 mg/mL. Ajouter de l’azolectine à 5 mg/mL et 0,85 (p/p) de Zwittergent 3-14.
REMARQUE : L’azolectine et le Zwittergent 3-14 doivent être ajoutés goutte à goutte. - Homogénéiser la suspension et centrifuger (100 000 × g) à 4 °C pendant 60 min. Prélever le surnageant et diluer 1:2 avec 2 mM d’EGTA (pH 7,2).
- Verser délicatement la suspension sur un gradient discontinu de concentration de tréhalose (45, 40, 35 et 30 % p/v) dans 10 mM de Tris-HCl, pH 7,0, 1 mM d’EDTA, 0,1 % de désoxycholate et 1 mg/mL d’azolectine. Centrifugeuse (100 000 × g) à 4 °C pendant 14 h. Récupérez la pastille transparente, légèrement jaunâtre, formée au fond des tubes.
REMARQUE : La formation du gradient de tréhalose et l’ajout de la suspension de l’échantillon doivent être effectués à l’aide de techniques appropriées. Une centrifugation de nuit est recommandée. - Suspendre doucement la pastille dans un petit volume (<1,5 mL) de 25 mM de Tris-HCl, pH 7,5, 0,3 de M KCl, 45 % de glycérol et 2 mM d’EDTA. Déterminer la concentration en protéines. Ajuster la concentration en protéines par dilution à 2 mg/mL. Prélever des aliquotes (~50 μL) de la suspension et stocker le SERCA purifié à -72 °C jusqu’à son utilisation.
- Réaliser la fiche de données de sécurité du SERCA72 purifié, et des échantillons de protéines prélevés aux différentes étapes de la purification. Teignez le gel avec Coomassie Blue72.
REMARQUE : L’utilisation de 0,6 M de tréhalose au lieu de glycérol dans le tampon de suspension augmente la stabilité de l’enzyme pendant le stockage et les cycles de congélation-décongélation28,29.
3. Dosage de l’activité ATPase (Timing, 1 - 2 h)
- Dosage enzymatique : ATPase-Piruvate kinase-lactate déshydrogénase
- Préparez le mélange réactionnel (1 mL) : 50 mM MOPS (pH 7,0), 1 mM EGTA, 80 mM KCl, 5 mM MgCl2, 3 mM CaCl2, 5 mM de phosphoénolpyruvate, 250 μM de NADH. Inclure des concentrations variables d’ATP (0,001 à 0,25 mM).
- Homogénéiser soigneusement le test de réaction par vortex. Incuber le test de réaction à 37 °C pendant 10 min. Ajouter 0,9 U de L-lactate déshydrogénase (LDH) et 1,5 U de pyruvate kinase (PK).
- Initier la réaction ATPase par l’ajout de 10 μg de SERCA.
- Déterminer la formation du NADH. Suivez l’évolution de l’intensité de l’absorbance toutes les secondes pendant 600 s à une longueur d’onde (λ) de 340 nm au fil du temps à l’aide d’un spectrophotomètre muni d’un support de cellule thermostaté.
REMARQUE : L’intervalle de temps pour la mesure de l’absorbance peut varier, mais essayez d’enregistrer autant de points de données que possible pour tracer la ligne droite initiale de la réaction ATPase.
- Déterminer le taux d’hydrolyse de l’ATP
REMARQUE : Pour plus de détails sur la procédure, veuillez consulter Rivera-Morán et al.46.- Calculez la valeur de la pente (Δabs/min) de la portion linéaire dans chaque courbe formée. Utilisez le coefficient d’extinction molaire (ε) du NADH (λ = 6 220 M−1·cm−1) pour déterminer l’activité de l’ATPase (μmoles ATP hydrolysées/min.mg prot.)
- Déterminer les paramètres cinétiques K,m et Vmax.
- Tracez les données de vitesse en fonction de la concentration d’ATP à l’aide d’un logiciel d’analyse de données et de graphiques.
- Ajustez les données par régression non linéaire à l’équation de Michaelis-Menten (équation 1)46 :
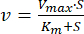
où v est la vitesse, Vmax est la vitesse maximale, [S] est la concentration d’ATP et Km est la constante de Michaelis-Menten. - Déterminez K,m et Vmax par itération.
REMARQUE : L’inclusion de DOC peut augmenter la réponse du taux d’ATPase ; il est recommandé de tester différentes concentrations de COD pour trouver l’optimum.
4. Test de marquage de SERCA avec de l’isothiocyanate de fluorescéine (FITC) (Timing, 30 - 45 min, uniquement pour le marquage FITC)
REMARQUE : Pour plus de détails sur la procédure, reportez-vous à 54,75,76.
- Mettre en suspension SERCA (20 μg) dans 50 μL (volume final) de tampon d’étiquetage (100 mM de KCl, 5 mM de MgCl2 et 30 mM de Tris-HCl, pH 8,9) contenant 1 mM de FITC. Mélanger par vortex.
- Incuber les échantillons à différents moments (15 min, 10 min, 7 min, 5 min et 2 min) dans l’obscurité et à température ambiante.
- Arrêtez la réaction de marquage en ajoutant 1 volume de tampon d’arrêt glacé (480 mM de saccharose et 48 mM de MOPS, pH 7,0), contenant de l’ATP (5 mM). Incuber sur glace pendant 5 min dans l’obscurité.
- Soumettre le SERCA labellisé FITC à la FDS-PAGE72. Exposez les gels transparents à la lumière UV (λ = 302 nm). Documentez le résultat avec une photo.
- Teindre le gel avec du bleu Coomassie. Documentez le résultat avec une photo.
REMARQUE : L’exécution du marquage FITC dans une pièce sombre peut améliorer la qualité du signal de fluorescence.
5. Spectres de dichroïsme circulaire (CD) (Timing, 30 min)
REMARQUE : Pour plus de détails sur la procédure, reportez-vous à11,77.
- Suspendre SERCA (3 μM) dans 400 μL de tampon phosphate 10 mM (pH 7,0) à 25 °C.
- Chargez l’échantillon dans une cellule de 0,1 cm de longueur de trajet. Réglez la plage λ des spectres UV lointains entre 190 et 260 nm. Réglez la résolution interne et la bande passante sur 1 nm.
- Enregistrez le signal CD à 50 nm/min à 25 °C.
Résultats
FDS-PAGE de SERCA à différents stades de purification (Figure 1). Le gel coloré en bleu de Coomassie montre l’enrichissement de la bande protéique SERCA (poids moléculaire apparent >100 kDa) au fur et à mesure de la progression du protocole de purification. La bande protéique correspondant à SERCA présente une pureté de >90 % après centrifugation sur un gradient de concentration de tréhalose. Graphique du taux d’hydrolyse de l’ATP en f...
Discussion
La plupart des molécules et des ions ne peuvent pas traverser librement les membranes cellulaires, par exemple, le proton (H+) nécessite un transporteur membranaire dans la membrane plasmique d’une variété d’organismes et d’organites tels que les mitochondries83,84. Les membranes cellulaires sont sélectives, et les molécules et les ions qui traversent les membranes cellulaires sont diversifiés, de sorte qu...
Déclarations de divulgation
L’auteur déclare qu’il n’a pas d’intérêts financiers concurrents.
Remerciements
L’auteur remercie Edmundo Mata-Morales pour l’aide qu’il a apportée au montage de la vidéo, à VM, à Valentín de la Cruz-Torres pour la purification des SRV, à Miguel A. Rivera-Moran pour la purification et l’analyse du SERCA, et à Juan C. Gonzalez-Castro, Franco E. Juarez, Alejandra Nevarez, Nicolas Rocha-Vizuet et Jocelin I. Ramírez-Alonso pour la production vidéo. Aucun financement, subvention ou autre soutien n’a été reçu pour mener cette étude.
matériels
| Name | Company | Catalog Number | Comments |
| ATP | Sigma-Aldrich Corp | A2383 | |
| Azolectin from soybean | Sigma-Aldrich Corp | 44924 | |
| Benchtop UV transilluminator | Cole-Parmer | EW-97623-08 | Dual intensity High setting is ideal for analytical documentation. Low setting reduces photonicking or photobleaching of gel samples while doing preparative work. |
| CaCl2 • 2H2O | Sigma-Aldrich Corp | 223506 | |
| Coolpix B500 camera | Nikon Corp | S210 | |
| Coomassie brilliant blue G-250 | Bio-Rad | 1610406 | |
| Dodecyl maltoside | Sigma-Aldrich Corp | D4641 | |
| Ethylene glycol-bis(2-aminoethylether)-N,N,N′,N′-tetraacetic acid | Sigma-Aldrich Corp | E4378 | |
| Ethylenediaminetetraacetic acid tetrasodium salt dihydrate | Sigma-Aldrich Corp | E6511 | |
| Fluorescein isothiocyanate | Sigma-Aldrich Corp | F3651 | |
| KCl | JT Baker | 7447-40-7 | |
| MgCl2 • 6H2O | Sigma-Aldrich Corp | M9272 | |
| MOPS | Sigma-Aldrich Corp | M1254 | |
| NADH | Chem-Impex International Inc | 230 | |
| N-tetradecyl-N,N- dimethyl-3-ammonium-1-propanesulfonate | Sigma-Aldrich Corp | D0431 | |
| Phosphoenolpyruvate (PEP) | Chem-Impex International Inc | 9711 | |
| Rabbit muscle lactate dehydrogenase | Roche | 10003557103 | |
| Rabbit muscle pyruvate kinase | Sigma-Aldrich Corp | P1506 | |
| Sodium deoxycholate | Sigma-Aldrich Corp | D6750 | |
| Sodium dodecyl sulfate | Bio-Rad | 1610302 | |
| Spectropolarimeter | Jasco Corp. | Jasco J1500 | |
| Sucrose | Sigma-Aldrich Corp | 84100 | |
| Trehalose | Sigma-Aldrich Corp | T0167 | Dihydrate |
| Tris(hydroxymethyl)aminomethane hydrochloride | Sigma-Aldrich Corp | 857645 | |
| Ultracentrifuge | Beckman | Optima XPN | |
| UV/VIS spectrophotometer | Agilent Technologies | 8453 | The Agilent 8453 UV-Vis Spectrophotometer uses a photodiode array for simultaneous measurement of the complete ultra-violet to visible light spectrum |
| WiseStir HS- 30E | Daihan Scientific Co. | DH.WOS01010 | Ideal for all disperging and homogenizing applications, designed for tissue grinders. |
Références
- Schuberth, C., Wedlich-Söldner, R. Building a patchwork - The yeast plasma membrane as model to study lateral domain formation. Biochim Biophys Acta Mol Cell Res. 1853 (4), 767-774 (2015).
- Douglas, L. M., Konopka, J. B. Fungal membrane organization: The eisosome concept. Annu Rev Microbiol. 68 (1), 377-393 (2014).
- Borrell, J. H., Domènech, &. #. 2. 1. 0. ;., Keough, K. M. W. . Membrane protein - Lipid interactions: Physics and Chemistry in the Bilayer. , (2016).
- Moraes, I., Evans, G., Sanchez-Weatherby, J., Newstead, S., Stewart, P. D. . The next generation in membrane protein structure determination. , (2016).
- White, S. H. Biophysical dissection of membrane proteins. Nature. 459 (7245), 344-346 (2009).
- Lacapère, J. -. J. Membrane protein structure determination. Methods Mol Biol. , 459 (1002).
- Palmgren, M. G. Plant plasma membrane H+-ATPases: Powerhouses for nutrient uptake. Annu Rev Plant Physiol Plant Mol Biol. 52 (1), 817-845 (2001).
- Yatime, L., et al. P-type ATPases as drug targets: Tools for medicine and science. Biochim Biophys Acta Bioenerg. 1787 (4), 207-220 (2009).
- Drews, J. Tetrahydrobiopterin biosynthesis as an off-target of sulfa drugs. Science. 287 (5460), 1960-1964 (2000).
- Xu, Z., Meshcheryakov, V. A., Poce, G., Chng, S. -. S. MmpL3 is the flippase for mycolic acids in mycobacteria. Proc Natl Acad Sci USA. 114 (30), 7993-7998 (2017).
- Siligardi, G., Hussain, R., Patching, S. G., Phillips-Jones, M. K. Ligand- and drug-binding studies of membrane proteins revealed through circular dichroism spectroscopy. Biochim Biophys Acta Biomembr. 1838 (1 PARTA), 34-42 (2014).
- Rawlings, A. E. Membrane proteins: Always an insoluble problem. Biochem Soc Trans. 44 (3), 790-795 (2016).
- Popot, J. -. L. . Membrane proteins in aqueous solutions. , 708 (2018).
- Palmgren, M. G., Sommarin, M., Ulvskov, P., Larsson, C. Effect of detergents on the H+-ATPase activity of inside-out and right-side-out plant plasma membrane vesicles. Biochim Biophys Acta Biomembr. 1021 (2), 133-140 (1990).
- McBride, Z., Chen, D., Reick, C., Xie, J., Szymanski, D. B. Global analysis of membrane-associated protein oligomerization using protein correlation profiling. Mol Cell Proteomics. 16 (11), 1972-1989 (2017).
- Yoneda, J. S., et al. Multimeric species in equilibrium in detergent-solubilized Na, K-ATPase. Int J Biol Macromol. 89, 238-245 (2016).
- Ohlendieck, K. Extraction of membrane proteins. Protein Purif Protocols. 59, 283-294 (2004).
- Champeil, P., et al. A robust method to screen detergents for membrane protein stabilization, revisited. Anal Biochem. 511, 31-35 (2016).
- Gimpl, K., Klement, J., Keller, S. Characterising protein/detergent complexes by triple-detection size-exclusion chromatography. Biol Proced Online. 18, 4 (2016).
- le Maire, M., et al. Gel chromatography and analytical ultracentrifugation to determine the extent of detergent binding and aggregation, and Stokes radius of membrane proteins using sarcoplasmic reticulum Ca2+-ATPase as an example. Nat Protoc. 3 (11), 1782-1795 (2008).
- Du, M., et al. Progress, applications, challenges and prospects of protein purification technology. Front Bioeng Biotechnol. 10, 1-26 (2022).
- Slotboom, D. J., Duurkens, R. H., Olieman, K., Erkens, G. B. Static light scattering to characterize membrane proteins in detergent solution. Methods. 46 (2), 73-82 (2008).
- Harding, S. E. Analytical ultracentrifugation as a matrix-free probe for the study of kinase related cellular and bacterial membrane proteins and glycans. Molecules. 26, 6080 (2021).
- Chen, Y. -. C., et al. Thermal stability, storage and release of proteins with tailored fit in silica. Sci Rep. 7 (1), 46568 (2017).
- Montigny, C. C., Arnou, B., Marchal, E., Champeil, P. Use of glycerol-containing media to study the intrinsic fluorescence properties of detergent-solubilized native or expressed SERCA1a. Biochemistry. 47 (46), 12159-12174 (2008).
- Simongini, M., Puglisi, A., Genovese, F., Hochkoeppler, A. Trehalose counteracts the dissociation of tetrameric rabbit lactate dehydrogenase induced by acidic pH conditions. Arch Biochem Biophys. 740, 109584 (2023).
- Shivanna, B. D., Rowe, E. S. Preservation of the native structure and function of Ca2+-ATPase from sarcoplasmic reticulum: Solubilization and reconstitution by new short-chain phospholipid detergent 1,2-diheptanoyl-sn-phosphatidylcholine. Biochem J. 325 (2), 533-542 (1997).
- Sampedro, J. G., Guerra, G., Pardo, J. P., Uribe, S. Trehalose-mediated protection of the plasma membrane H+-ATPase from Kluyveromyces lactis during freeze-drying and rehydration. Cryobiology. 37 (2), 131-138 (1998).
- Sampedro, J. G., Cortés, P., Muñoz-Clares, R. A., Fernández, A., Uribe, S. Thermal inactivation of the plasma membrane H+-ATPase from Kluyveromyces lactis. Protection by Trehalose. Biochim Biophys Acta Proteins Proteomics. 1544 (1-2), 64-73 (2001).
- Sampedro, J. G., et al. Fluorescence quenching by nucleotides of the plasma membrane H+-ATPase from Kluyveromyces lactis. Biochemistry. 46 (18), 5616-5622 (2007).
- Ruiz-Granados, Y. G., De La Cruz-Torres, V., Sampedro, J. G. The oligomeric state of the plasma membrane H+-ATPase from Kluyveromyces lactis. Molecules. 24 (5), 958 (2019).
- Zhao, P., Zhao, C., Chen, D., Yun, C., Li, H., Bai, L. Structure and activation mechanism of the hexameric plasma membrane H+-ATPase. Nat Commun. 12 (1), 6439 (2021).
- Auer, M., Scarborough, G. A., Kühlbrandt, W. Surface crystallization of the plasma membrane H+-ATPase on a carbon support film for electron crystallography. J Mol Biol. 287 (5), 961-968 (1999).
- Hennessey, J. P., Scarborough, G. A. Secondary structure of the Neurospora CRASSA plasma membrane H+-ATPase as estimated by circular dichroism. J Biol Chem. 263 (7), 3123-3130 (1988).
- Scarborough, G. A. Crystallization, structure and dynamics of the proton-translocating P-type ATPase. J Exp Biol. 203 (Pt 1), 147-154 (2000).
- Sumida, K. H., et al. Improving protein expression, stability, and function with proteinMPNN. J Am Chem Soc. 146 (3), 2054-2061 (2024).
- Eleutherio, E., Panek, A., De Mesquita, J. F., Trevisol, E., Magalhães, R. Revisiting yeast trehalose metabolism. Curr Genet. 61 (3), 263-274 (2015).
- Attfield, P. V. Trehalose accumulates in Saccharomyces cerevisiae during exposure to agents that induce heat shock response. FEBS Lett. 225 (1-2), 259-263 (1987).
- Marunde, M. R., et al. Improved tolerance to salt and water stress in Drosophila melanogaster cells conferred by late embryogenesis abundant protein. J Insect Physiol. 59 (4), 377-386 (2013).
- Crowe, J. H. Anhydrobiosis: An unsolved problem with applications in human welfare. Subcell Biochem. 71, 263-280 (2015).
- Crowe, J. H., Crowe, L. M., Chapman, D. Preservation of membranes in anhydrobiotic organisms: The role of trehalose. Science. 223 (4637), 701-703 (1984).
- Sampedro, J. G., Rivera-Moran, M. A., Uribe-Carvajal, S. Kramers' theory and the dependence of enzyme dynamics on trehalose-mediated viscosity. Catalysts. 10 (6), 1-19 (2020).
- Sampedro, J. G., Uribe, S. Trehalose-enzyme interactions result in structure stabilization and activity inhibition. The role of viscosity. Mol Cell Biochem. 256 - 257 (1-2), 319-327 (2004).
- Crowe, J. H. Trehalose as a "chemical chaperone": Fact and fantasy. Adv Exp Med Biol. 594, 143-158 (2007).
- Crowe, J. H., Carpenter, J. F., Crowe, L. M., Anchordoguy, T. J. Are freezing and dehydration similar stress vectors? A comparison of modes of interaction of stabilizing solutes with biomolecules. Cryobiology. 27 (3), 219-231 (1990).
- Rivera-Morán, M. A., Sampedro, J. G. Isolation of the sarcoplasmic reticulum Ca2+-ATPase from rabbit fast-twitch muscle. Methods Protoc. 6 (5), 102 (2023).
- Páez-Pérez, E. D., De La Cruz-Torres, V., Sampedro, J. G. Nucleotide binding in an engineered recombinant Ca2+-ATPase N-domain. Biochemistry. 55 (49), 6751-6765 (2016).
- Ramírez-Alonso, J. I., Sampedro, J. G. Effect of cations on ATP binding to the N-domain of Na+, K+-ATPase. J Fluoresc. , (2024).
- De la Cruz-Torres, V., Cataño, V., Olivo-Rodríguez, M., Sampedro, J. G. ANS Interacts with the Ca2+-ATPase nucleotide binding site. J Fluoresc. 30 (3), 483-496 (2020).
- Sampedro, J. G., Nájera, H., Uribe-Carvajal, S., Ruiz-Granados, Y. G. Mapping the ATP binding site in the plasma membrane H+-ATPase from Kluyveromyces lactis. J Fluoresc. 24 (6), 1849-1859 (2014).
- Kiani, A. K., et al. Ethical considerations regarding animal experimentation. J Prev Med Hyg. 63 (2 Suppl 3), E255-E266 (2022).
- Ruiz-Granados, Y., De La Cruz-Torres, V., Sampedro, J. The oligomeric state of the plasma membrane H+-ATPase from Kluyveromyces lactis. Molecules. 24 (5), 958 (2019).
- Wang, S., et al. Structural basis for sarcolipin's regulation of muscle thermogenesis by the sarcoplasmic reticulum Ca2+-ATPase. Sci Adv. 7 (48), eabi7154 (2021).
- Champeil, P., et al. ATP regulation of sarcoplasmic reticulum Ca2+-ATPase. Metal-free ATP and 8-bromo-ATP bind with high affinity to the catalytic site of phosphorylated ATPase and accelerate dephosphorylation. J Biol Chem. 263 (25), 12288-12294 (1988).
- Møller, J. V., Olesen, C. Preparation of Ca(2+)-ATPase1a enzyme from rabbit sarcoplasmic reticulum. Methods Mol Biol. 1377, 11-17 (2016).
- Wuytack, F., De Schutter, G., Casteels, R. Partial purification of (Ca2+ + Mg2+)-dependent ATPase from pig smooth muscle and reconstitution of an ATP-dependent Ca2+-transport system. Biochem J. 198 (2), 265-271 (1981).
- Mandal, A., et al. Solubilization, purification and reconstitution of Ca2+-ATPase from bovine pulmonary artery smooth muscle microsomes by different detergents: Preservation of native structure and function of the enzyme by DHPC. Biochim Biophys Acta Gen Subj. 1760 (1), 20-31 (2006).
- Inesi, G., Ma, H., Hua, S., Toyoshima, C. Characterization of Ca2+ ATPase residues involved in substrate and cation binding. Ann NY Acad Sci. 986 (1), 63-71 (2003).
- Van Winkle, W. B., Pitts, B. J., Entman, M. L. Rapid purification of canine cardiac sarcoplasmic reticulum Ca2+-ATPase. J Biol Chem. 253 (24), 8671-8673 (1978).
- Ruiz-Granados, Y. G., De La Cruz-Torres, V., Sampedro, J. G. The oligomeric state of the plasma membrane H+-ATPase from Kluyveromyces lactis. Molecules. 24 (5), 958 (2019).
- Teramoto, N., Sachinvala, N. D., Shibata, M. Trehalose and trehalose-based polymers for environmentally benign, biocompatible, and bioactive materials. Molecules. 13 (8), 1773-1816 (2008).
- Dao, T. T., et al. Demethoxycurcumin is a potent inhibitor of P-type ATPases from diverse kingdoms of life. PLOS ONE. 11 (9), e0163260 (2016).
- Bleeker, N. P., Cornea, R. L., Thomas, D. D., Xing, C. A novel SERCA inhibitor demonstrates synergy with classic SERCA inhibitors and targets multidrug-resistant AML. Mol Pharmaceutics. 10 (11), 4358-4366 (2013).
- Michelangeli, F., East, J. M. A diversity of SERCA Ca2+ pump inhibitors. Biochem Soc Trans. 39 (3), 789-797 (2011).
- Tadini-Buoninsegni, F., Smeazzetto, S., Gualdani, R., Moncelli, M. R. Drug interactions with the Ca2+-ATPase from Sarco(Endo)Plasmic Reticulum (SERCA). Front Mol Biosci. 5, 36 (2018).
- Clausen, J. D., et al. Elucidation of antimicrobial activity and mechanism of action by N-substituted carbazole derivatives. Bioorg Med Chem Lett. 27 (19), 4564-4570 (2017).
- Leary, S., et al. . AVMA guidelines for the euthanasia of animals: 2020 Edition, Members of the Panel on Euthanasia AVMA Staff Consultants. , (2020).
- de Aluja, A. S. Laboratory animals and official Mexican norms (NOM-062-ZOO-1999). Gac Med Mex. 138 (3), 295-298 (2002).
- Champeil, P., Buschlen-Boucly, S., Bastide, F., Gary-Bobo, C. Sarcoplasmic reticulum ATPase. Spin labeling detection of ligand-induced changes in the relative reactivities of certain sulfhydryl groups. J Biol Chem. 253 (4), 1179-1186 (1978).
- Skalec, A., Janeczek, M., Czerski, A. Anatomy and histology of the rabbit common calcanean tendon. J Vet Med Series C: Anat Histol Embryol. 48 (5), 466-475 (2019).
- Janes, L. E., Mioton, L. M., Fracol, M. E., Ko, J. H. An in vivo comparison: Novel mesh suture versus traditional suture-based repair in a rabbit tendon model. J Hand Surg Glob Online. 4 (1), 32-39 (2022).
- Bollag, D. M., Rozycki, M. D., Edelstein, S. J. . Protein Methods. , (1996).
- Gagné, F. Tissue preparation and subcellular fractionation techniques. Biochem Ecotoxicol. , 21-31 (2014).
- Lowry, O. H., Rosebrough, N. J., Farr, A. L., Randall, R. J. Protein measurement with the Folin phenol reagent. J Biol Chem. 193 (1), 265-275 (1951).
- Autry, J. M., Rubin, J. E., Svensson, B., Li, J. L., Thomas, D. D. Nucleotide activation of the Ca-ATPase. J Biol Chem. 287 (46), 39070-39082 (2012).
- Winters, D. L., Autry, J. M., Svensson, B., Thomas, D. D. Interdomain fluorescence resonance energy transfer in SERCA probed by cyan-fluorescent protein fused to the actuator domain. Biochemistry. 47 (14), 4246-4256 (2008).
- Miles, A. J., Wallace, B. A. Circular dichroism spectroscopy of membrane proteins. Chem Soc Rev. 14 (45), 1130-1135 (2016).
- Scofano, H. M., Vieyra, A., de Meis, L. Substrate regulation of the sarcoplasmic reticulum ATPase. Transient kinetic studies. J Biol Chem. 254 (20), 10227-10231 (1979).
- Yamamoto, T., Tonomura, Y. Reaction mechanism of the Ca++-dependent ATPase of sarcoplasmic reticulum from skeletal muscle: I. Kinetic studies. J Biochem. 62 (5), 558-575 (1967).
- Murphy, A. J. Affinity labeling of the active site of the Ca2+-ATPase of sarcoplasmic reticulum. Biochim Biophys Acta - Biomembr. 946 (1), 57-65 (1988).
- Csermely, P., Katopis, C., Wallace, B. A., Martonosi, A. The E1→E2 transition of Ca2+-transporting ATPase in sarcoplasmic reticulum occurs without major changes in secondary structure. A circular-dichroism study. Biochem J. 241 (3), 663-669 (1987).
- Abràmoff, M. D., Magalhães, P. J., Ram, S. J. Image processing with ImageJ. Biophotonics Int. 11 (7), 36-42 (2004).
- Scarborough, G. A. The plasma membrane proton-translocating ATPase. Cell Mol Life Sci. 57 (6), 871-883 (2000).
- Llopis, J., McCaffery, J. M., Miyawaki, A., Farquhar, M. G., Tsien, R. Y. Measurement of cytosolic, mitochondrial, and Golgi pH in single living cells with green fluorescent proteins. Proc Natl Acad Sci. 95 (12), 6803-6808 (1998).
- Kanai, R., Vilsen, B., Cornelius, F., Toyoshima, C. Crystal structures of Na+, K+-ATPase reveal the mechanism that converts the K+-bound form to Na+-bound form and opens and closes the cytoplasmic gate. FEBS Lett. 597 (15), 1957-1976 (2023).
- Palmgren, M. P-type ATPases: Many more enigmas left to solve. J Biol Chem. 299 (11), 105352 (2023).
- Frauenfelder, H. Ask not what physics can do for biology-Ask what biology can do for physics. Phys Biol. 11 (5), 053004 (2014).
- Frauenfelder, H. The physics of proteins. Annu Rev Biophys Biomol Struct. 36, (2010).
- Narhi, L. O. . Biophysics for Therapeutic Protein Development. , (2013).
- East, J. M. Purification of a membrane protein (Ca2+/Mg2+-ATPase) and its reconstitution into lipid vesicles. Methods Mol Biol. 27, 87-94 (1994).
- Lee, E. -. H., et al. Enhancement of enzyme activity and stability by poly(γ-glutamic acid). Polymer J. 42 (10), 818-822 (2010).
- Norrild, R. K., et al. Increasing protein stability by inferring substitution effects from high-throughput experiments. Cell Rep Methods. 2 (11), 100333 (2022).
- Song, Z., Zhang, Q., Wu, W., Pu, Z., Yu, H. Rational design of enzyme activity and enantioselectivity. Front Bioeng Biotechnol. 11, 1-14 (2023).
- Jidenko, M., Lenoir, G., Fuentes, J. M., le Maire, M., Jaxel, C. Expression in yeast and purification of a membrane protein, SERCA1a, using a biotinylated acceptor domain. Protein Expr Purif. 48 (1), 32-42 (2006).
- Hua, S., Ma, H., Lewis, D., Inesi, G., Toyoshima, C. Functional role of "N" (nucleotide) and "P" (phosphorylation) domain interactions in the sarcoplasmic reticulum (SERCA) ATPase. Biochemistry. 41 (7), 2264-2272 (2002).
- Pang, Y. -. H., Chen, J. -. W. Anisodamine causes the changes of structure and function in the transmembrane domain of the Ca2+-ATPase from sarcoplasmic reticulum. Biosci Biotechnol Biochem. 68 (1), 126-131 (2004).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationThis article has been published
Video Coming Soon