A subscription to JoVE is required to view this content. Sign in or start your free trial.
Method Article
تنقية الشبكة الإندوبلازمية ساركو Ca2 + -ATPase من عضلة الأرانب
In This Article
Summary
يصف هذا البروتوكول طريقة تنقية SERCA المحسنة ، والتي تتضمن ثلاثي السكاريد في خطوة الطرد المركزي النهائية. تعمل هذه الكربوهيدرات على استقرار البروتينات في ظل ظروف قاسية. كانت SERCA المنقاة نشطة تحفيزيا وأظهرت نقاء عاليا ، مما يجعلها مناسبة للدراسات الهيكلية والوظيفية.
Abstract
بعض ATPases من النوع P ، مثل sarco / الشبكة الإندوبلازمية Ca2 + -ATPase (SERCA) ، هي بطبيعتها بروتينات غشائية قابلة للتغيير تتطلب ظروفا فيزيائية وكيميائية محددة أثناء التنقية للحصول عليها بنقاء عال وجودة هيكلية وفي شكل نشط تحفيزيا. ثلاثياز ثنائي السكاريد هو مذاب متوافق يتم تصنيعه وتراكمه بتركيزات عالية في سيتوبلازم الخميرة لتثبيت الأغشية والبروتينات. ينتج عن استخدام تريهالوز كمادة مضافة في بروتوكول تنقية غشاء البلازما H + -ATPase مستحضر عالي الجودة ، يظهر هيكله السداسي بالطرق التحليلية البيوكيميائية. لذلك ، يمكن استخدام Trehalose كمادة مضافة مثبتة لتنقية بروتينات الغشاء (P-ATPases). يصف هذا البروتوكول تعديل البروتوكول الكلاسيكي لتنقية SERCA عن طريق إخضاع SERCA للطرد المركزي على تدرج تركيز تريهالوز. أدى إدراج هذه الكربوهيدرات إلى تنقية SERCA في شكل نشط تحفيزيا بنقاوة عالية ، والأهم من ذلك ، في شكل مستقر. أظهر التوصيف الكيميائي الحيوي الجزئي ل SERCA المنقى (SDS-Page ، حركية الإنزيم ، وضع العلامات FITC ، التحليل الطيفي الدائري لثنائي اللون) أن الإنزيم مناسب للدراسات الوظيفية والهيكلية. يقترح استخدام تريهالوز في بروتوكول تنقية ATPases من النوع P وبروتينات الغشاء المتقلب الأخرى (والخلوية).
Introduction
تعتبر بروتينات / إنزيمات الغشاء مكونات بيولوجية أساسية للخلايا لأنها تلعب أدوارا حاسمة في العمليات المختلفة1،2،3. قد تشمل بعض الوظائف نقل الأيونات والجزيئات داخل وخارج الخلية / المقصورات الداخلية (إما نشطة / سلبية) ، والتعرف على الخلايا الخلوية ، والارتباط بين الخلايا ، والتثبيت / التعلق ، واستشعار البيئة الخارجية من خلال التكامل مع آلية نقل الإشارة في ظل الظروف الفيزيائية والكيميائية العادية والقاسية (الملح العالي ، الماء المنخفض ، درجة الحرارة العالية ، مقاومة الأدوية ، إلخ)3 لذلك ، أصبح تحديد الهيكل ثلاثي الأبعاد (3D) لبروتينات الغشاء و / أو الإنزيمات ذا أهمية كبيرة لكل من البحث الأساسي والتطبيقي4،7،6. الأهم من ذلك ، تم استخدام بروتينات / إنزيمات الغشاء على نطاق واسع كأهداف لاكتشاف الأدوية (سواء كانت طبيعية أو حسب التصميم)7،8،9. أي أن بروتينات الغشاء لها أهمية متأصلة في الصحة9،10،11.
الطابع الكارهة للماء للبروتينات / الإنزيمات الغشائية هو الخاصية الفيزيائية والكيميائية الأكثر تحديا من الناحية الفنية للمختبر التجريبي12،13،14 ، وأكثر من ذلك عند العمل مع بروتينات الغشاء المتكاملة قليلة القلة و / أو شديدة القابليةللتغيير 15،16. من المستحسن للغاية عزل الكميات المناسبة من بروتين / إنزيم الغشاء بأعلى جودة ممكنة للمقايسات التجريبية الوظيفية والدراسات الهيكلية17. من الصعب جدا تنقية بروتينات الغشاء ، كونها بطبيعتها كارهة للماء ، وعادة ما يكون اختيار المنظف أحد أهم القضايا التي يجب مراعاتها17،18،19،20. في هذا الصدد ، عادة ما تسبب الطرق المعملية المستخدمة لعزل بروتينات الغشاء درجة معينة من الضرر للترتيب الهيكلي ثلاثي الأبعاد للبروتين21. تتضمن بعض هذه الطرق استخدام (أ) صوتنة (موجات فوق صوتية عالية التردد / الطاقة) ، (ب) المنظفات الذائبة للبروتين (إما قاسية أو متوسطة أو لطيفة) 22 ، (ج) ضغوط عالية نسبيا في كروماتوغرافيا العمود والطرد المركزي الفائق عاليالسرعة 23 ، (د) الجزيئات المترسبة ، (ه) الإنزيمات الهاضمة ، وغيرها21. يمكن أن تساهم كل هذه العمليات في أو تكون السبب الرئيسي لزعزعة استقرار البروتين أثناء التنقية21. في هذا الصدد ، يبدو أن بعض البروتوكولات تعمل بشكل جيد نسبيا مع بروتين غشاء معين. ومع ذلك ، فإن التحسين مرحب به دائما عند استخدام فحوصات أو طرق حديثة جديدة تتطلب جودة أعلى لتحضير البروتين الغشائي للحصول على نتائج مرضية24. قد تشمل خطوات التحسين ، على سبيل المثال لا الحصر ، تحسين تصميم البناء ، وإيجاد الظروف المثلى للتعبير عن البروتين الغشائي ، وإنشاء ظروف معالجة أفضل (على سبيل المثال ، الأس الهيدروجيني ، ودرجة الحرارة ، وما إلى ذلك) ، والعثور على أفضل منظف متوافق ، وضبط خطوات التنقية مثل وقت الصوتنة ، وسرعة الطرد المركزي ، وإعادة صياغة الحلول العازلة عن طريق إضافة عوامل التثبيت ، وما إلى ذلك.25،26،27. لذلك ، فإن أي تغيير في منهجية التنقية يؤدي إلى زيادة جودة (نقاء) ونشاط بروتين / إنزيم الغشاء المنقى مهم.
في عائلة ATPase من النوع P ، يبدو أن غشاء بلازما الخميرة H + -ATPase هو أحد أكثر الأعضاء قابليةللمرونة 28،29. يفقد H + -ATPase نشاط التحلل المائي ATP عند الجفاف (التجفيف بالتجميد) ، والصدمة الحرارية ، وما إلى ذلك.28،29. تم اختبار استخدام تريهالوز كمثبت للبروتين في عزل غشاء البلازما H + -ATPase من الخميرة K. lactis30،31 ؛ كان مستحضر H + -ATPase الذي تم الحصول عليه عالي النقاء ونشطا تحفيزيا. الأهم من ذلك ، أن هذا سمح بحل حالة القلة للإنزيم بالطرق الكيميائية الحيوية ، مما كشف أنه سداسي وتم تأكيده لاحقا بواسطة المجهر الإلكترونيبالتبريد 31،32،33،34،35. لذلك ، يبدو من المحتمل أن الترتيب الدقيق ثلاثي الأبعاد (3D) لبروتينات الغشاء يمكن أن يضيع في ظل ظروف قاسية نسبيا أثناء التنقية36. لوحظ حركية التعطيل ثنائية الطور ل H + -ATPase أثناء التعطيل بوساطة الحرارة29. في خلايا الخميرة ، كما هو الحال في العديد من الكائنات الحية الأخرى ، يتراكم تريهالوز ثنائي السكاريد بتركيزات عالية في ظل ظروف الإجهاد البيئي37،38،39. يحافظ Trehalose على سلامة الغشاء ووظيفة النقل عن طريق تثبيت البروتينات (المضمنة في الغشاء والخلايا على حد سواء) وأغشية الخلايا40،41. تمت دراسة آلية تثبيت التريهالوز على نطاق واسع من قبل عدة مجموعات ومختبرنا42،43،44،45. أظهرت التجارب التي أجريت على H +-ATPase والإنزيمات الأخرى أن التريهالوز هو أكثر مثبتات البروتين فعالية بين السكريات الأحادية والسكرياتثنائية السكاريد 28،29. أدى ذلك إلى إدراجه في بروتوكول تنقية H +-ATPase30. في الآونة الأخيرة ، تم استخدام التريهالوز أيضا في تنقية الشبكة الساركوبلازمية Ca2 + -ATPase (SERCA) من عضلة الأرانب سريعة الارتعاش مع نتائج جيدة في نقاء البروتين والنشاط46. لذلك ، يبدو أن التريهالوز مادة مضافة جيدة ومناسبة لتنقية ATPases من النوع P وربما البروتينات الغشائية والعصارة الخلوية الأخرى.
بالنسبة ل P-ATPases ، يعد وجود مجالات السيتوبلازمية الهيكلية ميزة تجريبية ، خاصة بالنسبة لدراسات تفاعل الركيزة / الترابط47 ؛ تمت دراسة ربط ATP في مجالات N المؤتلفة عاليةالنقاء 47،48،49 ، وبالتالي القضاء على الاعتبارات الفنية لتنقية إنزيمات الغشاء الكامل47،48،50 ، من بين أمور أخرى51. لسوء الحظ ، لا تزال بعض الدراسات الوظيفية (التحويل التحفيزي / الطاقة) والهيكلية (ترتيب الوحدات الفرعية والتفاعل مع البروتينات الأخرى) تتطلب P-ATPase52،53 بالكامل. في هذا الصدد ، تم تحقيق تنقية SERCA من قبل العديد من المجموعات البحثية54،55،56،57،58. ومع ذلك ، لا يزال من الممكن تنفيذ التحسينات46 ، على سبيل المثال ، زيادة سلامة ATPase59 المنقى ، وتجنب تمسخ البروتين / تعطيل مجمعات البروتين25 ، وزيادة الذوبان دون تمسخ البروتين الغشائي (أي تجنب تكوين المجاميع الجزيئية الكبيرة) 60 ، مما يؤدي إلى توافق أفضل مع المقايسات والطرق التحليلية النهائيةالأخرى 61 علاوة على ذلك ، نظرا لظهور الاستراتيجيات التجريبية الجديدة ، والمواد المضافة ، ومثبطات الإنزيم ، وما إلى ذلك في الأدبيات العلمية61،62،63،64،65،66 ، فإنها تحتاج أحيانا إلى الاختبار باستخدام P-ATPase بأكمله. يصف هذا العمل بروتوكول تنقية SERCA واستخدام تريهالوز كمادة مضافة لتحقيق الاستقرار في بنية البروتين ونشاط ATPase. أي ، بالإضافة إلى زيادة جودة الإنزيم (الهيكلي) ، يساعد تريهالوز على منع فقدان بنية الإنزيم ونشاطه أثناء عزل الإنزيم وتخزينه ، مما يساعد على حفظ المواد البيولوجية وبالتالي تقليل عدد تنقية الإنزيم.
Protocol
تم تنفيذ جميع الإجراءات الحيوانية وفقا للإرشادات الدولية والمحلية (NORMA Oficial Mexicana NOM-062-ZOO-1999) للتعامل مع في المختبرات التجريبية51،67،68. تم الحصول على الأنسجة العضلية من النوع البري Oryctolagus cuniculus من وحدة مناولة المحلية (INE/CITES/DGVS-ZOO-E0055-SLP-98)46. أجرى طبيب بيطري من ذوي الخبرة في إدارة المختبر تشريح العضلات الأولي ومعالجته. تفاصيل الكواشف والمعدات المستخدمة في هذه الدراسة مدرجة في جدول المواد.
1. عزل الشبكة الساركوبلازمية من عضلة الأرانب سريعة الارتعاش (التوقيت ، 7-9 ساعات)
ملاحظة: للحصول على تفاصيل حول الإجراء ، راجع Champeil et al.69.
- احصل على أنسجة عضلية سريعة الارتعاش (~ 70 جم من كلا الساقين الخلفيتين لكل) من النوع البري Oryctolagus cuniculus.
ملاحظة: مطلوب تحديد بصري مسبق للعضلات المراد تشريحها70،71. - نقع العضلة سريعة الارتعاش في الخلاط عن طريق تعليقها في ثلاثة أحجام من 100 ملي مولار من KCl ، مع 1 دقيقة تشغيل و 1 دقيقة في راحة مثلجة ، كرر ثلاث مرات72.
- قم بإزالة بقايا الأنسجة عن طريق الطرد المركزي (1500 × جم) عند 4 درجات مئوية لمدة 5 دقائق ؛ يتم إجراء الطرد المركزي الثاني (4200 × جم) إذا لزم الأمر.
- اجمع المادة الطافية وتجانسها باستخدام مطحنة الأنسجة (10 ضربات بسرعة منخفضة ، أي 500-1,500 دورة في الدقيقة)72،73. جهاز الطرد المركزي المتجانس (10,000 × جم) عند 4 درجات مئوية لمدة 15 دقيقة.
ملاحظة: يجب توخي الحذر عند التجانس ، لأن استخدام سرعة عالية نسبيا يمكن أن يؤدي إلى تلف النظام والمشغل. - جهاز الطرد المركزي المادة الطافية (التي تحتوي على الشبكة الساركوبلازمية) (33,000 × جم) عند 4 درجات مئوية لمدة 120 دقيقة.
- الكريات في 40 مل من 0.5 متر من السكروز والطرد المركزي (12,000 × جم) عند 4 درجات مئوية لمدة 15 دقيقة. قم بتخفيف المادة الطافية (0.6 متر من KCl و 0.15 م من السكروز) وقم بالطرد المركزي للتعليق (34,000 × جم) عند 4 درجات مئوية لمدة 165 دقيقة.
- الحبيبات التي تحتوي على الحويصلات الشبكية الساركوبلازمية (SRVs) في 0.3 متر من السكروز ، 0.1 متر من KCl ، و 5 ملي مولار من Tris-HCl ، درجة الحموضة 7.0. حدد تركيز البروتين باستخدام مقايسة لوري74 وألبومين المصل البشري كمعيار للبروتين.
- Aliquot SRVs (30-32 مجم / مل) في حجم 1 مل. قم بتخزين عينات SRVs عند -72 درجة مئوية لمزيد من المعالجة.
ملاحظة: إنتاجية البروتين هي 100-110 مجم SRVs لكل.
2. تنقية SERCA (التوقيت ، 18 - 20 ساعة)
ملاحظة: للحصول على تفاصيل حول الإجراء ، راجع Rivera-Morán et al.46.
- خفف معلق SRVs إلى 2 مجم بروتين / مل باستخدام 75 ملي مولار من Tris-HCl ، ودرجة الحموضة 7.2 ، و 0.6 متر من KCl ، و 6 ملي مولار من EDTA ، و 1 ملي من EGTA ، و 0.1٪ (وزن / حجم) ديوكسيكولات (DOC). احتضن تعليق SRVs على الجليد لمدة 10 دقائق وتحريك لطيف. جهاز طرد مركزي تعليق SRVs (100,000 × ز) عند 4 درجة مئوية لمدة 60 دقيقة.
ملاحظة: يجب إضافة DOC قطرة قطرة. - الكريات في <3 مل من 25 ملي من Tris-HCl ، ودرجة الحموضة 7.5 ، و 0.3 م من KCl ، و 45٪ جلسرين (حجم / حجم) ، و 2 ملي مولار EDTA. قم بتجانس التعليق واضبطه على الحجم النهائي البالغ 10 مل. حدد تركيز البروتين ثم اضبطه على 6.5 مجم / مل. أضف azolectin بمعدل 5 مجم / مل و 0.85 (وزن / وزن) Zwittergent 3-14.
ملاحظة: يجب إضافة Azolectin و Zwittergent 3-14 قطرة قطرة. - تجانس التعليق وأجهزة الطرد المركزي (100,000 × جم) عند 4 درجات مئوية لمدة 60 دقيقة. اجمع المادة الطافية وخفف 1: 2 مع 2 ملي مولار من EGTA (درجة الحموضة 7.2).
- صب المعلق برفق على تدرج تركيز تريهالوز متقطع (45 و 40 و 35 و 30٪ وزن / حجم) في 10 ملي مولار من Tris-HCl ، ودرجة الحموضة 7.0 ، و 1 ملي من EDTA ، و 0.1٪ ديوكسيكولات و 1 مجم / مل من الأزولكتين. جهاز طرد مركزي (100,000 × ز) عند 4 درجات مئوية لمدة 14 ساعة. اجمع الحبيبات الشفافة المصفرة قليلا المتكونة في قاع الأنابيب.
ملاحظة: يجب إجراء كل من تكوين تدرج التريهالوز وإضافة معلق العينة باستخدام التقنيات المناسبة. يوصى بالطرد المركزي بين عشية وضحاها. - الحبيبات برفق في حجم صغير (<1.5 مل) من 25 ملي من Tris-HCl ، ودرجة الحموضة 7.5 ، و 0.3 من M KCl ، و 45٪ جلسرين ، و 2 ملي مولار من EDTA. تحديد تركيز البروتين. اضبط تركيز البروتين عن طريق التخفيف إلى 2 مجم / مل. خذ كميات (~ 50 ميكرولتر) من المعلق وقم بتخزين SERCA المنقى عند -72 درجة مئوية حتى استخدامه.
- قم بإجراء SDS-PAGE من SERCA72 المنقى ، وعينات البروتين المأخوذة في خطوات مختلفة من التنقية. لطخ الجل ب Coomassie Blue72.
ملاحظة: يزيد استخدام 0.6 M trehalose بدلا من الجلسرين في المخزن المؤقت المعلق من استقرار الإنزيم أثناء التخزين ودورات التجميدوالذوبان 28،29.
3. فحص نشاط ATPase (التوقيت ، 1 - 2 ساعة)
- الفحص المقترن بالإنزيم: نازعة هيدروجين ATPase-Piruvate كيناز لاكتات
- تحضير خليط التفاعل (1 مل): 50 ملي مولار MOPS (الرقم الهيدروجيني 7.0) ، 1 ملي مولار EGTA ، 80 ملي كلوريد الكودميوم ، 5 ملي ملي MgCl2 ، 3 ملي CaCl2 ، 5 ملي فوسفوينول بيروفات ، 250 ميكرومتر NADH. قم بتضمين تركيزات ATP المتغيرة (0.001 إلى 0.25 مليمتر).
- تجانس بعناية مقايسة التفاعل عن طريق الدوامة. احتضان مقايسة التفاعل عند 37 درجة مئوية لمدة 10 دقائق. أضف 0.9 U من نازعة هيدروجين L-lactate (LDH) و 1.5 U بيروفات كيناز (PK).
- ابدأ تفاعل ATPase بإضافة 10 ميكروغرام من SERCA.
- تحديد تكوين NADH. تابع التغير في شدة الامتصاص كل ثانية لمدة 600 ثانية بطول موجي (λ) يبلغ 340 نانومتر بمرور الوقت باستخدام مقياس الطيف الضوئي مع حامل خلية ترموستات.
ملاحظة: يمكن أن يختلف الفاصل الزمني لقياس الامتصاص ، ولكن حاول تسجيل أكبر عدد ممكن من نقاط البيانات لرسم الخط المستقيم الأولي لتفاعل ATPase.
- تحديد معدل التحلل المائي ATP
ملاحظة: للحصول على تفاصيل حول الإجراء ، راجع Rivera-Morán et al.46.- احسب قيمة المنحدر (Δabs / دقيقة) للجزء الخطي في كل منحنى متكون. استخدم معامل الانقراض المولي (ε) ل NADH (λ = 6،220 M−1·cm−1) لتحديد نشاط ATPase (μmoles ATP متحلل / min.mg prot.)
- تحديد معلمات الحركية Km و Vmax
- ارسم بيانات السرعة مقابل تركيز ATP باستخدام برامج تحليل البيانات والرسوم البيانية.
- قم بملاءمة البيانات عن طريق الانحدار غير الخطي لمعادلة Michaelis-Menten (المعادلة 1)46:
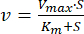
حيث v هي السرعة ، Vmax هي السرعة القصوى ، [S] هي تركيز ATP ، و Km هي ثابت Michaelis-Menten. - حدد Km و Vmax عن طريق التكرار.
ملاحظة: قد يؤدي إدراج DOC إلى زيادة استجابة معدل ATPase؛ يوصى باختبار تركيزات مختلفة من DOC للعثور على المستوى الأمثل.
4. مقايسة وضع العلامات على SERCA باستخدام Fluorescein Isothiocyanate (FITC) (التوقيت ، 30-45 دقيقة ، فقط لوضع العلامات على FITC)
ملاحظة: للحصول على تفاصيل حول الإجراء ، راجع54،75،76.
- قم بتعليق SERCA (20 ميكروغرام) في 50 ميكرولتر (الحجم النهائي) من المخزن المؤقت لوضع العلامات (100 ملي مولار من KCl ، و 5 ملي مولار MgCl2 ، و 30 ملي من Tris-HCl ، درجة الحموضة 8.9) تحتوي على 1 ملي مولار من FITC. تخلط عن طريق الدوامة.
- احتضان العينات في أوقات مختلفة (15 دقيقة و 10 دقائق و 7 دقائق و 5 دقائق و 2 دقيقة) في الظلام وفي درجة حرارة الغرفة.
- أوقف تفاعل وضع العلامات عن طريق إضافة حجم واحد من المخزن المؤقت للتوقف المثلج (480 ملي مولار من السكروز و 48 ملي مولار من MOPS ، الرقم الهيدروجيني 7.0) ، الذي يحتوي على ATP (5 مليمتر). احتضن على الثلج لمدة 5 دقائق في الظلام.
- أخضع SERCA المسمى FITC إلى SDS-PAGE72. قم بتعريض المواد الهلامية الشفافة للأشعة فوق البنفسجية (λ = 302 نانومتر). صورة توثيق النتيجة.
- وصمة عار الجل مع Coomassie blue. صورة توثيق النتيجة.
ملاحظة: قد يؤدي إجراء وضع العلامات على FITC في غرفة مظلمة إلى تحسين جودة إشارة التألق.
5. أطياف ثنائية اللون الدائرية (CD) (توقيت ، 30 دقيقة)
ملاحظة: للحصول على تفاصيل حول الإجراء ، راجع11،77.
- قم بتعليق SERCA (3 ميكرومتر) في 400 ميكرولتر من محلول الفوسفات 10 ملي مولار (الرقم الهيدروجيني 7.0) عند 25 درجة مئوية.
- قم بتحميل العينة في خلية بطول المسار 0.1 سم. تعيين نطاق أطياف الأشعة فوق البنفسجية البعيدة λ بين 190-260 نانومتر. اضبط الدقة الداخلية وعرض النطاق الترددي على 1 نانومتر.
- سجل إشارة القرص المضغوط عند 50 نانومتر / دقيقة عند 25 درجة مئوية.
النتائج
SDS-PAGE من SERCA في مراحل مختلفة من التنقية (الشكل 1). يظهر جل Coomassie الملون باللون الأزرق إثراء نطاق بروتين SERCA (الوزن الجزيئي الظاهر >100 كيلو دالتون) مع تقدم بروتوكول التنقية. يظهر نطاق البروتين المقابل ل SERCA نقاء بنسبة >90٪ بعد الطرد المركزي على تدرج تركيز تريهالو?...
Discussion
لا تستطيع معظم الجزيئات والأيونات عبور أغشية الخلايا بحرية ، على سبيل المثال ، يتطلب البروتون (H +) ناقلا غشيا في غشاء البلازما لمجموعة متنوعة من الكائنات الحية والعضيات مثل الميتوكوندريا83،84. أغشية الخلايا انتقائية ، والجزيئات والأ?...
Disclosures
ويعلن صاحب البلاغ أنه ليس لديه مصالح مالية متنافسة.
Acknowledgements
يعترف المؤلف بمساعدة إدموندو ماتا موراليس في تحرير الفيديو ، VM ، Valentín de la Cruz-Torres في تنقية SRVs ، ميغيل أ. ريفيرا موران في تنقية وتحليل SERCA ، وخوان سي غونزاليس كاسترو ، وفرانكو إي خواريز ، وأيخاندرا نيفاريز ، ونيكولاس روشا فيزويت ، وجوسلين آي راميريز ألونسو في إنتاج الفيديو. لم يتم تلقي أي أموال أو منح أو أي دعم آخر لإجراء هذه الدراسة.
Materials
| Name | Company | Catalog Number | Comments |
| ATP | Sigma-Aldrich Corp | A2383 | |
| Azolectin from soybean | Sigma-Aldrich Corp | 44924 | |
| Benchtop UV transilluminator | Cole-Parmer | EW-97623-08 | Dual intensity High setting is ideal for analytical documentation. Low setting reduces photonicking or photobleaching of gel samples while doing preparative work. |
| CaCl2 • 2H2O | Sigma-Aldrich Corp | 223506 | |
| Coolpix B500 camera | Nikon Corp | S210 | |
| Coomassie brilliant blue G-250 | Bio-Rad | 1610406 | |
| Dodecyl maltoside | Sigma-Aldrich Corp | D4641 | |
| Ethylene glycol-bis(2-aminoethylether)-N,N,N′,N′-tetraacetic acid | Sigma-Aldrich Corp | E4378 | |
| Ethylenediaminetetraacetic acid tetrasodium salt dihydrate | Sigma-Aldrich Corp | E6511 | |
| Fluorescein isothiocyanate | Sigma-Aldrich Corp | F3651 | |
| KCl | JT Baker | 7447-40-7 | |
| MgCl2 • 6H2O | Sigma-Aldrich Corp | M9272 | |
| MOPS | Sigma-Aldrich Corp | M1254 | |
| NADH | Chem-Impex International Inc | 230 | |
| N-tetradecyl-N,N- dimethyl-3-ammonium-1-propanesulfonate | Sigma-Aldrich Corp | D0431 | |
| Phosphoenolpyruvate (PEP) | Chem-Impex International Inc | 9711 | |
| Rabbit muscle lactate dehydrogenase | Roche | 10003557103 | |
| Rabbit muscle pyruvate kinase | Sigma-Aldrich Corp | P1506 | |
| Sodium deoxycholate | Sigma-Aldrich Corp | D6750 | |
| Sodium dodecyl sulfate | Bio-Rad | 1610302 | |
| Spectropolarimeter | Jasco Corp. | Jasco J1500 | |
| Sucrose | Sigma-Aldrich Corp | 84100 | |
| Trehalose | Sigma-Aldrich Corp | T0167 | Dihydrate |
| Tris(hydroxymethyl)aminomethane hydrochloride | Sigma-Aldrich Corp | 857645 | |
| Ultracentrifuge | Beckman | Optima XPN | |
| UV/VIS spectrophotometer | Agilent Technologies | 8453 | The Agilent 8453 UV-Vis Spectrophotometer uses a photodiode array for simultaneous measurement of the complete ultra-violet to visible light spectrum |
| WiseStir HS- 30E | Daihan Scientific Co. | DH.WOS01010 | Ideal for all disperging and homogenizing applications, designed for tissue grinders. |
References
- Schuberth, C., Wedlich-Söldner, R. Building a patchwork - The yeast plasma membrane as model to study lateral domain formation. Biochim Biophys Acta Mol Cell Res. 1853 (4), 767-774 (2015).
- Douglas, L. M., Konopka, J. B. Fungal membrane organization: The eisosome concept. Annu Rev Microbiol. 68 (1), 377-393 (2014).
- Borrell, J. H., Domènech, &. #. 2. 1. 0. ;., Keough, K. M. W. . Membrane protein - Lipid interactions: Physics and Chemistry in the Bilayer. , (2016).
- Moraes, I., Evans, G., Sanchez-Weatherby, J., Newstead, S., Stewart, P. D. . The next generation in membrane protein structure determination. , (2016).
- White, S. H. Biophysical dissection of membrane proteins. Nature. 459 (7245), 344-346 (2009).
- Lacapère, J. -. J. Membrane protein structure determination. Methods Mol Biol. , 459 (1002).
- Palmgren, M. G. Plant plasma membrane H+-ATPases: Powerhouses for nutrient uptake. Annu Rev Plant Physiol Plant Mol Biol. 52 (1), 817-845 (2001).
- Yatime, L., et al. P-type ATPases as drug targets: Tools for medicine and science. Biochim Biophys Acta Bioenerg. 1787 (4), 207-220 (2009).
- Drews, J. Tetrahydrobiopterin biosynthesis as an off-target of sulfa drugs. Science. 287 (5460), 1960-1964 (2000).
- Xu, Z., Meshcheryakov, V. A., Poce, G., Chng, S. -. S. MmpL3 is the flippase for mycolic acids in mycobacteria. Proc Natl Acad Sci USA. 114 (30), 7993-7998 (2017).
- Siligardi, G., Hussain, R., Patching, S. G., Phillips-Jones, M. K. Ligand- and drug-binding studies of membrane proteins revealed through circular dichroism spectroscopy. Biochim Biophys Acta Biomembr. 1838 (1 PARTA), 34-42 (2014).
- Rawlings, A. E. Membrane proteins: Always an insoluble problem. Biochem Soc Trans. 44 (3), 790-795 (2016).
- Popot, J. -. L. . Membrane proteins in aqueous solutions. , 708 (2018).
- Palmgren, M. G., Sommarin, M., Ulvskov, P., Larsson, C. Effect of detergents on the H+-ATPase activity of inside-out and right-side-out plant plasma membrane vesicles. Biochim Biophys Acta Biomembr. 1021 (2), 133-140 (1990).
- McBride, Z., Chen, D., Reick, C., Xie, J., Szymanski, D. B. Global analysis of membrane-associated protein oligomerization using protein correlation profiling. Mol Cell Proteomics. 16 (11), 1972-1989 (2017).
- Yoneda, J. S., et al. Multimeric species in equilibrium in detergent-solubilized Na, K-ATPase. Int J Biol Macromol. 89, 238-245 (2016).
- Ohlendieck, K. Extraction of membrane proteins. Protein Purif Protocols. 59, 283-294 (2004).
- Champeil, P., et al. A robust method to screen detergents for membrane protein stabilization, revisited. Anal Biochem. 511, 31-35 (2016).
- Gimpl, K., Klement, J., Keller, S. Characterising protein/detergent complexes by triple-detection size-exclusion chromatography. Biol Proced Online. 18, 4 (2016).
- le Maire, M., et al. Gel chromatography and analytical ultracentrifugation to determine the extent of detergent binding and aggregation, and Stokes radius of membrane proteins using sarcoplasmic reticulum Ca2+-ATPase as an example. Nat Protoc. 3 (11), 1782-1795 (2008).
- Du, M., et al. Progress, applications, challenges and prospects of protein purification technology. Front Bioeng Biotechnol. 10, 1-26 (2022).
- Slotboom, D. J., Duurkens, R. H., Olieman, K., Erkens, G. B. Static light scattering to characterize membrane proteins in detergent solution. Methods. 46 (2), 73-82 (2008).
- Harding, S. E. Analytical ultracentrifugation as a matrix-free probe for the study of kinase related cellular and bacterial membrane proteins and glycans. Molecules. 26, 6080 (2021).
- Chen, Y. -. C., et al. Thermal stability, storage and release of proteins with tailored fit in silica. Sci Rep. 7 (1), 46568 (2017).
- Montigny, C. C., Arnou, B., Marchal, E., Champeil, P. Use of glycerol-containing media to study the intrinsic fluorescence properties of detergent-solubilized native or expressed SERCA1a. Biochemistry. 47 (46), 12159-12174 (2008).
- Simongini, M., Puglisi, A., Genovese, F., Hochkoeppler, A. Trehalose counteracts the dissociation of tetrameric rabbit lactate dehydrogenase induced by acidic pH conditions. Arch Biochem Biophys. 740, 109584 (2023).
- Shivanna, B. D., Rowe, E. S. Preservation of the native structure and function of Ca2+-ATPase from sarcoplasmic reticulum: Solubilization and reconstitution by new short-chain phospholipid detergent 1,2-diheptanoyl-sn-phosphatidylcholine. Biochem J. 325 (2), 533-542 (1997).
- Sampedro, J. G., Guerra, G., Pardo, J. P., Uribe, S. Trehalose-mediated protection of the plasma membrane H+-ATPase from Kluyveromyces lactis during freeze-drying and rehydration. Cryobiology. 37 (2), 131-138 (1998).
- Sampedro, J. G., Cortés, P., Muñoz-Clares, R. A., Fernández, A., Uribe, S. Thermal inactivation of the plasma membrane H+-ATPase from Kluyveromyces lactis. Protection by Trehalose. Biochim Biophys Acta Proteins Proteomics. 1544 (1-2), 64-73 (2001).
- Sampedro, J. G., et al. Fluorescence quenching by nucleotides of the plasma membrane H+-ATPase from Kluyveromyces lactis. Biochemistry. 46 (18), 5616-5622 (2007).
- Ruiz-Granados, Y. G., De La Cruz-Torres, V., Sampedro, J. G. The oligomeric state of the plasma membrane H+-ATPase from Kluyveromyces lactis. Molecules. 24 (5), 958 (2019).
- Zhao, P., Zhao, C., Chen, D., Yun, C., Li, H., Bai, L. Structure and activation mechanism of the hexameric plasma membrane H+-ATPase. Nat Commun. 12 (1), 6439 (2021).
- Auer, M., Scarborough, G. A., Kühlbrandt, W. Surface crystallization of the plasma membrane H+-ATPase on a carbon support film for electron crystallography. J Mol Biol. 287 (5), 961-968 (1999).
- Hennessey, J. P., Scarborough, G. A. Secondary structure of the Neurospora CRASSA plasma membrane H+-ATPase as estimated by circular dichroism. J Biol Chem. 263 (7), 3123-3130 (1988).
- Scarborough, G. A. Crystallization, structure and dynamics of the proton-translocating P-type ATPase. J Exp Biol. 203 (Pt 1), 147-154 (2000).
- Sumida, K. H., et al. Improving protein expression, stability, and function with proteinMPNN. J Am Chem Soc. 146 (3), 2054-2061 (2024).
- Eleutherio, E., Panek, A., De Mesquita, J. F., Trevisol, E., Magalhães, R. Revisiting yeast trehalose metabolism. Curr Genet. 61 (3), 263-274 (2015).
- Attfield, P. V. Trehalose accumulates in Saccharomyces cerevisiae during exposure to agents that induce heat shock response. FEBS Lett. 225 (1-2), 259-263 (1987).
- Marunde, M. R., et al. Improved tolerance to salt and water stress in Drosophila melanogaster cells conferred by late embryogenesis abundant protein. J Insect Physiol. 59 (4), 377-386 (2013).
- Crowe, J. H. Anhydrobiosis: An unsolved problem with applications in human welfare. Subcell Biochem. 71, 263-280 (2015).
- Crowe, J. H., Crowe, L. M., Chapman, D. Preservation of membranes in anhydrobiotic organisms: The role of trehalose. Science. 223 (4637), 701-703 (1984).
- Sampedro, J. G., Rivera-Moran, M. A., Uribe-Carvajal, S. Kramers' theory and the dependence of enzyme dynamics on trehalose-mediated viscosity. Catalysts. 10 (6), 1-19 (2020).
- Sampedro, J. G., Uribe, S. Trehalose-enzyme interactions result in structure stabilization and activity inhibition. The role of viscosity. Mol Cell Biochem. 256 - 257 (1-2), 319-327 (2004).
- Crowe, J. H. Trehalose as a "chemical chaperone": Fact and fantasy. Adv Exp Med Biol. 594, 143-158 (2007).
- Crowe, J. H., Carpenter, J. F., Crowe, L. M., Anchordoguy, T. J. Are freezing and dehydration similar stress vectors? A comparison of modes of interaction of stabilizing solutes with biomolecules. Cryobiology. 27 (3), 219-231 (1990).
- Rivera-Morán, M. A., Sampedro, J. G. Isolation of the sarcoplasmic reticulum Ca2+-ATPase from rabbit fast-twitch muscle. Methods Protoc. 6 (5), 102 (2023).
- Páez-Pérez, E. D., De La Cruz-Torres, V., Sampedro, J. G. Nucleotide binding in an engineered recombinant Ca2+-ATPase N-domain. Biochemistry. 55 (49), 6751-6765 (2016).
- Ramírez-Alonso, J. I., Sampedro, J. G. Effect of cations on ATP binding to the N-domain of Na+, K+-ATPase. J Fluoresc. , (2024).
- De la Cruz-Torres, V., Cataño, V., Olivo-Rodríguez, M., Sampedro, J. G. ANS Interacts with the Ca2+-ATPase nucleotide binding site. J Fluoresc. 30 (3), 483-496 (2020).
- Sampedro, J. G., Nájera, H., Uribe-Carvajal, S., Ruiz-Granados, Y. G. Mapping the ATP binding site in the plasma membrane H+-ATPase from Kluyveromyces lactis. J Fluoresc. 24 (6), 1849-1859 (2014).
- Kiani, A. K., et al. Ethical considerations regarding animal experimentation. J Prev Med Hyg. 63 (2 Suppl 3), E255-E266 (2022).
- Ruiz-Granados, Y., De La Cruz-Torres, V., Sampedro, J. The oligomeric state of the plasma membrane H+-ATPase from Kluyveromyces lactis. Molecules. 24 (5), 958 (2019).
- Wang, S., et al. Structural basis for sarcolipin's regulation of muscle thermogenesis by the sarcoplasmic reticulum Ca2+-ATPase. Sci Adv. 7 (48), eabi7154 (2021).
- Champeil, P., et al. ATP regulation of sarcoplasmic reticulum Ca2+-ATPase. Metal-free ATP and 8-bromo-ATP bind with high affinity to the catalytic site of phosphorylated ATPase and accelerate dephosphorylation. J Biol Chem. 263 (25), 12288-12294 (1988).
- Møller, J. V., Olesen, C. Preparation of Ca(2+)-ATPase1a enzyme from rabbit sarcoplasmic reticulum. Methods Mol Biol. 1377, 11-17 (2016).
- Wuytack, F., De Schutter, G., Casteels, R. Partial purification of (Ca2+ + Mg2+)-dependent ATPase from pig smooth muscle and reconstitution of an ATP-dependent Ca2+-transport system. Biochem J. 198 (2), 265-271 (1981).
- Mandal, A., et al. Solubilization, purification and reconstitution of Ca2+-ATPase from bovine pulmonary artery smooth muscle microsomes by different detergents: Preservation of native structure and function of the enzyme by DHPC. Biochim Biophys Acta Gen Subj. 1760 (1), 20-31 (2006).
- Inesi, G., Ma, H., Hua, S., Toyoshima, C. Characterization of Ca2+ ATPase residues involved in substrate and cation binding. Ann NY Acad Sci. 986 (1), 63-71 (2003).
- Van Winkle, W. B., Pitts, B. J., Entman, M. L. Rapid purification of canine cardiac sarcoplasmic reticulum Ca2+-ATPase. J Biol Chem. 253 (24), 8671-8673 (1978).
- Ruiz-Granados, Y. G., De La Cruz-Torres, V., Sampedro, J. G. The oligomeric state of the plasma membrane H+-ATPase from Kluyveromyces lactis. Molecules. 24 (5), 958 (2019).
- Teramoto, N., Sachinvala, N. D., Shibata, M. Trehalose and trehalose-based polymers for environmentally benign, biocompatible, and bioactive materials. Molecules. 13 (8), 1773-1816 (2008).
- Dao, T. T., et al. Demethoxycurcumin is a potent inhibitor of P-type ATPases from diverse kingdoms of life. PLOS ONE. 11 (9), e0163260 (2016).
- Bleeker, N. P., Cornea, R. L., Thomas, D. D., Xing, C. A novel SERCA inhibitor demonstrates synergy with classic SERCA inhibitors and targets multidrug-resistant AML. Mol Pharmaceutics. 10 (11), 4358-4366 (2013).
- Michelangeli, F., East, J. M. A diversity of SERCA Ca2+ pump inhibitors. Biochem Soc Trans. 39 (3), 789-797 (2011).
- Tadini-Buoninsegni, F., Smeazzetto, S., Gualdani, R., Moncelli, M. R. Drug interactions with the Ca2+-ATPase from Sarco(Endo)Plasmic Reticulum (SERCA). Front Mol Biosci. 5, 36 (2018).
- Clausen, J. D., et al. Elucidation of antimicrobial activity and mechanism of action by N-substituted carbazole derivatives. Bioorg Med Chem Lett. 27 (19), 4564-4570 (2017).
- Leary, S., et al. . AVMA guidelines for the euthanasia of animals: 2020 Edition, Members of the Panel on Euthanasia AVMA Staff Consultants. , (2020).
- de Aluja, A. S. Laboratory animals and official Mexican norms (NOM-062-ZOO-1999). Gac Med Mex. 138 (3), 295-298 (2002).
- Champeil, P., Buschlen-Boucly, S., Bastide, F., Gary-Bobo, C. Sarcoplasmic reticulum ATPase. Spin labeling detection of ligand-induced changes in the relative reactivities of certain sulfhydryl groups. J Biol Chem. 253 (4), 1179-1186 (1978).
- Skalec, A., Janeczek, M., Czerski, A. Anatomy and histology of the rabbit common calcanean tendon. J Vet Med Series C: Anat Histol Embryol. 48 (5), 466-475 (2019).
- Janes, L. E., Mioton, L. M., Fracol, M. E., Ko, J. H. An in vivo comparison: Novel mesh suture versus traditional suture-based repair in a rabbit tendon model. J Hand Surg Glob Online. 4 (1), 32-39 (2022).
- Bollag, D. M., Rozycki, M. D., Edelstein, S. J. . Protein Methods. , (1996).
- Gagné, F. Tissue preparation and subcellular fractionation techniques. Biochem Ecotoxicol. , 21-31 (2014).
- Lowry, O. H., Rosebrough, N. J., Farr, A. L., Randall, R. J. Protein measurement with the Folin phenol reagent. J Biol Chem. 193 (1), 265-275 (1951).
- Autry, J. M., Rubin, J. E., Svensson, B., Li, J. L., Thomas, D. D. Nucleotide activation of the Ca-ATPase. J Biol Chem. 287 (46), 39070-39082 (2012).
- Winters, D. L., Autry, J. M., Svensson, B., Thomas, D. D. Interdomain fluorescence resonance energy transfer in SERCA probed by cyan-fluorescent protein fused to the actuator domain. Biochemistry. 47 (14), 4246-4256 (2008).
- Miles, A. J., Wallace, B. A. Circular dichroism spectroscopy of membrane proteins. Chem Soc Rev. 14 (45), 1130-1135 (2016).
- Scofano, H. M., Vieyra, A., de Meis, L. Substrate regulation of the sarcoplasmic reticulum ATPase. Transient kinetic studies. J Biol Chem. 254 (20), 10227-10231 (1979).
- Yamamoto, T., Tonomura, Y. Reaction mechanism of the Ca++-dependent ATPase of sarcoplasmic reticulum from skeletal muscle: I. Kinetic studies. J Biochem. 62 (5), 558-575 (1967).
- Murphy, A. J. Affinity labeling of the active site of the Ca2+-ATPase of sarcoplasmic reticulum. Biochim Biophys Acta - Biomembr. 946 (1), 57-65 (1988).
- Csermely, P., Katopis, C., Wallace, B. A., Martonosi, A. The E1→E2 transition of Ca2+-transporting ATPase in sarcoplasmic reticulum occurs without major changes in secondary structure. A circular-dichroism study. Biochem J. 241 (3), 663-669 (1987).
- Abràmoff, M. D., Magalhães, P. J., Ram, S. J. Image processing with ImageJ. Biophotonics Int. 11 (7), 36-42 (2004).
- Scarborough, G. A. The plasma membrane proton-translocating ATPase. Cell Mol Life Sci. 57 (6), 871-883 (2000).
- Llopis, J., McCaffery, J. M., Miyawaki, A., Farquhar, M. G., Tsien, R. Y. Measurement of cytosolic, mitochondrial, and Golgi pH in single living cells with green fluorescent proteins. Proc Natl Acad Sci. 95 (12), 6803-6808 (1998).
- Kanai, R., Vilsen, B., Cornelius, F., Toyoshima, C. Crystal structures of Na+, K+-ATPase reveal the mechanism that converts the K+-bound form to Na+-bound form and opens and closes the cytoplasmic gate. FEBS Lett. 597 (15), 1957-1976 (2023).
- Palmgren, M. P-type ATPases: Many more enigmas left to solve. J Biol Chem. 299 (11), 105352 (2023).
- Frauenfelder, H. Ask not what physics can do for biology-Ask what biology can do for physics. Phys Biol. 11 (5), 053004 (2014).
- Frauenfelder, H. The physics of proteins. Annu Rev Biophys Biomol Struct. 36, (2010).
- Narhi, L. O. . Biophysics for Therapeutic Protein Development. , (2013).
- East, J. M. Purification of a membrane protein (Ca2+/Mg2+-ATPase) and its reconstitution into lipid vesicles. Methods Mol Biol. 27, 87-94 (1994).
- Lee, E. -. H., et al. Enhancement of enzyme activity and stability by poly(γ-glutamic acid). Polymer J. 42 (10), 818-822 (2010).
- Norrild, R. K., et al. Increasing protein stability by inferring substitution effects from high-throughput experiments. Cell Rep Methods. 2 (11), 100333 (2022).
- Song, Z., Zhang, Q., Wu, W., Pu, Z., Yu, H. Rational design of enzyme activity and enantioselectivity. Front Bioeng Biotechnol. 11, 1-14 (2023).
- Jidenko, M., Lenoir, G., Fuentes, J. M., le Maire, M., Jaxel, C. Expression in yeast and purification of a membrane protein, SERCA1a, using a biotinylated acceptor domain. Protein Expr Purif. 48 (1), 32-42 (2006).
- Hua, S., Ma, H., Lewis, D., Inesi, G., Toyoshima, C. Functional role of "N" (nucleotide) and "P" (phosphorylation) domain interactions in the sarcoplasmic reticulum (SERCA) ATPase. Biochemistry. 41 (7), 2264-2272 (2002).
- Pang, Y. -. H., Chen, J. -. W. Anisodamine causes the changes of structure and function in the transmembrane domain of the Ca2+-ATPase from sarcoplasmic reticulum. Biosci Biotechnol Biochem. 68 (1), 126-131 (2004).
Reprints and Permissions
Request permission to reuse the text or figures of this JoVE article
Request PermissionExplore More Articles
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. All rights reserved