Zum Anzeigen dieser Inhalte ist ein JoVE-Abonnement erforderlich. Melden Sie sich an oder starten Sie Ihre kostenlose Testversion.
Method Article
Aufreinigung des Sarko-endoplasmatischen Retikulums Ca2+-ATPase aus dem Kaninchenmuskel
In diesem Artikel
Zusammenfassung
Dieses Protokoll beschreibt eine verbesserte SERCA-Aufreinigungsmethode, bei der das Disaccharid Trehalose in den letzten Zentrifugationsschritt einbezogen wird. Dieses Kohlenhydrat stabilisiert Proteine unter rauen Bedingungen. Das gereinigte SERCA war katalytisch aktiv und wies eine hohe Reinheit auf, wodurch es sich für strukturelle und funktionelle Studien eignete.
Zusammenfassung
Einige P-Typ-ATPasen, wie z. B. Sarko-/endoplasmatisches RetikulumCa-2+-ATPase (SERCA), sind inhärent labile Membranproteine, die während der Reinigung spezifische physikalisch-chemische Bedingungen erfordern, um sie mit hoher Reinheit und struktureller Qualität und in katalytisch aktiver Form zu erhalten. Das Disaccharid Trehalose ist ein kompatibler gelöster Stoff, der synthetisiert und in hohen Konzentrationen im Zytoplasma der Hefe akkumuliert wird, um die Membranen und Proteine zu stabilisieren. Die Verwendung von Trehalose als Additiv im Protokoll zur Reinigung der Plasmamembran H+-ATPase führt zu einem hochwertigen Präparat, dessen hexamere Struktur durch biochemische Analysemethoden gezeigt wird. Trehalose kann daher als stabilisierendes Additiv für die Aufreinigung von Membranproteinen (P-ATPasen) eingesetzt werden. Dieses Protokoll beschreibt die Modifikation des klassischen Protokolls für die SERCA-Reinigung, indem SERCA einer Zentrifugation an einem Trehalose-Konzentrationsgradienten unterzogen wird. Die Aufnahme dieses Kohlenhydrats führte zur Reinigung von SERCA in einer katalytisch aktiven Form mit hoher Reinheit und vor allem in einer stabilen Form. Eine partielle biochemische Charakterisierung des gereinigten SERCA (SDS-PAGE, Enzymkinetik, FITC-Markierung, Zirkulardichroismusspektroskopie) zeigte, dass das Enzym für funktionelle und strukturelle Studien geeignet ist. Die Verwendung von Trehalose im Aufreinigungsprotokoll von P-Typ-ATPasen und anderen labilen Membran- (und zytosolischen) Proteinen wird vorgeschlagen.
Einleitung
Membranproteine/Enzyme sind essentielle biologische Bestandteile von Zellen, da sie in verschiedenen Prozessen eine entscheidende Rolle spielen 1,2,3. Einige der Funktionen können den Transport von Ionen und Molekülen in und aus der Zelle/den inneren Kompartimenten (entweder aktiv/passiv), die Zell-Zell-Erkennung, die interzelluläre Bindung, die Verankerung/Bindung und die Wahrnehmung der äußeren Umgebung durch Integration mit der Signaltransduktionsmaschinerie unter normalen und rauen physikalischen und chemischen Bedingungen (hoher Salzgehalt, niedriger Wassergehalt, hohe Temperatur, Arzneimittelresistenz, usw.)3 Daher ist die Bestimmung der dreidimensionalen (3D) Struktur von Membranproteinen und/oder Enzymen sowohl für die Grundlagenforschung als auch für die angewandte Forschung von großer Bedeutung geworden 4,7,6. Wichtig ist, dass Membranproteine/-enzyme in großem Umfang als Ziele für die Wirkstoffforschung (ob natürlich oder beabsichtigt) verwendet werden7,8,9. Das heißt, Membranproteine haben eine inhärente Bedeutung für die Gesundheit 9,10,11.
Der hydrophobe Charakter von Membranproteinen/-enzymen ist die technisch anspruchsvollste physikalisch-chemische Eigenschaft für das Experimentallabor 12,13,14, umso mehr bei der Arbeit mit oligomeren und/oder hochlabilen integralen Membranproteinen 15,16. Die Isolierung geeigneter Mengen des Membranproteins/Enzyms mit höchstmöglicher Qualität für funktionelle experimentelle Assays und Strukturstudien ist sehr wünschenswert17. Membranproteine, die von Natur aus hydrophob sind, sind sehr schwer zu reinigen, und die Wahl des Detergens ist in der Regel eines der wichtigsten Punkte, die berücksichtigt werden müssen 17,18,19,20. In dieser Hinsicht verursachen die Labormethoden, die zur Isolierung von Membranproteinen verwendet werden, typischerweise einen gewissen Grad an Schädigung der 3D-Strukturanordnung des Proteins21. Einige dieser Verfahren umfassen die Verwendung von (a) Beschallung (hochfrequente/energiereiche Ultraschallwellen), (b) proteinlösliche Detergenzien (entweder scharf, mittel oder schonend)22, (c) relativ hohe Drücke in der Säulenchromatographie und Hochgeschwindigkeits-Ultrazentrifugation23, (d) fällende Moleküle, (e) Verdauungsenzymeund andere 21. All diese Prozesse können zur Destabilisierung von Proteinen während der Reinigung beitragen oder die Hauptursache dafür sein21. In dieser Hinsicht scheinen einige Protokolle für ein bestimmtes Membranprotein relativ gut zu funktionieren. Eine Optimierung ist jedoch immer willkommen, wenn neue moderne Assays oder Methoden verwendet werden, die eine höhere Qualität der Membranproteinpräparation erfordern, um zufriedenstellende Ergebnisse zu erzielen24. Zu den Optimierungsschritten gehören unter anderem die Verbesserung des Konstruktdesigns, die Suche nach optimalen Bedingungen für die Expression von Membranproteinen, die Etablierung besserer Handhabungsbedingungen (d. h. pH-Wert, Temperatur usw.), die Suche nach dem am besten kompatiblen Detergensmittel, die Anpassung von Reinigungsschritten wie Beschallungszeit, Zentrifugationsgeschwindigkeit, Neuformulierung von Pufferlösungen durch Zugabe von Stabilisierungsmitteln usw.25,26,27. Daher ist jede Änderung der Reinigungsmethodik, die zu einer Steigerung der Qualität (Reinheit) und Aktivität des gereinigten Membranproteins/Enzyms führt, wichtig.
In der Familie der P-Typ-ATPase scheint die Hefe-Plasmamembran H+-ATPase eines der labilsten Mitglieder zu sein28,29. Die H+-ATPase verliert ihre hydrolysierende ATP-Aktivität bei Dehydratisierung (Gefriertrocknung), Hitzeschock usw.28,29. Die Verwendung von Trehalose als Proteinstabilisator wurde bei der Isolierung der Plasmamembran H+-ATPase aus der Hefe K. lactis30,31 getestet; Das erhaltene H+-ATPase-Präparat war von hoher Reinheit und katalytisch aktiv. Wichtig ist, dass dies ermöglichte, den oligomeren Zustand des Enzyms durch biochemische Methoden aufzulösen, wodurch es sich als Hexamer entpuppte und später durch Kryo-Elektronenmikroskopie bestätigt wurde 31,32,33,34,35. Daher scheint es wahrscheinlich, dass die empfindliche dreidimensionale (3D) Anordnung von Membranproteinen unter relativ rauen Bedingungen während der Reinigung verloren gehenkann 36. Eine biphasische Inaktivierungskinetik wird für H+-ATPase während der hitzevermittelten Inaktivierung beobachtet29. In Hefezellen, wie in vielen anderen Organismen, reichert sich das Disaccharid Trehalose in hohen Konzentrationen unter Umweltstressbedingungen an 37,38,39. Trehalose erhält die Integrität der Membran und die Transportfunktion aufrecht, indem sie Proteine (sowohl membraneingebettete als auch zytosolische) und Zellmembranen stabilisiert40,41. Der stabilisierende Mechanismus von Trehalose wurde von mehreren Gruppen und von unserem Labor ausführlich untersucht 42,43,44,45. Experimente mit der H+-ATPase und anderen Enzymen haben gezeigt, dass Trehalose der effektivste Proteinstabilisator unter den Mono- und Disacchariden ist28,29. Dies führte zur Aufnahme in das H+-ATPase-Aufreinigungsprotokoll30. In jüngster Zeit wurde Trehalose auch bei der Reinigung des sarkoplasmatischen Retikulums Ca2+-ATPase (SERCA) aus schnell zuckenden Kaninchenmuskeln mit guten Ergebnissen in Bezug auf Proteinreinheit und -aktivität verwendet46. Daher scheint Trehalose ein gutes und geeignetes Additiv für die Aufreinigung von P-Typ-ATPasen und wahrscheinlich anderen Membran- und zytosolischen Proteinen zu sein.
Für P-ATPasen ist das Vorhandensein struktureller zytoplasmatischer Domänen ein experimenteller Vorteil, insbesondere für Substrat/Liganden-Interaktionsstudien47; Die ATP-Bindung wurde in hochreinen rekombinanten N-Domänen untersucht 47,48,49, wodurch die technischen Überlegungen zur Reinigung ganzer Membranenzyme 47,48,50 u.a. 51 entfallen. Leider benötigen einige funktionelle (katalytische/Energieumwandlung) und strukturelle (Anordnung der Untereinheiten und Wechselwirkung mit anderen Proteinen) Studien immer noch die gesamte P-ATPase52,53. In dieser Hinsicht wurde die Reinigung von SERCA von mehreren Forschungsgruppen erreicht 54,55,56,57,58. Es können jedoch immer noch Verbesserungen umgesetzt werden46, z. B. die Erhöhung der Unversehrtheit der gereinigten ATPase59, die Vermeidung der Denaturierung/Zerstörung von Proteinkomplexen25, die Erhöhung der Solubilisierung ohne Denaturierung des Membranproteins (d. h. die Vermeidung der Bildung makromolekularer Aggregate)60, die zu einer besseren Kompatibilität mit Assays und anderen nachgelagerten Analysemethodenführen 61 Darüber hinaus müssen neue experimentelle Strategien, Additive, Enzyminhibitoren usw. in der wissenschaftlichen Literatur 61,62,63,64,65,66 manchmal mit der gesamten P-ATPase getestet werden. Diese Arbeit beschreibt das Protokoll für die Aufreinigung von SERCA und die Verwendung von Trehalose als Additiv zur Stabilisierung der Proteinstruktur und der ATPase-Aktivität; D.h. neben der Erhöhung der (strukturellen) Qualität des Enzyms trägt Trehalose dazu bei, den Verlust der Enzymstruktur und -aktivität während der Enzymisolierung und -lagerung zu verhindern, was dazu beiträgt, biologisches Material zu sparen und somit die Anzahl der Enzymreinigungen zu reduzieren.
Protokoll
Alle Tierversuche wurden in Übereinstimmung mit internationalen und lokalen (NORMA Oficial Mexicana NOM-062-ZOO-1999) Richtlinien für den Umgang mit Tieren in Versuchslaboratorien 51,67,68 durchgeführt. Das Muskelgewebe wurde von Wildtyp-Oryctolagus cuniculus aus einer lokalen Tierhandhabungseinheit gewonnen (INE/CITES/DGVS-ZOO-E0055-SLP-98)46. Ein Tierarzt mit Erfahrung in der Haltung von Versuchstieren führte die erste Muskeldissektion und -aufbereitung durch. Die Einzelheiten zu den Reagenzien und der Ausrüstung, die in dieser Studie verwendet wurden, sind in der Materialtabelle aufgeführt.
1. Isolierung des sarkoplasmatischen Retikulums aus dem schnell zuckenden Kaninchenmuskel (Timing, 7 - 9 h)
HINWEIS: Einzelheiten zum Verfahren finden Sie in Champeil et al.69.
- Gewinnen Sie schnell zuckendes Muskelgewebe (~70 g von beiden Hinterbeinen pro Tier) von Wildtyp Oryctolagus cuniculus.
HINWEIS: Eine vorherige visuelle Identifizierung der zu präparierenden Muskeln ist erforderlich70,71. - Mazerieren Sie den schnell zuckenden Muskel in einem Mixer, indem Sie ihn in drei Volumina von 100 mM KCl suspendieren, mit 1 Minute an und 1 Minute in eiskalter Ruhe, wiederholen Sie dreimal72.
- Entfernen Sie die Gewebereste durch Zentrifugation (1500 × g) bei 4 °C für 5 Minuten; Bei Bedarf wird eine zweite Zentrifugation (4200 × g) durchgeführt.
- Der Überstand wird aufgefangen und mit einer Gewebemühle homogenisiert (10 Hübe bei niedriger Geschwindigkeit, d. h. 500-1.500 U/min)72,73. Das Homogenat (10.000 × g) bei 4 °C 15 min zentrifugieren.
HINWEIS: Beim Homogenisieren ist Vorsicht geboten, da die Verwendung einer relativ hohen Drehzahl zu Schäden an der Anlage und dem Bediener führen kann. - Der Überstand (mit dem sarkoplasmatischen Retikulum) (33.000 × g) wird 120 min lang bei 4 °C zentrifugiert.
- Die Pellets in 40 mL mit 0,5 M Saccharose suspendieren und 15 min (12.000 × g) bei 4 °C zentrifugieren. Der Überstand (0,6 M KCl und 0,15 M Saccharose) wird verdünnt und die Suspension (34.000 × g) bei 4 ΰC 165 min lang zentrifugiert.
- Suspendieren Sie das Pellet mit den Vesikeln des sarkoplasmatischen Retikulums (SRVs) in 0,3 M Saccharose, 0,1 M KCl und 5 mM Tris-HCl, pH 7,0. Bestimmen Sie die Proteinkonzentration unter Verwendung des Lowry-Assays74 und humanem Serumalbumin als Proteinstandard.
- Aliquotieren Sie die SRVs (30-32 mg/ml) in 1 ml Volumen. Lagern Sie die SRV-Proben zur weiteren Verarbeitung bei -72 °C.
HINWEIS: Die Proteinausbeute beträgt 100-110 mg SRVs pro Tier.
2. SERCA-Reinigung (Zeitmessung, 18 - 20 h)
HINWEIS: Für Einzelheiten zum Verfahren siehe Rivera-Morán et al.46.
- Verdünnen Sie die SRV-Suspension auf 2 mg Protein/ml mit eiskalten 75 mM Tris-HCl, pH 7,2, 0,6 M KCl, 6 mM EDTA, 1 mM EGTA und 0,1 % (w/v) Desoxycholat (DOC). Inkubieren Sie die SRV-Suspension 10 Minuten lang auf Eis und unter leichtem Rühren. Die SRV-Suspension (100.000 × g) wird 60 min lang bei 4 °C zentrifugiert.
HINWEIS: DOC sollte tropfenweise hinzugefügt werden. - Suspendieren Sie die Pellets in <3 ml 25 mM Tris-HCl, pH 7,5, 0,3 M KCl, 45 % Glycerin (v/v) und 2 mM EDTA. Homogenisieren Sie die Suspension und stellen Sie sie auf ein Endvolumen von 10 mL ein. Bestimmen Sie die Proteinkonzentration und stellen Sie sie dann auf 6,5 mg/ml ein. Fügen Sie Azolectin in einer Menge von 5 mg/ml und 0,85 (w/w) Zwittergent 3-14 hinzu.
HINWEIS: Azolectin und Zwittergent 3-14 sollten tropfenweise hinzugefügt werden. - Suspension und Zentrifuge (100.000 × g) bei 4 °C für 60 min homogenisieren. Den Überstand auffangen und 1:2 mit 2 mM EGTA (pH 7,2) verdünnen.
- Gießen Sie die Suspension vorsichtig auf einen diskontinuierlichen Trehalose-Konzentrationsgradienten (45, 40, 35 und 30 % w/v) in 10 mM Tris-HCl, pH 7,0, 1 mM EDTA, 0,1 % Desoxycholat und 1 mg/ml Azolektin. Zentrifugieren (100.000 × g) bei 4 °C für 14 h. Sammeln Sie das durchsichtige, leicht gelbliche Kügelchen, das sich am Boden der Röhrchen gebildet hat.
HINWEIS: Sowohl die Bildung des Trehalose-Gradienten als auch die Zugabe der Probensuspension müssen mit geeigneten Techniken durchgeführt werden. Eine Zentrifugation über Nacht wird empfohlen. - Suspendieren Sie das Pellet vorsichtig in einem kleinen Volumen (<1,5 ml) von 25 mM Tris-HCl, pH 7,5, 0,3 M KCl, 45 % Glycerin und 2 mM EDTA. Bestimmen Sie die Proteinkonzentration. Passen Sie die Proteinkonzentration durch Verdünnung auf 2 mg/ml an. Nehmen Sie Aliquote (~50 μL) der Suspension und lagern Sie das gereinigte SERCA bei -72 °C bis zu seiner Verwendung.
- Durchführung einer SDS-PAGE des gereinigten SERCA72 und von Proteinproben, die in verschiedenen Reinigungsschritten entnommen wurden. Färben Sie das Gel mit Coomassie Blue72.
HINWEIS: Die Verwendung von 0,6 M Trehalose anstelle von Glycerin im Suspensionspuffer erhöht die Stabilität des Enzyms während der Lagerung und der Gefrier-Tau-Zyklen28,29.
3. Assay für ATPase-Aktivität (Timing, 1 - 2 h)
- Enzymgekoppelter Assay: ATPase-Piruvat-Kinase-Lactat-Dehydrogenase
- Bereiten Sie das Reaktionsgemisch (1 mL) vor: 50 mM MOPS (pH 7,0), 1 mM EGTA, 80 mM KCl, 5 mM MgCl2, 3 mM CaCl2, 5 mM Phosphoenolpyruvat, 250 μM NADH. Variable ATP-Konzentrationen (0,001 bis 0,25 mM) einschließen.
- Der Reaktionsassay wird vorsichtig durch Vortexen homogenisiert. Inkubieren Sie den Reaktionsassay 10 Minuten lang bei 37 μC. Fügen Sie 0,9 U L-Lactat-Dehydrogenase (LDH) und 1,5 U Pyruvatkinase (PK) hinzu.
- Initiieren Sie die ATPase-Reaktion durch Zugabe von 10 μg SERCA.
- Bestimme die Bildung von NADH. Die Änderung der Absorptionsintensität wird jede Sekunde für 600 s bei einer Wellenlänge (λ) von 340 nm über die Zeit mit einem Spektralphotometer mit einem thermostatisierten Küvettenhalter verfolgt.
HINWEIS: Das Zeitintervall für die Absorptionsmessung kann variieren, aber versuchen Sie, so viele Datenpunkte wie möglich aufzuzeichnen, um die anfängliche Gerade der ATPase-Reaktion zu zeichnen.
- Bestimmen Sie die Geschwindigkeit der ATP-Hydrolyse
HINWEIS: Für Einzelheiten zum Verfahren siehe Rivera-Morán et al.46.- Berechnen Sie den Steigungswert (Δabs/min) des linearen Anteils in jeder gebildeten Kurve. Verwenden Sie den molaren Extinktionskoeffizienten (ε) von NADH (λ = 6.220 M-1·cm-1), um die ATPase-Aktivität zu bestimmen (μmol ATP hydrolysiert/min.mg prot.)
- Bestimmung der Kinetikparameter K, m und Vmax
- Plotten Sie die Geschwindigkeitsdaten im Vergleich zur ATP-Konzentration mithilfe von Datenanalyse- und Grafiksoftware.
- Passen Sie die Daten durch nichtlineare Regression an die Michaelis-Menten-Gleichung (Gl. 1)46 an:
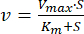
wobei v die Geschwindigkeit, Vmax die maximale Geschwindigkeit, [S] die ATP-Konzentration und Km die Michaelis-Menten-Konstante ist. - Bestimmen Sie Km und Vmax durch Iteration.
HINWEIS: Die Aufnahme von DOC kann die ATPase-Ratenreaktion erhöhen; Es wird empfohlen, verschiedene DOC-Konzentrationen zu testen, um das Optimum zu finden.
4. Markierungsassay von SERCA mit Fluorescein-Isothiocyanat (FITC) (Timing, 30 - 45 min, nur für FITC-Markierung)
HINWEIS: Einzelheiten zum Verfahren finden Sie unter 54,75,76.
- SERCA (20 μg) wird in 50 μl (Endvolumen) Markierungspuffer (100 mM KCl, 5 mM MgCl2 und 30 mM Tris-HCl, pH 8,9) mit 1 mM FITC suspendiert. Durch Vortexen mischen.
- Inkubieren Sie die Proben zu verschiedenen Zeiten (15 min, 10 min, 7 min, 5 min und 2 min) im Dunkeln und bei Raumtemperatur.
- Stoppen Sie die Markierungsreaktion durch Zugabe von 1 Volumen eiskaltem Stopppuffer (480 mM Saccharose und 48 mM MOPS, pH 7,0), der ATP (5 mM) enthält. 5 min im Dunkeln auf Eis inkubieren.
- Unterwerfen Sie die mit dem FITC-Etikett versehene SERCA SDS-PAGE72. Setzen Sie die klaren Gele UV-Licht (λ = 302 nm) aus. Fotodokumentieren Sie das Ergebnis.
- Färben Sie das Gel mit Coomassie blue. Fotodokumentieren Sie das Ergebnis.
HINWEIS: Die Durchführung der FITC-Markierung in einem dunklen Raum kann die Qualität des Fluoreszenzsignals verbessern.
5. Spektren des Zirkulardichroismus (CD) (Timing, 30 min)
HINWEIS: Einzelheiten zum Verfahren finden Sie unter11,77.
- SERCA (3 μM) in 400 μl 10 mM Phosphatpuffer (pH 7,0) bei 25 °C suspendieren.
- Laden Sie die Probe in eine Zelle mit einer Schichtdicke von 0,1 cm. Stellen Sie den Bereich der Fern-UV-Spektren λ zwischen 190 und 260 nm ein. Stellen Sie die interne Auflösung und die Bandbreite auf 1 nm ein.
- Nehmen Sie das CD-Signal mit 50 nm/min bei 25 °C auf.
Ergebnisse
SDS-PAGE von SERCA in verschiedenen Stadien der Reinigung (Abbildung 1). Das blau gefärbte Coomassie-Gel zeigt die Anreicherung der SERCA-Proteinbande (scheinbares Molekulargewicht >100 kDa) im Verlauf des Aufreinigungsprotokolls. Die Proteinbande, die SERCA entspricht, zeigt nach Zentrifugation auf einem Trehalose-Konzentrationsgradienten eine Reinheit von >90%. Diagramm der ATP-Hydrolyserate im Vergleich zur ATP-Konzentration (
Diskussion
Die meisten Moleküle und Ionen können die Zellmembranen nicht frei passieren, z. B. benötigt das Proton (H+) einen Membrantransporter in der Plasmamembran einer Vielzahl von Organismen und Organellen wie Mitochondrien83,84. Zellmembranen sind selektiv, und die Moleküle und Ionen, die die Zellmembranen durchqueren, sind vielfältig, so dass verschiedene Arten von Membranproteinen in der Zelle gefunden werden können...
Offenlegungen
Der Autor erklärt, dass er keine konkurrierenden finanziellen Interessen hat.
Danksagungen
Der Autor dankt Edmundo Mata-Morales bei der Bearbeitung des Videos, VM, Valentín de la Cruz-Torres bei der Reinigung der SRVs, Miguel A. Rivera-Moran bei der SERCA-Reinigung und -Analyse und Juan C. Gonzalez-Castro, Franco E. Juarez, Alejandra Nevarez, Nicolas Rocha-Vizuet und Jocelin I. Ramírez-Alonso bei der Videoproduktion. Für die Durchführung dieser Studie wurden keine Gelder, Zuschüsse oder andere Unterstützung erhalten.
Materialien
| Name | Company | Catalog Number | Comments |
| ATP | Sigma-Aldrich Corp | A2383 | |
| Azolectin from soybean | Sigma-Aldrich Corp | 44924 | |
| Benchtop UV transilluminator | Cole-Parmer | EW-97623-08 | Dual intensity High setting is ideal for analytical documentation. Low setting reduces photonicking or photobleaching of gel samples while doing preparative work. |
| CaCl2 • 2H2O | Sigma-Aldrich Corp | 223506 | |
| Coolpix B500 camera | Nikon Corp | S210 | |
| Coomassie brilliant blue G-250 | Bio-Rad | 1610406 | |
| Dodecyl maltoside | Sigma-Aldrich Corp | D4641 | |
| Ethylene glycol-bis(2-aminoethylether)-N,N,N′,N′-tetraacetic acid | Sigma-Aldrich Corp | E4378 | |
| Ethylenediaminetetraacetic acid tetrasodium salt dihydrate | Sigma-Aldrich Corp | E6511 | |
| Fluorescein isothiocyanate | Sigma-Aldrich Corp | F3651 | |
| KCl | JT Baker | 7447-40-7 | |
| MgCl2 • 6H2O | Sigma-Aldrich Corp | M9272 | |
| MOPS | Sigma-Aldrich Corp | M1254 | |
| NADH | Chem-Impex International Inc | 230 | |
| N-tetradecyl-N,N- dimethyl-3-ammonium-1-propanesulfonate | Sigma-Aldrich Corp | D0431 | |
| Phosphoenolpyruvate (PEP) | Chem-Impex International Inc | 9711 | |
| Rabbit muscle lactate dehydrogenase | Roche | 10003557103 | |
| Rabbit muscle pyruvate kinase | Sigma-Aldrich Corp | P1506 | |
| Sodium deoxycholate | Sigma-Aldrich Corp | D6750 | |
| Sodium dodecyl sulfate | Bio-Rad | 1610302 | |
| Spectropolarimeter | Jasco Corp. | Jasco J1500 | |
| Sucrose | Sigma-Aldrich Corp | 84100 | |
| Trehalose | Sigma-Aldrich Corp | T0167 | Dihydrate |
| Tris(hydroxymethyl)aminomethane hydrochloride | Sigma-Aldrich Corp | 857645 | |
| Ultracentrifuge | Beckman | Optima XPN | |
| UV/VIS spectrophotometer | Agilent Technologies | 8453 | The Agilent 8453 UV-Vis Spectrophotometer uses a photodiode array for simultaneous measurement of the complete ultra-violet to visible light spectrum |
| WiseStir HS- 30E | Daihan Scientific Co. | DH.WOS01010 | Ideal for all disperging and homogenizing applications, designed for tissue grinders. |
Referenzen
- Schuberth, C., Wedlich-Söldner, R. Building a patchwork - The yeast plasma membrane as model to study lateral domain formation. Biochim Biophys Acta Mol Cell Res. 1853 (4), 767-774 (2015).
- Douglas, L. M., Konopka, J. B. Fungal membrane organization: The eisosome concept. Annu Rev Microbiol. 68 (1), 377-393 (2014).
- Borrell, J. H., Domènech, &. #. 2. 1. 0. ;., Keough, K. M. W. . Membrane protein - Lipid interactions: Physics and Chemistry in the Bilayer. , (2016).
- Moraes, I., Evans, G., Sanchez-Weatherby, J., Newstead, S., Stewart, P. D. . The next generation in membrane protein structure determination. , (2016).
- White, S. H. Biophysical dissection of membrane proteins. Nature. 459 (7245), 344-346 (2009).
- Lacapère, J. -. J. Membrane protein structure determination. Methods Mol Biol. , 459 (1002).
- Palmgren, M. G. Plant plasma membrane H+-ATPases: Powerhouses for nutrient uptake. Annu Rev Plant Physiol Plant Mol Biol. 52 (1), 817-845 (2001).
- Yatime, L., et al. P-type ATPases as drug targets: Tools for medicine and science. Biochim Biophys Acta Bioenerg. 1787 (4), 207-220 (2009).
- Drews, J. Tetrahydrobiopterin biosynthesis as an off-target of sulfa drugs. Science. 287 (5460), 1960-1964 (2000).
- Xu, Z., Meshcheryakov, V. A., Poce, G., Chng, S. -. S. MmpL3 is the flippase for mycolic acids in mycobacteria. Proc Natl Acad Sci USA. 114 (30), 7993-7998 (2017).
- Siligardi, G., Hussain, R., Patching, S. G., Phillips-Jones, M. K. Ligand- and drug-binding studies of membrane proteins revealed through circular dichroism spectroscopy. Biochim Biophys Acta Biomembr. 1838 (1 PARTA), 34-42 (2014).
- Rawlings, A. E. Membrane proteins: Always an insoluble problem. Biochem Soc Trans. 44 (3), 790-795 (2016).
- Popot, J. -. L. . Membrane proteins in aqueous solutions. , 708 (2018).
- Palmgren, M. G., Sommarin, M., Ulvskov, P., Larsson, C. Effect of detergents on the H+-ATPase activity of inside-out and right-side-out plant plasma membrane vesicles. Biochim Biophys Acta Biomembr. 1021 (2), 133-140 (1990).
- McBride, Z., Chen, D., Reick, C., Xie, J., Szymanski, D. B. Global analysis of membrane-associated protein oligomerization using protein correlation profiling. Mol Cell Proteomics. 16 (11), 1972-1989 (2017).
- Yoneda, J. S., et al. Multimeric species in equilibrium in detergent-solubilized Na, K-ATPase. Int J Biol Macromol. 89, 238-245 (2016).
- Ohlendieck, K. Extraction of membrane proteins. Protein Purif Protocols. 59, 283-294 (2004).
- Champeil, P., et al. A robust method to screen detergents for membrane protein stabilization, revisited. Anal Biochem. 511, 31-35 (2016).
- Gimpl, K., Klement, J., Keller, S. Characterising protein/detergent complexes by triple-detection size-exclusion chromatography. Biol Proced Online. 18, 4 (2016).
- le Maire, M., et al. Gel chromatography and analytical ultracentrifugation to determine the extent of detergent binding and aggregation, and Stokes radius of membrane proteins using sarcoplasmic reticulum Ca2+-ATPase as an example. Nat Protoc. 3 (11), 1782-1795 (2008).
- Du, M., et al. Progress, applications, challenges and prospects of protein purification technology. Front Bioeng Biotechnol. 10, 1-26 (2022).
- Slotboom, D. J., Duurkens, R. H., Olieman, K., Erkens, G. B. Static light scattering to characterize membrane proteins in detergent solution. Methods. 46 (2), 73-82 (2008).
- Harding, S. E. Analytical ultracentrifugation as a matrix-free probe for the study of kinase related cellular and bacterial membrane proteins and glycans. Molecules. 26, 6080 (2021).
- Chen, Y. -. C., et al. Thermal stability, storage and release of proteins with tailored fit in silica. Sci Rep. 7 (1), 46568 (2017).
- Montigny, C. C., Arnou, B., Marchal, E., Champeil, P. Use of glycerol-containing media to study the intrinsic fluorescence properties of detergent-solubilized native or expressed SERCA1a. Biochemistry. 47 (46), 12159-12174 (2008).
- Simongini, M., Puglisi, A., Genovese, F., Hochkoeppler, A. Trehalose counteracts the dissociation of tetrameric rabbit lactate dehydrogenase induced by acidic pH conditions. Arch Biochem Biophys. 740, 109584 (2023).
- Shivanna, B. D., Rowe, E. S. Preservation of the native structure and function of Ca2+-ATPase from sarcoplasmic reticulum: Solubilization and reconstitution by new short-chain phospholipid detergent 1,2-diheptanoyl-sn-phosphatidylcholine. Biochem J. 325 (2), 533-542 (1997).
- Sampedro, J. G., Guerra, G., Pardo, J. P., Uribe, S. Trehalose-mediated protection of the plasma membrane H+-ATPase from Kluyveromyces lactis during freeze-drying and rehydration. Cryobiology. 37 (2), 131-138 (1998).
- Sampedro, J. G., Cortés, P., Muñoz-Clares, R. A., Fernández, A., Uribe, S. Thermal inactivation of the plasma membrane H+-ATPase from Kluyveromyces lactis. Protection by Trehalose. Biochim Biophys Acta Proteins Proteomics. 1544 (1-2), 64-73 (2001).
- Sampedro, J. G., et al. Fluorescence quenching by nucleotides of the plasma membrane H+-ATPase from Kluyveromyces lactis. Biochemistry. 46 (18), 5616-5622 (2007).
- Ruiz-Granados, Y. G., De La Cruz-Torres, V., Sampedro, J. G. The oligomeric state of the plasma membrane H+-ATPase from Kluyveromyces lactis. Molecules. 24 (5), 958 (2019).
- Zhao, P., Zhao, C., Chen, D., Yun, C., Li, H., Bai, L. Structure and activation mechanism of the hexameric plasma membrane H+-ATPase. Nat Commun. 12 (1), 6439 (2021).
- Auer, M., Scarborough, G. A., Kühlbrandt, W. Surface crystallization of the plasma membrane H+-ATPase on a carbon support film for electron crystallography. J Mol Biol. 287 (5), 961-968 (1999).
- Hennessey, J. P., Scarborough, G. A. Secondary structure of the Neurospora CRASSA plasma membrane H+-ATPase as estimated by circular dichroism. J Biol Chem. 263 (7), 3123-3130 (1988).
- Scarborough, G. A. Crystallization, structure and dynamics of the proton-translocating P-type ATPase. J Exp Biol. 203 (Pt 1), 147-154 (2000).
- Sumida, K. H., et al. Improving protein expression, stability, and function with proteinMPNN. J Am Chem Soc. 146 (3), 2054-2061 (2024).
- Eleutherio, E., Panek, A., De Mesquita, J. F., Trevisol, E., Magalhães, R. Revisiting yeast trehalose metabolism. Curr Genet. 61 (3), 263-274 (2015).
- Attfield, P. V. Trehalose accumulates in Saccharomyces cerevisiae during exposure to agents that induce heat shock response. FEBS Lett. 225 (1-2), 259-263 (1987).
- Marunde, M. R., et al. Improved tolerance to salt and water stress in Drosophila melanogaster cells conferred by late embryogenesis abundant protein. J Insect Physiol. 59 (4), 377-386 (2013).
- Crowe, J. H. Anhydrobiosis: An unsolved problem with applications in human welfare. Subcell Biochem. 71, 263-280 (2015).
- Crowe, J. H., Crowe, L. M., Chapman, D. Preservation of membranes in anhydrobiotic organisms: The role of trehalose. Science. 223 (4637), 701-703 (1984).
- Sampedro, J. G., Rivera-Moran, M. A., Uribe-Carvajal, S. Kramers' theory and the dependence of enzyme dynamics on trehalose-mediated viscosity. Catalysts. 10 (6), 1-19 (2020).
- Sampedro, J. G., Uribe, S. Trehalose-enzyme interactions result in structure stabilization and activity inhibition. The role of viscosity. Mol Cell Biochem. 256 - 257 (1-2), 319-327 (2004).
- Crowe, J. H. Trehalose as a "chemical chaperone": Fact and fantasy. Adv Exp Med Biol. 594, 143-158 (2007).
- Crowe, J. H., Carpenter, J. F., Crowe, L. M., Anchordoguy, T. J. Are freezing and dehydration similar stress vectors? A comparison of modes of interaction of stabilizing solutes with biomolecules. Cryobiology. 27 (3), 219-231 (1990).
- Rivera-Morán, M. A., Sampedro, J. G. Isolation of the sarcoplasmic reticulum Ca2+-ATPase from rabbit fast-twitch muscle. Methods Protoc. 6 (5), 102 (2023).
- Páez-Pérez, E. D., De La Cruz-Torres, V., Sampedro, J. G. Nucleotide binding in an engineered recombinant Ca2+-ATPase N-domain. Biochemistry. 55 (49), 6751-6765 (2016).
- Ramírez-Alonso, J. I., Sampedro, J. G. Effect of cations on ATP binding to the N-domain of Na+, K+-ATPase. J Fluoresc. , (2024).
- De la Cruz-Torres, V., Cataño, V., Olivo-Rodríguez, M., Sampedro, J. G. ANS Interacts with the Ca2+-ATPase nucleotide binding site. J Fluoresc. 30 (3), 483-496 (2020).
- Sampedro, J. G., Nájera, H., Uribe-Carvajal, S., Ruiz-Granados, Y. G. Mapping the ATP binding site in the plasma membrane H+-ATPase from Kluyveromyces lactis. J Fluoresc. 24 (6), 1849-1859 (2014).
- Kiani, A. K., et al. Ethical considerations regarding animal experimentation. J Prev Med Hyg. 63 (2 Suppl 3), E255-E266 (2022).
- Ruiz-Granados, Y., De La Cruz-Torres, V., Sampedro, J. The oligomeric state of the plasma membrane H+-ATPase from Kluyveromyces lactis. Molecules. 24 (5), 958 (2019).
- Wang, S., et al. Structural basis for sarcolipin's regulation of muscle thermogenesis by the sarcoplasmic reticulum Ca2+-ATPase. Sci Adv. 7 (48), eabi7154 (2021).
- Champeil, P., et al. ATP regulation of sarcoplasmic reticulum Ca2+-ATPase. Metal-free ATP and 8-bromo-ATP bind with high affinity to the catalytic site of phosphorylated ATPase and accelerate dephosphorylation. J Biol Chem. 263 (25), 12288-12294 (1988).
- Møller, J. V., Olesen, C. Preparation of Ca(2+)-ATPase1a enzyme from rabbit sarcoplasmic reticulum. Methods Mol Biol. 1377, 11-17 (2016).
- Wuytack, F., De Schutter, G., Casteels, R. Partial purification of (Ca2+ + Mg2+)-dependent ATPase from pig smooth muscle and reconstitution of an ATP-dependent Ca2+-transport system. Biochem J. 198 (2), 265-271 (1981).
- Mandal, A., et al. Solubilization, purification and reconstitution of Ca2+-ATPase from bovine pulmonary artery smooth muscle microsomes by different detergents: Preservation of native structure and function of the enzyme by DHPC. Biochim Biophys Acta Gen Subj. 1760 (1), 20-31 (2006).
- Inesi, G., Ma, H., Hua, S., Toyoshima, C. Characterization of Ca2+ ATPase residues involved in substrate and cation binding. Ann NY Acad Sci. 986 (1), 63-71 (2003).
- Van Winkle, W. B., Pitts, B. J., Entman, M. L. Rapid purification of canine cardiac sarcoplasmic reticulum Ca2+-ATPase. J Biol Chem. 253 (24), 8671-8673 (1978).
- Ruiz-Granados, Y. G., De La Cruz-Torres, V., Sampedro, J. G. The oligomeric state of the plasma membrane H+-ATPase from Kluyveromyces lactis. Molecules. 24 (5), 958 (2019).
- Teramoto, N., Sachinvala, N. D., Shibata, M. Trehalose and trehalose-based polymers for environmentally benign, biocompatible, and bioactive materials. Molecules. 13 (8), 1773-1816 (2008).
- Dao, T. T., et al. Demethoxycurcumin is a potent inhibitor of P-type ATPases from diverse kingdoms of life. PLOS ONE. 11 (9), e0163260 (2016).
- Bleeker, N. P., Cornea, R. L., Thomas, D. D., Xing, C. A novel SERCA inhibitor demonstrates synergy with classic SERCA inhibitors and targets multidrug-resistant AML. Mol Pharmaceutics. 10 (11), 4358-4366 (2013).
- Michelangeli, F., East, J. M. A diversity of SERCA Ca2+ pump inhibitors. Biochem Soc Trans. 39 (3), 789-797 (2011).
- Tadini-Buoninsegni, F., Smeazzetto, S., Gualdani, R., Moncelli, M. R. Drug interactions with the Ca2+-ATPase from Sarco(Endo)Plasmic Reticulum (SERCA). Front Mol Biosci. 5, 36 (2018).
- Clausen, J. D., et al. Elucidation of antimicrobial activity and mechanism of action by N-substituted carbazole derivatives. Bioorg Med Chem Lett. 27 (19), 4564-4570 (2017).
- Leary, S., et al. . AVMA guidelines for the euthanasia of animals: 2020 Edition, Members of the Panel on Euthanasia AVMA Staff Consultants. , (2020).
- de Aluja, A. S. Laboratory animals and official Mexican norms (NOM-062-ZOO-1999). Gac Med Mex. 138 (3), 295-298 (2002).
- Champeil, P., Buschlen-Boucly, S., Bastide, F., Gary-Bobo, C. Sarcoplasmic reticulum ATPase. Spin labeling detection of ligand-induced changes in the relative reactivities of certain sulfhydryl groups. J Biol Chem. 253 (4), 1179-1186 (1978).
- Skalec, A., Janeczek, M., Czerski, A. Anatomy and histology of the rabbit common calcanean tendon. J Vet Med Series C: Anat Histol Embryol. 48 (5), 466-475 (2019).
- Janes, L. E., Mioton, L. M., Fracol, M. E., Ko, J. H. An in vivo comparison: Novel mesh suture versus traditional suture-based repair in a rabbit tendon model. J Hand Surg Glob Online. 4 (1), 32-39 (2022).
- Bollag, D. M., Rozycki, M. D., Edelstein, S. J. . Protein Methods. , (1996).
- Gagné, F. Tissue preparation and subcellular fractionation techniques. Biochem Ecotoxicol. , 21-31 (2014).
- Lowry, O. H., Rosebrough, N. J., Farr, A. L., Randall, R. J. Protein measurement with the Folin phenol reagent. J Biol Chem. 193 (1), 265-275 (1951).
- Autry, J. M., Rubin, J. E., Svensson, B., Li, J. L., Thomas, D. D. Nucleotide activation of the Ca-ATPase. J Biol Chem. 287 (46), 39070-39082 (2012).
- Winters, D. L., Autry, J. M., Svensson, B., Thomas, D. D. Interdomain fluorescence resonance energy transfer in SERCA probed by cyan-fluorescent protein fused to the actuator domain. Biochemistry. 47 (14), 4246-4256 (2008).
- Miles, A. J., Wallace, B. A. Circular dichroism spectroscopy of membrane proteins. Chem Soc Rev. 14 (45), 1130-1135 (2016).
- Scofano, H. M., Vieyra, A., de Meis, L. Substrate regulation of the sarcoplasmic reticulum ATPase. Transient kinetic studies. J Biol Chem. 254 (20), 10227-10231 (1979).
- Yamamoto, T., Tonomura, Y. Reaction mechanism of the Ca++-dependent ATPase of sarcoplasmic reticulum from skeletal muscle: I. Kinetic studies. J Biochem. 62 (5), 558-575 (1967).
- Murphy, A. J. Affinity labeling of the active site of the Ca2+-ATPase of sarcoplasmic reticulum. Biochim Biophys Acta - Biomembr. 946 (1), 57-65 (1988).
- Csermely, P., Katopis, C., Wallace, B. A., Martonosi, A. The E1→E2 transition of Ca2+-transporting ATPase in sarcoplasmic reticulum occurs without major changes in secondary structure. A circular-dichroism study. Biochem J. 241 (3), 663-669 (1987).
- Abràmoff, M. D., Magalhães, P. J., Ram, S. J. Image processing with ImageJ. Biophotonics Int. 11 (7), 36-42 (2004).
- Scarborough, G. A. The plasma membrane proton-translocating ATPase. Cell Mol Life Sci. 57 (6), 871-883 (2000).
- Llopis, J., McCaffery, J. M., Miyawaki, A., Farquhar, M. G., Tsien, R. Y. Measurement of cytosolic, mitochondrial, and Golgi pH in single living cells with green fluorescent proteins. Proc Natl Acad Sci. 95 (12), 6803-6808 (1998).
- Kanai, R., Vilsen, B., Cornelius, F., Toyoshima, C. Crystal structures of Na+, K+-ATPase reveal the mechanism that converts the K+-bound form to Na+-bound form and opens and closes the cytoplasmic gate. FEBS Lett. 597 (15), 1957-1976 (2023).
- Palmgren, M. P-type ATPases: Many more enigmas left to solve. J Biol Chem. 299 (11), 105352 (2023).
- Frauenfelder, H. Ask not what physics can do for biology-Ask what biology can do for physics. Phys Biol. 11 (5), 053004 (2014).
- Frauenfelder, H. The physics of proteins. Annu Rev Biophys Biomol Struct. 36, (2010).
- Narhi, L. O. . Biophysics for Therapeutic Protein Development. , (2013).
- East, J. M. Purification of a membrane protein (Ca2+/Mg2+-ATPase) and its reconstitution into lipid vesicles. Methods Mol Biol. 27, 87-94 (1994).
- Lee, E. -. H., et al. Enhancement of enzyme activity and stability by poly(γ-glutamic acid). Polymer J. 42 (10), 818-822 (2010).
- Norrild, R. K., et al. Increasing protein stability by inferring substitution effects from high-throughput experiments. Cell Rep Methods. 2 (11), 100333 (2022).
- Song, Z., Zhang, Q., Wu, W., Pu, Z., Yu, H. Rational design of enzyme activity and enantioselectivity. Front Bioeng Biotechnol. 11, 1-14 (2023).
- Jidenko, M., Lenoir, G., Fuentes, J. M., le Maire, M., Jaxel, C. Expression in yeast and purification of a membrane protein, SERCA1a, using a biotinylated acceptor domain. Protein Expr Purif. 48 (1), 32-42 (2006).
- Hua, S., Ma, H., Lewis, D., Inesi, G., Toyoshima, C. Functional role of "N" (nucleotide) and "P" (phosphorylation) domain interactions in the sarcoplasmic reticulum (SERCA) ATPase. Biochemistry. 41 (7), 2264-2272 (2002).
- Pang, Y. -. H., Chen, J. -. W. Anisodamine causes the changes of structure and function in the transmembrane domain of the Ca2+-ATPase from sarcoplasmic reticulum. Biosci Biotechnol Biochem. 68 (1), 126-131 (2004).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten