Se requiere una suscripción a JoVE para ver este contenido. Inicie sesión o comience su prueba gratuita.
Method Article
Purificación del retículo sarcoendoplásmico Ca2+-ATPasa a partir de músculo de conejo
En este artículo
Resumen
Este protocolo describe un método de purificación SERCA mejorado, que incluye el disacárido trehalosa en el paso final de centrifugación. Este carbohidrato estabiliza las proteínas en condiciones adversas. El SERCA purificado era catalíticamente activo y mostraba una alta pureza, lo que lo hacía adecuado para estudios estructurales y funcionales.
Resumen
Algunas ATPasas de tipo P, como la Ca2+-ATPasa del retículo sarco/endoplásmico (SERCA), son proteínas de membrana inherentemente lábiles que requieren condiciones fisicoquímicas específicas durante la purificación para obtenerlas con alta pureza y calidad estructural y en una forma catalíticamente activa. El disacárido trehalosa es un soluto compatible que se sintetiza y acumula en altas concentraciones en el citoplasma de la levadura para estabilizar las membranas y las proteínas. El uso de trehalosa como aditivo en el protocolo para la purificación de la membrana plasmática H+-ATPasa da como resultado una preparación de alta calidad, cuya estructura hexámera se muestra mediante métodos analíticos bioquímicos. Por lo tanto, la trehalosa puede utilizarse como aditivo estabilizador para la purificación de proteínas de membrana (P-ATPasas). Este protocolo describe la modificación del protocolo clásico para la purificación de SERCA mediante el sometimiento de SERCA a centrifugación en un gradiente de concentración de trehalosa. La inclusión de este carbohidrato condujo a la purificación de SERCA en una forma catalíticamente activa con alta pureza y, lo que es más importante, en una forma estable. La caracterización bioquímica parcial del SERCA purificado (SDS-PAGE, cinética de la enzima, marcaje FITC, espectroscopia de dicroísmo circular) mostró que la enzima es adecuada para estudios funcionales y estructurales. Se sugiere el uso de trehalosa en el protocolo de purificación de ATPasas de tipo P y otras proteínas de membrana lábil (y citosólicas).
Introducción
Las proteínas/enzimas de membrana son componentes biológicos esenciales de las células, ya que desempeñan un papel fundamental en diversos procesos 1,2,3. Algunas de las funciones pueden incluir el transporte de iones y moléculas dentro y fuera de la célula/compartimentos internos (ya sea activo/pasivo), reconocimiento célula-célula, unión intercelular, anclaje/unión y detección del entorno externo a través de la integración con la maquinaria de transducción de señales en condiciones físicas y químicas normales y duras (alta sal, poca agua, alta temperatura, resistencia a los medicamentos, etc.)3 Por lo tanto, la determinación de la estructura tridimensional (3D) de las proteínas y/o enzimas de membrana se ha vuelto de gran importancia tanto para la investigación básica como para la aplicada 4,7,6. Es importante destacar que las proteínas/enzimas de membrana se han utilizado ampliamente como objetivos para el descubrimiento de fármacos (ya sea natural o por diseño)7,8,9. Es decir, las proteínas de membrana tienen una importancia inherente en la salud 9,10,11.
El carácter hidrofóbico de las proteínas/enzimas de membrana es la propiedad fisicoquímica técnicamente más desafiante para el laboratorio experimental 12,13,14, más aún cuando se trabaja con proteínas de membrana integrales oligoméricas y/o altamente lábiles 15,16. Es muy deseable el aislamiento de cantidades apropiadas de la proteína/enzima de membrana con la mayor calidad posible para ensayos experimentales funcionales y estudios estructurales17. Las proteínas de membrana, al ser inherentemente hidrofóbicas, son muy difíciles de purificar, y la elección del detergente suele ser una de las cuestiones más importantes a tener en cuenta 17,18,19,20. En este sentido, los métodos de laboratorio utilizados para aislar las proteínas de membrana suelen causar algún grado de daño en la disposición estructural 3D de la proteína21. Algunos de estos métodos incluyen el uso de (a) sonicación (ondas ultrasónicas de alta frecuencia/energía), (b) detergentes solubilizadores de proteínas (ya sean fuertes, medios o suaves)22, (c) presiones relativamente altas en cromatografía en columna y ultracentrifugación de alta velocidad23, (d) moléculas precipitantes, (e) enzimas digestivas y otros21. Todos estos procesos pueden contribuir o ser la causa principal de la desestabilización de proteínas durante la purificación21. En este sentido, algunos protocolos parecen funcionar relativamente bien para una proteína de membrana determinada. Sin embargo, la optimización siempre es bienvenida cuando se utilizan nuevos ensayos modernos o métodos que requieren una mayor calidad de preparación de proteínas de membrana para obtener resultados satisfactorios24. Los pasos de optimización pueden incluir, entre otros, mejorar el diseño del constructo, encontrar las condiciones óptimas para la expresión de proteínas de membrana, establecer mejores condiciones de manejo (es decir, pH, temperatura, etc.), encontrar el detergente más compatible, ajustar los pasos de purificación como el tiempo de sonicación, la velocidad de centrifugación, reformular las soluciones tampón mediante la adición de agentes estabilizadores, etc.25,26,27. Por lo tanto, es importante cualquier cambio en la metodología de purificación que conduzca a un aumento en la calidad (pureza) y la actividad de la proteína/enzima de membrana purificada.
En la familia de las ATPasas de tipo P, la membrana plasmática de levadura H+-ATPasa parece ser uno de los miembros más lábiles28,29. La H+-ATPasa pierde su actividad hidrolizante de ATP tras la deshidratación (liofilización), choque térmico, etc.28,29. El uso de trehalosa como estabilizador de proteínas se probó en el aislamiento de la membrana plasmática H+-ATPasa de la levadura K. lactis30,31; la preparación de H+-ATPasa obtenida fue de alta pureza y catalíticamente activa. Es importante destacar que esto permitió resolver el estado oligomérico de la enzima por métodos bioquímicos, revelando que se trataba de un hexámero y posteriormente confirmado por criomicroscopía electrónica 31,32,33,34,35. Por lo tanto, parece probable que la delicada disposición tridimensional (3D) de las proteínas de membrana pueda perderse en condiciones relativamente duras durante la purificación36. Se observa una cinética de inactivación bifásica para la H+-ATPasa durante la inactivación mediada por calor29. En las células de levadura, como en muchos otros organismos, el disacárido trehalosa se acumula en altas concentraciones en condiciones de estrés ambiental 37,38,39. La trehalosa mantiene la integridad de la membrana y la función de transporte mediante la estabilización de las proteínas (tanto incluidas en la membrana como citosólicas) y las membranas celulares40,41. El mecanismo estabilizador de la trehalosa ha sido ampliamente estudiado por varios grupos y por nuestro laboratorio 42,43,44,45. Los experimentos con la H+-ATPasa y otras enzimas han demostrado que la trehalosa es el estabilizador proteico más eficaz entre los monosacáridos y disacáridos28,29. Esto llevó a su inclusión en el protocolo de purificación de H+-ATPasa30. Recientemente, la trehalosa también se ha utilizado en la purificación del retículo sarcoplásmico Ca2+-ATPasa (SERCA) del músculo de contracción rápida de conejo con buenos resultados en pureza proteica y actividad46. Por lo tanto, la trehalosa parece ser un aditivo bueno y apropiado para la purificación de ATPasas de tipo P y probablemente otras proteínas de membrana y citosólicas.
En el caso de las P-ATPasas, la existencia de dominios citoplasmáticos estructurales es una ventaja experimental, especialmente para los estudios de interacción sustrato/ligando47; La unión de ATP se ha estudiado en dominios N recombinantes de alta pureza 47,48,49, eliminando así las consideraciones técnicas para la purificación de enzimas de membrana completa 47,48,50, entre otras51. Desafortunadamente, algunos estudios funcionales (catalíticos/conversión de energía) y estructurales (disposición de subunidades e interacción con otras proteínas) aún requieren la P-ATPasa52,53 completa. En este sentido, la purificación de SERCA ha sido lograda por varios grupos de investigación 54,55,56,57,58. Sin embargo, aún se pueden implementar mejoras46, por ejemplo, aumentar la integridad de la ATPasa59 purificada, evitar la desnaturalización/disrupción de los complejos proteicos25, aumentar la solubilización sin desnaturalización de la proteína de membrana (es decir, evitar la formación de agregados macromoleculares)60, conducir a una mejor compatibilidad con ensayos y otros métodos analíticos posteriores61 Además, a medida que aparecen en la literatura científica nuevas estrategias experimentales, aditivos, inhibidores enzimáticos, etc. 61,62,63,64,65,66, en ocasiones es necesario probarlas con la P-ATPasa completa. En este trabajo se describe el protocolo para la purificación de SERCA y el uso de trehalosa como aditivo para estabilizar la estructura proteica y la actividad de la ATPasa; Es decir, además de aumentar la calidad (estructural) de la enzima, la trehalosa ayuda a prevenir la pérdida de estructura y actividad enzimática durante el aislamiento y almacenamiento de la enzima, lo que ayuda a ahorrar material biológico y, por lo tanto, a reducir el número de purificaciones enzimáticas.
Protocolo
Todos los procedimientos con animales se realizaron de acuerdo con los lineamientos internacionales y locales (NORMA Oficial Mexicana NOM-062-ZOO-1999) para el manejo de animales en laboratorios experimentales 51,67,68. El tejido muscular se obtuvo de Oryctolagus cuniculus de tipo silvestre de una unidad local de manejo de animales (INE/CITES/DGVS-ZOO-E0055-SLP-98)46. Un veterinario con experiencia en el manejo de animales de laboratorio realizó la disección y el procesamiento muscular inicial. Los detalles de los reactivos y el equipo utilizado en este estudio se enumeran en la Tabla de Materiales.
1. Aislamiento del retículo sarcoplásmico del músculo de conejo de contracción rápida (Temporización, 7 - 9 h)
NOTA: Para más detalles sobre el procedimiento, consultar Champeil et al.69.
- Obtenga tejido muscular de contracción rápida (~70 g de ambas patas traseras por animal) del Oryctolagus cuniculus de tipo salvaje.
NOTA: Se requiere la identificación visual previa de los músculos a diseccionar70,71. - Macerar el músculo de contracción rápida en una licuadora suspendiéndolo en tres volúmenes de 100 mM de KCl, con 1 min encendido y 1 min en reposo helado, repetir tres veces72.
- Eliminar los restos de tejido por centrifugación (1500 × g) a 4 °C durante 5 min; Si es necesario, se realiza una segunda centrifugación (4200 × g).
- Recoger el sobrenadante y homogeneizarlo con una trituradora de tejidos (10 golpes a baja velocidad, es decir, 500-1.500 rpm)72,73. Centrifugar el homogeneizado (10.000 × g) a 4 °C durante 15 min.
NOTA: Se debe tener cuidado al homogeneizar, ya que el uso de una velocidad relativamente alta puede provocar daños en el sistema y en el operador. - Centrifugar el sobrenadante (que contiene el retículo sarcoplásmico) (33.000 × g) a 4 °C durante 120 min.
- Suspender los pellets en 40 mL de 0,5 M de sacarosa y centrifugar (12.000 × g) a 4 °C durante 15 min. Diluir el sobrenadante (0,6 M de KCl y 0,15 M de sacarosa) y centrifugar la suspensión (34.000 × g) a 4 °C durante 165 min.
- Suspenda el pellet que contiene las hojas de las vesículas del retículo sarcoplásmico (SRV) en 0,3 M de sacarosa, 0,1 M de KCl y 5 mM de Tris-HCl, pH 7,0. Determine la concentración de proteínas utilizando el ensayo de Lowry74 y la albúmina sérica humana como patrón de proteína.
- Alícuota los SRV (30-32 mg/mL) en volumen de 1 mL. Almacene las muestras de SRV a -72 °C para su posterior procesamiento.
NOTA: El rendimiento de proteína es de 100-110 mg SRV por animal.
2. Purificación SERCA (Temporización, 18 - 20 h)
NOTA: Para más detalles sobre el procedimiento, consultar Rivera-Morán et al.46.
- Diluya la suspensión de SRV a 2 mg de proteína/mL usando 75 mM de Tris-HCl helado, pH 7.2, 0.6 M de KCl, 6 mM de EDTA, 1 mM de EGTA y 0.1% (p/v) de desoxicolato (DOC). Incubar la suspensión de SRV en hielo durante 10 minutos y agitar suavemente. Centrifugar la suspensión del SRV (100.000 × g) a 4 °C durante 60 min.
NOTA: El DOC debe agregarse gota a gota. - Suspenda los gránulos en <3 mL de 25 mM de Tris-HCl, pH 7.5, 0.3 M de KCl, 45% de glicerol (v/v) y 2 mM de EDTA. Homogeneizar la suspensión y ajustar a un volumen final de 10 mL. Determine la concentración de proteínas y luego ajústela a 6.5 mg/mL. Añadir azolectina a 5 mg/mL y Zwittergent 3-14 (p/p) 0,85 (p/p).
NOTA: Azolectin y Zwittergent 3-14 deben agregarse gota a gota. - Homogeneizar la suspensión y la centrífuga (100.000 × g) a 4 °C durante 60 min. Recoger el sobrenadante y diluir 1:2 con 2 mM de EGTA (pH 7,2).
- Vierta la suspensión suavemente sobre un gradiente de concentración de trehalosa discontinuo (45, 40, 35 y 30% p/v) en 10 mM de Tris-HCl, pH 7,0, 1 mM de EDTA, 0,1% de desoxicolato y 1 mg/mL de azolectina. Centrífuga (100.000 × g) a 4 °C durante 14 h. Recoja la bolita transparente y ligeramente amarillenta que se forma en la parte inferior de los tubos.
NOTA: Tanto la formación del gradiente de trehalosa como la adición de la suspensión de la muestra deben realizarse utilizando técnicas adecuadas. Se recomienda la centrifugación durante la noche. - Suspenda suavemente el pellet en un pequeño volumen (<1,5 mL) de 25 mM de Tris-HCl, pH 7,5, 0,3 de M KCl, 45% de glicerol y 2 mM de EDTA. Determinar la concentración de proteínas. Ajustar la concentración de proteínas por dilución a 2 mg/mL. Tome alícuotas (~50 μL) de la suspensión y almacene el SERCA purificado a -72 °C hasta su uso.
- Realizar SDS-PAGE del SERCA72 purificado, y de muestras de proteínas tomadas en diferentes etapas de purificación. Tiñe el gel con Coomassie Blue72.
NOTA: El uso de trehalosa 0,6 M en lugar de glicerol en el tampón de suspensión aumenta la estabilidad de la enzima durante los ciclos de almacenamiento y congelación-descongelación28,29.
3. Ensayo de la actividad de la ATPasa (Temporización, 1 - 2 h)
- Ensayo acoplado a enzimas: ATPasa-piruvato quinasa-lactato deshidrogenasa
- Prepare la mezcla de reacción (1 mL): 50 mM MOPS (pH 7,0), 1 mM EGTA, 80 mM KCl, 5 mM MgCl2, 3 mM CaCl2, 5 mM fosfoenolpiruvato, 250 μM NADH. Incluyen concentraciones variables de ATP (0,001 a 0,25 mM).
- Homogeneizar cuidadosamente el ensayo de reacción por vórtice. Incubar el ensayo de reacción a 37 ◦C durante 10 min. Añadir 0,9 U de L-lactato deshidrogenasa (LDH) y 1,5 U de piruvato quinasa (PK).
- Iniciar la reacción de ATPasa mediante la adición de 10 μg de SERCA.
- Determinar la formación de NADH. Siga el cambio en la intensidad de la absorbancia cada segundo durante 600 s a una longitud de onda (λ) de 340 nm a lo largo del tiempo utilizando un espectrofotómetro con un soporte de celda termostático.
NOTA: El intervalo de tiempo para la medición de la absorbancia puede variar, pero intente registrar tantos puntos de datos como sea posible para dibujar la línea recta inicial de la reacción de ATPasa.
- Determinar la tasa de hidrólisis de ATP
NOTA: Para más detalles sobre el procedimiento, consultar Rivera-Morán et al.46.- Calcule el valor de la pendiente (Δabs/min) de la porción lineal en cada curva formada. Utilice el coeficiente de extinción molar (ε) de NADH (λ = 6,220 M−1·cm−1) para determinar la actividad de la ATPasa (μmoles de ATP hidrolizado/min.mg prot.)
- Determinar los parámetros cinéticos K,m y Vmax
- Trace los datos de velocidad frente a la concentración de ATP utilizando software de análisis de datos y gráficos.
- Ajuste los datos por regresión no lineal a la ecuación de Michaelis-Menten (Ec. 1)46:
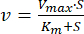
donde v es la velocidad, Vmax es la velocidad máxima, [S] es la concentración de ATP y Km es la constante de Michaelis-Menten. - Determine K, m y Vmax por iteración.
NOTA: La inclusión de DOC puede aumentar la respuesta de la tasa de ATPasa; Se recomienda probar diferentes concentraciones de DOC para encontrar el óptimo.
4. Ensayo de marcaje de SERCA con isotiocianato de fluoresceína (FITC) (Tiempo, 30 - 45 min, solo para el etiquetado FITC)
NOTA: Para obtener detalles sobre el procedimiento, consulte 54,75,76.
- Suspender SERCA (20 μg) en 50 μL (volumen final) de tampón de etiquetado (100 mM de KCl, 5 mM de MgCl2 y 30 mM de Tris-HCl, pH 8,9) que contenga 1 mM de FITC. Mezclar por vórtice.
- Incubar las muestras en diferentes momentos (15 min, 10 min, 7 min, 5 min y 2 min) en la oscuridad y a temperatura ambiente.
- Detenga la reacción de etiquetado añadiendo 1 volumen de tampón de parada helado (480 mM de sacarosa y 48 mM de MOPS, pH 7,0), que contenga ATP (5 mM). Incubar en hielo durante 5 minutos en la oscuridad.
- Sumeta el SERCA etiquetado por FITC a SDS-PÁGINA72. Exponga los geles transparentes a la luz ultravioleta (λ = 302 nm). Fotodocumenta el resultado.
- Tiñe el gel con azul Coomassie. Fotodocumenta el resultado.
NOTA: Realizar el marcaje FITC en una habitación oscura puede mejorar la calidad de la señal de fluorescencia.
5. Espectros de dicroísmo circular (CD) (Temporización, 30 min)
NOTA: Para obtener más información sobre el procedimiento, consulte11,77.
- Suspender SERCA (3 μM) en 400 μL de tampón fosfato de 10 mM (pH 7,0) a 25 °C.
- Cargue la muestra en una celda de 0,1 cm de longitud de trayectoria. Establezca el rango de espectros UV lejanos λ entre 190-260 nm. Establezca la resolución interna y el ancho de banda en 1 nm.
- Grabe la señal de CD a 50 nm/min a 25 °C.
Resultados
SDS-PAGE de SERCA en diferentes etapas de purificación (Figura 1). El gel teñido de azul de Coomassie muestra el enriquecimiento de la banda de proteína SERCA (peso molecular aparente >100 kDa) a medida que avanza el protocolo de purificación. La banda de proteínas correspondiente a SERCA muestra una pureza del >90% después de la centrifugación en un gradiente de concentración de trehalosa. Gráfico de la tasa de hidrólisis de ATP frente a ...
Discusión
La mayoría de las moléculas e iones no pueden atravesar libremente las membranas celulares, por ejemplo, el protón (H+) requiere un transportador de membrana en la membrana plasmática de una variedad de organismos y orgánulos como las mitocondrias83,84. Las membranas celulares son selectivas y las moléculas e iones que atraviesan las membranas celulares son diversos, por lo que se pueden encontrar varios tipos de...
Divulgaciones
El autor declara que no tiene intereses financieros contrapuestos.
Agradecimientos
El autor agradece la ayuda de Edmundo Mata-Morales en la edición del video, VM, Valentín de la Cruz-Torres en la purificación de los SRV, Miguel A. Rivera-Moran en la purificación y análisis de SERCA, y Juan C. González-Castro, Franco E. Juárez, Alejandra Nevarez, Nicolás Rocha-Vizuet y Jocelin I. Ramírez-Alonso en la producción del video. No se recibieron fondos, subvenciones u otro tipo de apoyo para llevar a cabo este estudio.
Materiales
| Name | Company | Catalog Number | Comments |
| ATP | Sigma-Aldrich Corp | A2383 | |
| Azolectin from soybean | Sigma-Aldrich Corp | 44924 | |
| Benchtop UV transilluminator | Cole-Parmer | EW-97623-08 | Dual intensity High setting is ideal for analytical documentation. Low setting reduces photonicking or photobleaching of gel samples while doing preparative work. |
| CaCl2 • 2H2O | Sigma-Aldrich Corp | 223506 | |
| Coolpix B500 camera | Nikon Corp | S210 | |
| Coomassie brilliant blue G-250 | Bio-Rad | 1610406 | |
| Dodecyl maltoside | Sigma-Aldrich Corp | D4641 | |
| Ethylene glycol-bis(2-aminoethylether)-N,N,N′,N′-tetraacetic acid | Sigma-Aldrich Corp | E4378 | |
| Ethylenediaminetetraacetic acid tetrasodium salt dihydrate | Sigma-Aldrich Corp | E6511 | |
| Fluorescein isothiocyanate | Sigma-Aldrich Corp | F3651 | |
| KCl | JT Baker | 7447-40-7 | |
| MgCl2 • 6H2O | Sigma-Aldrich Corp | M9272 | |
| MOPS | Sigma-Aldrich Corp | M1254 | |
| NADH | Chem-Impex International Inc | 230 | |
| N-tetradecyl-N,N- dimethyl-3-ammonium-1-propanesulfonate | Sigma-Aldrich Corp | D0431 | |
| Phosphoenolpyruvate (PEP) | Chem-Impex International Inc | 9711 | |
| Rabbit muscle lactate dehydrogenase | Roche | 10003557103 | |
| Rabbit muscle pyruvate kinase | Sigma-Aldrich Corp | P1506 | |
| Sodium deoxycholate | Sigma-Aldrich Corp | D6750 | |
| Sodium dodecyl sulfate | Bio-Rad | 1610302 | |
| Spectropolarimeter | Jasco Corp. | Jasco J1500 | |
| Sucrose | Sigma-Aldrich Corp | 84100 | |
| Trehalose | Sigma-Aldrich Corp | T0167 | Dihydrate |
| Tris(hydroxymethyl)aminomethane hydrochloride | Sigma-Aldrich Corp | 857645 | |
| Ultracentrifuge | Beckman | Optima XPN | |
| UV/VIS spectrophotometer | Agilent Technologies | 8453 | The Agilent 8453 UV-Vis Spectrophotometer uses a photodiode array for simultaneous measurement of the complete ultra-violet to visible light spectrum |
| WiseStir HS- 30E | Daihan Scientific Co. | DH.WOS01010 | Ideal for all disperging and homogenizing applications, designed for tissue grinders. |
Referencias
- Schuberth, C., Wedlich-Söldner, R. Building a patchwork - The yeast plasma membrane as model to study lateral domain formation. Biochim Biophys Acta Mol Cell Res. 1853 (4), 767-774 (2015).
- Douglas, L. M., Konopka, J. B. Fungal membrane organization: The eisosome concept. Annu Rev Microbiol. 68 (1), 377-393 (2014).
- Borrell, J. H., Domènech, &. #. 2. 1. 0. ;., Keough, K. M. W. . Membrane protein - Lipid interactions: Physics and Chemistry in the Bilayer. , (2016).
- Moraes, I., Evans, G., Sanchez-Weatherby, J., Newstead, S., Stewart, P. D. . The next generation in membrane protein structure determination. , (2016).
- White, S. H. Biophysical dissection of membrane proteins. Nature. 459 (7245), 344-346 (2009).
- Lacapère, J. -. J. Membrane protein structure determination. Methods Mol Biol. , 459 (1002).
- Palmgren, M. G. Plant plasma membrane H+-ATPases: Powerhouses for nutrient uptake. Annu Rev Plant Physiol Plant Mol Biol. 52 (1), 817-845 (2001).
- Yatime, L., et al. P-type ATPases as drug targets: Tools for medicine and science. Biochim Biophys Acta Bioenerg. 1787 (4), 207-220 (2009).
- Drews, J. Tetrahydrobiopterin biosynthesis as an off-target of sulfa drugs. Science. 287 (5460), 1960-1964 (2000).
- Xu, Z., Meshcheryakov, V. A., Poce, G., Chng, S. -. S. MmpL3 is the flippase for mycolic acids in mycobacteria. Proc Natl Acad Sci USA. 114 (30), 7993-7998 (2017).
- Siligardi, G., Hussain, R., Patching, S. G., Phillips-Jones, M. K. Ligand- and drug-binding studies of membrane proteins revealed through circular dichroism spectroscopy. Biochim Biophys Acta Biomembr. 1838 (1 PARTA), 34-42 (2014).
- Rawlings, A. E. Membrane proteins: Always an insoluble problem. Biochem Soc Trans. 44 (3), 790-795 (2016).
- Popot, J. -. L. . Membrane proteins in aqueous solutions. , 708 (2018).
- Palmgren, M. G., Sommarin, M., Ulvskov, P., Larsson, C. Effect of detergents on the H+-ATPase activity of inside-out and right-side-out plant plasma membrane vesicles. Biochim Biophys Acta Biomembr. 1021 (2), 133-140 (1990).
- McBride, Z., Chen, D., Reick, C., Xie, J., Szymanski, D. B. Global analysis of membrane-associated protein oligomerization using protein correlation profiling. Mol Cell Proteomics. 16 (11), 1972-1989 (2017).
- Yoneda, J. S., et al. Multimeric species in equilibrium in detergent-solubilized Na, K-ATPase. Int J Biol Macromol. 89, 238-245 (2016).
- Ohlendieck, K. Extraction of membrane proteins. Protein Purif Protocols. 59, 283-294 (2004).
- Champeil, P., et al. A robust method to screen detergents for membrane protein stabilization, revisited. Anal Biochem. 511, 31-35 (2016).
- Gimpl, K., Klement, J., Keller, S. Characterising protein/detergent complexes by triple-detection size-exclusion chromatography. Biol Proced Online. 18, 4 (2016).
- le Maire, M., et al. Gel chromatography and analytical ultracentrifugation to determine the extent of detergent binding and aggregation, and Stokes radius of membrane proteins using sarcoplasmic reticulum Ca2+-ATPase as an example. Nat Protoc. 3 (11), 1782-1795 (2008).
- Du, M., et al. Progress, applications, challenges and prospects of protein purification technology. Front Bioeng Biotechnol. 10, 1-26 (2022).
- Slotboom, D. J., Duurkens, R. H., Olieman, K., Erkens, G. B. Static light scattering to characterize membrane proteins in detergent solution. Methods. 46 (2), 73-82 (2008).
- Harding, S. E. Analytical ultracentrifugation as a matrix-free probe for the study of kinase related cellular and bacterial membrane proteins and glycans. Molecules. 26, 6080 (2021).
- Chen, Y. -. C., et al. Thermal stability, storage and release of proteins with tailored fit in silica. Sci Rep. 7 (1), 46568 (2017).
- Montigny, C. C., Arnou, B., Marchal, E., Champeil, P. Use of glycerol-containing media to study the intrinsic fluorescence properties of detergent-solubilized native or expressed SERCA1a. Biochemistry. 47 (46), 12159-12174 (2008).
- Simongini, M., Puglisi, A., Genovese, F., Hochkoeppler, A. Trehalose counteracts the dissociation of tetrameric rabbit lactate dehydrogenase induced by acidic pH conditions. Arch Biochem Biophys. 740, 109584 (2023).
- Shivanna, B. D., Rowe, E. S. Preservation of the native structure and function of Ca2+-ATPase from sarcoplasmic reticulum: Solubilization and reconstitution by new short-chain phospholipid detergent 1,2-diheptanoyl-sn-phosphatidylcholine. Biochem J. 325 (2), 533-542 (1997).
- Sampedro, J. G., Guerra, G., Pardo, J. P., Uribe, S. Trehalose-mediated protection of the plasma membrane H+-ATPase from Kluyveromyces lactis during freeze-drying and rehydration. Cryobiology. 37 (2), 131-138 (1998).
- Sampedro, J. G., Cortés, P., Muñoz-Clares, R. A., Fernández, A., Uribe, S. Thermal inactivation of the plasma membrane H+-ATPase from Kluyveromyces lactis. Protection by Trehalose. Biochim Biophys Acta Proteins Proteomics. 1544 (1-2), 64-73 (2001).
- Sampedro, J. G., et al. Fluorescence quenching by nucleotides of the plasma membrane H+-ATPase from Kluyveromyces lactis. Biochemistry. 46 (18), 5616-5622 (2007).
- Ruiz-Granados, Y. G., De La Cruz-Torres, V., Sampedro, J. G. The oligomeric state of the plasma membrane H+-ATPase from Kluyveromyces lactis. Molecules. 24 (5), 958 (2019).
- Zhao, P., Zhao, C., Chen, D., Yun, C., Li, H., Bai, L. Structure and activation mechanism of the hexameric plasma membrane H+-ATPase. Nat Commun. 12 (1), 6439 (2021).
- Auer, M., Scarborough, G. A., Kühlbrandt, W. Surface crystallization of the plasma membrane H+-ATPase on a carbon support film for electron crystallography. J Mol Biol. 287 (5), 961-968 (1999).
- Hennessey, J. P., Scarborough, G. A. Secondary structure of the Neurospora CRASSA plasma membrane H+-ATPase as estimated by circular dichroism. J Biol Chem. 263 (7), 3123-3130 (1988).
- Scarborough, G. A. Crystallization, structure and dynamics of the proton-translocating P-type ATPase. J Exp Biol. 203 (Pt 1), 147-154 (2000).
- Sumida, K. H., et al. Improving protein expression, stability, and function with proteinMPNN. J Am Chem Soc. 146 (3), 2054-2061 (2024).
- Eleutherio, E., Panek, A., De Mesquita, J. F., Trevisol, E., Magalhães, R. Revisiting yeast trehalose metabolism. Curr Genet. 61 (3), 263-274 (2015).
- Attfield, P. V. Trehalose accumulates in Saccharomyces cerevisiae during exposure to agents that induce heat shock response. FEBS Lett. 225 (1-2), 259-263 (1987).
- Marunde, M. R., et al. Improved tolerance to salt and water stress in Drosophila melanogaster cells conferred by late embryogenesis abundant protein. J Insect Physiol. 59 (4), 377-386 (2013).
- Crowe, J. H. Anhydrobiosis: An unsolved problem with applications in human welfare. Subcell Biochem. 71, 263-280 (2015).
- Crowe, J. H., Crowe, L. M., Chapman, D. Preservation of membranes in anhydrobiotic organisms: The role of trehalose. Science. 223 (4637), 701-703 (1984).
- Sampedro, J. G., Rivera-Moran, M. A., Uribe-Carvajal, S. Kramers' theory and the dependence of enzyme dynamics on trehalose-mediated viscosity. Catalysts. 10 (6), 1-19 (2020).
- Sampedro, J. G., Uribe, S. Trehalose-enzyme interactions result in structure stabilization and activity inhibition. The role of viscosity. Mol Cell Biochem. 256 - 257 (1-2), 319-327 (2004).
- Crowe, J. H. Trehalose as a "chemical chaperone": Fact and fantasy. Adv Exp Med Biol. 594, 143-158 (2007).
- Crowe, J. H., Carpenter, J. F., Crowe, L. M., Anchordoguy, T. J. Are freezing and dehydration similar stress vectors? A comparison of modes of interaction of stabilizing solutes with biomolecules. Cryobiology. 27 (3), 219-231 (1990).
- Rivera-Morán, M. A., Sampedro, J. G. Isolation of the sarcoplasmic reticulum Ca2+-ATPase from rabbit fast-twitch muscle. Methods Protoc. 6 (5), 102 (2023).
- Páez-Pérez, E. D., De La Cruz-Torres, V., Sampedro, J. G. Nucleotide binding in an engineered recombinant Ca2+-ATPase N-domain. Biochemistry. 55 (49), 6751-6765 (2016).
- Ramírez-Alonso, J. I., Sampedro, J. G. Effect of cations on ATP binding to the N-domain of Na+, K+-ATPase. J Fluoresc. , (2024).
- De la Cruz-Torres, V., Cataño, V., Olivo-Rodríguez, M., Sampedro, J. G. ANS Interacts with the Ca2+-ATPase nucleotide binding site. J Fluoresc. 30 (3), 483-496 (2020).
- Sampedro, J. G., Nájera, H., Uribe-Carvajal, S., Ruiz-Granados, Y. G. Mapping the ATP binding site in the plasma membrane H+-ATPase from Kluyveromyces lactis. J Fluoresc. 24 (6), 1849-1859 (2014).
- Kiani, A. K., et al. Ethical considerations regarding animal experimentation. J Prev Med Hyg. 63 (2 Suppl 3), E255-E266 (2022).
- Ruiz-Granados, Y., De La Cruz-Torres, V., Sampedro, J. The oligomeric state of the plasma membrane H+-ATPase from Kluyveromyces lactis. Molecules. 24 (5), 958 (2019).
- Wang, S., et al. Structural basis for sarcolipin's regulation of muscle thermogenesis by the sarcoplasmic reticulum Ca2+-ATPase. Sci Adv. 7 (48), eabi7154 (2021).
- Champeil, P., et al. ATP regulation of sarcoplasmic reticulum Ca2+-ATPase. Metal-free ATP and 8-bromo-ATP bind with high affinity to the catalytic site of phosphorylated ATPase and accelerate dephosphorylation. J Biol Chem. 263 (25), 12288-12294 (1988).
- Møller, J. V., Olesen, C. Preparation of Ca(2+)-ATPase1a enzyme from rabbit sarcoplasmic reticulum. Methods Mol Biol. 1377, 11-17 (2016).
- Wuytack, F., De Schutter, G., Casteels, R. Partial purification of (Ca2+ + Mg2+)-dependent ATPase from pig smooth muscle and reconstitution of an ATP-dependent Ca2+-transport system. Biochem J. 198 (2), 265-271 (1981).
- Mandal, A., et al. Solubilization, purification and reconstitution of Ca2+-ATPase from bovine pulmonary artery smooth muscle microsomes by different detergents: Preservation of native structure and function of the enzyme by DHPC. Biochim Biophys Acta Gen Subj. 1760 (1), 20-31 (2006).
- Inesi, G., Ma, H., Hua, S., Toyoshima, C. Characterization of Ca2+ ATPase residues involved in substrate and cation binding. Ann NY Acad Sci. 986 (1), 63-71 (2003).
- Van Winkle, W. B., Pitts, B. J., Entman, M. L. Rapid purification of canine cardiac sarcoplasmic reticulum Ca2+-ATPase. J Biol Chem. 253 (24), 8671-8673 (1978).
- Ruiz-Granados, Y. G., De La Cruz-Torres, V., Sampedro, J. G. The oligomeric state of the plasma membrane H+-ATPase from Kluyveromyces lactis. Molecules. 24 (5), 958 (2019).
- Teramoto, N., Sachinvala, N. D., Shibata, M. Trehalose and trehalose-based polymers for environmentally benign, biocompatible, and bioactive materials. Molecules. 13 (8), 1773-1816 (2008).
- Dao, T. T., et al. Demethoxycurcumin is a potent inhibitor of P-type ATPases from diverse kingdoms of life. PLOS ONE. 11 (9), e0163260 (2016).
- Bleeker, N. P., Cornea, R. L., Thomas, D. D., Xing, C. A novel SERCA inhibitor demonstrates synergy with classic SERCA inhibitors and targets multidrug-resistant AML. Mol Pharmaceutics. 10 (11), 4358-4366 (2013).
- Michelangeli, F., East, J. M. A diversity of SERCA Ca2+ pump inhibitors. Biochem Soc Trans. 39 (3), 789-797 (2011).
- Tadini-Buoninsegni, F., Smeazzetto, S., Gualdani, R., Moncelli, M. R. Drug interactions with the Ca2+-ATPase from Sarco(Endo)Plasmic Reticulum (SERCA). Front Mol Biosci. 5, 36 (2018).
- Clausen, J. D., et al. Elucidation of antimicrobial activity and mechanism of action by N-substituted carbazole derivatives. Bioorg Med Chem Lett. 27 (19), 4564-4570 (2017).
- Leary, S., et al. . AVMA guidelines for the euthanasia of animals: 2020 Edition, Members of the Panel on Euthanasia AVMA Staff Consultants. , (2020).
- de Aluja, A. S. Laboratory animals and official Mexican norms (NOM-062-ZOO-1999). Gac Med Mex. 138 (3), 295-298 (2002).
- Champeil, P., Buschlen-Boucly, S., Bastide, F., Gary-Bobo, C. Sarcoplasmic reticulum ATPase. Spin labeling detection of ligand-induced changes in the relative reactivities of certain sulfhydryl groups. J Biol Chem. 253 (4), 1179-1186 (1978).
- Skalec, A., Janeczek, M., Czerski, A. Anatomy and histology of the rabbit common calcanean tendon. J Vet Med Series C: Anat Histol Embryol. 48 (5), 466-475 (2019).
- Janes, L. E., Mioton, L. M., Fracol, M. E., Ko, J. H. An in vivo comparison: Novel mesh suture versus traditional suture-based repair in a rabbit tendon model. J Hand Surg Glob Online. 4 (1), 32-39 (2022).
- Bollag, D. M., Rozycki, M. D., Edelstein, S. J. . Protein Methods. , (1996).
- Gagné, F. Tissue preparation and subcellular fractionation techniques. Biochem Ecotoxicol. , 21-31 (2014).
- Lowry, O. H., Rosebrough, N. J., Farr, A. L., Randall, R. J. Protein measurement with the Folin phenol reagent. J Biol Chem. 193 (1), 265-275 (1951).
- Autry, J. M., Rubin, J. E., Svensson, B., Li, J. L., Thomas, D. D. Nucleotide activation of the Ca-ATPase. J Biol Chem. 287 (46), 39070-39082 (2012).
- Winters, D. L., Autry, J. M., Svensson, B., Thomas, D. D. Interdomain fluorescence resonance energy transfer in SERCA probed by cyan-fluorescent protein fused to the actuator domain. Biochemistry. 47 (14), 4246-4256 (2008).
- Miles, A. J., Wallace, B. A. Circular dichroism spectroscopy of membrane proteins. Chem Soc Rev. 14 (45), 1130-1135 (2016).
- Scofano, H. M., Vieyra, A., de Meis, L. Substrate regulation of the sarcoplasmic reticulum ATPase. Transient kinetic studies. J Biol Chem. 254 (20), 10227-10231 (1979).
- Yamamoto, T., Tonomura, Y. Reaction mechanism of the Ca++-dependent ATPase of sarcoplasmic reticulum from skeletal muscle: I. Kinetic studies. J Biochem. 62 (5), 558-575 (1967).
- Murphy, A. J. Affinity labeling of the active site of the Ca2+-ATPase of sarcoplasmic reticulum. Biochim Biophys Acta - Biomembr. 946 (1), 57-65 (1988).
- Csermely, P., Katopis, C., Wallace, B. A., Martonosi, A. The E1→E2 transition of Ca2+-transporting ATPase in sarcoplasmic reticulum occurs without major changes in secondary structure. A circular-dichroism study. Biochem J. 241 (3), 663-669 (1987).
- Abràmoff, M. D., Magalhães, P. J., Ram, S. J. Image processing with ImageJ. Biophotonics Int. 11 (7), 36-42 (2004).
- Scarborough, G. A. The plasma membrane proton-translocating ATPase. Cell Mol Life Sci. 57 (6), 871-883 (2000).
- Llopis, J., McCaffery, J. M., Miyawaki, A., Farquhar, M. G., Tsien, R. Y. Measurement of cytosolic, mitochondrial, and Golgi pH in single living cells with green fluorescent proteins. Proc Natl Acad Sci. 95 (12), 6803-6808 (1998).
- Kanai, R., Vilsen, B., Cornelius, F., Toyoshima, C. Crystal structures of Na+, K+-ATPase reveal the mechanism that converts the K+-bound form to Na+-bound form and opens and closes the cytoplasmic gate. FEBS Lett. 597 (15), 1957-1976 (2023).
- Palmgren, M. P-type ATPases: Many more enigmas left to solve. J Biol Chem. 299 (11), 105352 (2023).
- Frauenfelder, H. Ask not what physics can do for biology-Ask what biology can do for physics. Phys Biol. 11 (5), 053004 (2014).
- Frauenfelder, H. The physics of proteins. Annu Rev Biophys Biomol Struct. 36, (2010).
- Narhi, L. O. . Biophysics for Therapeutic Protein Development. , (2013).
- East, J. M. Purification of a membrane protein (Ca2+/Mg2+-ATPase) and its reconstitution into lipid vesicles. Methods Mol Biol. 27, 87-94 (1994).
- Lee, E. -. H., et al. Enhancement of enzyme activity and stability by poly(γ-glutamic acid). Polymer J. 42 (10), 818-822 (2010).
- Norrild, R. K., et al. Increasing protein stability by inferring substitution effects from high-throughput experiments. Cell Rep Methods. 2 (11), 100333 (2022).
- Song, Z., Zhang, Q., Wu, W., Pu, Z., Yu, H. Rational design of enzyme activity and enantioselectivity. Front Bioeng Biotechnol. 11, 1-14 (2023).
- Jidenko, M., Lenoir, G., Fuentes, J. M., le Maire, M., Jaxel, C. Expression in yeast and purification of a membrane protein, SERCA1a, using a biotinylated acceptor domain. Protein Expr Purif. 48 (1), 32-42 (2006).
- Hua, S., Ma, H., Lewis, D., Inesi, G., Toyoshima, C. Functional role of "N" (nucleotide) and "P" (phosphorylation) domain interactions in the sarcoplasmic reticulum (SERCA) ATPase. Biochemistry. 41 (7), 2264-2272 (2002).
- Pang, Y. -. H., Chen, J. -. W. Anisodamine causes the changes of structure and function in the transmembrane domain of the Ca2+-ATPase from sarcoplasmic reticulum. Biosci Biotechnol Biochem. 68 (1), 126-131 (2004).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoThis article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados