Method Article
Open-Source Droplet Microfluidics를 이용한 플라스미드 안정성 분석
요약
접근 가능한 오픈 소스 미세유체(microfluidic) 워크플로우는 박테리아의 플라스미드 보유에 대한 병렬 분석을 위해 제공됩니다. 형광 현미경 검사를 사용하여 겔 미세방울 내 단일 세포 미세군체의 플라스미드 존재를 정량화함으로써 이 방법은 기존 플레이트 카운팅에 대한 정확하고 접근 가능하며 확장 가능한 대안을 제공합니다.
초록
플라스미드는 다양한 유기체에서 외래 유전자의 도입 및 발현을 가능하게 함으로써 합성 생물학에서 중요한 역할을 하며, 이를 통해 세포 집단 내부 및 세포 집단 사이의 생물학적 회로 및 경로 구축을 촉진합니다. 많은 응용 분야에서 항생제 선택 없이 기능적 플라스미드를 유지하는 것이 중요합니다. 이 연구는 겔 미세방울에서 단세포를 배양하고 형광 현미경을 사용하여 미세콜로니를 정량화하여 플라스미드 보유를 분석하기 위한 개방형 하드웨어 기반 미세유체 워크플로우를 소개합니다. 이 접근 방식을 사용하면 수많은 액적 및 미세군체를 동시에 분석할 수 있어 기존 플레이트 카운팅에 비해 더 큰 통계적 성능을 제공하고 분석을 다른 액적 미세유체 워크플로우에 통합할 수 있습니다. 비특이적 형광 DNA 염색과 함께 형광 단백질을 발현하는 플라스미드를 사용함으로써, 플라스미드 손실 또는 형광 마커 발현을 기반으로 단일 콜로니를 식별하고 구별할 수 있습니다. 특히, 오픈 소스 하드웨어로 구현된 이 고급 워크플로우는 샘플과 미세유체 칩 모두의 정확한 흐름 제어 및 온도 관리를 제공합니다. 이러한 기능은 워크플로의 사용 편의성, 견고성 및 접근성을 향상시킵니다. 이 연구는 실험 모델로서 대장균 에 초점을 맞추고 있지만, 이 방법의 진정한 잠재력은 다양성에 있습니다. 플라스미드 또는 염색체의 형광 신호 정량화가 필요한 다양한 연구뿐만 아니라 다른 응용 분야에도 적용할 수 있습니다. 오픈 소스 하드웨어의 채택은 다양한 연구 환경에서 접근 가능한 기술을 사용하여 고처리량 생물 분석을 수행할 수 있는 가능성을 넓힙니다.
서문
플라스미드는 원핵 세포의 필수 유전 요소로, 측면 DNA 전달을 통한 미생물 진화와 환경 변화에 대한 빠른 적응에 크게 기여합니다 1,2. 이러한 염색체외 DNA 분자는 항생제 내성, 대사 기능 및 독성 인자와 같은 유리한 특성을 제공하는 유전자를 가지고 있어 미생물학 연구, 합성 생물학 및 진화 연구에 유용합니다2. 그러나 세포 집단에서 플라스미드 유지는 복제 및 분리의 대사 부담으로 인해 까다롭고, 종종 선택 압력 없이 플라스미드 손실이 발생합니다3. 또한 안정적인 유전을 위해서는 독소-항독소 및 파티셔닝 시스템과 같은 메커니즘이 필요하므로 플라스미드 유지 관리가 복잡해집니다. 다양한 조건에서 플라스미드 안정성을 평가하는 것은 플라스미드를 주요 연구 요소로 사용하는 기초 연구와 실제 응용 분야 모두에 중요합니다 4,5. 플라스미드 안정성을 평가하기 위한 대부분의 최신 방법은 상당한 한계가 있습니다: 유세포 분석 기반 방법은 간접적인 집단 수준의 데이터를 제공하고, 고가의 장비가 필요하며, 콜로니에 대한 직접적인 시각화가 부족합니다6. 벌크 전사체학 및 단백질체학 방법은 비용이 많이 들고 평균적인 세포 반응만 제공하며 개별 콜로니에서 플라스미드 보유를 직접 정량화할 수 없습니다6. 연속 희석 및 계대 태동과 같은 기존 방법은 간단하지만 시간이 많이 걸리고 정밀도와 표현성이 부족합니다7. 전반적으로, 시간 경과에 따라 또는 선택적 압력에 따라 특정 기능성 플라스미드를 유지하는 콜로니의 수를 정량적으로 추론하거나 예측하는 것은 여전히 어려운 일입니다.
이러한 문제를 해결하기 위해 개방형 하드웨어 연구 장비를 활용하는 새로운 미세유체 워크플로우를 제시하여 대장균 을 모델로 사용하여 여러 고립된 박테리아 군체의 형광 신호를 정량화합니다. 이 방법을 사용하면 다양한 조건 또는 선택적 압력에서 플라스미드 머무름을 고처리량으로 정밀하게 분석할 수 있습니다. 단일 세포 분해능 분석은 분리된 콜로니를 조작할 수 있는 정밀한 방법을 제공하여 머무름 및 손실률을 평가하는 데 도움이 될 수 있는 플라스미드 정량화에 대한 민감한 데이터를 산출합니다4.
미세유체역학, 특히 액적 미세유체역학(microfluidics)은 통제된 환경8에서 개별 세포를 캡슐화하고 조작하기 위한 강력한 도구로 부상했습니다. 특히, 마이크로겔 액적은 오일9에 부유하는 액적을 유지할 필요 없이 고처리량 및 정밀 분석을 위해 단일 세포를 캡슐화할 수 있으므로 정의된 미세환경에서 플라스미드 역학에 대한 통제된 연구가 가능합니다. 피펫 팁10에서 직접 세포 현탁액을 캡슐화한 후, 형광 기술을 사용하여 액적 내 미세군집의 성장을 모니터링할 수 있으며, 이를 통해 다양한 선택 압력3에서 플라스미드 머무름 및 편석을 자세히 분석할 수 있습니다.
기존 벌크 배양 기술에 비해 이 방법의 장점으로는 정밀도 향상, 변동성 감소, 고처리량 분석 수행 능력 등이 있습니다. 오픈 소스 미세유체역학 기술은 접근성, 적응성 및 유지 관리 문제와 같은 값비싼 독점 시스템의 한계를 극복하여 종종 연구 진행을 방해합니다 11,12,13. 오픈 소스 기기를 사용하여 마이크로겔에 플라스미드 머무름 분석의 고급 실험 워크플로우를 적용하는 방법을 시연함으로써 플라스미드 생물학, 합성 생물학 응용 분야 및 미세유체 액적 분석 기술 연구를 위한 접근 가능하고 신뢰할 수 있는 방법을 제공합니다.
요약하면, 이 논문은 높은 통계적 검정력으로 E. coli 의 플라스미드 보유를 정량적으로 평가하기 위한 접근 가능한 방법을 제시합니다. 이 방법의 기능은 플라스미드 생물학에 대한 이해를 증진하고 합성 생물학 응용 분야를 개선하는 데 유용한 도구입니다.
프로토콜
그림 1은 E. coli 에서 플라스미드 안정성을 평가하기 위한 개략도를 제공합니다.시약 및 사용된 장비에 대한 자세한 내용은 재료 표에 나열되어 있습니다. 원시 데이터 및 시각화 스크립트는 https://doi.org/10.17605/OSF.IO/6YWJK 에서 사용할 수 있습니다.
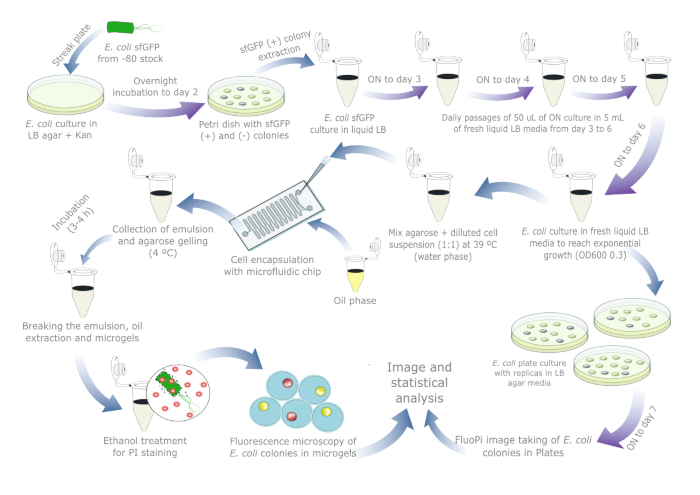
그림 1: E. coli에서 플라스미드 안정성을 평가하기 위한 일일 프로토콜. 파란색 화살표는 낮 동안의 단계를 나타내고 보라색 화살표는 밤새 부화를 나타냅니다. 모든 액체 및 한천 배양은 별도의 음성 대조 튜브/플레이트와 함께 37°C에서 수행되었습니다. 플라스미드 손실이 이미 발생했을 수 있는 실제 샘플에는 세포 배양 준비 및 계대가 필요하지 않으므로 플레이트 참조 배양이 포함된 경우 프로토콜을 하루나 이틀로 줄여야 합니다. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
1. 미세유체 칩 준비
참고: 이 프로토콜에서는 점적 영역에서 직경이 100μm 미만인 유중수 방울을 생성할 수 있는 세포 캡슐화에 다양한 상용 또는 맞춤형 칩 설계를 사용할 수 있습니다. 이 연구를 위해, 칩은 이전에 발표된 보고서(14)에서 보고된 것과 동일한 설계 및 제조 방법에 따라 설계 및 제작되었다(기사 데이터 https://doi.org/10.17605/OSF.IO/6YWJK 참조).
- 겔-마이크로액적 생성을 위해 설계된 마스터 몰드를 사용하여 PDMS-on-glass 미세유체 칩을 구하거나 준비합니다.
- 발수 용액(플루오로알킬 실란)을 칩에 주입하여 내부 마이크로채널을 소수성으로 만듭니다. 용액을 주입구에 주입하고 모든 채널이 유체로 채워졌는지 확인합니다. 채워진 채널을 약 30-60초 동안 그대로 둡니다.
- 내부 마이크로 채널로 공기를 배출하여 장치에서 용액을 제거합니다. 빈 공기 주사기를 사용하여 공기를 씻어내고 다른 포트를 흡수성 닦아서 튀는 것을 방지합니다.
- 처리된 장치를 65°C에서 핫플레이트에서 15분 동안 굽고 과도한 용액을 증발시킵니다. 또는 장치를 냉장고(4°C)에 밤새 보관하십시오.
알림: 미세유체 칩을 사용할 준비가 되었습니다. 여기에서 프로토콜을 일시 중지할 수 있습니다.
2. 시료 준비
- 세포 채취
참고: 액적의 세포 배양은 플라스미드 역학에 대한 관련 데이터를 제공할 수 있습니다. 실험적 박테리아 모델로서, E. coli TOP10 균주는 superfolder green 형광 단백질(sfGFP) 및 kanamycin-resistance15를 암호화하는 plasmid pCA_Odd1(기탁된 데이터 참조)와 함께 사용됩니다(https://doi.org/10.17605/OSF.IO/6YWJK 기탁된 데이터 참조). 박테리아, 플라스미드 및 성장 배지는 실험 시스템에 따라 달라질 수 있습니다.- 미리 혼합된 LB 25g과 한천 12g을 증류수 800mL(dH2O)에 용해시켜 Luria Bertoni(LB)-한천을 준비합니다. 또한 사전 혼합된 LB 25g을 800mL의 dH2O에 용해시켜 액체 LB 매체를 준비합니다. 용액을 오토클레이브하고 약 60°C로 냉각시킵니다. 다음 단계를 위해 플로우 후드와 같은 멸균 환경으로 이동합니다.
참고: 미리 혼합된 LB 분말에는 트립톤 10g, 효모 추출물 5g, NaCl 10g이 포함되어 있습니다. 자체 혼합 LB-한천 매체도 사용할 수 있습니다. - 50μL의 카나마이신(100mg/mL로 준비)을 50mL의 액체 LB-한천(최종 농도: 100μg/mL)에 추가합니다. 튜브를 몇 번 뒤집어 용액을 혼합합니다.
- 페트리 접시(90mm x 15mm)당 약 15mL의 액체 LB-한천을 붓습니다. 두 개의 플레이트를 준비합니다: 하나는 음성 대조군(오염 모니터링)용이고 다른 하나는 실험 배양용입니다. LB-한천 용액을 식히고 두 플레이트의 색상이 어두운 색에서 투명한 색으로 바뀔 때까지 굳히십시오.
- 멸균 루프를 사용하여 줄무늬 플레이트 기법을 사용하여 배양 플레이트에 E. coli 균주를 퍼뜨립니다. 글리세롤 스톡 E. coli 를 사용 직후 -80 °C에 다시 놓습니다. 두 플레이트를 모두 닫고 37°C에서 밤새 배양합니다(1일차).
- 2일차에는 오염 신호로 음극 대조판에 집락이 있는지 확인합니다(그렇다면 2.1.4의 단계를 반복). 액체 배양 스톡을 준비하기 위해 청색광 투과광기를 사용하여 E. coli 플레이트의 단일 형광 콜로니를 식별합니다.
- 멸균 조건에서 플레이트를 열고 멸균 루프 또는 200μL 피펫 팁을 사용하여 선택한 콜로니를 선택하고 5mL의 신선한 액체 LB 배지와 5μL의 카나마이신(100mg/mL로 준비)이 있는 배양 튜브로 옮깁니다. 오염 모니터링을 위해 접종 없이 negative control tube를 준비합니다. 220rpm으로 흔들면서 37°C에서 밤새 배양 튜브를 배양합니다.
- 항생제를 투여하지 않은 배지에서 추가로 3일 동안 샘플을 통과시켜 플라스미드 손실이 발생할 수 있는 조건을 시뮬레이션합니다. 3일, 4일, 5일째에 하룻밤 배양액 50μL를 항생제 없이 5mL의 액체 LB가 들어 있는 새로운 배양 튜브로 옮깁니다. 37°C에서 하룻밤 동안 220rpm으로 흔들면서 배양합니다. 6일째까지 이 단계를 반복하여 총 4번의 계대 세척 후 최종 5mL 배양을 얻습니다.
- 최종 야간 배양에서 50μL를 2mL의 액체 LB를 포함하는 새로운 배양 튜브로 옮깁니다. 분광 광도계를 사용하여 배양물이 0.3의 광학 밀도(OD600)에 도달하도록 합니다(약 3–4시간).
알림: 박테리아가 로그 성장 단계에 있는지 확인하기 위해 OD600이 증가하기 전에 신선한 배양을 사용해야 합니다. 원하는 농도에 도달하지 못하면 2.1.8단계부터 반복합니다.
- 미리 혼합된 LB 25g과 한천 12g을 증류수 800mL(dH2O)에 용해시켜 Luria Bertoni(LB)-한천을 준비합니다. 또한 사전 혼합된 LB 25g을 800mL의 dH2O에 용해시켜 액체 LB 매체를 준비합니다. 용액을 오토클레이브하고 약 60°C로 냉각시킵니다. 다음 단계를 위해 플로우 후드와 같은 멸균 환경으로 이동합니다.
- 캡슐화를 위한 세포-아가로스 혼합물
참고: 농도 제어는 단일 세포 액적 캡슐화를 보장하는 데 필수적입니다. 필요한 세포 농도는 다음 예와 같이 목표 캡슐화 속도 및 특정 액적 부피에 대해 계산할 수 있습니다.
액적 부피(Vdrop): 50μm 액적이 생성되는 경우
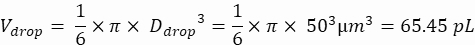
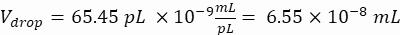
액적당 원하는 세포(Cpd): 평균 5개의 액적당 1개의 세포(0.2 cells/droplet)

희석 계수: 초기 세포 농도는 2.1.8단계에서 구합니다.
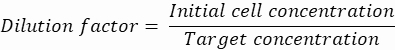
여기에서 50μm 또는 100μm(액적 분할 없음) 액적(65–520pL)은 5개의 액적당 약 1개의 세포가 캡슐화되거나 분열 없이 액적당 1.6개의 세포가 생성됩니다. 대장균의 경우 1 OD600 단위의 변환 계수를 7.8 x 108 cells/mL16≈ 사용합니다. 2.1.8단계의 OD600 값에 변환 계수를 곱하여 배양물의 초기 농도(cells/mL)를 구합니다.- 6.2e+6 cells/mL(직경 50μm 방울에 대한 목표 농도)의 농도로 kanamycin이 없는 액체 LB에 준비된 배양 스톡에서 E. coli 를 다시 현탁시킵니다. 아가로스와 혼합될 때까지 박테리아 현탁액을 실온에서 유지하십시오.
참고: 항생제는 음성의 미세집락 형성을 억제하므로 플라스미드 손실 실험을 위해 배지에서 제외하는 것이 필수적입니다. - 초저겔화 온도 아가로스를 2%(w/v)의 농도로 액체 LB에서 90°C로 가열하여 준비합니다. 온도 조절이 가능한 셰이커에서 혼합물을 10분 동안 흔듭니다.
- 써모 셰이커의 온도를 39°C로 낮추어 아가로스 용액을 식힙니다. 동시에 박테리아 현탁 튜브를 써모 셰이커에 4분 동안 넣어 39°C로 예열합니다.
- 박테리아와 아가로스 현탁액을 1:1 비율로 혼합하여 3.1e+6 cell/mL의 세포 현탁액으로 1%(w/v)의 아가로스 농도를 얻습니다. 박테리아 현탁액 대신 액체 LB를 사용하여 동일한 아가로스 농도의 negative control solution(오염 모니터링)을 준비합니다.
알림: 아가로스 세포 현탁액은 박테리아 성장으로 인한 농도 변화를 피하기 위해 신속하게 사용해야 합니다. 액적 생성을 위해 팁 히터에 로드될 때까지 아가로스 액체를 유지하기 위해 열 셰이커를 39°C로 유지하십시오.
- 6.2e+6 cells/mL(직경 50μm 방울에 대한 목표 농도)의 농도로 kanamycin이 없는 액체 LB에 준비된 배양 스톡에서 E. coli 를 다시 현탁시킵니다. 아가로스와 혼합될 때까지 박테리아 현탁액을 실온에서 유지하십시오.
3. High-throughput 단 하나 식민지 경작
- 실험 설정
- 가스 압력 드라이버 및 유량 센서를 포함한 전체 오픈 소스 유량 플랫폼(https://doi.org/10.17605/OSF.IO/6YWJK 참조)을 구축하거나 확보합니다. 또는 더 간단한 오픈 소스 하드웨어 스트로브 강화 현미경 단계를 빌드합니다(빌드 지침 https://wenzel-lab.github.io/strobe-enhanced-microscopy-stage/ 및 프로젝트 리포지토리 https://github.com/wenzel-lab/strobe-enhanced-microscopy-stage 참조).
참고: 상업용 현미경, 고속 카메라 및 샘플 가열 기능과 함께 기존의 미세유체 설정을 사용할 수도 있습니다. - 오픈 소스 Raspberry Pi 기반 압력 및 유량 컨트롤러 시스템을 지정된 저장소(https://github.com/wenzel-lab/modular-microfluidics-workstation-controller)에 문서화된 대로 통합합니다.
참고: 모듈 컨트롤이 표시되어 데이터 저장소(https://doi.org/10.17605/OSF.IO/6YWJK. 대안으로 기존의 압력 컨트롤러 또는 고토크 주사기 펌프를 사용할 수 있습니다. - 설정에 유리 슬라이드와 피펫 팁 히터(https://github.com/wenzel-lab/modular-microfluidics-workstation-controller/tree/master/module-heating-and-stirring)를 포함하여 아가로스 셀 샘플이 칩에 들어갈 때 샘플의 온도를 제어할 수 있습니다.
참고: 데이터 저장소(https://github.com/wenzel-lab/flow-microscopy-platform 및 https://doi.org/10.17605/OSF.IO/6YWJK)에 표시된 이러한 히터는 아가로스 작업을 가능하게 하는 오픈 소스 컨트롤러 시스템의 중요한 기능이며 다른 상용 시스템에서는 사용할 수 없습니다.
- 가스 압력 드라이버 및 유량 센서를 포함한 전체 오픈 소스 유량 플랫폼(https://doi.org/10.17605/OSF.IO/6YWJK 참조)을 구축하거나 확보합니다. 또는 더 간단한 오픈 소스 하드웨어 스트로브 강화 현미경 단계를 빌드합니다(빌드 지침 https://wenzel-lab.github.io/strobe-enhanced-microscopy-stage/ 및 프로젝트 리포지토리 https://github.com/wenzel-lab/strobe-enhanced-microscopy-stage 참조).
- Single-cell encapsulation (단일 셀 캡슐화)
- 스트로브 강화 현미경 스테이지에 미세유체 칩을 배치하여 액적 생성 접합부(수성 상과 오일 상의 교차점)가 보이도록 합니다.
- 제어 소프트웨어 인터페이스를 사용하여 피펫 팁 히터와 유리 슬라이드 히터를 40°C로 설정합니다.
- 튜빙 및 PDMS 플러그가 있는 주사기를 사용하여 세포 현탁액이 있는 1% 아가로스 혼합물을 200μL 피펫 팁에 로드합니다. 팁을 팁 히터에 삽입하고 미세유체 칩의 수성 상 입구에 놓습니다. 팁의 PDMS 씰을 흐름 제어 시스템 튜브에 연결된 씰로 교체하고 세포 현탁액의 주입을 시작합니다.
- 출구 튜브 끝을 폐기물 튜브에 삽입하고 사용자 인터페이스에서 두 단계의 유량 또는 압력을 설정하여 유체를 미세유체 채널에 천천히 전달합니다. 수성상에는 200μL/h(180mbar)를, 오일상에는 1700μL/h(320mbar)를 사용합니다. 액적 생성의 안정화를 위해 1분을 허용합니다.
참고: 압력 값은 칩 설계의 채널 크기에 따라 달라지며 유량 값은 다른 액적 생성 접합 설계에 맞게 조정해야 할 수 있습니다. - 액적 생성이 안정되면 폐기물과 수집 튜브를 수집 튜브로 옮깁니다. 샘플 저장소가 비워질 때까지 액적을 계속 수집합니다. 3.2.3-3.2.5단계를 반복하여 2.2.4단계의 음성 대조 용액을 캡슐화합니다.
참고: 샘플 수집은 15분 이내에 완료되어야 합니다. - 액적이 생성되는 동안 수집 튜브를 얼음에 보관하거나 실험 후 4시간 동안 1°C에 두어 아가로스가 액적 내부에서 겔화되도록 합니다.
참고: 미세유체 칩은 마이크로채널이 막히지 않은 상태로 유지되고 동일한 현탁액이 로드되는 경우 재사용할 수 있습니다. 에멀젼 생성을 완료한 후 피펫 팁을 폐기합니다(단계 3.2.3–3.2.5).
- 군집 성장과 에멀젼으로부터의 방출
- 박테리아가 포함된 겔 미세방울과 방울의 음성 대조군을 37°C로 설정된 배양 챔버로 옮깁니다.
- 충분한 군체가 자랄 수 있도록 최소 4시간 또는 밤새 미세 방울을 배양합니다. 음성 대조군에서 오염 징후가 보이지 않는지 명시야 현미경을 통해 확인합니다.
- 에멀젼에서 콜로니를 분리하려면 피펫 또는 바늘이 있는 주사기를 사용하여 젤 마이크로드롭릿 에멀젼 아래에서 가능한 한 많은 오일을 제거합니다(여기서는 21G 크기의 바늘이 사용됨).
- 50μL의 겔 미세입자를 새 마이크로튜브로 옮기고 추가 액적 분석을 위해 4°C에 보관합니다. 나머지 에멀젼에 에멀젼과 동일한 부피로 1H, 1H, 2H, 2H-perfluoro-1-octanol (PFO)과 불소 오일의 1 : 1 혼합물을 첨가합니다.
- 에멀젼 위에 약 200μL의 인산염 완충 식염수(PBS) 완충액 또는 0.9% w/v NaCl 완충액을 추가합니다. 혼합물을 소용돌이치고 고정 속도 원심 분리기에서 잠시 회전시킵니다.
- 액체 계면 바닥에서 유상을 조심스럽게 제거하고 상단에서 100μL의 PBS를 버립니다. 3.3.4–3.3.5단계를 반복하여 오일 잔류물이 최소화되거나 전혀 없는 PBS 완충액에서 세척된 마이크로겔을 얻습니다.
참고: 마이크로겔은 액체 계면에 침전합니다. 오일 상태와 함께 제거하지 마십시오.
4. 단일 식민지 분석
- 세포 염색
참고: 이 프로토콜에는 다양한 염색 조합이 작동합니다. 본질적으로 플라스미드에 의해 인코딩된 형광 단백질과 구별되는 색을 가진 DNA 또는 세포벽 염색을 선택해야 하며 형광 현미경에서 사용 가능한 필터 조합을 사용하여 분석할 수 있습니다. 여기서, 세포의 DNA는 플라스미드에 암호화된 녹색 형광 단백질과 형광을 구별하기 위해 Propidium Iodide(PI)로 염색되었지만, 다른 많은 DNA 염색을 사용할 수 있습니다.
주의: PI는 잠재적인 발암 물질이므로 적절한 개인 보호 장비와 함께 취급해야 합니다. 염료를 현지 규정에 따라 안전하게 폐기하십시오.- 세척된 마이크로겔을 실온에서 약 80 x g 으로 5분 동안 원심분리합니다. 피펫을 사용하여 상등액을 폐기합니다.
- 50 uL의 마이크로겔을 새로운 마이크로튜브로 옮겨 에탄올 처리를 위한 음성 대조군 역할을 합니다.
참고: PI 염색은 에탄올 처리의 영향을 받는 세포와 같이 손상된 막이 있는 세포에만 침투합니다. - 나머지 마이크로겔에 동일한 부피의 70% 에탄올을 첨가하고 소용돌이와 짧게 혼합합니다. PI 염색을 위해 박테리아 막을 투과화하기 위해 실온에서 15분 동안 배양합니다. 4.1.1단계를 반복합니다.
- 동일한 부피의 0.9% w/v NaCl을 마이크로겔에 추가하고 간단히 와류를 일으킵니다. 4.1.1단계를 반복합니다.
- 두 마이크로튜브 샘플에 2μL의 PI(1mg/mL)를 추가합니다. 잘 섞어 어두운 곳에서 실온에서 15분 동안 배양합니다.
참고: 음성 대조군이 형광 현미경 검사를 통해 적색 형광 신호를 나타내는 경우, 이전 단계에서 다른 용액에 노출되는 동안 집락의 무결성이 손상되었을 수 있습니다.
- 현미경 검사 법
참고: 액적과 마이크로겔은 도립 에피 형광 현미경으로 이미지화하여 마이크로겔 내 박테리아 군집의 액적 크기 분포와 형광을 얻습니다(그림 2). 여기서 오픈 소스 도립 현미경 플랫폼(https://github.com/wenzel-lab/SQUID-bioimaging-platform)17 은 10x 0.3NA 대물렌즈, 명시야 조명용 백색 LED 어레이, 여기용 470nm LED와 함께 사용됩니다. 상업용 에피 형광 현미경은 액적과 마이크로겔을 이미지화하는 데 사용할 수 있습니다. 조명과 필터는 사용되는 모델, 브랜드 및 형광 단백질에 따라 다르기 때문에 보정이 필요합니다.- 2 μL의 겔 마이크로액적을 이미징 챔버 슬라이드로 옮기고 5 μL의 불소 오일을 첨가하여 최적의 이미징을 위한 단층 액적을 형성합니다.
참고: 세포 계수 챔버 또는 간단한 미세유체 챔버는 에멀젼을 얇게 펴고 건조 과정을 늦추는 데 도움이 될 수 있습니다. - 현미경의 명시야 이미징을 위해 상단에서 백색 LED 매트릭스 조명을 활성화합니다. 준비된 슬라이드를 장착하고 샘플에 초점을 맞추고 물방울의 단층을 찾습니다. 명시야 이미지를 캡처합니다.
- 샘플을 이동하지 않고 여기를 위해 470nm LED로 전환하여 집락의 형광 이미지를 캡처합니다. sfGFP 이미징을 위해 녹색 파장 필터와 정렬되도록 필터 휠을 조정합니다. 액적 단층이 포함된 모든 영역을 스캔하고 4.2.2–4.2.3 단계를 반복하여 액적 분석에 대한 통계적 견고성을 보장합니다.
- 염색된 마이크로겔 2 μL를 이미징 챔버 칩으로 옮기고 0.9% w/v NaCl 5 μL를 전달하여 마이크로겔 단층을 형성합니다. 이미징 중 증발을 방지하기 위해 칩의 입구와 출구를 밀봉합니다.
참고: 증발은 형광 라벨의 공동 국소화에 영향을 줄 수 있습니다. 미세유체 챔버는 장시간 이미징을 위해 권장됩니다. - 4.2.2–4.2.3단계를 반복합니다. PI 이미징을 위해 빨간색 파장 간격 필터를 조정하고 해당 이미지를 캡처합니다.
- 한 위치에서 이미징을 완료한 후 마이크로젤 단층으로 슬라이드에서 다음으로 적합한 영역을 찾고 이미징 프로세스를 반복하여 샘플의 통계적 견고성과 포괄적인 분석을 보장합니다.
- 2 μL의 겔 마이크로액적을 이미징 챔버 슬라이드로 옮기고 5 μL의 불소 오일을 첨가하여 최적의 이미징을 위한 단층 액적을 형성합니다.
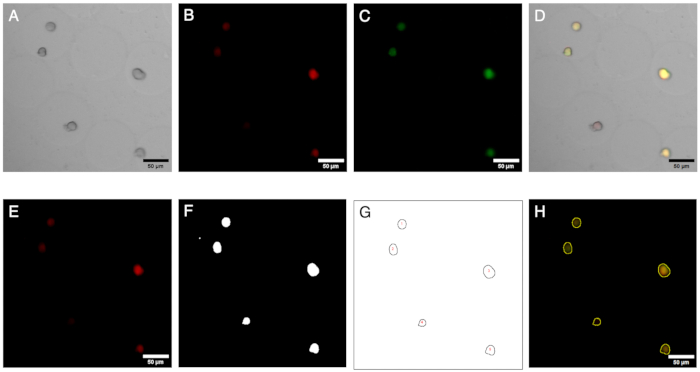
그림 2: 현미경 이미지 및 분석. 형광 이미징 및 마이크로겔 내 콜로니 분석. (가-씨) 도립 현미경을 사용한 명시야 및 형광 현미경을 통해 획득한 이미지 채널. 합성 이미지(D)는 마이크로겔 내에 음성 콜로니(적색 형광만 해당)의 존재를 보여줍니다. (E–H) 이미지 분석 워크플로우의 결과. ROI를 생성하면 빨간색 및 녹색 채널에서 콜로니를 식별할 수 있으며 신호를 정량화하여 음의 콜로니의 존재를 정의할 수 있습니다. 스케일 바: 50μm. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
- 이미지 분석
참고: 캡슐화된 집락의 형광 신호를 분석하고 희귀 이벤트를 식별하기 위해 명시야 및 형광 이미징의 이미지를 Fiji/ImageJ로 처리할 수 있습니다(그림 3). 이러한 단계는 매크로 스크립트로 구현할 수 있으며 매개변수 값은 광학 구성에 따라 달라질 수 있습니다.- 녹색 및 빨간색 채널에서 이미지를 엽니다. 좌표 (500, 500)에서 시작하여 너비와 높이가 각각 2000픽셀인 직사각형 관심 영역(ROI)을 정의합니다.
참고: 이러한 매개변수는 3000 x 3000 픽셀 이미지에 적용되며 ROI는 더 나은 조명으로 영역을 정의합니다. - 정의된 사각형 ROI로 이미지를 자릅니다. 각 픽셀의 강도에서 상수 값 10을 빼고 이미지에 매끄럽게 하는 필터를 적용하여 배경 노이즈를 줄이고 관심 있는 개체를 더 뚜렷하게 만듭니다.
참고: 이 영역만 추가 분석에 사용됩니다. - red 채널에서 이미지를 복제하고 바이너리 마스크로 변환하여 플라스미드와 관련된 희귀 이벤트를 식별합니다. 임계값을 10에서 255 사이로 설정합니다. 이 범위 내의 픽셀은 전경(관심 개체)으로 간주되고 다른 픽셀은 배경으로 처리됩니다.
- 형태학적 연산을 수행하여 작은 간격을 좁히고 관심 객체 내의 구멍을 채웁니다. watershed 알고리즘을 적용하여 이진 마스크 내에서 겹치는 개체를 분리합니다.
- 바이너리 마스크의 입자를 분석합니다. 크기가 30픽셀보다 크고 원형도가 0.50에서 1.00 사이인 입자만 고려하십시오. 결과가 요약되어 결과 테이블에 추가됩니다. 추가 시각화를 위해 입자 분석에서 감지된 ROI 세트를 저장합니다.
- 녹색 채널과 빨간색 채널의 영상에 저장된 ROI를 표시합니다. 이 이미지 내에서 ROI의 강도 또는 기타 속성을 측정합니다. 결과 테이블에 측정값을 기록하고 추가 통계 분석을 위해 결과를 별도의 CSV 파일에 저장합니다.
- 녹색 및 빨간색 채널에서 이미지를 엽니다. 좌표 (500, 500)에서 시작하여 너비와 높이가 각각 2000픽셀인 직사각형 관심 영역(ROI)을 정의합니다.
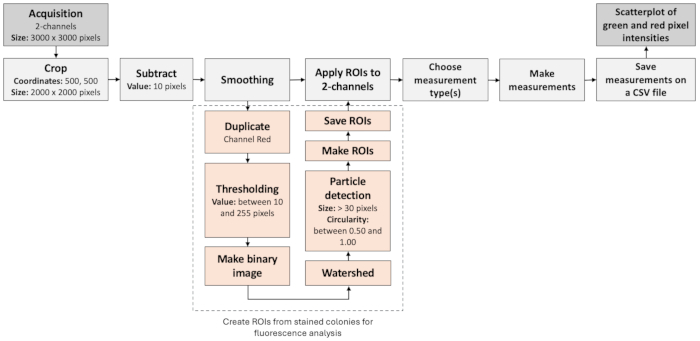
그림 3: 음성 콜로니를 식별하기 위한 이미지 분석 워크플로우. 이 그림은 형광 이미지를 자동으로 처리하고 평가하는 단계별 워크플로우를 보여줍니다. 워크플로우는 형광 라벨의 공동 국소화(colocaliation)와 입자 분석을 기반으로 합니다. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
5. 한천 플레이트 비교 분석
참고: 액적 방법을 기존 플레이트 분석법과 비교하기 위해 2.1.8단계에서 동일한 E. coli 균주의 형광 콜로니를 정량화했습니다. 페트리 접시를 사용하여 수행되었습니다. 이는 sfGFP 플라스미드 안정성을 측정하기 위한 아날로그 제어 방법으로 사용되었습니다. 그림 1의 방법 그림도 참조하십시오.
- Cell Harvesting 섹션의 2.1.1단계에서 설명한 대로 LB-한천(항생제 제외)을 준비합니다. 한천을 60 °C 이하로 냉각하고 균질화합니다.
- 멸균 환경에서 15mL의 한천 배지를 각 페트리 접시에 붓습니다. 사용할 준비가 될 때까지 뚜껑을 부분적으로 열어 플레이트가 굳어지도록 합니다.
- LB 배지에서 E. coli 배양을 각각 10μL씩 3개의 플레이트에 0.007 ± 0.002의 OD600을 접종합니다. L자형 스프레더를 사용하여 골고루 펴 바릅니다. 플레이트를 37 ° C에서 닫고 뒤집어 배양합니다. 동일한 조건에서 박테리아가 없는 제어판을 준비합니다.
- 24시간 후, 오픈 소스 형광 이미징 시스템(https://github.com/RudgeLab/FluoPi)인 FluoPi를 사용하여 플레이트의 형광 이미지를 캡처합니다15.
참고: FluoPi는 470nm를 중심으로 하는 파장의 청색 여기광이 있는 Raspberry Pi 카메라와 아크릴 여기 및 방출 필터로 구성됩니다. - 캡처된 이미지를 사용하여 각 플레이트의 형광 및 비형광 콜로니를 수동으로 계산합니다.
결과
cell encapsulation 및 microcolony formation의 검증
세포 캡슐화는 에멀젼을 분해하고 마이크로겔을 세척하기 전에 겔 미세방울에 대해 명시야 현미경 검사를 수행하여 육안으로 확인할 수 있습니다. 이 단계에서 에멀젼의 대표적인 결과는 그림 4에 나와 있습니다.
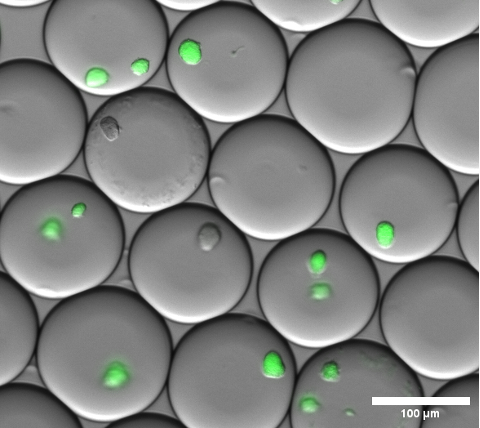
그림 4: 형광 현미경 검사 오버레이 이미지의 단면. 하룻밤 배양 후, 겔 미세방울 내부에서 E. coli 집락을 발현하는 sfGFP의 대표적인 미세군집체. 10배 배율과 0.30 NA의 현미경 대물렌즈를 사용했습니다. 눈금 막대 : 100 μm. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
이미지 분석 결과
마이크로겔이 염색되고 명시야가 지정되고 여러 위치에서 형광 채널이 획득되면 원본 이미지에서 음성으로 식별된 콜로니를 시각화할 수 있습니다( 그림 5A 참조). 한 실험의 모든 이미지에서 추출한 데이터를 플롯하여 다양한 콜로니의 형광 비율을 보여주고 플라스미드로 인코딩된 형광을 잃은 콜로니를 강조할 수 있습니다( 그림 5B 참조). 결과는 총 2785개의 분석된 마이크로 콜로니에서 100개의 콜로니가 플라스미드 또는 플라스미드 기능을 상실했으며, 이는 3.6%에 해당함을 나타냅니다.
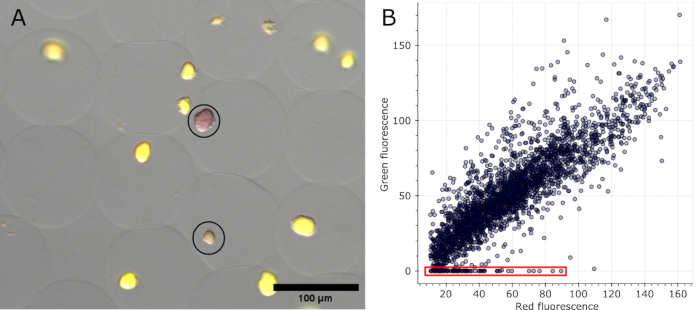
그림 5: negative microcolonies의 정량화. (A) 형광 현미경 검사 오버레이 이미지의 단면. 기름 제거 및 염색 후, sfGFP를 발현하는 마이크로겔의 대표적인 콜로니와 두 개의 음성 콜로니는 DNA 염색의 적색 형광(검은색 원으로 표시)을 보여주었습니다. 눈금 막대: 100μm. (B) 16개의 다중 채널 현미경 이미지에서 추출한 개별 미세군집의 형광 값의 산점도. 녹색 형광이 없는 집락은 그래프에서 빨간색으로 표시된 것처럼 음성으로 계산되었습니다. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
한천 플레이트 정량화
삼중 플레이트의 이미지는 그림 6에 나와 있으며, 비형광 콜로니는 흰색 화살표로 표시됩니다. 첫 번째 플레이트(그림 6A)에는 총 213개의 콜로니가 표시되었으며 그 중 1개는 형광이 아니었습니다. 두 번째 플레이트(그림 6B)에는 총 49개의 콜로니가 있으며 비형광 콜로니가 없습니다. 세 번째 플레이트(그림 6C)는 총 252개의 콜로니를 보여주었으며 그 중 6개는 형광이 아니었습니다. 이러한 결과는 평균 콜로니 플라스미드 손실률 2.3%에 해당하며, 표준 편차는 3.2입니다.
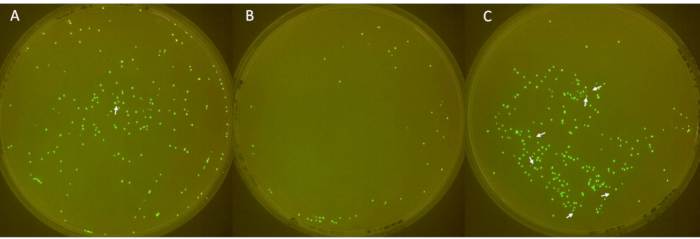
그림 6: 플레이트의 음성 콜로니 식별. (가-씨) LB-한천 플레이트의 형광 및 비형광 대장 균 집락(직경: 90mm, 높이: 15mm). -80°C 스톡에서 sfGFP를 함유한 대장 균에서 유래한 접종물을 1일째에 줄무늬를 만들고, 2일째에 항생제로 배양하고, 3일째부터 6일까지 매일 1:100씩 희석하여 플라스미드 손실을 허용했습니다. 콜로니를 37°C에서 24시간 동안 배양하고 FluoPi 챔버에서 이미지화했습니다. 비형광 집락은 김프로 강화되고 흰색 화살표로 표시되었습니다. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
토론
겔 마이크로드롭릿 기반 방법은 sfGFP와 같은 형광 단백질의 플라스미드 인코딩 유전자 발현이 있거나 없는 콜로니를 효과적으로 식별하고 정량화하는 것으로 입증되었습니다. 플라스미드 산물을 충분히 발현하지 않는 콜로니는 모든 콜로니를 염색하고 다른 방출 파장을 특징으로 하는 형광 DNA 염색(여기서는 Propidium Iodide)을 사용하여 식별합니다. 오픈 소스 기술을 활용하는 액적 미세유체역학(droplet microfluidics), 겔화(gelling) 및 형광 현미경(fluorescence microscopy)의 이러한 통합은 많은 연구 환경에서 고급 워크플로우를 실행할 수 있도록 합니다11,13. 겔-마이크로액적의 성공적인 생성은 세포 용해, 단일 게놈 증폭, 대사 세포 상호 작용 스크리닝, 배지 교환 등을 포함한 고급 분자 생물학 단일 세포 워크플로우를 가능하게 합니다 8,9. 이러한 장점은 이 프로토콜에서 사용되어 기존의 플레이트 기반 분석보다 더 확장 가능한 방식으로 미세군체를 성장, 염색 및 분석하는 데 사용됩니다.
중요한 단계
캡슐화 프로세스는 프로토콜의 중요하고 섬세한 부분입니다. 성분 농도, 유속 및 압력의 정밀한 제어는 특정 크기 범위 내에서 균일한 마이크로겔을 생성하고 액적당 평균 세포 수를 제어하는 데 필요합니다. 또한, 세포-아가로스 혼합물의 농도와 온도를 유지하면 응집 또는 조기 겔화를 방지할 수 있습니다. 피펫 팁에서 액체 아가로스 셀 현탁액의 온도 제어는 주사기 펌프 및 튜브의 온도를 제어하려는 노력에 비해 훨씬 쉽고 강력한 마이크로겔 생성을 제공하는 오픈 소스 하드웨어 미세유체역학 워크스테이션의 특히 유리한 구현입니다. 세포는 캡슐화 및 배양 전에 아가로스 성장 배지와 혼합되기 때문에 주요 세포 농도 변화를 피하기 위해 아가로스 마이크로겔을 빠르게 생성해야 합니다. 이를 위해 Abate 등에서 영감을 받은 액적 분할 미세유체 칩 설계가 최적화되었습니다18.
수정 및 문제 해결
원래 프로토콜을 개선하기 위해 몇 가지 보정 및 수정이 필요했습니다. 아가로스의 캡슐화는 일반 유중수 방울보다 훨씬 더 까다롭기 때문에 수성 상 흐름이 균일한 입자 크기 범위를 달성하도록 보장하면서 아가로스를 액체 상태로 유지하기 위한 시스템 설계가 필요합니다. 겔화로 인한 아가로스 점도의 변화는 유속에 영향을 미쳐 입자 크기가 커집니다. 현미경 검사에서는 명확한 차별화를 위해 겹치지 않는 여기 및 방출 신호를 보장하기 위해 필터와 광원을 신중하게 선택해야 합니다. 초기에는 DAPI가 박테리아를 염색하기 위해 선택되었지만, 방출 신호가 sfGFP와 겹쳐 청색 검출 채널에서 sfGFP가 검출되었습니다. PI로 전환한 이유는 PI의 방출이 장파장(적색광)에서 sfGFP와 잘 분리되어 있기 때문입니다.
제안된 방법을 사용하여 플라스미드 손실을 정량화했지만, 사용된 sfGFP 플라스미드는 의외로 안정적이었으며, pH9 배지 및 40°C에서의 배양과 같은 스트레스 조건에서도 항생제 없이 배양된 1세대 세포에서 플라스미드 손실 사례가 거의 나타나지 않았습니다. 이러한 관찰은 다른 연구 그룹의 연구 결과와 일치한다 1,19. 플라스미드 안정성은 초기 세포 배양 세대에 대한 이 방법의 완전한 정량 능력에 대한 입증을 제한했지만, 이 방법이 플라스미드 머무름의 작은 차이도 감지할 수 있을 만큼 충분히 민감하다는 것을 보여주었습니다. 초기 세대에서 높은 플라스미드 안정성의 관찰은 표적 박테리아 억제와 같은 음성 선택 분석을 사용하는 액적 미세유체 스크리닝에 중요한 의미를 갖습니다. 이는 selection target의 plasmid loss가 false positive selection result의 낮은 원인임을 의미합니다. 액적 미세유체 스크리닝은 일반적으로 피펫팅 로봇 워크플로우와 같은 다른 고처리량 스크리닝을 처리량의 크기만큼 능가하기 때문에 이러한 드문 이벤트를 평가하고 고려해야 합니다.
제한
장점에도 불구하고 제시된 방법에는 한계가 있습니다. 미세유체 장치 제작에는 전문 지식과 세부 사항에 대한 세심한 주의가 필요하며, 결정론적 캡슐화 효율성을 보장하기 위해 유속의 엄격한 실험적 제어가 필요합니다. 이러한 측면은 다양한 실험 설정에 대한 최적화가 필요할 수 있습니다. 이 방법은 신호 감지를 위해 형광 현미경에 의존하기 때문에 적절한 이미징 장비에 액세스해야 하지만, 이 장비는 오픈 소스 하드웨어를 사용하여 제작할 수 있으므로 접근성이 더 좋습니다. 또한 마이크로겔은 대형 노즐을 사용하여 상용 유세포 분석에서 처리할 수 있어 접근성과 실험 처리량을 더욱 향상시킬 수 있습니다. 이 세포 분석에는 액적 분류기도 사용할 수 있습니다.
또한, 이 방법은 플라스미드, 염색제 또는 기타 마커에서 형광 신호를 검출하도록 설계되었지만, 형광 표지가 가능한 세포로 제한되며, 이는 모든 박테리아 균주 또는 실험 조건에 적용되지 않을 수 있습니다. 그러나 이 방법은 위상차 또는 명시야 현미경과 같은 다른 유형의 현미경을 통합하도록 조정할 수 있으므로 형광 이외의 표현형 분석 응용 분야가 가능합니다. 또한 FTIR 또는 라만 분광법과 같은 분광 기술과 결합하여 캡슐화된 세포의 화학 조성 및 구조 정보를 분석하는 기능을 확장할 수 있습니다. 이러한 적응은 적용 범위를 넓혀 다양한 연구 환경을 위한 다목적 도구가 됩니다.
중요성과 응용 프로그램
플라스미드 손실19 에 대한 전통적인 분석법은 발현을 잃은 세포의 비율, 즉 실험법 설계 및 다양한 생물학적 응용에서 매우 중요할 수 있는 정보를 양호하게 정량화할 수 없습니다. 일반적으로 콜로니 유형은 한천 플레이트 분석에서 열거되며, 여기서 그림 4와 같이 잘 정의된 격리된 콜로니를 얻을 수 있습니다. 그러나 겹치는 군체는 자신 있게 식별하기 어렵습니다. 우리 손에서 항상 최적의 콜로니 밀도를 얻을 수 있는 것은 아니며, 저주파 플라스미드 손실 이벤트에 대한 좋은 통계를 얻기 위해서는 많은 플레이트가 필요합니다. 제안된 방법은 미세방울에서 콜로니가 개별적으로 발달하고 더 작으며 이미징 챔버에 쉽게 로드할 수 있어 큰 콜로니 수에 대한 현미경 또는 유세포 분석 기반 정량화가 가능하기 때문에 한천 플레이트 아날로그 방법보다 더 많은 수의 콜로니를 가진 고립된 콜로니에서 오는 형광 신호를 정확하게 정량화할 수 있는 보다 강력한 접근 방식을 제공합니다. 이를 통해 분석법의 통계적 표현을 크게 개선하고 다른 겔-마이크로액적 워크플로우에 통합할 수 있습니다.
오픈 소스 하드웨어11,20의 사용을 통해 연구원은 미세 유체 워크 스테이션 설계를 사용자 정의하고 흐름 랫을 정확하게 조정할 수 있습니다. 따라서 입자 크기는 다양한 세포 유형과 실험 조건을 지원합니다. 이러한 유연성은 위상차 또는 분광법과 같은 다른 현미경 유형을 잠재적으로 통합하는 것으로 확장되어 방법의 적용 가능성을 넓힙니다. 다양한 조건에서 플라스미드 안정성을 평가할 수 있는 이 방법의 용량은 항생제 선택 없이 플라스미드 잔류가 필요한 응용 분야, 특정 스트레스 조건 또는 다양한 배양 세대에 매우 중요합니다. 제시된 방법의 다양성과 적응성은 합성 생물학, 환경 모니터링 및 임상 진단을 포함한 분야의 다양한 연구 응용 분야에 가치가 있습니다2.
공개
저자들은 어떠한 경쟁적인 재정적 이해관계나 개인적 관계도 이 논문에 보고된 연구에 영향을 미칠 수 없었다고 선언한다.
감사의 말
이 작업은 ANID FONDECYT Regular 1241621와 Chang Zuckerberg Initiative 프로젝트 'Latin American Hub for Bioimaging Through Open Hardware'에서 T.W.에 부여한 자금 지원 프로젝트의 일부입니다. T.W.는 또한 CIFAR MacMillan Multiscale Human 프로그램의 Azrieli Global Scholar로서 CIFAR의 자금 지원에 감사드립니다.
자료
| Name | Company | Catalog Number | Comments |
| 1H,1H,2H,2H-Perfluoro-1-octanol | Sigma-Aldrich | 370533-25G | For breaking emulsion |
| 70% ethanol | For cell permeabilization | ||
| Agar-Agar | Winkler | 9002-18-0 | |
| Biopsy Punch | 0.75 mm and 1.8 mm | ||
| Blue LED transilluminator | IO Rodeo | ||
| Culture tube | 15 mL | ||
| Desiccator | With vacuum pump | ||
| Disposable cup | For mixing PDMS | ||
| Disposable fork | For mixing PDMS | ||
| E. coli TOP10 strain | |||
| FluoPi microscope | https://github.com/wenzel-lab/FluoPi | Green fluorescence imaging system for analyzing plates | |
| Fluorinated Oil | 3M | Novec 7500 | |
| Glass slide heater | https://github.com/wenzel-lab/modular-microfluidics-workstation-controller/tree/master/module-heating-and-stirring | For controlling the temperature at 40 °C of microfluidic chip | |
| Glass Slides | |||
| Hotplate | Mechanic | For evaporating Aquapel | |
| Image analysis software | Fiji/ImageJ | 2.14.0/1.54f | |
| Incubator | Mundo Lab | MLAB Scientific / For incubation of plates and microgels | |
| Isopropanol | For cleaning glass slides | ||
| Kanamycin | 100 ug/mL concentration | ||
| L-shaped spreader | For spreading bacteria on agar plates | ||
| Master mold | Chip design on silicone or glass wafer | ||
| Microtubes | 2 mL | ||
| NaCl solution | Sodium chloride 0.9% w/v | ||
| Open-source hardware strobe-enhanced microscopy stage | https://github.com/wenzel-lab/flow-microscopy-platform | For bright-field microscopy | |
| Petri dish | Citotest | 2303-1090 | 90 x 15 mm |
| Pipette tip heater | https://github.com/wenzel-lab/modular-microfluidics-workstation-controller/tree/master/module-heating-and-stirring | For controlling the temperature at 40 °C of pipette tip | |
| Plasma Cleaner | Diener Electronic | 117056 | For bonding PDMS with a glass slide |
| Plasmid pCA_Odd1 | Encodes sfGFP and kanamycin resistance | ||
| Polytetrafluoroethylene (PTFE) tubing | Adtech Polymer Engineering Ltd | ||
| Pre-mixed Luria Bertoni medium | US Biological Life Science | L1520 | |
| Propidium iodide (PI) | For staining | ||
| Raspberry Pi-based pressure and flow controller system | https://github.com/wenzel-lab/modular-microfluidics-workstation-controller | For controlling pressure and flow rates | |
| Silicone elastomer base | Sylgard | PDMS kit - 184 Silicone Elastomer Kit | |
| Silicone elastomer curing agent | Sylgard | PDMS kit - 184 Silicone Elastomer Kit | |
| Spectrophotometer | For measuring absorbance | ||
| SQUID microscope | https://github.com/wenzel-lab/SQUID-bioimaging-platform | Multi-fluorescence imaging system for analyzing stained cells | |
| Sterile loop | For picking a colony and streaking plating | ||
| Surfactant | Sphere Fluidics | Pico-Surf | |
| Syringes | NIPRO | With filters and tubing | |
| Temperature-controlled shaker | Mundo Lab | DLAB HCM100-Pro | |
| Tweezer | |||
| Ultra-low gelling temperature agarose | Sigma-Aldrich | A2576-5G | For generating hydrogel beads |
| Water repelent solution (fluoroalkyl silane) | Aquapel | For treating microchannels of PDMS device |
참고문헌
- Wein, T., Hülter, N. F., Mizrahi, I., Dagan, T. Emergence of plasmid stability under non-selective conditions maintains antibiotic resistance. Nat Commun. 10 (1), 2595 (2019).
- Rodríguez-Beltrán, J., DelaFuente, J., León-Sampedro, R., MacLean, R. C., Millán, &. #. 1. 9. 3. ;. S. Beyond horizontal gene transfer: The role of plasmids in bacterial evolution. Nat Rev Microbiol. 19 (6), 347 (2021).
- Wein, T., Dagan, T. Plasmid evolution. Curr Biol. 30 (19), R1158-R1163 (2020).
- Chen, S., Larsson, M., Robinson, R. C., Chen, S. L. Direct and convenient measurement of plasmid stability in lab and clinical isolates of E. coli. Sci Rep. 7 (1), 4788 (2017).
- Rouches, M. V., Xu, Y., Cortes, L. B. G., Lambert, G. A plasmid system with tunable copy number. Nat Commun. 13 (1), 3908 (2022).
- Silva, F., Queiroz, J. A., Domingues, F. C. Evaluating metabolic stress and plasmid stability in plasmid DNA production by Escherichia coli. Biotechnol Adv. 30 (3), 691-708 (2012).
- Wang, R., et al. Construction of novel pJRD215-derived plasmids using chloramphenicol acetyltransferase (cat) gene as a selection marker for Acidithiobacillus caldus. PLoS ONE. 12 (8), e0183307 (2017).
- Moragues, T., et al. Droplet-based microfluidics. Nat Rev Methods Primers. 3 (1), 32 (2023).
- Vitalis, C., Wenzel, T. Leveraging interactions in microfluidic droplets for enhanced biotechnology screens. Current Opinion in Biotechnology. 82, 102966 (2023).
- Sinha, N., Subedi, N., Wimmers, F., Soennichsen, M., Tel, J. A pipette-tip based method for seeding cells to droplet microfluidic platforms. J Vis Exp. (144), e57848 (2019).
- Wenzel, T. Open hardware: From DIY trend to global transformation in access to laboratory equipment. PLOS Biol. 21 (1), e3001931 (2023).
- Murillo, L. F. R., Wenzel, T. Welcome to the journal of open hardware. J Open Hardware. 1 (1), (2017).
- Shin, J. H., Choi, S. Open-source and do-it-yourself microfluidics. Sens Actuators B Chem. 347, 130624 (2021).
- Pryszlak, A., et al. Enrichment of gut microbiome strains for cultivation-free genome sequencing using droplet microfluidics. Cell Rep Methods. 2 (1), 100137 (2021).
- Pollak, B., et al. Universal loop assembly: open, efficient and cross-kingdom DNA fabrication. Biology. 5 (1), (2020).
- Volkmer, B., Heinemann, M. Condition-dependent cell volume and concentration of Escherichia coli to facilitate data conversion for systems biology modeling. PLoS ONE. 6 (7), e23126 (2011).
- Li, H., et al. Squid: Simplifying quantitative imaging platform development and deployment. bioRxiv. , (2020).
- Abate, A. R., Weitz, D. A. Faster multiple emulsification with drop splitting. Lab Chip. 11 (11), 1911-1915 (2011).
- Lau, B. T. C., Malkus, P., Paulsson, J. New quantitative methods for measuring plasmid loss rates reveal unexpected stability. Plasmid. 70 (3), 353-361 (2013).
- Oellermann, M., et al. Open hardware in science: The benefits of open electronics. Integr Comp Biol. 62 (4), 1061-1075 (2022).
재인쇄 및 허가
JoVE'article의 텍스트 или 그림을 다시 사용하시려면 허가 살펴보기
허가 살펴보기더 많은 기사 탐색
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. 판권 소유