Method Article
Séparation et caractérisation différentielle des vésicules extracellulaires microbiennes intestinales chez des rats sensibles au sel dans des conditions de régime riche en sel
* Ces auteurs ont contribué à parts égales
Dans cet article
Résumé
Le protocole décrit l’isolement des VE microbiennes intestinales de rats sensibles au sel nourris avec des HSD à l’aide de la centrifugation à gradient de densité. Les VE ont été caractérisées par le suivi des nanoparticules, le TEM, les tests LPS/BCA et le séquençage de l’ARNr 16S pour analyser la taille, la morphologie, la composition et l’origine du microbiote.
Résumé
Une consommation élevée de sel est un facteur de risque majeur d’hypertension, et son mécanisme sous-jacent peut être étroitement lié aux vésicules extracellulaires (VE) sécrétées par le microbiote intestinal. Ces VE, produits par le microbiote intestinal, portent divers composants bioactifs qui peuvent jouer un rôle crucial dans le développement de l’hypertension induite par un régime riche en sel (HSD). Pour étudier ce mécanisme, nous avons développé une méthode d’extraction efficace basée sur la centrifugation à gradient de densité pour isoler les VE du microbiote intestinal de rats sensibles au sel nourris avec un HSD. Grâce à l’analyse granulométrique, à la microscopie électronique à transmission (MET) et à la détection de lipopolysaccharides (LPS), nous avons identifié la distribution du gradient des VE du microbiote intestinal et obtenu une extraction précise. De plus, le séquençage du gène de l’ARNr 16S a été utilisé pour analyser l’origine et les différences de composition des VE entre les groupes normaux et HSD, révélant l’impact d’une consommation élevée de sel sur les caractéristiques génétiques des VE du microbiote intestinal. Cette étude fournit des outils précieux et des informations scientifiques sur les mécanismes du microbiote intestinal sous-jacents à l’hypertension induite par le sel et offre de nouvelles perspectives pour la prévention et le traitement des maladies connexes.
Introduction
Le microbiote intestinal, également connu sous le nom de microflore du microbiote intestinal ou microécologie intestinale, est un complexe de dizaines de milliers de micro-organismes situés dans le tractus gastro-intestinal biologique et joue un rôle crucial dans le maintien de la santé humaine1. Ces dernières années, grâce à d’autres recherches, il a été constaté que le microbiote intestinal peut produire des vésicules extracellulaires (VE)2. Les VE sont de petites vésicules libérées par les cellules, qui transportent diverses molécules dans la cellule, telles que des protéines, des acides nucléiques et des lipides 3,4. Ils peuvent interagir avec d’autres microbes5, des cellules épithéliales intestinales et même des tissus et des organes distants6, affectant ainsi la santé du corps humain 7,8. Il existe un lien étroit entre les VE produites par ces microbiotes intestinaux et l’alimentation9.
Les VE produites par le microbiote intestinal peuvent être des agents importants par lesquels un régime riche en sel (HSD) affecte la santé du corps. Non seulement la HSD perturbe directement l’équilibre du microbiote intestinal10, entraînant une réduction significative du nombre de bactéries bénéfiques (comme les Lactobacillus)11, mais favorise également la prolifération de bactéries nocives (comme les Bacteroides, etc.)12. Ce déséquilibre réduit la fonction de barrière intestinale et augmente le risque d’inflammation intestinale. De plus, un HSD affecte également l’équilibre acido-basique et l’absorption des nutriments dans l’intestin en modifiant les activités métaboliques13 du microbiote intestinal, telles que la réduction de la production d’acides gras à chaîne courte14,15 avec de multiples fonctions physiologiques.
Ces changements ont non seulement un impact sur la santé intestinale, mais peuvent également réguler indirectement la production et la libération de VE et modifier la composition et la fonction des VE. L’environnement à forte teneur en sel peut affecter les fonctions physiologiques normales des cellules intestinales, y compris la libération et le transport des VE, perturbant ainsi le rôle des VE dans la transmission de l’information intercellulaire et la régulation immunitaire. Dans le même temps, l’inflammation intestinale peut favoriser une variété de VE ayant des fonctions spéciales16 et se propager à l’ensemble du corps par l’axe intestin-organe et d’autres manières17,18, ce qui est étroitement lié à l’apparition et au développement de l’hypertension19,20, des maladies cardiovasculaires21 et cérébrovasculaires22,23, de l’obésité 24,25, du diabète26 et d’autres maladies chroniques.
Par conséquent, l’objectif global de cette étude était de développer une méthode efficace et fiable pour extraire les VE du microbiote intestinal de rats sensibles au sel nourris avec un HSD et d’étudier systématiquement leurs propriétés physiques, leur composition et leurs fonctions. En raison des caractéristiques de l’augmentation significative de la pression artérielle après un régime riche en sel, des rats sensibles au sel ont été sélectionnés et ont révélé l’effet de la HSD sur l’EV du microbiote intestinal en construisant une méthode d’extraction efficace. La méthode était basée sur la centrifugation à gradient de densité et combinait diverses techniques d’identification dynamique telles que la détection de la taille des particules, la mesure LPS/BCA, la microscopie électronique à transmission et l’analyse protéomique. Le protocole vise à révéler les effets de la HSD sur l’EV du microbiote intestinal et ses mécanismes dans les maladies cardiovasculaires. Grâce à sa grande efficacité, à sa reproductibilité et à sa large applicabilité, cette approche fournit non seulement un outil important pour explorer le mécanisme des VE du microbiote intestinal dans l’hypertension induite par le sel, mais jette également les bases théoriques pour développer des stratégies d’intervention contre les maladies basées sur les VE. Grâce à cette étude, nous espérons ouvrir de nouvelles voies pour la prévention et le traitement des maladies cardiovasculaires, telles que l’hypertension27,28.
Protocole
Cette étude expérimentale sur les animaux est conforme aux directives éthiques pertinentes et aux normes internationales. Les études impliquant des animaux ont été approuvées par le Comité d’éthique et de bien-être des animaux de laboratoire de l’Université de médecine chinoise de Chengdu (institution : Université de médecine chinoise de Chengdu ; numéro de protocole : 2018-21).
1. Préparation des animaux et régime alimentaire
- Acclimater des rats mâles de 6 semaines sensibles au sel Dahl, poids corporel 180-200 g, en les élevant à 50 % ± 10 % d’humidité ; cycle d’éclairage de 12 h / 12 h ; 20,0 ± 2,0 °C pendant 1 semaine dans des conditions spécifiques exemptes d’agents pathogènes.
- Par la suite, divisez les rats à parts égales en deux groupes (n = 6). Fournissez à chaque groupe un régime différent qui dure 6 semaines : un régime salé normal (NSD) avec 0,5 % de chlorure de sodium et un HSD avec 8 % de chlorure de sodium, qui est couramment utilisé dans les études publiées 29,30,31. Les souris ont eu un accès illimité à la nourriture et à l’eau tout au long de l’étude.
2. Surveillance de la pression artérielle
REMARQUE : La pléthysmographie du brassard de queue a été utilisée comme méthode non invasive pour la mesure de la pression artérielle, et l’enregistrement de la pression volumétrique (VPR) a été utilisé lorsque la pression artérielle a été mesurée à partir du volume sanguin de la queue.
- Placez les rats individuellement dans de petites cages avec de la sciure de bois, chauffées pendant 10 minutes à environ 37 °C et placez-les dans une retenue pour rongeurs personnalisée avec un nez réglable et une porte pour l’entrée des animaux. Placez les rats sur un coussin chauffant en position couchée pour maintenir leur température corporelle à 37 °C.
- Connectez le capteur VPR au bas de la queue et réglez au moins 10 cycles d’adaptation pour stabiliser le BP28.
- Remplissez le brassard gonflable sur la racine de la queue et placez le capteur pléthysmographique (ou capteur photoélectrique) en dessous. Gonflez lentement jusqu’à ce qu’elle dépasse la pression systolique (généralement 200 mmHg), puis relâchez lentement. L’instrument détecte automatiquement la valeur de pression de la disparition des ondes de pouls (pression artérielle systolique) et de la récupération (pression artérielle diastolique). Mesurez 3 à 5 fois consécutives et prenez la valeur moyenne.
- Entraînez tous les rats à s’adapter à la procédure de contention et effectuez cette étude dans un laboratoire silencieux vers 9 heures du matin pour minimiser les effets de la variation circadienne.
- Mesurez la pression artérielle systolique (TAS) et la pression artérielle diastolique (TAD) tous les deux jours avant le sacrifice. Estimation de la pression artérielle moyenne (PAM) à partir de la TAS et de la TAD
MAP = (TAS + 2 TAD)/3
3. Extraction des VE
- Prélèvement d’échantillons
- Trempez un coton-tige stérile dans de l’alcool à 75 % et utilisez-le pour stimuler l’anus du rat. La stimulation visait à favoriser le péristaltisme intestinal et à détendre le sphincter anal, favorisant ainsi la défécation. Une fois les matières fécales expulsées, collectez-les à l’aide d’une pince stérile dans des récipients stériles de taille appropriée et rangez-les immédiatement à -80 °C.
- Préparation des échantillons
- À l’aide d’une cuillère, transférez 5 g de matières fécales dans un tube à centrifuger prépesé de 50 ml. Ajouter 50 mL de PBS préchauffé à 37 °C sans endotoxines (concentration maximale de l’échantillon de 10 %) dans le tube et tourner pendant 30 min. Refroidissez la centrifugeuse à grande vitesse à 4 °C.
- Centrifugeuse à 8 000 x g pendant 15 min après avoir placé les échantillons symétriquement pour permettre à la centrifugeuse de s’équilibrer. Après la centrifugation, aspirez le surnageant et transférez-le dans un nouveau tube à centrifuger stérile.
- Centrifuger à nouveau à 8 000 x g pendant 15 min. Aspirez le surnageant et utilisez-le pour une analyse plus approfondie.
- Préparation de l’extrait brut
- Utilisez une unité de filtration stérile de 0,22 μm. Placez l’unité de filtration sur de la glace et tenez fermement la pompe à vide. Transférez le surnageant obtenu sur le dessus du filtre. Allumez la pompe à vide pour collecter les échantillons filtrés.
- Transvaser le filtrat dans un filtre centrifuge (10 kDa, 15 ml), centrifuger à 4 °C, 3 000 x g pendant 30 min et concentrer l’échantillon à au moins 1400 μL.
- Prélever l’échantillon concentré et, si nécessaire, le diluer à 1400 μL à l’aide de PBS pré-refroidi sans endotoxines. Conservez l’échantillon sur de la glace immédiatement après la dilution.
REMARQUE : L’extrait brut peut être utilisé immédiatement ou stocké à -80 °C pendant plusieurs mois.
- Séparation des VE
- Préparez le tampon de dégradé A et le tampon de dégradé B comme décrit ci-dessous. Préparez le tampon un jour à l’avance, car il faut plusieurs heures pour que le composé se dissolve complètement. Le tampon préparé peut être conservé à 4 °C pendant 6 mois.
- Préparation du tampon à gradient A : Dissoudre 0,25 mM de saccharose, 6 mM d’EDTA et 60 mM de Tris dans 800 mL d’eau exempte d’endotoxines par mélange magnétique, le pH étant ajusté à 7,4 à l’aide d’acide chlorhydrique. Diluer à 1 L avec de l’eau exempte d’endotoxines. Utilisez un filtre de 0,22 μm pour filtrer le tampon.
- Préparation du tampon à gradient B : Dissoudre 0,25 M de saccharose, 1 mM d’EDTA et 10 mM de Tris dans 800 mL d’eau exempte d’endotoxines par mélange magnétique, avec un pH ajusté à 7,4 avec de l’acide chlorhydrique. Dilué à 1 L avec de l’eau sans endotoxines. Utilisez un filtre à bouteille de 0,22 μm pour filtrer le tampon.
- Mélangez 1 volume de tampon de gradient A et 5 volumes de milieu de gradient de densité (le milieu est une solution d’iodixanol à 60 %) pour préparer la solution de travail.
- Préparation de la solution d’iodixanol (10 %, 20 %, 40 %) : Pour préparer une solution d’iodixanol à 10 %, mélangez 1 unité de solution de travail et 4 unités de tampon B ; pour une solution d’iodixanol à 20 %, mélanger 2 unités de solution de travail et 3 unités de tampon B ; pour une solution d’iodixanol à 40 %, mélanger 4 unités de solution de travail et 1 unité de tampon B. Préparez chaque solution fraîche et conservez-la sur de la glace.
REMARQUE : De nouvelles solutions fonctionnelles doivent être préparées pour chaque expérience, et un volume supplémentaire de 10 % de solutions peut être préparé pour tenir compte de l’erreur expérimentale. - Mélanger la solution préparée à l’étape 3.3.3 avec 7 mL de milieu à gradient de densité pour obtenir une solution d’iodixanol à 50 %.
- Pour préparer un gradient de densité, ajoutez une solution de bleu de trypan à une solution à 40 % et une solution d’iodixanol à 10 % (en poids/vol) pour obtenir un contour clair entre les différentes couches.
- Transvaser 8 mL de solution d’iodixanol à 50 % au fond du tube à centrifuger en polypropylène à paroi mince. Inclinez le tube à 70° et transférez 8 mL de solution d’iodixanol à 40 % sur la surface liquide. Ajouter 8 ml de solution d’iodixanol à 20 %, puis 7 ml de solution d’iodixanol à 10 %, et enfin 2 ml de PBS sans endotoxines.
REMARQUE : La préparation d’un gradient de densité nécessite de la pratique. La qualité du gradient de densité a un grand impact sur les résultats expérimentaux. Ne placez pas le tube à la verticale entre les différentes étapes du processus d’ajout, car cela a un impact négatif sur la qualité du gradient de densité. - Prérefroidissez la centrifugeuse à l’avance et placez le gradient de densité préparé dans l’ultracentrifugeuse. Paramètres d’entrée pour commencer, valeurs des paramètres : 100 000 x g, 20 h, 10 °C.
- Pour la séparation manuelle de la densité par gradient, prélevez lentement 2 ml de la solution à partir du centre de la surface du système contenant un total de 34 ml de liquide. Chaque 2 ml est un gradient, divisant ainsi la solution de gradient de densité en 17 gradients.
- Transférez les fractions de densité dans un tube d’échantillon stérile à l’aide d’une pipette et placez-les immédiatement sur de la glace. Clampez le tube de centrifugation entre le pouce et l’index pour vous assurer qu’il reste droit.
- Préparez le tampon de dégradé A et le tampon de dégradé B comme décrit ci-dessous. Préparez le tampon un jour à l’avance, car il faut plusieurs heures pour que le composé se dissolve complètement. Le tampon préparé peut être conservé à 4 °C pendant 6 mois.
- Récupération des VE
- À l’aide d’un système d’extraction entièrement automatisé des VE qui applique une oscillation en pression négative à travers un système d’oscillation à ultrasons à double couplage sur la puce d’ultrafiltration, éliminer les impuretés d’acide nucléique et de protéine dans l’échantillon à travers le nanopore et intercepter les VE, conduisant à l’enrichissement et à la purification des VE.
- Retirez le filtrat à l’étape 3.4.7 et ajoutez-le à la puce d’ultrafiltration.
- Faites fonctionner l’instrument conformément aux instructions de l’instrument et prélevez le filtrat filtré, à savoir la solution enrichie en exosomes. Le processus principal est illustré à la figure 1.
- À l’aide d’un système d’extraction entièrement automatisé des VE qui applique une oscillation en pression négative à travers un système d’oscillation à ultrasons à double couplage sur la puce d’ultrafiltration, éliminer les impuretés d’acide nucléique et de protéine dans l’échantillon à travers le nanopore et intercepter les VE, conduisant à l’enrichissement et à la purification des VE.
4. Identification des VE
- Détection de la taille et de la concentration des particules
- Évaluez la distribution de taille et le potentiel zêta à l’aide d’un compteur nanocoulométrique basé sur la détection d’impulsions résistives (RPS). Sélectionnez des puces nanoporeuses avec une plage de mesure de 60 à 200 nm pour cette expérience. De plus, évaluez la distribution granulométrique et le potentiel zêta des VE à l’aide d’un compteur de nanobibliothèques.
- Prenez une quantité appropriée de solution EVs et diluez-la (1/10, 1/100, 1/1000, 1/10000).
- Différentes dilutions de solutions de VE ont été placées sur l’instrument pour les tests.
- Le multiple de dilution avec une meilleure stabilité a été sélectionné et testé à nouveau pour obtenir des données plus précises.
- Évaluez la distribution de taille et le potentiel zêta à l’aide d’un compteur nanocoulométrique basé sur la détection d’impulsions résistives (RPS). Sélectionnez des puces nanoporeuses avec une plage de mesure de 60 à 200 nm pour cette expérience. De plus, évaluez la distribution granulométrique et le potentiel zêta des VE à l’aide d’un compteur de nanobibliothèques.
- Dosage LPS et BCA
- Dosage BCA : Lyser les VE à l’aide du tampon RIPA, puis séparer les protéines. Déterminer la teneur en protéines des VE à l’aide de la trousse de détermination de la concentration en protéines BCA.
- Test LPS : À l’aide du kit de détection d’endotoxines, ajoutez des échantillons, un détecteur d’endotoxines et chromogènes selon les étapes du kit. Mesurer l’absorbance à 545 nm et calculer l’expression LPS des vésicules externes selon la courbe standard.
- Évaluation TEM : Pour la coloration négative et l’analyse TEM, appliquer des échantillons sur une grille de cuivre luminescente recouverte d’un film de carbone continu et colorer avec 0,75 % de formiate d’uranyle. Pour la cryo-EM, absorbez les VE dans une grille de carbone EM de 300 mailles avec une surface hydrophile et congelez-les dans de l’azote liquide. Observez et analysez les grilles à l’aide du Cryo-TEM et enregistrez les images à 22 000 grossissements32.
- Caractérisation des protéines : Lyser les VE à l’aide d’un tampon RIPA puis séparer les protéines. Déterminer la concentration en protéines à l’aide du kit de dosage des protéines BCA, séparer des quantités égales de protéines de chaque échantillon par SDS-PAGE et colorer avec une solution de colorant bleu de Coomassie après électrophorèse pour visualiser les profils protéiques33,34.
- Séquençage et analyse S : Extraire l’ADN génomique des échantillons fécaux à l’aide d’un kit d’extraction d’ADN fécal. Évaluer l’intégrité et la concentration de l’ADN par électrophorèse sur gel d’agarose et spectrophotomètre. Amplifiez la région V3 - V4 du gène de l’ARNr 16S avec des amorces universelles, purifiez les produits de PCR, quantifiez-les et séquencez-les sur la plateforme Illumina. Effectuez l’analyse des données à l’aide du pipeline QIIME235.
- Classifiez les variantes de séquences d’amplicon (ASV) à l’aide de DADA2 et comparez-les à la base de données Silva à l’aide de l’algorithme VSEARCH. Utilisez l’analyse de la diversité α pour évaluer la richesse et la diversité des espèces au sein des échantillons, principalement à l’aide d’indices tels que l’indice de Shannon et l’indice Chao1. L’analyse β-diversité compare la composition de la communauté microbienne entre les échantillons et est généralement visualisée par l’analyse des coordonnées principales (PCoA). Utilisez des méthodes d’analyse différentielle, telles que l’analyse linéaire discriminante de taille d’effet (LEfSe) et l’analyse de parcelles volcaniques, pour identifier les taxons microbiens présentant des conditions significativement différentes ou entre les groupes.
Résultats
Les concentrations de VE ont été déterminées en différentes fractions (figure 2A). Les résultats expérimentaux ont montré que la concentration des VE présentait un modèle de distribution normal typique dans une série de solutions de gradient de densité (figure 2B). Plus précisément, dans la fraction 9, la concentration de VE a atteint son point culminant (3,85 x 109), ce qui suggère que la principale fraction de distribution des VE pourrait être 936.
Pour déterminer la teneur en protéines, des kits d’ACB ont été utilisés pour évaluer la teneur en protéines de différentes fractions (figure 2C). Dans cette méthode, la teneur en protéines de la fraction 9 était de 0,417 μg/μL, ce qui démontre une fois de plus la distribution des VE. Étant donné que le LPS est un composant unique des bactéries à Gram négatif37,38, des kits de détection d’endotoxines ont également été utilisés pour déterminer l’expression du LPS (figure 2D) afin d’évaluer la distribution des VE dans différentes fractions. Les résultats expérimentaux ont montré que dans les fractions 9 et 10, l’expression du LPS était significativement plus élevée que celle des autres fractions (absorbance pour la fraction 9 = 0,8086, pour la fraction 10 = 0,8515), et sa distribution présentait une distribution normale, ce qui prouvait que les EV sont principalement distribués dans la fraction 9. Des quantités relativement plus importantes de protéines dans la fraction 9 ont également été observées dans l’électrophorèse sur gel SDS-PAGE de VE isolés de différentes fractions (figure 2E).
Cette étude a examiné les VE à l’aide de la microscopie électronique à transmission (TEM ; Figure 2F). Grâce à l’imagerie à haute résolution par TEM, il a été possible de visualiser clairement la structure morphologique des VE, avec des structures circulaires semblables à des membranes, ce qui est crucial pour comprendre la biologie des VE.
Les changements de pression artérielle ont été déterminés dans les groupes NSD et HSD après 2 mois d’élevage dans les groupes NSD et HSD des rats. Nous pouvons voir que la TAS (figure 3A), la TAD (figure 3B) et la MBP (figure 3C) ont été significativement augmentées dans le groupe HSD, ce qui indique que le modèle d’hypertension de cette étude a été construit avec succès.
Dans cette étude, la concentration de VE a été détectée dans différents groupes (figure 3D), et les résultats ont montré que la concentration de VE dans le groupe HSD était significativement plus élevée que celle des rats dans le groupe NSD. Cela suggère que la HSD affecte le niveau de VE, potentiellement en raison de changements dans la composition du microbiote intestinal d’où proviennent les VE.
La taille des particules des VE a ensuite été mesurée dans cette étude. Les résultats de mesure indiquent que la taille des particules des VE est principalement d’environ 60 nm (Figure 3E), et que la taille des particules du groupe HSD est légèrement inférieure à celle du groupe NSD. Les données mesurées fournissent des informations importantes pour évaluer la distribution de taille et l’homogénéité des VE, facilitant ainsi la conception expérimentale ultérieure et le développement de l’application.
Ensuite, dans cette étude, la quantité d’expression LPS des VE dans le groupe HSD et le groupe NSD a été examinée (Figure 3F). Les résultats ont montré que l’expression du LPS par les VE dans le groupe HSD était également significativement plus élevée que celle dans le groupe NSD. Cela peut être dû à l’augmentation des bactéries à Gram négatif dans le microbiote intestinal dérivé de l’exovésicule ou à la concentration plus élevée de VE dans le groupe HSD que dans le groupe NSD.
Pour étayer davantage les différences entre les VE dans les groupes HSD et NSD, cette étude a examiné le microbiote intestinal dérivé et a effectué un séquençage de l’ARNr 16S d’échantillons de VE des groupes NSD et HSD afin de clarifier davantage l’effet de la HSD sur les VE dans le microbiote intestinal chez la souris.
L’analyse de la diversité α a montré que la diversité du microbiote intestinal était significativement réduite dans le groupe HSD, les indices de Shannon et d’ECA diminuant (Figure 4A). β -L’analyse de la diversité, basée sur le PCoA (Figure 4B), a permis de distinguer les phénotypes microbiens entre les groupes au niveau de l’ASV. L’intervention à haute teneur en sel a partiellement inversé les changements phénotypiques de la microflore intestinale et a révélé des différences significatives dans la composition du microbiote intestinal dérivé des vésicules externes entre les groupes (p = 0,001).
Après avoir filtré les bactéries de faible abondance et normalisé les données, l’annotation taxonomique a permis d’identifier différentes gammes de communautés microbiennes dans les échantillons. Le microbiote intestinal dérivé d’exovésicules se composait principalement de protéobactéries (93,19 %), de firmicutes (4,57 %) et de bactérodotes (1,19 % ; Figure 4C). Le groupe HSD a significativement augmenté l’abondance des vésicules externes de Firmicutes (Figure 4D). Au niveau du genre, les résultats ont montré que des genres tels que Nevskia et Acinetobacter étaient plus abondants dans le groupe NSD, tandis que Delftia, Burkholderia_Ca et Clostridium_sen étaient plus abondants dans le groupe HSD (Figure 4E). De plus, cette étude a également utilisé des cartes thermiques pour montrer les différences dans les VE du microbiote intestinal entre le groupe HSD et le groupe NSD (Figure 4F). Après l’intervention à forte teneur en sel, l’abondance de certaines bactéries, Delftia et Burkholderia_Ca, a considérablement augmenté, et Nevskia et Acinetobacter a diminué de manière significative (figure 4G). En conclusion, l’intervention à haute teneur en sel a considérablement modifié la différence dans la production de VE dérivées de la microflore intestinale, principalement parce qu’elle a modifié la distribution de leurs bactéries parentales, et les principales caractéristiques étaient une diminution de la diversité, des modifications de la structure d’équilibre et des modifications de l’abondance des différentes bactéries.
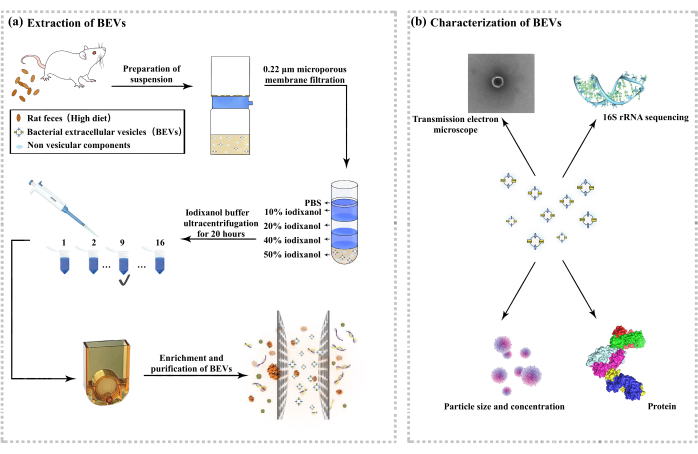
Figure 1 : Extraction et caractérisation des vésicules externes dérivées du microbien intestinal. (A) Organigramme de l’extraction des vésicules externes. (B) Caractérisation des vésicules externes. Veuillez cliquer ici pour voir une version agrandie de cette figure.
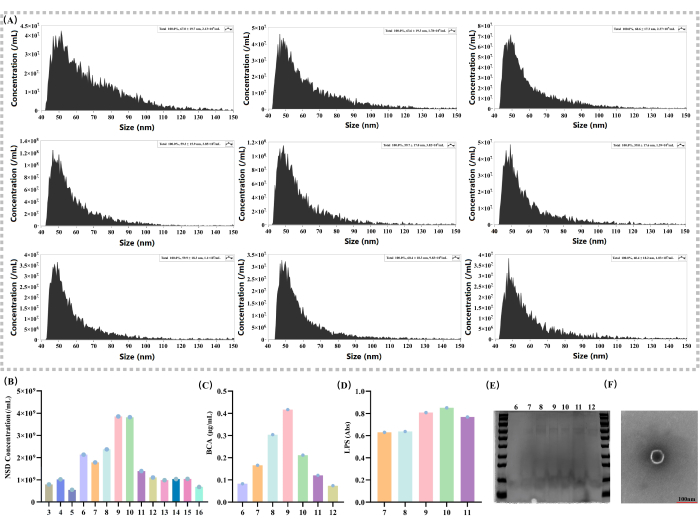
Figure 2 : Caractérisation de base des VE et analyse du gradient de densité local. (A) Mesures de la concentration extravésiculaire et de la taille des particules avec différents gradients de densité (nm ; particules/mL). (B) Comparaison des concentrations des vésicules externes en solution par gradient de densité 3 - 16 (particules/mL). (C) Détermination de la teneur en protéines de la solution sur le gradient de densité 6-12 par le kit BCA (μg/mL). (D) Mesure de l’expression de la LPS en solution par gradient de densité 7-11 avec un kit de détection d’endotoxines (Abs). (E) Les solutions avec un gradient de densité de 6 à 12 ont été soumises à une électrophorèse sur gel SDS-PAGE et colorées au bleu brillant de Coomassie. F) TEM : La barre d’échelle est de 100 nm ; Des vésicules individuelles sont montrées dans l’image. Veuillez cliquer ici pour voir une version agrandie de cette figure.
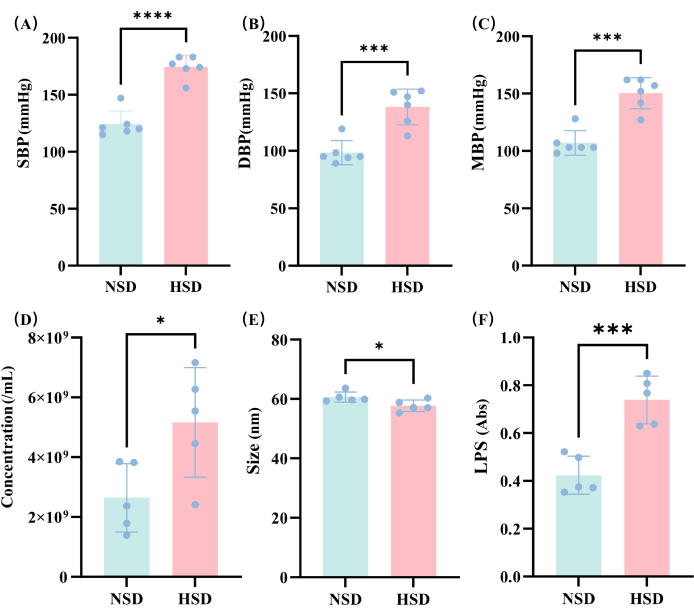
Figure 3 : Analyse de l’évolution de la pression artérielle et des VE sous différents régimes. a) SBP ; b) Le DBP ; (C) MBP ; (D) Comparaison des concentrations de vésicules externes dans les groupes HSD et NSD ; (E) Comparaison de la taille des vésicules externes dans les groupes HSD et NSD. (F) LPS dans les échantillons NSD et HSD déterminés par spectrophotométrie. *p < 0,05, ** p < 0,01, *** p < 0,001, et NS signifie aucune signification, test t. Veuillez cliquer ici pour voir une version agrandie de cette figure.

Figure 4 : Analyse du test de l’ARNr 16s dans les différents régimes. (A) L’analyse de la diversité α ; (B) Analyse des coordonnées principales ; (C) l’abondance du microbiote intestinal au niveau des embranchements ; (D) Changements dans les bactéries au niveau de l’embranchement ; (E) Résultat LEfSe ; (F) l’analyse de la carte thermique des grappes basée sur les genres bactériens différentiels ; (G) Changements dans les bactéries au niveau du genre. *p < 0,05, ** p < 0,01, *** p < 0,001, et NS signifie aucune signification, test t. Veuillez cliquer ici pour voir une version agrandie de cette figure.
Discussion
Dans cette étude, nous nous sommes concentrés sur les VE du microbiote intestinal chez des rats sensibles au sel sur un HSD et avons réalisé une série de réalisations clés. Tout d’abord, une méthode d’extraction efficace basée sur la centrifugation à gradient de densité a été mise au point avec succès pour isoler les VE du microbiote intestinal de rat sensible au sel sur HSD, et la plupart des composants non VE ont été isolés grâce à un processus méticuleux et standardisé de manipulation expérimentale sur des animaux et de traitement des échantillons. La méthode d’extraction des VE de centrifugation à gradient de densité est très efficace et reproductible, meilleure que la méthode d’ultracentrifugation conventionnelle, et peut mieux conserver l’intégrité et la fonctionnalité des VE, garantissant la qualité de l’échantillon et la faisabilité de l’étude.
Deuxièmement, les VE ont été identifiés de manière exhaustive grâce à diverses technologies dynamiques : la détection de la taille et de la concentration des particules indique que les VE ont une distribution de taille spécifique ; Les mesures de LPS et de BCA quantifient la teneur en protéines et l’expression des VE par le LPS ; La MET montre clairement la structure morphologique des VE ; et les caractéristiques protéiques définissent le spectre protéique. Ces résultats d’identification révèlent de manière exhaustive les propriétés physiques et biochimiques des VE, fournissant des données fiables à l’appui d’études ultérieures.
De plus, la technologie de séquençage du gène de l’ARNr 16S utilisant une analyse approfondie des différences entre l’origine et la composition des VE normaux et HSD, ainsi que l’analyse de la diversité α et l’analyse de la diversité β ont montré qu’une consommation élevée de sel affecte de manière significative les caractéristiques génétiques des VE microbiennes intestinales, y compris la structure de la communauté microbienne, la richesse des espèces et la diversité, afin de résoudre de manière plus complète les effets d’un régime riche en sel sur les VE du microbiote intestinal. Fournit plusieurs niveaux de preuves pour les études mécanistes.
Bien que cette méthode présente certains avantages par rapport à la méthode conventionnelle de centrifugation à survitesse en termes de récupération et d’intégrité des exosomes, elle présente encore certaines limites, telles qu’une forte demande d’échantillons et la difficulté de distinguer l’hôte de la source de microflore. Cependant, par rapport aux méthodes existantes, la centrifugation par gradient de densité présente des avantages uniques dans le maintien de l’intégrité fonctionnelle des exosomes, ce qui fournit un soutien technique fiable pour une enquête plus approfondie sur le mécanisme par lequel un régime riche en sel affecte la pression artérielle de l’hôte par le biais de la flore intestinale. De plus, pour la stratification de gradient peu claire rencontrée au cours de l’expérience, nous avons également amélioré en allongeant le temps de centrifugation ou en remplaçant le rotor plus performant.
À l’avenir, nous pourrons explorer davantage le mécanisme des VE du microbiote intestinal dans l’hypertension induite par un régime riche en sel, en particulier son interaction avec le système immunitaire, comme les lymphocytes T pro-inflammatoires39,40 ; ou utiliser des produits d’origine végétale pour intervenir dans les VE du microbiote intestinal41,42, intervenir davantage dans les maladies et explorer leur rôle dans la régulation du métabolisme de l’hôte et de la réponse immunitaire.
En conclusion, cette étude fournit non seulement un outil important pour explorer le mécanisme de l’hypertension du microbiote intestinal induite par le sel, à travers le modèle de rat sensible au sel, approfondir la compréhension de la relation entre HSD, le microbiote intestinal et les VE, et a également ouvert une nouvelle voie pour la prévention et le traitement des maladies cardiovasculaires telles que l’hypertension, fournit une perspective unique pour l’étude de l’interaction alimentation-microbe-hôte.
Déclarations de divulgation
Les auteurs déclarent qu’ils n’ont pas d’intérêts financiers concurrents connus ou de relations personnelles qui auraient pu sembler influencer les travaux rapportés dans cet article.
Remerciements
Ce travail a été soutenu par la Fondation nationale des sciences naturelles de Chine (82205240), la Fondation des sciences naturelles de la province du Sichuan (2025ZNSFSC1836) et le Projet clinique de base de l’Hôpital orthopédique provincial du Sichuan (PY202414).
matériels
| Name | Company | Catalog Number | Comments |
| Essential Supplies | |||
| Centrifugal Filter (10nkDa 2 mL) | Millipore | UFC903096 | |
| Centrifuge Tude(50 mL) | BKMAN | 20220404 | |
| Centrifuge Tudes | BECKMAN COULTER | Z30815SCA | |
| Vacuum Filtration System | Biosharp | 24902581 | |
| Reagents | |||
| Chromogenic LAL Endotoxin Assay Kit | Beyotime | 022124240705 | |
| Coomassie Blue Fast Staining Solution | Beyotime | Z972241010 | |
| EDTA | Damas-beta | P3117308 | |
| Enhanced BCA Protein Assay Kit | Beyotime | A006241112 | |
| Ethanol | KESH | ||
| HCl | |||
| OptiPrep (60% wt/vol, iodixanol) | Serumwerk | 00124 | |
| PBS | Labshark | 130114005 | |
| phosphotungstic acid | RUIXIN | ||
| Sucrose | Damas-beta | P1917057 | |
| Tris (VWR) | Damas-beta | P3061764 | |
| Trypan blue staining solution (0.4%) | Beyotime | BD07242904 | |
| Equipment | |||
| Absorbance Microplate Reader | SpectraMax | ABP01690 | |
| Biomicroscope | Motic | BA210Digital | |
| Desk centrifuge | Cence | CHT210R | |
| Desktop high-speed micro centrifuge | DLAB | D3024 | |
| Fixed Angle Aluminum Rotor + 500 mL Centrifugal Cup | Cence | ||
| High precision electronic balance | SKR | BN-200 | |
| Laminar flow cabinet | Nantong Hunan Scientific Instrument Co., Ltd. | SW-CJ-2FDS | |
| SW 32.1 Ti Swing bucket turn+ SW 32.1 Ti Rotor bucket | BECKMAN COULTER | ||
| Transmission electron microscope | JEOL | JEM-1400FLASH | |
| Tube rotator | |||
| Ultracentrifuge | BECKMAN COULTER | Optima XE-100 | |
| Ultra-pure water system | ULPHW | UPR-II-15TNZ | |
| Water-Cieculation Multifunction Vacuum Pump | Qiang Qiang | SHZ-D(III) |
Références
- Thursby, E., Juge, N. Introduction to the human gut microbiota. Biochem J. 474 (11), 1823-1836 (2017).
- Aschtgen, M. S., et al. Rotation of Vibrio fischeri Flagella Produces Outer Membrane Vesicles That Induce Host Development. J Bacteriol. 198 (16), 2156-2165 (2016).
- Gurung, S., Perocheau, D., Touramanidou, L., Baruteau, J. The exosome journey: from biogenesis to uptake and intracellular signalling. Cell Comm Signal. 19 (1), 47(2021).
- Avila-Calderón, E. D., et al. Outer Membrane Vesicles of Gram-Negative Bacteria: An Outlook on Biogenesis. Front Microbiol. 12, 557902(2021).
- Melo-Marques, I., Cardoso, S. M., Empadinhas, N. Bacterial extracellular vesicles at the interface of gut microbiota and immunity. Gut Microbes. 16 (1), 2396494(2024).
- Kim, N. Y., et al. Effect of gut microbiota-derived metabolites and extracellular vesicles on neurodegenerative disease in a gut-brain axis chip. Nano Converg. 11 (1), 7(2024).
- Ahmadi Badi, S., et al. Microbiota-Derived Extracellular Vesicles as New Systemic Regulators. Front Microbiol. 8, 1610(2017).
- Barathan, M., Ng, S. L., Lokanathan, Y., Ng, M. H., Law, J. X. The Profound Influence of Gut Microbiome and Extracellular Vesicles on Animal Health and Disease. Int J Mol Sci. 25 (7), 4024(2024).
- Maukonen, J., Saarela, M. Human gut microbiota: does diet matter. Proc Nutri Soc. 74 (1), 23-36 (2015).
- Bier, A., et al. A High Salt Diet Modulates the Gut Microbiota and Short Chain Fatty Acids Production in a Salt-Sensitive Hypertension Rat Model. Nutrients. 10 (9), 1154(2018).
- Aghamohammad, S., et al. Anti-inflammatory and immunomodulatory effects of Lactobacillus spp. as a preservative and therapeutic agent for IBD control. Immun Inflamm Dis. 10 (6), e635(2022).
- Dong, Z., et al. The Effects of High-Salt Gastric Intake on the Composition of the Intestinal Microbiota in Wistar Rats. Med Sci Monit. 26, e922160(2020).
- Yan, X., et al. Intestinal Flora Modulates Blood Pressure by Regulating the Synthesis of Intestinal-Derived Corticosterone in High Salt-Induced Hypertension. Circ Res. 126 (7), 839-853 (2020).
- Wang, X., Lang, F., Liu, D. High-Salt Diet and Intestinal Microbiota: Influence on Cardiovascular Disease and Inflammatory Bowel Disease. Biology. 13 (9), 674(2024).
- Qi, L., et al. Microbiome-metabolome analysis insight into the effects of high-salt diet on hemorheological functions in SD rats. Front Nutri. 11, 1408778(2024).
- Wu, Q., et al. Insights into the unique roles of extracellular vesicles for gut health modulation: Mechanisms, challenges, and perspectives. Curr Res Microb Sci. 7, 100301(2024).
- Zhang, H., et al. Effects of bacterial extracellular vesicles derived from oral and gastrointestinal pathogens on systemic diseases. Microbiol Res. 285, 127788(2024).
- Wang, H. X., Wang, Y. P. Gut Microbiota-brain Axis. Chinese Med J. 129 (19), 2373-2380 (2016).
- Huang, S., et al. A cross-tissue transcriptome association study identifies key genes in essential hypertension. Front Genet. 14, 1114174(2023).
- Gao, H., et al. Microbial DNA Enrichment Promotes Adrenomedullary Inflammation, Catecholamine Secretion, and Hypertension in Obese Mice. J Am Heart Assoc. 11 (4), e024561(2022).
- Chen, P. Gut Microbiota and Pathogenesis of Organ Injury. , Springer. Singapore. (2020).
- Xie, N., et al. hPSCs-derived brain organoids for disease modeling, toxicity testing and drug evaluation. Exp Neurol. 385, 115110(2024).
- Liu, N., et al. The underlying mechanisms of DNA methylation in high salt memory in hypertensive vascular disease. Sci Rep. 14 (1), 925(2024).
- Li, M., et al. Wheat β-glucan reduces obesity and hyperlipidemia in mice with high-fat and high-salt diet by regulating intestinal flora. Int J Biol Macromol. 288, 138754(2024).
- Kerem, G., et al. Small intestinal microbiota composition altered in obesity-T2DM mice with high salt fed. Sci Rep. 13 (1), 8256(2023).
- Díez-Sainz, E., Milagro, F. I., Riezu-Boj, J. I., Lorente-Cebrián, S. Effects of gut microbiota-derived extracellular vesicles on obesity and diabetes and their potential modulation through diet. J Physiol Biochem. 78 (2), 485-499 (2022).
- Wu, S., et al. Liuzijue training improves hypertension and modulates gut microbiota profile. Front Cardiovasc Med. 10, 1075084(2023).
- Qi, L. M., et al. Salvia miltiorrhiza bunge extract improves the Th17/Treg imbalance and modulates gut microbiota of hypertensive rats induced by high-salt diet. J Funct Foods. 117, 106211(2024).
- Wilck, N., et al. Salt-responsive gut commensal modulates T(H)17 axis and disease. Nature. 551 (7682), 585-589 (2017).
- Jiang, X., et al. Intestinal Gastrin/CCKBR (Cholecystokinin B Receptor) Ameliorates Salt-Sensitive Hypertension by Inhibiting Intestinal Na(+)/H(+) Exchanger 3 Activity Through a PKC (Protein Kinase C)-Mediated NHERF1 and NHERF2 Pathway. Hypertension. 79 (8), 1668-1679 (2022).
- Zheng, T., et al. Hypertension of liver-yang hyperactivity syndrome induced by a high salt diet by altering components of the gut microbiota associated with the glutamate/GABA-glutamine cycle. Front Nutr. 9, 964273(2022).
- Mulligan, S. K., et al. Multiplexed TEM Specimen Preparation and Analysis of Plasmonic Nanoparticles. Microsc Microanal. 21 (4), 1017-1025 (2015).
- Olson, B. Assays for Determination of Protein Concentration. Curr Protoc Pharmacol. 73, A.3a.1-a.3a.32 (2016).
- Matsumoto, H., Haniu, H., Komori, N. Determination of Protein Molecular Weights on SDS-PAGE. Methods Mol Biol. 1855, 101-105 (2019).
- Sanschagrin, S., Yergeau, E. Next-generation Sequencing of 16S Ribosomal RNA Gene Amplicons. J Vis Exp. (90), e51709(2014).
- Tulkens, J., De Wever, O., Hendrix, A. Analyzing bacterial extracellular vesicles in human body fluids by orthogonal biophysical separation and biochemical characterization. Nat Protoc. 15 (1), 40-67 (2020).
- Ruhal, R., Kataria, R. Biofilm patterns in gram-positive and gram-negative bacteria. Microbiol Res. 251, 126829(2021).
- Maldonado, R. F., Sá-Correia, I., Valvano, M. A. Lipopolysaccharide modification in Gram-negative bacteria during chronic infection. FEMS Microbio Rev. 40 (4), 480-493 (2016).
- Honda, K., Littman, D. R. The microbiota in adaptive immune homeostasis and disease. Nature. 535 (7610), 75-84 (2016).
- Guzik, T. J., et al. Role of the T cell in the genesis of angiotensin II induced hypertension and vascular dysfunction. J Exp Med. 204 (10), 2449-2460 (2007).
- Huang, J., et al. Extracellular vesicles as a novel mediator of interkingdom communication. Cytokine Growth Factor Rev. 73, 173-184 (2023).
- Li, W., et al. Alleviation of colitis by honeysuckle MIR2911 via direct regulation of gut microbiota. J Control Release. 376, 123-137 (2024).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationThis article has been published
Video Coming Soon