Method Article
Стратегия двойной CRISPR-интерференции для нацеливания на синтетические летальные взаимодействия между некодирующими РНК в раковых клетках
В этой статье
Резюме
В этом исследовании представлена двойная система CRISPRi, нацеленная на длинные некодирующие РНК в клетках меланомы. Он позволяет проводить комбинаторный нокдаун генов и синтетический летальный скрининг, выявляя специфические для рака взаимодействия lncRNA для потенциальных терапевтических стратегий.
Аннотация
Длинные некодирующие РНК (днРНК) представляют собой обширный и функционально разнообразный класс молекул РНК, более 100 000 из которых предсказаны в геноме человека. Хотя некодирующие белки менее консервативны у разных видов по сравнению с генами, кодирующими белки, они играют важную роль в регуляции генов, взаимодействии хроматина и прогрессировании рака. Их вовлеченность в рак делает их перспективными терапевтическими мишенями. CRISPR интерференция (CRISPRi), использующая каталитически неактивный Cas9, слитый с транскрипционным репрессором, таким как KRAB-MeCP2, предлагает точный метод нацеливания на ядерные некодирующие РНК и оценки их функций. В этом исследовании представлена двойная система CRISPRi с использованием ортогональных технологий CRISPRi из Staphylococcus aureus и Streptococcus pyogenes на основе dCas9-KRAB-MeCP2, оптимизированная для комбинаторного нацеливания на некодирующие РНК в клетках меланомы человека. Протокол облегчает комбинаторный нокдаун генов или синтетический летальный скрининг пар некодирующих РНК, предоставляя новый инструмент для исследования рака. Исследуя синтетическую летальность между некодирующими РНК, этот подход может помочь выявить взаимодействия некодирующих РНК, критически важные для выживания раковых клеток, предлагая новые терапевтические стратегии. Демонстрируется функциональность двойной системы, подчеркивая ее потенциал в выявлении критических взаимодействий lncRNA, специфичных для рака.
Введение
Хотя менее 3% генома человека кодирует белки, примерно 80% генома транскрибируется 1,2. Среди некодирующих транскрипционных единиц десятки тысяч классифицируются как длинные некодирующие РНК (днРНК), превышающие 200 нуклеотидов, а общее количество днРНК человека, по оценкам, превышает 100 000 3,4. В отличие от кодирующих генов, некодирующие РНК менее консервативны у разных видов. Учитывая, что люди на 99% разделяют свой геном с приматами, такими как шимпанзе, предполагается, что некодирующие РНК оказывают гораздо большее влияние на фенотипическуюэволюцию. Эти результаты указывают на важные клеточные функции некодирующих РНК. Несмотря на то, что регуляция некодирующих РНК и их взаимодействие с РНК-связывающими белками и другими РНК остаются не до конца изученными, а многие некодирующие РНК еще не полностью аннотированы, ясно, что некодирующие РНК демонстрируют специфические для клеток и тканей паттерны экспрессии в здоровом и болезненном состоянии, таких как рак 7,8,9,10. Они участвуют в различных функциях, включая регуляцию транскрипции генов, участие во взаимодействиях хроматина11, процессинг РНК12, стабилизацию РНК13 и регуляцию трансляции14.
При раке очень разнообразные типоспецифичные для типов клеток некодифичные некодирующие РНК влияют на развитие опухоли и метастазирование, регулируя экспрессию генов, что подчеркивает их потенциал в качестве ценных терапевтических мишеней15. Помимо обнаружения некодирующих РНК в качестве биомаркеров в образцах опухолей16, нацеливание на опухолеспецифичные некодиальные РНК с целью нарушения их последующих функций имеет значительный потенциал как в клиническом применении, так и в фундаментальных исследованиях. Основанные на РНК подходы к выяснению роли некодирующих РНК включают антисмысловые олигонуклеотиды (АСО), короткие шпильчатые РНК и малые интерферирующие РНК (миРНК)17,18. В то время как миРНК обычно используется для скрининга подавления экспрессии генов, нокдаун на основе миРНК ограничен цитоплазмой19. Тем не менее, некодирующая РНК часто действует в ядре.
В качестве альтернативы, для ингибирования некодиальных РНК враковых опухолях человека можно использовать кластеризованные регулярно чередующиеся короткие палиндромные повторы (CRISPRi). Кроме того, полногеномные CRISPRi-скрининги могут быть легко запрограммированы и нацелены на широкий спектр кодирующих и некодирующих генов для изучения их функционального влияния21,22. В CRISPRi каталитически дефицитный Cas9 (dCas9) сливается с доменом транскрипционного репрессора, таким как домен23 Krüppel-ассоциированного бокса (KRAB). Репрессия генов dCas9-KRAB направляется направляющей РНК (гРНК) в интересующую область. CRISPRi контролирует гены на уровне ДНК, что приводит к более высокой эффективности и желаемым фенотипам потери функции в отличие от РНК-интерференции, которая активна на посттранскрипционном уровне24. В ответ на ограниченную эффективность KRAB в подавлении мишени, в качестве болееэффективной стратегии подавления было введено слияние KRAB и MeCP2 с dCas9.
Несмотря на то, что сайленсинг одного гена может повлиять на жизнеспособность рака, синтетические двойные или множественные летальные взаимодействия могут спасти раковые клеткиот гибели клеток. Синтетическая летальность включает в себя два или более гена, каждый из которых может компенсировать функцию другого. Для решения проблем, связанных со скринингом нокдауна одного гена, двойные стратегии CRISPR, нацеленные на пары генов, кодирующих летальные белки, предлагают многообещающийподход.
В данной работе мы представляем протокол комбинаторного использования ортогонального нацеливания на основе CRISPRi lncRNA или других некодирующих пар РНК с использованием Staphylococcus aureus и Streptococcus pyogenes dCas9, слитых с KRAB-MeCP2 в клетках меланомы человека (см. рис. 1). Протокол может быть использован для комбинаторного классического нокдауна CRISPR или в качестве скрининга синтетических летальных пар при раке на основе CRISPRi. Использование двух различных видов dCas9, SpCas9 и SaCas9, в подходе CRISPRi позволяет независимо нацеливаться на различные геномные локусы с минимальной перекрестной реактивностью, повышая специфичность и гибкость при обеспечении высокой селективности по отношению к мишени. Используются различные протоспейсерные смежные мотивы (PAM): NGG для SpCas9 и NNGRRT для SaCas9. Двойная система dCas9 решает такие проблемы, как ограниченная совместимость sgРНК, обеспечивая точную одновременную модуляцию и снижая конкуренцию комплекса sgRNA-RNP при использовании одного типа dCas9. Эта инновация повышает надежность и универсальность скрининга библиотек двух sgRNA. В заключение мы предлагаем полнофункциональную двойную систему CRISPRi в клетках меланомы в качестве модели рака.
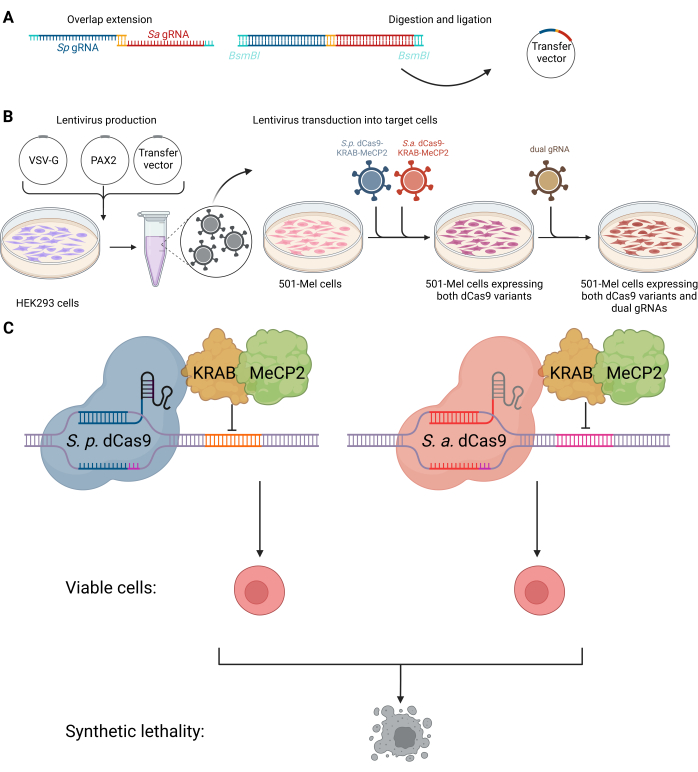
Рисунок 1: Схема двойной системы CRISPRi для нацеливания на синтетические летальные взаимодействия некодирующих РНК. (А) Процедура клонирования вектора двойной гРНК. (B) Клетки HEK293 трансфицировали конвертирующей плазмидой VSV-G, упаковывающей плазмидой PAX2 и вектором переноса для получения лентивирусов для последующей трансдукции в клетки 501-mel. Лентивирусы, содержащие генетическую информацию о SpdCas9-KRAB-MeCP2 (синий) и SadCas9-KRAB-MeCP2 (красный), были интегрированы в клетки 501-mel одновременно. После выбора антибиотика была проведена вторая лентивирусная трансдукция для интеграции желаемых двойных гРНК (оливкового цвета). (C) 501-mel клетки, экспрессирующие варианты dCas9, взаимодействуют со своими соответствующими гРНК, подавляя экспрессию генов-мишеней, что приводит к гибели раковых клеток. Создано с помощью BioRender.com. Пожалуйста, нажмите здесь, чтобы просмотреть увеличенную версию этой цифры.
протокол
Клеточная линия меланомы человека 501-mel (RRID: CVCL_4633) была любезно предоставлена лабораторией Aifantis (Нью-Йоркский университет). Клеточная линейка Lenti-X 293T HEK была приобретена у компании Takara Bio. Эти клеточные линии культивировали в модифицированной среде Дульбекко с высоким содержанием глюкозы (DMEM) с добавлением 10% фетальной бычьей сыворотки (FBS) при 37 °C в атмосфере 5%CO2 в стерильных условиях. Клетки пропускали до тех пор, пока они не достигали 80%-90% конфлюенции. Клеточные линии регулярно проверялись на контаминацию микоплазмой.
1. Конструирование гРНК с помощью онлайн-платформы для проектирования гРНК
- Разработайте одну гРНК, специфичную для SpCas9, и одну гРНК, специфичную для SaCas9, для каждой целевой lncRNA, чтобы оценить, какой вариант дает лучшие результаты нокдауна.
- Нацельтесь на нужные lncРНК, в данном случае RP11-120D5.1 (RP11) и XLOC_030781 (XLOC), используя наиболее эффективные гРНК для каждого S. pyogenes (gRNA-sp) Cas9 и S. aureus (gRNA-sa) Cas9.
ПРИМЕЧАНИЕ: РНК S. pyogenes могут быть выделены из Petroulia et al.28. - Следуйте рекомендациям, специфичным для сайта, чтобы отдавать приоритет самой высокой целевой активности, выражающейся в высоких оценках по цели, при этом сводя к минимуму нецелевые совпадения, как предлагают инструменты проектирования гРНК.
- Выберите геном GRCh38 (hg, Homo sapiens). Выберите сайт PAM для конкретных SpCas9 (NGG) и SaCas9 (NNGRRT).
- Используйте метод нецелевого скоринга от Hsu et al.29. Расположите гРНК в диапазоне от -150.о. до +50.о. вокруг сайта начала транскрипции для достижения оптимальной производительности.
- Выбирайте гРНК с наивысшим показателем «по цели» и «по цели» (см. дополнительную таблицу 1).
- Объедините нецелевую или скремблированную контрольную гРНК, в данном случае против Rosa26, с каждой целевой гРНК, чтобы создать двойной вектор гРНК, нацеленный на одну некодирующую РНК. Комбинация гРНК-мишеней в одной плазмиде позволяет сайт-специфичную модификацию обоих сайтов-мишеней.
ПРИМЕЧАНИЕ: Двойной вектор гРНК, содержащий контрольную гРНК и целевую гРНК, обеспечивает нокдаун одной целевой некодирующей РНК, в то время как векторы двойной гРНК, содержащие гРНК, нацеленные на сайты, позволяют одновременно нокдаунить целевые некодирующие РНК.
2. Клонирование гРНК
- Проведите ПЦР с расширением перекрытия с использованием двух фрагментов ДНК, содержащих последовательность гРНК. Используйте прямой праймер U6 и обратный праймер H1 (см. дополнительную таблицу 2) для амплификации, как описано в Najm et al.27.
- Организуйте ПЦР-реакцию объемом 50 мкл с 25 мкл 2x высокоточной мастер-смеси для ПЦР с HF-буфером и 1 мкл каждого фрагмента ДНК, содержащего либо гРНК S. pyogenes, либо S. aureus . Реакцию протекают в следующих циклических условиях: начальная денатурация при 98 °C в течение 30 с, затем 15 циклов денатурации при 98 °C в течение 5 с, отжиг при 55 °C в течение 10 с и удлинение при 72 °C в течение 15 с.
- Добавьте 2,5 мкл прямого праймера U6 и 2,5 мкл обратного праймера H1 после завершения начальных 15 циклов и подвергните реакцию еще 20 циклам в тех же условиях, с окончательным продлением в течение 5 мин при 72 °C.
- Очистите продукты ПЦР с помощью набора для очистки ПЦР в соответствии с инструкциями производителя.
- Расщепляют 1 г модифицированной плазмиды с использованием 1 мкл фермента рестрикции BsmB1 и 1 мкл 10-кратного буфера в течение 30 мин при 37 °C в реакции 10 мкл.
- Клонируйте продукты ПЦР в расщепленную плазмиду с двойной гРНК-Zeo-GFP (см. Дополнительный рисунок 1 и Дополнительный файл 1) с использованием протокола реакции сборки ДНК HiFi, как описано в другихработах 30.
- Преобразуйте химически компетентные клетки E. coli, смешав 50 μл бактериальной культуры с 4 μл сборочного продукта в соответствии с инструкциями производителя.
3. Производство лентивирусов
ВНИМАНИЕ: Обрабатывайте активные лентивирусы на всех этапах, соблюдая соответствующие правила безопасности. Всегда выполняйте работу с вирусами в защитном шкафу класса 2, чтобы предотвратить опасность неизбежного образования аэрозоля. Все работы вне защитного шкафа, такие как центрифугирование, проводите в аэрозоленепроницаемых контейнерах и роторах, одобренных для использования с микроорганизмами группы риска 2. Постоянно носите соответствующую защитную одежду, включая лабораторный халат, одноразовые перчатки и защитные очки, в рабочей зоне. Рабочие поверхности должны быть обработаны дезинфицирующим раствором, а другие отходы также должны быть обработаны дезинфицирующим раствором, что позволит им оставаться в герметичном шкафу под ультрафиолетовым светом не менее 240 минут. Использованные серологические пипетки и наконечники для пипеток необходимо промыть в дезинфицирующем растворе перед утилизацией в автоклавируемых бутылках. После этого отходы следует утилизировать в контейнере для отходов S2. Отходы S2 будут инактивированы путем автоклавирования. Трансдуцированные клеточные линии можно обрабатывать в условиях S1 не ранее, чем через 2 суток после трансдукции и только после не менее двух полных смен питательной среды на безвирусную среду.
- Подсчитайте клетки Lenti-X 293T HEK, смешав 10 мкл клеток с 10 мкл раствора трипанового синего в гемоцитометре. Пластина 4 x 106 элементов Lenti-X 293T HEK на 10 см пластины для достижения ~80% слияния на следующий день.
- Начиная с этого этапа, работайте в лаборатории класса 2. Смешайте 500 мкл подходящей трансфекционной среды с вектором переноса SpdCas9-KRAB-MeCP2 массой 11,25 мкг (см. Дополнительный файл 2), огибающей плазмидой VSV-G массой 5,5 мкг и вектором pPAX2 массой 16,5 мкг. Выдерживать 5 минут при комнатной температуре. Для второго варианта dCas9 используйте вектор переноса SadCas9-KRAB-MeCP2 (см. Дополнительный файл 3).
- В отдельной пробирке смешайте 500 мкл среды с восстановленной сывороткой с 36 мкл реагента PEI (запас: 1 мг/мл). Выдерживать 5 минут при комнатной температуре.
- Соедините смеси и выдержите 15 минут при комнатной температуре. Осторожно добавьте смесь по каплям в клеточную среду (6 мл) и инкубируйте в течение ночи при 37 °C и 5%CO2. Убедитесь, что клетки остаются прикрепленными, чтобы избежать снижения выхода лентивирусов.
- Слейте среду в автоклавируемую бутылку для отходов с помощью серологической пипетки через 12-15 часов. С этого момента расходные материалы, в том числе серологические пипетки и наконечники для пипеток, промывайте дезинфицирующим раствором перед утилизацией в отходах автоклава. Добавьте 5 мл свежей среды DMEM в трансфицированные клетки с помощью новой пипетки.
- Сбор лентивирусов путем переноса надосадочной жидкости с помощью серологической пипетки в каноническую пробирку объемом 50 мл через 48 ч после трансфекции. Храните каноническую трубку в холодильнике. Добавьте в клетки 5 мл свежей среды и инкубируйте клетки в течение еще 24 часов.
- Повторите сбор лентивирусов, перенеся надосадочную жидкость в ту же каноническую пробирку объемом 50 мл, в которой был собран первый урожай. Снова добавьте 5 мл свежей среды, инкубируйте в течение 24 ч, а затем перенесите надосадочную жидкость в каноническую пробирку объемом 50 мл. Храните комбинированные надосадочные жидкости первого, второго и третьего урожаев в канонической пробирке объемом 50 мл при температуре 4 °C до фильтрации и центрифугирования.
- Комбинированный надосадочный жидкость центрифугировать в течение 5 мин при 500 x g и 4 °C в аэрозоленепроницаемых контейнерах и роторах, разрешенных для использования с организмами группы риска 2, для гранулирования отделившихся клеток и мусора. Профильтруйте надосадочную жидкость через стерильные шприцевые фильтры для культивирования клеток 0,2 мкм.
- Сконцентрируйте отфильтрованную надосадочную жидкость при концентрации 1000 x g и 4 °C с использованием центробежной фильтрующей установки с соответствующей молекулярной массой от 100 кДа до приблизительно 500 μL. Сохраняйте аликвоты вируса в объеме 50-100 μL при -80 °C до дальнейшего использования.
4. Трансдукция лентивируса и отбор стабильных трансфицированных клеток
ВНИМАНИЕ: Всегда выполняйте работу с вирусами в защитном шкафу класса 2.
- Планшет 501-mel помещают клетки в 6-луночный планшет в соотношении 2 x 105 клеток/лунку с конечным объемом культивирования 2 мл, за 1 день до трансдукции.
- Замените питательную среду 2 мл предварительно подогретой среды DMEM с добавлением полибрена в конечной концентрации 6 мкг/мл.
- С этого момента работа в лаборатории класса 2 обязательна. Добавьте в клетки одновременно 50 мкл лентивирусов, содержащих SpdCas9-KRAB-MeCP2 и SadCas9-KRAB-MeCP2. Промойте расходные материалы, в том числе серологические пипетки и наконечники для пипеток, дезинфицирующим раствором перед тем, как утилизировать их в отходах автоклава. Осторожно встряхните планшет и инкубируйте при температуре 37 °C в атмосфере 5%CO2 .
- Слейте среду в автоклавируемый флакон с помощью серологической пипетки и добавьте 2 мл свежей среды, содержащей бластицидин (10 г/мл) и пуромицин (2 мкг/мл) через 16 ч после трансдукции.
- Продолжайте отбор, заменив среду свежей средой объемом 2 мл, содержащей бластицидин (10 мкг/мл) и пуромицин (2 мкг/мл) через 2 дня. Обрабатывайте трансдуцированные клетки в условиях S1 через 7 дней после трансдукции.
Примечание: Выбор антибиотика должен проводиться до тех пор, пока все нетрансдуцированные контрольные клетки не погибнут. Альтернативные стратегии выбора могут быть использованы в зависимости от кассеты сопротивления в векторе передачи. При необходимости оптимизируйте концентрацию антибиотиков с помощью кривой убийства. Для получения клеточных популяций, экспрессирующих слияния dCas9 на достаточных уровнях, может потребоваться сложный отбор клонов одиночных клеток с помощью проточной цитометрии или серийного разведения и гальванирования с последующим количественным определением белка.
5. Количественное определение белка и вестерн-блоттинг
- Подсчитывайте стабильно трансфицированные клетки 501-mel, смешивая 10 мкл клеток с 10 мкл раствора Trypan Blue в гемоцитометре. Центрифугируйте 1 x 106 элементов в течение 5 мин при 500 x g и 4 °C. Выбросьте надосадочную жидкость и повторно суспендируйте клеточную гранулу в 1 мл ледяного PBS.
- Повторите центрифугирование и повторно суспендируйте клеточную гранулу в 50 мкл буфера RIPA, содержащего ингибиторы протеазы. Инкубировать на льду в течение 15 минут и удалить клеточный мусор центрифугированием при 13 000 x g в течение 10 минут при 4 °C. Храните надосадочную жидкость при температуре -20 °C или используйте немедленно для количественного определения белка, как описано в других работах31.
- Смешайте 10 г белка с 4 x буфером LDS, 50 mM DTT и залейте до 20 μL дистиллированной водой. Разделите белки на 10% разделительном геле с помощью SDS-PAGE на основе триса/глицина при напряжении 135 В в течение 1 ч, как описано ранее32. Перенесите белки на мембрану PVDF при напряжении 90 В в течение 90 минут с помощью переноса в мокром баке.
- Инкубируйте мембрану в блокирующем буфере (5% обезжиренного молока в TBS-T) в течение 1 ч при 180 об/мин при комнатной температуре.
- Добавьте 1:10 000 моноклонального антитела мыши против GAPDH в блокирующий буфер в качестве внутреннего контроля. Добавьте первичное антитело anti-Cas9 (S. pyogenes) или анти-Cas9 (S. aureus) в соотношении 1:1000 в блокирующий буфер и инкубируйте в течение ночи при 4 °C с легким встряхиванием.
- Промойте мембрану 3 раза с буфером TBST по 10 минут каждая. Инкубировать со вторичными антителами к HRP против мышей (1:10 000) в течение 1 ч при комнатной температуре.
- Промойте 3 раза с TBST буфером по 10 минут каждый. Обнаружение сигналов с помощью ECL Substrate и системы формирования изображений.
6. Анализ жизнеспособности с помощью двойной гнРНК
ВНИМАНИЕ: Всегда выполняйте работу с вирусами в защитном шкафу класса 2.
- Затравка 0,1 x 105 клеток, содержащих стабильные трансфицированные SadCas9-KRAB и SpdCas9-KRAB, через 7 дней после трансдукции в 96-луночном планшете в 100 мкл предварительно подогретой среды DMEM за 1 день до трансдукции.
- В день трансдукции замените питательную среду 100 мкл предварительно подогретой среды DMEM с добавлением полибрена в конечной концентрации 6 мкг/мл. С этого момента работа в лаборатории класса 2 обязательна. Добавьте в клетки соответствующий объем лентивирусов.
Примечание: Объем лентивирусов, содержащих двойной вектор гРНК, должен быть изучен с помощью GFP для построения линейной кривой путем титрования лентивирусов. - Замените среду свежей средой, содержащей 500 мкг/мл цеоцина, 5 мкг/мл бластицидина и 1 мкг/мл пуромицина, через 16 ч после трансдукции.
- Выполните обнаружение люминесценции через 5 дней после трансдукции с помощью анализа жизнеспособности клеток в соответствии с инструкциями производителя.
Результаты
Экспрессирующие кассеты SadCas9-KRAB-MeCP2 и SpdCas9-KRAB-MeCP2 были интегрированы в клетки 501-mel одновременно с помощью трансдукции с равными количествами лентивирусов. Поскольку этот протокол CRISPRi основан на экспрессии вариантов dCas9-KRAB-MeCP2, вестерн-блоттинг идеально подходит для подтверждения их присутствия на белковом уровне. После обогащения положительно трансфицированных клеток были собраны образцы для вестерн-блоттинга, чтобы подтвердить наличие ортологов dCas9-KRAB-MeCP2. После успешной трансдукции ожидается появление одной полосы, соответствующей гибридному белку SpdCas9-KRAB-MeCP2 (202 кДа) и гибридному белку SadCas9-KRAB-MeCP2 (170 кДа), что было подтверждено, как показано на рисунке 2A, с использованием антител, специфичных к соответствующим ортолологам Cas9. Однако, если вестерн-блоттинг невозможен, можно использовать кПЦР для проверки гибридных белков на уровне мРНК.
После получения 501-мельных клеток, стабильно экспрессирующих функциональные репрессоры, была проведена вторая лентивирусная трансдукция с использованием двойного вектора гРНК. Ранее было показано, что RP11 и XLOC активируются в краткосрочных культурах метастатической меланомы, а индивидуальное нацеливание CRISPRi продемонстрировало их влияние на пролиферацию и выживаемость клеток. Таким образом, мы выбрали эту пару некодирующих РНК для оценки в качестве экспериментальной синтетической комбинации летальных некодирующих РНК, предвосхищая кумулятивный, легко наблюдаемый фенотип роста в модифицированных клетках.
Чтобы исследовать эффекты двойного CRISPRi в раковых клетках с использованием анализов жизнеспособности, обе целевые некодирующие РНК должны быть эффективно подавлены. КПЦР является наиболее подходящим методом для подтверждения этой репрессии путем измерения уровня их транскриптов. Мы оценили функциональность разработанного двойного протокола CRISPRi путем определения уровней РНК и изучения жизнеспособности клеток при нацеливании на потенциально летальную пару RP11 и XLOC. Поэтому для снижения целевых уровней РНК были введены различные гРНК, нацеленные на RP11 и XLOC. Репрессия РНК была исследована с использованием гРНК, специфичных для мишеней SadCas9-KRAB-MeCP2 или SpdCas9-KRAB-MeCP2, которые тестировались по отдельности и в комбинации вместе с контрольной гРНК, нацеленной на Rosa26 (см. рис. 2B). Хотя гРНК, нацеленные на XLOC или RP11 по отдельности, эффективно подавляли свои соответствующие мишени, одновременный нокдаун как XLOC, так и RP11 был достигнут только с парой XLOC-sa/RP11-sp.
Поскольку уровни РНК XLOC и RP11 были успешно снижены в клеточной линии 501-mel с выбранной парой гРНК XLOC-sa/RP11-sp, был проведен анализ жизнеспособности двойной репрессии lncРНК (см. рис. 2C). Через 5 дней жизнеспособность клеток составила 81% и 89% от контроля Rosa26 при использовании целевых гРНК по отдельности. Комбинация RP11-sp и XLOC-sa привела к дальнейшему значительному снижению по сравнению с RP11-sp, при этом жизнеспособность клеток составила 59% от контрольной Rosa26.

Рисунок 2: Функциональность системы репрессии двойной lncRNA. (A) Вестерн-блоттинг был проведен для обнаружения вариантов dCas9-KRAB-MeCP2 в модифицированных клетках 501-mel с использованием антител против Cas9. В качестве контроля использовали 501-mel клетки дикого типа (WT) без трансдукции (см. Дополнительный рисунок 3, Дополнительный рисунок 4, Дополнительный рисунок 5, Дополнительный рисунок 6 для сырого блоттинга). (B) анализ уровней RP11 и XLOC РНК на основе количественной ПЦР относительно контрольных клеток Rosa26 после трансдукции двойных гРНК в модифицированные клетки 501-mel. В качестве гена ведения хозяйства использовался GAPDH. Данные выражаются в виде среднего ± SD. (C) Анализ жизнеспособности репрессии двух некодирующих РНК был проведен после интеграции двойных гРНК в модифицированные клетки 501-mel в трех независимых трансдукциях. Значения люминесценции нормализовали по контролю Rosa26. Результаты жизнеспособности выражаются в виде среднего (n = 3) ± SEM. Был проведен непарный t-критерий. Звездочка (*) указывает на значимую разницу при p < 0,05. Пожалуйста, нажмите здесь, чтобы просмотреть увеличенную версию этой цифры.
Дополнительный рисунок 1: Плазмидная карта. Карта была создана с помощью Benchling (https://benchling.com/). Используемые в протоколе плазмиды были модифицированы из Addgene #96921 и #110824. Пожалуйста, нажмите здесь, чтобы загрузить этот файл.
Дополнительный рисунок 2: Дополнительный рисунок 2: Вестерн-блоттинг
Выражение dCas9-KRAB. (A) Экспрессия SadCas9-KRAB в клетках 501-mel через 6 дней после трансфекции. (B) Последовательная трансдукция SadCas9-KRAB и SpdCas9-KRAB в клетках 501-mel привела к снижению экспрессии SadCas9-KRAB через 3 недели после трансфекции. (C) Одновременная трансдукция SadCas9-KRAB и SpdCas9-KRAB в клетках 501-mel привела к снижению экспрессии SadCas9-KRAB через 3 недели после трансфекции. (D) Экспрессия SpdCas9-KRAB может наблюдаться после одновременной трансдукции SadCas9-KRAB и SpdCas9-KRAB. LV. Объем используемых лентивирусов в μL. Пожалуйста, нажмите здесь, чтобы скачать этот файл.
Дополнительный рисунок 3: Исходные данные вестерн-блоттинга SaCas9-KRAB. Пожалуйста, нажмите здесь, чтобы загрузить этот файл.
Дополнительный рисунок 4: Исходные данные SpCas9-KRAB по вестерн-блоттингу. Пожалуйста, нажмите здесь, чтобы загрузить этот файл.
Дополнительный рисунок 5: Исходные данные GAPDH_SpCas9-KRAB. Пожалуйста, нажмите здесь, чтобы скачать этот файл.
Дополнительный рисунок 6: Исходные данные GAPDH_SaCas9-KRAB. Пожалуйста, нажмите здесь, чтобы загрузить этот файл.
Дополнительная таблица 1: Используемые последовательности направляющей РНК. Пожалуйста, нажмите здесь, чтобы загрузить этот файл.
Дополнительная таблица 2: Используемые последовательности ДНК. Пожалуйста, нажмите здесь, чтобы загрузить этот файл.
Дополнительный файл 1: FASTA файла dual-grna-zeo-gfp. Пожалуйста, нажмите здесь, чтобы загрузить этот файл.
Дополнительный файл 2: FASTA-файл dSpCas9-krab-mecp2. Пожалуйста, нажмите здесь, чтобы загрузить этот файл.
Дополнительный файл 3: FASTA-файл dSaCas9-krab-mecp2. Пожалуйста, нажмите здесь, чтобы загрузить этот файл.
Обсуждение
В этом исследовании мы реализовали стратегию нацеливания на двойную lncRNA в клетках меланомы с использованием CRISPRi на основе dCas9-KRAB-MeCP2 из двух ортогональных видов. Используя эту систему, мы успешно подавили синтетическую летальную пару некодирующих РНК, RP11 и XLOC, что привело к увеличению гибели клеток, в отличие от случаев, когда они были подавлены по отдельности. Тем не менее, системы CRISPRi несут риск подавления генов вне мишени, что потенциально влияет на интерпретацию результатов. Несмотря на то, что экспериментальная валидация не проводилась, мы свели к минимуму потенциальные побочные эффекты во время дизайна гРНК in silico.
Изменчивость эффективности трансдукции может повлиять на воспроизводимость. Поэтому мы оптимизировали трансдукцию путем применения равных количеств лентивирусов, определенных с помощью стандартной калибровки кривой с использованием различных концентраций лентивирусов. Тем не менее, увеличенный размер груза нового вектора может наложить ограничения на эффективность лентивирусной упаковки, потенциально снижая эффективность трансдукции, особенно в труднопереносимых клеточных линиях. В результате, для достижения достаточной трансдукции и устойчивой экспрессии dCas9 в некоторых случаях могут потребоваться высокие титры вируса. Кроме того, недостаточный нокдаун можно обойти путем частого мониторинга уровней белка dCas9-KRAB-MeCP2 и экспрессии РНК целевых некодирующих РНК. В качестве альтернативы, существуют версии dCas9 с только одной системой репрессоров для преодоления этого ограничения, однако, со стоимостью снижения эффективности25.
Успешную активность продемонстрировала только комбинация XLOC-sa-RP11-sp, в то время как обратная конфигурация XLOC-sp-RP11-sa оказалась неэффективной. Правдоподобным объяснением этой разницы в эффективности нокдауна является взаимодействие между специфичностью PAMи доступностью хроматина. Учитывая, что SaCas9 и SpCas9 распознают различные последовательности PAM, сайт-мишень для SpCas9 в ориентации XLOC-sp-RP11-sa может не иметь оптимального PAM, тем самым снижая его аффинность связывания и последующую активность. Кроме того, пространственное расположение двух вариантов гибридного белка dCas9 может влиять на архитектуру хроматина и доступность34,35, потенциально ограничивая рекрутирование транскрипционного механизма или связанных с ним регуляторных факторов в конфигурации XLOC-sp-RP11-sa. Эти результаты свидетельствуют о том, что относительное расположение SaCas9 и SpCas9 в данном геномном контексте может критически повлиять на их функциональную эффективность, что требует дальнейших исследований для оптимизированных стратегий модуляции на основе CRISPR.
Разработанный протокол может быть адаптирован для нацеливания или скрининга других синтетических летальных некодирующих пар РНК путем модификации двойного вектора гРНК. Как только исследуемая клеточная линия стабильно экспрессирует оба варианта dCas9, становятся возможными различные подходы. В то время как РНК-интерференции, АСО и шРНК могут быть использованы для подавления экспрессии генов, CRISPRi предлагает более простую методологию скрининга, и его комбинация с небольшими химическими соединениями или ингибиторами также возможна. Заменяя вектор двойной гРНК библиотекой с двумя гРНК, этот протокол позволяет проводить систематический скрининг синтетических летальных пар или других генетических зависимостей в раковых клетках, тем самым повышая точность нацеливания на раковые клетки.
Несмотря на то, что мы добились интеграции вариантов dCas9-KRAB-MeCP2 с помощью лентивирусной трансдукции, альтернативные методы стабильной трансфекции также могут быть использованы для включения желаемых ферментов в клетки 501-mel. Это особенно актуально, поскольку, по нашему опыту, сайленсинг больших конструкций, таких как dCas9-KRAB-MeCP2, может происходить с течением времени36,37. В качестве альтернативы, dCas9-KRAB был показан как меньшая альтернатива23. По нашему опыту, трансдукция меньшими по размеру гибридными белками dCas9-KRAB, полученными из ортологов Cas9, приводила к снижению экспрессии белка при длительном культивировании (см. дополнительный рисунок 2). Таким образом, следует регулярно контролировать постоянную экспрессию белка в повторяющиеся моменты времени. Кроме того, трансдукция двух больших гибридных белков вместе с селекционными маркерами и двойными кассетами экспрессии гРНК может индуцировать клеточный стресс или может достигать емкости лентивирусной упаковки.
Достижение 100% эффективности нокдауна при двойном CRISPRi может быть сложной задачей из-за неоптимального дизайна гРНК или недостаточной экспрессии dCas9. Чтобы решить эту проблему, необходимо протестировать несколько гРНК на мишень с использованием передовых инструментов проектирования, чтобы определить наиболее эффективные последовательности. Кроме того, оптимизация экспрессии dCas9-KRAB-MeCP2 за счет улучшенного векторного дизайна и характеризации клональных клеточных линий может повысить эффективность нокдауна.
Альтернативные стратегии интеграции системы CRISPRi включают использование транспозазных векторов или CRISPR-направленную интеграцию для достижения стабильной вставки в безопасные геномные сайты, которые, как известно, поддерживают стабильную экспрессиюс течением времени. Тем не менее, многие типы клеток, включая клетки меланомы, трудно трансфектировать39, что делает лентивирусную трансдукцию предпочтительным методом. Электропорация ферментов на основе транспозазы или Cas может предложить альтернативные способы интеграции конструкций dCas9-KRAB-MeCP2 в клетки меланомы40. Реализация системы индуцируемого нокаута была бы выгодна для активации экспрессии dCas9-KRAB-MeCP2 по мере необходимости, а не конститутивной экспрессии для минимизации клеточного стресса.
Наконец, разработанный подход не ограничивается клетками меланомы, но может быть распространен на любой тип клеток, где исследуется синтетическая летальность. Этот подход явно предназначен для масштабирования в сторону модальности скрининга библиотеки гРНК. Однако из-за повышенной комбинаторной сложности, например, при наличии более трех гРНК на пару генов, включая контрольную группу, и по меньшей мере 300-кратного охвата, на практике можно нацеливаться только на субтранскриптомную фракцию экспрессированных некодирующих РНК. Таким образом, пользователи должны тщательно отбирать кандидатов, учитывая такие факторы, как уровни экспрессии некодирующей РНК и их известную значимость в конкретных клеточных контекстах, прежде чем приступать к библиотечному скринингу. Тем не менее, комбинаторная универсальность этого подхода делает его ценным инструментом для исследования синтетических летальных взаимодействий и других генетических зависимостей некодирующих РНК при различных типах рака и, возможно, других заболеваниях. Это расширяет доступный в настоящее время инструментарий для изучения этих сетевых взаимодействий в клетках.
Раскрытие информации
Йохен Имиг в настоящее время финансируется CGCIII компанией Pfizer Inc. CGCIII спонсируется компаниями Pfizer Inc., Merck KGaA и AstraZeneca PLC. Спонсоры не играли никакой роли в разработке, исполнении, интерпретации или написании исследования. У всех авторов нет конфликта интересов, который можно было бы раскрыть.
Благодарности
Выражаем признательность Стефану Раунсеру (Институт молекулярной физиологии Макса Планка, Дортмунд) за доступ к лабораторному пространству уровня биобезопасности 2. Особая благодарность Эрику Вангу за его интеллектуальный вклад, а также всем бывшим и нынешним членам лаборатории Имиг за их ценные дискуссии. Йохен Имиг в настоящее время финансируется CGCIII компанией Pfizer Inc. в CGC III.
Материалы
| Name | Company | Catalog Number | Comments |
| Amicon Ultra Centrifugal Filter, 100 kDa MWCO | Millipore | UFC910024 | |
| anti-Cas9 (S. aureus) (6H4) mouse monoclonal antibody | Cell Signaling Technology | 48989 | |
| anti-Cas9 (S. pyogenes) (7A9-3A3) monoclonal antibody | Cell Signaling Technology | 14697 | |
| anti-GAPDH mouse monoclonal antibody | Sigma Aldrich | G8795 | |
| Anti-Mouse IgG (whole molecule)–Peroxidase antibody produced in rabbit | Sigma Aldrich | A9044 | |
| Bio-Rad ChemiDoc MP Imaging System | Bio-Rad | ||
| Blasticidine S hydrochloride | Sigma Aldrich | 15205-25MG | |
| CellTiter-Glo Luminescent Cell Viability Assay | Promega | G7570 | |
| Centrifuge | Eppendorf | 5804R | |
| Centrifuge | Eppendorf | 5415R | |
| CFX Connect Real-Time PCR Detection System | Bio-Rad | ||
| Clarity Western ECL Substrate, 500 mL | Bio-Rad | 1705061 | |
| CO2 Incubator Model CB 170 | Binder | ||
| cOmplete Protease Inhibitor Cocktail | Roche | 11697498001 | |
| Esp3I (BsmB1) restriction enzyme | Thermo Scientific | ER0451 | |
| Falcon 10 mL Serological Pipet | Corning | 356551 | |
| Falcon 25 mL Serological Pipet | Corning | 357525 | |
| Falcon 5 mL Serological Pipet | Corning | 356543 | |
| Fetal Bovine Serum | Sigma Aldrich | F9665-500ML | |
| Gibco Dulbecco’s modified Eagle’s medium (DMEM), high glucose, pyruvate | Gibco | 41966029 | |
| Human melanoma cell line 501-mel | was kindly provided by the Aifantis Lab (New York University) | RRID: CVCL_4633 | |
| Immobilon -P PVDF Membrane | Millipore | IPVH00010 | |
| Lenti-X 293T HEK cell line | Takara Bio | 632180 | |
| Mini Trans-Blot Cell system | Bio-Rad | ||
| Mini-PROTEAN Tetra Handcast System | Bio-Rad | ||
| NEBuilder HiFi DNA Assembly Reaction | New England Biolabs | E2621 | |
| Non-fat milk powder | Biomol | 54650 | |
| NuPAGE LDS Sample Buffer (4x) | Invitrogen | NP0008 | |
| One Shot Stbl Chemically Competent E. coli | Invitrogen | C737303 | |
| Opti-MEM I Reduced Serum Medium | Gibco | 31985062 | |
| PCR Tubes 0.5 ml (Flat Cap) | VWR International | 732-3207 | |
| Phosphate-Buffered Saline (PBS) | Corning | 45000-446 | |
| Phusion High-Fidelity PCR Master Mix with HF Buffer | Thermo Scientific | F531L | |
| Pierce BCA Protein Assay Kit | Thermo Scientific | 23225 | |
| Polybrene | Sigma Aldrich | TR-1003-G | |
| Polyethylenimine, branched | Sigma Aldrich | 408727 | |
| Puromycin dihydrochloride | Santa Cruz Biotechnology | sc-108071A | |
| QIAquick PCR Purification Kit | Qiagen | 28104 | |
| SafeSeal Microcentrifuge Tube 1.5 mL | Sarstedt | 72,706 | |
| Sodium chloride, 5 M Aqua Solution, RNase Free | Alfa Aesar | J60434.AE | |
| Sodium Dodecyl Sulfate (SDS) | Sigma Aldrich | L3771 | |
| Syringe Filter PES 33mm 0.2 μM | Fisher Scientific | 15206869 | |
| TC Dish 100, Standard | Sarstedt | 8,33,902 | |
| TC Plate 6 Well, Standard, F | Sarstedt | 83,39,20,005 | |
| Tris base | Roche | 10708976001 | |
| TWEEN 20 | Sigma Aldrich | P9416-50ML |
Ссылки
- Lander, E. S., et al. Initial sequencing and analysis of the human genome. Nature. 409 (6822), 860-921 (2001).
- ENCODE Project Consortium. An integrated encyclopedia of DNA elements in the human genome. Nature. 489 (7414), 57-74 (2012).
- Szcześniak, M. W., Wanowska, E., Mukherjee, N., Ohler, U., Makałowska, I. Towards a deeper annotation of human lncRNAs. Biochim Biophys Acta Gene Regul Mech. 1863 (4), 194385(2020).
- Fang, S., et al. NONCODEV5: a comprehensive annotation database for long non-coding RNAs. Nucl Acids Res. 46 (D1), D308-D314 (2018).
- Lin, J., et al. Human-specific lncRNAs contributed critically to human evolution by distinctly regulating gene expression. eLife. 12, RP89001.2(2023).
- Sarropoulos, I., Marin, R., Cardoso-Moreira, M., Kaessmann, H. Developmental dynamics of lncRNAs across mammalian organs and species. Nature. 571 (7766), 510-514 (2019).
- Melé, M., et al. Human genomics. The human transcriptome across tissues and individuals. Science. 348 (6235), 660-665 (2015).
- Cabili, M. N., et al. Integrative annotation of human large intergenic noncoding RNAs reveals global properties and specific subclasses. Genes Dev. 25 (18), 1915-1927 (2011).
- Liu, S. J., Dang, H. X., Lim, D. A., Feng, F. Y., Maher, C. A. Long noncoding RNAs in cancer metastasis. Nat Rev Cancer. 21 (7), 446-460 (2021).
- Ahmad, M., Weiswald, L. B., Poulain, L., Denoyelle, C., Meryet-Figuiere, M. Involvement of lncRNAs in cancer cells migration, invasion and metastasis: cytoskeleton and ECM crosstalk. J Exp Clin Cancer Res. 42 (1), 173(2023).
- Jain, A. K., et al. LncPRESS1 Is a p53-Regulated LncRNA that Safeguards Pluripotency by Disrupting SIRT6-Mediated De-acetylation of Histone H3K56. Mol Cell. 64 (5), 967-981 (2016).
- Bergmann, J. H., et al. Regulation of the ESC transcriptome by nuclear long noncoding RNAs. Genome Res. 25 (9), 1336-1346 (2015).
- Kretz, M., et al. Control of somatic tissue differentiation by the long non-coding RNA TINCR. Nature. 493 (7431), 231-235 (2013).
- Yoon, J. H., et al. LincRNA-p21 suppresses target mRNA translation. Mol Cell. 47 (4), 648-655 (2012).
- Huarte, M. The emerging role of lncRNAs in cancer. Nat Med. 21 (11), 1253-1261 (2015).
- Badowski, C., He, B., Garmire, L. X. Blood-derived lncRNAs as biomarkers for cancer diagnosis: the Good, the Bad and the Beauty. NPJ Precis Oncol. 6 (1), 40(2022).
- Zong, X., et al. Knockdown of nuclear-retained long noncoding RNAs using modified DNA antisense oligonucleotides. Meth Mol Biol. 1262, 321-331 (2015).
- Gagnon, K. T., Li, L., Chu, Y., Janowski, B. A., Corey, D. R. RNAi factors are present and active in human cell nuclei. Cell Rep. 6 (1), 211-221 (2014).
- Zeng, Y., Cullen, B. R. RNA interference in human cells is restricted to the cytoplasm. RNA. 8 (7), 855-860 (2002).
- Yang, J., et al. CRISPR/Cas9-mediated noncoding RNA editing in human cancers. RNA Biol. 15 (1), 35-43 (2018).
- Liu, S. J., et al. CRISPRi-based genome-scale identification of functional long noncoding RNA loci in human cells. Science. 355 (6320), aah7111(2017).
- Tsung, K., et al. CRISPRi screen of long non-coding RNAs identifies LINC03045 regulating glioblastoma invasion. PLoS Genet. 20 (6), e1011314(2024).
- Gilbert, L. A., et al. CRISPR-mediated modular RNA-guided regulation of transcription in eukaryotes. Cell. 154 (2), 442-451 (2013).
- Boettcher, M., McManus, M. T. Choosing the right tool for the job: RNAi, TALEN, or CRISPR. Mol Cell. 58 (4), 575-585 (2015).
- Yeo, N. C., et al. An enhanced CRISPR repressor for targeted mammalian gene regulation. Nat Meth. 15 (8), 611-616 (2018).
- Parrish, P. C. R., et al. Discovery of synthetic lethal and tumor suppressor paralog pairs in the human genome. Cell Rep. 36 (9), 109597(2021).
- Najm, F. J., et al. Orthologous CRISPR-Cas9 enzymes for combinatorial genetic screens. Nat Biotechnol. 36 (2), 179-189 (2018).
- Petroulia, S., et al. CRISPR-inhibition screen for lncRNAs linked to melanoma growth and metastasis. bioRxiv. , (2024).
- Hsu, P. D., et al. DNA targeting specificity of RNA-guided Cas9 nucleases. Nat Biotechnol. 31 (9), 827-832 (2013).
- New England Biolabs. NEBuilder HiFi DNA Assembly Reaction (E2621) v2. , (2020).
- Cortés-Ríos, J., et al. Protein quantification by bicinchoninic acid (BCA) assay follows complex kinetics and can be performed at short incubation times. Anal Biochem. 608, 113904(2020).
- Junior, N. Polyacrylamide Gel Electrophoresis (SDS-PAGE) v1. , (2019).
- Chen, X., Liu, J., Janssen, J. M., Gonçalves, M. A. F. V. The chromatin structure differentially impacts high-specificity CRISPR-Cas9 Nuclease strategies. Mol Ther Nucl Acids. 8, 558-563 (2017).
- Schep, R., et al. Impact of chromatin context on Cas9-induced DNA double-strand break repair pathway balance. Mol Cell. 81 (10), 2216-2230.e10 (2021).
- Daer, R. M., Cutts, J. P., Brafman, D. A., Haynes, K. A. The impact of chromatin dynamics on Cas9-Mediated genome editing in human cells. ACS Syn Biol. 6 (3), 428-438 (2017).
- Chavez, M., Rane, D. A., Chen, X., Qi, L. S. Stable expression of large transgenes via the knock-in of an integrase-deficient lentivirus. Nat Biomed Eng. 7 (5), 661-671 (2023).
- Ellis, J. Silencing and variegation of gammaretrovirus and lentivirus vectors. Human Gene Ther. 16 (11), 1241-1246 (2005).
- Shrestha, D., et al. Genomics and epigenetics guided identification of tissue-specific genomic safe harbors. Genome Biol. 23 (1), 199(2022).
- Chu, Z., et al. Enhanced gene transfection and induction of apoptosis in melanoma cells by branched poly(β-amino ester)s with uniformly distributed branching units. J Control Release. 367, 197-208 (2024).
- Han, S. Y., et al. Nucleofection is a highly effective gene transfer techfnique for human melanoma cell lines. Exp Dermatol. 17 (5), 405-411 (2008).
Перепечатки и разрешения
Запросить разрешение на использование текста или рисунков этого JoVE статьи
Запросить разрешениеСмотреть дополнительные статьи
This article has been published
Video Coming Soon
Авторские права © 2025 MyJoVE Corporation. Все права защищены