Method Article
Estratégia de interferência dupla de CRISPR para direcionar interações letais sintéticas entre RNAs não codificantes em células cancerígenas
Neste Artigo
Resumo
Este estudo apresenta um sistema CRISPRi duplo direcionado a RNAs longos não codificantes em células de melanoma. Ele permite o knockdown de genes combinatórios e a triagem letal sintética, identificando interações de lncRNA específicas do câncer para possíveis estratégias terapêuticas.
Resumo
Os RNAs longos não codificantes (lncRNAs) representam uma classe vasta e funcionalmente diversa de moléculas de RNA, com mais de 100.000 previstas no genoma humano. Embora os lncRNAs sejam menos conservados entre as espécies em comparação com os genes codificadores de proteínas, eles desempenham papéis críticos na regulação gênica, interações da cromatina e progressão do câncer. Seu envolvimento no câncer os torna alvos terapêuticos promissores. A interferência CRISPR (CRISPRi), utilizando Cas9 cataliticamente inativo fundido com um repressor transcricional como o KRAB-MeCP2, oferece um método preciso para direcionar lncRNAs nucleares e avaliar suas funções. Este estudo apresenta um sistema CRISPRi duplo usando tecnologias CRISPRi ortogonais de Staphylococcus aureus e Streptococcus pyogenes com base em dCas9-KRAB-MeCP2, otimizado para direcionamento combinatório de lncRNAs em células de melanoma humano. O protocolo facilita o knockdown de genes combinatórios ou a triagem letal sintética de pares de lncRNA, fornecendo uma nova ferramenta para a pesquisa do câncer. Ao explorar a letalidade sintética entre lncRNAs, essa abordagem pode ajudar a identificar interações de lncRNA críticas para a sobrevivência das células cancerígenas, oferecendo novas estratégias terapêuticas. A funcionalidade do sistema duplo é demonstrada, destacando seu potencial na identificação de interações críticas de lncRNA específicas do câncer.
Introdução
Embora menos de 3% do genoma humano codifique proteínas, aproximadamente 80% do genoma é transcrito 1,2. Entre as unidades transcricionais não codificantes, dezenas de milhares são classificadas como RNAs não codificantes longos (lncRNAs) excedendo 200 nucleotídeos, e estima-se que o número total de lncRNAs humanos exceda 100.000 3,4. Em contraste com os genes codificantes, os lncRNAs são menos conservados entre as espécies. Dado que os humanos compartilham 99% de seu genoma com primatas como os chimpanzés, acredita-se que os lncRNAs tenham uma influência muito maior na evolução fenotípica 5,6. Esses achados indicam importantes funções celulares dos lncRNAs. Embora a regulação dos lncRNAs e suas interações com proteínas de ligação ao RNA e outros RNAs permaneçam incompletamente compreendidas, e muitos lncRNAs ainda não tenham sido totalmente anotados, é claro que os lncRNAs exibem padrões de expressão específicos de células e tecidos na saúde e na doença, como o câncer 7,8,9,10. Eles estão implicados em diversas funções, incluindo regulação da transcrição gênica, envolvimento nas interações da cromatina11, processamento de RNA12, estabilização de RNA13 e regulação da tradução14.
No câncer, lncRNAs específicos de tipos de células altamente diversos influenciam o desenvolvimento e a metástase do tumor, regulando a expressão gênica, destacando seu potencial como alvos terapêuticos valiosos15. Além da detecção de lncRNAs como biomarcadores em amostras de tumor16, direcionar lncRNAs específicos do tumor para interromper suas funções a jusante tem um potencial significativo em aplicações clínicas e pesquisa básica. As abordagens baseadas em RNA para elucidar os papéis dos lncRNAs incluem oligonucleotídeos antisense (ASOs), RNAs em grampo curto e pequenos RNAs interferentes (siRNAs) 17 , 18 . Enquanto o siRNA é comumente utilizado para telas de silenciamento de genes, o knockdown baseado em siRNA é restrito ao citoplasma19. No entanto, o lncRNA freqüentemente opera dentro do núcleo.
Alternativamente, a interferência de repetições palindrômicas curtas agrupadas regularmente interespaçadas (CRISPRi) pode ser usada para inibir lncRNAs em cânceres humanos20. Além disso, as telas CRISPRi em todo o genoma podem ser facilmente programadas e direcionadas a uma ampla gama de genes codificantes e não codificantes para examinar seu impacto funcional21,22. No CRISPRi, um Cas9 deficiente catalítico (dCas9) é fundido a um domínio repressor transcricional, como o domínio da caixa associada a Krüppel (KRAB)23. A repressão gênica por dCas9-KRAB é guiada por um RNA guia (gRNA) para a região de interesse. O CRISPRi controla genes nos níveis de DNA, levando a uma maior eficiência e fenótipos de perda de função desejados em contraste com a interferência do RNA, que é ativa no nível pós-transcricional24. Em resposta à eficácia limitada do KRAB no silenciamento de alvos, uma fusão de KRAB e MeCP2 com dCas9 foi introduzida como uma estratégia de repressão mais eficaz25.
Embora o silenciamento de um único gene possa afetar a viabilidade do câncer, interações letais duplas ou múltiplas sintéticas podem resgatar células cancerígenas da morte celular26. A letalidade sintética envolve dois ou mais genes, cada um dos quais pode compensar a função do outro. Para superar problemas com telas de knockdown de gene único, estratégias CRISPR duplas direcionadas a pares de genes codificadores de proteínas letais oferecem uma abordagem promissora27.
Aqui, apresentamos um protocolo para o uso combinatório de direcionamento ortogonal baseado em CRISPRi de lncRNA ou outros pares de RNA não codificantes usando Staphylococcus aureus e Streptococcus pyogenes dCas9 fundidos a KRAB-MeCP2 em células de melanoma humano (ver Figura 1). O protocolo pode ser utilizado para knockdown CRISPR clássico combinatório ou como triagem baseada em CRISPRi de pares letais sintéticos no câncer. O uso de duas espécies diferentes de dCas9, SpCas9 e SaCas9, na abordagem CRISPRi permite o direcionamento independente de loci genômicos distintos com reatividade cruzada mínima, aumentando a especificidade e a flexibilidade, garantindo uma alta seletividade no alvo. Diferentes motivos adjacentes ao protoespaçador (PAMs) são utilizados: NGG para SpCas9 e NNGRRT para SaCas9. O sistema dCas9 duplo aborda desafios como compatibilidade limitada de sgRNA, permitindo modulação precisa e simultânea e reduzindo a competição do complexo sgRNA-RNP ao usar um único tipo de dCas9. Essa inovação melhora a robustez e a versatilidade da triagem de biblioteca de sgRNA dupla. Em conclusão, fornecemos um sistema CRISPRi duplo totalmente funcional em células de melanoma como um modelo de câncer.
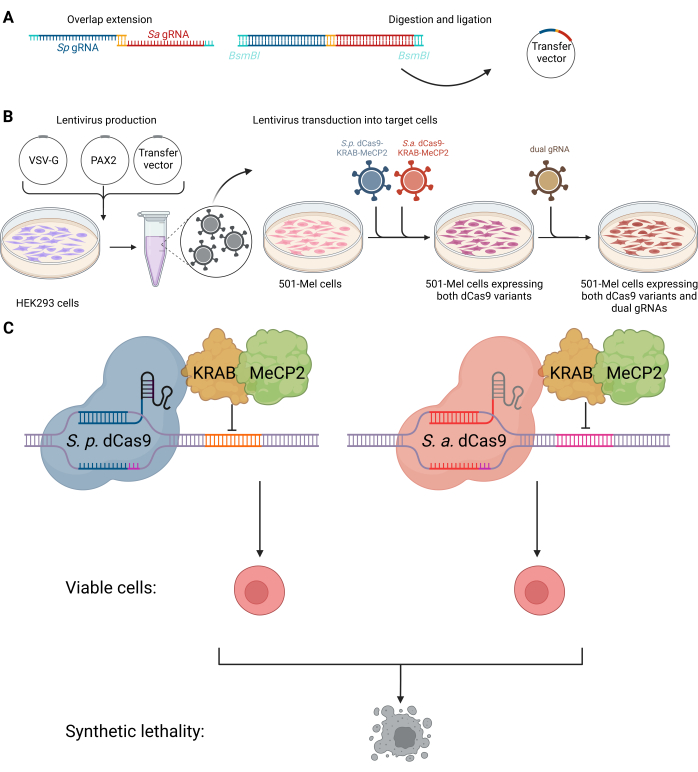
Figura 1: Esquema do sistema CRISPRi duplo para direcionar interações letais sintéticas de RNAs não codificantes. (A) Procedimento de clonagem do vetor duplo de gRNA. (B) As células HEK293 foram transfectadas com plasmídeo de envelope VSV-G, plasmídeo de empacotamento PAX2 e vetor de transferência para produzir lentivírus para posterior transdução em células 501-mel. Lentivírus contendo a informação genética para SpdCas9-KRAB-MeCP2 (azul) e SadCas9-KRAB-MeCP2 (vermelho) foram integrados em células 501-mel simultaneamente. Após a seleção do antibiótico, uma segunda transdução lentiviral foi realizada para integrar os gRNAs duplos desejados (verde oliva). (C) As células 501-mel que expressam as variantes dCas9 interagem com seus gRNAs correspondentes para silenciar a expressão do gene alvo, resultando na morte das células cancerígenas. Criado com BioRender.com. Clique aqui para ver uma versão maior desta figura.
Protocolo
A linha celular de melanoma humano 501-mel (RRID: CVCL_4633) foi gentilmente cedida pelo Aifantis Lab (Universidade de Nova York). A linha celular Lenti-X 293T HEK foi adquirida da Takara Bio. Essas linhagens celulares foram cultivadas em meio de Eagle modificado (DMEM) de Dulbecco com alta glicose suplementada com 10% de soro fetal bovino (FBS) a 37 °C em uma atmosfera de 5% de CO2 sob condições estéreis. As células foram passadas até atingirem 80%-90% de confluência. As linhagens celulares foram testadas regularmente para contaminação por micoplasma.
1. Projetando gRNAs usando uma plataforma de design de gRNA online
- Projete um gRNA específico para SpCas9 e um gRNA específico para SaCas9 para cada lncRNA alvo para avaliar qual variante produz resultados superiores de knockdown.
- Direcione os lncRNAs desejados, aqui RP11-120D5.1 (RP11) e XLOC_030781 (XLOC), usando o gRNA mais eficiente para cada S. pyogenes (gRNA-sp) Cas9 e S. aureus (gRNA-sa) Cas9.
NOTA: Os gRNAs de S. pyogenes podem ser extraídos de Petroulia et al.28. - Siga as recomendações específicas do local para priorizar a maior atividade no alvo, expressa por altas pontuações no alvo, minimizando as correspondências fora do alvo, conforme sugerido pelas ferramentas de design de gRNA.
- Escolha o genoma GRCh38 (hg, Homo sapiens). Selecione o site do PAM para o SpCas9 (NGG) e o SaCas9 (NNGRRT) específicos.
- Use o método de pontuação fora do alvo de Hsu et al.29. Posicione os gRNAs dentro de uma janela de -150 pb a +50 pb ao redor do local de início da transcrição para um desempenho ideal.
- Escolha gRNAs com a maior pontuação no alvo e fora do alvo (consulte a Tabela Suplementar 1).
- Combine um gRNA de controle não direcionado ou embaralhado, aqui contra Rosa26, com cada gRNA alvo para criar um vetor de gRNA duplo, visando um lncRNA. A combinação de gRNAs alvo em um plasmídeo permite a modificação específica do local de ambos os locais-alvo.
NOTA: O vetor de gRNA duplo que abriga o gRNA de controle e o gRNA alvo permite o knockdown de um lncRNA alvo, enquanto os vetores de gRNA duplos contendo gRNAs direcionados a locais permitem o knockdown simultâneo de lncRNAs direcionados.
2. Clonagem de gRNA
- Realize uma PCR de extensão de sobreposição usando dois fragmentos de DNA contendo a sequência de gRNA. Use o primer direto U6 e o primer reverso H1 (ver Tabela Suplementar 2) para amplificação, conforme descrito por Najm et al.27.
- Configure uma reação de PCR de 50 μL com 25 μL de mistura mestre de PCR de alta fidelidade 2x com tampão HF e 1 μL de cada fragmento de DNA contendo o gRNA de S. pyogenes ou S. aureus . Execute a reação nas seguintes condições de ciclagem: desnaturação inicial a 98 ° C por 30 s, seguida por 15 ciclos de desnaturação a 98 ° C por 5 s, recozimento a 55 ° C por 10 s e alongamento a 72 ° C por 15 s.
- Adicione 2,5 μL de primer direto U6 e 2,5 μL de primer reverso H1 após completar os 15 ciclos iniciais e submeta a reação a mais 20 ciclos nas mesmas condições, com uma extensão final por 5 min a 72 ° C.
- Purifique os produtos de PCR usando um kit de purificação de PCR de acordo com as instruções do fabricante.
- Digerir 1 μg do plasmídeo modificado utilizando 1 μl de enzima de restrição BsmB1 e 1 μl de tampão 10x durante 30 min a 37 °C numa reacção de 10 μL.
- Clone os produtos de PCR no plasmídeo dual-gRNA-Zeo-GFP digerido (ver Figura Suplementar 1 e Arquivo Suplementar 1) usando um protocolo de reação de montagem de DNA HiFi conforme descrito por outros30.
- Transforme células de E. coli quimicamente competentes misturando 50 μL de cultura de bactérias com 4 μL de produto de montagem de acordo com as instruções do fabricante.
3. Produção de lentivírus
CUIDADO: Manuseie lentivírus ativos em todas as etapas, seguindo as diretrizes de segurança apropriadas. Sempre trabalhe com vírus em um gabinete de segurança Classe 2 para evitar riscos de formação inevitável de aerossóis. Realize todo o trabalho fora do gabinete de segurança, como centrifugação, em recipientes e rotores à prova de aerossóis aprovados para uso com organismos do Grupo de Risco 2. Use roupas de proteção adequadas, incluindo jaleco, luvas descartáveis e óculos de segurança, em todos os momentos na área de trabalho. As superfícies de trabalho devem ser tratadas com uma solução desinfetante, e outros resíduos também devem ser tratados com uma solução desinfetante, permitindo que permaneçam no gabinete selado sob luz ultravioleta por pelo menos 240 min. As pipetas sorológicas utilizadas e as pontas das pipetas devem ser enxaguadas em uma solução desinfetante antes do descarte em frascos autoclaváveis. Em seguida, os resíduos devem ser descartados no recipiente de resíduos S2. Os resíduos S2 serão inativados por meio de autoclavagem. As linhagens celulares transduzidas podem ser manuseadas em condições S1 não antes de 2 dias após a transdução e somente após pelo menos duas trocas completas do meio de cultura com um meio livre de vírus.
- Conte as células Lenti-X 293T HEK misturando 10 μL de células com 10 μL de solução de azul de tripano em um hemocitômetro. Placa 4 x 106 células Lenti-X 293T HEK por placa de 10 cm para atingir ~ 80% de confluência no dia seguinte.
- Trabalhe em um laboratório de Classe 2 a partir desta etapa. Misture 500 μL de um meio de transfecção adequado com o vetor de transferência de 11,25 μg SpdCas9-KRAB-MeCP2 (ver Arquivo Suplementar 2), plasmídeo de envelope de 5,5 μg VSV-G e vetor pPAX2 de 16,5 μg. Incube por 5 min em temperatura ambiente. Para a segunda variante dCas9, use o vetor de transferência SadCas9-KRAB-MeCP2 (consulte o Arquivo Suplementar 3).
- Em um tubo separado, misture 500 μL de soro reduzido com 36 μL de reagente PEI (estoque: 1 mg / mL). Incube por 5 min em temperatura ambiente.
- Combine as misturas e incube por 15 min em temperatura ambiente. Adicione a mistura cuidadosamente gota a gota ao meio celular (6 ml) e incube durante a noite a 37 °C e 5% de CO2. Certifique-se de que as células permaneçam presas para evitar a redução do rendimento lentiviral.
- Descarte o meio em um frasco de lixo autoclavável usando uma pipeta sorológica após 12-15 h. A partir de agora, enxágue os consumíveis, incluindo pipetas sorológicas e pontas de pipeta, com a solução desinfetante antes de descartá-los no lixo da autoclave. Adicione 5 mL de meio DMEM fresco às células transfectadas usando uma pipeta nova.
- Colha lentivírus transferindo o sobrenadante usando uma pipeta sorológica para um tubo canônico de 50 mL 48 h após a transfecção. Guarde o tubo canônico na geladeira. Adicione 5 ml de meio fresco às células e incube as células durante mais 24 h.
- Repita a coleta de lentivírus transferindo o sobrenadante para o mesmo tubo canônico de 50 mL contendo a primeira colheita. Novamente, adicione 5 mL de meio fresco, incube por 24 h e transfira o sobrenadante depois para o tubo canônico de 50 mL. Armazenar sobrenadantes combinados da primeira, segunda e terceira colheitas no tubo canónico de 50 ml a 4 °C até filtração e centrifugação.
- Centrifugar o sobrenadante combinado durante 5 minutos a 500 x g e 4 °C em recipientes e rotores estanques a aerossóis aprovados para utilização com organismos do grupo de risco 2 para pulverizar células e detritos destacados. Filtrar o sobrenadante através de filtros de seringa de grau de cultura de células estéreis de 0,2 μm.
- Concentrar o sobrenadante filtrado a 1.000 x g e 4 °C utilizando uma unidade de filtragem centrífuga com um limite de peso molecular adequado de 100 kDa a cerca de 500 μL. Armazenar alíquotas de vírus de 50-100 μL a -80 °C até nova utilização.
4. Transdução de lentivírus e seleção de células transfectadas estáveis
CUIDADO: Sempre trabalhe com vírus em um gabinete de segurança Classe 2.
- Placa de células 501-mel em uma placa de 6 poços a 2 x 105 células/poço com um volume de cultura final de 2 mL, 1 dia antes da transdução.
- Substituir o meio de cultura por 2 ml de meio DMEM pré-aquecido suplementado com polibreno a uma concentração final de 6 μg/ml.
- A partir de agora, é necessário trabalhar no laboratório da Classe 2. Adicione 50 μL de lentivírus contendo SpdCas9-KRAB-MeCP2 e SadCas9-KRAB-MeCP2 simultaneamente às células. Enxágue os consumíveis, incluindo pipetas sorológicas e pontas de pipeta, com a solução desinfetante antes de descartá-los no lixo da autoclave. Agitar suavemente a placa e incubar a 37 °C numa atmosfera de CO2 a 5%.
- Descarte o meio em um frasco autoclavável usando uma pipeta sorológica e adicione 2 mL de meio fresco contendo blasticidina (10 μg / mL) e puromicina (2 μg / mL) 16 h após a transdução.
- Continue a seleção substituindo o meio por meio fresco de 2 mL contendo blasticidina (10 μg / mL) e puromicina (2 μg / mL) após 2 dias. Manuseie as células transduzidas sob condições S1 após 7 dias após a transdução.
NOTA: A seleção de antibióticos deve ser realizada até que todas as células de controle não transduzidas tenham morrido. Estratégias alternativas de seleção podem ser usadas dependendo do de resistência no vetor de transferência. Se necessário, otimize as concentrações de antibióticos usando uma curva de matança. A seleção elaborada de clones de célula única por meio de citometria de fluxo ou diluição e plaqueamento em série, seguida de quantificação de proteínas, pode ser necessária para obter populações de células que expressem fusões de dCas9 em níveis suficientes.
5. Quantificação de proteínas e análise de Western blot
- Conte células 501-mel transfectadas estáveis misturando 10 μL de células com 10 μL de solução de azul de tripano em um hemocitômetro. Centrifugue 1 x 106 das células por 5 min a 500 x g e 4 ° C. Descarte o sobrenadante e ressuspenda o pellet celular em 1 mL de PBS gelado.
- Repita a centrifugação e ressuspenda o pellet celular em 50 μL de tampão RIPA contendo inibidores de protease. Incubar em gelo por 15 min e remover os detritos celulares por centrifugação a 13.000 x g por 10 min a 4 ° C. Conservar o sobrenadante a -20 °C ou utilizá-lo imediatamente para a quantificação das proteínas, tal como descrito por outros31.
- Misture 10 μg de proteína com 4 x tampão LDS, 50 mM DTT e encha até 20 μL com água destilada. Separe as proteínas em um gel de separação a 10% usando SDS-PAGE à base de Tris / glicina a 135 V por 1 h, conforme descrito anteriormente32. Transfira proteínas para uma membrana de PVDF a 90 V por 90 min com uma transferência de tanque úmido.
- Incubar a membrana em tampão de bloqueio (5% de leite desnatado em TBS-T) por 1 h a 180 rpm em temperatura ambiente.
- Adicione anticorpo monoclonal de camundongo anti-GAPDH 1:10.000 no tampão de bloqueio como controle interno. Adicionar o anticorpo primário anti-Cas9 (S. pyogenes) ou anti-Cas9 (S. aureus) numa diluição de 1:1 000 no tampão de bloqueio e incubar durante a noite a 4 °C com agitação suave.
- Lave a membrana 3x com tampão TBST por 10 min cada. Incubar com anticorpo HRP anti-camundongo secundário (1:10.000) por 1 h em temperatura ambiente.
- Lave 3x com tampão TBST por 10 min cada. Detecte sinais usando o substrato ECL e um sistema de imagem.
6. Ensaio de viabilidade de repressão de LncRNA duplo
CUIDADO: Sempre trabalhe com vírus em um gabinete de segurança Classe 2.
- Semear 0,1 x 105 células contendo SadCas9-KRAB e SpdCas9-KRAB transfectados estáveis 7 dias após a transdução em uma placa de 96 poços em 100 μL de meio DMEM pré-aquecido 1 dia antes da transdução.
- No dia da transdução, substituir o meio de cultura por 100 μl de meio DMEM pré-aquecido suplementado com polibreno a uma concentração final de 6 μg/ml. A partir de agora, é necessário trabalhar no laboratório da Classe 2. Adicione o volume apropriado de lentivírus às células.
NOTA: O volume de lentivírus contendo o vetor gRNA duplo deve ser examinado usando GFP para gerar uma curva linear titulando lentivírus. - Substitua o meio por meio fresco contendo 500 μg / mL de zeocina, 5 μg / mL de blasticidina e 1 μg / mL de puromicina 16 h após a transdução.
- Realize a detecção de luminescência 5 dias após a transdução usando um ensaio de viabilidade celular de acordo com as instruções do fabricante.
Resultados
Os de expressão de SadCas9-KRAB-MeCP2 e SpdCas9-KRAB-MeCP2 foram integrados em células 501-mel simultaneamente usando transdução com quantidades iguais de lentivírus. Como este protocolo CRISPRi depende da expressão de variantes dCas9-KRAB-MeCP2, a análise de Western blot é ideal para confirmar sua presença no nível da proteína. Após o enriquecimento das células transfectadas positivamente, amostras foram coletadas para análise de Western blot para confirmar a presença dos ortólogos dCas9-KRAB-MeCP2. Após a transdução bem-sucedida, uma única banda correspondente à proteína de fusão SpdCas9-KRAB-MeCP2 (202 kDa) e à proteína de fusão SadCas9-KRAB-MeCP2 (170 kDa) é esperada e foi confirmada, conforme mostrado na Figura 2A, usando anticorpos específicos para os respectivos ortólogos Cas9. No entanto, se o Western blot não for viável, a qPCR pode ser usada para verificar as proteínas de fusão no nível do mRNA.
Uma vez que as células 501-mel expressando de forma estável os repressores funcionais foram geradas, a segunda transdução lentiviral foi realizada usando um vetor gRNA duplo. RP11 e XLOC foram previamente mostrados como regulados positivamente em culturas de melanoma metastático de curto prazo, e o direcionamento individual de CRISPRi demonstrou seu impacto na proliferação e sobrevivência celular28. Assim, selecionamos este par de lncRNA para avaliação como uma combinação de RNA não codificante letal sintético de prova de conceito, antecipando um fenótipo de crescimento cumulativo e facilmente observável nas células modificadas.
Para investigar os efeitos do CRISPRi duplo em células cancerígenas usando ensaios de viabilidade, ambos os lncRNAs direcionados devem ser efetivamente reprimidos. qPCR é o método mais adequado para confirmar essa repressão medindo seus níveis de transcrição. Avaliamos a funcionalidade do protocolo CRISPRi duplo desenvolvido determinando os níveis de RNA e examinando a viabilidade celular ao direcionar o par RP11 e XLOC potencialmente letal. Portanto, diferentes gRNAs direcionados a RP11 e XLOC foram introduzidos para reduzir os níveis de RNA alvo. A repressão do RNA foi investigada usando gRNAs específicos do alvo para SadCas9-KRAB-MeCP2 ou SpdCas9-KRAB-MeCP2, testados individualmente e em combinação com um gRNA de controle direcionado a Rosa26 (ver Figura 2B). Embora os gRNAs direcionados a XLOC ou RP11 individualmente tenham suprimido efetivamente seus respectivos alvos, o knockdown simultâneo de XLOC e RP11 foi alcançado apenas com o par XLOC-sa / RP11-sp.
Como os níveis de RNA XLOC e RP11 foram reduzidos com sucesso na linha celular 501-mel com o par de gRNA XLOC-sa / RP11-sp selecionado, foi realizado um ensaio de viabilidade de repressão de lncRNA duplo (ver Figura 2C). Após 5 dias, a viabilidade celular foi de 81% e 89% do controle Rosa26 quando os gRNAs alvo foram usados individualmente. A combinação de RP11-sp e XLOC-sa resultou em uma redução significativa adicional em comparação com RP11-sp sozinho, com viabilidade celular em 59% do controle Rosa26.

Figura 2: Funcionalidade do sistema de repressão de lncRNA duplo. (A) A análise de Western blot foi realizada para detectar as variantes dCas9-KRAB-MeCP2 em células 501-mel modificadas usando anticorpos anti-Cas9. Células do tipo selvagem (WT) 501-mel sem transdução foram usadas como controle (ver Figura Suplementar 3, Figura Suplementar 4, Figura Suplementar 5, Figura Suplementar 6 para o blot bruto). (B) análise baseada em qPCR dos níveis de RNA RP11 e XLOC em relação às células de controle Rosa26 após a transdução de gRNAs duplos em células 501-mel modificadas. O GAPDH foi usado como o gene de limpeza. Os dados são expressos como ± média SD. (C) O ensaio de viabilidade de repressão de lncRNA duplo foi realizado após a integração de gRNAs duplos em células 501-mel modificadas em três transduções independentes. Os valores de luminescência foram normalizados para o controle Rosa26. Os resultados de viabilidade são expressos como a média (n = 3) ± EPM. Foi realizado um teste t não pareado. Asterisco (*) indica uma diferença significativa em p < 0,05. Clique aqui para ver uma versão maior desta figura.
Figura suplementar 1: Mapa de plasmídeo. O mapa foi criado com Benchling (https://benchling.com/). Os plasmídeos utilizados no protocolo foram modificados a partir de Addgene #96921 e #110824. Clique aqui para baixar este arquivo.
Figura Suplementar 2: Figura Suplementar 2: Análise de Western blot de
Expressão dCas9-KRAB. (A) Expressão de SadCas9-KRAB em células 501-mel 6 dias após a transfecção. (B) A transdução sequencial de SadCas9-KRAB e SpdCas9-KRAB em células 501-mel levou à redução da expressão de SadCas9-KRAB 3 semanas após a transfecção. (C) A transdução simultânea de SadCas9-KRAB e SpdCas9-KRAB em células 501-mel resultou em expressão reduzida de SadCas9-KRAB 3 semanas após a transfecção. (D) A expressão de SpdCas9-KRAB pode ser observada após a transdução simultânea de SadCas9-KRAB e SpdCas9-KRAB. LV. O volume de lentivírus usado em μL. Clique aqui para baixar este arquivo.
Figura suplementar 3: Dados brutos Western blot SaCas9-KRAB. Clique aqui para baixar este arquivo.
Figura suplementar 4: Dados brutos de Western blot SpCas9-KRAB. Clique aqui para baixar este arquivo.
Figura 5 suplementar: Dados brutos GAPDH_SpCas9-KRAB. Clique aqui para baixar este arquivo.
Figura 6 suplementar: Dados brutos GAPDH_SaCas9-KRAB. Clique aqui para baixar este arquivo.
Tabela Suplementar 1: Sequências de RNA guia utilizadas. Clique aqui para baixar este arquivo.
Tabela Suplementar 2: Sequências de DNA utilizadas. Clique aqui para baixar este arquivo.
Arquivo suplementar 1: Arquivo FASTA de dual-grna-zeo-gfp. Clique aqui para baixar este arquivo.
Arquivo Suplementar 2: Arquivo FASTA de dSpCas9-krab-mecp2. Clique aqui para baixar este arquivo.
Arquivo Suplementar 3: Arquivo FASTA de dSaCas9-krab-mecp2. Clique aqui para baixar este arquivo.
Discussão
Neste estudo, implementamos uma estratégia dupla de direcionamento de lncRNA em células de melanoma usando CRISPRi baseado em dCas9-KRAB-MeCP2 de duas espécies ortogonais. Utilizando este sistema, reprimimos com sucesso um par letal sintético de lncRNAs, RP11 e XLOC, levando ao aumento da morte celular, ao contrário de quando foram reprimidos individualmente. No entanto, os sistemas CRISPRi apresentam um risco de repressão gênica fora do alvo, afetando potencialmente a interpretação dos resultados. Embora a validação experimental não tenha sido realizada, minimizamos os potenciais efeitos fora do alvo durante o projeto de gRNA in silico.
A variabilidade na eficiência da transdução pode afetar a reprodutibilidade. Portanto, otimizamos a transdução aplicando quantidades iguais de lentivírus, determinadas por meio de uma calibração de curva padrão usando concentrações variáveis de lentivírus. No entanto, o aumento do tamanho da carga do vetor recém-projetado pode impor limitações à eficiência do empacotamento lentiviral, reduzindo potencialmente a eficiência da transdução, particularmente em linhagens celulares difíceis de transduzir. Como resultado, alcançar transdução suficiente e expressão robusta de dCas9 pode exigir altos títulos virais em certos casos. Além disso, o knockdown insuficiente pode ser contornado monitorando frequentemente os níveis de proteína dCas9-KRAB-MeCP2 e a expressão de RNA de lncRNAs direcionados. Alternativamente, existem versões dCas9 disponíveis com apenas um sistema repressor para superar essa limitação, porém, com o custo de diminuição da eficiência25.
Apenas a combinação XLOC-sa-RP11-sp demonstrou atividade bem-sucedida, enquanto a configuração reversa XLOC-sp-RP11-sa foi ineficaz. Uma explicação plausível para essa diferença na eficácia do knockdown é a interação entre a especificidade do PAM e a acessibilidade da cromatina33. Dado que SaCas9 e SpCas9 reconhecem sequências PAM distintas, o local alvo para SpCas9 na orientação XLOC-sp-RP11-sa pode não ter um PAM ideal, reduzindo assim sua afinidade de ligação e atividade subsequente. Além disso, o arranjo espacial das duas variantes da proteína de fusão dCas9 pode influenciar a arquitetura e acessibilidade da cromatina34,35, potencialmente restringindo o recrutamento da maquinaria transcricional ou fatores regulatórios associados na configuração XLOC-sp-RP11-sa. Esses achados sugerem que o posicionamento relativo de SaCas9 e SpCas9 dentro de um determinado contexto genômico pode impactar criticamente sua eficácia funcional, um aspecto que justifica uma investigação mais aprofundada para estratégias otimizadas de modulação baseadas em CRISPR.
O protocolo desenvolvido é adaptável para direcionar ou rastrear outros pares de RNA não codificantes letais sintéticos, modificando o vetor gRNA duplo. Uma vez que uma linha celular de interesse está expressando de forma estável ambas as variantes dCas9, uma variedade de abordagens se torna viável. Embora RNAi, ASOs e shRNAs possam ser empregados para silenciamento de genes, o CRISPRi oferece uma metodologia de triagem mais direta, e sua combinação com pequenos compostos químicos ou inibidores também é concebível. Ao substituir o vetor de gRNA duplo por uma biblioteca de gRNA dupla, este protocolo permite a triagem sistemática de pares letais sintéticos ou outras dependências genéticas em células cancerígenas, aumentando assim a precisão do direcionamento de células cancerígenas.
Embora tenhamos alcançado a integração das variantes dCas9-KRAB-MeCP2 por meio da transdução lentiviral, métodos alternativos de transfecção estável também podem ser usados para incorporar as enzimas desejadas nas células 501-mel. Isso é particularmente relevante, pois, em nossa experiência, o silenciamento de grandes construtos como dCas9-KRAB-MeCP2 pode ocorrer ao longo do tempo36,37. Alternativamente, dCas9-KRAB mostrou-se uma alternativa menor23. Em nossa experiência, a transdução com as proteínas de fusão dCas9-KRAB menores derivadas de ortólogos Cas9 levou à redução da expressão da proteína durante o cultivo prolongado (ver Figura 2 suplementar). Portanto, a expressão proteica consistente deve ser monitorada regularmente em pontos de tempo recorrentes. Além disso, a transdução de duas grandes proteínas de fusão junto com marcadores de seleção e duplos de expressão de gRNA pode induzir estresse celular ou atingir capacidades de empacotamento lentiviral.
Alcançar 100% de eficiência de knockdown em CRISPRi duplo pode ser um desafio devido ao design de gRNA abaixo do ideal ou à expressão insuficiente de dCas9. Para resolver isso, vários gRNAs por alvo devem ser testados usando ferramentas avançadas de design para identificar as sequências mais eficazes. Além disso, otimizar a expressão de dCas9-KRAB-MeCP2 por meio de um design de vetor aprimorado e caracterização de linhagens celulares clonais pode aumentar a eficiência do knockdown.
Estratégias alternativas para integrar o sistema CRISPRi incluem o uso de vetores de transposase ou integração direcionada por CRISPR para obter inserção estável em locais genômicos de porto seguro, que são conhecidos por manter a expressão estável ao longo do tempo38. No entanto, muitos tipos de células, incluindo células de melanoma, são difíceis de transfectar39, tornando a transdução lentiviral o método preferido. A eletroporação de enzimas baseadas em transposase ou Cas pode oferecer um meio alternativo para integrar as construções dCas9-KRAB-MeCP2 em células de melanoma40. A implementação de um sistema de nocaute induzível seria vantajosa para ativar a expressão de dCas9-KRAB-MeCP2 conforme necessário, em vez da expressão constitutiva para minimizar o estresse celular.
Finalmente, a abordagem desenvolvida não se limita às células de melanoma, mas pode ser estendida a qualquer tipo de célula onde a letalidade sintética esteja sendo investigada. Essa abordagem deve ser explicitamente ampliada para a modalidade de triagem da biblioteca de gRNA. No entanto, devido ao aumento da complexidade combinatória, por exemplo, com mais de três gRNAs por par de genes, incluindo controles e pelo menos 300 x de cobertura, apenas uma fração subtranscriptômica de lncRNAs expressos pode ser direcionada na prática. Portanto, os usuários devem pré-selecionar cuidadosamente os candidatos, considerando fatores como os níveis de expressão de lncRNA e sua relevância conhecida em contextos celulares específicos antes de prosseguir com a triagem da biblioteca. No entanto, a versatilidade combinatória dessa abordagem a torna uma ferramenta valiosa para investigar interações letais sintéticas e outras dependências genéticas de lncRNAs em vários tipos de câncer e potencialmente outras doenças. Isso expande o kit de ferramentas atualmente disponível para estudar essas interações de rede nas células.
Divulgações
Jochen Imig é atualmente financiado pela CGCIII pela Pfizer Inc. A CGCIII é patrocinada pela Pfizer Inc., Merck KGaA e AstraZeneca PLC. Os patrocinadores não tiveram nenhum papel na concepção, execução, interpretação ou redação do estudo. Todos os autores não têm conflitos de interesse a divulgar.
Agradecimentos
Agradecemos a Stefan Raunser (Instituto Max Planck de Fisiologia Molecular, Dortmund) pelo acesso ao espaço do laboratório de Nível 2 de Biossegurança. Agradecimentos especiais a Eric Wang por suas contribuições intelectuais e a todos os membros do passado e do presente do laboratório Imig por suas valiosas discussões. Jochen Imig é atualmente financiado pela CGCIII pela Pfizer Inc. no CGC III.
Materiais
| Name | Company | Catalog Number | Comments |
| Amicon Ultra Centrifugal Filter, 100 kDa MWCO | Millipore | UFC910024 | |
| anti-Cas9 (S. aureus) (6H4) mouse monoclonal antibody | Cell Signaling Technology | 48989 | |
| anti-Cas9 (S. pyogenes) (7A9-3A3) monoclonal antibody | Cell Signaling Technology | 14697 | |
| anti-GAPDH mouse monoclonal antibody | Sigma Aldrich | G8795 | |
| Anti-Mouse IgG (whole molecule)–Peroxidase antibody produced in rabbit | Sigma Aldrich | A9044 | |
| Bio-Rad ChemiDoc MP Imaging System | Bio-Rad | ||
| Blasticidine S hydrochloride | Sigma Aldrich | 15205-25MG | |
| CellTiter-Glo Luminescent Cell Viability Assay | Promega | G7570 | |
| Centrifuge | Eppendorf | 5804R | |
| Centrifuge | Eppendorf | 5415R | |
| CFX Connect Real-Time PCR Detection System | Bio-Rad | ||
| Clarity Western ECL Substrate, 500 mL | Bio-Rad | 1705061 | |
| CO2 Incubator Model CB 170 | Binder | ||
| cOmplete Protease Inhibitor Cocktail | Roche | 11697498001 | |
| Esp3I (BsmB1) restriction enzyme | Thermo Scientific | ER0451 | |
| Falcon 10 mL Serological Pipet | Corning | 356551 | |
| Falcon 25 mL Serological Pipet | Corning | 357525 | |
| Falcon 5 mL Serological Pipet | Corning | 356543 | |
| Fetal Bovine Serum | Sigma Aldrich | F9665-500ML | |
| Gibco Dulbecco’s modified Eagle’s medium (DMEM), high glucose, pyruvate | Gibco | 41966029 | |
| Human melanoma cell line 501-mel | was kindly provided by the Aifantis Lab (New York University) | RRID: CVCL_4633 | |
| Immobilon -P PVDF Membrane | Millipore | IPVH00010 | |
| Lenti-X 293T HEK cell line | Takara Bio | 632180 | |
| Mini Trans-Blot Cell system | Bio-Rad | ||
| Mini-PROTEAN Tetra Handcast System | Bio-Rad | ||
| NEBuilder HiFi DNA Assembly Reaction | New England Biolabs | E2621 | |
| Non-fat milk powder | Biomol | 54650 | |
| NuPAGE LDS Sample Buffer (4x) | Invitrogen | NP0008 | |
| One Shot Stbl Chemically Competent E. coli | Invitrogen | C737303 | |
| Opti-MEM I Reduced Serum Medium | Gibco | 31985062 | |
| PCR Tubes 0.5 ml (Flat Cap) | VWR International | 732-3207 | |
| Phosphate-Buffered Saline (PBS) | Corning | 45000-446 | |
| Phusion High-Fidelity PCR Master Mix with HF Buffer | Thermo Scientific | F531L | |
| Pierce BCA Protein Assay Kit | Thermo Scientific | 23225 | |
| Polybrene | Sigma Aldrich | TR-1003-G | |
| Polyethylenimine, branched | Sigma Aldrich | 408727 | |
| Puromycin dihydrochloride | Santa Cruz Biotechnology | sc-108071A | |
| QIAquick PCR Purification Kit | Qiagen | 28104 | |
| SafeSeal Microcentrifuge Tube 1.5 mL | Sarstedt | 72,706 | |
| Sodium chloride, 5 M Aqua Solution, RNase Free | Alfa Aesar | J60434.AE | |
| Sodium Dodecyl Sulfate (SDS) | Sigma Aldrich | L3771 | |
| Syringe Filter PES 33mm 0.2 μM | Fisher Scientific | 15206869 | |
| TC Dish 100, Standard | Sarstedt | 8,33,902 | |
| TC Plate 6 Well, Standard, F | Sarstedt | 83,39,20,005 | |
| Tris base | Roche | 10708976001 | |
| TWEEN 20 | Sigma Aldrich | P9416-50ML |
Referências
- Lander, E. S., et al. Initial sequencing and analysis of the human genome. Nature. 409 (6822), 860-921 (2001).
- ENCODE Project Consortium. An integrated encyclopedia of DNA elements in the human genome. Nature. 489 (7414), 57-74 (2012).
- Szcześniak, M. W., Wanowska, E., Mukherjee, N., Ohler, U., Makałowska, I. Towards a deeper annotation of human lncRNAs. Biochim Biophys Acta Gene Regul Mech. 1863 (4), 194385(2020).
- Fang, S., et al. NONCODEV5: a comprehensive annotation database for long non-coding RNAs. Nucl Acids Res. 46 (D1), D308-D314 (2018).
- Lin, J., et al. Human-specific lncRNAs contributed critically to human evolution by distinctly regulating gene expression. eLife. 12, RP89001.2(2023).
- Sarropoulos, I., Marin, R., Cardoso-Moreira, M., Kaessmann, H. Developmental dynamics of lncRNAs across mammalian organs and species. Nature. 571 (7766), 510-514 (2019).
- Melé, M., et al. Human genomics. The human transcriptome across tissues and individuals. Science. 348 (6235), 660-665 (2015).
- Cabili, M. N., et al. Integrative annotation of human large intergenic noncoding RNAs reveals global properties and specific subclasses. Genes Dev. 25 (18), 1915-1927 (2011).
- Liu, S. J., Dang, H. X., Lim, D. A., Feng, F. Y., Maher, C. A. Long noncoding RNAs in cancer metastasis. Nat Rev Cancer. 21 (7), 446-460 (2021).
- Ahmad, M., Weiswald, L. B., Poulain, L., Denoyelle, C., Meryet-Figuiere, M. Involvement of lncRNAs in cancer cells migration, invasion and metastasis: cytoskeleton and ECM crosstalk. J Exp Clin Cancer Res. 42 (1), 173(2023).
- Jain, A. K., et al. LncPRESS1 Is a p53-Regulated LncRNA that Safeguards Pluripotency by Disrupting SIRT6-Mediated De-acetylation of Histone H3K56. Mol Cell. 64 (5), 967-981 (2016).
- Bergmann, J. H., et al. Regulation of the ESC transcriptome by nuclear long noncoding RNAs. Genome Res. 25 (9), 1336-1346 (2015).
- Kretz, M., et al. Control of somatic tissue differentiation by the long non-coding RNA TINCR. Nature. 493 (7431), 231-235 (2013).
- Yoon, J. H., et al. LincRNA-p21 suppresses target mRNA translation. Mol Cell. 47 (4), 648-655 (2012).
- Huarte, M. The emerging role of lncRNAs in cancer. Nat Med. 21 (11), 1253-1261 (2015).
- Badowski, C., He, B., Garmire, L. X. Blood-derived lncRNAs as biomarkers for cancer diagnosis: the Good, the Bad and the Beauty. NPJ Precis Oncol. 6 (1), 40(2022).
- Zong, X., et al. Knockdown of nuclear-retained long noncoding RNAs using modified DNA antisense oligonucleotides. Meth Mol Biol. 1262, 321-331 (2015).
- Gagnon, K. T., Li, L., Chu, Y., Janowski, B. A., Corey, D. R. RNAi factors are present and active in human cell nuclei. Cell Rep. 6 (1), 211-221 (2014).
- Zeng, Y., Cullen, B. R. RNA interference in human cells is restricted to the cytoplasm. RNA. 8 (7), 855-860 (2002).
- Yang, J., et al. CRISPR/Cas9-mediated noncoding RNA editing in human cancers. RNA Biol. 15 (1), 35-43 (2018).
- Liu, S. J., et al. CRISPRi-based genome-scale identification of functional long noncoding RNA loci in human cells. Science. 355 (6320), aah7111(2017).
- Tsung, K., et al. CRISPRi screen of long non-coding RNAs identifies LINC03045 regulating glioblastoma invasion. PLoS Genet. 20 (6), e1011314(2024).
- Gilbert, L. A., et al. CRISPR-mediated modular RNA-guided regulation of transcription in eukaryotes. Cell. 154 (2), 442-451 (2013).
- Boettcher, M., McManus, M. T. Choosing the right tool for the job: RNAi, TALEN, or CRISPR. Mol Cell. 58 (4), 575-585 (2015).
- Yeo, N. C., et al. An enhanced CRISPR repressor for targeted mammalian gene regulation. Nat Meth. 15 (8), 611-616 (2018).
- Parrish, P. C. R., et al. Discovery of synthetic lethal and tumor suppressor paralog pairs in the human genome. Cell Rep. 36 (9), 109597(2021).
- Najm, F. J., et al. Orthologous CRISPR-Cas9 enzymes for combinatorial genetic screens. Nat Biotechnol. 36 (2), 179-189 (2018).
- Petroulia, S., et al. CRISPR-inhibition screen for lncRNAs linked to melanoma growth and metastasis. bioRxiv. , (2024).
- Hsu, P. D., et al. DNA targeting specificity of RNA-guided Cas9 nucleases. Nat Biotechnol. 31 (9), 827-832 (2013).
- New England Biolabs. NEBuilder HiFi DNA Assembly Reaction (E2621) v2. , (2020).
- Cortés-Ríos, J., et al. Protein quantification by bicinchoninic acid (BCA) assay follows complex kinetics and can be performed at short incubation times. Anal Biochem. 608, 113904(2020).
- Junior, N. Polyacrylamide Gel Electrophoresis (SDS-PAGE) v1. , (2019).
- Chen, X., Liu, J., Janssen, J. M., Gonçalves, M. A. F. V. The chromatin structure differentially impacts high-specificity CRISPR-Cas9 Nuclease strategies. Mol Ther Nucl Acids. 8, 558-563 (2017).
- Schep, R., et al. Impact of chromatin context on Cas9-induced DNA double-strand break repair pathway balance. Mol Cell. 81 (10), 2216-2230.e10 (2021).
- Daer, R. M., Cutts, J. P., Brafman, D. A., Haynes, K. A. The impact of chromatin dynamics on Cas9-Mediated genome editing in human cells. ACS Syn Biol. 6 (3), 428-438 (2017).
- Chavez, M., Rane, D. A., Chen, X., Qi, L. S. Stable expression of large transgenes via the knock-in of an integrase-deficient lentivirus. Nat Biomed Eng. 7 (5), 661-671 (2023).
- Ellis, J. Silencing and variegation of gammaretrovirus and lentivirus vectors. Human Gene Ther. 16 (11), 1241-1246 (2005).
- Shrestha, D., et al. Genomics and epigenetics guided identification of tissue-specific genomic safe harbors. Genome Biol. 23 (1), 199(2022).
- Chu, Z., et al. Enhanced gene transfection and induction of apoptosis in melanoma cells by branched poly(β-amino ester)s with uniformly distributed branching units. J Control Release. 367, 197-208 (2024).
- Han, S. Y., et al. Nucleofection is a highly effective gene transfer techfnique for human melanoma cell lines. Exp Dermatol. 17 (5), 405-411 (2008).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados