Method Article
Monitoramento da dinâmica conformacional de proteínas únicas não modificadas usando pinças plasmônicas
Neste Artigo
Resumo
As pinças plasmônicas usam ressonância plasmônica de superfície localizada em nanoestruturas de ouro para capturar nanopartículas únicas, incluindo proteínas, dentro de um campo óptico em escala nanométrica. Mudanças no sinal espalhado revelam a presença de proteínas e dinâmica conformacional, permitindo o monitoramento sem modificações de fluoróforos ou amarração de superfície.
Resumo
As técnicas atuais de molécula única para caracterizar proteínas normalmente requerem rótulos, amarras ou o uso de condições de solução não nativas. Tais mudanças podem alterar a biofísica das proteínas e reduzir a utilidade dos dados adquiridos. As pinças plasmônicas são uma técnica que usa ressonância plasmônica de superfície localizada (LSPR) em nanoestruturas de ouro para aumentar o campo elétrico dentro de uma região de hotspot confinada. Esse aprimoramento de campo permite o uso de baixas potências de laser para capturar nanopartículas únicas muito menores do que as pinças ópticas convencionais, com apenas alguns nanômetros de diâmetro, como proteínas individuais. O aprisionamento de moléculas de proteína únicas dentro da região do hotspot induz uma mudança no índice de refração local (proteína n > nágua), alterando o espalhamento de luz como um produto da polarizabilidade da molécula, que é afetada por seu volume, forma anisotropia e índice de refração. Um fotodiodo de avalanche (APD) coleta as mudanças subsequentes no espalhamento da luz. Essas alterações podem então ser analisadas para determinar mudanças na molécula aprisionada, incluindo seu tamanho, conformação global e dinâmica da mudança conformacional ao longo do tempo. A incorporação de microfluídica dentro do sistema permite mudanças ambientais controladas e monitoramento em tempo real de seus efeitos subsequentes na molécula. Neste protocolo, demonstramos as etapas para capturar moléculas de proteína únicas, alterar suas condições de solução ambiental e monitorar suas mudanças conformacionais correspondentes usando um sistema de pinças plasmônicas.
Introdução
As técnicas atuais de molécula única para interrogar a dinâmica conformacional de proteínas incluem métodos baseados em rotulagem, como transferência de energia de ressonância de fluorescência (FRET) 1 , 2 , abordagens baseadas em amarração, como pinças ópticas3 , 4 e microscopia de força atômica (AFM ) 5, técnicas baseadas em interferência, como microscopia de espalhamento de interferência (iSCAT) 6 ou técnicas baseadas em nanofluídicas, como nanoporos7,8,9. Embora esses métodos tenham muitas vantagens; Algumas limitações importantes os impedem de fornecer dados sobre a dinâmica conformacional de proteínas não modificadas. FRET e pinças ópticas requerem marcação de fluoróforo ou amarração a uma superfície, o que pode afetar as propriedades biofísicas das proteínas 10,11,12. O iSCAT, embora tecnicamente livre de rótulos, também requer interação entre a proteína e uma superfície para observar a interferência gerada entre os dois, o que potencialmente afeta as propriedades das proteínas. Além disso, limitado por sua relação sinal-ruído, o iSCAT só pode detectar proteínas >40 kDa devido ao ruído do equipamento e flutuações de fundo semelhantes a manchas13. Embora esse limite de tamanho possa ser aliviado por meio do aprendizado de máquina, os componentes do buffer são limitados, pois podem afetar as propriedades ópticas, resultando em dados ruidosos13,14. Os nanoporos apresentam tempos de translocação rápidos de proteínas através do poro (geralmente dentro de 5 μs), tornando-os incapazes de detectar dinâmicas conformacionais mais lentas15,16, embora pesquisas para aliviar essas limitações, como o uso de origami de DNA em uma armadilha eletro-osmótica de nanoporos17 ou incorporação de plasmônica18,19,20,21. Além disso, altas concentrações de sal, normalmente em torno de 1 M, podem reduzir a aplicabilidade dos dados para o trabalho in vivo 15,22. A técnica ideal de molécula única para caracterização de proteínas deve monitorar proteínas em tempo real e capturar a dinâmica conformacional por períodos mais longos (ou seja, milissegundos) sem a necessidade de modificações nas condições de proteína ou solução não nativa.
As pinças plasmônicas são semelhantes às pinças ópticas convencionais, no sentido de que usam a luz para prender a matéria. As pinças plasmônicas, no entanto, utilizam ressonância plasmônica de superfície localizada (LSPR) para aumentar o campo elétrico em várias ordens de magnitude para gerar uma força de gradiente forte o suficiente para prender nanopartículas únicas23. Além disso, a partícula aprisionada desempenha um papel ativo no aumento da força da armadilha, conhecida como aprisionamento de ação reversa auto-induzida (SIBA) para estruturas de nanoabertura24. Esse aprisionamento SIBA permite que baixas potências de laser (ou seja, miliwatts) prendam pequenas partículas de até apenas alguns nanômetros de diâmetro, como proteínas 25,26,27. A captura de moléculas de proteína única dentro da região do hotspot causa uma mudança no índice de refração local (proteína n > nágua), alterando o espalhamento de luz com base na polarizabilidade da molécula, que é influenciada pelo volume da proteína, anisotropia de forma e índice de refração28. Um fotodiodo de avalanche (APD) detecta essas informações para monitorar as mudanças subsequentes no espalhamento de luz. Além disso, as pinças plasmônicas permitem o monitoramento das proteínas aprisionadas em tempo real sem marcação, amarras e condições adversas de solução por longos períodos de tempo (ou seja, minutos a horas) 29 , preenchendo os critérios para uma técnica ideal de molécula única para proteínas. Usando uma estrutura de nanoburaco duplo (DNH), as pinças plasmônicas demonstraram sua capacidade de capturar várias proteínas e elucidar informações importantes delas, incluindo transições conformacionais29, cinética de desmontagem30, paisagens de energia31, rastreamento de difusão32 e ligação de ligante33,34. Além das estruturas DNH, geometrias de estrutura alternativas foram demonstradas para capturar partículas com pequenos tamanhos de partícula35,36. Neste protocolo, são apresentadas as etapas fundamentais para configurar e executar uma configuração de pinças plasmônicas com um sistema microfluídico integrado. Esperamos que este protocolo ajude a aumentar a acessibilidade e a compreensão das pinças plasmônicas para pesquisadores, particularmente aqueles nas áreas de biologia estrutural e biofísica.
Protocolo
CUIDADO: Leia todas as fichas de dados de segurança (SDS) relevantes para todos os produtos químicos usados e siga todas as práticas de segurança apropriadas e use equipamentos de proteção individual (óculos de segurança a laser, jalecos, luvas) conforme necessário.
1. Construindo a configuração das pinças plasmônicas
NOTA: A configuração óptica é baseada no kit Modular Optical Tweezers System (OTKB) utilizando um laser e APD diferentes (consulte a Tabela de Materiais). Use apenas equipamento óptico em uma mesa óptica adequada para reduzir o impacto de vibrações externas no sistema. O laser no kit era de 976 nm, mas como o comprimento de onda de ressonância de pico para a ressonância em cunha das estruturas DNH é de cerca de 740-760 nm33. Escolhemos um laser NIR (852 nm) por estar próximo ao pico de ressonância, induzir LSPR e também ter uma melhor taxa de rendimento de detecção pelo APD à base de silício. Lasers comcomprimentos de onda 20 ou18 mais longos têm sido usados para capturar biomoléculas.
- Configure o laser no suporte de laser de fotodiodo e alimente-o em um colimador, o que leva a um feixe de laser colimado com largura de 1,7 mm para nossa configuração.
- Adicione uma placa de meia onda ao caminho da luz para ajustar a polarização. Ajuste usando um polarizador Glan-Taylor para garantir que a polarização vertical (polarização S) tenha a maior intensidade de luz.
NOTA: Usar a polarização correta é vital para garantir o máximo aprimoramento do campo elétrico das estruturas DNH, pois elas dependem da polarização. - Foque a luz através de uma configuração de expansor de feixe que consiste em uma lente plano-côncava (f = -50 mm) seguida por uma lente plano-convexa (f = 150 mm) para aumentar a largura do feixe para cerca de 5 mm para preencher a abertura traseira total da objetiva inferior.
- Use um espelho dicróico (passagem curta de 805 nm) para refletir o laser para o local desejado. Configure a câmera (CCD) atrás dela.
- Redirecione a luz para a objetiva inferior (100x/1.25 NA) e posicione a objetiva superior (4x/0.1 NA) até a distância confocal para coletar a luz transmitida.
- Adicione outro espelho dicróico (passagem curta de 805 nm) para refletir a luz do laser em direção ao APD através de uma lente plano-convexa posicionada em sua distância focal para focar a luz no APD. Adicione a fonte de luz branca (WLS) atrás do espelho dicróico para que ela possa passar de volta pela configuração para alcançar a câmera. A Figura 1 mostra um esquema da configuração das pinças plasmônicas totalmente montadas.
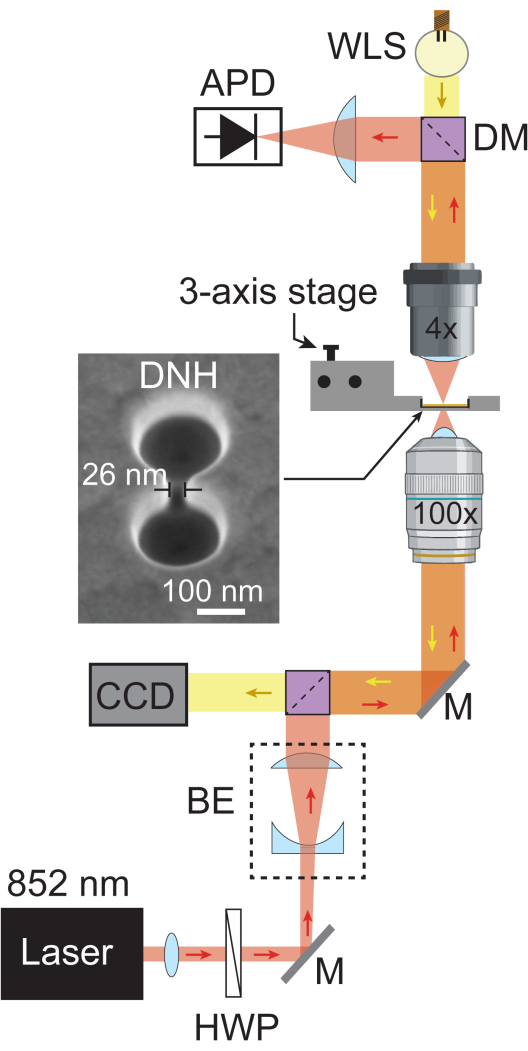
Figura 1: Configuração de pinças plasmônicas. O esquema mostra o caminho óptico completo da configuração das pinças plasmônicas. Um feixe de laser de 852 nm (vermelho) passa por um colimador e uma placa de meia onda, depois é expandido pelo expansor de feixe (BE) para ~ 5,1 mm. Posteriormente, é focado na amostra usando uma objetiva de 100x. A luz laser transmitida é coletada por uma objetiva de 4x e registrada por um APD a uma taxa de amostragem de 1 MHz. A luz branca do WLS (amarelo) passa pela objetiva de 4x, amostra e objetiva de 100x antes de atingir o CCD, que fornece uma imagem de campo brilhante da amostra. As áreas onde ambos os caminhos de luz se cruzam são representadas em laranja. Imagem SEM de DNH tirada com inclinação de 20°. Abreviaturas: BE = Expansor de feixe, CCD = Dispositivo de carga acoplada, DM = Espelho dicróico, HWP = Placa de meia onda, M = Espelho prateado e WLS = Fonte de luz branca. Clique aqui para ver uma versão maior desta figura.
2. Fabricando as estruturas DNH
- Depositar 5 nm de Ti seguido de 100 nm de Au em uma bolacha de sílica fundida de 550 μm de espessura usando evaporação por feixe eletrônico, conforme descrito anteriormente 29,33. Corte o wafer em cubos em amostras de 1 cm x 1 cm prontas para uso.
NOTA: A evaporação do feixe E foi escolhida porque produz uma camada de ouro com baixa rugosidade superficial, produzindo estruturas DNH de alta qualidade com alta eficiência de aprisionamento37. - Pegue a amostra de filme de ouro em branco e monte-a em uma configuração de feixe de íons focado em microscópio eletrônico de varredura (SEM-FIB) à temperatura ambiente usando uma fonte de íons de gálio.
- Certifique-se de que as nanoaberturas FIB estejam alinhadas com o SEM para produzir DNH em posições precisas.
- Crie uma caixa de marcação usando um feixe de íons de alta corrente (por exemplo, 100 pA). Este marcador servirá como um guia de referência para localizar a posição das estruturas DNH sob a configuração óptica.
- Para criar a estrutura DNH, dois círculos com uma distância de centro a centro de 200 nm e 160 nm de diâmetro são ligados por um retângulo (3 nm x 40 nm). Para um tamanho de lacuna de cerca de 20-30 nm, feche os círculos com um fator de dose de 0,09, enquanto a dose retangular está entre 0,300 a 0,350 usando uma energia de sonda de 30 kV e corrente de feixe de 1 pA.
NOTA: O SEM-FIB usado tem uma resolução de 3 nm nesta energia de sonda, permitindo a fabricação confiável de DNH com tamanhos de folga entre 20-30 nm. Não usaríamos uma resolução mais baixa, pois isso produzirá tamanhos de lacuna maiores, reduzindo a eficiência de captura do DNH. Se um FIB for inviável, um método alternativo para produzir estruturas DNH é usar litografia de nanoesferas de poliestireno38. Devido à alta precisão necessária para fabricar estruturas DNH, a mesma receita pode não produzir o mesmo tamanho de lacuna entre os usos de SEM-FIB. Altere a dose e a altura do retângulo (dose de 0,280 a 0,350 e altura de 2 a 4 nm) e meça o tamanho da lacuna, visando cerca de 20 nm.
3. Revestimento de amostras DNH
- Use um recipiente resistente a solventes, como um prato cristalizador feito de vidro borossilicato 3.3, e adicione 20 mL de etanol.
CUIDADO: O etanol é altamente inflamável e irritante. Use apenas em uma hotte. Armazene em local fresco e bem ventilado. Manuseie com luvas e jaleco de laboratório. - Pese 32 mg de poli (etilenoglicol) metil éter tiol (PEG-tiol; peso molecular médio 800 g / mol) e misture-o ao etanol, garantindo que todo o PEG-tiol tenha se dissolvido para produzir uma solução com uma concentração de cerca de 2 mM para maximizar a densidade da monocamada39.
CUIDADO: PEG-tiol é um irritante; manuseie-o com EPI apropriado. - Adicione os chips de amostra contendo as nanoestruturas à mistura usando uma pinça reta, cubra e incube durante a noite (~ 16 h) à temperatura ambiente para que o PEG-tiol forme uma monocamada auto-montada na superfície do ouro.
- Após a incubação, lavar as amostras segurando-as com uma pinça recta sobre um recipiente adequado na hotte. Use uma garrafa de esguicho para borrifar etanol em cada lado completamente. Seque completamente com uma pistola de ar antes do armazenamento ou montagem subsequente nas pinças plasmônicas.
NOTA: As amostras podem ser armazenadas a 4 °C para melhorar a longevidade, pois podem se degradar com o tempo.
4. Montagem da amostra revestida com PEG em uma célula de fluxo
- Coloque a amostra revestida na célula de fluxo impressa em 3D (impressa por uma impressora Form 2 com resina Clear V4) usando uma pinça reta com a camada de ouro voltada para cima (consulte a Figura 1 suplementar para obter os principais parâmetros e valores em nosso projeto de célula de fluxo).
- Descasque um lado da tampa da fita dupla face de plástico PET transparente usando uma pinça reta e coloque-a na amostra e na célula de fluxo, garantindo que as nanoestruturas e os orifícios de entrada/saída na célula de fluxo permaneçam descobertos. Pressione suavemente as bordas da fita com uma pinça arredondada para garantir que ela tenha aderido adequadamente à célula de fluxo e à amostra.
- Retire o outro lado da fita e coloque delicadamente uma lamínula de vidro (espessura de 0,17 mm) sobre a amostra usando uma pinça arredondada. Pressione suavemente as bordas da lamínula com a pinça arredondada para garantir que ela tenha aderido corretamente. Isso cria um canal de líquido (altura = 50 μm, volume = 3,5 μL) dentro da célula de fluxo.
NOTA: Não pressione a lamínula no local ou próximo ao local onde as nanoestruturas estão localizadas, pois podem ser danificadas. Se a lamínula estiver suja, lave-a com etanol e seque-a com uma pistola de ar antes de montá-la. - Misture partes iguais de A e B das soluções de silicone duplicadas na proporção de 1:1 (ou de acordo com as instruções do fabricante) em uma lâmina de microscópio usando uma pequena ponta de pipeta.
- Preencha as lacunas entre a lamínula e a célula de fluxo com o silicone de duplicação misto, empurrando-o suavemente sob a lamínula. Segure a célula de fluxo de cabeça para baixo e coloque cuidadosamente o silicone duplicado ao redor da parede interna do orifício antes de usar a ponta da pipeta para movê-la suavemente para as bordas visíveis da parte inferior da sílica fundida da amostra (consulte a Figura Suplementar 2).
- Deixe secar com a camada de ouro voltada para cima até que o silicone duplicador esteja totalmente endurecido, de acordo com as instruções do fabricante.
NOTA: Tome cuidado para não permitir que o silicone cubra as nanoestruturas quando aplicado na parte inferior da amostra. Tome cuidado para não forçar o silicone duplicado sob a lamínula, pois ele pode entrar nos orifícios de entrada/saída da célula de fluxo ou nas nanoestruturas, que podem ser difíceis de limpar (Figura 3 suplementar).
5. Conectando o sistema microfluídico
NOTA: Certifique-se de usar tubos limpos para o sistema. Aqui, tubos de PTFE de diâmetro interno pequeno: 0,18 mm de diâmetro interno e tubos de PTFE de diâmetro interno grande: 0,8 mm serão usados, mas outros valores de diâmetro interno funcionarão.
- Configure a bomba de seringa e conecte-a à válvula solenóide de 3/2 vias usando o tubo grande de PTFE. Conecte uma porta da válvula ao recipiente tampão e a outra a uma bobina de retenção, com cerca de 1 mL de volume, feita de um grande tubo de identificação para evitar o refluxo para a seringa.
NOTA: A válvula 3/2 pode ser controlada através de um controlador de válvula, que determina se a solução é infundida/retirada do recipiente tampão ou da bobina de retenção. - Conecte a bobina de retenção à válvula central da válvula microfluídica bidirecional rotativa 12/1. Conecte recipientes para soluções usando tubos de identificação pequenos para amostras escassas ou valiosas e tubos de identificação grandes para o resto. Use uma válvula apenas para infundir em um recipiente de resíduos e outra apenas para infundir na célula de fluxo; use tubos pequenos de identificação para a válvula de infusão de célula de fluxo.
- Conecte a tubulação de diâmetro interno grande da saída da célula de fluxo ao recipiente de resíduos. A Figura 2 mostra um esquema do sistema microfluídico totalmente conectado.
NOTA: A tubulação de PTFE de ID pequena é preferível para a entrada da célula de fluxo e para válvulas que serão usadas para infusão de proteínas devido ao menor volume onde a amostra permanecerá na tubulação. A tubulação de PTFE de diâmetro interno grande é preferível para materiais mais baratos ou mais abundantes, como tampões. Tente usar tubos mínimos para reduzir ainda mais esse volume morto.
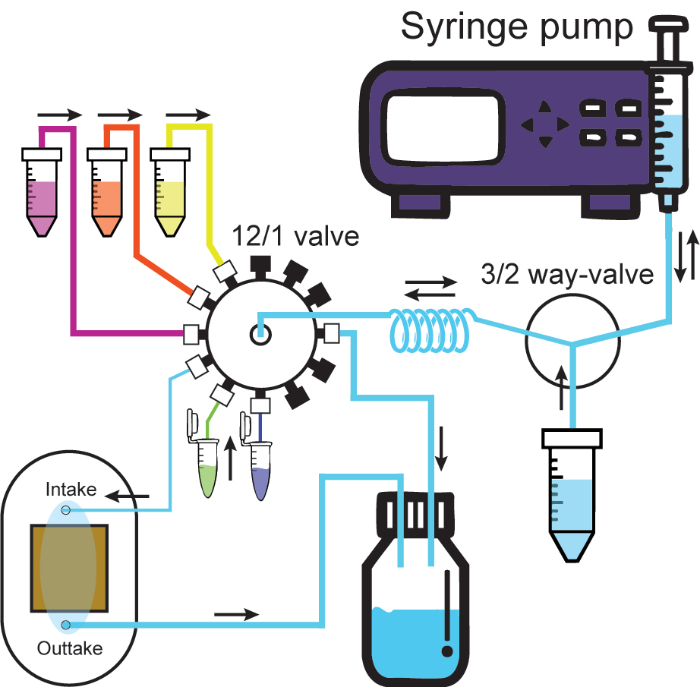
Figura 2: Sistema microfluídico. O esquema ilustra o sistema microfluídico. A bomba de seringa infunde ou retira soluções através de uma porta da válvula de 3/2 vias, seja o recipiente da solução ou a bobina de retenção. As soluções conectadas à válvula 12/1 sempre passam pela bobina de retenção quando retiradas e podem então ser infundidas pelo canal desejado. A infusão no canal de entrada conectado à célula de fluxo empurrará a solução da célula de fluxo para o recipiente de resíduos. Tubos grossos e finos representam tubos de identificação grandes e pequenos do protocolo. As tampas pretas na válvula 12/1 representam canais selados. As direções do fluxo são rotuladas por setas pretas. Clique aqui para ver uma versão maior desta figura.
6. Preparando o sistema microfluídico
- Carregue a interface do usuário do sistema microfluídico no PC e verifique se os componentes estão conectados corretamente. Selecione o ícone de reprodução ao lado do fio e do distribuidor para abrir suas respectivas interfaces de usuário. Para contornar a válvula de 3/2 vias, gire a porta 1 no fio.
- Certifique-se de que as válvulas que serão usadas no sistema sejam limpas por infusão de isopropanol (IPA) e, em seguida, lave a tubulação várias vezes com tampão(s) de escolha ou água destilada para remover o ar e o IPA dentro do sistema. A infusão pode ser feita selecionando a válvula desejada na interface do usuário do distribuidor e acionando a bomba de seringa, selecionando infundir/retirar e escolhendo o volume e a vazão desejados (por exemplo, volume de 0.5 mL e vazão de 0.2 mL/min).
NOTA: Não infunda IPA nas células de fluxo, pois isso degradará a célula e a cola, o que pode causar vazamentos.
7. Montagem da célula de fluxo em pinças plasmônicas e verificação de vazamentos
- Prenda os tubos de entrada e saída à parte apropriada da célula de fluxo antes de colocá-los em um tecido limpo com uma camada de ouro voltada para cima.
- Infundir tampão na célula de fluxo com uma alta taxa de fluxo (~ 0,3 mL / min) e verifique se o fluido se move através da amostra na célula de fluxo e se nenhum fluido é visível na parte externa ou inferior da célula de fluxo. Taxas de fluxo mais altas podem ser usadas, desde que não ocorra vazamento. Se a amostra vazar, desmonte e remonte.
- Adicione 1-2 gotas de óleo de imersão na objetiva 100x antes de colocar a célula de fluxo no estágio de pinça plasmônica com a camada de ouro voltada para baixo. Coloque clipes de metal sobre os ímãs da célula de fluxo e trave a platina para ajudar a mantê-la no lugar.
CUIDADO: O óleo objetivo é irritante, perigoso para a saúde e tóxico para a vida aquática, manuseie apenas com luvas.
8. Localizando nanoestruturas na amostra
- Ligue a fonte de luz branca. Abra o software da câmera e aumente o ganho e o tempo de exposição até que as nanoestruturas fiquem visíveis. Ligue o laser com potência de laser relativamente alta e ajuste o eixo z manualmente até que o ponto do laser esteja visível. Priorize o aumento do tempo de exposição e do ganho em vez de aumentar a potência do laser para encontrar o ponto do laser.
CUIDADO: Os lasers representam um sério risco potencial para os usuários. Certifique-se de que sejam usados EPI apropriados, como óculos de segurança a laser com densidade óptica suficiente dentro da faixa de comprimento de onda necessária. - Ligue o controlador piezoelétrico e selecione as configurações apropriadas para a porta COM e a tensão máxima. Defina os valores dos eixos x, y e z para metade da tensão máxima para permitir uma boa faixa para alinhar o stage em todas as direções.
NOTA: A porta COM do controlador piezoelétrico pode ser encontrada e alterada no gerenciador de dispositivos na guia portas. - Mova o ponto do laser para sobrepor com um dos DNH usando os botões de controle dos eixos x, y e z do estágio principal. Certifique-se de que o APD esteja ligado e, em seguida, feche suavemente o gabinete para as pinças plasmônicas.
NOTA: O uso de uma ferramenta de marcação no software da câmera, se disponível, ajudará a mover o ponto do laser sobre uma nanoestrutura. Defina o marcador no centro do ponto do laser e desligue o laser, mas deixe a fonte de luz branca ligada, para permitir um alinhamento mais fácil com uma nanoestrutura. O APD pode ser supersaturado e danificado se muita luz laser o atingir. Certifique-se de que a amostra esteja no caminho do laser e aumente gradualmente a potência do laser para garantir que a saturação não seja atingida. Filtros de densidade neutra (ND) podem ser colocados antes do APD para reduzir a luz que o atinge, se necessário.
9. Alinhamento ideal do laser com a nanoestrutura desejada
- Abra o software associado à gravação APD, como uma interface de usuário Labview caseira, e defina a frequência de corte para 1 kHz. Nomeie e defina o caminho do arquivo desejado e o formato de nomenclatura dos arquivos que serão salvos.
- Desligue a fonte de luz branca e ligue o laser novamente. Defina para uma potência de laser apropriada (por exemplo, ~ 20 mW) e use os controles piezoelétricos para ajustar os eixos x, y e z até que o sinal APD seja o mais alto possível, evitando a saturação de APD e com desvio padrão mínimo (SD) do traço.
NOTA: O APD provavelmente terá uma faixa de sensibilidade ideal para transmissão (consulte o manual do produto), ideal para ficar por perto, por exemplo, 1000-2000 mV. Filtros ND podem ser usados para manter a transmissão em torno dessa faixa. A principal consideração é que a transmissão não esteja próxima do ponto de saturação da APD, pois o aprisionamento pode aumentar a transmissão até o limite de saturação, levando à perda de dados.
10. Infusão de proteínas na célula de fluxo
- Desligue o laser para preservar a vida útil das nanoestruturas e opere a unidade de controle da bomba de seringa e a interface do usuário do distribuidor para ajustar a válvula e retirar a quantidade desejada de proteína (por exemplo, 30 μL). Para retirar usando o tubo de diâmetro interno pequeno, uma taxa de fluxo mais baixa ajuda a garantir que todas as soluções sejam retiradas (~ 0,01 - 0,1 mL / min). Taxas de fluxo mais altas podem ser usadas para reduzir o tempo gasto esperando, se desejado.
NOTA: O volume ideal de proteína a ser usado depende de quão valiosa é a proteína e do que o experimento espera alcançar. A concentração típica de proteína que usamos é de 1 μM e um volume de alíquota de 100 μL. Se a amostra de proteína for abundante, concentrações mais altas podem ser usadas para reduzir o tempo médio de captura. No entanto, se as concentrações forem muito altas, o risco de prender duas das mesmas proteínas dentro da nanoestrutura aumentará. Se o experimento exigir uma infusão de um ligante/proteína diferente após a captura da proteína inicial, um volume de alíquota menor pode ser preferido para minimizar o desperdício e o tempo necessário para infundir a próxima solução. Certifique-se de que a tubulação permaneça submersa em solução para evitar que o ar entre no sistema. Deve ter-se especial cuidado com as alíquotas proteicas se o tubo não atingir o fundo do recipiente ou se a solução for retirada em excesso. - Usando a interface do usuário microfluídica, infundir a solução proteica a uma taxa de fluxo semelhante à taxa de retirada (~ 0,01 - 0,1 mL / min). Deixe em infusão a essa taxa até que a solução de proteína atinja a célula de fluxo e, em seguida, reduza a taxa de fluxo para ≤0,001 mL / min. Verifique o volume e o tempo na bomba de seringa para certificar-se de que correspondem ao volume esperado (por exemplo, 1 min para 1 mL de volume a 1 mL/min).
NOTA: O volume necessário para isso pode ser calculado com base no volume da tubulação do sistema microfluídico para a célula de fluxo. Se o comprimento e o ID da tubulação forem conhecidos, uma calculadora online para o volume de um cilindro pode ser usada. A taxa de infusão pode ser ajustada conforme desejado, por exemplo, >0.001 mL/min para infusão de proteínas para reduzir o tempo médio de espera para uma armadilha. No entanto, recomenda-se cautela, pois se a taxa de fluxo for muito alta, isso pode impedir que as proteínas entrem na nanoestrutura.
11. Coleta de dados
- Enquanto a solução de proteína está sendo infundida na célula de fluxo, inicie o registro de dados do sinal APD. Ajuste os eixos x, y e z conforme necessário usando a interface do usuário do controlador piezoelétrico, pois o sistema provavelmente se desviará com o tempo. Os traços de captura ideais têm um padrão geral consistente seguindo o traço da Figura 3.
- Ao observar uma grande mudança na transmissão e S.D. semelhante ao traço de captura exemplar, anote o tempo em que isso ocorre para classificação de dados futura (consulte a Figura 4B , por exemplo.).
NOTA: A deriva no sistema pode levar a mudanças na transmissão e um aumento no SD, às vezes repentinamente, o que pode ser confundido com uma armadilha de proteína. Certifique-se de que o salto de sinal exiba o mesmo padrão que a armadilha exemplar e que nenhuma interferência externa esteja presente quando o salto ocorreu, como desvio de alinhamento ou ruídos altos captados pelo sistema. - Se a proteína precisar ser liberada como parte do experimento, desligue o laser por ~ 5 s e ligue-o novamente. O traço deve ter uma grande mudança na transmissão e um DP significativamente menor, indicando um retorno ao estado basal.
NOTA: Se uma grande mudança na transmissão e / ou S.D. não for observada, ou um traço semelhante à proteína que está sendo aprisionada for observado, a proteína provavelmente está presa à superfície da amostra (consulte a Figura 5B, por exemplo).
12. Desmontagem da amostra
- Depois de realizar o experimento desejado, desligue o laser, retire a célula de fluxo do estágio de 3 eixos e desconecte a tubulação do sistema microfluídico.
- Coloque a célula de fluxo no tecido limpo com a camada dourada da amostra voltada para cima. Usando um bisturi, corte cuidadosamente a cola sob a lamínula de vidro e retire-a suavemente com uma pinça arredondada. Descarte-o na lixeira de vidro quebrada designada.
CUIDADO: Vidros quebrados e o uso de objetos pontiagudos podem causar ferimentos. Certifique-se de que óculos de segurança, luvas e jaleco sejam usados. - Segure a célula de fluxo em um ângulo com a camada de ouro ainda voltada para cima e use uma pinça arredondada para remover cuidadosamente a cola na parte inferior da célula de fluxo para liberar a amostra. A duplicação de silicone permite fácil remoção sem danificar as estruturas DNH.
- Usando uma pinça reta, pegue a amostra e enxágue abundantemente com IPA, depois regue antes de secar com uma pistola de ar. Se reutilizável, armazene a amostra em um recipiente adequado com a camada de ouro voltada para cima.
NOTA: As amostras normalmente têm um período utilizável de cerca de 1-2 semanas se manuseadas corretamente, embora o tamanho das nanoestruturas mude com o tempo. Se estiver muito danificado, mude para uma nova amostra para o próximo experimento. Os danos podem ser na forma de arranhões presentes na superfície do ouro perto das nanoestruturas ou de baixo desempenho experimental, indicando degradação das nanoestruturas29.
13. Preparando o sistema para uso futuro
- Use lenço de papel dobrado para limpar o óleo da objetiva em uma direção, levantando-o e repetindo-o com outra parte limpa do tecido.
NOTA: As objetivas são propensas a manchas e arranhões, o que pode prejudicar significativamente o desempenho. Use sempre luvas durante a limpeza e evite tocar em material orgânico. Use apenas lenços de limpeza de lentes novos para evitar contaminação e limpe em uma única direção com um movimento. Evite esfregar a lente e limpar o lenço para frente e para trás para minimizar os danos. Se a lavagem a seco for insuficiente, aplique uma pequena quantidade de etanol no tecido, limpe o óleo como mencionado anteriormente e finalize com um lenço seco. - Se as mesmas soluções de proteína/tampão forem usadas em seguida, a tubulação pode não precisar ser trocada. Se forem usadas soluções diferentes, substitua a tubulação para reduzir o risco de contaminação de soluções anteriores.
NOTA: Não use o mesmo tubo por mais de 1 semana, mesmo que esteja realizando experimentos com as mesmas soluções, pois ele ficará sujo com o tempo.
Resultados
Após a aquisição de dados, a análise de dados pode ser realizada nos dados brutos usando o código MATLAB para gerar rastreamentos a partir dos dados brutos coletados pelo APD. A Figura 3 mostra um traço de aprisionamento exemplar, incluindo a linha de base antes do aprisionamento, o evento de aprisionamento em que uma grande mudança na transmissão (ΔT / T0) e desvio padrão é observada antes que o laser seja desligado por cerca de 5s antes de ser ligado novamente. Uma redução significativa no desvio padrão e o retorno da transmissão a níveis semelhantes aos da linha de base indicam a liberação de proteína. O desvio linear é removido do traço usando a função MATLAB detrend.m e, em seguida, o valor médio dos dados é adicionado de volta ao traço sem tendência. Ocasionalmente, precisamos diminuir a tendência do traço à medida que a configuração se desloca ao longo do tempo, causando uma diminuição linear na transmissão (veja o traço cinza na Figura 3). Pequenas mudanças nos traços da linha de base antes e depois do trapping são devidas ao ajuste do estágio para otimizar a linha de base com desvio padrão mínimo, demonstrado na Figura 4A. Às vezes, as moléculas de proteína são visíveis no traço sem ficarem presas, denominadas proteínas passageiras. As proteínas que passam aparecem como uma mudança brusca na transmissão, semelhante a uma armadilha típica (Figura 4B), mas com uma duração significativamente mais curta, como mostrado na Figura 4A. A densidade espectral de potência (PSD) apresenta outra análise para confirmar o aprisionamento de proteínas, fornecendo intensidade de sinal em várias frequências. Os movimentos conformacionais de proteínas são normalmente vistos na faixa de >1 μs por métodos de espectroscopia de molécula única40. A Figura 4C demonstra que, em comparação com a linha de base, o aprisionamento de uma proteína leva a uma maior intensidade de sinal, pelo menos dentro da faixa de 10 kHz (> 100 μs). Também destaca a importância de alinhar o estágio a uma linha de base otimizada, pois uma linha de base ruim pode aumentar o ruído em frequências entre 50-500 Hz, uma faixa de frequência sobreposta com movimentos conformacionais de proteínas.
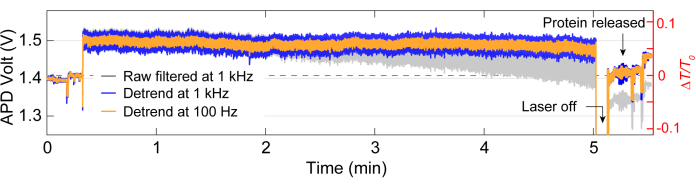
Figura 3: Traço de captura completo para uma única proteína. Traço representativo para uma armadilha completa, incluindo a linha de base, o aprisionamento de uma proteína e a liberação da proteína. Os saltos no rastreamento antes e depois do trapping são devidos ao alinhamento. Clique aqui para ver uma versão maior desta figura.

Figura 4: Eventos de rastreamento comuns. (A) Exemplos de um alinhamento de uma linha de base ruim para uma boa e uma proteína passando perto do ponto quente. (B) Traço de captura mostrando o processo desde a linha de base quando o hotspot DNH está vazio até quando a proteína está presa. (C) Gráfico de densidade espectral de potência (PSD) entre as linhas de base boas e ruins representadas em (A) e a proteína presa em (B). Valores PSD mais altos indicam maior ruído em frequências específicas. Clique aqui para ver uma versão maior desta figura.
A maioria dos eventos de captura segue o mesmo padrão geral do traço na Figura 3, embora problemas ocasionais possam surgir durante os experimentos. Para a maioria dos experimentos, a proteína deve ser liberada manualmente, desligando o laser assim que o experimento desejado for concluído. Em alguns casos, no entanto, a proteína pode deixar a armadilha sem intervenção, como mostrado na Figura 5A. Por outro lado, às vezes as proteínas podem permanecer no local de captura mesmo depois de desligar o laser, provavelmente devido à proteína aderir à amostra. Essa aderência resulta em um traço ruidoso após desligar e ligar o laser (consulte a Figura 5B). A probabilidade de isso ocorrer depende da proteína, pois algumas proteínas são mais propensas à adsorção superficial41,42. O uso de um revestimento como o PEG-tiol pode reduzir as chances de aderência da proteína39,43. A menos que desejado, como estudar interações proteína-proteína, outro problema é o aprisionamento duplo, onde uma segunda proteína é presa após a primeira armadilha. Isso é caracterizado por outro aumento acentuado na transmissão, semelhante ao primeiro trap, e uma mudança no desvio padrão (ver Figura 5C).
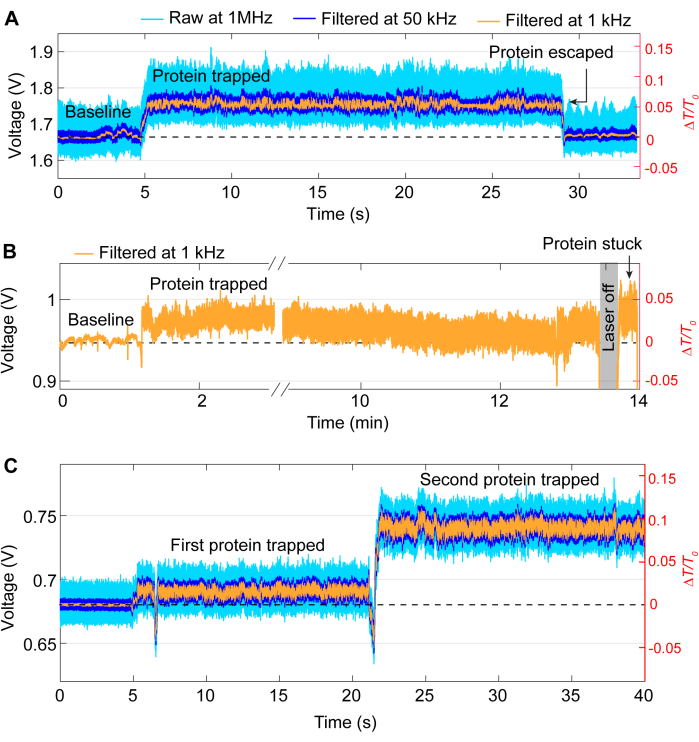
Figura 5: Exemplos de eventos de armadilhagem indesejáveis. (A) Liberação não intencional de uma proteína do hotspot DNH. (B) Exemplo de proteína que fica presa na superfície da amostra no hotspot DNH. (C) O salto de traços ocorre quando uma segunda proteína é presa enquanto a primeira ainda permanece no hotspot DNH. Clique aqui para ver uma versão maior desta figura.
Um experimento representativo realizado sobre o carregamento de ferro in situ em uma molécula de apoferritina demonstra o uso de pinças plasmônicas como ferramenta para investigar a dinâmica conformacional de proteínas29. A ferritina é uma proteína transportadora de ferro que existe em dois estados: apo-ferritina, que não contém ferro, e holoferritina, que é preenchida com ferro44,45. O ferro ferroso entra na proteína através de canais de 3 vezes, onde é oxidado em ferro férrico e armazenado no núcleo da proteína46. A Figura 6A mostra um traço típico de aprisionamento de apoferritina com uma solução ferrosa infundida por mais de 20 minutos enquanto a proteína está presa. Os traços de 20 s obtidos ao longo de todo o traço nos pontos b-e fornecem informações sobre as mudanças que ocorrem na proteína ao longo do tempo. Na Figura 6B, a apoferritina está presa em um tampão PBS padrão e nenhuma mudança significativa é observada no traço. A Figura 6C, D mostra flutuações no S.D dos traços, que são causadas pelo carregamento de ferro na proteína através de seus canais de 3 vezes, resultando em um estado mais dinâmico (apo-) onde os canais estão abertos e um estado mais compacto (holo-) com os canais fechados. Após a molécula de ferritina ser preenchida com ferro, ela fez a transição para sua holoforma, resultando em um traço de aprisionamento estável, conforme mostrado na Figura 6E. As funções de densidade de probabilidade (PDF) nas Figuras 6B-E mostram ainda mais as mudanças que a proteína sofre após a exposição a diferentes condições de solução ao longo do tempo.

Figura 6: Carga de ferro in situ em uma apoferritina presa. (A) Traço de transmissão completo de um DNH com uma molécula de apoferritina presa, seguido pela injeção de uma solução ferrosa no local de aprisionamento para observar as mudanças conformacionais da ferritina associadas à carga de ferro. (B) Traço de captura de 20 s de uma apoferritina presa antes que a solução ferrosa atingisse o ponto quente. (C, D) Traços de aprisionamento de 20 s após a molécula de apoferritina ter sido exposta à solução ferrosa. Segmentos azuis e roxos marcam o S.D superior e inferior do traço, indicando conformações flexíveis e rígidas de ferritina, respectivamente. (E) Traço de captura de 20 s após a apoferritina ter sido exposta à solução ferrosa por >20 minutos. Os gráficos da função de densidade de probabilidade (PDF) à direita mostram a distribuição da transmissão e são codificados por cores para os segmentos azul e roxo. Este número foi modificado de29. Clique aqui para ver uma versão maior desta figura.
Figura suplementar 1: Amostra DNH de ouro montada na célula de fluxo impressa em 3D. A amostra é colocada em uma ranhura especial e aderida à célula de fluxo usando fita adesiva PET dupla face. Os principais parâmetros e medições associadas para nosso projeto de célula de fluxo são rotulados. Clique aqui para baixar este arquivo.
Figura suplementar 2: Parte traseira da célula de fluxo com amostra DNH dourada montada e parede interna rotulada. A amostra é selada na célula de fluxo usando silicone duplicado. Clique aqui para baixar este arquivo.
Figura suplementar 3: Diagrama da célula de fluxo com DNH dourado montado com orifícios de entrada e saída rotulados. Clique aqui para baixar este arquivo.
Discussão
Uma etapa crucial no protocolo é garantir que a célula de fluxo não vaze antes de ser colocada no palco, que deve ser testado fora da montagem em uma alta taxa de fluxo de antemão. O vazamento após a montagem da amostra pode danificar os componentes ópticos, em particular a objetiva inferior.
O alinhamento pode se desviar de sua posição ideal ao longo do tempo durante um experimento, causando variação de sinal devido à sensibilidade das pinças plasmônicas. Quando isso ocorrer, realinhe usando os controles piezoelétricos para transmissão máxima e desvio padrão mínimo da linha de base, pois uma linha de base ruidosa reduz a qualidade dos dados. Deve-se tomar cuidado para fazer anotações meticulosas de quando os alinhamentos são feitos para remover o risco de confundir a interferência do usuário com um evento de captura. Se ocorrer um grande desvio, alinhe suavemente o estágio para minimizar o risco de liberação da proteína e observe o tempo de ajuste.
Modificações e alterações na técnica apresentada podem ser feitas com base em necessidades experimentais específicas. Por exemplo, um estágio com temperatura controlada pode ajudar a resfriar/aquecer a amostra conforme desejado, em vez de usar o aquecimento a laser para aumentar a temperatura31,33. Outras técnicas, por exemplo, a microscopia de espalhamento interferométrico (iSCAT), podem fornecer interferência da proteína dentro do campo de espalhamento do DNH, obtendo sinal adicional proporcional à polarizabilidade da proteína, que está associada ao tamanho de partícula47,48.
As pinças plasmônicas são puramente uma técnica de detecção temporal, pois os dados são registrados pelo APD (um detector de pixel único). Essa técnica não fornece informações diretas sobre as mudanças estruturais de uma proteína, como quais regiões da proteína estão envolvidas em uma mudança conformacional ou onde um ligante ou outra proteína pode se ligar à proteína. Além disso, a técnica é limitada a uma faixa temporal de >1 μs devido à taxa de amostragem do cartão de aquisição de dados (1 MHz). Considerando a frequência de Nyquist, onde a maior faixa possível é a metade da taxa de aquisição, neste caso, 2 μs em condições perfeitas.
Neste protocolo, descrevemos o processo de criação de um experimento de pinças plasmônicas para capturar uma única proteína e monitorar mudanças em sua dinâmica conformacional ao longo do tempo. A técnica pode ser desenvolvida em qualquer microscópio caseiro ou comercial. Ao contrário das abordagens baseadas em fluorescência ou tethering, como smFRET e pinças ópticas, a técnica pode capturar proteínas sem rótulos ou amarras e ainda atingir a sensibilidade de uma única molécula. As condições da solução podem ser alteradas enquanto a proteína é capturada usando um sistema microfluídico, permitindo o monitoramento em tempo real do efeito de diferentes soluções na proteína. Essas características tornam as pinças plasmônicas uma ferramenta promissora nos campos da biofísica e do biossensor, particularmente para proteínas que as técnicas convencionais lutam para interrogar em seus estados nativos. Aplicações futuras se concentrarão na decodificação da dinâmica conformacional de proteínas mais dinâmicas, como proteínas intrinsecamente desordenadas e proteínas contendo regiões intrinsecamente desordenadas e proteínas de membrana, cuja dinâmica e até mesmo estrutura escapam às técnicas atuais.
Divulgações
Os autores não têm nada a divulgar.
Agradecimentos
S.Z. reconhece o apoio da Parceria de Treinamento de Doutorado do Conselho de Pesquisa em Biotecnologia e Ciências Biológicas (BBSRC DTP) (BB/T0083690/1). Os autores reconhecem o financiamento da Iniciativa de Educação e Pesquisa Reino Unido-Índia (UKIERI). M.R. agradece o apoio da Royal Society e da Wolfson Foundation.
Materiais
| Name | Company | Catalog Number | Comments |
| 100x Objective (NA = 1.25) | Olympus | PLN100XO | Clean oil only with lens cleaning tissue in one motion |
| 12-1 rotary bidirectional microfluidic valve | Elveflow | ||
| 3/2 way solenoid valve | Elveflow | ||
| 4x Objective (NA = 0.1) | Olympus | PLN4XP | |
| Airgun | RS Components | 666-6772 | |
| Avalanche photodiode (APD) | Thorlabs | APD120A/M | Do not oversaturate, 50 MHz bandwidth, static sensitive |
| Butterfly laser diode | Thorlabs | FPL852S | |
| Charge-coupled device (camera) | Hikrobot | MV-CE200-10UC | |
| Crystallising dish | VWR | 216-0065 | |
| Data acquisition card | National instruments | USB-6361 | 2 MHz sampling rate |
| Double-sided tape | Adhesive Research Inc | ARcare92712 | Cut appropriate shape for flow cell with a laser cutter |
| Ethanol | Fume hood only, use gloves | ||
| Fibreport collimator | Thorlabs | PAF2-A7B | |
| Glass microscope coverslips (thickness 0.17 mm) | |||
| Half wave plate | Thorlabs | AHWP10M-980 | |
| Ideal-tek 120 mm, stainless steel, straight tweezers | RS Components | 282-7472 | For holding sample and peeling double sided tape |
| Isopropanol (IPA) | Fume hood only, use gloves | ||
| Lens cleaning tissue | Thorlabs | MC-5 | |
| Metrosil rapid duplicating silicone parts A and B | Metrodent | MSILR/1 | |
| Microscope slides (75 mm x 26 mm) | |||
| Modular Optical Tweezers System | Thorlabs | OTKB/M-CUSTOM | |
| Objective oil | Olympus | Use gloves, store between 2oC-8oC | |
| Photodiode laser mount | Thorlabs | CLD1015 | |
| Piezoelectric controller | Thorlabs | MDT693B | |
| Piezoelectric stage | Thorlabs | Nanomax 300 | |
| Planoconcave lens (f = -50 mm) | Thorlabs | LC1715-B | |
| Planoconvex lens (f = 150 mm) | Thorlabs | LA1433-B | |
| Planoconvex lens (f = 60 mm) | Thorlabs | LA1134-B | |
| Poly(ethylene glycol) methyl ether thiol (PEG-thiol) 800 MW | Sigma Aldrich | 729108 | Store between 2oC-8oC |
| PTFE tubing (ID: 0.18 mm) | Vici Jour | JR-T-6805-M25 | |
| PTFE tubing (ID: 0.8 mm) | Cole-Parmer | WZ-21942-72 | |
| SEM-FIB | Zeiss | Crossbeam 550 | Gallium ion source |
| Shortpass dichroic mirror 805 nm | Thorlabs | DMSP805 | |
| Silver mirror | Thorlabs | PF-10-03-P01 | |
| Syringe pump | Harvard Apparatus | 70-4511 | |
| Weller Erem 120 mm stainless steel, flat, rounded tweezers | RS Components | 176-1150 | For holding glass coverslip and peeling glue from flow cell |
Referências
- Peter Lu, H. P. Sizing up single-molecule enzymatic conformational dynamics. Chem Soc Rev. 43 (4), 1118-1143 (2014).
- Mazal, H., Haran, G. Single-molecule FRET methods to study the dynamics of proteins at work. Curr Opin Biomed Eng. 12, 8-17 (2019).
- Ritchie, D. B., Woodside, M. T. Probing the structural dynamics of proteins and nucleic acids with optical tweezers. Curr Opin Str Biol. 34, 43-51 (2015).
- Neupane, K., Solanki, A., Sosova, I., Belov, M., Woodside, M. T. Diverse metastable structures formed by small oligomers of α-Synuclein probed by force spectroscopy. PLoS One. 9 (1), e86495 (2014).
- Hughes, M. L., Dougan, L. The physics of pulling polyproteins: a review of single molecule force spectroscopy using the AFM to study protein unfolding. Rep Prog Phys. 79 (7), 076601-076601 (2016).
- Young, G., et al. Quantitative mass imaging of single biological macromolecules. Science. 360 (6387), 423-427 (2018).
- Schmid, S., Dekker, C. Nanopores: a versatile tool to study protein dynamics. Essays Biochem. 65 (1), 93-107 (2021).
- Van Meervelt, V., et al. Real-time conformational changes and controlled orientation of native proteins inside a protein nanoreactor. J Am Chem Soc. 139 (51), 18640-18646 (2017).
- Awasthi, S., Ying, C., Li, J., Mayer, M. Simultaneous determination of the size and shape of single α-Synuclein oligomers in solution. ACS Nano. 17 (13), 12325-12335 (2023).
- Sµnchez-Rico, C., Von Vithenberg, L., Warner, L., Lamb, D. C., Sattler, M. Effects of fluorophore attachment on protein conformation and dynamics studied by spFRET and NMR spectroscopy. Chemistry. 23 (57), 14267-14277 (2017).
- Yin, L., et al. How does fluorescent labeling affect the binding kinetics of proteins with intact cells. Biosens Bioelectr. 66, 412-416 (2015).
- Berkovich, R., et al. Rate limit of protein elastic response is tether dependent. Proc Natl Acad Sci. 109 (36), 14416-14421 (2012).
- Dahmardeh, M., Mirzaalian Dastjerdi, H., Mazal, H., Köstler, H., Sandoghdar, V. Self-supervised machine learning pushes the sensitivity limit in label-free detection of single proteins below 10 kDa. Nat Meth. 20 (3), 442-447 (2023).
- Becker, J., et al. A quantitative description for optical mass measurement of single biomolecules. ACS Photon. 10 (8), 2699-2710 (2023).
- Houghtaling, J., et al. Estimation of shape, volume, and dipole moment of individual proteins freely transiting a synthetic nanopore. ACS Nano. 13 (5), 5231-5242 (2019).
- Plesa, C., et al. Fast translocation of proteins through solid state nanopores. Nano Lett. 13 (2), 658-663 (2013).
- Schmid, S., Stömmer, P., Dietz, H., Dekker, C. Nanopore electro-osmotic trap for the label-free study of single proteins and their conformations. Nat Nanotechnol. 16 (11), 1244-1250 (2021).
- Shi, X., Verschueren, D. V., Dekker, C. Active delivery of single DNA molecules into a plasmonic nanopore for label-free optical sensing. Nano Lett. 18 (12), 8003-8010 (2018).
- Verschueren, D. V., et al. Label-free optical detection of DNA translocations through plasmonic nanopores. ACS Nano. 13 (1), 61-70 (2019).
- Verschueren, D., Shi, X., Dekker, C. Nano-optical tweezing of single proteins in plasmonic nanopores. Small Meth. 3 (5), 1800465 (2019).
- Peri, S. S. S., et al. Quantification of low affinity binding interactions between natural killer cell inhibitory receptors and targeting ligands with a self-induced back-action actuated nanopore electrophoresis (SANE) sensor. Nanotechnology. 32 (4), 045501 (2020).
- Leong, I. W., Tsutsui, M., Yokota, K., Taniguchi, M. Salt gradient control of translocation dynamics in a solid-state nanopore. Anal Chem. 93 (49), 16700-16708 (2021).
- Juan, M. L., Righini, M., Quidant, R. Plasmon nano-optical tweezers. Nat Photon. 5 (6), 349-356 (2011).
- Juan, M. L., Gordon, R., Pang, Y., Eftekhari, F., Quidant, R. Self-induced back-action optical trapping of dielectric nanoparticles. Nat Phys. 5 (12), 915-919 (2009).
- Pang, Y., Gordon, R. Optical trapping of 12 nm dielectric spheres using double-nanoholes in a gold film. Nano Lett. 11 (9), 3763-3767 (2011).
- Pang, Y., Gordon, R. Optical trapping of a single protein. Nano Lett. 12 (1), 402-406 (2012).
- Kotnala, A., Gordon, R. Quantification of high-efficiency trapping of nanoparticles in a double nanohole optical tweezer. Nano Lett. 14 (2), 853-856 (2014).
- Booth, L. S., et al. Modelling of the dynamic polarizability of macromolecules for single-molecule optical biosensing. Sci Rep. 12 (1), 1995 (2022).
- Yousefi, A., et al. Optical monitoring of in situ Iron loading into single, native ferritin proteins. Nano Lett. 23 (8), 3251-3258 (2023).
- Yousefi, A., et al. Structural Flexibility and Disassembly Kinetics of Single Ferritin Molecules Using Optical Nanotweezers. ACS Nano. 18 (24), 15617-15626 (2024).
- Peters, M., et al. Energy landscape of conformational changes for a single unmodified protein. NPJ Biosens. 1 (1), 14-14 (2024).
- Peters, M., McIntosh, D., Branzan Albu, A., Ying, C., Gordon, R. Label-free tracking of proteins through plasmon-enhanced interference. ACS Nanosci Au. 4 (1), 69-75 (2024).
- Ying, C., et al. Watching single unmodified enzymes at work. arXiv. , (2021).
- Yang-Schulz, A., et al. Direct observation of small molecule activator binding to single PR65 protein. NPJ Biosensing. 2 (1), 1-10 (2025).
- Yang, W., van Dijk, M., Primavera, C., Dekker, C. FIB-milled plasmonic nanoapertures allow for long trapping times of individual proteins. iScience. 24 (11), 103237 (2021).
- Koya, A. N., et al. Novel plasmonic nanocavities for optical trapping-assisted biosensing applications. Adv Optical Mater. 8 (7), 1901481 (2020).
- Goswami, A., Umashankar, R., Gupta, A. K., Aravindan, S., Rao, P. V. Development of a microstructured surface using the FIB. J Micromanufact. 1 (1), 53-61 (2018).
- Onuta, T. D., Waegele, M., DuFort, C. C., Schaich, W. L., Dragnea, B. Optical field enhancement at cusps between adjacent nanoapertures. Nano Lett. 7 (3), 557-564 (2007).
- Al-Ani, A., et al. The influence of PEG-thiol derivatives on controlling cellular and bacterial interactions with gold surfaces. Appl Surf Sci. 462, 980-990 (2018).
- Schuler, B., Hofmann, H. Single-molecule spectroscopy of protein folding dynamics-expanding scope and timescales. Curr Opin Str Biol. 23 (1), 36-47 (2013).
- Kopac, T. Protein corona, understanding the nanoparticle-protein interactions and future perspectives: A critical review. Int J Biol Macromol. 169, 290-301 (2021).
- Li, X., Guo, W., Xu, R., Song, Z., Ni, T. The interaction mechanism between gold nanoparticles and proteins: Lysozyme, trypsin, pepsin, γ-globulin, and hemoglobin. Spectrochim Acta A Mol Biomol Spectrosc. 272, 120983 (2022).
- Emilsson, G., et al. Strongly stretched protein resistant poly(ethylene glycol) brushes prepared by grafting-To. ACS Appl Mater Inter. 7 (14), 7505-7515 (2015).
- Theil, E. C., Liu, X. S., Tosha, T. Gated pores in the ferritin protein nanocage. Inorg Chim Acta. 361 (4), 868-874 (2008).
- Theil, E. C., et al. The ferritin iron entry and exit problem. Inorg Chim Acta. 297 (1), 242-251 (2000).
- Takahashi, T., Kuyucak, S. Functional properties of threefold and fourfold channels in Ferritin D=deduced from electrostatic calculations. Biophys J. 84 (4), 2256-2263 (2003).
- Gemeinhardt, A., et al. Label-free imaging of single proteins secreted from living cells via iSCAT microscopy. J Vis Exp. (141), e58486 (2018).
- Piliarik, M., Sandoghdar, V. Direct optical sensing of single unlabelled proteins and super-resolution imaging of their binding sites. Nat Comm. 5 (1), 4495 (2014).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoExplore Mais Artigos
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados