Method Article
Monitorización de la dinámica conformacional de proteínas individuales no modificadas mediante nanopinzas plasmónicas
En este artículo
Resumen
Las nanopinzas plasmónicas utilizan la resonancia de plasmones de superficie localizada en nanoestructuras de oro para atrapar nanopartículas individuales, incluidas proteínas, dentro de un campo óptico a escala nanométrica. Los cambios en la señal dispersa revelan la presencia de proteínas y la dinámica conformacional, lo que permite el monitoreo sin modificaciones de fluoróforos o anclaje de superficie.
Resumen
Las técnicas actuales de una sola molécula para caracterizar proteínas generalmente requieren etiquetas, ataduras o el uso de condiciones de solución no nativas. Tales cambios pueden alterar la biofísica de las proteínas y reducir la utilidad de los datos adquiridos. Las nanopinzas plasmónicas son una técnica que utiliza la resonancia de plasmones de superficie localizada (LSPR) en nanoestructuras de oro para mejorar el campo eléctrico dentro de una región de punto caliente confinada. Esta mejora del campo permite el uso de bajas potencias láser para atrapar nanopartículas individuales mucho más pequeñas que las pinzas ópticas convencionales, de hasta solo unos pocos nanómetros de diámetro, como las proteínas individuales. El atrapamiento de moléculas de proteína individuales dentro de la región del punto caliente induce un cambio en el índice de refracción local (nproteína >n agua), alterando la dispersión de la luz como producto de la polarizabilidad de la molécula, que se ve afectada por su volumen, forma anisotropía e índice de refracción. Un fotodiodo de avalancha (APD) recoge los cambios posteriores en la dispersión de la luz. Estas alteraciones se pueden analizar para determinar los cambios en la molécula atrapada, incluido su tamaño, conformación global y dinámica del cambio conformacional a lo largo del tiempo. La incorporación de la microfluídica dentro del sistema permite controlar los cambios ambientales y monitorizar en tiempo real sus efectos posteriores sobre la molécula. En este protocolo, demostramos los pasos para atrapar moléculas de proteína individuales, alterar sus condiciones de solución ambiental y monitorear sus cambios conformacionales correspondientes utilizando un sistema de nanotweezers plasmónicos.
Introducción
Las técnicas actuales de una sola molécula para interrogar la dinámica conformacional de las proteínas incluyen métodos basados en el marcaje, como la transferencia de energía por resonancia fluorescente (FRET)1,2, enfoques basados en anclaje, como las pinzas ópticas 3,4 y la microscopía de fuerza atómica (AFM)5, técnicas basadas en interferencias, como la microscopía de dispersión de interferencias (iSCAT)6, o técnicas basadas en nanofluidos, como los nanoporos7,Puesto 8,9. Si bien estos métodos tienen muchas ventajas; Algunas limitaciones clave les impiden proporcionar datos sobre la dinámica conformacional de proteínas no modificadas. Las pinzas FRET y ópticas requieren un marcaje o anclaje de fluoróforos a una superficie, lo que puede afectar las propiedades biofísicas de las proteínas 10,11,12. iSCAT, aunque técnicamente no tiene marcas, también requiere la interacción entre la proteína y una superficie para observar la interferencia generada entre las dos que potencialmente afecta las propiedades de las proteínas. Además, limitado por su relación señal-ruido, iSCAT solo puede detectar proteínas de >40 kDa debido al ruido del equipo y a las fluctuaciones de fondo en forma de motas13. Aunque este límite de tamaño puede aliviarse mediante el aprendizaje automático, los componentes del búfer son limitados, ya que pueden afectar a las propiedades ópticas, lo que da lugar a datos ruidosos13,14. Los nanoporos presentan tiempos de translocación rápidos de las proteínas a través del poro (generalmente dentro de los 5 μs), lo que los hace incapaces de detectar dinámicas conformacionales más lentas15,16, aunque las investigaciones para aliviar estas limitaciones, como el uso de origami de ADN en una trampa electro-osmótica de nanoporos17 o la incorporación de plasmónicos 18,19,20,21. Además, las altas concentraciones de sal, típicamente alrededor de 1 M, pueden reducir la aplicabilidad de los datos para el trabajo in vivo 15,22. La técnica ideal de una sola molécula para la caracterización de proteínas debe monitorear las proteínas en tiempo real y capturar la dinámica conformacional durante períodos más largos (es decir, milisegundos) sin la necesidad de modificaciones en las condiciones de la proteína o de la solución no nativa.
Las nanopinzas plasmónicas son similares a las pinzas ópticas convencionales, en el sentido de que utilizan la luz para atrapar la materia. Las nanopinzas plasmónicas, sin embargo, utilizan la resonancia plasmónica de superficie localizada (LSPR) para mejorar el campo eléctrico en varios órdenes de magnitud para generar una fuerza de gradiente lo suficientemente fuerte como para atraparnanopartículas individuales. Además, la partícula atrapada desempeña un papel activo en la mejora de la resistencia de la trampa, lo que se conoce como atrapamiento de acción trasera autoinducida (SIBA) para estructuras de nanoapertura24. Este atrapamiento SIBA permite que las potencias láser bajas (es decir, milivatios) atrapen partículas pequeñas de hasta solo unos pocos nanómetros de diámetro, como las proteínas 25,26,27. El atrapamiento de moléculas de proteína individuales dentro de la región del punto caliente provoca un cambio en el índice de refracción local (nproteína >n agua), alterando la dispersión de la luz en función de la polarizabilidad de la molécula, que está influenciada por el volumen de la proteína, la forma anisotropía y el índice de refracción28. Un fotodiodo de avalancha (APD) detecta esta información para monitorear los cambios posteriores en la dispersión de la luz. Además, las nanopinzas plasmónicas permiten el monitoreo de las proteínas atrapadas en tiempo real sin marcaje, ataduras y condiciones de solución duras durante largos períodos de tiempo (es decir, de minutos a horas)29, cumpliendo con los criterios para una técnica ideal de una sola molécula para proteínas. Utilizando una estructura de doble nanoagujero (DNH), las nanopinzas plasmónicas han demostrado su capacidad para atrapar varias proteínas y dilucidar información clave de ellas, incluidas las transiciones conformacionales29, la cinética de desmontaje30, los paisajes energéticos31, el seguimiento de difusión32 y la unión de ligandos33,34. Además de las estructuras DNH, se ha demostrado que geometrías de estructuras alternativas atrapan partículas con tamaños de partícula pequeños35,36. En este protocolo se presentan los pasos fundamentales para configurar y ejecutar una configuración de nanotweezers plasmónicas con un sistema de microfluídica integrado. Esperamos que este protocolo ayude a aumentar la accesibilidad y la comprensión de las nanotweezers plasmónicas para los investigadores, en particular los de los campos de la biología estructural y la biofísica.
Protocolo
PRECAUCIÓN: Lea todas las hojas de datos de seguridad (SDS) relevantes para todos los productos químicos utilizados y cumpla con todas las prácticas de seguridad apropiadas, y use equipo de protección personal (gafas de seguridad láser, batas de laboratorio, guantes) según sea necesario.
1. Construcción de la configuración de las nanotpinzas plasmónicas
NOTA: La configuración óptica se basa en el kit del sistema de pinzas ópticas modulares (OTKB) que utiliza un láser y un APD diferentes (consulte la tabla de materiales). Utilice únicamente equipos ópticos en una mesa óptica adecuada para reducir el impacto de las vibraciones externas en el sistema. El láser en el kit era de 976 nm, pero como la longitud de onda de resonancia máxima para la resonancia en cuña de las estructuras DNH es de alrededor de 740-760 nm33. Elegimos un láser NIR (852 nm) porque está cerca del pico de resonancia, induce LSPR y también tiene una mejor tasa de rendimiento de detección por el APD basado en silicio. Se han utilizado láseres con longitudes de onda más largas de20 o18 más cortas para atrapar biomoléculas.
- Configure el láser en la montura láser de fotodiodo y alimentécelo a un colimador, que conduce a un rayo láser colimado con un ancho de 1,7 mm para nuestra configuración.
- Añade una placa de media onda a la trayectoria de la luz para ajustar la polarización. Ajuste con un polarizador Glan-Taylor para garantizar que la polarización vertical (polarización S) tenga la intensidad de luz más alta.
NOTA: El uso de la polarización correcta es vital para garantizar la máxima mejora del campo eléctrico de las estructuras DNH, ya que dependen de la polarización. - Enfoca la luz a través de una configuración de expansor de haz que consta de una lente plano-cóncava (f = -50 mm) seguida de una lente plano-convexa (f = 150 mm) para aumentar el ancho del haz a alrededor de 5 mm para llenar la apertura trasera completa del objetivo inferior.
- Utilice un espejo dicroico (paso corto de 805 nm) para reflejar el láser en la ubicación deseada. Coloque la cámara (CCD) detrás de él.
- Redirija la luz hacia el objetivo inferior (100x/1,25 NA) y coloque el objetivo superior (4x/0,1 NA) a la distancia confocal para recoger la luz transmitida.
- Agregue otro espejo dicroico (paso corto de 805 nm) para reflejar la luz láser hacia el APD a través de una lente plano-convexa colocada en su distancia focal para enfocar la luz en el APD. Agregue la fuente de luz blanca (WLS) detrás del espejo dicroico para que pueda volver a pasar a través de la configuración para llegar a la cámara. La Figura 1 muestra un esquema de la configuración de las nanotweezers plasmónicas completamente ensambladas.
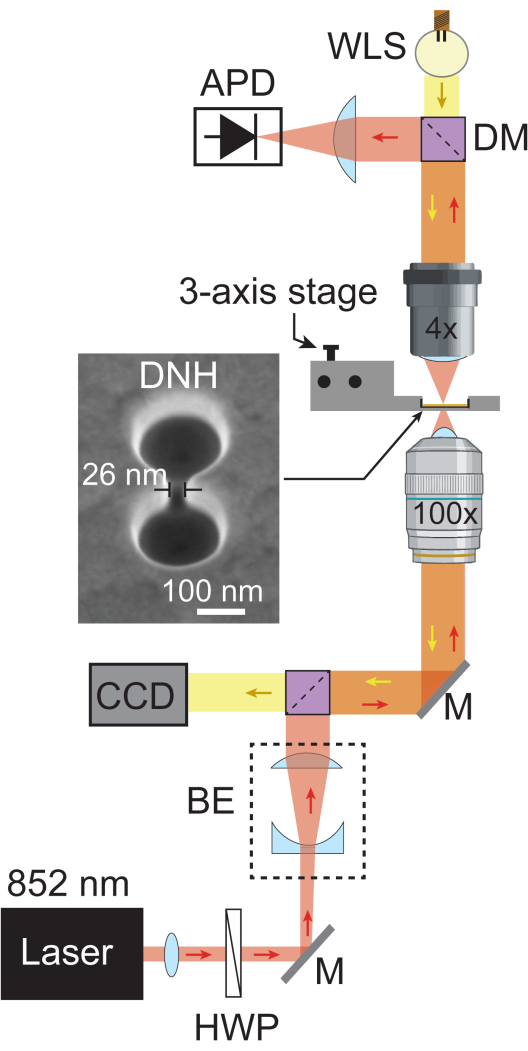
Figura 1: Configuración de las nanotpinzas plasmónicas. El esquema muestra la vía óptica completa de la configuración de las nanotweezers plasmónicas. Un rayo láser de 852 nm (rojo) pasa a través de un colimador y una placa de media onda, luego se expande mediante el expansor de haz (BE) a ~5,1 mm. Posteriormente, se enfoca en la muestra utilizando un objetivo de 100x. La luz láser transmitida es recogida por un objetivo 4x y registrada por un APD a una frecuencia de muestreo de 1 MHz. La luz blanca de WLS (amarillo) pasa a través del objetivo 4x, la muestra y el objetivo 100x antes de llegar al CCD, que proporciona una imagen de campo claro de la muestra. Las áreas donde se cruzan ambas trayectorias de luz se representan en naranja. Imagen SEM de DNH tomada con una inclinación de 20°. Abreviaturas: BE = Expansor de haz, CCD = Dispositivo de carga acoplada, DM = Espejo dicroico, HWP = Placa de media onda, M = Espejo plateado y WLS = Fuente de luz blanca. Haga clic aquí para ver una versión más grande de esta figura.
2. Fabricación de las estructuras DNH
- Deposite 5 nm de Ti seguido de 100 nm de Au en una oblea de sílice fundida de 550 μm de espesor utilizando evaporación por haz de electrones como se describió anteriormente29,33. Corta la oblea en dados de 1 cm x 1 cm en muestras listas para usar.
NOTA: Se eligió la evaporación por haz de electrones porque produce una capa de oro con baja rugosidad superficial, produciendo estructuras de DNH de alta calidad con alta eficiencia de atrapamiento37. - Tome la muestra de película de oro en blanco y móntela en una configuración de haz de iones enfocado en microscopio electrónico de barrido (SEM-FIB) a temperatura ambiente utilizando una fuente de iones de galio.
- Asegúrese de que las nanoaperturas FIB estén alineadas con SEM para producir DNH en posiciones precisas.
- Cree un cuadro de marcadores utilizando un haz de iones de alta corriente (por ejemplo, 100 pA). Este marcador servirá como guía de referencia para localizar la posición de las estructuras DNH bajo la configuración óptica.
- Para crear la estructura DNH, dos círculos con una distancia de centro a centro de 200 nm y 160 nm de diámetro están unidos por un rectángulo (3 nm x 40 nm). Para un tamaño de espacio de alrededor de 20-30 nm, grabe círculos con un factor de dosis de 0,09, mientras que la dosis rectangular está entre 0,300 y 0,350 utilizando una energía de sonda de 30 kV y una corriente de haz de 1 pA.
NOTA: El SEM-FIB utilizado tiene una resolución de 3 nm a esta energía de sonda, lo que permite la fabricación confiable de DNH con tamaños de espacio entre 20 y 30 nm. No usaríamos una resolución más baja, ya que esto producirá espacios de mayor tamaño, lo que reducirá la eficiencia de captura del DNH. Si un FIB es inviable, un método alternativo para producir estructuras de DNH es el uso de litografía de nanoesferas de poliestireno38. Debido a la alta precisión requerida para fabricar estructuras de DNH, es posible que la misma receta no produzca el mismo tamaño de espacio entre los usos de SEM-FIB. Modifique la dosis y la altura del rectángulo (dosis de 0,280 a 0,350 y altura de 2 a 4 nm) y mida el tamaño del espacio, apuntando a alrededor de 20 nm.
3. Recubrimiento de muestras de DNH
- Utilice un recipiente resistente a los disolventes, como un plato de cristalización hecho de vidrio de borosilicato 3.3, y agregue 20 ml de etanol.
PRECAUCIÓN: El etanol es altamente inflamable e irritante. Úselo solo en una campana extractora. Almacenar en un lugar fresco y bien ventilado. Mango con guantes y bata de laboratorio. - Pesar 32 mg de poli(etilenglicol) metil éter tiol (PEG-tiol; peso molecular promedio 800 g / mol) y mezclarlo con el etanol, asegurándose de que todo el PEG-tiol se haya disuelto para producir una solución con una concentración de alrededor de 2 mM para maximizar la densidad de la monocapa39.
PRECAUCIÓN: El PEG-tiol es un irritante; manéjelo con el EPI adecuado. - Agregue los chips de muestra que contienen las nanoestructuras a la mezcla con pinzas rectas, cubra e incube durante la noche (~ 16 h) a temperatura ambiente para que el PEG-tiol forme una monocapa autoensamblada en la superficie de oro.
- Después de la incubación, enjuague las muestras sosteniéndolas con pinzas rectas sobre un recipiente adecuado en la campana extractora. Use una botella rociadora para rociar etanol en cada lado a fondo. Secar completamente con una pistola de aire comprimido antes de su posterior almacenamiento o montaje en las nanotweezers plasmónicas.
NOTA: Las muestras se pueden almacenar a 4 °C para mejorar la longevidad, ya que pueden degradarse con el tiempo.
4. Montaje de la muestra recubierta de PEG en una celda de flujo
- Coloque la muestra recubierta en la celda de flujo impresa en 3D (impresa por una impresora Form 2 con resina Clear V4) utilizando pinzas rectas con la capa de oro hacia arriba (consulte la Figura complementaria 1 para conocer los parámetros y valores clave en nuestro diseño de celda de flujo).
- Despegue un lado de la cubierta de cinta adhesiva de doble cara de plástico PET transparente con pinzas rectas y colóquela en la muestra y la celda de flujo, asegurándose de que las nanoestructuras y los orificios de entrada/salida de la celda de flujo permanezcan descubiertos. Presione suavemente alrededor de los bordes de la cinta con pinzas redondeadas para asegurarse de que se haya adherido correctamente a la celda de flujo y a la muestra.
- Despegue el otro lado de la cinta y coloque suavemente un cubreobjetos de vidrio (grosor de 0,17 mm) sobre la muestra con pinzas redondeadas. Presione suavemente alrededor de los bordes del cubreobjetos con las pinzas redondeadas para asegurarse de que se haya adherido correctamente. Esto crea un canal de líquido (altura = 50 μm, volumen = 3,5 μL) dentro de la celda de flujo.
NOTA: No presione el cubreobjetos en o cerca de donde se encuentran las nanoestructuras, ya que pueden dañarse. Si el cubreobjetos está sucio, lávelo con etanol y séquelo con una pistola de aire antes de montarlo. - Mezcle partes iguales de A y B de las soluciones de silicona duplicadas en una proporción de 1:1 (o según las instrucciones del fabricante) en un portaobjetos de microscopio con una punta de pipeta pequeña.
- Rellene los espacios entre el cubreobjetos y la celda de flujo con la silicona duplicada mezclada, empujándola suavemente debajo del cubreobjetos. Sostenga la celda de flujo boca abajo y coloque con cuidado la silicona duplicada alrededor de la pared interna del orificio antes de usar la punta de la pipeta para moverla suavemente sobre los bordes visibles de la parte inferior de la sílice fundida de la muestra (consulte la Figura complementaria 2).
- Dejar secar con la capa de oro hacia arriba hasta que la silicona duplicada se haya fraguado por completo, según las instrucciones del fabricante.
NOTA: Tenga cuidado de no permitir que la silicona cubra las nanoestructuras cuando se aplique en la parte inferior de la muestra. Tenga cuidado de no forzar la silicona duplicada debajo del cubreobjetos, ya que puede entrar en los orificios de entrada/salida de la celda de flujo o en las nanoestructuras, que pueden ser difíciles de limpiar (Figura complementaria 3).
5. Conexión del sistema de microfluídica
NOTA: Asegúrese de usar tubos limpios para el sistema. En este caso, se utilizará un tubo de PTFE de diámetro interior pequeño: 0,18 mm de diámetro interno y un tubo de PTFE de diámetro interior grande: 0,8 mm, pero otros valores de diámetro interno funcionarán.
- Configure la bomba de jeringa y conéctela a la válvula solenoide de 3/2 vías utilizando el tubo grande de PTFE. Conecte un puerto de la válvula al contenedor de almacenamiento intermedio y el otro a una bobina de retención, de alrededor de 1 ml de volumen, hecha del tubo de diámetro interior grande para evitar el reflujo hacia la jeringa.
NOTA: La válvula 3/2 se puede controlar a través de un controlador de válvula, que determina si la solución se infunde/retira del recipiente tampón o de la bobina de retención. - Conecte la bobina de retención a la válvula central de la válvula microfluídica bidireccional rotativa 12/1. Conecte los recipientes para soluciones que utilizan tubos de diámetro interno pequeños para muestras escasas o valiosas y tubos de diámetro interno grandes para el resto. Use una válvula solo para infundir en un contenedor de desechos y otra solo para infundir en la celda de flujo; use un tubo de diámetro interno pequeño para la válvula de infusión de celda de flujo.
- Conecte un tubo de diámetro interno grande desde la salida de la celda de flujo hasta el contenedor de desechos. La Figura 2 muestra un esquema del sistema microfluídico completamente conectado.
NOTA: Es preferible un tubo de PTFE de diámetro interno pequeño para la entrada de la celda de flujo y para las válvulas que se utilizarán para la infusión de proteínas debido al menor volumen donde la muestra permanecerá en el tubo. Los tubos de PTFE de diámetro interior grandes son preferibles para materiales menos costosos o más abundantes, como los tampones. Trate de usar un tubo mínimo para reducir aún más este volumen muerto.
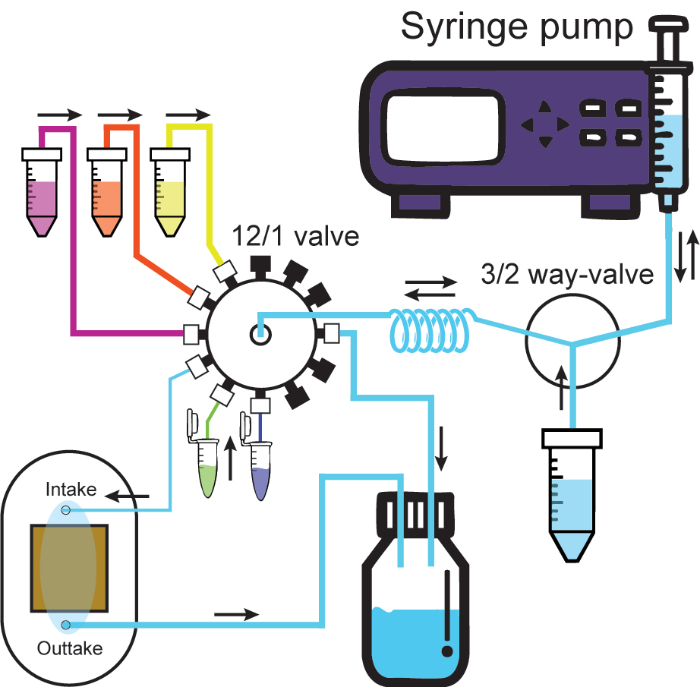
Figura 2: Sistema microfluídico. El esquema ilustra el sistema microfluídico. La bomba de jeringa infunde o extrae soluciones a través de un puerto de la válvula de 3/2 vías, ya sea el recipiente de solución o la bobina de retención. Las soluciones conectadas a la válvula 12/1 siempre pasan a través de la bobina de retención cuando se retiran y luego se pueden infundir a través del canal deseado. La infusión al canal de entrada conectado a la celda de flujo empujará la solución desde la celda de flujo hacia el contenedor de desechos. Los tubos gruesos y delgados representan tubos de diámetro interno grandes y pequeños del protocolo. Las tapas negras de la válvula 12/1 representan canales sellados. Las direcciones de flujo están etiquetadas con flechas negras. Haga clic aquí para ver una versión más grande de esta figura.
6. Preparación del sistema de microfluídica
- Cargue la interfaz de usuario del sistema de microfluídica en la PC y verifique que los componentes estén conectados correctamente. Seleccione el icono de reproducción junto a la conexión y el distribuidor para abrir su respectiva interfaz de usuario. Para omitir la válvula de 3/2 vías, gire el puerto 1 del cable.
- Asegúrese de que las válvulas que se utilizarán dentro del sistema se limpien mediante la infusión de isopropanol (IPA) y luego lave los tubos varias veces con tampones de su elección o agua destilada para eliminar el aire y el IPA dentro del sistema. La infusión se puede realizar seleccionando la válvula deseada en la interfaz de usuario del distribuidor y haciendo funcionar la bomba de jeringa, seleccionando infundir/extraer y eligiendo el volumen y el caudal deseados (por ejemplo, 0,5 mL de volumen y 0,2 mL/min de caudal).
NOTA: No infunda alcohol isopropílico en las celdas de flujo, ya que esto degradará la celda y el pegamento, lo que puede provocar fugas.
7. Montaje de la celda de flujo en nanopinzas plasmónicas y verificación de fugas
- Conecte los tubos de entrada y salida a la parte adecuada de la celda de flujo antes de colocarlos sobre un pañuelo limpio con una capa de oro hacia arriba.
- Infunda tampón en la celda de flujo con un caudal alto (~0,3 mL/min) y verifique que el fluido se mueva a través de la muestra en la celda de flujo y que no haya fluido visible en el exterior o la parte inferior de la celda de flujo. Se pueden utilizar caudales más altos, siempre que no se produzcan fugas. Si la muestra tiene fugas, desmonte y vuelva a montar.
- Agregue 1-2 gotas de aceite de inmersión en el objetivo 100x antes de colocar la celda de flujo en la etapa de nanotweezers plasmónicas con la capa de oro hacia abajo. Coloque clips de metal sobre los imanes de la celda de flujo y bloquee la platina para ayudar a mantenerla en su lugar.
PRECAUCIÓN: El aceite objetivo es irritante, peligroso para la salud y tóxico para la vida acuática, solo manéjelo con guantes.
8. Localización de nanoestructuras en la muestra
- Encienda la fuente de luz blanca. Abra el software de la cámara y aumente la ganancia y el tiempo de exposición hasta que las nanoestructuras sean visibles. Encienda el láser con una potencia láser relativamente alta y ajuste el eje z manualmente hasta que el punto láser sea visible. Priorice el aumento del tiempo de exposición y la ganancia sobre el aumento de la potencia del láser para encontrar el punto láser.
PRECAUCIÓN: Los láseres representan un grave riesgo potencial para los usuarios. Asegúrese de utilizar los EPI adecuados, como las gafas de seguridad láser, con suficiente densidad óptica dentro del rango de longitud de onda requerido. - Encienda el controlador piezoeléctrico y seleccione la configuración adecuada para el puerto COM y el voltaje máximo. Establezca los valores de los ejes x, y y z a la mitad del voltaje máximo para permitir un buen rango para alinear el escenario en todas las direcciones.
NOTA: El puerto COM del controlador piezoeléctrico se puede encontrar y cambiar en el administrador de dispositivos en la pestaña de puertos. - Mueva el punto láser para superponerlo con uno de los DNH usando las perillas de control de los ejes x, y y z de la etapa principal. Asegúrese de que el APD esté encendido y, a continuación, cierre suavemente la carcasa de las nanopinzas plasmónicas.
NOTA: El uso de una herramienta de marcado en el software de la cámara, si está disponible, ayudará a mover el punto láser sobre una nanoestructura. Coloque el marcador en el centro del punto láser y apague el láser, pero deje la fuente de luz blanca encendida, para permitir una alineación más fácil con una nanoestructura. El APD puede estar sobresaturado y dañado si le llega demasiada luz láser. Asegúrese de que la muestra esté en el camino de la trayectoria del láser y aumente gradualmente la potencia del láser para garantizar que no se alcance la saturación. Se pueden colocar filtros de densidad neutra (N.D) antes del APD para reducir la luz que le llega si es necesario.
9. Alinear de forma óptima el láser con la nanoestructura deseada
- Abra el software asociado con la grabación de APD, como una interfaz de usuario casera de Labview, y establezca la frecuencia de corte en 1 kHz. Asigne un nombre y establezca la ruta de archivo deseada y el formato de nomenclatura de los archivos que se guardarán.
- Apague la fuente de luz blanca y vuelva a encender el láser. Ajuste a una potencia láser adecuada (por ejemplo, ~ 20 mW) y use los controles piezoeléctricos para ajustar los ejes x, y y z hasta que la señal APD sea lo más alta posible, evitando la saturación de APD y con una desviación estándar mínima (SD) de la traza.
NOTA: Es probable que el APD tenga un rango de sensibilidad óptimo para la transmisión (consulte el manual del producto) que es ideal para mantenerse, por ejemplo, 1000-2000 mV. Se pueden utilizar filtros N.D. para mantener la transmisión alrededor de este rango. La consideración principal es que la transmisión no esté cerca del punto de saturación de APD, ya que el atrapamiento puede aumentar la transmisión hasta el límite de saturación, lo que provoca la pérdida de datos.
10. Infusión de proteínas en la celda de flujo
- Apague el láser para preservar la vida útil de las nanoestructuras y haga funcionar la unidad de control de la bomba de jeringa y la interfaz de usuario del distribuidor para configurar la válvula y extraer la cantidad deseada de proteína (por ejemplo, 30 μL). Para la extracción con el tubo de diámetro interior pequeño, un caudal más bajo ayuda a garantizar que se extraigan todas las soluciones (~0,01 - 0,1 mL/min). Se pueden utilizar caudales más altos para reducir el tiempo de espera si se desea.
NOTA: El volumen óptimo de proteína a utilizar depende de lo valiosa que sea la proteína y de lo que se espera lograr en el experimento. La concentración típica de proteína que utilizamos es de 1 μM y un volumen alícuota de 100 μL. Si la muestra de proteína es abundante, se pueden utilizar concentraciones más altas para reducir el tiempo medio de captura. Sin embargo, si las concentraciones son demasiado altas, aumentará el riesgo de atrapar dos de las mismas proteínas dentro de la nanoestructura. Si el experimento requiere una infusión de un ligando/proteína diferente después de atrapar la proteína inicial, se puede preferir un volumen de alícuota más bajo para minimizar el desperdicio y el tiempo necesario para infundir la siguiente solución. Asegúrese de que el tubo permanezca sumergido en la solución para evitar que entre aire en el sistema. Se debe tener especial cuidado con las alícuotas de proteínas si el tubo no llega al fondo del recipiente o si la solución se extrae en exceso. - Usando la interfaz de usuario de microfluídica, infunda la solución de proteína a una velocidad de flujo similar a la tasa de extracción (~0,01 - 0,1 mL/min). Deje infundir a esta velocidad hasta que la solución de proteína llegue a la celda de flujo, luego reduzca el caudal a ≤0.001 mL / min. Verifique el volumen y el tiempo de la bomba de jeringa para asegurarse de que coincidan con el volumen esperado (p. ej., 1 min para un volumen de 1 mL a 1 mL/min).
NOTA: El volumen requerido para esto se puede calcular en función del volumen de la tubería desde el sistema microfluídico hasta la celda de flujo. Si se conocen la longitud y el diámetro interior de la tubería, se puede utilizar una calculadora en línea para el volumen de un cilindro. La velocidad de infusión se puede ajustar según se desee, por ejemplo, >0,001 mL/min para la infusión de proteínas para reducir el tiempo medio de espera de una trampa. Sin embargo, se recomienda precaución, ya que si el caudal es demasiado alto, esto puede evitar que las proteínas ingresen a la nanoestructura.
11. Recopilación de datos
- Mientras la solución de proteína se infunde en la celda de flujo, inicie el registro de datos de la señal APD. Ajuste los ejes x, y y z según sea necesario utilizando la interfaz de usuario del controlador piezoeléctrico, ya que es probable que el sistema se desvíe con el tiempo. Las trazas de reventado ideales tienen un patrón general coherente que sigue el de la traza de la Figura 3.
- Al observar un gran cambio en la transmisión y la S.D. similar a la traza de captura del ejemplo, anote el momento en que esto ocurre para la clasificación de datos futura (consulte la Figura 4B para ver el ejemplo).
NOTA: La deriva en el sistema puede provocar cambios en la transmisión y un aumento de la S.D., a veces de forma bastante repentina, lo que podría confundirse con una trampa de proteínas. Asegúrese de que el salto de señal muestre el mismo patrón que la trampa ejemplar y que no haya interferencias externas cuando se produjo el salto, como la deriva de alineación o los ruidos fuertes que capta el sistema. - Si es necesario liberar la proteína como parte del experimento, apague el láser durante ~ 5 s y vuelva a encenderlo. La traza debe tener un gran cambio en la transmisión y una S.D. significativamente más baja, lo que indica un retorno al estado de referencia.
NOTA: Si no se observa un gran cambio en la transmisión y/o la S.D., o se observa un rastro similar a la proteína que está atrapando, es probable que la proteína esté adherida a la superficie de la muestra (consulte la Figura 5B, por ejemplo).
12. Desmontaje de la muestra
- Después de realizar el experimento deseado, apague el láser, saque la celda de flujo de la etapa de 3 ejes y desconecte la tubería del sistema microfluídico.
- Coloque la celda de flujo sobre un pañuelo limpio con la capa de oro de la muestra hacia arriba. Con un bisturí, corte con cuidado el pegamento debajo del cubreobjetos de vidrio y levántelo suavemente con pinzas redondeadas. Deséchelo en el contenedor de vidrio roto designado.
PRECAUCIÓN: Los vidrios rotos y el uso de objetos afilados pueden causar lesiones. Asegúrese de usar gafas de seguridad, guantes y una bata de laboratorio. - Sostenga la celda de flujo en ángulo con la capa de oro aún hacia arriba y use pinzas redondeadas para quitar con cuidado el pegamento en la parte inferior de la celda de flujo para liberar la muestra. La silicona duplicada permite una fácil extracción sin dañar las estructuras de DNH.
- Con unas pinzas rectas, recoja la muestra y enjuague bien con IPA, luego riegue antes de secar con una pistola de aire. Si es reutilizable, almacene la muestra en un recipiente adecuado con la capa de oro hacia arriba.
NOTA: Las muestras suelen tener un lapso utilizable de alrededor de 1-2 semanas si se manejan correctamente, aunque el tamaño de las nanoestructuras cambia con el tiempo. Si está demasiado dañado, cambie a una muestra nueva para el siguiente experimento. El daño puede ser en forma de arañazos presentes en la superficie del oro cerca de las nanoestructuras o por un rendimiento experimental deficiente, lo que indica la degradación de las nanoestructuras29.
13. Preparación del sistema para su uso futuro
- Use un pañuelo de papel doblado para limpiar el aceite del objetivo en una dirección, levantándolo y repitiéndolo con otra parte limpia del pañuelo.
NOTA: Los objetivos son propensos a mancharse y rayarse, lo que puede afectar significativamente el rendimiento. Siempre use guantes durante la limpieza y evite tocar material orgánico. Use solo un pañuelo de limpieza de lentes nuevo para evitar la contaminación y limpie en una sola dirección con un solo movimiento. Evite frotar la lente y el pañuelo de limpieza de un lado a otro para minimizar el daño. Si la limpieza en seco no es suficiente, aplique una pequeña cantidad de etanol al pañuelo, limpie el aceite como se mencionó anteriormente y termine con un pañuelo seco. - Si a continuación se van a utilizar las mismas soluciones de proteína/tampón, es posible que no sea necesario cambiar el tubo. Si se utilizan diferentes soluciones, reemplace el tubo para reducir el riesgo de contaminación de soluciones anteriores.
NOTA: No use el mismo tubo durante más de 1 semana, incluso si realiza experimentos con las mismas soluciones, ya que se ensuciará con el tiempo.
Resultados
Después de la adquisición de datos, el análisis de datos se puede realizar en los datos sin procesar utilizando código de MATLAB para generar seguimientos a partir de los datos sin procesar recopilados por el APD. La Figura 3 muestra un ejemplo de traza de atrapamiento que incluye la línea de base antes del atrapado, el evento de atrapamiento donde se observa un gran cambio en la transmisión (ΔT / T0) y la desviación estándar antes de que el láser se apague durante aproximadamente 5 segundos antes de volver a encenderse. Una reducción significativa en la desviación estándar y el retorno de la transmisión a niveles similares a los basales indica la liberación de proteínas. El desfase lineal se elimina del rastreo mediante la función detrend.m de MATLAB y, a continuación, el valor medio de los datos se vuelve a agregar al rastreo sin tendencia. De vez en cuando, necesitamos eliminar la tendencia de la traza a medida que la configuración se desplaza con el tiempo, lo que provoca una disminución lineal en la transmisión (consulte la traza gris en la Figura 3). Los pequeños cambios en las trazas de referencia antes y después del reventado se deben al ajuste de la etapa para optimizar la línea de base con una desviación estándar mínima, como se muestra en la Figura 4A. A veces, las moléculas de proteína son visibles en el rastro sin ser atrapadas, lo que se denomina proteínas de paso. Las proteínas que pasan aparecen como un cambio brusco en la transmisión, similar a una trampa típica (Figura 4B), pero con una duración significativamente más corta, como se muestra en la Figura 4A. La densidad espectral de potencia (PSD) presenta otro análisis para confirmar el atrapamiento de proteínas al proporcionar intensidad de señal a varias frecuencias. Los movimientos conformacionales de las proteínas se observan típicamente en el rango de >1 μs mediante métodos de espectroscopia de molécula única40. La Figura 4C demuestra que, en comparación con la línea de base, el atrapamiento de una proteína conduce a una mayor intensidad de señal, al menos dentro del rango de 10 kHz (> 100 μs). También destaca la importancia de alinear la etapa con una línea de base optimizada, ya que una línea de base incorrecta podría aumentar el ruido a frecuencias entre 50 y 500 Hz, un rango de frecuencia superpuesto con movimientos conformacionales de proteínas.
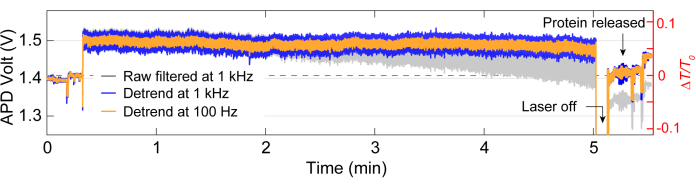
Figura 3: Traza de captura completa para una sola proteína. Traza representativa de una trampa completa, incluida la línea de base, la captura de una proteína y la liberación de la proteína. Los saltos en la traza antes y después del atrapado se deben a la alineación. Haga clic aquí para ver una versión más grande de esta figura.

Figura 4: Eventos de rastreo comunes. (A) Ejemplos de una alineación de una línea de base mala a una buena y una proteína que pasa cerca del punto caliente. (B) Traza de atrapamiento que muestra el proceso desde la línea de base cuando el punto caliente de DNH está vacío hasta cuando la proteína está atrapada. (C) Gráfico de densidad espectral de potencia (PSD) entre las líneas de base buenas y malas representadas en (A) y la proteína atrapada en (B). Los valores más altos de PSD indican un mayor ruido a frecuencias particulares. Haga clic aquí para ver una versión más grande de esta figura.
La mayoría de los eventos de reventado siguen el mismo patrón general que el seguimiento de la figura 3, aunque pueden surgir problemas ocasionales durante los experimentos. Para la mayoría de los experimentos, la proteína debe liberarse manualmente apagando el láser una vez que se complete el experimento deseado. En algunos casos, sin embargo, la proteína puede salir de la trampa sin intervención, como se muestra en la Figura 5A. Por el contrario, a veces las proteínas pueden permanecer en el sitio de captura incluso después de apagar el láser, probablemente debido a que la proteína se adhiere a la muestra. Este pegado da como resultado un rastro ruidoso después de apagar y encender el láser (consulte la Figura 5B). La probabilidad de que esto ocurra depende de la proteína, ya que algunas proteínas son más propensas a la adsorción superficial41,42. El uso de un recubrimiento como el PEG-tiol puede reducir las posibilidades de que las proteínas se peguen39,43. A menos que se desee, como el estudio de las interacciones proteína-proteína, otro problema es el doble atrapamiento, donde una segunda proteína queda atrapada después de la primera. Esto se caracteriza por otro aumento brusco en la transmisión, similar a la primera trampa, y un cambio en la desviación estándar (ver Figura 5C).
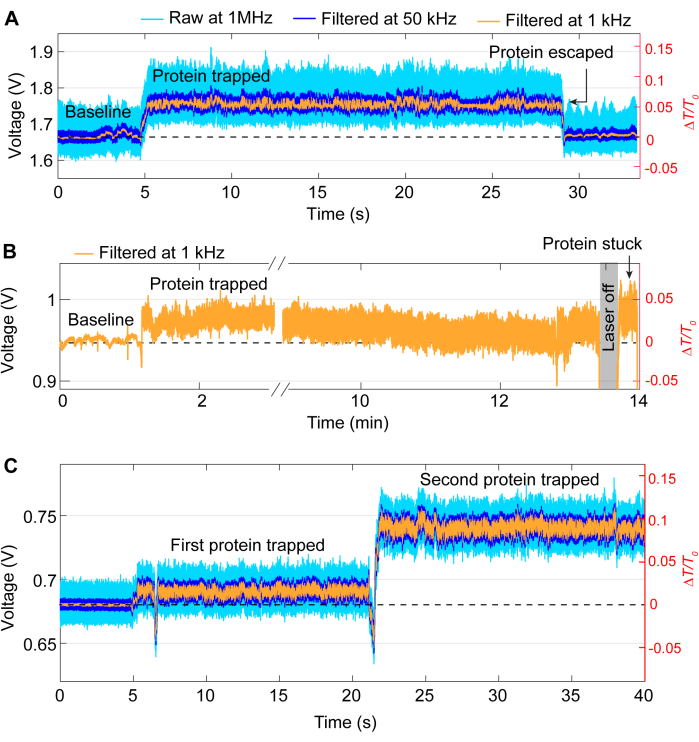
Figura 5: Ejemplos de eventos de atrapamiento no deseados. (A) Liberación no intencional de una proteína desde el punto caliente de DNH. (B) Ejemplo de proteína que se atasca en la superficie de la muestra en el punto caliente de DNH. (C) El salto de traza ocurre cuando una segunda proteína queda atrapada mientras la primera aún permanece en el punto caliente de DNH. Haga clic aquí para ver una versión más grande de esta figura.
Un experimento representativo llevado a cabo sobre la carga de hierro in situ en una molécula de apo-ferritina demuestra el uso de nanotweezers plasmónicas como herramienta para investigar la dinámica conformacional de las proteínas29. La ferritina es una proteína transportadora de hierro que existe en dos estados: la apo-ferritina, que no contiene hierro, y la holoferritina, que está llena de hierro44,45. El hierro ferroso ingresa a la proteína a través de canales 3 veces donde se oxida a hierro férrico y se almacena en el núcleo de la proteína46. La Figura 6A muestra una traza típica de atrapamiento de apo-ferritina con una solución ferrosa infundida durante más de 20 minutos mientras la proteína está atrapada. Las trazas de 20 s tomadas a lo largo de toda la traza en los puntos b-e proporcionan información sobre los cambios que se producen en la proteína a lo largo del tiempo. En la Figura 6B, la apo-ferritina está atrapada en un tampón PBS estándar y no se observan cambios significativos en la traza. La figura 6C, D muestra fluctuaciones en el S.D de las trazas, que son causadas por la carga de hierro en la proteína a través de sus 3 canales, lo que resulta en un estado más dinámico (apo-) donde los canales están abiertos, y un estado más compacto (holo-) con los canales cerrados. Al llenarse la molécula de ferritina con hierro, pasó a su holoforma, lo que resultó en un rastro de atrapamiento estable, como se muestra en la Figura 6E. Las funciones de densidad de probabilidad (PDF) en las Figuras 6B-E muestran aún más los cambios que experimenta la proteína al exponerse a diferentes condiciones de solución a lo largo del tiempo.

Figura 6: Carga de hierro in situ en una apoferritina atrapada. (A) Traza de transmisión completa de un DNH con una molécula de apoferritina atrapada, seguida de la inyección de una solución ferrosa en el sitio de captura para observar los cambios conformacionales de la ferritina asociados con la carga de hierro. (B) Traza de atrapamiento de 20 segundos de una apoferritina atrapada antes de que la solución ferrosa alcanzara el punto caliente. (C, D) Trazas de atrapamiento de 20 s después de que la molécula de apoferritina se expuso a la solución ferrosa. Los segmentos azul y púrpura marcan la S.D superior e inferior de la traza, lo que indica conformaciones flexibles y rígidas de ferritina, respectivamente. (E) Trazas de atrapamiento de 20 s después de que la apoferritina se expuso a la solución ferrosa durante >20 minutos. Los gráficos de la función de densidad de probabilidad (PDF) a la derecha muestran la distribución de la transmisión y están codificados por colores para los segmentos azul y morado. Esta cifra ha sido modificada de29. Haga clic aquí para ver una versión más grande de esta figura.
Figura complementaria 1: Muestra de oro DNH montada en la celda de flujo impresa en 3D. La muestra se coloca en una ranura especial y se adhiere a la celda de flujo con cinta adhesiva de PET de doble cara. Los parámetros clave y las mediciones asociadas para el diseño de nuestra celda de flujo están etiquetados. Haga clic aquí para descargar este archivo.
Figura complementaria 2: Parte posterior de la celda de flujo con muestra de DNH dorado montada y pared interior etiquetada. La muestra se sella en la celda de flujo utilizando silicona duplicada. Haga clic aquí para descargar este archivo.
Figura complementaria 3: Diagrama de la celda de flujo con DNH dorado montado con orificios de entrada y salida etiquetados. Haga clic aquí para descargar este archivo.
Discusión
Un paso crucial en el protocolo es garantizar que la celda de flujo no tenga fugas antes de colocarla en la etapa, que debe probarse fuera del soporte a un caudal alto de antemano. Las fugas después de montar la muestra pueden dañar los componentes ópticos, en particular el objetivo inferior.
La alineación puede desviarse de su posición óptima con el tiempo durante un experimento, causando una variación de la señal debido a la sensibilidad de las nanotpinzas plasmónicas. Cuando esto ocurre, realinee usando los controles piezoeléctricos a la transmisión máxima y la desviación estándar mínima de la línea de base, ya que una línea de base ruidosa reduce la calidad de los datos. Se debe tener cuidado de tomar notas meticulosas de cuándo se realizan las alineaciones para eliminar el riesgo de confundir la interferencia del usuario con un evento de atrapamiento. Si se produce una deriva importante, alinee suavemente la etapa para minimizar el riesgo de liberación de la proteína y anote el tiempo de ajuste.
Las modificaciones y alteraciones de la técnica presentada se pueden realizar en función de las necesidades experimentales específicas. Por ejemplo, una etapa de temperatura controlada puede ayudar a enfriar/calentar la muestra según se desee en lugar de utilizar el calentamiento láser para aumentar la temperatura31,33. Otras técnicas, por ejemplo, la microscopía de dispersión interferométrica (iSCAT), pueden proporcionar interferencia de la proteína dentro del campo de dispersión del DNH, obteniendo una señal adicional proporcional a la polarizabilidad de la proteína, que se asocia con el tamaño de partícula47,48.
Las nanopinzas plasmónicas son puramente una técnica de detección temporal, ya que los datos son registrados por el APD (un detector de un solo píxel). Esta técnica no proporciona información directa sobre los cambios estructurales de una proteína, como qué regiones de la proteína están involucradas en un cambio conformacional o dónde un ligando u otra proteína puede unirse a la proteína. Además, la técnica está limitada a un rango temporal de >1 μs debido a la frecuencia de muestreo de la tarjeta de adquisición de datos (1 MHz). Considerando la frecuencia de Nyquist, donde el rango más alto posible es la mitad de la tasa de adquisición, en este caso, 2 μs en condiciones perfectas.
En este protocolo, hemos descrito el proceso de configuración de un experimento de nanotweezers plasmónicos para atrapar una sola proteína y monitorear los cambios en su dinámica conformacional a lo largo del tiempo. La técnica se puede desarrollar en cualquier microscopio casero o comercial. A diferencia de los enfoques basados en fluorescencia o anclaje, como smFRET y pinzas ópticas, la técnica puede atrapar proteínas sin etiquetas ni ataduras y aún así lograr la sensibilidad de una sola molécula. Las condiciones de la solución se pueden cambiar mientras la proteína está atrapada mediante un sistema de microfluídica, lo que permite el seguimiento en tiempo real del efecto de las diferentes soluciones en la proteína. Estas características hacen que las nanopinzas plasmónicas sean una herramienta prometedora dentro de los campos de la biofísica y la biodetección, particularmente para las proteínas que las técnicas convencionales luchan por interrogar en sus estados nativos. Las aplicaciones futuras se centrarán en decodificar la dinámica conformacional de proteínas más dinámicas, como las proteínas intrínsecamente desordenadas y las proteínas que contienen regiones intrínsecamente desordenadas, y las proteínas de membrana, cuya dinámica e incluso estructura eluden las técnicas actuales.
Divulgaciones
Los autores no tienen nada que revelar.
Agradecimientos
S.Z. agradece el apoyo de la Asociación de Formación Doctoral del Consejo de Investigación en Biotecnología y Ciencias Biológicas (BBSRC DTP) (BB/T0083690/1). Los autores agradecen la financiación de la Iniciativa de Educación e Investigación del Reino Unido-India (UKIERI). M.R. agradece el apoyo de la Royal Society y de la Fundación Wolfson.
Materiales
| Name | Company | Catalog Number | Comments |
| 100x Objective (NA = 1.25) | Olympus | PLN100XO | Clean oil only with lens cleaning tissue in one motion |
| 12-1 rotary bidirectional microfluidic valve | Elveflow | ||
| 3/2 way solenoid valve | Elveflow | ||
| 4x Objective (NA = 0.1) | Olympus | PLN4XP | |
| Airgun | RS Components | 666-6772 | |
| Avalanche photodiode (APD) | Thorlabs | APD120A/M | Do not oversaturate, 50 MHz bandwidth, static sensitive |
| Butterfly laser diode | Thorlabs | FPL852S | |
| Charge-coupled device (camera) | Hikrobot | MV-CE200-10UC | |
| Crystallising dish | VWR | 216-0065 | |
| Data acquisition card | National instruments | USB-6361 | 2 MHz sampling rate |
| Double-sided tape | Adhesive Research Inc | ARcare92712 | Cut appropriate shape for flow cell with a laser cutter |
| Ethanol | Fume hood only, use gloves | ||
| Fibreport collimator | Thorlabs | PAF2-A7B | |
| Glass microscope coverslips (thickness 0.17 mm) | |||
| Half wave plate | Thorlabs | AHWP10M-980 | |
| Ideal-tek 120 mm, stainless steel, straight tweezers | RS Components | 282-7472 | For holding sample and peeling double sided tape |
| Isopropanol (IPA) | Fume hood only, use gloves | ||
| Lens cleaning tissue | Thorlabs | MC-5 | |
| Metrosil rapid duplicating silicone parts A and B | Metrodent | MSILR/1 | |
| Microscope slides (75 mm x 26 mm) | |||
| Modular Optical Tweezers System | Thorlabs | OTKB/M-CUSTOM | |
| Objective oil | Olympus | Use gloves, store between 2oC-8oC | |
| Photodiode laser mount | Thorlabs | CLD1015 | |
| Piezoelectric controller | Thorlabs | MDT693B | |
| Piezoelectric stage | Thorlabs | Nanomax 300 | |
| Planoconcave lens (f = -50 mm) | Thorlabs | LC1715-B | |
| Planoconvex lens (f = 150 mm) | Thorlabs | LA1433-B | |
| Planoconvex lens (f = 60 mm) | Thorlabs | LA1134-B | |
| Poly(ethylene glycol) methyl ether thiol (PEG-thiol) 800 MW | Sigma Aldrich | 729108 | Store between 2oC-8oC |
| PTFE tubing (ID: 0.18 mm) | Vici Jour | JR-T-6805-M25 | |
| PTFE tubing (ID: 0.8 mm) | Cole-Parmer | WZ-21942-72 | |
| SEM-FIB | Zeiss | Crossbeam 550 | Gallium ion source |
| Shortpass dichroic mirror 805 nm | Thorlabs | DMSP805 | |
| Silver mirror | Thorlabs | PF-10-03-P01 | |
| Syringe pump | Harvard Apparatus | 70-4511 | |
| Weller Erem 120 mm stainless steel, flat, rounded tweezers | RS Components | 176-1150 | For holding glass coverslip and peeling glue from flow cell |
Referencias
- Peter Lu, H. P. Sizing up single-molecule enzymatic conformational dynamics. Chem Soc Rev. 43 (4), 1118-1143 (2014).
- Mazal, H., Haran, G. Single-molecule FRET methods to study the dynamics of proteins at work. Curr Opin Biomed Eng. 12, 8-17 (2019).
- Ritchie, D. B., Woodside, M. T. Probing the structural dynamics of proteins and nucleic acids with optical tweezers. Curr Opin Str Biol. 34, 43-51 (2015).
- Neupane, K., Solanki, A., Sosova, I., Belov, M., Woodside, M. T. Diverse metastable structures formed by small oligomers of α-Synuclein probed by force spectroscopy. PLoS One. 9 (1), e86495 (2014).
- Hughes, M. L., Dougan, L. The physics of pulling polyproteins: a review of single molecule force spectroscopy using the AFM to study protein unfolding. Rep Prog Phys. 79 (7), 076601-076601 (2016).
- Young, G., et al. Quantitative mass imaging of single biological macromolecules. Science. 360 (6387), 423-427 (2018).
- Schmid, S., Dekker, C. Nanopores: a versatile tool to study protein dynamics. Essays Biochem. 65 (1), 93-107 (2021).
- Van Meervelt, V., et al. Real-time conformational changes and controlled orientation of native proteins inside a protein nanoreactor. J Am Chem Soc. 139 (51), 18640-18646 (2017).
- Awasthi, S., Ying, C., Li, J., Mayer, M. Simultaneous determination of the size and shape of single α-Synuclein oligomers in solution. ACS Nano. 17 (13), 12325-12335 (2023).
- Sµnchez-Rico, C., Von Vithenberg, L., Warner, L., Lamb, D. C., Sattler, M. Effects of fluorophore attachment on protein conformation and dynamics studied by spFRET and NMR spectroscopy. Chemistry. 23 (57), 14267-14277 (2017).
- Yin, L., et al. How does fluorescent labeling affect the binding kinetics of proteins with intact cells. Biosens Bioelectr. 66, 412-416 (2015).
- Berkovich, R., et al. Rate limit of protein elastic response is tether dependent. Proc Natl Acad Sci. 109 (36), 14416-14421 (2012).
- Dahmardeh, M., Mirzaalian Dastjerdi, H., Mazal, H., Köstler, H., Sandoghdar, V. Self-supervised machine learning pushes the sensitivity limit in label-free detection of single proteins below 10 kDa. Nat Meth. 20 (3), 442-447 (2023).
- Becker, J., et al. A quantitative description for optical mass measurement of single biomolecules. ACS Photon. 10 (8), 2699-2710 (2023).
- Houghtaling, J., et al. Estimation of shape, volume, and dipole moment of individual proteins freely transiting a synthetic nanopore. ACS Nano. 13 (5), 5231-5242 (2019).
- Plesa, C., et al. Fast translocation of proteins through solid state nanopores. Nano Lett. 13 (2), 658-663 (2013).
- Schmid, S., Stömmer, P., Dietz, H., Dekker, C. Nanopore electro-osmotic trap for the label-free study of single proteins and their conformations. Nat Nanotechnol. 16 (11), 1244-1250 (2021).
- Shi, X., Verschueren, D. V., Dekker, C. Active delivery of single DNA molecules into a plasmonic nanopore for label-free optical sensing. Nano Lett. 18 (12), 8003-8010 (2018).
- Verschueren, D. V., et al. Label-free optical detection of DNA translocations through plasmonic nanopores. ACS Nano. 13 (1), 61-70 (2019).
- Verschueren, D., Shi, X., Dekker, C. Nano-optical tweezing of single proteins in plasmonic nanopores. Small Meth. 3 (5), 1800465 (2019).
- Peri, S. S. S., et al. Quantification of low affinity binding interactions between natural killer cell inhibitory receptors and targeting ligands with a self-induced back-action actuated nanopore electrophoresis (SANE) sensor. Nanotechnology. 32 (4), 045501 (2020).
- Leong, I. W., Tsutsui, M., Yokota, K., Taniguchi, M. Salt gradient control of translocation dynamics in a solid-state nanopore. Anal Chem. 93 (49), 16700-16708 (2021).
- Juan, M. L., Righini, M., Quidant, R. Plasmon nano-optical tweezers. Nat Photon. 5 (6), 349-356 (2011).
- Juan, M. L., Gordon, R., Pang, Y., Eftekhari, F., Quidant, R. Self-induced back-action optical trapping of dielectric nanoparticles. Nat Phys. 5 (12), 915-919 (2009).
- Pang, Y., Gordon, R. Optical trapping of 12 nm dielectric spheres using double-nanoholes in a gold film. Nano Lett. 11 (9), 3763-3767 (2011).
- Pang, Y., Gordon, R. Optical trapping of a single protein. Nano Lett. 12 (1), 402-406 (2012).
- Kotnala, A., Gordon, R. Quantification of high-efficiency trapping of nanoparticles in a double nanohole optical tweezer. Nano Lett. 14 (2), 853-856 (2014).
- Booth, L. S., et al. Modelling of the dynamic polarizability of macromolecules for single-molecule optical biosensing. Sci Rep. 12 (1), 1995 (2022).
- Yousefi, A., et al. Optical monitoring of in situ Iron loading into single, native ferritin proteins. Nano Lett. 23 (8), 3251-3258 (2023).
- Yousefi, A., et al. Structural Flexibility and Disassembly Kinetics of Single Ferritin Molecules Using Optical Nanotweezers. ACS Nano. 18 (24), 15617-15626 (2024).
- Peters, M., et al. Energy landscape of conformational changes for a single unmodified protein. NPJ Biosens. 1 (1), 14-14 (2024).
- Peters, M., McIntosh, D., Branzan Albu, A., Ying, C., Gordon, R. Label-free tracking of proteins through plasmon-enhanced interference. ACS Nanosci Au. 4 (1), 69-75 (2024).
- Ying, C., et al. Watching single unmodified enzymes at work. arXiv. , (2021).
- Yang-Schulz, A., et al. Direct observation of small molecule activator binding to single PR65 protein. NPJ Biosensing. 2 (1), 1-10 (2025).
- Yang, W., van Dijk, M., Primavera, C., Dekker, C. FIB-milled plasmonic nanoapertures allow for long trapping times of individual proteins. iScience. 24 (11), 103237 (2021).
- Koya, A. N., et al. Novel plasmonic nanocavities for optical trapping-assisted biosensing applications. Adv Optical Mater. 8 (7), 1901481 (2020).
- Goswami, A., Umashankar, R., Gupta, A. K., Aravindan, S., Rao, P. V. Development of a microstructured surface using the FIB. J Micromanufact. 1 (1), 53-61 (2018).
- Onuta, T. D., Waegele, M., DuFort, C. C., Schaich, W. L., Dragnea, B. Optical field enhancement at cusps between adjacent nanoapertures. Nano Lett. 7 (3), 557-564 (2007).
- Al-Ani, A., et al. The influence of PEG-thiol derivatives on controlling cellular and bacterial interactions with gold surfaces. Appl Surf Sci. 462, 980-990 (2018).
- Schuler, B., Hofmann, H. Single-molecule spectroscopy of protein folding dynamics-expanding scope and timescales. Curr Opin Str Biol. 23 (1), 36-47 (2013).
- Kopac, T. Protein corona, understanding the nanoparticle-protein interactions and future perspectives: A critical review. Int J Biol Macromol. 169, 290-301 (2021).
- Li, X., Guo, W., Xu, R., Song, Z., Ni, T. The interaction mechanism between gold nanoparticles and proteins: Lysozyme, trypsin, pepsin, γ-globulin, and hemoglobin. Spectrochim Acta A Mol Biomol Spectrosc. 272, 120983 (2022).
- Emilsson, G., et al. Strongly stretched protein resistant poly(ethylene glycol) brushes prepared by grafting-To. ACS Appl Mater Inter. 7 (14), 7505-7515 (2015).
- Theil, E. C., Liu, X. S., Tosha, T. Gated pores in the ferritin protein nanocage. Inorg Chim Acta. 361 (4), 868-874 (2008).
- Theil, E. C., et al. The ferritin iron entry and exit problem. Inorg Chim Acta. 297 (1), 242-251 (2000).
- Takahashi, T., Kuyucak, S. Functional properties of threefold and fourfold channels in Ferritin D=deduced from electrostatic calculations. Biophys J. 84 (4), 2256-2263 (2003).
- Gemeinhardt, A., et al. Label-free imaging of single proteins secreted from living cells via iSCAT microscopy. J Vis Exp. (141), e58486 (2018).
- Piliarik, M., Sandoghdar, V. Direct optical sensing of single unlabelled proteins and super-resolution imaging of their binding sites. Nat Comm. 5 (1), 4495 (2014).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoExplorar más artículos
This article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados