Method Article
Identificação e classificação de variantes missense da subunidade do receptor GABAA específicas da posição para seu papel nos neurônios piramidais do hipocampo
Neste Artigo
Resumo
Este estudo apresenta uma estrutura multiescala, abrangendo desde o DNA até a função da proteína e o comportamento neural. Ele apresenta uma nova abordagem para investigar mutações patogênicas previstas na subunidade do receptor GABAA , levantando a hipótese de que mutações epileptogênicas e mutações proximais, previstas como patogênicas, podem produzir efeitos semelhantes no modelo de neurônio piramidal CA1.
Resumo
Compreender os efeitos de variantes funcionalmente desconhecidas em genes associados à epilepsia é crucial para elucidar a fisiopatologia da doença e desenvolver terapêuticas personalizadas. Com uma estrutura multiescala, abrangendo desde a sequência de DNA até a função da proteína e o comportamento neural, descrevemos uma nova abordagem para prever e investigar mutações patogênicas, levantando a hipótese de que mutações epileptogênicas na subunidade do receptor GABAA e mutações previstas próximas podem produzir efeitos semelhantes no modelo de neurônio piramidal CA1. Ao explorar as relações características entre mutações patogênicas previstas e mutações epileptogênicas proximais, o estudo visa estimar os efeitos das mutações previstas com base nos efeitos das mutações epileptogênicas nas simulações de neurônios piramidais do hipocampo.
A metodologia começa com a coleta de dados genéticos da subunidade γ2 do receptor GABAA , seguida de limpeza e formatação dos dados realizada em R usando um script personalizado. Em seguida, os preditores de conjunto serão aplicados para identificar e priorizar as variantes patogênicas missense da subunidade γ2 . O mapeamento de uma variante patogênica específica (prevista) para os domínios estruturais da subunidade compartilhados por mutações epileptogênicas será ilustrado, acompanhado de modelagem molecular de seus efeitos e consideração da conservação evolutiva. Em seguida, será realizada meta-análise específica da variante e normalização de parâmetros, seguida de análise de correlação para identificar quaisquer relações significativas entre as mutações previstas e as mutações epileptogênicas proximais. Usando um simulador neural baseado em Python, será descrito um modelo de neurônio baseado em condutância multicompartimental, refletindo o efeito de mutantes do tipo selvagem e epileptogênicos. A simulação das respostas neurais geradas pelo subtipo de receptor GABAA epileptogênico será considerada para a estimativa aproximada do efeito das variantes patogênicas previstas na resposta neural. Até onde sabemos, este é o primeiro protocolo que explora uma estrutura multiescala para estimar os efeitos das variantes do receptor GABAA no comportamento neuronal, crucial para a pesquisa da epilepsia. Este protocolo pode servir como base para melhorar as previsões de fenótipos celulares causados por variantes potencialmente patogênicas dos receptores GABAA associados à epilepsia.
Introdução
Para quase todas as doenças humanas, a variação genética desempenha um papel significativo na suscetibilidade individual. Portanto, entender como as variações de sequência se relacionam com o risco de doença oferece uma maneira valiosa de descobrir os principais processos envolvidos no desenvolvimento da doença e identificar novas abordagens para prevenção e tratamento1. Isso também se aplica aos distúrbios do neurodesenvolvimento, que estão entre as condições médicas crônicas mais prevalentes na atenção primária pediátrica2. Condições como transtorno do espectro autista, deficiência intelectual e epilepsia ilustram como a variação genética influencia significativamente a suscetibilidade individual durante o desenvolvimento3.
O cérebro em desenvolvimento é mais suscetível a crises epilépticas do que o cérebro adulto devido à incompatibilidade de neurodesenvolvimento geneticamente programada no equilíbrio crítico entre excitação e inibição4. Como o GABA (ácido gama-aminobutírico), o principal neurotransmissor inibitório no cérebro adulto, é excitatório durante o desenvolvimento embrionário e pós-natal inicial, isso não é favorável à estabilidade necessária para prevenir convulsões em cérebros jovens. Esse estado temporário, causado pela falta de expressão suficiente de cotransportadores K-Cl5, pode contribuir para um risco aumentado de atividade convulsiva na presença de receptores GABAA disfuncionais. Os receptores GABAA medeiam as ações excitatórias e inibitórias do GABA, dependendo da concentração intracelular do íon Cl-6. Assim, à medida que o cérebro amadurece, mutações nos genes codificadores do receptor GABAA, bem como em outros canais iônicos, distorcem a excitabilidade, e mutações em genes envolvidos no metabolismo neuronal, sinalização celular e formação de sinapses7 podem causar condições como epilepsia do tipo ausência da infância8.
As intervenções clínicas estão cada vez mais alavancando a análise genética para melhorar a precisão no tratamento de distúrbios do neurodesenvolvimento2. O teste genético na epilepsia pediátrica apresenta alvos potenciais para abordagens de medicina de precisão9, destacando a importância das variantes genéticas na orientação das decisões de tratamento. Além disso, ~ 25% dos pacientes com epilepsia com mutações de novo recebem diagnósticos genéticos que identificam alvos potenciais para a medicina de precisão, ressaltando o valor significativo das variantes genéticas na orientação das decisões de tratamento10. Isso foi alimentado por avanços nas tecnologias de sequenciamento de próxima geração, como painéis de genes direcionados, sequenciamento de exoma completo e sequenciamento de genoma completo, que aceleraram drasticamente as descobertas genéticas11. No entanto, o número crescente de novas descobertas de genes vem com um desafio quando os resultados produzem uma variante de significado desconhecido (VUS), uma classificação que reflete evidências conflitantes ou informações insuficientes sobre o papel molecular da variante na patogênese da doença. As variantes classificadas como VUS correspondem a uma categoria dentro do sistema de classificação de variantes de cinco níveis proposto pelo American College of Medical Genetics and Genomics (ACMG) e pela Association for Molecular Pathology (AMP)12.
Enfrentar o desafio de variantes genéticas funcionalmente desconhecidas requer esforços em duas dimensões principais: prática clínica e pesquisa. Clinicamente, a incerteza em torno da USV pode complicar o manejo e a tomada de decisãodo paciente 13. Do ponto de vista da pesquisa científica, identificar variantes patogênicas entre o número crescente de variantes de significado incerto e determinar seus papéis na fisiopatologia da doença e nos efeitos fenotípicos são cruciais1. Um cenário ideal envolveria prever com precisão os efeitos moleculares, neuronais e de nível de rede de todas as variantes funcionalmente não caracterizadas, minimizando assim os recursos, tempo e esforço necessários para investigações baseadas em laboratório. Esses aspectos ressaltam a importância de classificar com precisão as variantes genéticas para permitir o diagnóstico preciso de epilepsias genéticas, apoiar o tratamento personalizado e facilitar a descoberta de potenciais alvos farmacológicos. As ferramentas preditivas atuais 14,15,16,17 são relativamente precisas, mas normalmente fornecem apenas classificações binárias (patogênicas vs. benignas) e carecem de informações específicas da doença sobre fisiopatologia molecular, consequências fenotípicas e mecanismos subjacentes. Concentrando-se nas variantes missense desconhecidas de genes codificadores de subunidades do receptor GABAA selecionados, este artigo apresenta uma estrutura destinada a melhorar a orientação da pesquisa, incorporando fatores contextuais de variantes como aspectos moleculares, evolutivos e estruturais, bem como simulações de patologia neural derivadas de dados biofísicos in vitro de mutações associadas à epilepsia. Nossa metodologia aborda a identificação de variantes patogênicas desconhecidas da subunidade γ2 do receptor GABAA, uma subunidade-chave envolvida na fisiopatologia da epilepsia 18,19,20. Isso é seguido pela exploração da correspondência específica da posição dessas variantes previstas com as mutações associadas à epilepsia caracterizadas por dados estruturais e eletrofisiológicos. Esses dados são então usados para estimar o efeito variante em um modelo de neurônio piramidal hipocampal expressando um subtipo de receptor GABAA, composto por subunidades γ2, α1 e β3 (receptores γ2-GABAA), responsáveis pela rápida inibição sináptica6. É importante notar que os receptores GABAA se reúnem a partir de um grande pool de subunidades (α1-α6, β1-β3, γ1-γ3, δ, Ε, θ, π e ρ1-ρ3) e, dependendo da composição da subunidade, os receptores GABAA diferem em sua modulação, características biofísicas, bem como padrões de expressão regional, celular e subcelular acoplados a funções específicas 6,21,22,23, 24,25. Assim, o presente estudo se concentra apenas nos receptores γ2-GABAA ou γ2 contendo GABAA.
As subunidades do receptor GABAA são compostas por características estruturais características - um longo domínio extracelular N-terminal (ECD), quatro domínios transmembranares (TM1 a TM4), um ligante intracelular conectando o TM1 e o TM2, um ligante extracelular conectando o TM2 e o TM3, uma grande alça intracelular entre o TM3 e o TM4 (alça TM3-TM4) e um terminal C extracelular curto 6,26, 27. Sugere-se que o receptorGABA A funcione por meio de um mecanismo complexo de "bloqueio e puxão", onde a ligação do GABA bloqueia as subunidades β e α, fazendo com que elas puxem os domínios extracelulares (ECDs) das subunidades, girando-as no sentido anti-horário27. Esse movimento dobra os domínios transmembranares (TMDs), abrindo assim o canal iônico27. Assim, a atividade do canal parece ser coordenada junto com estruturais dentro dos receptores GABAA. Acontece que as mutações da epilepsia causam disfunção na atividade do canal por meio da distorção desses estruturais28. Consequentemente, nosso estudo é baseado na ideia de que variantes patogênicas preditas nas proximidades de mutações epileptogênicas funcionalmente identificadas nos estruturais específicos das subunidades do receptor GABAA podem exibir padrões semelhantes de distorção eletrofisiológica ou biofísica na função do canal, como observado nos casos dessas mutações epileptogênicas. Embora a presença de estruturais epileptogênicos nas subunidades do receptor GABAA 28 apoie indiretamente essa noção, nosso estudo demonstra a complexidade e o desafio de correlacionar parâmetros biofísicos de mutações epileptogênicas com os de mutações patogênicas previstas. Para desmascarar essas relações complexas, nossa estrutura é significativa, pois destaca uma abordagem multiescala que vai do DNA à função da proteína e ao comportamento neural crítico para a pesquisa da epilepsia. Essa abordagem integra genética computacional com modelagem molecular e simulações neurais, ao mesmo tempo em que enfatiza a importância de métodos complementares, como aprendizado de máquina treinado em grandes conjuntos de dados, que podem capturar os efeitos das mutações na estrutura do canal, atividade e excitabilidade neural. Além disso, a simulação da atividade do receptor γ2-GABAA epileptogênico no modelo de neurônio piramidal do hipocampo permite a replicação do fenótipo celular in vitro associado à canalopatia do receptor GABAA e a demonstração de respostas alteradas de neurônio único no centro da disfunção da rede.
Protocolo
1. Predição in silico de variantes patogênicas
- Coleta de dados de variantes
- Usando o banco de dados ClinVar29, pesquise variantes de significado incerto (VUS) na região codificadora do gene de interesse através do site: https://www.ncbi.nlm.nih.gov/clinvar/. Digite o símbolo do gene (por exemplo, GABRG2) na barra de pesquisa e filtre os resultados para incluir apenas os tipos desejados de variantes, como nucleotídeo único, variantes missense com significado incerto. Baixe e salve os dados como data.xlxs (Arquivo Suplementar 4: Tabela Suplementar S1). Registre a data dos dados baixados.
NOTA: No presente protocolo, será analisada a subunidade γ2 humana do receptor GABAA, especificamente a subunidade do receptor do ácido gama-aminobutírico tipo A gama2 (GABRG2), variante de transcrição 1, mRNA (NCBI Ref. seq.: NM_198904.4), também conhecida como γ2L. É importante registrar o transcrito de referência do gene de interesse, bem como outros identificadores correspondentes em diferentes bancos de dados (UniProt, ENSEMBL, PDB), uma vez que diferentes métodos computacionais podem exigir identificadores diferentes (Arquivo Suplementar 4: Tabela Suplementar S2). Caso o banco de dados ou ferramenta computacional não reconheça os números de versão dos identificadores de sequência, tente o ID com o número da versão (NM_198904.4) e sem o número da versão (NM_198904). - Informações básicas sobre proteínas de referência
- No https://www.ncbi.nlm.nih.gov/ do banco de dados do NCBI, selecione Nucleotídeo nas opções de pesquisa e insira o NCBI Ref. seq. ID do gene de interesse (NM_198904.4). Em seguida, rolando para baixo na coluna da direita, clique na Proteína na categoria Informações relacionadas para encontrar a proteína (NP_944494.1) codificada pela transcrição NM_198904.4. Usando as informações fornecidas para a proteína NP_944494.1, registre as posições de sequência das regiões específicas na forma de uma tabela (Arquivo Suplementar 4: Tabela Suplementar S3).
NOTA: É importante determinar as informações preliminares conhecidas para a posição da sequência de regiões, motivos ou resíduos funcional e estruturalmente críticos, como domínios de proteínas, locais de fosforilação, locais de ligação de ligantes e interfaces de interação molecular. Isso pode ser alcançado combinando banco de dados (NCBI, ENSEMBL, UniProt...) e pesquisas bibliográficas.
- No https://www.ncbi.nlm.nih.gov/ do banco de dados do NCBI, selecione Nucleotídeo nas opções de pesquisa e insira o NCBI Ref. seq. ID do gene de interesse (NM_198904.4). Em seguida, rolando para baixo na coluna da direita, clique na Proteína na categoria Informações relacionadas para encontrar a proteína (NP_944494.1) codificada pela transcrição NM_198904.4. Usando as informações fornecidas para a proteína NP_944494.1, registre as posições de sequência das regiões específicas na forma de uma tabela (Arquivo Suplementar 4: Tabela Suplementar S3).
- Usando o banco de dados ClinVar29, pesquise variantes de significado incerto (VUS) na região codificadora do gene de interesse através do site: https://www.ncbi.nlm.nih.gov/clinvar/. Digite o símbolo do gene (por exemplo, GABRG2) na barra de pesquisa e filtre os resultados para incluir apenas os tipos desejados de variantes, como nucleotídeo único, variantes missense com significado incerto. Baixe e salve os dados como data.xlxs (Arquivo Suplementar 4: Tabela Suplementar S1). Registre a data dos dados baixados.
- Organização de dados variantes
- Organize os dados para atender aos requisitos de entrada para os preditores escolhidos. Certifique-se de que o formato dos dados recuperados esteja organizado para corresponder aos requisitos do servidor dbNSFP http://database.liulab.science/dbNSFP. Para fazer isso, remova colunas desnecessárias do arquivo data.xlsx (Arquivo Suplementar 4: Tabela Suplementar S1 da etapa 1.1.1), mantendo apenas as seguintes colunas na ordem especificada:
"Cromossomo GRCh38", "Localização GRCh38", "Nome", "Alteração de proteína". - Salve o arquivo com um novo nome de arquivo: "data1.xlsx" (Tabela Suplementar S4). Formate o arquivo data1.xlsx em R executando o código (Arquivo Suplementar 1: Data_GABAA. R), que salvará os dados formatados como data1_output.xlsx (Arquivo Suplementar 4: Tabela Suplementar S5) no diretório de trabalho relevante para o projeto R.
NOTA: Diferentes métodos computacionais requerem diferentes tipos e formatos de dados. Coletar e organizar dados de acordo com requisitos de formato específicos, mesmo para uma dúzia de variantes, pode ser propenso a erros e demorado, portanto, essa etapa é importante, a menos que o pool de variantes seja composto por apenas algumas variantes. Então, a organização manual de dados pode ser possível.
- Organize os dados para atender aos requisitos de entrada para os preditores escolhidos. Certifique-se de que o formato dos dados recuperados esteja organizado para corresponder aos requisitos do servidor dbNSFP http://database.liulab.science/dbNSFP. Para fazer isso, remova colunas desnecessárias do arquivo data.xlsx (Arquivo Suplementar 4: Tabela Suplementar S1 da etapa 1.1.1), mantendo apenas as seguintes colunas na ordem especificada:
- Predição de patogenicidade
- Transfira o conteúdo do arquivo data1_output.xlsx para a versão acadêmica do servidor dbNSFP30,31 acessado via http://database.liulab.science/dbNSFP. Para fazer isso, copie/cole ou carregue diretamente o arquivo em .txt formato.
- Certifique-se de que as seguintes opções sejam pré-selecionadas e confirmadas no servidor: HG38 (build do genoma), ClinPred32 e BayesDEL33 antes do envio. Em alguns minutos, o servidor gerará os resultados.
NOTA: No presente protocolo, dois preditores de conjunto, BayesDEL33 e ClinPred32, foram selecionados para alta precisão34 e praticidade. No entanto, outros preditores, como o AlphaMissense, que está disponível no banco de dados dbNSFP 30,31 também podem ser selecionados. A seleção de ferramentas in silico depende de vários fatores, incluindo a geração de várias linhas suficientes de evidências computacionais para uma previsão poderosa12. Preditores de conjunto integrando a análise de vários algoritmos preditivos podem servir a esse propósito. - Baixe o arquivo de saída (um formato .txt) e salve-o como data2.xlsx (Arquivo Suplementar 4: Tabela Suplementar S6).
- Defina os filtros em data2.xlsx (Arquivo Suplementar 4: Tabela Suplementar S6) clicando na opção de filtro no menu e determinando as variantes de consenso em ambas as colunas filtrando por D. Isso fornecerá a lista das variantes mais patogênicas; salve-o (consulte a guia Consenso na Tabela Suplementar S6 [ Arquivo Suplementar 4]).
- Seleção de variantes
- Dentre as predições patogênicas consensuais, determinar as variantes na proximidade de mutações epileptogênicas obtidas da literatura. Certifique-se de que estes últimos tenham parâmetros estruturais e biofísicos adequados para modelagem de neurônios.
NOTA: Esta etapa é exploratória e também está relacionada ao levantamento da proteína de interesse em termos de seus parâmetros estruturais, físico-químicos e biofísicos. No presente estudo, esses dados foram obtidos de Brünger et al.35 e Guo et al.36, além de um levantamento de mutações associadas à epilepsia. Além disso, como opção, os escores do AlphaMissense37 foram acessados a partir do banco de dados dbNSFP30,31 repetindo a etapa 1.3 (Arquivo Suplementar 4: Tabela Suplementar S7). Mais detalhes são fornecidos nas seções 2.1.1 e 2.1.2 do protocolo e nos Resultados (consulte "Variantes de agrupamento para parâmetros estruturais e biofísicos"). - Para visualização básica, use os servidores Protter38 ((https://wlab.ethz.ch/protter/start/) e HOPE39 (https://www3.cmbi.umcn.nl/hope/) para examinar as variantes na etapa anterior no contexto de mutações selecionadas do gene GABRG2 : P302L40 e K328M (ou K289M41, ao excluir o peptídeo sinal de 39 resíduos).
NOTA: Devido à enorme complexidade, a avaliação estrutural dos efeitos das variantes deve ser conduzida em vários níveis de análise. Ferramentas como o Protter38 permitirão a visualização clara das variantes no contexto das características topológicas da proteína e servidores fáceis de usar, como o HOPE39 , fornecerão informações sobre o efeito da variante por modelagem molecular. Além disso, uma revisão abrangente da literatura sobre a proteína de interesse é fundamental para identificar e integrar as informações sobre mutações associadas à epilepsia. - Análise da conservação evolutiva e insights estruturais
- Abra o Jalview 42,43,44, um programa de código aberto para edição, visualização e análise de proteínas.
- Importe sequências para alinhamento. Clique em Arquivo no menu superior | Buscar sequências; selecione o banco de dados na caixa de diálogo (como UniProt); clique na guia recuperar IDs; e, conforme descrito na caixa de diálogo, insira os IDs de acesso UniProt do gene de interesse (GABRG2) de espécies humanas e de outros vertebrados: P18507, P22723, Q6PW52, A0A2I3TKX0, F1RR72, A0A8I3MDZ2, A0A8M1P4D6. Clique em OK.
NOTA: Os números de acesso UniProt de proteínas codificadas por GABRG2 são os seguintes: P18507 (P18507-2) para Homo sapiens, P22723 para Mus musculus, A0A2I3TKX0 para Pan troglodytes, F1RR72 para Sus scrofa, A0A8I3MDZ2 para Canis familiaris e A0A8M1P4D6 para Danio rerio. - Dependendo do gene de interesse, algumas sequências podem não ser anotadas; portanto, realize uma pesquisa BLAST para identificar informações relevantes e potenciais homólogos para uma melhor compreensão contextual. Nesse caso, carregue o formato FASTA de sequências de proteínas por meio da opção Adicionar sequências/caixa de texto De no menu Arquivo para produzir vários alinhamentos de sequência das sequências desejadas.
- Depois que o alinhamento for carregado, observe as sequências exibidas para comparação de várias sequências. Cada linha representa uma sequência e cada coluna representa uma posição no alinhamento. Para determinar o melhor método de alinhamento, use diferentes abordagens; por exemplo, clique nos serviços da Web no menu Sequência e selecione a opção Executar T-Coffee com predefinição , que permite o alinhamento ideal.
- Clique com o botão direito do mouse na sequência P18507 Homo sapiens (a sequência de referência no presente estudo) e defina-a como a sequência de referência. Escolha Formatar no menu superior e clique em Wrap para a visualização do alinhamento completo na tela. No mesmo menu Formatar, clique na escala acima para aprimorar a visualização de números de resíduos específicos. Para melhorar ainda mais a visualização, ajuste os esquemas de cores acessando Cor e selecionando diferentes opções (por exemplo, Cor Clustal, Propriedade Química); Modifique o tamanho da fonte, se necessário.
- Clique em Calcular na barra de menus e selecione Calcular consenso automaticamente para destacar as regiões conservadas.
- Concentre-se na posição das variantes de interesse identificadas na etapa de previsão in-silico e examine as posições específicas das variantes. Anote resíduos específicos clicando com o botão direito do mouse neles e selecionando Adicionar anotação. Escreva o rótulo (por exemplo, ID da variante) com o código de cor apropriado e salve.
NOTA: Na presente análise, P302L (roxo) e A303T (vermelho) foram selecionados para visualizá-los no alinhamento de sequências múltiplas junto com os dados estruturais (consulte a próxima seção).
- Reconstrução tridimensional da proteína completa mostrando os resíduos conservados selecionados
- No arquivo obtido na etapa anterior, clique com o botão direito do mouse na sequência de referência (GABRG2 humano) e selecione os dados da estrutura 3D.
- Identifique os dados estruturais apropriados (7QNE, Cadeia C)26 no menu suspenso e selecione Abrir nova visualização de estrutura com Jmol.
NOTA: Isso permitirá a incorporação dos resíduos selecionados no alinhamento de sequências múltiplas nos dados estruturais pelo Jmol, um visualizador baseado em Java de código aberto para estruturas químicas 3D.
- Dentre as predições patogênicas consensuais, determinar as variantes na proximidade de mutações epileptogênicas obtidas da literatura. Certifique-se de que estes últimos tenham parâmetros estruturais e biofísicos adequados para modelagem de neurônios.
2. Seleção de parâmetros e modelação biofísica
- Meta-análise específica da variante e normalização de parâmetros
- Pesquise a literatura atual para reunir variantes de subunidades identificadas com condutância eletrofisiológica do canal de dados (gGABAA), tempo de desativação (τ desativação), tempo de subida (τaumento) e amplitude máxima da corrente (Imáx.). Forneça as composições de subunidades, tipo de célula e medidas de tipo selvagem para cada caso. Rotule as variantes e seus controles de acordo (por exemplo, conhecido para variantes com características biofísicas identificadas e controle conhecido para as medições do tipo selvagem para cada variante).
- Obtenha pontuações de patogenicidade AlphaMissense para variantes com características biofísicas identificadas.
NOTA: Consulte a seção 1.3 do protocolo para obter mais detalhes. - Crie um quadro de dados com subunidade e posição de aminoácidos para cada variante, os aminoácidos originais e alterados, pontuação de patogenicidade e parâmetros biofísicos obtidos da literatura. Para evitar discrepâncias experimentais, normalize os parâmetros biofísicos para variantes identificadas como mudanças de x vezes em medições de tipo selvagem.
- Análise comparativa de variantes por características estruturais e funcionais
- Organize as variantes previstas em um quadro de dados; rotular de acordo (por exemplo, previsto para variantes sem literatura disponível sobre suas características biofísicas).
- Classifique as variantes por sua localização na sequência de aminoácidos e estrutura terciária. Adicione parâmetros de classificação estrutural (por exemplo, localização em alfa-hélices, bobinas, folhas beta, domínios extracelulares, intracelulares ou transmembrana, revestimento de poros, ligação de agonistas, interações proteína-proteína) no quadro de dados e forneça informações para cada variante em relação à sua posição de aminoácido.
- Classifique as variantes por sua distância ao centro da membrana e ao eixo dos poros. Adicione os parâmetros distância ao eixo dos poros e distância ao centro da membrana no quadro de dados.
- Analise a correlação entre parâmetros estruturais e biofísicos em relação a variantes conhecidas. Se possível, avalie as variantes previstas em relação às correlações obtidas.
- Construção de modelos de sinapses e neurônios
- Use o Brian245, um simulador neural de código aberto desenvolvido em Python para modelar e simular redes neurais de pico, para construir um modelo biofísico multicompartimental da sinapse GABAérgica em um neurônio piramidal do hipocampo baseado em condutância multicompartimental.
- Projete o modelo baseado em condutância definindo a cinética de passagem do canal iônico, parâmetros passivos e ativos e condutâncias pós-sinápticas. Defina o modelo baseado em condutância conforme fornecido no Arquivo Suplementar 2, que descreve as equações usadas no modelo.
- Defina a capacitância da membrana (Cm) como 1 μF / cm2 e a resistência intracelular (Ra) como 200 Ω.cm.
- Use as condutâncias modificadas do tipo Hodgkin-Huxley para neurônios piramidais do hipocampo39 com gL = 0,0003 S / cm2, gK = 0,036 S / cm2, EL = -76,5 mV, ENa = 50 mVe EK = -90 mV.
- Ajuste a distribuição de densidade dos canais de NaV sobre gNa como 0,05 S / cm2 para soma, 0,5 S / cm2 para segmento inicial de axônio (AIS) e nó de Ranvier (NR) e 0,005 S / cm2 para dendritos. Defina gK e gNa como 0 em segmentos mielinizados.
- Construa a cinética de passagem do canal iônico para NaV e KV conforme descrito no Arquivo Suplementar 2.
- Introduza as correntes sinápticas (Isyn) como a soma de todas as sinapses glutamatérgicas e GABAérgicas em um compartimento. Inclua a corrente mediada pelo receptor AMPA rápida (IAMPA) e a corrente mediada pelo receptor NMDA lenta (INMDA) na corrente glutamatérgica (Iglu). Inclua apenas corrente rápida mediada pelo receptor GABAA na corrente GABAérgica (IGABA). Suponha que uma quantidade constante de glutamato seja liberada para a sinapse para cada pico pré-sináptico; portanto, a ativação dos receptores é dependente do tempo de pico (sAMPA e sNMDA) e as condutâncias totais do receptor (gAMPA e gNMDA) refletem a quantidade de glutamato que é liberada por cada evento.
- Use o modelo sináptico conforme descrito no Arquivo Suplementar 2.
NOTA: Para obter uma explicação detalhada das equações, consulte o Arquivo Suplementar 2 que descreve as equações usadas no modelo.
- Obtenha o diâmetro medido experimentalmente para soma e neuritos e o comprimento de cada compartimento de neuritos e padrões de ramificação da literatura anterior46,47. Reduza a morfologia real do neurônio em um modelo multicompartimental, dividindo a célula em vários compartimentos, que preserva com precisão a estrutura de ramificação principal e mantém a simetria bilateral.
- Defina o morfológico (comprimento e diâmetro do segmento; ou seja, d_soma: 30 μm; l_AH: 5 μm; d_AH_i: 1,5 μm; d_AH_f: 1,3 μm; l_AIS: 40 μm; d_axon: 1 μm; l_myseg: 100 μm; l_NR: 2 μm; l_AxTer: 4 μm; d_AxTer: 2 μm; l_approx: 100 μm; l_apmed: 100 μm; l_apdis: 200 μm; d_approx_i: 4 μm; d_approx_f: 3 μm; d_apmed : 2 μm; d_apdis: 2 μm; l_apLM: 70 μm; d_apLM: 2 μm; l_nAcDbasal: 400 μm; d_nAcDbasal: 1,4 μm; l_nAcDbasal_stem: 20 μm; d_nAcDbasal_stem: 1,5 μm) e parâmetros biofísicos (conforme indicado na seção 2.3.2) para cada compartimento do modelo de neurônio piramidal46,47, conforme também detalhado no script Python (Arquivo Suplementar 3: GABAAvar.py).
- Determine os parâmetros biofísicos para o modelo de sinapse GABAérgica avaliando as medições de controle do tipo selvagem obtidas na etapa 2.1.1.
- Projetar a topologia do modelo de neurônios e atribuir parâmetros morfológicos e biofísicos, o que inclui especificar o arranjo espacial e as interconexões dos compartimentos, com base nas informações morfológicas e de ramificação obtidas anteriormente. Atribua os parâmetros morfológicos (por exemplo, comprimento e diâmetro do segmento) e biofísicos apropriados (Seção 2.3.2) a cada compartimento do modelo, conforme descrito no Arquivo Suplementar 3: GABAAvar.py.
- Construção de sinapses e injeção de corrente
- Crie a atividade pré-sináptica usando SpikeGeneratorGroup (uma classe da biblioteca Brian2) conforme fornecido em "GABAAvar.py" (Arquivo Suplementar 3). Conecte o gerador de pico ao compartimento alvo do neurônio modelo usando a classe Synapses para modelar conexões sinápticas.
- Defina uma corrente constante sustentada (Iinj) como 0,85 nA e coloque no soma para imitar a atividade subliminar impulsionada pela carga de corrente iônica de linha de base em um determinado momento, conforme descrito no Arquivo Suplementar 3: GABAAvar.py.
- Para criar monitores de gravação, registre rastreamentos de tensão de compartimentos de destino usando StateMonitor.
- Construa e execute a rede.
- Construa a rede com o neurônio modelo, conexões e monitores usando Network.
- Defina o intervalo de tempo da simulação por defaultclock.dt (por exemplo, 0,01 ms).
- Execute a simulação na rede com network.run(T*ms), em que T é definido como 1.000 ms no exemplo.
- Testando o impacto das mutações missense do receptor GABAA
- Defina o impacto de cada mutação missense na cinética do canal por meio dos parâmetros biofísicos coletados na etapa 2.1.1.
- Execute a estimulação alterando esses parâmetros e plote os resultados usando "matplotlib.pyplot" conforme fornecido em "GABAAvar.py" (Arquivo Suplementar 3).
- Teste combinações de parâmetros para analisar as mudanças nos padrões e taxas de disparo. Plote os resultados para comparações.
Resultados
Este estudo utiliza uma abordagem multiescala para prever e caracterizar as variantes patogênicas na subunidade γ2 do receptor GABAA , um componente chave na fisiopatologia da epilepsia. Por meio do uso de modelos preditivos, modelagem molecular, conservação evolutiva, exame estrutural, análise de correlação e simulações neurais, essa abordagem aprimora a classificação de variantes, com relevância significativa para a pesquisa de epilepsia e possivelmente para uso clínico. O resumo geral da metodologia é apresentado na Figura 1.
Avaliação comparativa de duas mutações adjacentes da subunidade γ2
Com base em nossa suposição de que mutações patogênicas previstas adjacentes a mutações epileptogênicas em subunidades do receptor GABAA podem produzir efeitos eletrofisiológicos semelhantes na função do canal e no comportamento neural, primeiro conduzimos um breve exame da relação entre uma mutação epileptogênica bem conhecida e uma mutação predita proximal da subunidade γ2.
Entre as variantes previstas como patogênicas (Tabela Suplementar S6), A303T (rs1581439874, ClinVar Accession: VCV000663033.6) é selecionada como exemplo. Além da previsão por modelos de conjunto, a patogenicidade de A303T foi confirmada pelos escores AlphaMissense (Arquivo Suplementar 4: Tabela Suplementar S7). A303T está no segundo domínio transmembrana da subunidade γ2 do receptor GABAA e localizado próximo à mutação epileptogênica P302L40, conforme mostrado na Figura 2. Conforme avaliado por modelagem molecular, as substituições γ2P302L e γ2A303T resultaram em aminoácidos com cadeias laterais maiores, conforme mostrado na Figura 3. Tanto o resíduo mutante quanto o do tipo selvagem são apolares na mutação γ2P302L, enquanto no γ2A303T, o resíduo mutante tem uma cadeia lateral polar e o resíduo do tipo selvagem tem uma cadeia lateral apolar. Tanto o P302 quanto o Ala303 estão localizados na interface de interação da subunidade com a subunidade β3 (observada em 7QNB e 7QNA, respectivamente). Tanto o P302 quanto o Ala303 têm área de superfície acessível ao solvente (SASA) comparável. Além disso, ambos os resíduos são 100% conservados ao longo da evolução dos vertebrados (Figura 4, painel superior). Ambos estão localizados nas proximidades da segunda região transmembrana (domínio TM2) da subunidade γ2, como mostrado em amarelo na reconstrução tridimensional da proteína receptora GABAA (7QNE26, onde A303, mostrado em vermelho, é o primeiro resíduo neste domínio (Figura 4). Com base nessas características comparáveis e usando um modelo de neurônio piramidal, a simulação de mutação epileptogênica proximal, como a mutação da subunidade γ2 P302L40 , pode ser usada para a caracterização preliminar do efeito da variante prevista (γ2A303T) na resposta neural. Na próxima etapa, expandimos nossa análise para um conjunto mais amplo de variantes dentro das subunidades do receptor GABAA .
Variantes de agrupamento para parâmetros estruturais e biofísicos
Após a avaliação comparativa de duas mutações adjacentes na seção anterior, implementamos uma abordagem sistemática para avaliar se características moleculares compartilhadas entre as variantes poderiam ser identificadas. Esta fase teve como objetivo explorar se padrões consistentes emergem em características estruturais, físico-químicas e biofísicas entre os aminoácidos e variantes, fornecendo assim mais suporte para nossa hipótese inicial.
O quadro de dados usado neste estudo e as referências são fornecidos no Arquivo Suplementar 4: Tabela Suplementar S840, 48,49,50,51,52,53,54,55,56,57,58,59,60, 61,62,63, Tabela SuplementarS936 e Tabela SuplementarS1035. Além disso, as correlações entre os parâmetros estruturais e biofísicos foram determinadas para cada subunidade e para todas as variantes sem distinção de subunidade (Arquivo Suplementar 4: Tabela Suplementar S11, Tabela Suplementar S12, Tabela Suplementar S13, Tabela Suplementar S14 e Tabela Suplementar S15). Informações sobre parâmetros estruturais (localização em alfa-hélices, bobinas, folhas beta; domínios extracelulares, intracelulares ou transmembrana; revestimento de poros, ligação agonista/alostérica e interações proteína-proteína) foram obtidas de Brünger et al.35. Os parâmetros biofísicos foram obtidos a partir de estudos de eletrofisiologia patch-clamp em células do Rim Embrionário Humano (HEK) 293. Os valores foram normalizados com as respectivas constantes de tempo de ativação do receptor selvagem (τr) e tempo de desativação (τd).
Uma vez que nosso estudo é baseado na ideia de que variantes de aminoácidos adjacentes ou próximas a mutações funcionalmente identificadas em subunidades do receptor GABAA podem exibir padrões semelhantes de alterações eletrofisiológicas na função do canal, como observado em casos dessas mutações, exploramos a possibilidade de uma relação entre parâmetros estruturais, físico-químicos e biofísicos. As localizações das variantes em relação às suas distâncias do centro da membrana e do eixo dos poros são dadas na Figura 5 e na Figura 6. Nesse contexto, também foram utilizados os escores (Arquivo Suplementar 4: Tabela Suplementar S7) do AlphaMissense37; alimentado pelo modelo de previsão de estrutura de proteína altamente preciso AlphaFold264, que pode utilizar a sequência básica de aminoácidos como entrada. O AlphaMissense pode fornecer pistas para os aspectos estruturais das substituições de aminoácidos únicos. A distribuição das pontuações AlphaMissense para variantes conhecidas (pretas) e previstas (vermelhas) em relação à posição da variante (posição dos aminoácidos, distância ao centro da membrana e distância ao eixo dos poros) das subunidades do receptor GABAA (subunidades α1, β3, γ2 codificadas pelos genes GABRA1, GABRB3, GABRG2, respectivamente) é dada na Figura 7.
A Figura 7A-C mostra a distribuição do escore AlphaMissense entre as posições dos aminoácidos, a Figura 7D-F mostra a distribuição do escore AlphaMissense na distância normalizada do eixo dos poros e a Figura 7G-I mostra a distribuição do escore AlphaMissense na distância do centro da membrana. A análise de correlação na Figura 7 indicou a dificuldade de determinar uma relação subjacente por meio de propriedades estruturais para prever o resultado para variantes recém-identificadas. As variantes da subunidade b2 (codificadas pelo gene GABRB2) foram incluídas nas seções de agrupamento e correlação para poder realizar uma análise mais ampla. No entanto, apenas as variantes da subunidade α1 codificada por GABRA1 (Figura 7A, D, G), subunidade β3 codificada por GABRB3 (Figura 7B, E, H) e subunidade γ2 codificada por GABRBG2 (Figura 7C, F, I) foram incluídas nos modelos biofísicos, uma vez que o modelo se concentra na função de um neurônio piramidal do hipocampo e na combinação α1β3γ2 de GABAA subunidades receptoras é a combinação mais difundida no hipocampo65. Da mesma forma, quaisquer variantes de α1, β3 ou γ2 para as quais a cinética do canal não tenha sido estudada em um receptor α1β3γ2 GABAA também foram excluídas das simulações. Houve uma correlação leve (Figura Suplementar S1 e Figura Suplementar S2) entre os escores AlphaMissense e os parâmetros biofísicos (tempos normalizados de ativação e desativação) derivados dos efeitos das mutações do receptor GABAA (Arquivo Suplementar 4 e Tabela Suplementar S8) na presente análise. Isso sugere que as mutações previstas como patogênicas (com base nas pontuações do AlphaMissense) também podem levar a mudanças mensuráveis e potencialmente perturbadoras na cinética do receptor (por exemplo, tempos de ativação e desativação). No entanto, a falta de correlação entre as correlações posicionais na Figura 7 dificulta a utilização dos escores do AlphaMissense para nossa suposição, que se baseia na ideia de que os aminoácidos adjacentes terão consequências semelhantes para as características biofísicas.
As distribuições da distância normalizada ao eixo dos poros em relação à cinética de ativação e desativação das variantes conhecidas são mostradas na Figura 8 e na Figura 9. Há uma correlação leve para a subunidade γ2 (Figura 8C), sugerindo a possibilidade de que nossa hipótese, que se baseia na suposição de que aminoácidos adjacentes terão consequências semelhantes, possa ser verdadeira em algumas regiões, especificamente na proximidade da área de poros do canal receptor, o domínio TM2. Esta região é adjacente à nossa mutação epileptogênica de referência (Figura 2 e Figura 4; γ2P302), tornando-a uma candidata relativamente boa para simulações neurais. Com base nisso, uma estimativa aproximada dos efeitos de mutações previstas adjacentes, como γ2A303T (Figura 2 e Figura 4) pode ser feita. Nossos resultados aqui apresentados consideram apenas as medidas em α1β3γ2; portanto, as variantes avaliadas em nosso modelo foram restritas às variantes fornecidas no Arquivo Suplementar 4: Tabela Suplementar S16.
Efeito de mutações na inibição mediada pelo receptor GABA A do disparo de neurônios piramidais CA1
O efeito das mutações na inibição mediada pelo receptor GABAA é demonstrado em um modelo de neurônio piramidal CA1 baseado em condutância multicompartimental. O impacto das variantes missense do receptor GABAA na função do neurônio piramidal do hipocampo pode ser explorado através do desvio GABAérgico de entradas apicais para o neurônio, a partir das projeções de CA3 e neurônios piramidais do córtex entorrinal (EC) III. Em outras palavras, uma maneira de simular a atividade dos receptores GABAA é assumir um contexto no qual a simulação represente suposições realistas sobre o significado fisiológico dos receptores, como a inibição do shunt, um dos mecanismos de inibição GABAérgica. Os neurônios piramidais do hipocampo CA1, tipicamente em seus dendritos apicais, têm receptores GABAA nessas zonas, que são direcionados pelas projeções dos neurônios do CA3 e EC III. Este arranjo é, portanto, adequado para a simulação. Esta questão de pesquisa requer um projeto de entrada com atrasos e intensidades variados. Portanto, três diferentes sinapses glutamatérgicas (GluS1/2/3) foram colocadas nos dendritos apical distal, apical medial e basal, conforme mostrado na Figura 10, e foram ativadas sequencialmente. Para avaliar o impacto das entradas sinápticas, a amplitude da corrente constante deve permanecer abaixo do limite mínimo de disparo de pico (Iinj < Imin). O modelo de neurônio piramidal com receptor GABAA do tipo selvagem ou mutante foi iniciado com uma injeção de corrente constante de 0,85 nA no soma. A sinapse GABAérgica foi então colocada no soma. A atividade pré-sináptica, imitada pelo gerador de picos, foi iniciada primeiro no dendrito apical distal. As entradas sinápticas nos dendritos apicais medial e basal foram atrasadas em 25 ms e 50 ms, respectivamente. A sinapse GABAérgica foi ativada com um atraso de 40 ms. A intensidade da inibição GABAérgica foi ajustada de forma que todo o trem de picos, exceto o primeiro pico, seja inibido. Em seguida, o impacto das variantes é explorado neste cenário, variando τaumento,τ desativação e gGABAA .
Os parâmetros para receptores selvagens e mutantes foram obtidos a partir da coleção descrita na etapa 2.1.1 do protocolo, especificamente para receptores compostos por α1β3γ2, que é a composição de subunidade mais abundante nos neurônios piramidais do hipocampo65. A distribuição dos parâmetros é dada no Arquivo Suplementar 4: Tabela Suplementar S16.
Cada mutação de subunidade foi testada em casos de sinapse glutamatérgica única, dupla e tripla. Em uma abordagem simples, o impacto das mutações pode ser avaliado sobre a taxa e o padrão de disparo. As médias e o desvio padrão de ΔtISI também podem ser estimados para avaliar ainda mais as mudanças no padrão de disparo, onde ΔtISI representa a mudança no intervalo entre picos. Os resultados para cada caso são dados como taxas de disparo e ΔtISI (média e desvio padrão) no Arquivo Suplementar 4: Tabela Suplementar S17 e Figura Suplementar S3. Os trens de pico e traços de tensão para as variantes que alteraram os padrões de disparo são fornecidos na Figura 11, Figura 12 e Figura 13.
Para ativação de sinapse glutamatérgica simples (GluS1) e tripla (GluS1-2-3), as mutações que alteraram a resposta dos neurônios foram apenas β3mutações N110D e γ2K328M. No caso de entrada glutamatérgica única, β3N110D levou a inibição prejudicada, e o padrão de disparo foi travado no trem de pico glutamatérgico após o 4ºpico pré-sináptico com um pequeno atraso (Figura 11). γ2K328M também prejudicou a inibição, embora apenas em torno do5º pico pré-sináptico, e introduziu um atraso maior no pico pós-sináptico em comparação com β3N110D (Figura 11). No caso de ativação de GluS1-2-3 (Figura 13), a resposta foi semelhante entre as mutações β3N110D e γ2K328M. Ambos os mutantes produziram um padrão de disparo onde quase todos os picos pré-sinápticos acumulados foram detectados e desencadearam uma resposta. Em ambos os casos, os modelos de neurônios dispararam com um par de espinhos em resposta à atividade pré-sináptica.
A dupla ativação da sinapse glutamatérgica produziu resultados distintos em comparação com as outras duas configurações (Figura 12). Neste caso, duas mutações na subunidade b3 do receptor GABAA (β3N110D e β3T288N) e duas mutações na subunidade γ2 do receptor GABAA (γ2P302L e γ2K328M) prejudicaram a inibição GABAérgica. O modelo de neurônio com mutante γ2P302L disparou quase em sincronia com o GluS2, que provavelmente foi uma resposta atrasada ao GluS1 com aproximadamente o mesmo atraso de picos pré-sinápticos entre GluS1-2. A mutação β3T288N produziu um resultado semelhante, com a distinção do segundo pico ainda em sincronia com GluS2. O modelo de neurônio com o mutante N110D β3respondeu a quase todas as entradas glutamatérgicas acumuladas, exceto para os dois primeiros picos pré-sinápticos de GluS1 / 2, que foram introduzidos com umΔt ISI mais curto. O padrão de disparo para γ2K328M foi novamente como β3N110D, com a distinção de segundo e terceiro picos pré-sinápticos sendo perdidos.
Esses resultados demonstram os diversos efeitos das mutações da subunidade b3 (codificada pelo gene GABRB3 ) e da subunidade γ2 (codificada pelo GABRG2) na atividade do neurônio piramidal do hipocampo. Curiosamente, as mutações β3L170R, β3A305V, β3E180G, β3D120N, β3Y302C e γ2R82Q não produziram nenhuma alteração na atividade neural. O comprometimento mais grave na inibição foi para β3N110D e γ2K328M, ambos comdesativação de τ significativamente menor e maioraumento de τ. Nossa análise preliminar também mostrou que mudanças noaumento de τ ou gGABAA por si só não são suficientes para prejudicar a inibição (dados não mostrados). Pode-se argumentar que as mutações que levam à diminuição significativa dadesativação do τ, juntamente com o aumento do τ, levam a um comprometimento mais significativo na inibição GABAérgica.
No caso em que todas as entradas excitatórias devem ser inibidas, qualquer mutação que resulte em disparo produzirá respostas neurais anormais e inespecíficas, que têm o potencial de serem exageradas em um circuito neural composto por neurônios com as mesmas mutações. O equilíbrio de excitação/inibição em uma rede neural pode ser significativamente afetado pelo feedback inibitório prejudicado resultante, que é um componente crucial de qualquer atividade de rede.
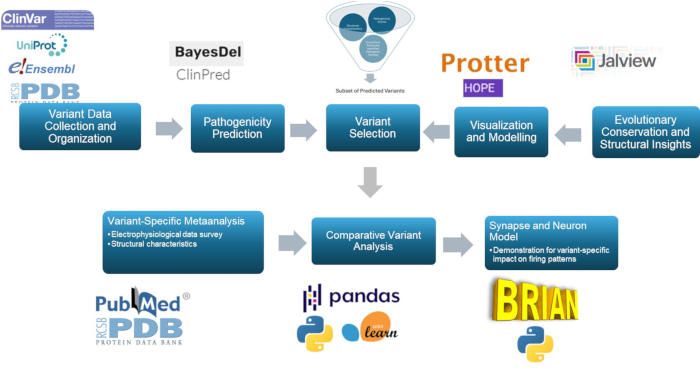
Figura 1: Visão geral da previsão e análise de efeitos variantes para fins clínicos e de pesquisa, com foco específico em análise in silico e simulações de resposta neural. Clique aqui para ver uma versão maior desta figura.
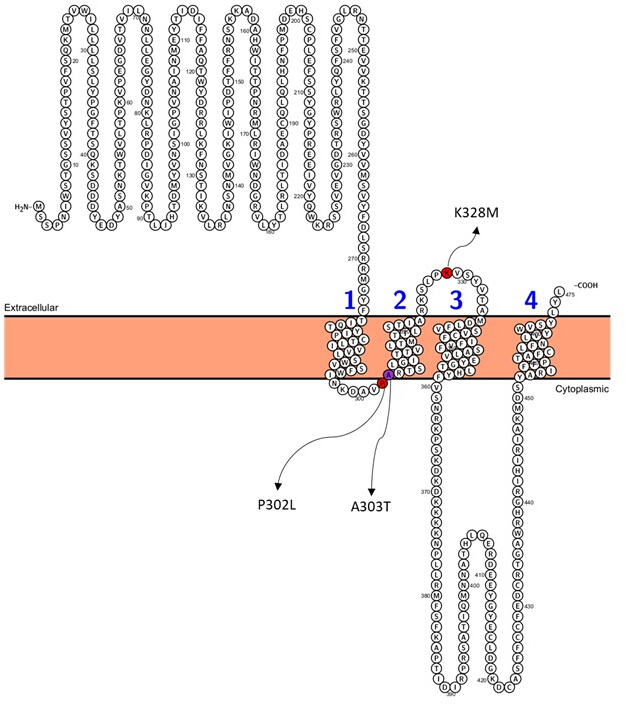
Figura 2: Posição do γ2A303T e mutações selecionadas do paciente;γ2P302L e γ2K328L usados para simulações neurais. Clique aqui para ver uma versão maior desta figura.
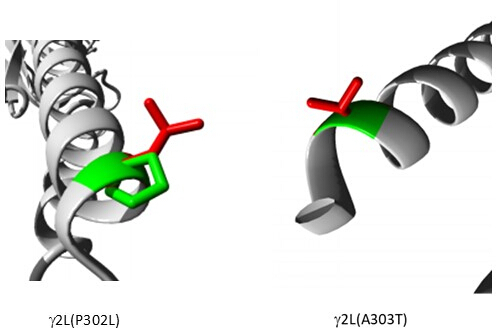
Figura 3: Modelagem comparativa da mutação do paciente γ2P302L e da variante adjacente γ2L (A303T) prevista como patogênica. Em ambos os modelos, o verde representa o tipo selvagem e o vermelho representa os resíduos mutantes. Clique aqui para ver uma versão maior desta figura.
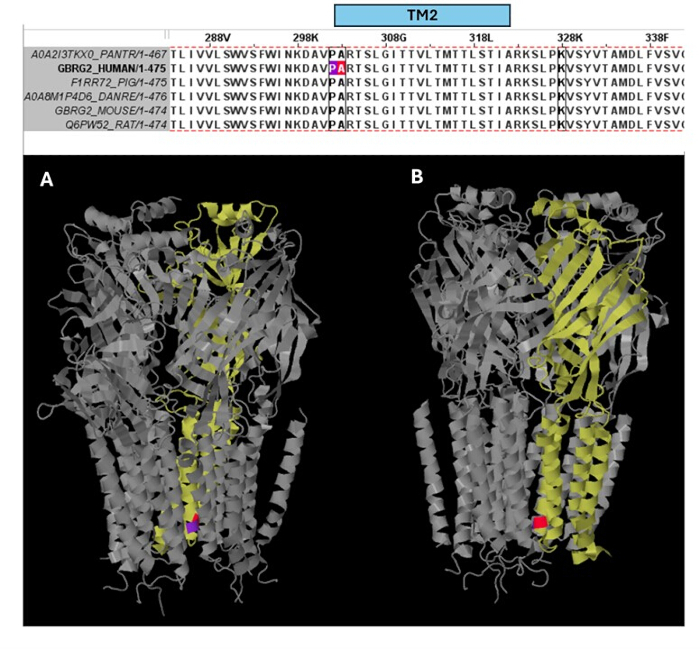
Figura 4: Alinhamento de múltiplas sequências e insights estruturais. O painel superior mostra a conservação evolutiva dos resíduos na posição da mutação do paciente (P302L) (roxo) na borda do TM2 e da variante patogênica A303T (cor vermelha) no início do TM2 da subunidade γ2. O painel inferior mostra a visualização desses resíduos conservados na estrutura tridimensional do receptor GABAA (7QNE) (A), onde a subunidade γ2 (Cadeia C no 7QNE) é mostrada em amarelo e de diferentes ângulos (A, B). Abreviatura: TM2 = segundo domínio transmembrana. Clique aqui para ver uma versão maior desta figura.
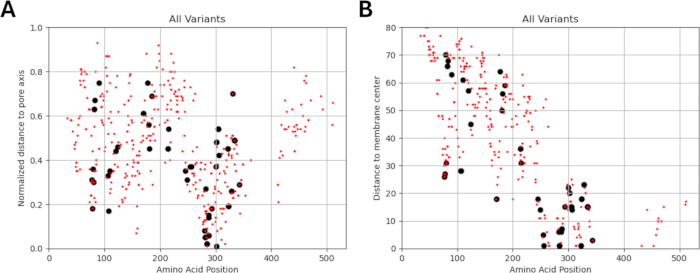
Figura 5: Localização de todas as variantes incluídas. As localizações das variantes conhecidas (pretas) e previstas (vermelhas) em relação à sua (A) distância normalizada do eixo dos poros e (B) distância do centro da membrana são mostradas. Clique aqui para ver uma versão maior desta figura.
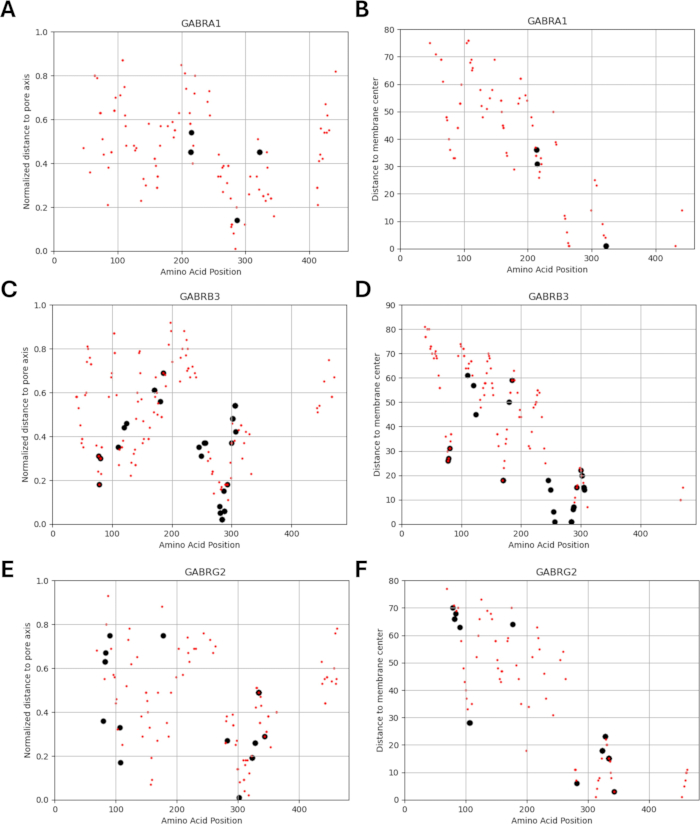
Figura 6: Localização de variantes para cada subunidade. As localizações das variantes conhecidas (pretas) e previstas (vermelhas) em relação à sua distância normalizada (A, C, E) do eixo dos poros e (B, D, F) distância do centro da membrana são mostradas. Clique aqui para ver uma versão maior desta figura.
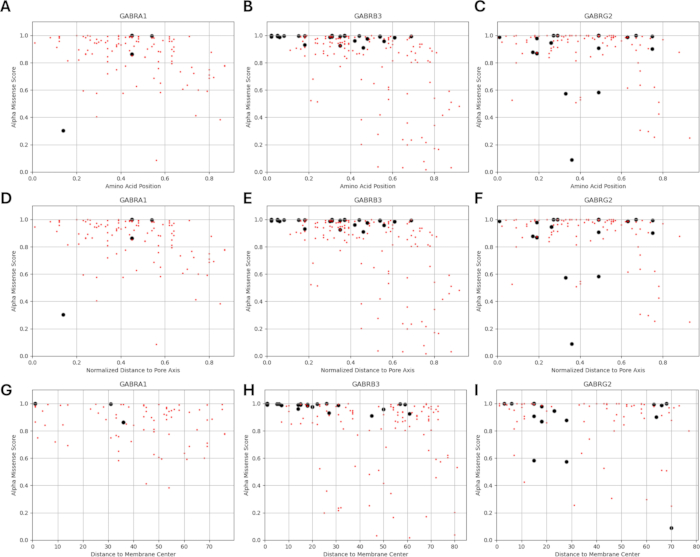
Figura 7: Distribuição da pontuação AlphaMissense sobre a localização da variante. (AC) A distribuição da pontuação AlphaMissense sobre a posição do aminoácido, (DF) distância normalizada do eixo dos poros e (GI) distância do centro da membrana são fornecidas para variantes conhecidas (pretas) e previstas (vermelhas). Clique aqui para ver uma versão maior desta figura.
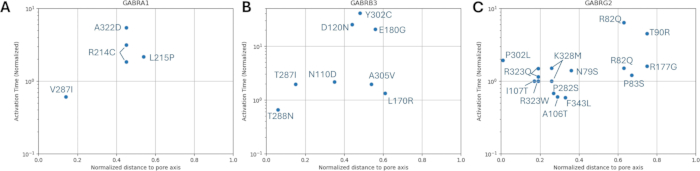
Figura 8: Tempo de ativação normalizado dos receptores GABAA com mutações na subunidade α1 (GABRA1), subunidade β3 (GABRB3) e subunidade γ2 (GABRG2). As constantes de tempo de ativação obtidas experimentalmente em relação à distância normalizada do eixo dos poros para cada mutação nas subunidades (A) α1, (B) β3 e (C) γ2 são exibidas. Os valores foram normalizados com o respectivo tempo de ativação do receptor selvagem. Clique aqui para ver uma versão maior desta figura.
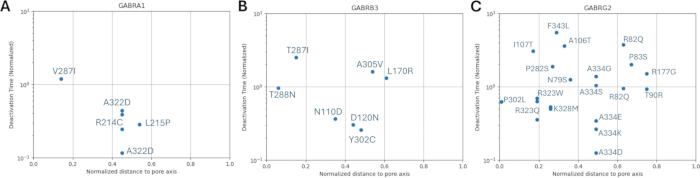
Figura 9: Tempo de desativação normalizado dos receptores GABAA com mutações na subunidade α1 (GABRA1) do receptor GABAA , subunidade β3 (GABRB3) do receptor GABAA e subunidade γ2 (GABRG2). As constantes de tempo de desativação obtidas experimentalmente em relação à distância normalizada do eixo dos poros para cada mutação nas subunidades (A) α1, (B) β3 e (C) γ2 são exibidas. Os valores foram normalizados com o respectivo tempo de desativação do receptor selvagem. Clique aqui para ver uma versão maior desta figura.

Figura 10: Modelo de neurônio piramidal CA1. O neurônio modelo consiste em (1) soma, (2) um dendrito apical com compartimentos proximal, medial e distal, terminando com dois ramos na lâmina molecular, (3) dois dendritos basais simetricamente compostos que se ramificam em duas seções após um curto caule dendrítico basal e (4) um axônio que começa com uma colina axônica cônica, seguida por um segmento inicial do axônio cilíndrico, segmentos mielinizados e nódulos de Ranvier, terminando com um terminal axônico esférico. Os triângulos verdes indicam a localização das sinapses glutamatérgicas e o triângulo vermelho representa a sinapse GABAérgica localizada no soma. Barra de escala = 100 μm. Clique aqui para ver uma versão ampliada desta figura.
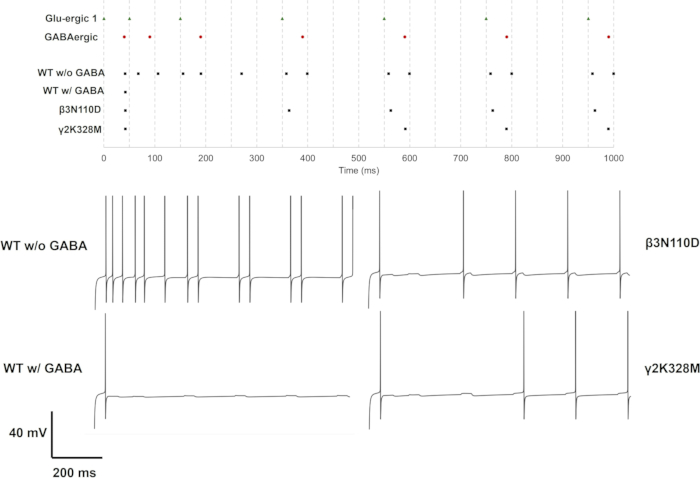
Figura 11: Padrão de disparo apenas com atividade GluS1. Os trens de pico para neurônios pré-sinápticos (GluS1 (triângulo verde) e GABAérgicos (círculo vermelho)) e os neurônios pós-sinápticos com receptores GABAA do tipo selvagem ou mutantes (quadrado preto) são fornecidos no painel superior. Traços de voltagem individuais para neurônios com receptor GABAA do tipo selvagem com ou sem inibição GABAérgica e para neurônios com receptores GABAA mutantes com inibição GABAérgica são exibidos nos painéis inferiores. Clique aqui para ver uma versão maior desta figura.

Figura 12: Padrão de disparo apenas com atividade GluS1 e GluS2. Os trens de pico para neurônios pré-sinápticos (GluS1 / 2 (triângulo verde) e GABAérgicos (círculo vermelho)) e os neurônios pós-sinápticos com receptores GABAA do tipo selvagem ou mutantes (quadrado preto) são fornecidos no painel superior. Traços de voltagem individuais para neurônios com receptor GABAA do tipo selvagem com ou sem inibição GABAérgica e para neurônios com receptores GABAA mutantes com inibição GABAérgica são exibidos nos painéis inferiores. Clique aqui para ver uma versão maior desta figura.
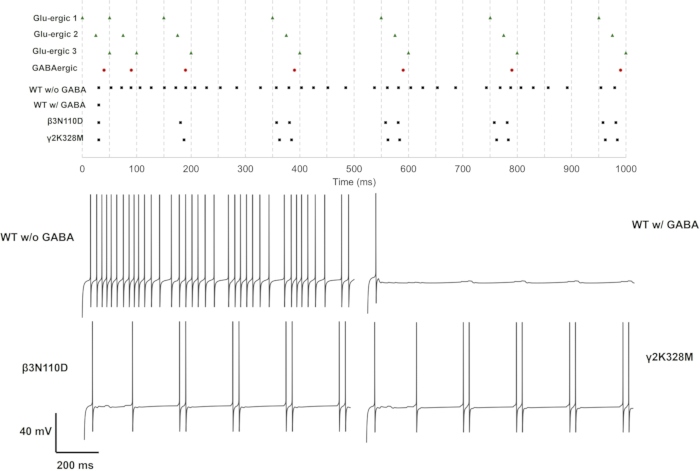
Figura 13: Padrão de disparo apenas com atividade GluS1, GluS2 e GluS3. Os trens de pico para neurônios pré-sinápticos (GluS1 / 2/3 (triângulo verde) e GABAérgicos (círculo vermelho) e os neurônios pós-sinápticos com receptores GABAA do tipo selvagem ou mutantes (quadrado preto) são fornecidos no painel superior. Traços de voltagem individuais para neurônios com receptor GABAA do tipo selvagem com ou sem inibição GABAérgica e para neurônios com receptores GABAA mutantes com inibição GABAérgica são exibidos nos painéis inferiores. Clique aqui para ver uma versão maior desta figura.
Figura Suplementar S1: Distribuição dos escores AlphaMissense e parâmetros biofísicos (Tempo de desativação normalizado; τd) normalizado de mutações na subunidade do receptor GABAA selecionadas no presente estudo. Veja também o Arquivo Suplementar 4: Tabela Suplementar 8. Clique aqui para baixar esta figura.
Figura Suplementar S2: Distribuição dos escores AlphaMissense e parâmetros biofísicos (Tempo de ativação normalizado; τr) normalizado de mutações na subunidade do receptor GABAA selecionadas no presente estudo. Veja também o Arquivo Suplementar 4: Tabela Suplementar 8. Clique aqui para baixar esta figura.
Figura S3 suplementar: Os intervalos entre espinhos para resposta neural com receptores GABAA do tipo selvagem e mutantes. O gráfico superior indica os intervalos de tempo entre espinhos para entrada glutamatérgica única. O gráfico do meio mostra dois, e o gráfico mais baixo mostra três sinapses glutamatérgicas ativas simultaneamente. Clique aqui para baixar esta figura.
Arquivo Suplementar 1: O arquivo "Data_GABAA. R" necessário para ser executado em R para formatação de dados. Clique aqui para baixar este arquivo.
Arquivo Suplementar 2: Equações usadas no projeto do Modelo Baseado em Condutância. Clique aqui para baixar este arquivo.
Arquivo Suplementar 3: GABAAvar.py necessário para executar no Brian2 para Simulação Neural. O arquivo contém os códigos Python para o modelo de neurônio multicompartimental baseado em Brian2 (função: CA1_Pyr), equações para modelos de neurônios e sinápticos baseados em condutância (função: model_eqns, syn_eqns) e parâmetros iniciais (função: biophys_param, morpho_param, syn_param). Clique aqui para baixar este arquivo.
Arquivo Suplementar 4: Uma pasta zip contendo todas as Tabelas Suplementares. Clique aqui para baixar este arquivo.
Tabela Suplementar S1: Variantes missense de significado desconhecido no gene GABRG2 baixadas do ClinVar como .txt arquivo e posteriormente salvas como "data.xlxs". Clique aqui para baixar esta tabela.
Tabela Suplementar S2: Identificadores das sequências usadas no estudo, o transcrito de referência do gene de interesse (NCBI Ref. seq.) e outros identificadores correspondentes em diferentes bancos de dados). Clique aqui para baixar esta tabela.
Tabela Suplementar S3: Posições das regiões estruturais e funcionais. As posições das regiões específicas da proteína da subunidade γ2 (Sequência de Referência NCBI: NP_944494.1) codificadas pelo transcrito de referência (NM_198904.4) Clique aqui para baixar esta tabela.
Tabela suplementar S4: O conteúdo do arquivo "data1.xlxs", que representa os dados ClinVar do GABRG2 que inclui apenas as colunas: "GRCh38Chromosome", "GRCh38Location", "Name", "Protein change". Clique aqui para baixar esta tabela.
Tabela suplementar S5: O conteúdo do arquivo "data1_output.xlsx" que contém a formatação necessária de variantes de sentido incorreto dos dados GABRG2 a serem carregados no servidor dbNSFP para previsão de efeito de variante. Clique aqui para baixar esta tabela.
Tabela suplementar S6: O conteúdo do arquivo "data2.xlsx" que contém a saída do servidor dbNSFP para a previsão do efeito variante para variantes missense desconhecidas do GABRG2. Clique aqui para baixar esta tabela.
Tabela Suplementar S7: Pontuações AlphaMissense para variantes da subunidade do receptor GABAA . Clique aqui para baixar esta tabela.
Tabela Suplementar S8: Características biofísicas das variantes. Os valores dos parâmetros biofísicos foram obtidos de estudos anteriores com experimentos de eletrofisiologia. As variantes são rotuladas com o tipo "S" (substituição), enquanto os parâmetros do receptor do tipo selvagem são dados para cada substituição e rotulados com "C" (controle). τd : Constante de tempo de desativação, PAberto : Probabilidade aberta, gGABA: Condutância do receptor, Imax: Corrente máxima, τr : Constante de tempo de ativação. Clique aqui para baixar esta tabela.
Tabela Suplementar S9: Características físico-químicas das variantes. As variantes previamente identificadas com parâmetros biofísicos são rotuladas com o tipo "S" (substituição), enquanto as variantes previstas são representadas com "P". H: Mudança na hidrofobicidade, VSC: Mudança no volume da cadeia lateral, P1: Mudança na polaridade, P2: Mudança na polarização, SASA: Mudança na superfície acessível ao solvente, NCISC: Mudança no índice de carga líquida. Os valores são obtidos para cada aminoácido original e variante de Guo et al.36 e a mudança em cada parâmetro é estimada conforme dado. Clique aqui para baixar esta tabela.
Tabela Suplementar S10: Características estruturais das variantes. As variantes previamente identificadas com parâmetros biofísicos são rotuladas com o tipo "S" (substituição), enquanto as variantes previstas são representadas com "P". A localização da variante em um domínio é representada com 1, caso contrário, 0. Todos os valores são obtidos de Brünger et al.35. Clique aqui para baixar esta tabela.
Tabela Suplementar S11: Correlações de parâmetros estruturais e biofísicos para todas as variantes conhecidas. Clique aqui para baixar esta tabela.
Tabela Suplementar S12: Correlações de parâmetros estruturais, físico-químicos e biofísicos para variantes conhecidas de GABRA1 . Clique aqui para baixar esta tabela.
Tabela Suplementar S13: Correlações de parâmetros estruturais, físico-químicos e biofísicos para variantes conhecidas de GABRB2 . Clique aqui para baixar esta tabela.
Tabela Suplementar S14: Correlações de parâmetros estruturais, físico-químicos e biofísicos para variantes conhecidas de GABRB3 . Clique aqui para baixar esta tabela.
Tabela Suplementar S15: Correlações de parâmetros estruturais, físico-químicos e biofísicos para variantes conhecidas de GABRG2 . Clique aqui para baixar esta tabela.
Tabela Suplementar S16: Parâmetros biofísicos para receptores α1β3γ2 GABAA de tipo selvagem e mutantes. Clique aqui para baixar esta tabela.
Tabela Suplementar S17: Taxa de disparo e intervalos entre espinhos em resposta a sinapses glutamatérgicas simples, duplas ou triplas com receptores GABAA do tipo selvagem e mutantes. Clique aqui para baixar esta tabela.
Discussão
Ao aplicar uma combinação de genética computacional, modelagem molecular e simulações neurais, a abordagem apresentada neste artigo tem o potencial de melhorar a classificação das variantes do receptor GABAA, oferecendo informações valiosas para pesquisas sobre epilepsia e aplicações clínicas. Uma análise abrangente para a identificação e priorização de mutações patogênicas previstas é apresentada e estendida em uma estrutura que potencialmente preenche a lacuna entre os efeitos variantes na proteína e no fenótipo celular. A avaliação do impacto da atividade epileptogênica do receptor GABAA na simulação do neurônio piramidal do hipocampo permite a replicação do fenótipo in vitro associado à disfunção do receptor GABAA e a demonstração da alteração da resposta de um único neurônio na raiz da disfunção da rede. Com base nessas simulações de respostas neurais geradas por mutações epileptogênicas, uma estimativa aproximada dos efeitos funcionais de mutações previstas estruturalmente proximais foi explorada. As previsões sobre o efeito das mutações previstas na cinética do canal requerem uma análise completa com conjuntos de variantes conhecidos. Análises comparativas, como as apresentadas neste artigo, e simulações de atividade neural fornecem insights críticos para a geração e melhoria de modelos preditivos com foco no efeito variante na função da proteína e na patologia neural. Além disso, nossa metodologia pode ser usada para selecionar e priorizar as variantes mais patogênicas entre as variantes desconhecidas para examinar os efeitos variantes ligados aos distúrbios do neurodesenvolvimento relacionados ao receptor GABAA. Por exemplo, subunidades receptoras marcadas com sondas fluorescentes 66,67,68,69,70 e carregando as mutações previstas podem ser expressas in vitro para estudar seu tráfego, expressão da superfície celular e neurofisiologia. Além disso, modelos animais como C. elegans podem ser considerados para validar os efeitos das mutações previstas. Por exemplo, a edição do gene CRISPR-Cas9 tem sido usada para gerar uma deleção de unc-49, um receptor GABAA de C. elegans, gerando assim mutações homozigóticas associadas à epilepsia em unc-49 ou subunidades do receptor GABAA humano71.
Em geral, a classificação das variantes se beneficia do uso de múltiplos níveis de evidência computacional, conforme preconizado pelo ACMG-AMP12. Essa abordagem fortalece a confiabilidade da classificação de variantes, integrando diferentes ferramentas preditivas e fontes de dados, aumentando a precisão das avaliações clínicas e melhorando o processo geral de tomada de decisão no diagnóstico genômico. Em nossa metodologia, a utilização de preditores de ensemble, que combinam as previsões de várias ferramentas, preenchendo assim o requisito de várias linhas de evidência computacional e eliminando a necessidade de usar diferentes ferramentas separadamente é uma vantagem. Essa abordagem também supera o desafio de lidar com resultados díspares de ferramentas individuais, simplificando assim o processo de previsão e aumentando a eficiência. No entanto, não há garantia quanto à precisão preditiva de análises centradas em genes ou específicas de variantes. Isso leva à conclusão de que as previsões centradas no gene ou específicas da variante devem ser realizadas sob condições específicas ajustadas para os contextos e objetivos específicos 15,72,73,74. Para intervenções clínicas, isso exigiria a avaliação da acurácia preditiva de ferramentas in silico para um gene específico ou subconjunto de genes no contexto de uma determinada doença, muitas vezes com otimização individualizada75. No entanto, a avaliação da precisão preditiva é frequentemente limitada pela falta de um número suficiente de variantes, o que pode afetar a confiabilidade da avaliação da precisão.
Diferentes ferramentas na literatura estão disponíveis e sua precisão é testada e validada em conjuntos de dados14. No entanto, esses resultados de precisão baseados em grandes conjuntos de dados não são necessariamente refletidos na previsão de algumas variantes desconhecidas para um determinado gene. Nesse contexto, a literatura acumulada sugere que os preditores de conjunto, que compilam e calculam os resultados de preditores individuais, são conhecidos por terem um desempenho melhor do que a concordância de preditores individuais 33,76,77,78 e, portanto, no presente estudo, optamos por usar preditores de conjunto, ou seja, BayesDEL33 e ClinPred 32 especificamente por seu desempenho superior32, 34 BayesDEL foi avaliado comparativamente para 4.094 variantes missense em genes clinicamente relevantes, incluindo genes que codificam proteínas transmembranares, como a subunidade alfa 5 do canal de sódio dependente de voltagem (SCN5A), e mostrou desempenho superior33. Em nosso protocolo de previsão de efeito variante, como primeiro passo, consideramos o consenso de dois preditores de conjunto (BayesDEL e ClinPred). O AlphaMissense37, um modelo de aprendizado profundo desenvolvido pelo Google DeepMind, é uma extensão do AlphaFold 64,79, utilizando assim o poder da previsão de estrutura de proteínas de alta precisão. Quando comparamos os resultados iniciais da previsão dos modelos de conjunto (BayesDEL e ClinPred, conforme descrito em nossa etapa 1.3 do protocolo) com os resultados do AlphaMissense, as previsões estavam parcialmente de acordo entre si (Arquivo Suplementar 4: Tabela Suplementar S15) e não se alinharam totalmente com as previsões dos modelos de conjunto (BayesDEL e ClinPred), que chegaram a um consenso de patogênicos ou associados a doenças, mostradas como linhas cor-de-rosa (Arquivo Suplementar 4: Tabela Suplementar S15). No entanto, as variantes desconhecidas (L81F, A303T e V329F) próximas às mutações GABRG2 R82Q, P302L e K328M, que usamos em nosso modelo de neurônios e previstas como patogênicas tanto por ClinPred quanto por BayesDEL, também foram previstas como patogênicas por AlphaMissense, conforme mostrado por destaques amarelos (Arquivo Suplementar 4: Tabela Suplementar S15).
Como o AlphaMissense29 usa previsão de sequência e contexto estrutural, em nosso estudo, também queríamos ver se havia alguma associação entre as pontuações do AlphaMissense e os locais de mutação do receptor GABAA com base em suas distâncias do centro da membrana e do eixo dos poros. Nossa hipótese é baseada na ideia de que o impacto funcional de variantes de aminoácidos adjacentes ou proximais às mutações funcionalmente identificadas das subunidades do receptor GABAA pode mostrar padrões semelhantes de alterações físico-químicas na função do canal observadas nos casos de mutações. Uma correlação entre as posições de mutação da subunidade do receptor GABAA e as pontuações do AlphaMissense nos ajudaria a identificar uma relação utilizável para construir uma estrutura para nossa hipótese, permitindo a previsão das consequências funcionais de novas variantes missense nas subunidades do receptor GABAA . No entanto, os escores do AlphaMissense não foram preditivos de alterações nesses parâmetros biofísicos (Figura 7). É importante notar que o tamanho limitado da amostra em nossa análise dificulta a obtenção de conclusões definitivas. No entanto, nossa análise descobriu que os escores do AlphaMissense não se correlacionaram com os parâmetros estruturais dos receptores GABAA . A falta de uma correlação posicional clara (por exemplo, entre as posições das mutações e as pontuações do AlphaMissense) desafia a validade de nossa suposição. Se os resíduos adjacentes estivessem realmente tendo efeitos semelhantes, esperaríamos ver uma correlação mais clara. Como esse não é o caso, isso enfraquece a capacidade de usar as pontuações do AlphaMissense como uma ferramenta confiável para testar nossa suposição.
Curiosamente, em nosso estudo, encontramos uma correlação leve entre a distância da variante ao eixo dos poros e o tempo de ativação do canal normalizado para os mutantes do gene GABRG2 . Assim, nossa suposição preliminar de que os aminoácidos adjacentes terão consequências semelhantes pode ser verdadeira em algumas regiões do canal, como regiões no poro ou em locais-chave envolvidos no gating, mas pode não ser tão clara em outras regiões. O pequeno conjunto de dados limita a capacidade de discernir essa variabilidade, mas dados futuros ou análises estruturais mais detalhadas podem ajudar a refinar esse aspecto de nossa hipótese. As simulações de dinâmica molecular80 podem servir como uma poderosa abordagem complementar para investigar melhor esses achados preliminares, especialmente no contexto da avaliação comparativa de duas mutações de subunidades γ2 adjacentes, a saber, a mutação epileptogênica γ2P302L40 e a mutação predita proximal γ2A303T (rs1581439874), realizada em nosso estudo. No futuro, essa abordagem pode permitir uma estimativa mais precisa do efeito da variante desconhecida no fenótipo celular, especialmente quando integrada às simulações neurais apresentadas em nosso estudo.
Além disso, será interessante explorar se as propriedades estruturais e físico-químicas das subunidades do receptor GABAA , juntamente com outras características, podem ser usadas para treinar modelos poderosos de aprendizado de máquina para a previsão funcional de novos efeitos variantes no canal, neurônio, rede e fenótipo da doença. Com o advento das abordagens automatizadas de aprendizado de máquina, chegamos a um ponto em que médicos e cientistas de laboratório úmido também podem desenvolver seus próprios modelos em um ambiente mais democratizado81. Portanto, a integração dessas tecnologias na prática clínica poderia potencialmente agilizar o processo, tornando a medicina personalizada mais acessível e reduzindo a dependência de conhecimentos altamente especializados para análise de variantes funcionais. Nesse contexto, nossa abordagem fornece insights sobre a dinâmica estrutural e funcional do receptor, potencialmente auxiliando em estudos futuros para a predição funcional do efeito variante.
Apesar do progresso atual na previsão da estrutura da proteína e do avanço representado pelo AlphaFold64, a previsão precisa do efeito das mutações e da função da proteína continua sendo um desafio devido à falta de dados necessários para treinar o modelo79. Para a predição do efeito variante, o AlphaMissense mostra desempenho superior em comparação com um subconjunto de modelos preditivos, mas os preditores de conjunto BayesDEL25 e ClinPred24, que foram usados em nosso estudo, não foram incluídos nesta comparação29. É importante notar que, em nosso estudo, as ferramentas in silico BayesDEL, ClinPred e AlphaMissense foram empregadas para propósitos distintos. Os preditores de conjunto, BayesDEL e ClinPred, foram usados principalmente para previsão de patogenicidade, enquanto o AlphaMissense foi usado especificamente para explorar a relação entre suas pontuações e os dados conhecidos para o impacto de mutações na subunidade γ2. Especificamente, nossa hipótese assume que as variantes patogênicas previstas, particularmente aquelas localizadas próximas ou adjacentes a mutações funcionalmente identificadas nas subunidades do receptor GABAA , exibirão parâmetros biofísicos semelhantes aos observados em mutações caracterizadas funcionalmente. Para investigar isso, escolhemos o AlphaMissense devido ao fato de ser alimentado pelo modelo AlphaFold264 altamente preciso, que utiliza a sequência básica de peptídeos para prever as consequências de substituições de aminoácidos únicos.
Consequentemente, uma grande limitação de nosso estudo é impulsionada principalmente pela disponibilidade limitada de dados experimentais. Por exemplo, nosso modelo de neurônio é baseado na expressão dos dados derivados da combinação de subunidades α1β3γ2 dos receptores GABAA , o que limita inerentemente as mutações estudadas na literatura às subunidades expressas como parte dessa combinação específica de receptores. Além disso, contamos com dados eletrofisiológicos derivados exclusivamente da expressão dessas subunidades em células HEK, estreitando ainda mais o escopo dos dados disponíveis na literatura. Nosso uso de modelagem neural para estimar os efeitos de variantes desconhecidas pressupõe que variantes desconhecidas (previstas como patogênicas em nosso fluxo de trabalho) localizadas próximas a mutações conhecidas exibirão padrões semelhantes nos parâmetros cinéticos do canal ou nas propriedades físico-químicas dos efeitos da mutação descritos na literatura. Essa suposição, juntamente com a necessidade de dados eletrofisiológicos para conjuntos de receptores específicos em células HEK293, reduz a quantidade de dados experimentais disponíveis para modelagem. Como resultado dessas restrições, os dados disponíveis nos permitiram modelar apenas um número limitado de variantes em subunidades α1β3γ2. No entanto, treinar o modelo de neurônios para diferentes conjuntos de subunidades, como combinações de subunidades α1β2γ2, combinações de subunidades α1β2δ ou α4β3δ, que têm implicações específicas de subunidades celulares, circuitos e níveis de rede, provavelmente mostrará aplicabilidade mais ampla a vários tipos de epilepsia e distúrbios do neurodesenvolvimento. No futuro, com o aumento de dados eletrofisiológicos disponíveis e estudos focados em mutações em conjuntos de receptores bem definidos e tipos de células específicas, pode ser possível aumentar a generalização e a precisão de nossa abordagem.
Os modelos de neurônios baseados em condutância multicompartimental fornecem uma ferramenta poderosa para gerar previsões para o significado funcional das variantes do receptor da resposta de um único neurônio. Essa ferramenta permite definições flexíveis de parâmetros celulares/sinápticos e suas localizações para testar qualquer pergunta específica. Geradores de pico simples usados neste protocolo podem ser substituídos por outros modelos de neurônios para estudar a atividade do microcircuito. A etapa crítica do protocolo também é a etapa mais limitante: a definição de qualquer variante do receptor em termos de cinética de canal alterada. As informações necessárias são idealmente fornecidas por estudos de eletrofisiologia de patch clamp; no entanto, a análise computacional de substituições de aminoácidos com significância clínica prevista e as comparações com substituições conhecidas também podem fornecer algumas dicas. Nosso estudo e protocolo descrito incorporam o uso de simulações de atividade neural não como uma ferramenta preditiva, mas sim como uma ferramenta para explorar os efeitos das mutações para apoiar uma visão mais ampla sobre as consequências das características biofísicas alteradas do receptor GABAA na atividade de um único neurônio. A dependência de dados experimentais em simulações neurais é uma limitação importante em nossa abordagem, que pode se beneficiar de modelagem molecular avançada para preencher a lacuna entre estrutura e função.
Em nosso protocolo, algumas etapas devem ser avaliadas criticamente. Primeiro, a escolha do modelo preditivo usado na primeira parte do nosso protocolo pode ser crítica. A seleção de ferramentas in silico depende de vários fatores, incluindo a geração de várias linhas suficientes de evidências computacionais para uma previsão poderosa12. Os preditores de conjunto que integram a análise de vários algoritmos preditivos se ajustam melhor a essa recomendação, portanto, preferidos em comparação com os preditores individuais. Existem diferentes preditores, e sua precisão geralmente é testada em grandes conjuntos de dados, o que não garante necessariamente a precisão do modelo preditivo usado para efeitos variantes desconhecidas localizados em um gene específico. Isso é compensado usando dois modelos de conjunto, que coletam e calculam os resultados da previsão de vários preditores. Além disso, os pontos de corte dos modelos preditivos podem ser ajustados para aumentar a especificidade se o objetivo for principalmente identificar as variantes mais patogênicas. Definir limites de corte apropriados é importante para equilibrar o trade-off entre sensibilidade e especificidade e garantir a classificação precisa das variantes. Em nosso estudo, usamos os pontos de corte padrão. Especialmente não alteramos o ponto de corte em favor da captura de variantes que podem ter maior probabilidade de serem patogênicas, pois isso diminuiria o escopo das variantes a serem examinadas em vários níveis de nossa análise, conforme descrito em nosso fluxo de trabalho. Também é importante ressaltar que, ao escolher os dados estruturais para a reconstrução tridimensional da proteína de interesse, há necessidade de revisão preliminar dos dados estruturais na literatura. O exame estrutural dos receptores GABAA ganhou força recentemente com estudos estruturais robustos relatando a estrutura tridimensional de diferentes conjuntos de receptores em diferentes condições 26,27,82,83,84,85. Dada a disponibilidade desses dados, em nosso estudo, nos concentramos nas estruturas determinadas experimentalmente para a reconstrução de dados estruturais. No entanto, a previsão do AlphaFold64 pode ser favorecida para a análise de outras proteínas que não possuem esses dados determinados experimentalmente. Para dados estruturais derivados de estudos experimentais, é importante prestar atenção à numeração de aminoácidos. A numeração PDB de aminoácidos pode ser diferente da numeração UniProt, uma vez que a primeira pode não incluir o peptídeo sinal que é removido durante a maturação da proteína. Além disso, proteínas quiméricas expressas no sistema experimental podem causar discrepâncias. Nesse caso, o alinhamento em pares da sequência de interesse com a sequência derivada dos dados estruturais ajudará a preservar a consistência. Na literatura, os dados estruturais para a proteína da subunidade γ2 são baseados em diferentes métodos, incluindo métodos experimentais como microscopia eletrônica (EM) e o método de predição de alta precisão do AlphaFold. Se o método experimental não cobrir totalmente a sequência desejada com alta resolução, as previsões do AlphaFold podem ser usadas. No presente estudo, a estrutura 7QNE26 foi escolhida por corresponder à estrutura microscópica eletrônica criogênica do receptor sináptico α1β3γ2 GABAA humano de comprimento total. Isso representa exatamente a combinação de subunidades de comprimento total, que foi o foco do presente estudo.
Além disso, para análise comparativa, o uso de parâmetros normalizados da cinética do canal deve ser preferido, pois os valores desses parâmetros podem variar dependendo da composição da subunidade do receptor e das configurações experimentais. Por exemplo, τaumento eτ desativação devem ser determinados sobre mudanças de x vezes sobre os valores de controle do tipo selvagem. Na etapa 2.5 do protocolo, para um baixo número de variantes conhecidas, a análise de correlação paramétrica ou categórica e a determinação dos valores de rho e p podem ser preferidas. Idealmente, espera-se que métodos como a análise de componentes principais produzam relações mais precisas, mas exijam um número maior de amostras.
O ambiente de simulação pode ser alterado. Neste estudo, Brian2 foi preferido pelos seguintes motivos: a classe de neurônios espaciais em Brian2 fornece uma ferramenta valiosa para simular a atividade neural. Brian2 tem uma vantagem significativa no uso de equações diferenciais para descrever dinâmicas contínuas e atualizar instruções para eventos discretos, em vez de depender de modelos de "caixa preta" predefinidos, e isso leva a uma excelente legibilidade e adaptabilidade do código, uma vez que todos os aspectos do modelo podem ser explicitamente definidos em um único script Python, com as equações do modelo declaradas em notação matemática e apenas uma pequena quantidade de vocabulário específico de Brian usado37. Como o modelo é explicitamente descrito, todos os recursos são documentados e podem ser encontrados no arquivo de descrição da simulação primária, o que elimina a necessidade dos modelos de "caixa preta" anteriormente confiáveis, conforme mencionado nos estudos de Stimberg et al.45,86.
O modelo de neurônio atual utiliza condutâncias modificadas do tipo Hodgkin-Huxley46 com Na+, K+ e corrente de fuga. Este modelo baseado em condutância pode ser expandido ainda mais com a inclusão de vários outros tipos de canais, como canais de Ca2+ . Para modelos de sinapse, é importante notar que esses parâmetros devem ser obtidos para composições de subunidades específicas, e apenas as variantes com parâmetros medidos com essas composições devem ser avaliadas. Neste estudo, a composição do receptor α1β3γ2 foi selecionada; portanto, apenas as variantes de α1, β3 ou γ2, com parâmetros cinéticos de canal medidos em um receptor α1β3γ2 GABAA , foram incluídas.
A estimativa da biofísica celular envolve a segmentação da célula em vários compartimentos cilíndricos, cada um potencialmente possuindo características de condutividade variadas. Apesar das irregularidades nos dendritos de um neurônio, eles podem ser vistos como fitas localmente homogêneas. Tais modelos ajudam a entender a estrutura complexa e o funcionamento das células. O design do modelo se concentra em uma versão simplificada da morfologia real que pode refletir esses recursos.
A localização e as características físico-químicas do aminoácido alterado determinam o impacto na cinética do canal. Por exemplo, uma substituição de aminoácidos resultando em um aminoácido com uma cadeia lateral maior diminuirá a condutância do canal se essa mudança ocorrer em um aminoácido que reveste o poro do canal. As substituições também podem afetar a abertura/fechamento do canal. Para simplificar neste modelo, a cinética de ligação do GABA é reduzida apenas a uma proporção de receptores disponíveis; no entanto, modelos mais detalhados podem ser projetados para incluir essa interação para estudar o possível impacto de substituições que alteram a afinidade de ligação do ligante.
Em conclusão, o presente estudo emprega métodos computacionais para prever variantes patogênicas nas subunidades do receptor GABAA e estimar qualitativamente o possível fenótipo celular com base na simulação de mutações associadas à epilepsia no modelo de neurônio piramidal do hipocampo. Até onde sabemos, este é o primeiro protocolo a explorar a aplicação combinada de genética computacional, modelagem molecular e simulações neurais para avaliar como as variantes genéticas podem contribuir para a disfunção do receptor GABAA em vários níveis de complexidade, do DNA à função da proteína e comportamento neural. Este protocolo pode fornecer uma base para melhorar a estimativa de variantes potencialmente deletérias e patologia neural associada na epilepsia. Além disso, pode ser utilizado no estudo de outras canalopatias, produzindo informações importantes sobre o mecanismo subjacente de distúrbios relevantes do neurodesenvolvimento e da rede. Com base nisso, ao incorporar as avaliações estruturais e físico-químicas do receptor GABAA para examinar o impacto funcional das variantes e sua integração na cinética do canal do receptor GABAA , uma análise mais precisa pode ser desenvolvida no futuro.
Divulgações
Todos os autores declaram não ter conflitos de interesse relacionados a este trabalho.
Agradecimentos
Agradecemos a Çağla Koca por sua ajuda na construção do neurônio modelo.
Materiais
| Name | Company | Catalog Number | Comments |
| Brian2 | Sorbonne Université, INSERM, CNRS, Institut de la Vision, France; Imperial College London, United Kingdom | 2.8.0.4 | Stimberg et al., 2019 (https://pypi.org/project/Brian2/ ) |
| dbNSFP server | Genos Bioinformatics LLC, USA | v3.0 | Liu et al., 2020 (http://database.liulab.science/dbNSFP) (https://sites.google.com/site/jpopgen/dbNSFP) |
| HOPE | Centre for Molecular and Biomolecular Informatics CMBI, Radboud University, Netherlands | 1.1.1 | Venselaar et al., 2010 (https://www3.cmbi.umcn.nl/hope/) |
| Jalview | University of Dundee, UK | JV2 | Waterhouse et al., 2009 (https://www.jalview.org/) |
| Jupyter Notebook | Project Jupyter, USA | https://jupyter.org/install | |
| Phyton | Python Software Foundation, USA | 3.13 | https://www.python.org/downloads/ |
| Protter | ETH Zurich, Switzerland | Version 1.0 | Omasits, et al., 2014 (https://wlab.ethz.ch/protter/start/) |
| R | The R Foundation for Statistical Computing, USA | R version 4.3.2 | https://www.r-project.org/ |
| RStudio | Posit software, PBC, USA | RStudio 2023.12.1+402 "Ocean Storm" Release | https://posit.co/downloads/ |
Referências
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados