Method Article
Umore acqueo come biopsia liquida per retinoblastoma: paracentesi corneale chiara e analisi genomica
In questo articolo
Riepilogo
L'umore acqueo è una biopsia liquida ad alto rendimento per il retinoblastoma, un cancro intraoculare che non può essere sottoposto a biopsia in vivo a causa del rischio di diffusione extraoculare. In questo articolo, viene presentato un metodo per estrarre in sicurezza l'umore acqueo tramite paracentesi corneale chiara e i passaggi per l'analisi genomica per identificare i biomarcatori prognostici.
Abstract
Esiste una significativa utilità clinica potenziale per l'applicazione di una piattaforma di biopsia liquida per il retinoblastoma, dato che la biopsia tumorale diretta è vietata in questi pazienti. L'umore acqueo (AH) si forma in un compartimento separato dal tumore ma è racchiuso all'interno dello stesso spazio oculare. Pertanto, è una fonte arricchita di informazioni genomiche tumorali specifiche per l'occhio che possono essere utilizzate come biopsia liquida o surrogato della biopsia tumorale per questa malattia. Questo manoscritto descrive in dettaglio una metodologia per estrarre in modo sicuro l'AH dagli occhi del retinoblastoma tramite paracentesi corneale chiara. Inoltre, vengono presentate le fasi dell'analisi genomica, tra cui l'isolamento e la purificazione del DNA libero da cellule, il sequenziamento di nuova generazione, l'analisi dell'alterazione del numero di copie somatiche (SCNA), l'identificazione della mutazione della variante a singolo nucleotide (SNV) RB1 e la stima della frazione tumorale. Sono state valutate la validità pre-analitica, analitica e clinica precoce della piattaforma per biopsia liquida AH; Tuttavia, non è privo di limitazioni. Questi sono in gran parte una conseguenza della quantità di DNA libero da cellule necessaria per alcune fasi del saggio. Rispetto ad altre piattaforme di biopsia liquida a base di sangue attualmente in fase di studio per il retinoblastoma, una piattaforma a base di AH è limitata dal volume di biofluido (e quindi dalla quantità di DNA) che può essere estratto dall'occhio; il vantaggio è che l'AH è specifico per l'occhio. La piattaforma qui discussa è unica in quanto rileva il DNA tumorale circolante nell'AH attraverso due meccanismi (SCNA e SNV RB1 ), producendo una maggiore sensibilità per l'identificazione delle informazioni genomiche tumorali. La biopsia liquida AH ha il potenziale per un'applicazione clinica diretta all'oncologia di precisione per i pazienti con retinoblastoma, con particolare importanza per i pazienti con malattia bilaterale poiché l'AH è specifica per i tumori di ciascun occhio. C'è una ricerca in corso con applicazioni di questa piattaforma anche a pazienti con altri tumori oculari.
Introduzione
Il retinoblastoma (RB) è un tumore raro e unico. Sebbene sia il tumore maligno intraoculare primario più comune che si forma nella retina in via di sviluppo di neonati e bambini piccoli, ci sono solo circa 7000 casi nel mondo ogni anno e circa 250-300 di questi sono negli Stati Uniti. Sebbene la sopravvivenza dei pazienti si avvicini al 98% nei paesi sviluppati1, la sopravvivenza oculare per gli occhi avanzati, che sono clinicamente classificati come International Intraocular RB Classification (IIRC)2 Group D/E o AJCC cT2b/3, è di gran lunga inferiore. Molti di questi occhi avanzati vengono enucleati principalmente (come primo trattamento) o secondariamente (dopo tentativi falliti di terapia di salvataggio del globo). Nell'attuale pratica clinica dell'oncologia oculare, non esistono biomarcatori molecolari oculari specifici derivati dal tumore che siano attualmente utilizzati clinicamente per assistere nella diagnosi, nella prognosi per la sopravvivenza oculare o nel monitoraggio del trattamento dei pazienti con RB. Ciò è dovuto, in gran parte, al fatto che il tessuto tumorale è disponibile solo per le analisi molecolari e genetiche da occhi enucleati perché la biopsia tumorale diretta nella RB è vietata a causa della preoccupazione per la diffusione extraocularedel tumore 3,4,5,6,7,8,9 . A causa di questo divieto, precedenti studi retrospettivi sulla genetica dei tumori RB e sulle loro correlazioni cliniche erano limitati all'analisi del tessuto tumorale ottenuto solo da occhi enucleati 10,11,12,13. Pertanto, c'è stata una mancanza di dati molecolari oggettivi derivati dal tumore disponibili alla diagnosi e durante la terapia di recupero dell'occhio. Ciò ha limitato non solo la comprensione della biologia tumorale in vivo e dei modi in cui questi tumori cambiano a livello molecolare durante la terapia, ma anche la capacità di sviluppare piani di trattamento personalizzati, specifici per l'occhio e basati sulla genomica per questi giovani pazienti oncologici.
A parte il divieto di biopsia, un altro aspetto unico di questo cancro è che la maggior parte dei tumori è iniziata dalla perdita bi-allelica del gene oncosoppressore RB1, che modula il ciclo cellulare. La retina in via di sviluppo è squisitamente sensibile a questa perdita. Nel 60% dei pazienti, questa inattivazione bi-allelica del gene RB1 si verifica nella retina solo come perdita somatica e provoca una malattia unilaterale. Tuttavia, nel 40% dei pazienti, la mutazione iniziale di RB1 avviene nella linea germinale, seguita da un secondo "colpo" nella retina. In questi bambini, ci sono spesso tumori multipli che colpiscono entrambi gli occhi. Infine, un sottogruppo molto piccolo (<2%) di tumori sembra essere guidato dall'amplificazione di MYCN senza mutazioni nel gene RB1. Mentre i tumori guidati da MYCN quasi universalmente falliscono il trattamento e richiedono l'enucleazione, attualmente non esiste un modo chiaro e oggettivo per identificare questo sottotipo di tumore aggressivo alla diagnosi14,15. Inoltre, il monitoraggio dell'attività intraoculare del tumore RB si basa quasi esclusivamente sull'imaging e sulle osservazioni cliniche di ciascun occhio da parte dell'oncologo oculare curante. Non esiste un mezzo oggettivo e quantitativo di diagnosi, prognosi o un modo per monitorare le dinamiche del tumore specifiche dell'occhio durante il trattamento. A causa di queste limitazioni uniche per la RB, la prospettiva di una piattaforma di biopsia liquida per questo cancro è allettante. Le biopsie liquide utilizzano fluidi corporei per isolare e sequenziare il DNA libero da cellule per determinare se è derivato dal tumore, noto come DNA tumorale circolante (ctDNA). Mentre il siero è ricercato per altri tumori, il ctDNA RB che si trova nel siero non è specifico per l'occhio, il che ha chiari limiti per il 40% dei pazienti che soffrono di malattia bilaterale. Inoltre, è stata descritta solo nel contesto di una malattia intraoculare o metastatica avanzata, generalmente con una bassa frazione tumorale (<5%)16.
Nel tentativo di affrontare questi problemi clinici reali che i pazienti e le famiglie devono affrontare, nel 2017 abbiamo dimostrato che l'umore acqueo (AH, il fluido chiaro davanti all'occhio), è una fonte ad alto rendimento di ctDNA che può essere utilizzato come biopsia liquida, o più come surrogato della biopsia tumorale, per valutare RB17. 18,19. Con oltre 200 campioni fino ad oggi, è stato identificato il ctDNA nel >95% dei campioni, compresi gli occhi IIRC meno avanzati dei gruppi A, B e C, nonché le differenze genomiche tra gli occhi nei pazienti bilaterali18,19. Questo DNA può essere isolato in modo sicuro ed efficace per il rilevamento di biomarcatori molecolari rilevanti in occhi che sono attivamente sottoposti a trattamento o naïve al trattamento 18,20,21. La piattaforma per biopsia liquida AH può essere utilizzata anche per identificare le varianti patogene diagnostiche di RB1 o l'amplificazione primaria di MYCN che avvia la tumorigenesi21,22. È importante sottolineare che abbiamo identificato una firma molecolare prognostica - la presenza di un guadagno del cromosoma 6p con un'ampiezza di ≥1,5 rispetto all'amplificazione mediana o focale di MYCN - che è associata a un aumento del 16,5% della probabilità di perdita del controllo intraoculare del tumore che richiede la rimozione dell'occhio 18,20,21. Infine, è stato dimostrato che i cambiamenti nella frazione tumorale del ctDNA (TFx) nell'AH sono correlati con la risposta terapeutica poiché livelli più elevati sono correlati con la malattia attiva e livelli decrescenti sono associati a una risposta positiva al trattamento23. Date queste applicazioni e la loro potenziale utilità clinica, abbiamo voluto delineare la metodologia per la biopsia e la valutazione dell'AH. Ciò include la paracentesi corneale chiara per l'acquisizione del campione e il protocollo per l'analisi genomica, in particolare la costruzione e il sequenziamento della libreria di cfDNA, la determinazione dell'ampiezza dello SCNA, l'identificazione della variante patogena RB1 e il calcolo del TFx.
Protocollo
Questa ricerca è condotta sotto l'approvazione del Children's Hospital di Los Angeles e della University of Southern California Institutional Review Board e aderisce ai principi della Dichiarazione di Helsinki. Il consenso informato scritto è sempre ottenuto dai tutori legali di tutti i partecipanti.
Uno schema per il flusso di lavoro della biopsia liquida AH può essere visto nella Figura 1.

Figura 1: Flusso di lavoro per la biopsia liquida dell'AH che accompagna il protocollo qui descritto. EUA, esame in anestesia; LBX, biopsia liquida; cfDNA, DNA libero da cellule; CNA, modifica del numero di copia; TFx, frazione tumorale; IVM, intravitreale melfalano. Clicca qui per visualizzare una versione più grande di questa figura.
1. Procedura chirurgica:
NOTA: Questa procedura viene eseguita durante l'esame di routine in anestesia (EUA) per la valutazione clinica dei pazienti con RB. La procedura di paracentesi per estrarre l'AH deve essere eseguita solo da un chirurgo oftalmico qualificato che abbia completato la formazione standard in chirurgia oculare.
- I criteri di selezione e inclusione dei pazienti sono i seguenti.
- Assicurarsi che la camera sia formata e sgombra senza essere superficiale dal tumore, dalle sinechie o da una lente catarattosa.
NOTA: In alcuni occhi avanzati, la camera è troppo bassa al momento della diagnosi per estrarre in sicurezza l'AH. In questi casi, si raccomanda di attendere fino a dopo il primo ciclo di chemioterapia sistemica o intra-arteriosa. Una volta che il tumore principale si restringe, la camera generalmente si approfondisce, il che consente un approccio sicuro. - Assicurarsi che ci sia una visione chiara di tutte le strutture e che la pressione sia di <22 mm Hg in modo che non vi sia un rapido abbassamento della camera a causa della dinamica della pressione. Questo è vero per tutti i punti temporali, ma è particolarmente critico quando l'AH viene assunta al momento della diagnosi.
- Assicurarsi che non vi sia un coinvolgimento diretto del segmento anteriore da parte del tumore, che viene valutato mediante visualizzazione diretta e biomicroscopia ecografica. Questo criterio di inclusione viene solitamente identificato durante l'EUA.
- Assicurarsi che la camera sia formata e sgombra senza essere superficiale dal tumore, dalle sinechie o da una lente catarattosa.
- Una volta che il paziente è stato ritenuto idoneo e acconsentito, portare il paziente in sala operatoria. Consentire all'anestesista curante di iniziare l'anestesia come di routine per l'EUA per la cura clinica dei pazienti con RB.
- Una volta che il paziente è stato intubato e sedato e tutti gli esami clinici, l'imaging e qualsiasi esigenza terapeutica locale (ad es. laser o crioterapia) relativi all'EUA, estrarre l'AH per la piattaforma di biopsia liquida. Preparare e drappeggiare l'occhio nel solito modo sterile. Questo può essere completato dall'infermiere in circolazione o dal chirurgo operativo.
- Prima dell'EUA, posizionare le gocce dilatanti abitualmente utilizzate presso l'istituto per un'EUA nell'occhio chirurgico.
NOTA: Qui vengono utilizzati colliri combinati composti da 2,5% fenilefrina, 1% ciclopentolato e 1% tropicamide. Tuttavia, sono sufficienti le gocce standard utilizzate per la dilatazione dell'EUA; l'occhio non deve essere dilatato semplicemente per estrarre l'AH. - Posizionare il 5% di betadina goccia nell'occhio chirurgico e applicarlo utilizzando una spugnetta partendo dal centro e muovendosi perifericamente. Includere i margini palpebrali superiori e inferiori nella preparazione chirurgica. Completare la preparazione chirurgica utilizzando betadine tre volte, quindi attendere almeno due minuti prima di procedere.
- Metti un asciugamano blu sterile sopra la testa del paziente e infilalo sotto la testa per evitare che cada. Asciugare le ciglia e la zona periorbitale con un asciugamano blu sterile o una garza.
- Lascia che il chirurgo operante si lavi dentro, indossi un camice e indossi i guanti.
- Coprire il paziente con teli blu sterili, lasciando scoperto l'occhio chirurgico. Posizionare uno speculum con coperchio sterile per una visualizzazione ottimale del campo sterile.
NOTA: Presso il nostro istituto non è richiesta un'anestesia topica aggiuntiva poiché il bambino è in anestesia generale per l'EUA. - Eseguire il time out secondo il protocollo istituzionale. In questo caso, viene effettuato un primo time out per l'EUA e un secondo time out per verificare l'occhio o gli occhi prima della procedura.
- Prima dell'EUA, posizionare le gocce dilatanti abitualmente utilizzate presso l'istituto per un'EUA nell'occhio chirurgico.
- Utilizzando un microscopio operatorio, eseguire una paracentesi corneale chiara con l'estrazione di 0,1 mL di AH con un ago da 32 G su una siringa da 1 cc.
- Prima di iniziare la procedura, bagnare l'occhio con una soluzione salina bilanciata sterile (ogni mL contenente cloruro di sodio 0,64%, cloruro di potassio 0,075%, cloruro di calcio diidrato 0,048%, cloruro di magnesio esaidrato 0,03%, acetato di sodio triidrato 0,39%, citrato di sodio diidrato 0,17%, idrossido di sodio e/o acido cloridrico (per regolare il pH) e acqua per preparazioni iniettabili) per mantenere la lubrificazione corneale; Questa è una preparazione commerciale standard.
- Assicurarsi che l'ago da 32 G sia luer-lock alla siringa da 1 cc e che non vi sia pressione nella siringa (muovendo lo stantuffo della siringa verso l'interno e verso l'esterno più volte prima dell'uso).
- Successivamente, far passare l'ago attraverso la cornea chiara al limbus (perpendicolare ad esso, come è standard per la paracentesi) e rimanere all'interno della camera anteriore sopra l'iride dilatata periferica.
- Durante l'estrazione, tenere la punta dell'ago smussata verso l'alto anteriormente all'iride sotto la visualizzazione diretta tramite il microscopio. Se si preferisce, stabilizzare l'occhio con una pinza da 0,12 mentre l'ago passa attraverso la cornea.
NOTA: Non deve mai esserci contatto tra la punta dell'ago e qualsiasi struttura oculare; Dovrebbe rimanere sopra l'iride periferica nella camera anteriore media. - Estrarre 0,1 mL di AH. Manipolare lo stantuffo della siringa con la mano non dominante del chirurgo (ma con la pratica) o da un assistente addestrato senza muovere l'ago. Non perdere la visualizzazione diretta della punta dell'ago. Dai la priorità all'anatomia dell'occhio e prendi meno AH se necessario per assicurarti che la camera rimanga formata.
- Dopo l'estrazione, rimuovere delicatamente l'ago dalla camera anteriore. Assicurarsi che la camera rimanga formata ma leggermente poco profonda; La pressione sarà morbida ma fisiologica. Una volta tolto l'ago dall'occhio, bagnare abbondantemente l'occhio con acqua sterile; L'acqua sterile è preferita a una soluzione salina bilanciata in questa fase come misura di sicurezza perché l'acqua lisa le cellule.
- Esaminare il sito dell'ago per eventuali perdite. In caso di perdite, utilizzare un applicatore con punta di cotone per applicare una leggera pressione sul sito di iniezione per 30 secondi prima di controllare nuovamente la presenza di perdite. Ripetere il processo se necessario.
NOTA: L'uso di metodi più invasivi per fermare eventuali perdite non è mai stato necessario con questo approccio. A volte questa procedura è seguita da un'iniezione intravitreale di chemioterapia; per questo documento sui metodi, il protocollo si concentra sull'estrazione dell'AH come procedura primaria.
- Esaminare il sito dell'ago per eventuali perdite. In caso di perdite, utilizzare un applicatore con punta di cotone per applicare una leggera pressione sul sito di iniezione per 30 secondi prima di controllare nuovamente la presenza di perdite. Ripetere il processo se necessario.
- Una volta che non ci sono perdite, la procedura è considerata completa. Rimuovere i teli sterili dal paziente e pulire la betadina dal viso.
- Rimuovere con cautela la punta dell'ago dalla siringa, utilizzare un tappo sterile per chiudere il campione tramite luer lock per non perdere alcun campione ed etichettare la siringa in modo appropriato.
NOTA: Questo dovrebbe essere fatto dal coordinatore della ricerca clinica o dal chirurgo mentre il bambino viene estubato.- Dopo l'estrazione del campione, conservare il campione in una siringa con tappo a -80 °C; I campioni possono essere posti su ghiaccio secco per essere trasportati in un congelatore appropriato dalla sala operatoria.
- Posizionare una singola goccia di collirio antibiotico a seconda delle preferenze del chirurgo sulla superficie dell'occhio chirurgico prima che il paziente venga inviato al recupero. Qui vengono utilizzati colliri steroidei-antibiotici combinati e l'occhio non viene patchato.
- Una volta completata la procedura, l'anestesista curante garantisce una corretta estubazione. Portare il paziente alla guarigione.
- Dimettere il paziente a casa seguendo il protocollo postoperatorio istituzionale.
- Informare il paziente che l'occhio non deve essere coperto con bende o coperture protettive.
- Informare il paziente di non immergere completamente la testa in una vasca da bagno o in piscina per 24 ore e i genitori di non strofinare l'occhio del paziente (bambino); Non ci sono altre limitazioni all'attività del paziente.
- Informare il paziente che è improbabile che abbia dolore a causa della paracentesi, ma che potrebbe avere dolore a causa delle procedure associate durante l'EUA. Il paziente può assumere paracetamolo o ibuprofene in caso di qualsiasi disagio, come suggerito dal chirurgo.
2. isolamento e purificazione del cfDNA
- Eseguire l'isolamento e l'elaborazione del DNA entro 72 ore dall'estrazione dell'AH.
- Scongelare l'AH a temperatura ambiente. Controllare frequentemente il campione e passare all'estrazione subito dopo il completamento dello scongelamento.
- Estrarre il cfDNA dall'AH ed eluirlo in 50 μL di soluzione AVE (inclusa nel kit) utilizzando il kit di isolamento e purificazione del cfDNA (vedere la Tabella dei materiali per i dettagli) secondo il manuale di istruzioni del produttore.
3. Sequenziamento di nuova generazione (NGS) e controllo qualità (QC)
- Costruire le librerie di DNA per il sequenziamento utilizzando il kit di sequenziamento della libreria di DNA (vedere la Tabella dei materiali) secondo il manuale di istruzioni del produttore. Amplifica il DNA della libreria con 14 cicli di reazione a catena della polimerasi (PCR) come descritto nel manuale del kit di sequenziamento della libreria del DNA.
NOTA: Le librerie finali possono essere conservate a -20 °C fino a 1 mese prima del sequenziamento finale per l'analisi genomica. - Sequenziare le librerie di DNA sulla piattaforma NGS (vedere la Tabella dei materiali) secondo il protocollo del produttore seguendo il protocollo single-end 50 cicli o il protocollo paired-end 150 cicli.
NOTA: In linea di principio, altre piattaforme NGS funzioneranno (anche se non sono state provate qui), con adattatori adatti per la cella a flusso di sequenziamento. - Eseguire il controllo qualità per tutti i campioni di AH prelevati al momento della diagnosi o dell'enucleazione primaria.
NOTA: Il controllo qualità non può essere eseguito per campioni prelevati in altri punti temporali poiché la massa del DNA sarà troppo bassa e fuori dall'intervallo di rilevamento per i passaggi descritti di seguito.- Utilizzare 1 μl di DNA estratto ciascuno per il saggio di quantificazione del DNA e il saggio di profilazione dimensionale (vedere la tabella dei materiali) secondo i protocolli del produttore.
NOTA: Il picco dovrebbe essere di circa 300 bp. Se è inferiore a 150 bp, il campione è stato compromesso. Vedere File supplementare 1A per un esempio di un campione che deve superare il controllo di qualità e File supplementare 1B per un esempio di uno che non deve superarlo.
- Utilizzare 1 μl di DNA estratto ciascuno per il saggio di quantificazione del DNA e il saggio di profilazione dimensionale (vedere la tabella dei materiali) secondo i protocolli del produttore.
4. Analisi dei dati di alterazione del numero di copie somatiche (SCNA)
- Mappare le letture ottenute da NGS (sezione 3) al genoma umano (hg19, Genome Reference Consortium GRCh37, University of California Santa Cruz Genome Browser database)24,25 con un allineatore.
- Rimuovere i duplicati PCR (samtools rmdup26).
- Normalizzare il contenuto di guanina-citosina calcolando la percentuale di basi di guanina e citosina in ciascun contenitore dal genoma di riferimento. Un programma di esempio è descritto in Baslan et al.27.
- Per ottenere il numero di copie del segmento di DNA, dividere il genoma in 5000 contenitori di lunghezza variabile e quindi calcolare il numero relativo di letture contenute in ciascun contenitore.
- Utilizzare rapporti log2 privi di riferimenti per determinare le stime del numero di copie. Prendiamo il conteggio mediano dei cromosomi autosomici normali.
- Esegui la segmentazione utilizzando la segmentazione binaria circolare con DNAcopy (Bioconductor28).
- Definire gli SCNA come positivi con una deflessione del 20% rispetto al basale (rapporto log2 = 0), il che significa perdite con rapporti log2 ≤ 0,2 (rapporto di 0,87 o inferiore definisce una delezione) e guadagni con rapporti log2 ≥ 0,2 (rapporto di 1,15 o superiore definisce un'amplificazione).
NOTA: Queste soglie sono coerenti con le analisi bioptiche liquide precedentemente stabilite27,29.
5. Analisi mutazionale RB1 di campioni di AH
- Inoltre, amplificare l'intera libreria del genoma a 500 ng ciascuna per NGS mirato basato sulla cattura per il rilevamento delle mutazioni secondo il protocollo del produttore.
- Esegui il sequenziamento single-end a 150 bp o 50 bp a piattaforma NGS sulle librerie acquisite a >100x per protocollo del produttore.
- Elaborare i dati NGS. In questo studio, viene utilizzata una pipeline interna basata sulla pipeline bcbio presso il CHLA Center for Personalized Medicine30.
- Taglia i dati fastq grezzi dagli adattatori e dalle basi di bassa qualità con Atropos31 e allineali al riferimento GRCh37 umano con BWA-MEM e NovoAlign (v3)32,33.
- Contrassegna i duplicati con FreeBayes34 e quindi determina la variante germinale.
NOTA: Negli strumenti di chiamata delle varianti FreeBayes, ogni posizione del genoma del campione viene confrontata con il genoma di riferimento e la conta degli alleli viene modellata matematicamente per ottenere le misure di verosimiglianza del genotipo34. - Esegui l'annotazione delle varianti con Ensembl Variant Effect Predictor (v96)35 seguendo i passaggi 5.3.4-5.3.5.
- Utilizzare VarDict per determinare le varianti somatiche nell'AH o nel tumore senza il campione di sangue normale accoppiato (cioè in cieco rispetto alla variante germinale)36.
- Perdita di eterozigosi se le varianti continue di una regione hanno il ≤3% di alleli alternativi.
NOTA: Il software di analisi genomica commerciale può essere utilizzato anche con i parametri sopra indicati.
6. Determinazione del cfDNA TFx
- Stimare il TFx per ogni campione di cfDNA AH sequenziato utilizzando il software ichorCNA (software di stima TFx basato su CNA)37.
NOTA: Questo è il software standard utilizzato nelle biopsie liquide a base di sangue per determinare il cfDNA TFx38. L'utilizzo del software su AH è stato precedentemente pubblicato21,23. - Esamina individualmente i profili genomici e le corrispondenti soluzioni TFx per verificare che la stima del TFx per ciascun campione sia appropriata39.
Risultati
Di seguito sono presentati i risultati completi di due occhi (casi 33 e 47). I numeri dei casi rimangono coerenti con le pubblicazioni precedenti a scopo di confronto 18,20,21. I medici curanti sono stati all'oscuro dei risultati della biopsia liquida dell'AH durante la terapia. Tutte le decisioni terapeutiche non sono state randomizzate e sono state prese secondo lo standard di cura di routine precedentemente pubblicato40,41. I dati sugli esiti clinici sono rimasti separati dai dati genomici fino all'analisi finale. I dati grezzi dei risultati presentati sono disponibili su richiesta all'autore corrispondente. Grazie ai finanziamenti degli NIH, i dati sono regolati dalla politica di condivisione dei dati genomici degli NIH e in futuro saranno disponibili per altri ricercatori tramite un repository di dati designato dagli NIH ad accesso controllato (dbGAP); È disponibile anche su richiesta dell'autore corrispondente.
I casi 33 e 47 sono entrambi occhi del gruppo D2 dell'IIRC che presentavano presentazioni cliniche molto simili. Pertanto, sono stati trattati con la terapia di salvataggio del globo a discrezione dei genitori e con il trattamento di oncologi oculari e medici, in particolare con chemioterapia sistemica per il caso 47 e chemioterapia intra-arteriosa per il caso 33. Sulla base della prognosi clinica attualmente accettata che si basa sul gruppo IIRC dell'occhio2, il successo previsto del salvataggio del globo per gli occhi di questi due pazienti sarebbe stato lo stesso: 65%-70% per il gruppo D è una media, anche se questa varia a seconda del centro di trattamento41. Tuttavia, sulla base dei dati del Gruppo D raccolti finora dalla profilazione molecolare diretta al tumore in questo centro, il successo previsto del salvataggio del globo sarebbe del 72% per il caso 47 (senza guadagno di 6p identificato nell'AH) e del 9% per il caso 33 (con un guadagno di 6p identificato)18,20,21. Questo è dimostrato di seguito.
Il caso 47 è un esempio di occhio con rilevamento riuscito di SCNA e SNV utilizzando la piattaforma di biopsia liquida AH al momento della diagnosi, insieme alle tendenze TFx corrispondenti alla risposta al trattamento longitudinalmente. La paziente è una femmina che si è presentata a 15 mesi di età con un IIRC Gruppo D di 14 mm x 9 mm, stadio cT2b RB con semina vitreale a sfera. Era negativa per la mutazione germinale RB1 come determinato dai test clinici di routine sui leucociti sierici. Dall'AH, all'atto della diagnosi sono stati identificati un guadagno di 1q e un guadagno di 6p nell'AH, oltre ad altri due SCNA RB non altamente ricorrenti con perdita di 7p e guadagno di 13q (Figura 2). Da notare che l'ampiezza del guadagno di 6p era di 1,2 e solo le ampiezze di ≥ rapporto di 1,5 rispetto alla mediana hanno dimostrato di far presagire una prognosi infausta. Dato che non c'era amplificazione focale di MYCN e che il guadagno di 6p era inferiore alla soglia di 1,5, la prognosi per il salvataggio con trattamento basato sulle caratteristiche molecolari era alta. Lo stesso campione di AH prelevato alla diagnosi è stato valutato anche per il rilevamento di varianti patogene RB1 , che hanno rivelato SNV c.958C>T, p.Arg320* all'interno del gene RB1 con una frequenza di alleli varianti dell'87,01% (intervallo di confidenza al 95%, 79,7%-94,6%). Questo paziente è stato trattato con sei cicli di carboplatino, etoposide e vincristina (CEV) con regressione della malattia, ma ha dimostrato una semina persistente simile a una sfera e a una polvere che ha richiesto tre iniezioni intravitreali di melfalan (IVM) sequenziali. Durante il trattamento IVM, i campioni di AH A-C (ciascuno separato da due settimane) hanno dimostrato una completa normalizzazione del profilo genomico, una diminuzione del TFx e una diminuzione della concentrazione di DNA, tutti in concomitanza con la regressione clinica della malattia (Figura 3). Dopo la diagnosi, i valori di TFx sono rimasti al di sotto del limite di rilevamento del 5% per il resto del trattamento. A 19 mesi di follow-up, l'occhio è rimasto stabile senza recidive tumorali o diffusione extraoculare della malattia.
Ciò è in contrasto con il caso 33. Questo maschio di 22 mesi aveva una presentazione clinica complessivamente simile con una massa retinica di 11 mm x 18 mm e una semina vitreale di tipo polveroso, coerente con il Gruppo D unilaterale IIRC, stadio cT2b. Era anche negativo per la mutazione germinale RB1 come determinato dai test clinici di routine sui leucociti sierici. L'AH preso alla diagnosi ha dimostrato un guadagno di 1q, 6p (in questo caso con un rapporto di ampiezza di 1,5 rispetto alla mediana) e una perdita di 16q insieme a una perdita focale di 6q (Figura 4A). Sulla base del profilo molecolare prognostico, questo occhio aveva una probabilità di enucleazione aumentata di 16,5 in base alla presenza di 6p ≥ 1,5 di ampiezza. In questo campione non è stato identificato alcun SNV RB1, nonostante la copertura completa dell'intera lunghezza del gene RB1. Uno dei motivi per cui un SNV RB1 potrebbe non essere identificato è nei tumori primari guidati da MYCN, dove non è sempre prevista una mutazione RB1 concomitante 14,15,42,43,44,45. Tuttavia, il caso 33 non ha dimostrato alcuna evidenza di amplificazione di MYCN in nessuno dei suoi campioni di AH o nel suo tessuto tumorale enucleato (Figura 5). Una spiegazione più probabile per un risultato negativo dell'SNV RB1 è che la tumorigenesi iniziale sia stata guidata da una disregolazione epigenetica (ad esempio, metilazione del promotore)46,47, un fenomeno noto in RB che non sarebbe stato identificato dal test qui descritto.
Il trattamento iniziale per questo paziente è stato di quattro cicli totali di chemioterapia intraarteriosa con melfalan seguiti da quattro iniezioni IVM a causa della semina vitreale persistente. Durante la terapia IVM sono stati ottenuti tre campioni di AH (ciascuno separato da quattro settimane) e hanno dimostrato gli stessi tre SCNA presenti alla diagnosi (Figura 5). I valori di TFx sono rimasti elevati durante il trattamento, nonostante la diminuzione del volume del tumore primario riflettesse i semi tumorali attivi nel vitreo. Ciò dimostra come TFx sia rappresentativo dello stato generale della malattia nell'occhio. Sei mesi dopo la diagnosi, a causa di una malattia attiva persistente, l'occhio è stato enucleato. Il profilo genomico ottenuto dal tessuto tumorale in quel momento ha dimostrato una concordanza del 92,81% con il campione di AH ottenuto al momento della diagnosi (Figura 4B).
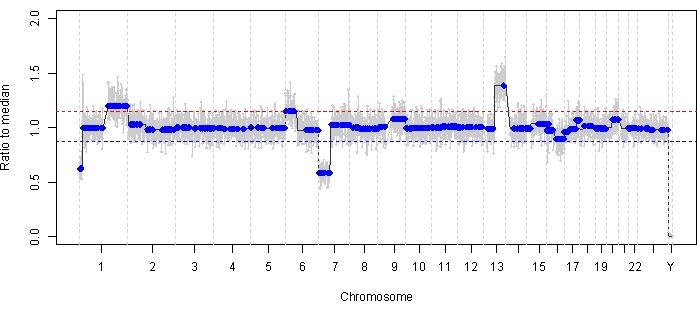
Figura 2: Profilo genomico alla diagnosi per il caso 47. Gli SCNA RB altamente ricorrenti con guadagno di 1q e 6p, insieme a SCNA RB non altamente ricorrenti con perdita di 7p e guadagno di 13q, sono stati identificati nell'AH prelevato al momento della diagnosi. La linea rossa rappresenta la soglia per un guadagno, mentre la linea blu rappresenta la soglia per una perdita. In particolare, l'ampiezza del guadagno di 6p era di <1,5 rispetto alla mediana, che è al di sotto della soglia della firma molecolare che fa presagire una prognosi infausta. Pertanto, sulla base della mancanza di biomarcatori negativi per il salvataggio dell'occhio, si prevede che questo occhio risponda alla terapia. Clicca qui per visualizzare una versione più grande di questa figura.
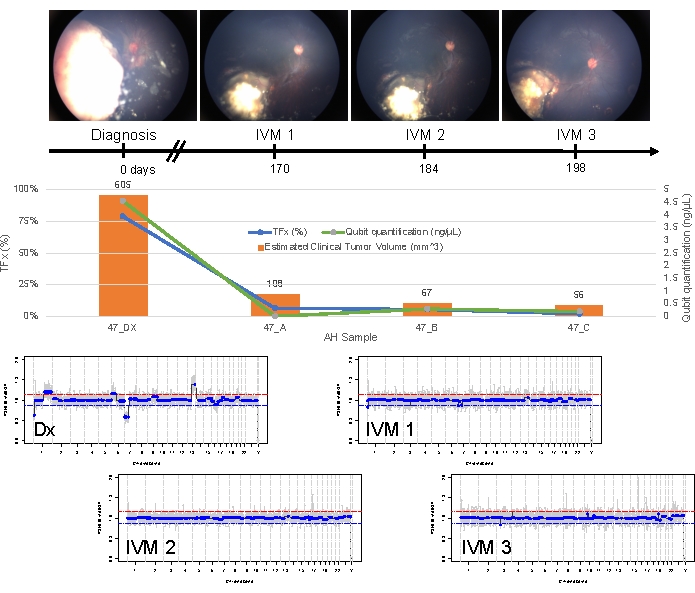
Figura 3: Informazioni longitudinali per il caso 47. Ciò include le foto del fondo oculare, la quantificazione del cfDNA, il volume clinico del tumore stimato dalle misurazioni del B-scan, i profili genomici e le stime TFx per ogni punto temporale clinico in cui l'AH è stato campionato (Dx = diagnosi, A = IVM1, B = IVM2, C = IVM3). Questo occhio ha risposto al trattamento ed è rimasto salvato a 19 mesi dal follow-up. Nel tempo è stata osservata una diminuzione della quantità di cfDNA, in linea con gli studi precedentemente pubblicati che mostravano la più alta resa ottenuta alla diagnosi. Anche il TFx è diminuito durante il trattamento, riflettendo la risoluzione della semina e la diminuzione del volume principale del tumore retinico (i volumi tumorali sono indicati sopra la barra arancione di ciascun punto temporale clinico). Come previsto con la regressione clinica della malattia, anche i profili genomici si sono normalizzati. Nei profili genomici, la linea rossa rappresenta la soglia per un guadagno, mentre la linea blu rappresenta la soglia per una perdita. Questa figura è stata ristampata con il permesso di Xu, L. et al.21. Clicca qui per visualizzare una versione più grande di questa figura.

Figura 4: Profili genomici per il caso 33. La linea rossa rappresenta la soglia per un guadagno, mentre la linea blu rappresenta la soglia per una perdita (A) Profilo genomico alla diagnosi per il caso 33. Alla diagnosi, oltre alla perdita focale di 6q, nell'AH sono stati identificati SCNA RB altamente ricorrenti con guadagno di 1q, guadagno di 6p e perdita di 16q. In particolare, l'ampiezza del guadagno di 6p era di 1,5 rispetto alla mediana, indicando un biomarcatore molecolare prognostico sfavorevole. Pertanto, sulla base di questa firma molecolare, prevediamo che questo occhio abbia una probabilità significativamente maggiore di fallimento del trattamento. (B) Profilo genomico ottenuto da tessuto tumorale enucleato, che era altamente concordante con il profilo ottenuto da AH alla diagnosi. A causa della mescolanza con il tessuto retinico normale, gli SCNA del tessuto tumorale possono dimostrare un'ampiezza inferiore rispetto all'AH a causa del TFx diluito. Clicca qui per visualizzare una versione più grande di questa figura.

Figura 5: Informazioni longitudinali per il caso 33. Ciò include le foto del fondo oculare, la quantificazione del DNA libero da cellule, il volume clinico del tumore stimato dalle misurazioni del B-scan (i volumi del tumore sono indicati sopra la barra arancione di ciascun punto temporale clinico), i profili genomici e le stime TFx per ogni punto temporale clinico in cui l'AH è stato campionato (Dx = diagnosi, A = IVM1, B = IVM2, C = IVM3, SE = enucleazione secondaria). Questo occhio non ha risposto al trattamento, richiedendo infine l'enucleazione secondaria (ES). Ciò era dovuto a una semina persistentemente attiva accompagnata da una recidiva del tumore apicale. La quantità di CfDNA è diminuita nel tempo, coerentemente con gli studi precedentemente pubblicati che mostrano la più alta resa ottenuta alla diagnosi (guadagno di 1q, guadagno di 6p, perdita di 16q e perdita focale di 6q). Tuttavia, il TFx è rimasto alto durante il trattamento, un prodotto della semina persistente che ancora rilascia cfDNA derivato dal tumore nell'AH. I profili genomici erano coerenti e mostravano gli stessi tre SCNA presenti alla diagnosi. Nell'AH ottenuto alla ES, sono stati osservati un nuovo guadagno di 2p (*) su larga scala e una perdita di 19q, suggerendo un'evoluzione clonale al momento della recidiva del tumore apicale. Nei profili genomici, la linea rossa rappresenta la soglia per un guadagno, mentre la linea blu rappresenta la soglia per una perdita. Questa figura è stata ristampata con il permesso di Xu, L. et al.21. Clicca qui per visualizzare una versione più grande di questa figura.
Fascicolo supplementare 1: Un esempio di (A) un campione che dovrebbe superare il controllo di qualità, con il picco intorno ai 300 bp e (B) un campione che non dovrebbe superare il controllo di qualità, con il picco intorno ai 150 bp. Clicca qui per scaricare questo file.
Discussione
La paracentesi corneale chiara è una procedura eseguita comunemente per molteplici indicazioni diagnostiche e/o terapeutiche in oftalmologia. In particolare per la RB, fa parte del protocollo standard di iniezione di chemioterapia intravitreale per ridurre la pressione intraoculare prima dell'iniezione per prevenire il reflusso al sito di iniezione48. Nonostante sia una procedura comune, non è del tutto priva di rischi; il dogma precedente era che un ago non dovrebbe mai entrare in un occhio con RB attivo. Questo dogma si è evoluto nell'ultimo decennio, prima attraverso il lavoro formativo di Patricia Chévez-Barrios sulla terapia con vettore adenovirale iniettato49, seguito da metodi progressivi di sicurezza migliorati per le iniezioni oculari in RB di Francis Munier48. Questo lavoro ha aperto la strada all'estrazione dell'AH da utilizzare come biopsia liquida, o surrogato della biopsia tumorale, per questo cancro. Ora siamo il primo centro a riportare l'utilità della piattaforma e i risultati preliminari di sicurezza dell'AH presi al momento della diagnosi. Parte dei metodi qui descritti ha lo scopo di garantire la sicurezza della selezione del paziente e gli aspetti procedurali dell'estrazione di AH per RB. In primo luogo, gli aghi possono entrare solo nella camera anteriore e non devono entrare in contatto con l'iride o il cristallino, poiché ciò può causare cicatrici dell'iride o cataratta che limitano la capacità del chirurgo di monitorare il tumore. È molto importante che l'ago non entri mai nella cavità vitreale (a meno che non sia combinato con la somministrazione di chemioterapia come nel caso dell'IVM), o che entri in contatto con il tumore, poiché ciò aumenta ipoteticamente il rischio di semina del tumore e di estensione extraoculare della malattia 3,4,5. Entrambe queste strutture sono posteriori alla camera anteriore e separate da essa dal cristallino e dall'iride. Per evitare danni alle strutture oculari, è importante mantenere sempre lo smusso dell'ago verso l'alto, sopra l'iride periferica e sempre sotto visualizzazione diretta con il microscopio operatorio. Esiste il rischio di una lieve fuoriuscita dell'AH dal sito dell'ago; Se ciò si verifica, si risolverà con una leggera pressione tramite un applicatore con punta di cotone. L'uso dell'ago di calibro più piccolo disponibile e l'assicurarsi che l'ago venga estratto lentamente dallo stesso tratto senza alcun taglio laterale ridurrà questo rischio. Sebbene sia previsto un leggero abbassamento della camera anteriore, la camera dovrebbe rimanere formata senza il contatto della cornea dell'iride e la pressione intraoculare dovrebbe essere morbida, ma fisiologica.
Per quanto riguarda le analisi genomiche, ci sono diversi passaggi critici coinvolti. La più importante è la manipolazione del campione di piccolo volume una volta estratto dall'occhio; è fondamentale che il campione rimanga congelato per tutto il tempo prima dell'elaborazione per prevenire la degradazione del DNA che può verificarsi con ripetuti cicli di congelamento e scongelamento50. Assicurarsi che i campioni siano posti su ghiaccio secco subito dopo l'estrazione e trasferiti in un congelatore a -80 °C aiuta a garantire ciò. Una volta che i campioni hanno iniziato l'elaborazione, le fasi di controllo della qualità sono l'opportunità principale per risolvere i problemi, garantendo che il cfDNA sia costruito in librerie di alta qualità. Utilizzando la quantificazione del DNA e il saggio di profilazione delle dimensioni dei frammenti di DNA, il controllo di qualità può essere eseguito su campioni prelevati durante l'enucleazione primaria o al momento della diagnosi a causa della maggiore resa di cfDNA presente in questi campioni18,21. Quando il picco ottenuto è intorno ai 300 bp, questo garantisce che il cfDNA sarà riconosciuto dalla piattaforma NGS. Se il picco ottenuto è inferiore a 150 bp, indicando che la maggior parte dei frammenti rilevati sono primer per la costruzione di librerie o oligo adattatori, il campione è stato compromesso e non deve essere ulteriormente elaborato per gli SCNA. Tuttavia, nella nostra esperienza nell'elaborazione di centinaia di campioni, meno del 5% deve essere rimosso per il controllo di qualità a causa dello scarso rapporto di allineamento delle letture20. Insieme ai metodi che abbiamo standardizzato per la raccolta, la manipolazione, la conservazione e l'elaborazione dei campioni, questa procedura dimostra la validità pre-analitica della piattaforma per biopsia liquida AH.
La biopsia liquida AH ha anche una validità analitica stabilita in base alla sua capacità di rilevare in modo accurato e affidabile mutazioni patogene RB1 e SCNA, con concordanze medie costantemente >95% tra i profili genomici generati da campioni di AH e il corrispondente tessuto tumorale 18,20,21,22,23. Nonostante la validità analitica dimostrata della piattaforma, non è priva di limitazioni. L'analisi mutazionale di RB1 può essere eseguita solo su campioni con >10 ng di cfDNA, che sono più frequentemente ottenuti in occhi naïve al trattamento al momento della diagnosi o dell'enucleazione primaria18,21. Ciò è dovuto alla minore concentrazione di cfDNA presente nei campioni di AH provenienti da occhi attivamente sottoposti a trattamento rispetto all'AH alla diagnosi o al momento dell'enucleazione primaria. Un ulteriore vincolo è che gli SCNA non possono essere rilevati a TFxs inferiori al 5%, il che impedisce il monitoraggio della malattia negli occhi in cui il carico tumorale è diminuito in modo significativo; Nella nostra esperienza, questo si verifica più comunemente negli occhi che rispondono al trattamento chemioterapico intravitreale23.
Al fine di determinare il TFx del cfDNA nell'AH, viene utilizzato un software di stima TFx basato su CNA. Questo software è uno strumento standard e accettato per il calcolo del TFx nelle biopsie liquide, e il suo algoritmo è stato descritto in dettaglio38,51. In breve, il software di stima TFx basato su CNA prevede SCNA su larga scala all'interno di cfDNA sequenziato utilizzando un modello di Markov nascosto. Le stime TFx sono derivate in base alla presenza di SCNA, tenendo conto delle differenze nella subclonalità e nella ploidia in ciascun locus, e da queste, il software di stima TFx basato su CNA sceglie una soluzione TFx ottimale38. Tuttavia, una limitazione intrinseca del software di stima TFx basato su CNA è che il TFx viene calcolato in base alla presenza di SCNA in un campione; pertanto, non è in grado di determinare il TFx in campioni senza SCNA (cioè con profili genomici piatti)38. Come è stato precedentemente dimostrato, non tutti i tumori RB hanno SCNA 14,15,18,21,23,45. Pertanto, un TFx molto basso, come determinato dal software di stima TFx basato su CNA, significa che 1) il campione di AH non contiene cfDNA derivato dal tumore misurabile o 2) il cfDNA tumorale è presente, ma non è rilevabile dal software di stima TFx basato su CNA a causa della mancanza di SCNA38. La frazione allelica variante (VAF) per SNV è un surrogato di TFx. Per questo motivo, stiamo lavorando per sviluppare una pipeline TFx basata su RB1 basata su VAF al fine di consentire agli occhi RB senza SCNA di ricevere anche il monitoraggio TFx longitudinale. Dato che tutti i tumori RB, con l'eccezione dei tumori primari guidati da MYCN 14,15,42,43,44,45, contengono mutazioni somatiche nel gene RB1, una pipeline non dipendente dagli SCNA amplierebbe l'applicazione del protocollo di biopsia liquida qui presentato. Inoltre, poiché gli SNV possono essere monitorati fino a TFx inferiori al 5%, ciò aumenterebbe la sensibilità della nostra piattaforma.
La piattaforma per biopsia liquida AH qui descritta non è l'unica piattaforma per biopsia liquida esistente in letteratura per RB, ma in particolare, è la prima a descrivere l'acquoso come fonte arricchita di DNA tumorale e la prima a descrivere la rilevazione del ctDNA attraverso due meccanismi (SCNA e SNV RB1) in qualsiasi biofluido sulla base del lavoro pubblicato fino ad oggi. Con la capacità di rilevare il ctDNA in due modi, la biopsia liquida AH ha una sensibilità maggiore rispetto ad altre piattaforme in letteratura. Ad esempio, un altro gruppo ha rilevato con successo il ctDNA RB nell'AH con gli SNV 52 di RB1. Tuttavia, questa piattaforma si basava su letture NGS mirate basate sulla conoscenza a priori del SNV. Al contrario, la piattaforma descritta in questo manoscritto utilizza WGS non distorti, dandogli la capacità di rilevare SCNA e SNV. Sono state provate anche biopsie liquide basate sul sangue, sebbene il cfDNA ottenuto sia stato costantemente al di sotto della soglia per il rilevamento di SCNA, che è la prognostica per la probabilità di salvataggio oculare (in questo momento, gli SNV RB1 non hanno dimostrato di essere prognostici per il salvataggio oculare, Tuttavia, potrebbe esserci un ruolo evolutivo per l'individuazione della malattia metastatica)19. Kothari et al. hanno descritto gli SNV RB1 nel plasma dei pazienti con RB, ma solo di quelli con una malattia intraoculare avanzata che richiede l'enucleazione16. La piattaforma per biopsia liquida AH qui descritta ha la capacità di rilevare il ctDNA nell'AH di occhi meno avanzati, anche senza la presenza di semina19,21. Inoltre, le biopsie liquide basate sul sangue non sono specifiche per l'occhio, poiché il ctDNA isolato dal siero può provenire da entrambi gli occhi in caso di RB bilaterale. Ciò limita l'utilità clinica delle piattaforme ematiche, in particolare nei pazienti bilaterali che costituiscono il 40% dei casi di RB, mentre l'AH rimane specifico per gli occhi e può dimostrare eterogeneità interoculare sia a livello di SNV che di SCNA53.
L'importanza della biopsia liquida AH per il campo della RB è fondamentale. La biopsia liquida AH non solo offre l'opportunità di comprendere meglio le dinamiche intratumorali negli occhi che sono attivamente sottoposti a terapia, ma ha anche il potenziale per migliorare la cura del paziente. Sulla base di studi precedenti, abbiamo stabilito una firma molecolare, basata sulla presenza di amplificazione MYCN o di guadagno del cromosoma 6p con un'ampiezza di ≥1,5 rispetto alla mediana, che è prognostica per una probabilità 16,5 volte maggiore di fallimento del trattamento che richiede l'enucleazione 18,20,21. Con queste conoscenze al momento della diagnosi, i medici sarebbero in grado di consigliare meglio le famiglie sulle opzioni di trattamento appropriate e sulla probabilità di salvataggio degli occhi con le terapie attuali. Sebbene la validità clinica della piattaforma per biopsia liquida AH per RB sia stata stabilita 17,18,19,20,21,22,23, è attualmente approvata solo per la ricerca; Sono necessari studi prospettici multicentrici più ampi prima che la biopsia liquida dell'AH possa essere implementata clinicamente per aiutare a indirizzare la cura del paziente per la RB. Indipendentemente da ciò, la biopsia liquida AH ha il potenziale per consentire un'oncologia di precisione in futuro, non solo per RB, ma anche per altri tumori oculari.
Divulgazioni
Jesse Berry, Liya Xu e James Hicks hanno depositato una domanda di brevetto intitolata Aqueous Humor Cell-Free DNA for Diagnostic and Prognostic Evaluation of Ophthalmic Disease. In caso contrario, gli autori non segnalano alcun potenziale conflitto di interessi.
Riconoscimenti
Questa ricerca è stata supportata dalle seguenti fonti: NCI del NIH Award K08CA232344 (a J. L. Berry); Hyundai Hope on Wheels RGA012351 (a J. L. Berry); Childhood Eye Cancer Trust (a J.L. Berry); American Cancer Society IRG-16-181-57 (a J. L. Berry); Fondazione Wright (a J.L. Berry e M.E. Kim); Knights Templar Eye Foundation (a J.L. Berry); La Fondazione Larry e Celia Moh (a J. L. Berry); l'Istituto per le Famiglie, Inc., Ospedale Pediatrico di Los Angeles (J. L. Berry); una sovvenzione dipartimentale illimitata da Research to Prevent Blindness (tutti); L'NCI P30CA014089 (tutti); Fondo di ricerca Vicky Joseph (a P. Kuhn); Carol Vassiliadis Research Fund (a P. Kuhn); e USC Dornsife College of Letters, Arts and Sciences (a P. Kuhn).
Materiali
| Name | Company | Catalog Number | Comments |
| 1 cc syringe | surgical grade, whatever available in hospital | ||
| 32 G needle | surgical grade, whatever available in hospital | ||
| Aligner | Authors use Bowtie2 (http://bowtie-bio.sourceforge.net/bowtie2/index.shtml) but other aligners such as BWA or GRCh38 will also work | ||
| Atropos | generic term: adapter remover. https://atropos.readthedocs.io/en/latest/index.html# | ||
| Bioanalyzer High Sensitivity DNA Kit | Agilent | 5067-4626 | generic term: DNA fragment size profiling assay |
| BWA-MEM | generic term: long sequence aligner. http://bio-bwa.sourceforge.net/bwa.shtml | ||
| DNAcopy | Bioconductor | generic term: DNA copy number data analysis. https://bioconductor.org/packages/release/bioc/html/DNAcopy.html | |
| dsDNA High Sensitivity Assay | Qubit | Q32851 | generic term: DNA quantification assay |
| FreeBayes | generic term: sequence variant determiner. https://github.com/freebayes/freebayes | ||
| ichorCNA software | generic term: CNA-based TFx estimation. https://github.com/broadinstitute/ichorCNA | ||
| Illumina platform | Illumina | generic term: NGS platform; please note that other NGS platforms will work in principle, but have not been trialed by these authors | |
| NovoAlign (v3) | Novocraft | generic term: mapping of short reads onto reference genome. http://www.novocraft.com/products/novoalign/ | |
| QIAamp Circulating Nucleic Acid Kit | Qiagen | 55114 | generic term: cfDNA isolation and purification kit |
| QIAseq Ultralow Input Library Kit | Qiagen | 180492 | generic term: DNA library sequencing kit |
| Samtools rmdup | generic term: tool to remove duplicate reads. http://www.htslib.org/doc/samtools-rmdup.html | ||
| VarDict | generic term: variant caller. https://github.com/AstraZeneca-NGS/VarDict | ||
| Variant Effect Predictor | Ensembl | generic term: variant effect determinator. https://uswest.ensembl.org/info/docs/tools/vep/index.html |
Riferimenti
- Fernandes, A. G., Pollock, B. D., Rabito, F. A. Retinoblastoma in the United States: A 40-year incidence and survival analysis. Journal of Pediatric Ophthalmology & Strabismus. 55 (3), 182-188 (2018).
- Linn, A. M. Intraocular retinoblastoma: the case for a new group classification. Ophthalmology Clinics of North America. 18 (1), 41-53 (2005).
- Shields, J. A., Shields, C. L., Ehya, H., Eagle, R. C., De Potter, P. Fine-needle aspiration biopsy of suspected intraocular tumors. The 1992 Urwick lecture. Ophthalmology. 100 (11), 1677-1684 (1993).
- Karcioglu, Z. A., Gordon, R. A., Karcioglu, G. L. Tumor seeding in ocular fine needle aspiration biopsy. Ophthalmology. 92 (12), 1763-1767 (1985).
- Karcioglu, Z. A. Fine needle aspiration biopsy (FNAB) for retinoblastoma. Retina. 22 (6), 707-710 (2002).
- Eide, N., Syrdalen, P., Walaas, L., Hagmar, B. Fine needle aspiration biopsy in selecting treatment for inconclusive intraocular disease. Acta Ophthalmologica Scandinavica. 77 (4), 448-452 (1999).
- Eide, N., Walaas, L. Fine-needle aspiration biopsy and other biopsies in suspected intraocular malignant disease: a review. Acta Ophthalmologica. 87 (6), 588-601 (2009).
- Eriksson, O., Hagmar, B., Ryd, W. Effects of fine-needle aspiration and other biopsy procedures on tumor dissemination in mice. Cancer. 54 (1), 73-78 (1984).
- Ali, M. J., Honavar, S. G., Vemuganti, G. K., Singh, A. D. Fine needle aspiration biopsy of retinal tumors. Monographs in Clinical Cytology. 21, 72-81 (2012).
- Di Nicolantonio, F. a. l., et al. The chemosensitivity profile of retinoblastoma. Recent Results in Cancer Research. 161, 73-80 (2003).
- Francis, J. H., et al. Efficacy and toxicity of second-course ophthalmic artery chemosurgery for retinoblastoma. Ophthalmology. 122 (5), 1016-1022 (2015).
- Francis, J. H. Efficacy and toxicity of intravitreous chemotherapy for retinoblastoma: Four-year experience. Ophthalmology. 124 (4), 488-495 (2017).
- Francis, J. H., et al. Current treatment of bilateral retinoblastoma: The impact of intraarterial and intravitreous chemotherapy. Neoplasia. 20 (8), 757-763 (2018).
- Rushlow, D. E., et al. Characterisation of retinoblastomas without RB1 mutations: genomic, gene expression, and clinical studies. The Lancet Oncology. 14 (4), 327-334 (2013).
- Afshar, A. R., et al. Next-generation sequencing of retinoblastoma identifies pathogenic alterations beyond RB1 inactivation that correlate with aggressive histopathologic features. Ophthalmology. 127 (6), 804-813 (2020).
- Kothari, P. Cell-free DNA profiling in retinoblastoma patients with advanced intraocular disease: An MSKCC experience. Cancer Medicine. 9 (17), 6093-6101 (2020).
- Berry, J. L., et al. Potential of aqueous humor as a surrogate tumor biopsy for retinoblastoma. JAMA Ophthalmology. 135 (11), 1221-1230 (2017).
- Berry, J. L., et al. Genomic cfDNA analysis of aqueous humor in retinoblastoma predicts eye salvage: the surrogate tumor biopsy for retinoblastoma. Molecular Cancer Research. 16 (11), 1701-1712 (2018).
- Berry, J. L., et al. Aqueous humor is superior to blood as a liquid biopsy for retinoblastoma. Ophthalmology. 127 (4), 552-554 (2020).
- Xu, L., et al. Chromosome 6p amplification in aqueous humor cell-free DNA Is a prognostic biomarker for retinoblastoma ocular survival. Molecular Cancer Research. 18 (8), 1166-1175 (2020).
- Xu, L., et al. Establishing the clinical utility of ctDNA analysis for diagnosis, prognosis, and treatment monitoring of retinoblastoma: The aqueous humor liquid biopsy. Cancers. 13 (6), (2021).
- Xu, L. Simultaneous identification of clinically relevant RB1 mutations and copy number alterations in aqueous humor of retinoblastoma eyes. Ophthalmic Genetics. 41 (6), 526-532 (2020).
- Polski, A., et al. Longitudinal aqueous humor sampling reflects treatment response in retinoblastoma patients. Investigative Ophthalmology & Visual Science. 61 (7), 1394 (2020).
- . Genome Browser Available from: https://genome.ucsc.edu/cgi-bin/hgTracks?db=hg19&lastVirtModeType=default&lastVirtModeExtraState=&virtModeType=default&3A15578261%_2D15621068&hgsid=1135765621_qIuA0U (2021)
- Navarro Gonzalez, J., et al. The UCSC genome browser database: 2021 update. Nucleic Acids Research. 49 (1), 1046-1057 (2021).
- Li, H. A statistical framework for SNP calling, mutation discovery, association mapping and population genetical parameter estimation from sequencing data. Bioinformatics. 27 (21), 2987-2993 (2011).
- Baslan, T., et al. Genome-wide copy number analysis of single cells. Nature Protocols. 7 (6), 1024-1041 (2012).
- Huber, W., et al. Orchestrating high-throughput genomic analysis with Bioconductor. Nature Methods. 12 (2), 115-121 (2015).
- Baslan, T., et al. Erratum: Genome-wide copy number analysis of single cells. Nature Protocols. 11 (3), 1024-1041 (2016).
- Didion, J. P., Martin, M., Collins, F. S. Atropos: specific, sensitive, and speedy trimming of sequencing reads. PeerJ. 5, 3720 (2017).
- Li, H. Aligning sequence reads, clone sequences and assembly contigs with BWA-MEM. arXiv. , (2013).
- Garrison, E., Marth, G. Haplotype-based variant detection from short-read sequencing. arXiv. , (2012).
- McLaren, W., et al. The ensembl variant effect predictor. Genome Biology. 17 (1), 122 (2016).
- Lai, Z., et al. VarDict: a novel and versatile variant caller for next-generation sequencing in cancer research. Nucleic Acids Research. 44 (11), 108 (2016).
- . GitHub Available from: https://github.com/broadinstitute/ichorCNA (2017)
- Adalsteinsson, V. A., et al. Scalable whole-exome sequencing of cell-free DNA reveals high concordance with metastatic tumors. Nature Communications. 8 (1), 1-13 (2017).
- . GitHub Available from: https://github.com/broadinstitute/ichorCNA/wiki/Interpreting-ichorCNA-results (2017)
- Berry, J. L., et al. Long-term outcomes of Group D retinoblastoma eyes during the intravitreal melphalan era. Pediatric Blood & Cancer. 64 (12), (2017).
- Berry, J. L., et al. Long-term outcomes of Group D eyes in bilateral retinoblastoma patients treated with chemoreduction and low-dose IMRT salvage. Pediatric Blood & Cancer. 60 (4), 688-693 (2013).
- Francis, J. H., et al. Molecular changes in retinoblastoma beyond RB1: Findings from next-generation sequencing. Cancers (Basel). 13 (1), (2021).
- Schwermer, M., et al. Comprehensive characterization of RB1 mutant and MYCN amplified retinoblastoma cell lines. Experimental Cell Research. 375 (2), 92-99 (2019).
- Davies, H. R. Whole-genome sequencing of retinoblastoma reveals the diversity of rearrangements disrupting RB1 and uncovers a treatment-related mutational signature. Cancers. 13 (4), 754 (2021).
- Lillington, D. M., et al. Comparative genomic hybridization of 49 primary retinoblastoma tumors identifies chromosomal regions associated with histopathology, progression, and patient outcome. Genes, Chromosomes and Cancer. 36 (2), 121-128 (2003).
- Greger, V., et al. Frequency and parental origin of hypermethylated RB1 alleles in retinoblastoma. Human Genetics. 94 (5), 491-496 (1994).
- Raizis, A. M., et al. DNA hypermethylation/boundary control loss identified in retinoblastomas associated with genetic and epigenetic inactivation of the RB1 gene promoter. Epigenetics. 1-15, (2020).
- Munier, F. L., et al. Profiling safety of intravitreal injections for retinoblastoma using an anti-reflux procedure and sterilisation of the needle track. British Journal of Ophthalmology. 96 (8), 1084-1087 (2012).
- Chévez-Barrios, P., et al. Response of retinoblastoma with vitreous tumor seeding to adenovirus-mediated delivery of thymidine kinase followed by ganciclovir. Journal of Clinical Oncology. 23 (31), 7927-7935 (2005).
- Shao, W., Khin, S., Kopp, W. C. Characterization of effect of repeated freeze and thaw cycles on stability of genomic DNA using pulsed field gel electrophoresis. Biopreservation and Biobanking. 10 (1), 4-11 (2012).
- Choudhury, A. D., et al. Tumor fraction in cell-free DNA as a biomarker in prostate cancer. JCI Insight. 3 (21), (2018).
- Gerrish, A., et al. Non-invasive diagnosis of retinoblastoma using cell-free DNA from aqueous humour. British Journal of Ophthalmology. 103 (5), 721-724 (2019).
- Wong, E. Y., Xu, L., Shen, L. Inter-eye genomic heterogeneity in bilateral retinoblastoma via aqueous humor liquid biopsy. NPJ Precis Oncol. 5 (1), 73 (2021).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneEsplora altri articoli
This article has been published
Video Coming Soon