Method Article
房水作为视网膜母细胞瘤的液体活检:透明角膜穿刺术和基因组分析
摘要
房水是一种针对视网膜母细胞瘤的高产液体活检,视网膜母细胞瘤是一种由于眼外扩散的风险而无法 在体内 活检的眼内癌。在此,提出了一种通过透明角膜穿刺术安全提取房水的方法以及基因组分析以识别预后生物标志物的步骤。
摘要
鉴于这些患者禁止直接进行肿瘤活检,因此应用液体活检平台治疗视网膜母细胞瘤具有显着的潜在临床效用。房水 (AH) 在与肿瘤不同的隔室中形成,但被封闭在相同的眼腔内。因此,它是眼睛特异性肿瘤基因组信息的丰富来源,可用作液体活检或替代该疾病的肿瘤活检。本手稿详细介绍了一种通过透明角膜穿刺术从视网膜母细胞瘤眼中安全提取 AH 的方法。此外,还介绍了基因组分析的步骤,包括游离 DNA 分离和纯化、下一代测序、体细胞拷贝数改变 (SCNA) 分析、 RB1 单核苷酸变异 (SNV) 突变鉴定和肿瘤分数估计。已经评估了 AH 液体活检平台的分析前、分析和早期临床有效性;但是,它并非没有限制。这些主要是由于检测的某些步骤所需的游离 DNA 数量的结果。与目前正在研究视网膜母细胞瘤的其他基于血液的液体活检平台相比,基于 AH 的平台受到可从眼睛中提取的生物液体积(以及 DNA 数量)的限制;好处是 AH 是眼睛特异性的。这里讨论的平台是独一无二的,因为它通过两种机制(SCNA 和 RB1 SNV)检测 AH 中的循环肿瘤 DNA,从而为识别肿瘤基因组信息产生更高的灵敏度。AH 液体活检有可能直接临床应用于视网膜母细胞瘤患者的精准肿瘤学,对双侧疾病患者尤其重要,因为 AH 对每只眼睛的肿瘤具有特异性。目前正在研究将该平台应用于其他眼部肿瘤患者。
引言
视网膜母细胞瘤 (RB) 是一种罕见且独特的癌症。虽然它是在婴幼儿发育中的视网膜中形成的最常见的原发性眼内恶性肿瘤,但世界上每年只有大约 7000 例病例,其中大约 250-300 例在美国。尽管发达国家的患者生存率接近 98%1,但临床分类为国际眼内 RB 分类 (IIRC)2 D/E 组或 AJCC cT2b/3 的高级眼的眼部存活率要低得多。许多这些先进的眼主要是(作为第一次治疗)或次要(在尝试挽救眼球疗法失败后)摘除的。在目前的眼肿瘤学临床实践中,目前临床上没有肿瘤来源的、眼睛特异性的分子生物标志物来辅助诊断、眼生存预后或RB患者的治疗监测。这在很大程度上是由于肿瘤组织只能用于去核眼的分子和遗传分析,因为由于担心眼外肿瘤扩散,禁止对 RB 进行直接肿瘤活检 3,4,5,6,7,8,9 .由于这一禁止,以前关于 RB 肿瘤遗传学及其临床相关性的回顾性研究仅限于分析从去核眼获得的肿瘤组织 10,11,12,13。因此,在诊断和整个抢眼治疗过程中一直缺乏客观的肿瘤衍生分子数据。这不仅限制了对体内肿瘤生物学的理解以及这些肿瘤在整个治疗过程中在分子水平上变化的方式,还限制了为这些年轻癌症患者制定个性化、眼睛特异性、基于基因组的治疗计划的能力。
除了禁止活检外,这种癌症的另一个独特之处在于,大多数肿瘤是由调节细胞周期的 RB1 肿瘤抑制基因的双等位基因丢失引发的。发育中的视网膜对这种损失非常敏感。在 60% 的患者中,RB1 基因的这种双等位基因失活仅以体细胞丢失的形式发生在视网膜中,并导致单侧疾病。然而,在 40% 的患者中,最初的 RB1 突变发生在种系中,然后是视网膜中的第二次“打击”。在这些儿童中,通常有多个肿瘤影响双眼。最后,极小的肿瘤亚群 (<2%) 似乎是由 MYCN 扩增驱动的,而 RB1 基因没有突变。虽然 MYCN 驱动的肿瘤几乎普遍治疗失败并需要摘除术,但目前没有明确、客观的方法来识别这种侵袭性肿瘤亚型14,15。此外,眼内 RB 肿瘤活动的监测几乎完全依赖于治疗眼肿瘤学家对每只眼睛的影像学和临床观察。在整个治疗过程中,没有金标准的客观、定量诊断、预后或监测眼特异性肿瘤动力学的方法。由于 RB 的这些独特局限性,针对这种癌症的液体活检平台的前景很诱人。液体活检利用体液分离和测序游离 DNA,以确定它是否是肿瘤来源的,称为循环肿瘤 DNA (ctDNA)。虽然血清被研究用于其他癌症,但在血清中发现的 RB ctDNA 不是眼睛特异性的,这对 40% 的双侧疾病患者有明显的局限性。此外,它仅在晚期眼内或转移性疾病的情况下被描述,通常肿瘤分数较低 (<5%)16。
为了解决患者和家属面临的这些实际临床问题,我们在 2017 年证明了房水(AH,眼前的透明液体)是一种高产量的 ctDNA 来源,可以用作液体活检——或者更确切地说是作为肿瘤活检的替代品——来评估 RB17,18,19.迄今为止,在 200 多个样本中,鉴定出 >95% 的样本中的 ctDNA,包括较不晚期的 IIRC A、B 和 C 组眼睛,以及双侧患者眼睛之间的基因组差异18,19。这种 DNA 可以安全有效地分离出来,用于检测正在积极接受治疗或未接受过治疗的眼睛中的相关分子生物标志物 18,20,21。AH 液体活检平台也可用于识别启动肿瘤发生的诊断性 RB1 致病性变异或原发性 MYCN 扩增21,22。重要的是,我们已经确定了一个预后分子特征——存在染色体 6p 增益,与正中或局灶性 MYCN 扩增的幅度为 ≥1.5——这与需要摘除眼睛的眼内肿瘤控制丧失的可能性增加 16.5 相关 18,20,21.最后,已经证明 AH 中 ctDNA 肿瘤分数 (TFx) 的变化与治疗反应相关,因为较高的水平与活动性疾病相关,而降低的水平与积极的治疗反应相关23。鉴于这些应用及其潜在的临床用途,我们想描述 AH 活检和评估的方法。这包括用于样本采集的透明角膜穿刺术和基因组分析方案,特别是 cfDNA 文库构建和测序、SCNA 振幅测定、RB1 致病性变异鉴定和 TFx 计算。
研究方案
这项研究是在洛杉矶儿童医院和南加州大学机构审查委员会的批准下进行的,并遵守赫尔辛基宣言的原则。书面知情同意书始终来自所有参与者的法定监护人。
AH 液体活检工作流程的示意图如图 1 所示。

图 1:AH 液体活检伴随本文所述方案的工作流程。 EUA,麻醉下检查;LBX,液体活检;cfDNA,游离 DNA;CNA,拷贝数改变;TFx,肿瘤分数;IVM,玻璃体内美法仑。 请单击此处查看此图的较大版本。
1. 手术程序:
注意:该程序是在麻醉下常规检查 (EUA) 期间进行的,用于 RB 患者的临床评估。提取 AH 的穿刺术只能由已完成眼外科标准培训的训练有素的眼科医生完成。
- 患者选择和纳入标准如下。
- 确保腔室形成且清晰,没有肿瘤、粘连或白内障晶状体的浅层。
注意:在一些高级眼睛中,诊断时腔室太浅,无法安全地提取 AH。在这些情况下,建议等到全身或动脉内化疗的第一个周期之后。一旦主要肿瘤缩小,心房通常会加深,这为安全的方法提供了途径。 - 确保所有结构都有清晰的视野,并且压力为 <22 mm Hg,这样就不会由于压力动力学而使腔室迅速变浅。这适用于所有时间点,但在诊断时服用 AH 时尤其重要。
- 确保肿瘤没有直接累及眼前节,这可以通过直接可视化和超声生物显微镜进行评估。此纳入标准通常在 EUA 期间确定。
- 确保腔室形成且清晰,没有肿瘤、粘连或白内障晶状体的浅层。
- 一旦认为患者合适并同意,将患者带到手术室。允许主治麻醉师开始麻醉,这是 EUA 对 RB 患者临床护理的常规。
- 一旦患者插管和镇静,并且所有与 EUA 相关的临床检查、影像学检查和任何局部治疗需求(例如,激光或冷冻疗法)都已完成,即可提取液体活检平台的 AH。以通常的无菌方式准备并覆盖眼睛。这可以由循环护士或手术外科医生完成。
- 在 EUA 之前,将机构常规用于 EUA 的散瞳滴剂滴入手术眼中。
注意:这里使用由 2.5% 去氧肾上腺素、1% 环喷托酯和 1% 托吡卡胺组成的组合滴眼液。然而,任何用于 EUA 扩张的标准滴剂都足够了;不必简单地为了提取 AH 而散瞳。 - 将 5% 优碘滴剂滴入手术眼中,并使用海绵从中心开始向周边移动。在手术准备中包括上眼睑缘和下眼睑缘。使用 betadine 完成手术准备 3 次,然后等待至少 2 分钟后再继续。
- 将一条无菌蓝色毛巾盖在患者的头上,并将其塞在头下以防止其掉落。用无菌的蓝色毛巾或纱布擦干睫毛和眶周区域。
- 让手术外科医生擦洗身体,穿上长袍,戴上手套。
- 用无菌的蓝色窗帘盖住患者,让手术眼暴露在外。放置无菌眼睑窥器,以实现无菌区域的最佳可视化。
注意:我们机构不需要额外的局部麻醉,因为孩子正在接受 EUA 的全身麻醉。 - 根据机构协议执行暂停。在这里,对 EUA 进行初始超时,并在手术前进行第二次超时以验证眼睛。
- 在 EUA 之前,将机构常规用于 EUA 的散瞳滴剂滴入手术眼中。
- 使用手术显微镜,进行透明的角膜穿刺术,在 1 cc 注射器上用 32 G 针头提取 0.1 mL AH。
- 手术开始前,用无菌平衡盐溶液(每毫升含氯化钠 0.64%、氯化钾 0.075%、氯化钙二水合物 0.048%、氯化镁 0.03%、乙酸钠三水合物 0.39%、柠檬酸钠二水合物 0.17%、氢氧化钠和/或盐酸(调节 pH 值)和注射用水)湿润眼睛,以保持角膜润滑;这是一种标准的商业制剂。
- 确保 32 G 针头鲁尔锁紧到 1 cc 注射器上,并且注射器中没有压力(使用前将注射器的柱塞移入和移出数次)。
- 之后,将针头穿过角膜缘处的透明角膜(垂直于角膜,这是穿刺术的标准),并停留在外周扩张虹膜的前房内。
- 在提取过程中,通过显微镜直接观察,保持针尖斜面向上。如果愿意,当针头穿过角膜时,用 0.12 把镊子稳定眼睛。
注意: 针尖和任何眼部结构之间不应有接触;它应该保持在中前房的周边虹膜上。 - 提取 0.1 mL AH。用外科医生的非惯用手(但要练习)或由训练有素的助手作注射器柱塞,而无需移动针头。不要失去针尖的直接观察。优先考虑眼睛的解剖结构,必要时减少服用 AH 以确保腔室保持形成。
- 提取后,轻轻地从前房中取出针头。确保腔室保持成型但略浅;压力将是柔和的,但是生理性的。将针头从眼睛中取出后,用无菌水充分沐浴眼睛;作为一项安全措施,在此步骤中,无菌水优于平衡盐溶液,因为水会裂解细胞。
- 检查针头部位是否有任何泄漏。如果有泄漏,请使用棉签涂抹器在注射部位轻轻按压 30 秒,然后再次检查是否有泄漏。如果需要,请重复该过程。
注意:这种方法从不需要使用更具侵入性的方法来阻止任何泄漏。有时,该手术之后是玻璃体内注射化疗;对于本方法论文,该协议侧重于 AH 提取作为主要程序。
- 检查针头部位是否有任何泄漏。如果有泄漏,请使用棉签涂抹器在注射部位轻轻按压 30 秒,然后再次检查是否有泄漏。如果需要,请重复该过程。
- 一旦没有泄漏,该过程就被认为是完整的。取下患者身上的无菌窗帘并清洁脸上的优碘。
- 小心地从注射器中取出针尖,使用无菌帽通过鲁尔锁关闭样品,以免丢失任何样品,并在注射器上贴上适当的标签。
注意:这应由临床研究协调员或外科医生在孩子拔管时完成。- 样品提取后,将样品储存在 -80 °C 的带盖注射器中;样本可以放在干冰上,从手术室运送到合适的冰箱。
- 在将患者送往康复中心之前,根据外科医生的喜好将一滴抗生素滴眼液滴在手术眼的表面。在这里,使用类固醇-抗生素复方滴眼液,并且不遮盖眼睛。
- 手术完成后,主治麻醉师会确保正确拔管。让患者康复。
- 按照机构术后方案将患者出院回家。
- 告诉患者,眼睛不需要用任何绷带或保护罩覆盖。
- 告知患者 24 小时内不要将头部完全浸入浴缸或游泳池中,父母不要揉搓患者(孩子)的眼睛;患者活动没有其他限制。
- 告诉患者,他/她/他们不太可能因穿刺术而疼痛,但在 EUA 期间可能会因相关手术而感到疼痛。按照外科医生的建议,如果出现任何不适,患者可以服用对乙酰氨基酚或布洛芬。
2. cfDNA 分离和纯化
- 在 AH 提取后 72 小时内进行 DNA 分离和处理。
- 在室温下解冻 AH。经常检查样品,并在解冻完成后立即进行提取。
- 根据制造商的说明手册,使用 cfDNA 分离和纯化试剂盒(详见 材料表 )从 AH 中提取 cfDNA 并将其洗脱到 50 μL AVE 溶液(包含在试剂盒中)中。
3. 下一代测序 (NGS) 和质量控制 (QC)
- 根据制造商的说明手册,使用 DNA 文库测序试剂盒(参见 材料表)构建用于测序的 DNA 文库。使用 DNA 文库测序试剂盒手册中详述的 14 个聚合酶链反应 (PCR) 循环扩增文库 DNA。
注意:最终文库可以在最终测序前在 -20 °C 下储存长达 1 个月以进行基因组分析。 - 按照制造商的方案,按照单端 50 个循环或双端 150 个循环方案,在 NGS 平台上对 DNA 文库进行测序(参见 材料表)。
注:其他 NGS 平台原则上可以工作(尽管此处未尝试),并为测序测序测序流通池提供合适的适配器。 - 对诊断或初次眼球摘除时采集的任何 AH 样本进行 QC。
注:不能对在其他时间点采集的样品进行 QC,因为 DNA 质量太低,超出下述步骤的检测范围。- 根据制造商的方案,分别使用 1 μL 提取的 DNA 进行 DNA 定量测定和大小分析测定(参见 材料表)。
注意:峰值应在 300 bp 左右。如果它短于 150 bp,则样品已受损。有关应通过 QC 的样品示例,请参阅 补充文件 1A ,有关不应通过 QC 的样品示例,请参阅 补充文件 1B 。
- 根据制造商的方案,分别使用 1 μL 提取的 DNA 进行 DNA 定量测定和大小分析测定(参见 材料表)。
4. 体细胞拷贝数改变 (SCNA) 数据分析
- 使用比对器将从 NGS(第 3 部分)获得的读数映射到人类基因组(hg19、基因组参考联盟 GRCh37、加州大学圣克鲁斯基因组浏览器数据库)24,25。
- 删除 PCR 重复项 (samtools rmdup26)。
- 通过计算参考基因组中每个 bin 中鸟嘌呤和胞嘧啶碱基的百分比,对鸟嘌呤-胞嘧啶含量进行标准化。Baslan et al.27 描述了一个示例程序。
- 为了获得 DNA 片段拷贝数,请将基因组分成 5000 个可变长度的 bins,然后计算每个 bin中包含的相对读数数。
- 使用无参考 log2 比率来确定拷贝数估计值。取正常常染色体的中位窗口计数。
- 使用带有 DNAcopy 的循环二元分割 (Bioconductor28) 进行分割。
- 将 SCNA 定义为相对于基线偏转 20% 时为阳性(log2 比率 = 0),这意味着 log2 比率≤ 0.2 的损失(比率为 0.87 或更低定义为缺失),log2 比率≥ 0.2 时的增益(比率为 1.15 或更高定义为扩增)。
注意:这些阈值与先前建立的液体活检分析27,29 一致。
5. AH 样本的 RB1 突变分析
- 此外,根据制造商的方案,将全基因组文库扩增至每个 500 ng,用于基于捕获的靶向 NGS,用于突变检测。
- 根据制造商的方案,对捕获的文库进行 NGS 平台双端 150 bp 或 50 bp 单端测序,>100 倍。
- 处理 NGS 数据。在这项研究中,使用了基于 CHLA 个性化医学中心 bcbio 管道的内部管道30.
- 使用 Atropos31 修剪接头和低质量碱基的原始 fastq 数据,并使用 BWA-MEM 和 NovoAlign (v3) 32,33 与人类 GRCh37 参考对齐。
- 用 FreeBayes34 标记重复项,然后确定种系变体。
注意:在 FreeBayes 变体调用工具中,将样本基因组的每个位置与参考基因组进行比较,并对等位基因计数进行数学建模以获得基因型似然测量34。 - 按照步骤 5.3.4-5.3.5 使用 Ensembl Variant Effect Predictor (v96)35 进行变体注释。
- 使用 VarDict 确定 AH 或肿瘤中的体细胞变异,而无需配对的正常血液样本(即,对种系变异不知情)36。
- 如果一个区域的连续变体具有 ≤3% 的替代等位基因,则检出杂合性丢失。
注:商业基因组分析软件也可以与上述参数一起使用。
6. cfDNA TFx 的测定
- 使用 ichorCNA 软件(基于 CNA 的 TFx 估计软件)估计每个测序的 AH cfDNA 样品的 TFx37。
注:这是基于血液的液体活检中用于测定 cfDNATFx 38 的标准软件。该软件在 AH 上的使用之前已发布21,23。 - 单独查看基因组图谱和相应的 TFx 解决方案,以验证每个样本的 TFx 估计值是否合适39。
结果
两只眼(病例 33 和 47)的综合结果如下所示。病例数与以前的出版物保持一致,用于比较目的 18,20,21。治疗医师在治疗期间对 AH 液体活检的结果不知情。所有治疗决策都是非随机的,并根据先前发布的常规护理标准做出40,41。在最终分析之前,临床结果数据与基因组数据保持分离。所提供结果的原始数据可应通讯作者的要求提供。由于 NIH 资助,这些数据受 NIH 基因组数据共享政策的约束,并将在未来通过受控访问 NIH 指定数据存储库 (dbGAP) 提供给其他研究人员;也可向通讯作者索取。
病例 33 和 47 都是 IIRC D 组2 眼,临床表现非常相似。因此,根据父母的决定,他们接受了眼球挽救疗法,并接受了眼部和内科肿瘤学家的治疗——特别是病例 47 的全身化疗和病例 33 的动脉内化疗。根据目前公认的依赖于 IIRC组的 2 眼的临床预后,这两名患者眼球挽救的预测成功率是相同的:D 组的平均成功率为 65%-70%,尽管这因治疗中心而异41。然而,根据迄今为止从该中心的肿瘤定向分子分析中收集的 D 组数据,病例 47 的预测眼球挽救成功率为 72%(在 AH 中未发现 6p 增益),病例 33 的预测成功率为 9%(已发现 6p 增益)18,20,21。这如下所示。
病例 47 是在诊断时使用 AH 液体活检平台成功检测 SCNA 和 SNV 的眼睛示例,以及与纵向治疗反应相对应的 TFx 趋势。患者是一名女性,在 15 个月大时出现 14 mm x 9 mm IIRC D 组 cT2b RB 伴球状玻璃体接种。通过常规临床血清白细胞检测确定她的 RB1 种系突变呈阴性。从 AH 中,除了另外两个非高度复发性 RB SCNA 7p 丢失和 13q 增益外,在诊断时在 AH 中确定了 RB SCNA 1q 增益和 6p 增益(图 2)。值得注意的是,6p 增益的振幅为 1.2,只有与中位数之比≥ 1.5 的振幅被证明预示着预后不良。鉴于没有局灶性 MYCN 扩增且 6p 增益低于 1.5 阈值,基于分子特征的治疗挽救预后很高。还评估了诊断时采集的相同 AH 样本用于检测 RB1 致病性变异,结果显示 RB1 基因内的 SNV c.958C>T、p.Arg320* 具有 87.01% 的变异等位基因频率(95% 置信区间,79.7%–94.6%)。该患者接受了 6 个周期的卡铂、依托泊苷和长春新碱 (CEV) 治疗,疾病消退,但表现出持续的球状和尘埃样接种,需要连续 3 次玻璃体内美法仑 (IVM) 注射。在 IVM 治疗期间,AH 样本 A-C(每个样本间隔两周)显示基因组图谱完全正常化,TFx 降低,DNA 浓度降低——所有这些都与疾病的临床消退同时进行(图 3)。诊断后,在剩余的治疗中,TFx 值仍低于 5% 的检测限。在 19 个月的随访中,眼睛保持稳定,没有肿瘤复发或疾病的眼外扩散。
这与情况 33 形成鲜明对比。这名 22 个月大的男性临床表现总体相似,视网膜肿块和尘埃型玻璃体播散,与单侧 IIRC D 组 cT2b 期一致。通过常规临床血清白细胞检测确定,他的 RB1 种系突变也呈阴性。诊断时采集的 AH 显示 RB SCNAs 1q 增益、6p 增益(在这种情况下,振幅与中位数的比率为 1.5)和 16q 损失以及局灶性 6q 损失(图 4A)。根据预后分子特征,基于 6p ≥ 1.5 振幅的存在,这只眼的眼眼摘除的几率增加了 16.5。尽管完全覆盖了 RB1 基因的整个长度,但在该样本中未鉴定出 RB1 SNV。可能无法识别 RB1 SNV 的一个原因是在原发性 MYCN 驱动的肿瘤中,并不总是预期并发 RB1 突变 14,15,42,43,44,45。然而,病例 33 在其任何 AH 样本或其去核肿瘤组织中均未显示 MYCN 扩增的证据(图 5)。RB1 SNV 阴性结果的更可能解释是,最初的肿瘤发生是由表观遗传失调(例如,启动子甲基化)驱动的46,47,这是 RB 中的一种已知现象,无法通过本文描述的测定来识别。
该患者的初始治疗是 4 个总周期的美法仑动脉内化疗,然后由于持续的玻璃体接种而进行 4 次 IVM 注射。在 IVM 治疗期间获得了 3 个 AH 样本 (每个样本间隔 4 周),并显示出与诊断时相同的 3 个 SCNA(图 5)。TFx 值在整个治疗过程中保持较高水平,尽管原发肿瘤体积减少反映了玻璃体中的活性肿瘤种子。这表明 TFx 如何代表眼睛的整体疾病状态。诊断后 6 个月,由于持续活动性疾病,眼睛被摘除。当时从肿瘤组织获得的基因组图谱显示与诊断时获得的 AH 样本有 92.81% 的一致性(图 4B)。
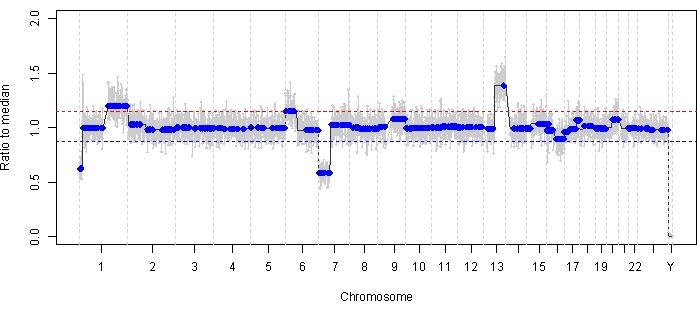
图 2:病例 47 诊断时的基因组图谱。 在诊断时采集的 AH 中发现了高度复发的 RB SCNAs 1q 增加和 6p 增加,以及非高度复发性的 RB SCNAs 7p 丢失和 13q 增加。红线表示收益阈值,而蓝线表示亏损阈值。值得注意的是,6p 增益的振幅与中位数的比率为 <1.5,低于预示不良预后的分子特征阈值。因此,基于缺乏用于眼睛挽救的阴性生物标志物,预计这只眼睛会对治疗产生反应。 请单击此处查看此图的较大版本。
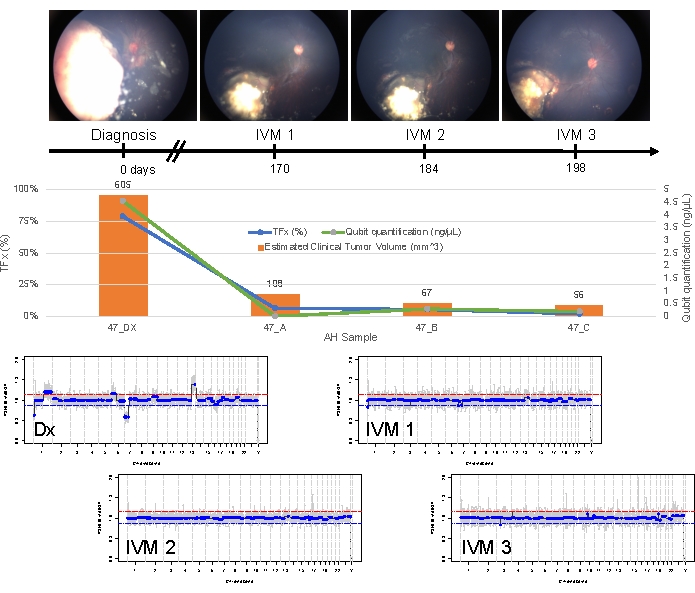
图 3:案例 47 的纵向信息。 这包括眼底照片、cfDNA 定量、B 扫描测量估计的临床肿瘤体积、基因组图谱以及对 AH 采样的每个临床时间点的 TFx 估计(Dx = 诊断,A = IVM1,B = IVM2,C = IVM3)。这只眼睛对治疗有反应,并在 19 个月的随访中保持挽救。随着时间的推移,观察到 cfDNA 数量减少,这与先前发表的研究一致,该研究显示诊断时获得最高产量。TFx 在治疗过程中也降低,反映了接种的分辨率和主要视网膜肿瘤体积的减少(肿瘤体积显示在每个临床时间点的橙色条上方)。正如预期的那样,疾病的临床消退也使基因组谱恢复正常。在基因组图谱中,红线表示增益阈值,而蓝线表示损失阈值。此图经 Xu, L. et al.21 许可转载。 请单击此处查看此图的较大版本。

图 4:案例 33 的基因组图谱。 红线表示增益阈值,而蓝线表示病例 33 诊断时损失 (A) 基因组图谱的阈值。除了局灶性 6q 丢失外,在诊断时在 AH 中发现了高度复发的 RB SCNAs 1q 增加、6p 增加和 16q 丢失。值得注意的是,6p 增益的幅度与中位数的比率为 1.5,表明预后不良的分子生物标志物。因此,根据这个分子特征,我们可以预测这只眼睛治疗失败的可能性显着增加。(B) 从去核肿瘤组织获得的基因组图谱,这与诊断时从 AH 获得的图谱高度一致。由于与正常视网膜组织混合,由于稀释了 TFx,与 AH 相比,来自肿瘤组织的 SCNA 可以表现出较低的振幅。 请单击此处查看此图的较大版本。

图 5:案例 33 的纵向信息。 这包括眼底照片、游离 DNA 定量、B 扫描测量估计的临床肿瘤体积(肿瘤体积显示在每个临床时间点的橙色条上方)、基因组图谱和对 AH 采样的每个临床时间点的 TFx 估计(Dx = 诊断,A = IVM1,B = IVM2,C = IVM3,SE = 二次眼球摘除)。这只眼睛对治疗没有反应,最终需要二次眼球摘除术 (ES)。这是由于持续活跃的接种伴随着根尖肿瘤复发。CfDNA 数量随着时间的推移而减少,与先前发表的研究一致,该研究显示诊断时获得的最高产量 (1q 增益、6p 增益、16q 丢失和局灶性 6q 丢失)。然而,TFx 在整个治疗过程中保持较高水平,这是持续接种的产物,仍然将肿瘤来源的 cfDNA 脱落到 AH 中。基因组图谱一致,并显示诊断时存在的相同 3 个 SCNA。在 ES 获得的 AH 中,观察到新的大规模 2p 增益 (*) 和 19q 丢失,表明根尖肿瘤复发时的克隆进化。在基因组图谱中,红线表示增益阈值,而蓝线表示损失阈值。此图经 Xu, L. et al.21 许可转载。 请单击此处查看此图的较大版本。
补充文件 1: 示例 (A) 应通过质量控制的样品,峰约为 300 bp,以及 (B) 不应通过质量控制的样品,峰约为 150 bp。 请点击此处下载此文件。
讨论
透明角膜穿刺术是眼科中通常用于多种诊断和/或治疗适应症的手术。特别是对于 RB,它是标准玻璃体内化疗注射方案的一部分,用于降低注射前的眼压以防止反流到注射部位48。尽管是一种常见的手术,但它并非完全没有风险;以前的教条是针头永远不应该进入 RB 活跃的眼睛。这一教条在过去十年中不断发展,首先是通过 Patricia Chévez-Barrios 关于注射腺病毒载体疗法的形成性工作49,然后是 Francis Munier48 对 RB 眼部注射的渐进式安全性增强方法。这项工作为提取 AH 用作这种癌症的液体活检或替代肿瘤活检铺平了道路。我们现在是第一个报告该平台的效用和诊断时 AH 的初步安全性结果的中心。本文描述的部分方法旨在确保 RB 的患者选择和程序方面的安全性。首先,针头只能进入前房,不应与虹膜或晶状体接触,因为这会导致虹膜瘢痕形成或白内障,从而限制外科医生监测肿瘤的能力。最重要的是,针头永远不会进入玻璃体腔(除非像 IVM 一样与化疗相结合)或接触肿瘤,因为这假设会增加肿瘤播散和疾病眼外扩展的风险 3,4,5。这两个结构都位于前房的后方,并通过晶状体和虹膜与前房隔开。为避免损伤任何眼部结构,重要的是要始终保持针头斜面向上,在周边虹膜上方,并始终使用手术显微镜进行直接观察。AH 有从针部位轻微泄漏的风险;如果发生这种情况,它将通过棉签涂抹器轻轻按压消退。使用最小的可用规格针头并确保针头从同一通道缓慢拔出,没有任何横向剪切,将降低这种风险。虽然预计前房会略微变浅,但前房应保持形成,无需虹膜角膜接触,眼压应较软,但符合生理作用。
关于基因组分析,涉及几个关键步骤。最重要的是处理从眼睛中提取的小体积样品;至关重要的是,样品在处理前的整个过程中保持冷冻状态,以防止反复冻融循环可能发生的 DNA 降解50。确保样品在提取后立即放在干冰上并转移到 -80 °C 冰箱中有助于确保这一点。一旦样品开始处理,质量控制步骤是解决问题的主要机会,可以保证 cfDNA 被构建到高质量的文库中。使用 DNA 定量和 DNA 片段大小分析分析,可以对初次摘除期间或诊断时采集的样品进行质量控制,因为这些样品中存在的 cfDNA 产量较高18,21。当获得的峰约为 300 bp 时,这保证了 gNGS 平台能够识别 cfDNA。如果获得的峰短于 150 bp,表明检测到的大多数片段是文库构建引物或衔接寡核苷酸,则样品已受损,不应进一步处理 SCNA。然而,根据我们处理数百个样品的经验,由于读数比对率不佳,需要去除不到 5% 的样品以进行质量控制20。除了我们标准化的标本采集、处理、储存和加工方法外,该程序还证明了 AH 液体活检平台的分析前有效性。
AH 液体活检还基于其准确可靠地检测 RB1 致病突变和 SCNA 的能力建立了分析有效性,AH 样本和相应肿瘤组织生成的基因组谱之间的平均一致性始终为 >95%18、20、21、22、23。尽管该平台证明了分析的有效性,但它并非没有限制。RB1 突变分析只能对含有 >10 ng cfDNA 的样本进行,这些 cfDNA 最常在诊断或初次摘除时在初治眼中获得18,21。这是因为与诊断时或初次摘除时的 AH 相比,正在积极接受治疗的眼睛的 AH 样本中存在的 cfDNA 浓度较低。另一个限制是 TFxs 低于 5% 时无法检测到 SCNA,这阻止了对肿瘤负荷显着下降的眼睛的疾病监测;根据我们的经验,这最常见于对玻璃体内化疗有反应的眼睛23。
为了确定 AH 中 cfDNA 的 TFx,使用了基于 CNA 的 TFx 估计软件。该软件是液体活检中 TFx 计算的标准和公认工具,其算法已详细描述38,51。简而言之,基于 CNA 的 TFx 估计软件利用隐藏马尔可夫模型预测测序 cfDNA 中的大规模 SCNA。TFx 估计是根据 SCNA 的存在得出的,同时考虑了每个基因座的亚克隆性和倍性差异,从这些中,基于 CNA 的 TFx 估计软件选择了最佳 TFx 解决方案38。然而,基于 CNA 的 TFx 估计软件的一个固有局限性是 TFx 是根据样本中 SCNA 的存在来计算的;因此,它无法确定不含 SCNA(即具有平坦基因组图谱)的样品中的 TFx38。如前所述,并非所有 RB 肿瘤都具有 SCNA14、15、18、21、23、45。因此,由基于 CNA 的 TFx 估计软件确定的非常低的 TFx 意味着 1) AH 样本中没有可测量的肿瘤来源的 cfDNA,或 2) 存在肿瘤 cfDNA,但由于缺乏 SCNA,基于 CNx 的 TFx 估计软件无法检测到38。SNV 的变异等位基因分数 (VAF) 是 TFx 的替代物。出于这个原因,我们正在努力开发基于 VAF 的基于 RB1 的 TFx 管道,以便让没有 SCNA 的 RB 眼也能接受纵向 TFx 监测。鉴于除原发性 MYCN 驱动的肿瘤14、15、42、43、44、45 外,所有 RB 肿瘤都含有 RB1 基因的体细胞突变,不依赖于 SCNA 的管道将拓宽本文提出的液体活检方案的应用。此外,由于 SNV 可以监测到 TFxs 低于 5%,这将提高我们平台的灵敏度。
此处描述的 AH 液体活检平台并不是文献中存在的唯一 RB 液体活检平台,但值得注意的是,它是第一个将房水描述为肿瘤 DNA 的富集来源,并且第一个描述通过两种机制(SCNA 和 RB1 SNV)检测 ctDNA 基于迄今为止已发表的工作在任何生物流体中。AH 液体活检能够以两种方式检测 ctDNA,因此比文献中的其他平台具有更高的灵敏度。例如,另一组在 AH 中成功检测到 RB ctDNA 和 RB1 SNV 52。然而,该平台依赖于基于 SNV 先验知识的靶向 NGS 读长。相比之下,本手稿中详述的平台利用无偏倚的 WGS,使其能够检测 SCNA 和 SNV。基于血液的液体活检也已被尝试,尽管获得的 cfDNA 一直低于 SCNA 检测的阈值,这是挽眼可能性的预后(目前,RB1 SNV 尚未显示对挽眼预后, 然而,检测转移性疾病的作用可能不断发展)19。Kothari 等人描述了 RB 患者血浆中的 RB1 SNV,但仅限于那些患有需要眼球摘除术的晚期眼内疾病的患者16。此处描述的 AH 液体活检平台能够检测较不高级眼睛的 AH 中的 ctDNA,即使不存在接种19,21。此外,基于血液的液体活检不是眼睛特异性的,因为在双侧 RB 病例中,从血清中分离的 ctDNA 可能来自双眼。这限制了基于血液的平台的临床效用,特别是在占 RB 病例 40% 的双侧患者中,而 AH 仍然是眼睛特异性的,并且可以在 SNV 和 SCNA 水平上表现出眼间异质性53。
AH 液体活检对 RB 领域的意义至关重要。AH 液体活检不仅提供了更好地了解正在积极接受治疗的眼睛的肿瘤内动力学的机会,而且还有可能改善患者护理。根据以前的研究,我们建立了一个分子特征——基于 MYCN 扩增或染色体 6p 增益的存在,振幅与中位数的比率为 ≥1.5——预后需要眼球摘除术的治疗失败的可能性增加 16.5 倍 18,20,21。在诊断时有了这些知识,临床医生将能够更好地就适当的治疗方案和当前疗法挽救眼睛的可能性向家庭提供咨询。尽管 RB 的 AH 液体活检平台的临床有效性已经确定17、18、19、20、21、22、23,但目前仅获准用于研究;在临床实施 AH 液体活检之前,需要进行更大规模的前瞻性多中心研究,以帮助指导 RB 的患者护理。无论如何,AH 液体活检有可能在未来实现精准肿瘤学,不仅适用于 RB,也适用于其他眼部肿瘤。
披露声明
Jesse Berry、Liya Xu 和 James Hicks 提交了一项名为“用于眼科疾病诊断和预后评估的房水游离 DNA”的专利申请。否则,作者报告没有潜在的利益冲突。
致谢
这项研究得到了以下来源的支持:NIH 奖 K08CA232344 的 NCI(授予 JL Berry);现代 Hope on Wheels RGA012351(给 J. L. Berry);儿童眼癌信托基金(J.L. Berry);美国癌症协会 IRG-16-181-57(致 JL Berry);莱特基金会(J.L. Berry 和 M.E. Kim);圣殿骑士眼基金会(J.L. Berry);Larry 和 Celia Moh 基金会(J. L. Berry 的);洛杉矶儿童医院家庭研究所 (J. L. Berry);来自 Research to Prevention Blindness 的无限制部门拨款(全部);NCI P30CA014089(全部);Vicky Joseph 研究基金(致 P. Kuhn);Carol Vassiliadis 研究基金(致 P. Kuhn);和 USC Dornsife College of Letters, Arts and Sciences(致 P. Kuhn)。
材料
| Name | Company | Catalog Number | Comments |
| 1 cc syringe | surgical grade, whatever available in hospital | ||
| 32 G needle | surgical grade, whatever available in hospital | ||
| Aligner | Authors use Bowtie2 (http://bowtie-bio.sourceforge.net/bowtie2/index.shtml) but other aligners such as BWA or GRCh38 will also work | ||
| Atropos | generic term: adapter remover. https://atropos.readthedocs.io/en/latest/index.html# | ||
| Bioanalyzer High Sensitivity DNA Kit | Agilent | 5067-4626 | generic term: DNA fragment size profiling assay |
| BWA-MEM | generic term: long sequence aligner. http://bio-bwa.sourceforge.net/bwa.shtml | ||
| DNAcopy | Bioconductor | generic term: DNA copy number data analysis. https://bioconductor.org/packages/release/bioc/html/DNAcopy.html | |
| dsDNA High Sensitivity Assay | Qubit | Q32851 | generic term: DNA quantification assay |
| FreeBayes | generic term: sequence variant determiner. https://github.com/freebayes/freebayes | ||
| ichorCNA software | generic term: CNA-based TFx estimation. https://github.com/broadinstitute/ichorCNA | ||
| Illumina platform | Illumina | generic term: NGS platform; please note that other NGS platforms will work in principle, but have not been trialed by these authors | |
| NovoAlign (v3) | Novocraft | generic term: mapping of short reads onto reference genome. http://www.novocraft.com/products/novoalign/ | |
| QIAamp Circulating Nucleic Acid Kit | Qiagen | 55114 | generic term: cfDNA isolation and purification kit |
| QIAseq Ultralow Input Library Kit | Qiagen | 180492 | generic term: DNA library sequencing kit |
| Samtools rmdup | generic term: tool to remove duplicate reads. http://www.htslib.org/doc/samtools-rmdup.html | ||
| VarDict | generic term: variant caller. https://github.com/AstraZeneca-NGS/VarDict | ||
| Variant Effect Predictor | Ensembl | generic term: variant effect determinator. https://uswest.ensembl.org/info/docs/tools/vep/index.html |
参考文献
- Fernandes, A. G., Pollock, B. D., Rabito, F. A. Retinoblastoma in the United States: A 40-year incidence and survival analysis. Journal of Pediatric Ophthalmology & Strabismus. 55 (3), 182-188 (2018).
- Linn, A. M. Intraocular retinoblastoma: the case for a new group classification. Ophthalmology Clinics of North America. 18 (1), 41-53 (2005).
- Shields, J. A., Shields, C. L., Ehya, H., Eagle, R. C., De Potter, P. Fine-needle aspiration biopsy of suspected intraocular tumors. The 1992 Urwick lecture. Ophthalmology. 100 (11), 1677-1684 (1993).
- Karcioglu, Z. A., Gordon, R. A., Karcioglu, G. L. Tumor seeding in ocular fine needle aspiration biopsy. Ophthalmology. 92 (12), 1763-1767 (1985).
- Karcioglu, Z. A. Fine needle aspiration biopsy (FNAB) for retinoblastoma. Retina. 22 (6), 707-710 (2002).
- Eide, N., Syrdalen, P., Walaas, L., Hagmar, B. Fine needle aspiration biopsy in selecting treatment for inconclusive intraocular disease. Acta Ophthalmologica Scandinavica. 77 (4), 448-452 (1999).
- Eide, N., Walaas, L. Fine-needle aspiration biopsy and other biopsies in suspected intraocular malignant disease: a review. Acta Ophthalmologica. 87 (6), 588-601 (2009).
- Eriksson, O., Hagmar, B., Ryd, W. Effects of fine-needle aspiration and other biopsy procedures on tumor dissemination in mice. Cancer. 54 (1), 73-78 (1984).
- Ali, M. J., Honavar, S. G., Vemuganti, G. K., Singh, A. D. Fine needle aspiration biopsy of retinal tumors. Monographs in Clinical Cytology. 21, 72-81 (2012).
- Di Nicolantonio, F. a. l., et al. The chemosensitivity profile of retinoblastoma. Recent Results in Cancer Research. 161, 73-80 (2003).
- Francis, J. H., et al. Efficacy and toxicity of second-course ophthalmic artery chemosurgery for retinoblastoma. Ophthalmology. 122 (5), 1016-1022 (2015).
- Francis, J. H. Efficacy and toxicity of intravitreous chemotherapy for retinoblastoma: Four-year experience. Ophthalmology. 124 (4), 488-495 (2017).
- Francis, J. H., et al. Current treatment of bilateral retinoblastoma: The impact of intraarterial and intravitreous chemotherapy. Neoplasia. 20 (8), 757-763 (2018).
- Rushlow, D. E., et al. Characterisation of retinoblastomas without RB1 mutations: genomic, gene expression, and clinical studies. The Lancet Oncology. 14 (4), 327-334 (2013).
- Afshar, A. R., et al. Next-generation sequencing of retinoblastoma identifies pathogenic alterations beyond RB1 inactivation that correlate with aggressive histopathologic features. Ophthalmology. 127 (6), 804-813 (2020).
- Kothari, P. Cell-free DNA profiling in retinoblastoma patients with advanced intraocular disease: An MSKCC experience. Cancer Medicine. 9 (17), 6093-6101 (2020).
- Berry, J. L., et al. Potential of aqueous humor as a surrogate tumor biopsy for retinoblastoma. JAMA Ophthalmology. 135 (11), 1221-1230 (2017).
- Berry, J. L., et al. Genomic cfDNA analysis of aqueous humor in retinoblastoma predicts eye salvage: the surrogate tumor biopsy for retinoblastoma. Molecular Cancer Research. 16 (11), 1701-1712 (2018).
- Berry, J. L., et al. Aqueous humor is superior to blood as a liquid biopsy for retinoblastoma. Ophthalmology. 127 (4), 552-554 (2020).
- Xu, L., et al. Chromosome 6p amplification in aqueous humor cell-free DNA Is a prognostic biomarker for retinoblastoma ocular survival. Molecular Cancer Research. 18 (8), 1166-1175 (2020).
- Xu, L., et al. Establishing the clinical utility of ctDNA analysis for diagnosis, prognosis, and treatment monitoring of retinoblastoma: The aqueous humor liquid biopsy. Cancers. 13 (6), (2021).
- Xu, L. Simultaneous identification of clinically relevant RB1 mutations and copy number alterations in aqueous humor of retinoblastoma eyes. Ophthalmic Genetics. 41 (6), 526-532 (2020).
- Polski, A., et al. Longitudinal aqueous humor sampling reflects treatment response in retinoblastoma patients. Investigative Ophthalmology & Visual Science. 61 (7), 1394 (2020).
- . Genome Browser Available from: https://genome.ucsc.edu/cgi-bin/hgTracks?db=hg19&lastVirtModeType=default&lastVirtModeExtraState=&virtModeType=default&3A15578261%_2D15621068&hgsid=1135765621_qIuA0U (2021)
- Navarro Gonzalez, J., et al. The UCSC genome browser database: 2021 update. Nucleic Acids Research. 49 (1), 1046-1057 (2021).
- Li, H. A statistical framework for SNP calling, mutation discovery, association mapping and population genetical parameter estimation from sequencing data. Bioinformatics. 27 (21), 2987-2993 (2011).
- Baslan, T., et al. Genome-wide copy number analysis of single cells. Nature Protocols. 7 (6), 1024-1041 (2012).
- Huber, W., et al. Orchestrating high-throughput genomic analysis with Bioconductor. Nature Methods. 12 (2), 115-121 (2015).
- Baslan, T., et al. Erratum: Genome-wide copy number analysis of single cells. Nature Protocols. 11 (3), 1024-1041 (2016).
- Didion, J. P., Martin, M., Collins, F. S. Atropos: specific, sensitive, and speedy trimming of sequencing reads. PeerJ. 5, 3720 (2017).
- Li, H. Aligning sequence reads, clone sequences and assembly contigs with BWA-MEM. arXiv. , (2013).
- Garrison, E., Marth, G. Haplotype-based variant detection from short-read sequencing. arXiv. , (2012).
- McLaren, W., et al. The ensembl variant effect predictor. Genome Biology. 17 (1), 122 (2016).
- Lai, Z., et al. VarDict: a novel and versatile variant caller for next-generation sequencing in cancer research. Nucleic Acids Research. 44 (11), 108 (2016).
- . GitHub Available from: https://github.com/broadinstitute/ichorCNA (2017)
- Adalsteinsson, V. A., et al. Scalable whole-exome sequencing of cell-free DNA reveals high concordance with metastatic tumors. Nature Communications. 8 (1), 1-13 (2017).
- . GitHub Available from: https://github.com/broadinstitute/ichorCNA/wiki/Interpreting-ichorCNA-results (2017)
- Berry, J. L., et al. Long-term outcomes of Group D retinoblastoma eyes during the intravitreal melphalan era. Pediatric Blood & Cancer. 64 (12), (2017).
- Berry, J. L., et al. Long-term outcomes of Group D eyes in bilateral retinoblastoma patients treated with chemoreduction and low-dose IMRT salvage. Pediatric Blood & Cancer. 60 (4), 688-693 (2013).
- Francis, J. H., et al. Molecular changes in retinoblastoma beyond RB1: Findings from next-generation sequencing. Cancers (Basel). 13 (1), (2021).
- Schwermer, M., et al. Comprehensive characterization of RB1 mutant and MYCN amplified retinoblastoma cell lines. Experimental Cell Research. 375 (2), 92-99 (2019).
- Davies, H. R. Whole-genome sequencing of retinoblastoma reveals the diversity of rearrangements disrupting RB1 and uncovers a treatment-related mutational signature. Cancers. 13 (4), 754 (2021).
- Lillington, D. M., et al. Comparative genomic hybridization of 49 primary retinoblastoma tumors identifies chromosomal regions associated with histopathology, progression, and patient outcome. Genes, Chromosomes and Cancer. 36 (2), 121-128 (2003).
- Greger, V., et al. Frequency and parental origin of hypermethylated RB1 alleles in retinoblastoma. Human Genetics. 94 (5), 491-496 (1994).
- Raizis, A. M., et al. DNA hypermethylation/boundary control loss identified in retinoblastomas associated with genetic and epigenetic inactivation of the RB1 gene promoter. Epigenetics. 1-15, (2020).
- Munier, F. L., et al. Profiling safety of intravitreal injections for retinoblastoma using an anti-reflux procedure and sterilisation of the needle track. British Journal of Ophthalmology. 96 (8), 1084-1087 (2012).
- Chévez-Barrios, P., et al. Response of retinoblastoma with vitreous tumor seeding to adenovirus-mediated delivery of thymidine kinase followed by ganciclovir. Journal of Clinical Oncology. 23 (31), 7927-7935 (2005).
- Shao, W., Khin, S., Kopp, W. C. Characterization of effect of repeated freeze and thaw cycles on stability of genomic DNA using pulsed field gel electrophoresis. Biopreservation and Biobanking. 10 (1), 4-11 (2012).
- Choudhury, A. D., et al. Tumor fraction in cell-free DNA as a biomarker in prostate cancer. JCI Insight. 3 (21), (2018).
- Gerrish, A., et al. Non-invasive diagnosis of retinoblastoma using cell-free DNA from aqueous humour. British Journal of Ophthalmology. 103 (5), 721-724 (2019).
- Wong, E. Y., Xu, L., Shen, L. Inter-eye genomic heterogeneity in bilateral retinoblastoma via aqueous humor liquid biopsy. NPJ Precis Oncol. 5 (1), 73 (2021).
转载和许可
请求许可使用此 JoVE 文章的文本或图形
请求许可探索更多文章
This article has been published
Video Coming Soon
版权所属 © 2025 MyJoVE 公司版权所有,本公司不涉及任何医疗业务和医疗服务。