Method Article
Un método eficiente para la producción de adenovirus
En este artículo
Resumen
Aquí, presentamos un protocolo para la producción de adenovirus utilizando el sistema pAdEasy. La tecnología incluye la recombinación de los plásmidos pAdTrack y pAdEasy-1, el empaquetado y la amplificación del adenovirus, la purificación de las partículas adenovirales del lisato celular y el medio de cultivo, la titulación viral y las pruebas funcionales del adenovirus.
Resumen
La transducción adenoviral tiene la ventaja de una inducción fuerte y transitoria de la expresión del gen de interés en una amplia variedad de tipos y órganos celulares. Sin embargo, la tecnología adenoviral recombinante es laboriosa, lenta y costosa. Aquí, presentamos un protocolo mejorado utilizando el sistema pAdEasy para obtener partículas adenovirales purificadas que pueden inducir una fuerte expresión de proteína verde fluorescente (GFP) en células transducidas. Las ventajas de este método mejorado son una preparación más rápida y una disminución del costo de producción en comparación con el método original desarrollado por Bert Vogelstein. Los pasos principales de la tecnología adenoviral son: (1) la recombinación de pAdTrack-GFP con el plásmido pAdEasy-1 en bacterias BJ5183; (2) el empaquetado de las partículas adenoviral; (3) la amplificación del adenovirus en las células AD293; (4) la purificación de las partículas adenovirales del lysate de la célula y del medio de cultivo; y (5) la titulación viral y la prueba funcional del adenovirus. Las mejoras al método original consisten en (i) la recombinación en BJ5183-que contiene pAdEasy-1 por la transformación química de bacterias; (ii) la selección de clones recombinantes por PCR "negativa" y "positiva"; iii) la transfección de células AD293 utilizando el sistema de transfección K2 para el envasado adenoviral; iv) la precipitación con sulfato de amonio de las partículas virales liberadas por las células AD293 en el medio de cultivo celular; y (v) la purificación del virus por ultracentrifugación discontinua de gradiente de cloruro de cesio de un solo paso. Se obtuvo una fuerte expresión del gen de interés (en este caso, GFP) en diferentes tipos de células transducidas (como hepatocitos, células endoteliales) de diversas fuentes (humanas, bovinas, murinas). La transferencia génica mediada por adenovirales representa una de las principales herramientas para el desarrollo de terapias génicas modernas.
Introducción
Los adenovirus son virus no envulados que contienen una nucleocáppsida y un genoma de ADN lineal de doble cadena1,2,3. Los adenovirus pueden infectar una amplia gama de tipos de células y la infección no depende de la división celular activa del huésped. Después de la infección, el adenovirus introduce su ADN genómico en el núcleo de la célula huésped, donde permanece epicromoso y se transcribe junto con los genes del huésped. Así, se alcanza un riesgo potencial mínimo de mutagénesis de inserción o regulación de oncogenes4,5,6. El genoma adenoviral no se replica junto con el genoma del huésped y, por lo tanto, los genes adenovirales se diluyen en una población de células en división. Entre las ventajas de la transducción adenoviral, hay: (i) altos niveles de expresión de transgenes; (ii) reducción de riesgos relacionados con la integración del ADN viral en el genoma del huésped, debido a la expresión episomal; iii) transducción de una amplia variedad de tipos de células en división y no en división. La mayoría de los adenovirus utilizados en la investigación biomédica no son replicativos, careciendo de la región E17,8,9. Para su producción, se requiere una línea celular que suministre la secuencia E1 (como HEK293). Además, se eliminó una región no esencial para el ciclo de vida viral (E3) para permitir la inserción de un transgén en el genoma viral; otras regiones (E2 y E4) fueron suprimidas más a fondo en algunos adenovirus, pero en estos casos, una producción disminuida de adenoviral y la expresión baja del transgene fueron divulgadas7.
Aquí, presentamos un protocolo mejorado para construir, empaquetar y purificar los adenovirus utilizando el sistema AdEasy. Estas mejoras permitieron el empaquetamiento del adenovirus de una manera más rápida y económica en comparación con el método original desarrollado por Bert Vogelstein2,10,debido a las siguientes ventajas: (i) la recombinación en BJ5183-que contiene pAdEasy-1 por transformación química de bacterias; ii) la selección de los clones recombinantes por PCR; iii) la transfección de células AD293 utilizando el sistema de transfección K2 para el envasado adenoviral; (iv) la precipitación de partículas adenovirales del medio de cultivo después del empaquetado y de la amplificación virales; (v) la purificación adenoviral usando la ultracentrifugación del gradiente del cloruro de cesio de un paso (CsCl).
El protocolo para la producción de adenovirus utilizando el sistema AdEasy (Figura 1) comprende los siguientes pasos:
(1) Recombinación de pAdTrack-GFP con pAdEasy-1 en bacterias BJ5183
(2) Empaquetado de las partículas adenovirales
(3) Amplificación del adenovirus
(4) Purificación de las partículas adenovirales del lisato celular y del medio de cultivo
(5) Titulación de adenovirus.
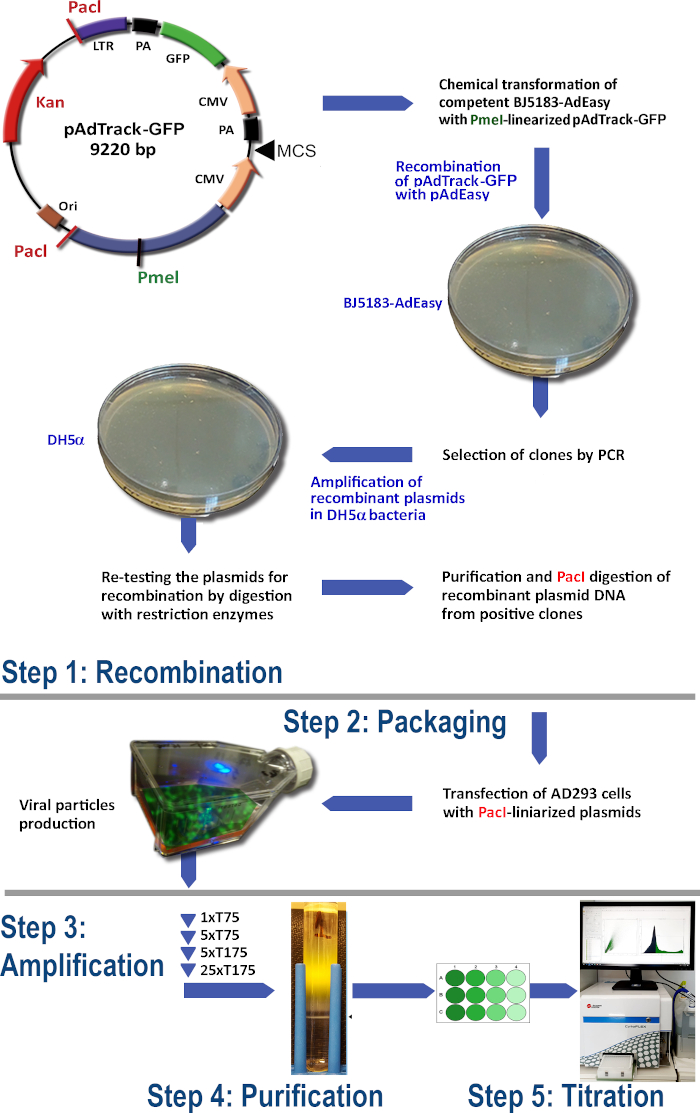
Figura 1: La tecnología de producción de adenovirus. Los principales pasos de la tecnología adenoviral son: (1) La recombinación del pAdTrack-GFP con el plásmido pAdEasy-1 en bacterias BJ5183. Los plásmidos recombinados seleccionados se amplifican en bacterias DH5α y luego se purifican; (2) El empaquetado de las partículas adenovirales en las células AD293, que están produciendo las proteínas adeno-E1; (3) La amplificación del adenovirus en las células AD293; (4) La purificación de las partículas adenovirales del lisiado celular y del medio de cultivo por ultracentrifugación en un gradiente de densidad CsCl; (5) La titulación del adenovirus y la prueba funcional. Haga clic aquí para ver una versión más amplia de esta figura.
En este protocolo, ejemplificamos la tecnología para la producción del adenovirus, que puede inducir la expresión de GFP en las células huésped. GFP ya está insertado en la columna vertebral del vector lanzadera pAdTrack-CMV (Addgene #16405), bajo un segundo promotor CMV y se utiliza como gen reportero (Figura 1). Por esta razón, aquí designamos el vector pAdTrack-CMV como pAdTrack-GFP y evaluamos la expresión de GFP con fines demostrativos. Además de la expresión de GFP, el sistema se puede utilizar para sobreexpresar un gen de interés, que puede ser clonado en los múltiples sitios de clonación del pAdTrack-CMV. Un gen o un minigén clonado en el pAdTrack-CMV suele ser más eficiente para la inducción de la expresión en comparación con el ADNc11. Los datos mostraron una fuerte expresión de GFP en células transducidas (como hepatocitos, células endoteliales) de diversas fuentes (humanas, bovinas, murinas). La transferencia génica mediada por adenovirales representa una de las principales herramientas para el desarrollo de terapias génicas modernas.
Protocolo
Nota de seguridad: En general, los adenovirus se clasifican como organismos de nivel 2 de bioseguridad y, por lo tanto, todas las manipulaciones deben ser realizadas en un gabinete de bioseguridad de Clase II por una persona capacitada, usando equipo de protección de riesgo biológico (incluidos guantes, máscara facial para aerosoles biológicos, bata de laboratorio, etc.). Todos los materiales sólidos contaminados con el adenovirus deben desinfectarse con una solución de lejía al 10% durante 30 min y en autoclave durante 30 min a 121 °C y 1 bar. Dependiendo del gen insertado, el adenovirus creado puede tener un potencial peligroso y puede clasificarse en otros niveles de bioseguridad.
1. Preparación experimental
- Utilice una campana de cultivo celular separada para las manipulaciones adenovirales y una incubadora separada para cada tipo de adenovirus. Utilizar frascos T con tapas de filtro para el envasado y amplificación viral; evitar tanto como sea posible experimentos de transducción en placas de Petri ventiladas.
- Vacíe la campana de cultivo celular después de cada uso y expóndala a los rayos UV durante 15 min.
- Autoclave periódicamente la ayuda de pipeta, pipetas y otros utensilios. Si es posible, cultíe en un laboratorio/capucha de cultivo celular separado las células para el envasado adenoviral (células AD293) y las células que se utilizarán en experimentos de transducción. Los lotes de diferentes adenovirus amplificados en el mismo período deben comprobarse para detectar la contaminación cruzada por PCR.
- Prepare las siguientes soluciones.
- Preparar sob (caldo super óptimo) medio: 20 g de triptona, 5 g de extracto de levadura, 0,5 g de NaCl (10 mM de concentración final), 2,5 mL de 1 M KCl (2,5 mM de concentración final), amdH2O a 1 L. Después del autoclave a 121 °C, añadir las siguientes soluciones estériles: 5 mL de 1 M MgCl2 y 5 mL de 1 M MgSO4.
- Preparar SOC (Caldo Super Óptimo con represión catabolita) medio: en 1 L SOB estéril añadir las siguientes soluciones estériles: 20 mL de glucosa 1 M, 5 mL de 1 M MgCl2, y 5 mL de 1 M MgSO4.
- Preparar solución de precipitación: disolver 29,5 g de acetato de potasio en 60 mL deH2O, añadir 11,5 mL de ácido acético, yH2O a 100 mL.
- Preparar tampón de resuspensión: 95 mL de glucosa al 20%, 5 mL de 1 M Tris-Cl pH 8, 4 mL de EDTA 0,5 M pH 8, y añadirH2O a 200 mL.
- Preparar solución de lisis: 4,8 mL de NaOH de 8,3 M, 10 mL de SDS al 20%, yH2O a 200 mL.
2. Recombinación del vector viral pAdTrack-GFP con plásmido pAdEasy-1 en bacterias BJ5183
- Linearización de pAdTrack-GFP y purificación del plásmido linealizado.
- Prepare la siguiente mezcla de digestión sobre hielo:
10 μg de pAdTrack-GFP
5 μL de tampón incoloro 10x
2 μL de Pme I
H2O de a un volumen final de 50 μL. - Incubar a 37 °C durante 3 horas en un baño de agua.
- Inactivar a 65 °C durante 20 min.
- Compruebe la eficiencia de la digestión de pAdTrack-GFP con Pme I: ejecute 1 μg del plásmido digerido en paralelo con 1 μg de plásmido no digerido en un gel de agarosa al 0,8%.
- Prepare la siguiente mezcla de digestión sobre hielo:
- Aislamiento y purificación del ADN
NOTA: Los pasos 1-6 deben realizarse en una campana extractora de humos.- Añadir un volumen igual de fenol/cloroformo/alcohol isoamilo (25:24:1) sobre la mezcla de digestión e invertir el tubo hasta que la mezcla sea homogénea.
- Centrífuga durante 3 min a 16.200 x g,y luego transferir la fase acuosa superior a un tubo de recolección.
- Añadir un volumen igual de fenol/cloroformo/alcohol isoamilo (25:24:1) sobre la fase orgánica inferior y el vórtice.
- Centrífuga durante 3 min a 16.200 x g,y luego transferir la fase superior al mismo tubo de recolección.
- Añadir un volumen igual de cloroformo sobre la fase acuosa cosechada en el tubo de recolección y el vórtice.
- Centrífuga durante 3 min a 16.200 x g,y luego transferir la fase acuosa superior a un nuevo tubo de recolección.
- Añadir un volumen de 1/10 de acetato de sodio 3 M, y 2 volúmenes de frío 100% etanol y vórtice.
- Incubar durante 1 hora a -70 °C o durante la noche a -20 °C.
- Descongelar la muestra sobre hielo y centrifugarla durante 10 min a 16.200 x g y 4 °C.
- Retiramos el sobrenadante y añadimos 750 μL de etanol al 75%.
- Centrífuga durante 3 min a 16.200 x g y 4 °C y retira el sobrenadante.
- Gire brevemente el tubo para eliminar todo el sobrenadante y seque el pellet en la capucha. No seque el pellet de ADN durante mucho tiempo porque es difícil de disolver.
- Disolver el pellet en 15 μL deH2O.
- Mida la concentración de ADN usando un espectrofotómetro (por ejemplo, Nanodrop).
- Transformación de bacterias AdEasier-1 con pAdTrack-GFP
NOTA: En este paso, tiene lugar la recombinación de pAdTrack-GFP con el plásmido pAdEasy-1.- Prepare las bacterias químicas competentes AdEasier-1 (BJ5183-containing pAdEasy-1, Addgene #16399), utilizando un kit de transformación comercial, siguiendo las instrucciones de los fabricantes. Mantenga las alícuotas de 100 μL de bacterias competentes a -80 °C.
- Descongelar una alícuota de bacterias AdEasier-1 competentes en el hielo y añadir 1 μg de Pme I purificado -digerido pAdTrack-GFP. Mezcle suavemente moviendo el tubo (no pipetee la mezcla). Incubar durante 10 min sobre hielo.
- Añadir 900 μL de medio SOC e incubar durante 1 hora a 37 °C con agitación.
- Microfuge durante 5 min a 600 x g.
- Retire 900 μL del sobrenadante, mezcle el pellet y el sobrenadante, y sisembra las bacterias transformadas en placas de agar LB con kanamicina.
- Incubar durante ~16 horas a 37 °C (no exceda las 18 horas).
- Selección de los posibles clones positivos por PCR
- Divida los palillos de dientes en dos mitades y esterilice los palillos de medio dientes en autoclave.
- Recoger colonias pequeñas y translúcidas con palillos de medio diente estériles.
- Brevemente, gire el medio palillo de dientes con bacterias en agua de 10 μL (en un tubo de PCR) y luego coloque el medio palillo de dientes en un tubo eppendorf de 1,5 mL que contenga un medio SOC de 100 μL con kanamicina. Incubar durante 4-6 horas a 37 °C, mientras se prueban los clones por PCR "negativa" y "positiva".
- Incubar los tubos de PCR que contengan 10 μL de agua con bacterias durante 5 min a 95 °C para obtener la muestra bacteriana y ejecutar en paralelo la PCR "negativa" y "positiva".
- PCR "negativa" - para probar la integridad de pAdTrack-GFP: Prepare la siguiente mezcla de PCR para la PCR negativa en el hielo.
5 μL de la muestra bacteriana
0,1 μL de cebador Forward (4631 F: 5'-CAGTAGTCGGTGCTCGTCCAG)
0,1 μL de cebador Inverso (5616 R: 5'-TATGGGGGCTGTAATGTTGTCTC)
0,1 μL de dNTP 10 mM
3 μL de 5x Buffer
1,5 μL de MgCl2 25mM
0,1 μL de gotaq polimerasa
H2O a un volumen final de 15 μL
NOTA: Se debe incluir el control positivo en el que la plantilla de ADN es el vector pAdTrack-GFP. - PCR "positiva" - para probar la presencia del gen de interés. Utilice cebadores específicos para el gen insertado y prepare la mezcla como en el paso anterior. Los cebadores utilizados para la GFP fueron los siguientes:
F: 5'-CAAGGACGACGGCAACTACA
R: 5'-ATGGGGGTGTTCTGCTGGTA - Ejecutar en paralelo la PCR "negativa" y la "positiva". El Programa de PCR es: 5 min, 95 °C; 40 ciclos de los siguientes pasos: 30 seg, 95 °C; 30 seg, 68 °C; 1 minuto, °C 72; elongación final: 10 min, 72 °C.
NOTA: Adaptar la temperatura de recocido para la amplificación del gen de interés. - Evaluar los productos de PCR en un gel de agarosa al 1% y hacer la selección de los clones.
- Considere para su posterior procesamiento los clones que no dan productos de PCR para la "PCR negativa" y el producto de PCR específico después de la "PCR positiva".
- Cultivar los cultivos bacterianos de clones recombinantes seleccionados
- Diluir los cultivos de los clones presuntamente positivos (resultado en el paso 2.4.3.) en 4 mL de medio SOC con kanamicina, e incubarlos durante la noche a 37°C con agitación.
- Aislamiento del ADN plásmido de las bacterias AdEasier-1 (Miniprep usando lisis alcalina)
- Transferir 1,5 mL de cultivo bacteriano en tubos de microcentrífuga, centrífuga durante 1 min a 16.200 x g,y retirar el sobrenadante.
- Transfiera otro cultivo bacteriano de 1,5 mL en el mismo tubo, repita la centrifugación y retire el sobrenadante.
- Añadir 200 μL de tampón de resuspensión (50 mM de glucosa, 10 mM de EDTA, 25 mM de Tris-HCl pH 8).
- Añadir 200 μL de solución de lisis (0,2 N NaOH, 1% SDS), mezclar suavemente invirtiendo el tubo.
- Añadir 200 μL de solución de precipitación (60 mL de acetato de potasio 5 M, 11,5 mL de ácido acético glacial, añadirH2O hasta 100 mL), y mezclar suavemente invirtiendo el tubo.
- Centrífuga durante 3 min a 16.200 x g.
- Transferir el sobrenadante en un nuevo tubo de microcentrífuga, añadir 500 μL de isopropanol, mezclar e incubar durante 20 min sobre hielo.
- Centrífuga durante 15 min a 16.200 x g y añadir 500 μL de etanol al 75%.
- Centrífuga durante 10 min a 16.200 x g y retira el sobrenadante.
- Centrífuga durante 3 min a 16.200 x g,retiramos el sobrenadante y añadimos 15 μL deH2O.
- Amplificación, aislamiento y re-prueba de los plásmidos recombinados
- Transformación de las bacterias DH5α con el ADN aislado de las células AdEasier-1.
- Prepare las bacterias competentes DH5α utilizando el kit de transformación comercial, siguiendo las instrucciones de los fabricantes.
- Descongelar 100 μL de bacterias competentes DH5α en el hielo, añadir el ADN recombinante, e incubar 10 min en el hielo. Luego sembra las bacterias en placas de agar LB con kanamicina.
- Incubar a 37 °C durante la noche.
- Recoger varias colonias y cultivar cada una en 2 mL de LB medio con kanamicina, a 37 °C, durante la noche, con agitación.
- Aislar el ADN (Miniprep utilizando lisis alcalina) y resuspend el ADN obtenido en 25 μL H2O.
- Confirmar los clones positivos por digestión enzimática.
- Prepare la siguiente mezcla sobre hielo:
5 μL de ADN recombinante
1,5 μL de búfer incoloro de 10x
0,5 μL de Hind III o Pst I
H2O a un volumen final de 15 μL - Incubar a 37 °C durante 30 min.
NOTA: Como control, digiera también los plásmidos pAdTrack-GFP y pAdEasy-1. - En cada muestra agregue 3 μL de búfer de carga Sx6 con RNasa A (si la ARNasa A no está presente en los búferes de miniprep).
- Ejecute los fragmentos de ADN digeridos en electroforesis en gel de agarosa al 1%.
NOTA: El patrón de digestión de un clon positivo incluye la mayoría de los fragmentos del plásmido pAdEasy digerido, revelando la recombinación pAdEasy con el vector pAdTrack. El gen de interés debe ser evidenciado por la digestión con las enzimas de restricción utilizadas para la clonación.
- Prepare la siguiente mezcla sobre hielo:
- Preparación de ADN plásmido (grado de transfección) para el envasado de adenovirus.
- Cultivar un cultivo de 200 mL de bacterias a partir de un clon positivo para aislar el ADN del plásmido.
- Aísle el ADN del plásmido usando un kit comercial para el ADN del plásmido Midiprep (por ejemplo, Qiagen Plasmid Midi Kit) siguiendo las instrucciones del fabricante.
- Transformación de las bacterias DH5α con el ADN aislado de las células AdEasier-1.
3. Empaquetar las partículas adenovirales
- Día 1. Semilla de las células AD293
- Lave las células AD293 con PBS e incubarlas con tripsina al 0,125% durante 2-5 min a 37 °C.
- Recoger las células en medio frío con suero.
- Centrífuga durante 5 min a 400 x g a 4 °C.
- Resuspend las células en medio con suero y sembrar las células a una densidad de ~2 x 106/T25 matraz. Preferiblemente, utilice un matraz con un filtro.
- Día 1. Digerir el ADN recombinante con Pac I
- Prepare la siguiente combinación:
6 μL de ADN recombinante (1 μg/μL)
2 μL de Pac I
2.5 μL de buffer incoloro 10x
H2O a un volumen final de 25 μL - Incubar durante 3 h (o durante la noche) a 37 °C, y luego inactivar la enzima a 65 °C durante 20 min.
- Precipitación de ADN con etanol: añadir 2,5 μL de (1/10 v/v) acetato de sodio 3 M y 2-3 volúmenes de etanol al 100%. Incubación durante 30 min a -70 °C o durante la noche a -20 °C.
- Centrífuga a 16.200 x g durante 30 min a 4 °C y resuspens el pellet en agua estéril.
- Prepare la siguiente combinación:
- Día 2: Transfección de células AD293 usando reactivo K2
- Añadir 40 μL de K2 Multiplicador sobre las células, dos horas antes de la transfección.
- Prepare soluciones A y B:
Solución A: Añadir 6 μg de PAC l-linearizado de ADN en 260 μL de Opti-MEM.
Solución B: Añadir 21,6 μL de reactivo K2 en 248,4 μL de Opti-MEM. - Añadir la solución A sobre la solución B y mezclar suavemente pipeteando.
- Incubar la mezcla durante 20 min a temperatura ambiente. Agregue la mezcla A y B gota a gota a las celdas.
- Día 3-11: Monitoree la expresión de GFP por microscopia de fluorescencia
NOTA: Las células deben aparecer verdes en microscopía de fluorescencia y deben desprenderse gradualmente. - Día 11: Cosechar las partículas adenovirales F1
- Recoja las células separadas y el medio en un tubo de 50 mL, raspe las células adherentes y agréguelas en el mismo tubo.
- Centrífuga durante 5 min a 400 x g, recoger el sobrenadante en un nuevo tubo y resuspend el pellet celular en 0,5 mL de PBS.
- Interrupción celular
- Transfiera la suspensión celular en un tubo de microcentrífuga.
- Realizar tres ciclos de congelación/descongelación (congelar en nitrógeno líquido o a -80 °C /descongelar a 37 °C durante un máximo de 7 min).
- Pase las células rotas a través de una aguja de jeringa de 23 G tres veces.
- Retire los restos de la célula por centrifugación a 9.600 x g durante 12 min.
- Transfiera el sobrenadante al tubo de 50 mL con el medio recogido.
4. Amplificación del adenovirus
NOTA: Si las células AD293 no alcanzaron la confluencia necesaria, las alícuotas de las cepas adenovirales (lisiado obtenido de las células productoras de virus) que se utilizarán para la infección pueden almacenarse a -80 °C.
- Prepare las partículas adenovirales F2.
- Sembrar las células AD293 en un matraz T75 (5 x 106 células/matraz).
- Infecte ~90% de células AD293 confluentes utilizando las partículas adenovirales F1: agregue el homogeneizado celular y el medio del matraz T25 sobre las células cultivadas en el matraz T75.
- Monitoree la expresión de GFP por microscopía de fluorescencia.
- Coseche las células productoras de virus cuando ~ 90% de los AD293 transducidos se desprenda (~ el5º día después de la transducción). Mantenga el medio de cultivo celular a 4 °C.
- Interrumpa las células (semejantemente con ésos para F1) en 1 mL PBS.
- Prepare las partículas adenovirales F3.
- Infecte ~90% de células AD293 confluentes sembradas en matraz T175 con partículas adenovirales F2 y el medio de cultivo celular de las partículas adenovirales F2.
- Cosechar las células (~ 5 días después de la transducción).
- Interrumpa las células (semejantemente con ésos para F1) en 2 mL de PBS.
- Prepare las partículas adenovirales F4.
- Infecte 5 frascos T175 que contienen ~90% de células AD293 confluentes con partículas adenovirales F3 y medio de cultivo celular.
- Cosechar las células (~ 5 días después de la transducción).
- Interrumpa las células (semejantemente con ésos para F1) en 3 mL de PBS.
- Prepare las partículas adenovirales F5.
- Infecte 25 frascos T175 que contienen ~90% de células AD293 confluentes con cepas adenovirales F4 y medio de cultivo celular.
5. Purificación del adenovirus a partir de lisato celular y medio de cultivo
- Cosecha de las células productoras de virus y el medio de cultivo.
- Cosechar las células AD293 de F5 después de 5 días desde la transducción.
- Guarde el medio en una botella estéril para la precipitación de las partículas adenovirales.
NOTA: Mantenga el medio en el refrigerador hasta la purificación del adenovirus. - Centrifugar las células a 400 x g, durante 5 min, a 4 oC.
- Resuspend el pellet final en 5 mL de 10 mM Tris HCl, pH 8 con 2 mM MgCl2.
- Alícuota la suspensión en tubos de 1,5 mL.
- Interrumpir las células (de manera similar con las de F1): tres ciclos de congelación /descongelación.
NOTA: Si la ultracentrifugación no se puede realizar inmediatamente, mantenga las muestras a -80 °C. - Pase la suspensión celular a través de una aguja de jeringa 23G por tres veces.
- Centrifugar el homogeneato a 9 600 x g, durante 12 min.
- Guarde el sobrenadante para la purificación de adenovirus por ultracentrifugación de gradiente CsCl.
- Precipitación del adenovirus liberado en el medio de cultivo.
- Traiga la botella con el medio de cultivo celular guardado a temperatura ambiente.
- Añadir 121 g de sulfato de amonio a cada 500 mL de medio de cultivo celular (la saturación de la solución debe estar entre 40 - 42%).
- Mezcle cuidadosamente hasta que el sulfato de amonio se disuelva por completo.
- Incubar durante un mínimo de 2,5 horas a temperatura ambiente.
- Centrífuga a 1600 x g, durante 15 min, a 22 oC y guardar el pellet.
- Resuspend el pellet en 4 mL de 10mM Tris HCl pH 8 con 2mM MgCl2; esta suspensión debe purificarse inmediatamente mediante ultracentrifugación de gradiente CsCl.
NOTA: Si la etapa de purificación no se puede realizar posteriormente, dializar durante la noche el pellet resuspended contra 10mM Tris HCl, pH 8 con 2mM MgCl2.
- Purificación del adenovirus por ultracentrifugación.
- Prepare un gradiente discontinuo de CsCl en tubos de polipropileno para el rotor SW41Ti. Añadir 3 mL de 765 mg/mL de CsCl (alta densidad: 1,4 g/L) en la parte inferior del tubo. Añadir lentamente 3 mL de 288,5 mg/mL de CsCl (baja densidad: 1,2 g/L) sobre la primera capa de CsCl.
- Superponga suavemente 3 - 4 mL de suspensión de partículas adenovirales liberadas de las células o precipitadas desde el medio de cultivo celular (como se describió anteriormente) en la parte superior del gradiente.
- Llene los tubos con aceite mineral y coloque los tubos en los cubos SW41Ti fríos.
- Equilibre los tubos. Asegúrese de que los tubos de polipropileno llenos se cargan simétricamente en el rotor. Coloque el rotor en la ultracentrífuga.
- Centrífuga a 210.000 x g y 4 °C, durante 18 horas, sin freno.
- Coloque los tubos de ultracentrífuga en un soporte con un papel negro detrás para obtener las bandas.
- Deseche la fase superior clara, los desechos de la célula y la banda superior en un contenedor de residuos con la solución de blanqueo.
- Coseche la banda más baja que contiene el adenovirus completo (~ 700 μL - 1 mL) en un tubo estéril de 1,5 mL y manténgalo en hielo.
- Pre-húmedo un casete de diálisis en tampón de diálisis (10 mM tampón Tris-Cl pH 8, 2 mM MgCl2).
- Inyecte el adenovirus purificado en el cassette de diálisis usando una jeringa de 2 ml.
- Dializar durante la noche contra 10 mM Tris-Cl tampón pH 8, 2 mM MgCl2 (cambiar el tampón de diálisis 3 - 4 veces).
- Cosechar el caldo adenoviral del cassette de diálisis en alícuotas de 10 - 100 μL.
- Añadir sacarosa al 4% de concentración final a las alícuotas virales (para la crioprotección).
- Guarde las alícuotas a -80 °C.
6. Titulación de adenovirus
- Día 1: Chapado de las células
- Sembrar las células AD293 a una densidad de 2,5 × 105 células por pozo (en una placa de cultivo de 12 pozos) en un medio de crecimiento completo de 1 mL, como se muestra en la Figura 2. Asegúrese de que las células se esparcen uniformemente en cada pozo para una determinación precisa del título.
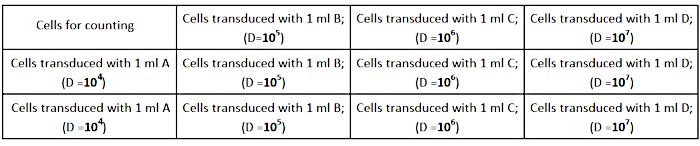
Figura 2: Diseño de la placa de titulación. Haga clic aquí para ver una versión más amplia de esta figura.
- Día 2: Transducción de células
- Desprenda las células de un pozo con tripsina y contadlas. Tenga en cuenta este número porque se utilizará para calcular el título viral.
- Realizar diluciones en serie (1/104; 1/105; 1/106; 1/107) del stock viral en 1 mL de medio de crecimiento completo de la siguiente manera:
- 1/103: dilución de la cepa de virus - añadir 2 μL de cepa viral a 1998 μL de medio completo.
- 1/104: Realizar una dilución 1:10 de 1/103 diluyendo 120 μL a 1080 μL de medio completo (A).
- 1/105: Realizar 1:10 dilución de B diluyendo 120 μL de A a 1080 μL de medio completo (B).
- 1/106: Realizar dilución 1:10 de C diluyendo 120 μL de B a 1080 μL de medio completo (C).
- 1/107: Realizar dilución 1:10 de D diluyendo 120 μL de C a 1080 μL de medio completo (D).
NOTA: Prepare 3 tubos de cada dilución (A, B, C, D) para realizar el experimento en triplicados. - Retire el medio de cultivo celular de los pocillos y agregue las diluciones preparadas del virus, como se muestra en la Figura 2.
- Día 3: Monitoreo de la expresión de GFP
- Revise los pozos para detectar la presencia de células verdes utilizando un microscopio de fluorescencia.
- Día 4: Análisis de citometría de flujo de células GFP-positivas
- Preparar y etiquetar doce tubos de 1,5 mL.
- Recoja el medio de cultivo celular (junto con las células separadas) en tubos de 1,5 mL y manténgalos en hielo.
- Añadir 200 μL de tripsina en cada pocillo.
- Incubar la placa durante 2 - 3 min a 37 °C en la incubadora de CO2.
- Coseche las células en los mismos tubos eppendorf con el medio de cultivo celular. Mantenga los tubos en hielo.
- Pellet de las células a 400 x g, durante 5 min, a 4 °C.
- Retire el sobrenadante; mantenga los tubos en hielo.
- Resuspend el pellet en 250 μL de PBS + 2% FBS; mantenga los tubos en hielo.
- Transfiera la suspensión celular en tubos o placas de citometría de flujo.
- Ejecute las muestras en un citómetro de flujo que registra la fluorescencia de las células que expresan GFP.
Cálculo del título: Las muestras con 5 - 20% de células GFP positivas de la población madre deben tenerse en cuenta para el cálculo del título viral utilizando la siguiente fórmula:
Título (TU/mL) = D x F/100 x C/V
D = factor de dilución
F = porcentaje de células positivas / 100
C = número de celdas / pozo
V = volumen de inóculo viral
7. Transducción adenoviral de células diana y pruebas de la expresión proteica inducida
- Día 1: Sembrando las células
- Sembrar las células diana asegurando que se esparcen uniformemente en los pozos.
- Día 2: Transducción de las células
- Separe las celdas de destino de un pozo y contadlas.
- Calcular el volumen apropiado de suspensión adenoviral necesaria para transducir las células con el número deseado de partículas infecciosas por célula.
- Agregue la cantidad correspondiente de suspensión viral a las células diana.
- Día 3: Eliminación de la suspensión viral y comprobación de la expresión de GFP
- Reemplace el medio de cultivo celular que contiene partículas adenovirales con un medio fresco.
- Compruebe la expresión de GFP en el microscopio de fluorescencia.
Resultados
Modificamos y mejoramos el vogelstein original' protocolo de s para lograr una producción más rápida y más eficiente del adenovirus. En primer lugar, revisamos la metodología para lograr una selección más fácil de recombinantes. Tras la recombinación, los clones bacterianos BJ5183 fueron probados por "PCR negativa" para evaluar la integridad de pAdTrack-GFP como indicador de la falta de recombinación(Figura 3A),o por "PCR positiva" para identificar el gen de interés, asimilado en nuestro caso a GFP(Figura 3B). Tanto en PCRs "negativos" como "positivos", utilizamos pAdTrack-GFP como plantilla de control, que dio una banda de 986 pb para la integridad de pAdTrack(Figura 3A,carril 1), y una banda de 264 pb para GFP(Figura 3B,carril 3). Las cartillas usadas para la "polimerización en cadena negativa" fueron diseñadas para amplificar un fragmento de 986 puntos de ebullición que contenía el sitio de PmeI en pAdTrack-GFP. Este fragmento de ADN se agranda drásticamente después de la recombinación y no se amplifica en los clones recombinantes positivos. Los clones negativos para recombinación, en los que pAdTrack-GFP permaneció intacto, se representan en la Figura 3A,carriles 3, 4 y 6. Los cebadores recocidos en las secuencias de ADN adyacentes al sitio de recombinación. Los clones recombinantes positivos potenciales(Figura 3A,carriles 2 y 5) expresaron GFP como se muestra en la Figura 3B,carril 1 y 2. El ADN plásmido de estos clones se aisló y se utilizó para la transformación DH5α para obtener una mayor cantidad de ADN. Estos plásmidos recombinantes preseleccionados amplificados en DH5α fueron probados por digestión enzimática. En la Figura 3C-E se ilustran los resultados de la digestión enzimática de un clon recombinante-positivo digerido con enzimas de restricción Hind III, PstI, BamHI(Figura 3C, D, E carril 2). Los patrones de digestión HindIII y PstI del clon recombinante fueron similares a los obtenidos para pAdEasy-1 ya que HindIII y PstI cortaron el plásmido pAdEasy-1 24 y 25 veces, respectivamente,(Figura 3C y D,carril 3); HindIII corta una vez y PstI corta cuatro veces el vector pAdTrack-GFP(Figura 3C y D,carril 1). BamHI cortó dos veces el vector pAdEasy-1(Figura 3C,carril 3), y una vez pAdTrack-GFP(Figura 3C,carril 1).
PacI cortó un fragmento de 4,5 kb del plásmido recombinante(Figura 3F,carril 2), un fragmento de 2863 pb de pAdTrack-GFP(Figura 3F,carril 1), y linealizó el vector pAdEasy-1(Figura 3F,carril 3). La escalera de ADN está representada en la Figura 3C-F,en los carriles 4. El plásmido recombinante fue digerido con Pac I para su uso posterior para la transfección AD293.
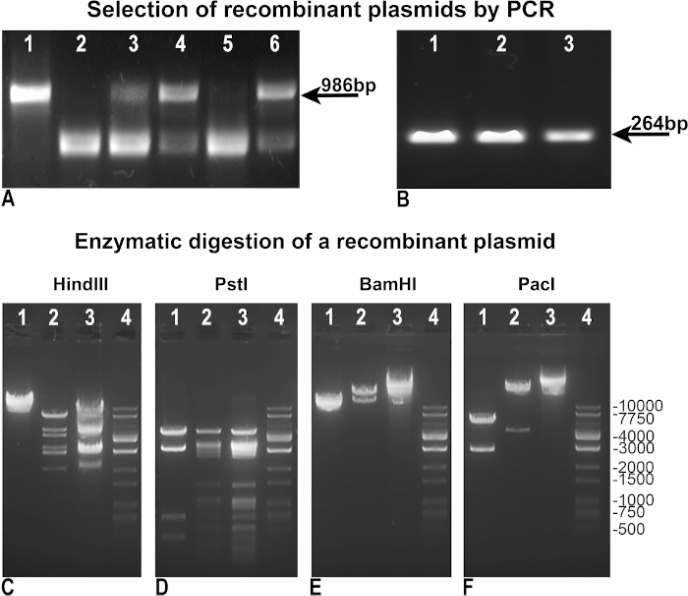
Figura 3: La recombinación de pAdTrack-GFP con el plásmido pAdEasy-1. Los plásmidos obtenidos después de la recombinación de pAdTrack-GFP y pAdEasy-1 fueron probados por PCR "negativa" para la integridad de pAdTrack-GFP (A). Las copias no recombinantes fueron evidenciadas por la presencia de una venda de 986 puntos de ebullición correspondiente a la secuencia amplificada del plásmido de pAdTrack-GFP (A, carriles 3, 4, y 6). Las copias potencialmente positivas para la recombinación (A, carriles 2 y 5) también fueron obtenidas. Cuando se utilizó el vector pAdTrack-GFP como plantilla, se obtuvo una banda de 986 pb para pAdTrack-GFP (A, carril 1). Las copias recombinantes potencialmente positivas fueron probadas para la expresión de GFP por la polimerización en cadena "positiva" (b); aparece una banda de 264 pb tanto para clones potencialmente recombinados (B, carril 1 y 2), como para el plásmido pAdTrack-GFP. La DNA a partir de una copia recombinante potencial fue probada con HindIII, PstI, BamHI, y la enzima de la restricción de PacI (C-F, carriles 2). En los controles, el vector pAdEasy-1 (C-F, carriles 3) y el plásmido pAdTrack-GFP (C-F, carriles 1) fueron digeridos con las mismas enzimas. La escalera de ADN está representada en el carril C-F 4. Haga clic aquí para ver una versión más amplia de esta figura.
El empaquetado y la amplificación adenoviral fueron realizados en las células AD293. Las partículas adenovirales (AdV-GFP) fueron purificadas del lysate de la célula AD293 así como del medio de cultivo celular, donde habían sido lanzadas por las células infectadas. Para concentrar el adenovirus encontrado en el medio de cultivo celular, las partículas se precipitaron con sulfato de amonio y luego se resuspendieron en 10 mM Tris HCl pH 8 con 2 mM MgCl2,el mismo tampón que el utilizado para la lisis celular. Posteriormente, las partículas adenovirales del lisiado celular y del medio de cultivo fueron purificadas por la ultracentrifugación discontinua del gradiente de CsCl. Después de la ultracentrifugación, se obtuvo una banda fuerte de AdV-GFP purificada, como se muestra en la Figura 4.

Figura 4: La purificación adenoviral por ultracentrifugación sobre un gradiente discontinuo de CsCl. El homogeneizado celular y el adenovirus precipitado desde el medio fueron sometidos a ultracentrifugación en un gradiente discontinuo formado por soluciones de CsCl de baja y alta densidad. Las vendas fuertes del adenovirus de GFP- fueron evidenciadas en ambos casos. Haga clic aquí para ver una versión más amplia de esta figura.
Para determinar el título viral expresado en unidades de transducción por un mL (TU/mL), las células AD293 fueron infectadas con diluciones seriales del AdV-GFP. Después de 48 horas, las células infectadas expresaron GFP, en una correlación inversa con el factor de dilución de la suspensión viral. Esto se observó mediante microscopía de fluorescencia y el porcentaje de células GFP positivas se determinó por citometría de flujo(Figura 5). Para calcular el título, se consideró la dilución viral que indujo 5 - 20% de las células GFP positivas (Figura 5C). Por lo general, obtenemos un título viral de ~ 1010 (TU / mL) para GFP-adenovirus.
A continuación, proporcionamos un ejemplo de un cálculo de título adenoviral para un lote adenoviral específico en el que 300000 células (C) fueron transducidas con 1 mL de solución adenoviral (V), a un factor de dilución de 106 (D), para el cual se obtuvieron células GFP positivas al 6%(F):
Título (TU/mL) = D x F/100 x C/V = 106 x 6/100 x 300000/1 = 1,8 x 1010 TU/mL
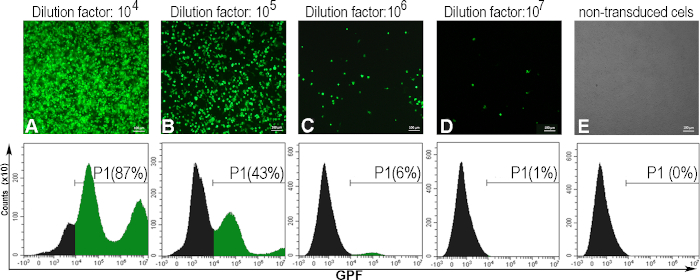
Figura 5: La evaluación del título adenoviral. Las células AD293 fueron infectadas con varias diluciones adenoviral. Cuarenta y ocho horas más adelante, las células fueron observadas por microscopia de la fluorescencia y analizadas por cytometry de flujo para determinar el porcentaje de células positivas de GFP inducidas por diversas diluciones adenovirales (A-D). Para establecer la puerta para la citometría de flujo, también se analizaron las células no transducidas (E). El título calculado para el factor de dilución 106,cuando el 6% de las células eran positivas para GFP fue de 1,8 x 1010 TU/mL. Para paneles A-E, barras: 100μm. Haga clic aquí para ver una versión más grande de esta figura.
Para probar el potencial de la transducción del adenovirus preparado, cuatro variedades de células fueron utilizadas: las células endoteliales humanas (EA.hy926), las células endoteliales aórticas bovinas (BAEC), los hepatocitos murine (Hepa 1-6), y las células stromal mesenquimales murine (MSC). Las células endoteliales (EA.hy926 y BAEC) fueron transducidas con 25 TU/cell, los hepatocitos fueron transducidos con 5 TU/cell y msc fueron transducidos con 250 TU/cell.
Aquí está un ejemplo de cómo el volumen de suspensión adenoviral necesitó infectar 3 x 106 células con 25 TU/cell, usando la suspensión adenoviral con 1,8 x 1010 TU/mL, era calculado.
Para 1 celda .............. 25 TU
3 x 106 celdas .............. x TU  x=75 x 106 TU
x=75 x 106 TU
Si el stock viral contiene
1.8 x 1010 TU .............. 1 mL
75 x 106 TU .............. y mL  y= 4,2 x 10-3 mL = 4,2μL de stock viral
y= 4,2 x 10-3 mL = 4,2μL de stock viral
Cuarenta y ocho horas después de la transducción, las células eran analizadas por microscopia de la fluorescencia. Como se muestra en la Figura 6,las células endoteliales humanas o bovinas se transdujeron con buena eficiencia (~ 50%) para 25 TU/célula (Figura 6 EA.hy926 y BAEC). Los hepatocitos murinos (Hepa 1-6) fueron transducidos eficientemente por el adenovirus en una cantidad baja de partículas del adenovirus (5 TU/cell), pero también son sensibles al adenovirus puesto que un porcentaje más alto de células muertas (células PI-positivas) fue registrado (el ~16%) en comparación con los otros tipos de celdas. Las células del estroma mesenquimal fueron las más difíciles de transducir(Figura 6),debido a la falta de receptores adenovirales específicos (datos no publicados).
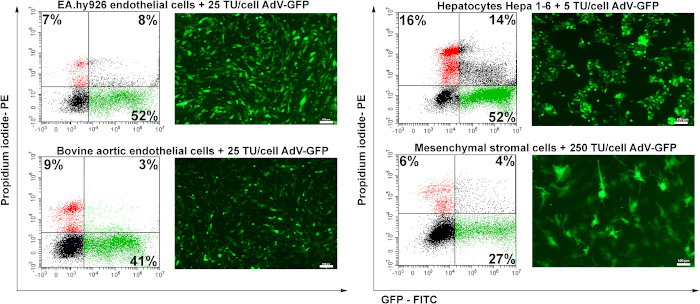
Figura 6: La infectividad del adenovirus y la inducción de la expresión de GFP en células transducidas. Las células endoteliales humanas (EA.hy926), las células endoteliales aórticas bovinas (BAEC), los hepatocitos murine (Hepa 1-6), y las células stromal mesenquimal murine (MSC) fueron transducidas con la cantidad indicada de adenovirus. GFP fue detectado por microscopia de la fluorescencia y el porcentaje de las células positivas de GFP era analizado por cytometry de flujo. Las células PI-positivas determinadas por citometría de flujo muestran la mortalidad celular determinada por transducción viral. Las células de EA.hy926, las células endoteliales aórticas bovinas, y las células de Hepa 1-6 fueron transducidas altamente por el adenovirus, la producción de la transducción que se extendía a partir del 41 - el 52%. Para el MSC, una cantidad más alta de virus (250 TU/cells) indujo el positivo del solamente 27% GFP de las células transducidas. Barras: 100μm. Haga clic aquí para ver una versión más grande de esta figura.
Discusión
Los adenovirus recombinantes son una herramienta versátil para la administración y expresióngénica 12,13,14. Para inducir una fuerte expresión de proteínas por transducción adenoviral, la secuencia de codificación del gen de interés se inserta en el genoma del adenovirus. El sistema adenoviral AdEasy, desarrollado en el laboratorio de Bert Vogelstein, comprende un plásmido troncal (pAdEasy-1) que contiene la mayor parte del genoma del serotipo 5 de adenovirus de tipo salvaje, y un vector lanzadera (pAdTrack), diseñado para la clonación degenes 2,10. La deleción de los genes adenovirales E1 (responsables del ensamblaje de partículas de virus infecciosos) y E3 (que codifican proteínas implicadas en la evasión de la inmunidad del huésped)creóun espacio en el genoma adenoviral, en el que se puede insertar un gen de interés de 6,5-7,5 kb2,3. Este tamaño es suficiente para muchos genes, especialmente para aquellos con intrones más cortos15,16,17. También hay investigadores que informan de la producción de adenovirus portadores del ADNc de untransgén 18,19,20. Sin embargo, obtuvimos un rendimiento más bajo de la expresión del transgén para los adenovirus cDNA-que llevan que para sus contrapartes que llevan un gene o un mini-gene (datos no mostrados).
Mejorar y adaptar los métodos anteriores2,10,14,18,21,la tecnología para la producción adenoviral requiere un menor tiempo, menor costo y menos esfuerzo. El ADN adenoviral de longitud completa se obtiene por recombinación entre el vector lanzadera y el plásmido pAdEasy-1 en la cepa de E. coli propensa a la recombinación homóloga, BJ5183. El protocolo implica la transformación química de las células AdEasier-1 (bacterias BJ5183 que contienen pAdEasy-1). Esta técnica no requiere un electroporador que puede no estar disponible en algunos laboratorios, es muy sencilla, aumenta el rendimiento de recombinación, y reduce el tiempo necesario para obtener células competentes y realizar la transformación. La preselección de clones recombinantes realizada por PCR acorta aún más el tiempo y facilita todo el procedimiento. Un procedimiento similar fue utilizado por Zhao y sus compañeros de trabajo22,sin embargo, en el protocolo, optimizamos las secuencias de los cebadores.
Para el empaquetado y la amplificación del GFP-adenovirus, una línea celular derivada HEK293 fue utilizada, a saber las células AD293, que son más adherentes a la placa de cultivo. Otras líneas celulares comúnmente utilizadas para la producción adenoviral son las siguientes: 911, 293FT, pTG6559 (derivado de A549), PER. C6 (derivado de HER), GH329 (derivado de HeLa), N52. E6, y HeLa-E123,24,25,26. En nuestras manos, no se obtuvo ninguna mejora en la producción adenoviral cuando se utilizaron 911 células (datos no mostrados). La transfección de células AD293 con el plásmido recombinante utilizando el reactivo K2 aumentó en gran medida la eficiencia de la etapa de envasado viral. Después de la producción de adenovirus, hasta ~ 70% del adenovirus todavía está dentro de las células y es liberado por tres ciclos de congelación y descongelación. Aumentar el número de ciclos no es adecuado porque destruye el adenovirus.
A lo largo del proceso de producción adenoviral de rutina, numerosas partículas virales se liberan en el medio de cultivo celular. El descarte de este medio de cultivo celular durante la cosecha de las células infectadas ad293 daría lugar a una pérdida viral importante. Optimizamos el protocolo descrito por Schagen y colaboradores para purificar las partículas adenovirales del medio de cultivo celular por precipitación con sulfato de amonio27. Este método tiene una mayor eficiencia en la recuperación de adenovirus del medio de cultivo celular en comparación con el método que utiliza polietilenglicol28. El adenovirus precipitado debe purificarse inmediatamente por ultracentrifugación o mantenerse en el refrigerador durante un par de días, pero solo después de la diálisis, para eliminar el exceso de sal. Mantener el precipitado más de unas pocas horas sin diálisis es perjudicial para el virus.
La purificación de las partículas adenovirales por ultracentrifugación realizada en un solo paso reduce la manipulación del stock adenoviral y facilita el procedimiento en comparación con los protocolos utilizando pasos sucesivos de ultracentrifugación14,29. La diálisis del adenovirus purificado es necesaria para eliminar el cloruro de cesio que puede afectar aún más a la transducción. En el protocolo, utilizamos el almacenador intermediario de Tris que contenía MgCl2 pero no sacarosa para la diálisis, puesto que requiere una cantidad enorme, injustificada de sacarosa que se necesite de otra manera como conservante para la congelación. Así, añadimos sacarosa más adelante, directamente en las acciones adenoviral preparadas para la congelación. Para evitar la congelación y descongelación frecuentes del adenovirus purificado, es aconsejable alícuota de las existencias adenovirales y almacenarlas a -80 °C. El título adenoviral fue evaluado por cytometry de flujo considerando el gene del reportero de GFP y el porcentaje de células transducidas para una dilución viral específica. Este método es más rápido en comparación con el clásico "ensayo de placa" y es más confiable en comparación con la evaluación de las proteínas de la cápside (por varios métodos como ELISA o citometría de flujo) que no revela la capacidad de infección de las partículas adenovirales. Sin embargo, la cuantificación basada en ELISA, Q-PCR o el ensayo de placa utilizando kits disponibles en el mercado son métodos alternativos, especialmente útiles para la titulación de adenovirus que no contienen un trazador fluorescente.
Teniendo en cuenta que los adenovirus pAdTrack se derivan del serotipo 5 de adenovirus humanos que es reconocido por Coxsackievirus y adenovirus receptores (CAR), hemos demostrado la capacidad del GFP-adenovirus para transducir células de origen humano (células endoteliales), pero también células de otros orígenes: bovinos (células endoteliales) y murinas (células del estroma mesenquimal y hepatocitos). Los datos mostraron que el GFP-adenovirus puede inducir un alto nivel de expresión de un transgén.
En conclusión, optimizamos esta laboriosa tecnología para reducir el tiempo, los costos y el esfuerzo necesario para obtener las partículas adenovirales. El adenovirus preparado es capaz de infectar varios tipos de células e inducir la expresión del gen de interés. Este protocolo se puede utilizar en una variedad de experimentos puesto que la transferencia adenoviral-mediada del gen representa una de las herramientas principales para desarrollar terapias génicas modernas.
ABREVIATURAS: AdV-GFP, partículas adenovirales; BAEC, células endoteliales aórticas bovinas; CsCl, cloruro de cesio; GFP, proteína verde fluorescente; MSC: células del estroma mesenquimal; TU, unidades transductoras.
Divulgaciones
Los autores no tienen nada que revelar.
Agradecimientos
Este trabajo fue apoyado por un proyecto cofinanciado por el Fondo Europeo de Desarrollo Regional a través del Programa Operativo de Competitividad 2014-2020 (POC-A.1-A.1.1.4-E-2015, ID: P_37_668; acrónimo DIABETER), una subvención del Ministerio rumano de Investigación e Innovación PCCDI- UEFISCDI, número de proyecto PN-III-P1-1.2-PCCDI-2017-0697 dentro de PNCD III y por la Academia Rumana. Los autores agradecen a Kyriakos Kypreos (Universidad de Patras, Grecia) por sus generosos y pertinentes consejos, a Ovidiu Croitoru (Universidad de Bellas Artes, Bucarest, Rumania) por el rodaje, la edición de películas y el diseño gráfico, y a Mihaela Bratu por la asistencia técnica.
Materiales
| Name | Company | Catalog Number | Comments |
| AD293 cells | Agilent Technologies | 240085 | |
| AdEasier-1 cells | Addgene | 16399 | |
| Agarose I (for electrophoresis) | Thermo Scientific | 17850 | |
| Ammonium sulfate | Sigma | A4418 | |
| Ampicillin sodium salt | Sigma | A0166 | |
| BamH I | Thermo Scientific | FD0054 | |
| Cell culture plates 100 mm | Eppendorf | 30702115 | |
| Cesium chloride | Sigma | L4036 | |
| DH5alpha bacteria | Thermo Scientific | 18265017 | |
| DMEM (GlutaMAX, 4.5g/L D-Glucose) | Gibco | 3240-027 | |
| EA.hy926 cells | ATCC | CRL-2922 | |
| EDTA | Sigma | E5134 | |
| Ethanol (99.8%) | Roth | 5054.2 | |
| Fetal Bovine Serum | Sigma | F7524 | |
| Flasks T25, T75, T175 | Eppendorf | 30712129 | |
| Glucose | Sigma | G7021 | |
| Hepa 1-6 murine hepatocytes | ATCC | CRL-1830 | |
| Hind III | Thermo Scientific | FD0504 | |
| Kanamycin Sulfate | Thermo Scientific | 15160054 | |
| K2 Transfection System | Biontex | T060-5.0 | |
| LB medium | Formedium | LBx0102 | |
| LB-agar | Formedium | LBx0202 | |
| Mix & Go E. coli Transformation kit | Zymo Research | T3001 | |
| Midori Green Advanced DNA stain | Nippon Genetics Europe | MG-04 | |
| NaOH | Sigma | S8045 | |
| Opti-MEM | Thermo Scientific | 31985070 | |
| Pac I | Thermo Scientific | FD2204 | |
| pAdEasy-1 | Addgene | 16400 | |
| pAdTrack-CMV | Addgene | 16405 | |
| Phenol:chloroform:isoamyl alcohol (24:24:1) | Invitrogen | 15593-031 | |
| Polymerase GoTaq | Promega | M3005 | |
| Pme I (Mss I) | Thermo Scientific | FD1344 | |
| Potassium acetate | VWR Chemicals | 43065P | |
| Pst I | Thermo Scientific | FD0614 | |
| Qiagen Midi Prep kit | Qiagen | 12125 | |
| Cell Scraper | TPP | 99003 | |
| SDS | Thermo Scientific | 28365 | |
| Slide-A-Lyzer dialysis cassettes | Thermo Scientific | 66330 | |
| Sodium pyruvate | SIGMA | P5280-100G | |
| Syringe with 23G neeedle | B Braun | 464BR | |
| Tris HCl | Sigma | 1185-53-1 | |
| Trypan blue | Roth | CN76.1 | |
| Tubes 50ml | TPP | 91050 | |
| Ultra-Clear Tubes (14x89 mm) | Beckman Coulter | 344059 | |
| Centrifuge (refrigerated) | Sigma Sartorius | 3-19KS | |
| HeraeusFresco 17 Microcentrifuge | Thermo Scientific | 75002420 | |
| Ultracentrifuge with SW41Ti rotor | Beckman Coulter | Optima L-80 XP | |
| Culture Hood | Thermo Scientific | Class II | |
| Pipettes (0-2µl, 1-10µl, 2-20µl, 10-100µl, 20-200µl, 100-1000µl) | Thermo Scientific | ||
| Dry Block Heating Thermostat | Biosan | TDB-120 | |
| Thermocycle | SensoQuest | 012-103 | |
| Water Bath | Memmert | WNB 14 |
Referencias
- Lee, C. S., et al. Adenovirus-Mediated Gene Delivery: Potential Applications for Gene and Cell-Based Therapies in the New Era of Personalized Medicine. Genes and Diseases. 4 (2), 43-63 (2017).
- He, T. C., et al. A simplified system for generating recombinant adenoviruses. Proceedings of the National Academy of Sciences of the United States of America. 95 (5), 2509-2514 (1998).
- Russell, W. C. Update on adenovirus and its vectors. The Journal of General Virology. 81, 2573-2604 (2000).
- Rauschhuber, C., Noske, N., Ehrhardt, A. New insights into stability of recombinant adenovirus vector genomes in mammalian cells. European Journal of Cell Biology. 91 (1), 2-9 (2012).
- Saha, B., Wong, C. M., Parks, R. J. The adenovirus genome contributes to the structural stability of the virion. Viruses. 6 (9), 3563-3583 (2014).
- Kreppel, F., Kochanek, S. Modification of adenovirus gene transfer vectors with synthetic polymers: a scientific review and technical guide. Molecular Therapy: the Journal of the American Society of Gene Therapy. 16 (1), 16-29 (2008).
- Dormond, E., Perrier, M., Kamen, A. From the first to the third generation adenoviral vector: what parameters are governing the production yield. Biotechnol Advances. 27 (2), 133-144 (2009).
- Parks, R. J., et al. A helper-dependent adenovirus vector system: removal of helper virus by Cre-mediated excision of the viral packaging signal. Proceedings of the National Academy of Sciences of the United States of America. 93 (24), 13565-13570 (1996).
- Jager, L., Ehrhardt, A. Emerging adenoviral vectors for stable correction of genetic disorders. Current Gene Therapy. 7 (4), 272-283 (2007).
- Luo, J., et al. A protocol for rapid generation of recombinant adenoviruses using the AdEasy system. Nature Protocols. 2 (5), 1236-1247 (2007).
- Dumitrescu, M., et al. Adenovirus-Mediated FasL Minigene Transfer Endows Transduced Cells with Killer Potential. International Journal of Molecular Sciences. 21 (17), (2020).
- Campos, S. K., Barry, M. A. Current advances and future challenges in Adenoviral vector biology and targeting. Current Gene Therapy. 7 (3), 189-204 (2007).
- Khare, R., Chen, C. Y., Weaver, E. A., Barry, M. A. Advances and future challenges in adenoviral vector pharmacology and targeting. Current Gene Therapy. 11 (4), 241-258 (2011).
- Jager, L., et al. A rapid protocol for construction and production of high-capacity adenoviral vectors. Nature Protocols. 4 (4), 547-564 (2009).
- Zvintzou, E., et al. Pleiotropic effects of apolipoprotein C3 on HDL functionality and adipose tissue metabolic activity. Journal of Lipid Research. 58 (9), 1869-1883 (2017).
- Karavia, E. A., et al. Apolipoprotein A-I modulates processes associated with diet-induced nonalcoholic fatty liver disease in mice. Molecular Medicine. 18, 901-912 (2012).
- Lampropoulou, A., Zannis, V. I., Kypreos, K. E. Pharmacodynamic and pharmacokinetic analysis of apoE4 [L261A, W264A, F265A, L268A, V269A], a recombinant apolipoprotein E variant with improved biological properties. Biochemical Pharmacology. 84 (11), 1451-1458 (2012).
- Zheng, S. Y., Li, D. C., Zhang, Z. D., Zhao, J., Ge, J. F. Adenovirus-mediated FasL gene transfer into human gastric carcinoma. World Journal of Gastroenterology. 11 (22), 3446-3450 (2005).
- Ambar, B. B., et al. Treatment of experimental glioma by administration of adenoviral vectors expressing Fas ligand. Human Gene Therapy. 10 (10), 1641-1648 (1999).
- Okuyama, T., et al. Efficient Fas-ligand gene expression in rodent liver after intravenous injection of a recombinant adenovirus by the use of a Cre-mediated switching system. Gene Therapy. 5 (8), 1047-1053 (1998).
- van Dijk, K. W., Kypreos, K. E., Fallaux, F. J., Hageman, J. Adenovirus-mediated gene transfer. Methods in Molecular Biology. 693, 321-343 (2011).
- Zhao, Y. D., Li, T., Huang, G. A simple negative selection method to identify adenovirus recombinants using colony PCR. Electronic Journal of Biotechnology, North America. 17 (1), 46-49 (2014).
- Kovesdi, I., Hedley, S. J. Adenoviral producer cells. Viruses. 2 (8), 1681-1703 (2010).
- Lin, X. Construction of new retroviral producer cells from adenoviral and retroviral vectors. Gene Therapy. 5 (9), 1251-1258 (1998).
- Fallaux, F. J., et al. Characterization of 911: a new helper cell line for the titration and propagation of early region 1-deleted adenoviral vectors. Human Gene Therapy. 7 (2), 215-222 (1996).
- Altaras, N. E., et al. Production and formulation of adenovirus vectors. Advances in Biochemical Engineering/ Biotechnology. 99, 193-260 (2005).
- Schagen, F. H., et al. Ammonium sulphate precipitation of recombinant adenovirus from culture medium: an easy method to increase the total virus yield. Gene Therapy. 7 (18), 1570-1574 (2000).
- Colombet, J., et al. Virioplankton 'pegylation': use of PEG (polyethylene glycol) to concentrate and purify viruses in pelagic ecosystems. Journal of Microbiological Methods. 71 (3), 212-219 (2007).
- Kypreos, K. E., van Dijk, K. W., van Der Zee, A., Havekes, L. M., Zannis, V. I. Domains of apolipoprotein E contributing to triglyceride and cholesterol homeostasis in vivo. Carboxyl-terminal region 203-299 promotes hepatic very low density lipoprotein-triglyceride secretion. Journal of Biological Chemistry. 276 (23), 19778-19786 (2001).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoThis article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados