Method Article
Nicht-virales Engineering von primären humanen T-Zellen durch Homologie-vermittelte Endverknüpfung und gezielte Integration großer DNA-Templates
In diesem Artikel
Zusammenfassung
Es wird ein detailliertes Protokoll für die Verwendung der CRISPR/Cas9-Technologie zur Erzielung eines hocheffizienten gezielten Knock-ins von großen, multicistronischen Konstrukten in primären humanen T-Zellen über den Homologie-vermittelten Endverbindungsweg (HMEJ) zur Verfügung gestellt. T-Zellen, die mit diesem cGMP-adaptierbaren Protokoll hergestellt wurden, behalten eine hervorragende Zellexpansion, Zytotoxizität und Zytokinproduktion bei.
Zusammenfassung
Viele der derzeitigen adoptiven zellulären Therapien stützen sich auf lenti- oder retrovirale Vektoren, um T-Zellen für die Expression eines chimären Antigenrezeptors (CAR) oder eines exogenen T-Zell-Rezeptors (TCR) zu entwickeln, der auf ein spezifisches tumorassoziiertes Antigen abzielt. Die Abhängigkeit von viralen Vektoren für die Produktion therapeutischer T-Zellen erhöht den Zeitplan, die Kosten und die Komplexität der Herstellung erheblich und schränkt gleichzeitig die Umsetzung neuer Therapien ein, insbesondere im akademischen Umfeld. Es wird ein Verfahren für effizientes nicht-virales Engineering von T-Zellen unter Verwendung von CRISPR/Cas9 und homologievermittelter Endverknüpfung vorgestellt, um eine gezielte Integration großer, multicistronischer DNA-Fracht zu erreichen. Dieser Ansatz hat Integrationsfrequenzen erreicht, die mit denen viraler Vektoren vergleichbar sind, und gleichzeitig hochfunktionelle T-Zellen hervorgebracht, die sowohl in vitro als auch in vivo eine starke Anti-Tumor-Wirksamkeit aufweisen können. Insbesondere lässt sich diese Methode schnell an die aktuellen guten Herstellungspraktiken (cGMP) und das klinische Scale-up anpassen und bietet eine kurzfristige Option für die Herstellung von therapeutischen T-Zellen für den Einsatz in klinischen Studien.
Einleitung
T-Zellen sind eine Schlüsselkomponente des adaptiven Immunsystems und besitzen direkte zytolytische Fähigkeiten, die Fähigkeit, die Immunantwort durch die Produktion von Zytokinen zu modulieren, die Lizenzierung von B-Zellen und dendritischen Zellen und die Etablierung eines immunologischen Gedächtnisses1. Sie spielen eine entscheidende Rolle bei der Immunentwicklung, der Homöostase und Überwachung, dem Schutz vor Krankheitserregern und der Prävention und Abwehr von Krebs sowie bei Allergien und Autoimmunität1. T-Zellen besitzen eine enorme Vielfalt an T-Zell-Rezeptoren (TCRs), die durch V(D)J-Rekombination erzeugt werden, was es T-Zellen ermöglicht, eine Vielzahl von Antigenen zu erkennen und wirksame Immunantworten gegen verschiedene Krankheitserreger zu starten 1,2. T-Zellen können im Allgemeinen in zwei Kategorien eingeteilt werden: CD4-T-Zellen, auch bekannt als T-Helferzellen, die in erster Linie andere Immunzellen, wie z. B. B-Zellen, bei der Koordination der Immunantwort unterstützen, und CD8-T-Zellen oder zytotoxische T-Zellen, die infizierte oder krebsartige Zellen direkt abtöten, indem sie bestimmte Antigene erkennen, die auf ihren Oberflächen vorhanden sind1.
Die Entwicklung von chimären Antigenrezeptoren (CAR) hat zu einem massiven Anstieg des Interesses am Genome Engineering von T-Zellen für Immuntherapien geführt. CARs sind technisch hergestellte Proteine, die von Antikörpern abgeleitete Antigenbindungsdomänen mit T-Zell-Signaldomänen verschmelzen, wodurch T-Zellen Zellen identifizieren und angreifen können, die das spezifische Epitop exprimieren, das vom Antikörperteil des CAR3 erkannt wird. Diese Rezeptoren wurden für eine Vielzahl von Immuntherapien verwendet, darunter Infektionskrankheiten und Autoimmunerkrankungen, aber die Technologie ist bei Krebsimmuntherapien am weitesten fortgeschritten.
CAR-T-Zellen waren bei der Behandlung von Leukämien und Lymphomen äußerst erfolgreich, zeigten jedoch eine begrenzte Wirksamkeit bei der Behandlung solider Tumoren 4,5. Dies hat zu einer Welle der Weiterentwicklung geführt, die darauf abzielt, die Wirksamkeit von CAR-T-Zellen für solide Tumorindikationen zu verbessern. Es wurden mehrere Ansätze entwickelt, darunter Zytokin-Armierung, Checkpoint-Gen-Knockout, dominante negative Rezeptoren, Chemokinrezeptoren, die Expression mehrerer CARs in einer Zelle, Modifikation des CAR zur Verbesserung der intrazellulären Signalübertragung und Integration in vorgegebene Loci, z. B. den TRAC-Locus, um die Regulationsmechanismen des Wirts zu nutzen, um Erschöpfung zu verhindern 6,7,8. Viele dieser Ansätze erfordern entweder eine größere genetische Fracht und/oder eine ortsspezifische Integration. Zu den alternativen Ansätzen gehört auch die Verwendung transgener TCRs, um es T-Zellen zu ermöglichen, intrazelluläre Neoantigene anzugreifen 9,10. Dies hat jedoch den wesentlichen Nachteil, dass der TCR Spezifität sowohl für das Neoantigen-Epitop als auch für das HLA-Molekül aufweisen muss, was die Verwendung des eventuellen Therapeutikums auf Patienten beschränkt, die das verwandte HLA exprimieren. Darüber hinaus verändern oder reduzieren viele Tumoren die HLA-Expression als Reaktion auf die Immuntherapie, wodurch die Wirksamkeit von T-Zellen, die transgene TCRs exprimieren, stark verringertwird 11.
Die meisten CAR-T- oder TCR-T-Zelltherapien in klinischen Studien werden unter Verwendung retroviraler Vektoren wie Lentiviren oder Gammaretroviren hergestellt, wodurch eine hohe Integrationsfrequenz bei mäßig großer Fracht erreicht wird. Virale Vektoren leiden jedoch unter langen Herstellungszeiträumen aufgrund der aktuellen Anforderungen der guten Herstellungspraxis (cGMP) und unspezifischer Integrationsprofile, die ein Risiko für eine Insertionsmutagenese darstellen12,13. Darüber hinaus kann es schwierig sein, transgene Retroviren bei hohen Titern zu produzieren, wenn die Fracht 5 kb14 überschreitet. Andere Vektoren, wie z. B. solche, die vom rekombinanten Adeno-assoziierten Virus (rAAV) abgeleitet sind, integrieren sich nicht auf natürliche Weise, können aber die DNA-Donator-Template in den Zellkern transportieren und können in Kombination mit CRISPR/Cas9 verwendet werden, um das traditionelle homologiegesteuerte Rekombinations-vermittelte Genom-Engineering (HDR) zu erleichtern. Diese Viren haben jedoch auch lange und komplizierte Produktionsabläufe und sind durch die Frachtgröße (<4,7 kb) und die Notwendigkeit, lange Homologiearme (500-1000 bp) einzubeziehen, begrenzt15,16,17,18.
Nicht-virales Genom-Engineering unter Verwendung von Transposons oder einer Kombination aus gezielten Nukleasen und einem DNA-Donor-Template wurde in primären menschlichen Lymphozyten berichtet 8,19,20. Diese Ansätze sind jedoch durch die toxische Reaktion auf nackte DNA-Moleküle im Zytoplasma nach der Erkennung durch zytoplasmatische DNA-Sensoren, die in Lymphozyten exprimiert werden, begrenzt21. Es wurden Versuche unternommen, niedermolekulare Inhibitoren dieser DNA-Sensorwege während der Transfektion zu verwenden, aber die Redundanz dieser Signalwege könnte ihre Verwendung in cGMP-Protokollen erschweren22. Bemerkenswert ist, dass Transposon-Vektoren wie Dornröschen, PiggyBac und Tc Buster die Integration großer genetischer Fracht mit hoher Effizienz ermöglichen, aber ein unspezifisches Integrationsprofil aufweisen23,24. Die nicht-virale, zielgerichtete Transgenintegration unter Verwendung von Plasmid-, linearen oder einzelsträngigen DNA-Templates in Kombination mit einer gezielten Nuklease für HDR ist eine attraktive Alternative, wurde aber durch eine schlechte Effizienz eingeschränkt, insbesondere bei immer größeren genetischen Frachten, wobei weniger als 10 % Effizienz bei der Verwendung von Fracht über 1,5 kb berichtet wurde 8,19.
Hier stellen wir das Schritt-für-Schritt-Protokoll für die nicht-virale, homologievermittelte Endverbindung (HMEJ)-Insertion großer DNA-Nutzlasten in primären menschlichen T-Zellen vor, wie in Webber, Johnson et al. beschrieben.25. HMEJ verwendet kurze 48-bp-Homologiearme, die von Cas9-gRNA-Zielstellen flankiert werden, um eine hochwirksame gezielte Integration großer DNA-Fracht im Vergleich zu herkömmlichem HDR zu ermöglichen. Ein Verfahren zur Verringerung der Zytotoxizität von Plasmid-DNA in primären T-Zellen besteht darin, Plasmide mit minimiertem Rückgrat, wie z. B. Minikreise oder Nanoplasmide25, zu verwenden. Minikreise sind miniaturisierte Plasmidvektoren, die durch Exzision des Ursprungs der Replikation und des Antibiotikaresistenzgens durch Rekombination nach Plasmidamplifikation hergestellt werden; Es wurde gezeigt, dass sie das nicht-virale Engineering von T-Zellen verbessern und die Zelltoxizität verringern23,24. Nanoplasmide weisen auch eine reduzierte Gesamtgröße auf, die durch die Verwendung eines minimalen Replikationsursprungs und eines nicht-traditionellen Selektionsmarkers erreicht wird26. Unserer Erfahrung nach bieten Minicircle- und Nanoplasmid-Vektorplattformen eine vergleichbare Effizienzverbesserung und reduzierte Toxizität im Vergleich zu herkömmlichen Plasmiden25.
Hier präsentieren wir ein detailliertes Protokoll, das die zeitliche Optimierung der Reagenzienabgabe und der Reagenzienzusammensetzung sowie die Verwendung von HMEJ und CRISPR/Cas9 zur Erzielung einer hohen Wirksamkeit, das ortsspezifische Genom-Engineering von primären humanen T-Zellen mit großen (>6,3 kb), multicistronischen DNA-Templates für den Einsatz in Immuntherapien und einer Vielzahl anderer Anwendungen kombiniert25. Wir erreichen eine höhere Integration mit HMEJ- und 48 bp-Homologiearmen als mit traditioneller HR unter Verwendung von 1 kb Homologiearmen, insbesondere mit genetischen Ladungen >1,5 kb25,27. Wichtig ist, dass T-Zellen, die durch HMEJ-Reparatur hergestellt wurden, eine hervorragende Zellexpansion, Zytotoxizität und Zytokinproduktion beibehalten, während sie einen nicht erschöpften Phänotypbeibehalten 25. Dieses Protokoll lässt sich leicht an cGMP-Standards anpassen und ist auf klinisch relevante Zellzahlen skalierbar, was einen schnellen Übergang zur zukünftigen Verwendung in einer Vielzahl von klinischen Studien ermöglicht25.
Protokoll
Alle Experimente wurden unter universellen Vorsichtsmaßnahmen für durch Blut übertragbare Krankheitserreger, mit steriler/aseptischer Technik, persönlicher Schutzausrüstung und geeigneter Ausrüstung der Biosicherheitsstufe 2 (BSL2) durchgeführt. Alle hier beschriebenen Experimente wurden vom Institutional Biosafety Committee (IBC) an der University of Minnesota genehmigt. Einzelheiten zu den in dieser Studie verwendeten Reagenzien und Geräten sind in der Materialtabelle aufgeführt.
1. Vorbereitung der Medien
- Bereiten Sie Nahrungsergänzungsmittel für T-Zell-Komplettmedien (TCM) vor.
- Rekonstituieren Sie rekombinantes humanes IL-2 auf eine Konzentration von 12000 IE/ml durch Zugabe von filtersterilisierter 100 mM Essigsäure und mischen Sie es durch Pipettieren gut. Dann weiter auf eine Konzentration von 6000 IE/ml mit filtersterilisiertem 0,2 % Rinderserumalbumin (BSA) in 1x phosphatgepufferter Kochsalzlösung (1x PBS) verdünnen und durch Pipettieren gut mischen, um eine erweiterte Lagerlösung herzustellen.
- Rekombinantes humanes IL-7 mit sterilem Wasser auf eine Konzentration von 200 ng/μl rekonstituieren und durch Pipettieren gut mischen. Dann weiter auf eine Konzentration von 100 ng/μl mit filtersterilisiertem 0,2 % BSA in PBS verdünnen und durch Pipettieren gut mischen, um eine erweiterte Lagerlösung herzustellen.
- Rekonstituiertes humanes IL-15 auf eine Konzentration von 200 ng/μl mit sterilem Wasser rekonstituieren und durch Pipettieren gut mischen. Dann weiter auf eine Konzentration von 100 ng/μl mit filtersterilisiertem 0,2 % BSA in PBS verdünnen und durch Pipettieren gut mischen, um eine erweiterte Lagerlösung herzustellen.
HINWEIS: Bewahren Sie jedes Zytokin in kleinen Aliquoten bis zu sechs Monate lang bei -20 °C bis -80 °C auf und vermeiden Sie wiederholte Gefrier-/Auftauzyklen.
- TCM-Medien vorbereiten.
- Bereiten Sie das Basalmedium vor, indem Sie 2,6 % T-Zell-Expansionsergänzungsmittel und 2,5 % Immunzellserumersatz zum T-Zellexpansions-Basalmedium hinzufügen. Weitere Informationen finden Sie in der Materialtabelle .
- Bereiten Sie die TCM vor, indem Sie 1 % L-Glutamin, 1 % Penicillin/Streptomycin, 10 mM N-Acetyl-L-Cystein, 300 I.E./ml rekombinantes humanes IL-2, 5 ng/ml rekombinantes humanes IL-7 und 5 ng/ml rekombinantes humanes IL-15 zu den Basalmedien hinzufügen.
- Sterilisieren Sie TCM, indem Sie das Medium durch einen 0,22-μm-Filter in eine sterile Medienflasche leiten.
- Halten Sie TCM bis zu 2 Wochen lang bei 4 °C.
- Bereiten Sie das Basalmedium vor, indem Sie 2,6 % T-Zell-Expansionsergänzungsmittel und 2,5 % Immunzellserumersatz zum T-Zellexpansions-Basalmedium hinzufügen. Weitere Informationen finden Sie in der Materialtabelle .
- Bereiten Sie das Wiederherstellungsmedium vor.
- Bereiten Sie das Basalmedium vor, indem Sie 2,6 % T-Zell-Expansionsergänzungsmittel und 2,5 % Immunzellserumersatz zum T-Zellexpansions-Basalmedium hinzufügen.
- Bereiten Sie das Wiederherstellungsmedium vor, indem Sie 1 % L-Glutamin, 10 mM N-Acetyl-L-Cystein, 300 I.E./ml rekombinantes humanes IL-2, 5 ng/ml rekombinantes humanes IL-7, 5 ng/ml rekombinantes humanes IL-15 zu Basalmedien und 1 μg/ml DNase hinzufügen.
HINWEIS: Fügen Sie Penicillin/Streptomycin nicht zu Wiederherstellungsmedien hinzu, da dies die Zellerholung nach der Elektroporation verringern kann. - Sterilisieren Sie das Rückgewinnungsmedium, indem Sie das Medium durch einen 0,22-μm-Filter in eine sterilisierte Medienflasche leiten.
- Bewahren Sie das Wiederherstellungsmedium bis zu 2 Wochen lang bei 4 °C auf.
- Bereiten Sie das Wiederherstellungsmedium vor, indem Sie 1 % L-Glutamin, 10 mM N-Acetyl-L-Cystein, 300 I.E./ml rekombinantes humanes IL-2, 5 ng/ml rekombinantes humanes IL-7, 5 ng/ml rekombinantes humanes IL-15 zu Basalmedien und 1 μg/ml DNase hinzufügen.
- Bereiten Sie das Basalmedium vor, indem Sie 2,6 % T-Zell-Expansionsergänzungsmittel und 2,5 % Immunzellserumersatz zum T-Zellexpansions-Basalmedium hinzufügen.
2. Auswahl der Knock-in-Site und Vorlagendesign
- Bestimmen Sie einen genomischen Zielort für den Knock-in.
HINWEIS: Ein Safe-Harbor-Standort, wie z. B. AAVS1, kann verwendet werden, wenn minimale Auswirkungen auf die Zielzelle gewünscht werden. Eine Zielstelle, die die Genexpression stört, kann verwendet werden, um gleichzeitig das Zielgen auszuschalten und das Konstrukt von Interesse in einem einzigen Schritt auszuschalten. Eine Zielstelle und ein Donor, die für die Integration im Frame in ein endogenes Gen ausgelegt sind, können verwendet werden, um Genfusionen zu erzeugen, oder in Kombination mit einer ribosomalen 2A-Skip-Sequenz, um das Konstrukt unter die transkriptionelle Kontrolle eines endogenen Promotors zu stellen (Abbildung 1). - Entwerfen Sie das Cargo-Template so, dass die Expressionskassette von 48-bp-Homologiearmen flankiert wird, die der Integrationsstelle entsprechen, und von linearisierenden gRNA-Zielstellen flankiert wird.
HINWEIS: Bei der plasmidlinearisierenden gRNA kann es sich um eine universelle gRNA28 (UgRNA) (GGGAGGCGUUCGGGCCACAG) handeln, die so konzipiert ist, dass sie auf eine Sequenz (GGGAGGCGTTCGGGCCACAG) abzielt, die im menschlichen oder murinen Genom25 nicht vorkommt, oder auf die genomische Ziel-gRNA-Sequenz, so dass eine einzelne gRNA an der genomischen Knock-in-Stelle schneiden und auch das Fracht-Template linearisieren kann (Abbildung 1). Als Vektoren für die Frachtvorlage können entweder Nanoplasmide oder Minikreise verwendet werden. Standardplasmide können anstelle von Nanoplasmiden oder Minikreisen für einige transformierte Linien verwendet werden, führen jedoch zu einem reduzierten Knock-in und einer reduzierten Zellexpansion in empfindlicheren Primärzellen wie T-Zellen.
3. Isolierung und Aktivierung von T-Zellen
- Beziehen Sie T-Zellen von einem kommerziellen Anbieter oder isolieren Sie T-Zellen aus PBMCs durch immunomagnetische Sortierung18.
- Bestimmen Sie die Anzahl der für das Experiment erforderlichen T-Zellen und tauen, waschen und resuspendieren Sie die T-Zellen dann in einer Konzentration von 1 × 106 Zellen pro ml in der TCM. Geben Sie 1 ml dieser Mischung in die Vertiefungen einer 24-Well-Platte.
- Wirbeln Sie die T-Zell-Aktivierungskügelchen auf, um die Kügelchen zu resuspendieren, und fügen Sie die Kügelchen in einem Verhältnis von 2:1 Kügelchen hinzu: Zelle zu jeder Vertiefung. Inkubieren Sie diese Zellen 36 Stunden lang in einem 37 °C und 5 % CO2 -Inkubator, bevor Sie mit dem T-Zell-Engineering beginnen.
HINWEIS: Eine Inkubationszeit von weniger als oder mehr als 36 Stunden nach der Aktivierung verringert die Knock-in-Effizienz, die Lebensfähigkeit und die Zellexpansion nach dem Engineering25.
- Wirbeln Sie die T-Zell-Aktivierungskügelchen auf, um die Kügelchen zu resuspendieren, und fügen Sie die Kügelchen in einem Verhältnis von 2:1 Kügelchen hinzu: Zelle zu jeder Vertiefung. Inkubieren Sie diese Zellen 36 Stunden lang in einem 37 °C und 5 % CO2 -Inkubator, bevor Sie mit dem T-Zell-Engineering beginnen.
4. T-Zell-Engineering
- Bestimmen Sie die experimentellen Bedingungen, die zum Abschluss des Experiments erforderlich sind, einschließlich experimenteller Kontrollen.
HINWEIS: Es ist wichtig, eine reine Plasmidbedingung als Kontrolle für die episomale Expression aufzunehmen. Als Negativkontrolle kann auch eine Elektroporationsbedingung mit der chemisch modifizierten Zielstelle gRNA28 und/oder Cas9 mRNA in Abwesenheit eines Plasmidspenders verwendet werden. - Bereiten Sie eine Wiederherstellungsplatte vor.
- Für jede Versuchsbedingung, einschließlich der experimentellen Kontrollen, werden 300 μl Rückgewinnungsmedium in eine Vertiefung einer 24-Well-Gewebekulturplatte gegeben und die Platte in einem Inkubator mit 37 °C und 5 % CO2 erwärmt.
HINWEIS: In Tabelle 1 finden Sie alternative Zellennummern, Wiederherstellungsvolumes und Bedingungen.
- Für jede Versuchsbedingung, einschließlich der experimentellen Kontrollen, werden 300 μl Rückgewinnungsmedium in eine Vertiefung einer 24-Well-Gewebekulturplatte gegeben und die Platte in einem Inkubator mit 37 °C und 5 % CO2 erwärmt.
- Bereiten Sie T-Zellen vor.
- Ernten Sie die stimulierte T-Zell/T-Zell-Aktivierungsbead-Mischung, zählen Sie lebensfähige Zellen und resuspendieren Sie die T-Zellen/T-Zell-Aktivierungsbeads in einer Konzentration von 1-5 × 106 Zellen/ml in TCM-Medien in einem 1,5-ml-Mikrozentrifugenröhrchen.
- Setzen Sie das Mikrozentrifugenröhrchen in einen Magneten (siehe Materialtabelle) und lassen Sie es 3 Minuten lang inkubieren.
HINWEIS: Eine Bead-Entfernung ist erforderlich, wenn T-Zell-Aktivierungsbeads zur Aktivierung von T-Zellen verwendet werden. Eine Raupenentfernung ist möglicherweise nicht erforderlich, wenn eine alternative Aktivierungsmethode verwendet wird. - Nach der Inkubation und ohne Entnahme aus dem Magneten das Medium und die T-Zellen in ein neues Röhrchen umfüllen. Dieses Röhrchen enthält stimulierte T-Zellen, bei denen die T-Zell-Aktivierungskügelchen entfernt wurden. Zählen Sie die T-Zellen neu und geben Sie sie entweder in konische 14-ml- oder 50-ml-Röhrchen. Füllen Sie das Röhrchen mit 1× PBS auf und bewahren Sie die Zellen in einem Inkubator mit 37 °C und 5 % CO2 auf, bis sie zentrifugiert werden können.
- Bereiten Sie die T-Zell-Mischung vor.
- Bestimmen Sie die benötigte Menge an Master-Mix-Puffer (82 % Primärzelllösung und 18 % Ergänzung), indem Sie die Anzahl der Elektroporationsbedingungen plus eins (n + 1) multiplizieren. (z. B. 6 geplante Elektroporationsbedingungen + 1 = 7,7 × 18 μl pro Bedingung = 126 μl Master-Mix-Puffer).
HINWEIS: Diese Volumina sind spezifisch für das in dieser Studie verwendete 4D-Elektroporationssystem (siehe Materialtabelle). Andere Elektroporationssysteme erfordern andere Volumina und Konzentrationen. Volumen der primären Zelllösung = Gesamtvolumen des benötigten Master-Mix-Puffers × 0,82 und Volumen des Ergänzungsmittels = Gesamtvolumen des benötigten Master-Mix-Puffers x 0,18. (z. B. 0,82 × 126 μl = 103,32 μl primäre Zelllösung und 0,18 × 126 μl = 22,68 μl). - Mischen Sie die Primärzelllösung und das Supplement zusammen, um einen Master-Mix-Puffer herzustellen.
- T-Zellen in 1x PBS für 10 min bei 200 × g (bei Raumtemperatur) zentrifugieren und den Überstand absaugen.
- Resuspendieren Sie die T-Zellen im Master-Mix-Puffer in einer Konzentration von 1 × 106 Zellen pro 18 μl Master-Mix-Puffer.
- Bestimmen Sie die benötigte Menge an Master-Mix-Puffer (82 % Primärzelllösung und 18 % Ergänzung), indem Sie die Anzahl der Elektroporationsbedingungen plus eins (n + 1) multiplizieren. (z. B. 6 geplante Elektroporationsbedingungen + 1 = 7,7 × 18 μl pro Bedingung = 126 μl Master-Mix-Puffer).
- Bereiten Sie die Reagenzien vor.
- Bestimmen Sie anhand der Konzentrationen der Stammlösungen das Volumen des HMEJ-Plasmids, der gRNA an der Zielstelle, der Plasmid-Linearisierungs-gRNA (UgRNA) und der Cas9-mRNA, die erforderlich sind, um die angegebene Masse pro Elektroporation zu erreichen (Tabelle 2).
- Geben Sie für jede Bedingung Reagenzien in eine Vertiefung einer 96-Well-Platte auf Eis.
- Bereiten Sie die Elektroporationsmischung vor.
- Geben Sie nach Bedarf 18 μl T-Zell-Mix in jede Vertiefung der 96-Well-Platte.
- Fügen Sie bei Bedarf zusätzlichen Master-Mix-Puffer hinzu, um sicherzustellen, dass alle Elektroporationsbedingungen ein endgültiges Gesamtvolumen von 22 μl erreichen.
HINWEIS: Das Volumen von 22 μl enthält ein Übervolumen von 10 %, um mögliche Pipettierverluste auszugleichen.
- Führen Sie eine Elektroporation durch.
- Schalten Sie das Gerät ein und laden Sie das Programm.
- Ermitteln Sie die Anzahl der benötigten Küvetten und öffnen Sie die gewünschte Anzahl an Küvetten. Markieren Sie ein Ende der Küvette und der Kappe und entfernen Sie die Kappe von der Küvette.
HINWEIS: Es gibt 16 Wells pro Küvette für 20 μl Reaktionen. - Pipettieren Sie vorsichtig 20 μl der Elektroporationsmischung 1-2 Mal aus den Vertiefungen der 96-Well-Platte auf und ab und füllen Sie sie in Küvetten.
- Verschließen Sie die Küvette mit der in Schritt 4.7.2 vorgenommenen Markierung, um zu vermeiden, dass der Deckel verkehrt herum aufgesetzt wird. Klopfen Sie 3-5 Mal auf die Küvette, um mögliche Luftblasen zu entfernen, und platzieren Sie dann das Gerät und elektropolieren Sie die Küvette.
- Nehmen Sie die Küvette vorsichtig aus dem Gerät, setzen Sie sie in die Haube ein und lassen Sie die Zellen 15 Minuten bei Raumtemperatur ruhen.
- Ziehen Sie nach der Pause 80 μl erwärmtes Rückgewinnungsmedium aus der Rückgewinnungsplatte und geben Sie es zu jeder Probe (an der Seite der Küvette, nicht direkt in die Zellen), pipettieren Sie einmal sehr vorsichtig auf und ab und übertragen Sie das Gesamtvolumen von 100 μl zurück auf die Auffangplatte.
HINWEIS: Zellen sind nach der Elektroporation sehr zerbrechlich. Eine schonende Handhabung ist von entscheidender Bedeutung. - Inkubieren Sie die Zellen bei 37 °C und 5% CO2 für 30 min.
- Fügen Sie TCM hinzu, um die Zellen auf eine Konzentration von 1 × 106 Zellen/ml zu verdünnen. Ersetzen Sie die Hälfte des Mediums am nächsten Tag und dann alle 3-4 Tage durch TCM mit 2× Zytokinkonzentrationen für die Dauer des Experiments.
- Nach der Verdünnung auf 1 × 106 Zellen/ml die Zellen durch Zugabe von T-Zell-Aktivierungskügelchen in einem Verhältnis von 0,5:1 Kügelchen: Zelle zu jeder Vertiefung erneut stimulieren.
HINWEIS: Zellen können sich schnell ausdehnen. Möglicherweise müssen halbe Medienwechsel durchgeführt werden, und die Zellen müssen möglicherweise in größere Vertiefungen verschoben werden. - Entfernen Sie an Tag 3 die T-Zell-Aktivierungskügelchen.
- Nach der gewünschten Wachstumsdauer ernten Sie die Zellen.
HINWEIS: Wenn sich unbenutzte Vertiefungen in der Küvette befinden, können die verwendeten Vertiefungen markiert werden, und die gesamte Küvette kann in einen konischen 50-ml-Topf gelegt und für die zukünftige Verwendung bei 4 °C gelagert werden.
Ergebnisse
Hier wurde ein großes (>6,3 kb) multicistronisches Template namens "Giant Minicircle"-Konstrukt mittels CRISPR/Cas9-Editierung in den TRAC-Locus in primären menschlichen T-Zellen integriert; eine TRAC-spezifische gRNA (TCTCTCAGCTGGGGC), die UgRNA und HMEJ-Nanoplasmid (Abbildung 2A) mit einer Erkrankung ohne TRAC-spezifische gRNA, die universelle gRNA und Cas9-mRNA, die als Negativkontrolle verwendet wurde. Proben, die das Giant Minicircle-Konstrukt, TRAC-spezifische gRNA, die universelle gRNA und Cas9-mRNA enthalten, hatten eine durchschnittliche Knock-in-Rate von 23,35 % bei der Messung der GFP-Expression (23,5 % ± 5,247), während die Negativkontrolle keine GFP-Expression zeigte (Abbildung 2B). Es gab keine signifikanten Unterschiede in der Faltenausdehnung (Abbildung 2C) und der Lebensfähigkeit (Abbildung 2D) zwischen den experimentellen Bedingungen. Diese Ergebnisse zeigen einen hocheffizienten Knock-in eines sehr großen Templates bei gleichzeitiger Beibehaltung einer hervorragenden Zellviabilität und Zellexpansion.

Abbildung 1: Schematische Darstellung des HMEJ-Konstruktdesigns. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
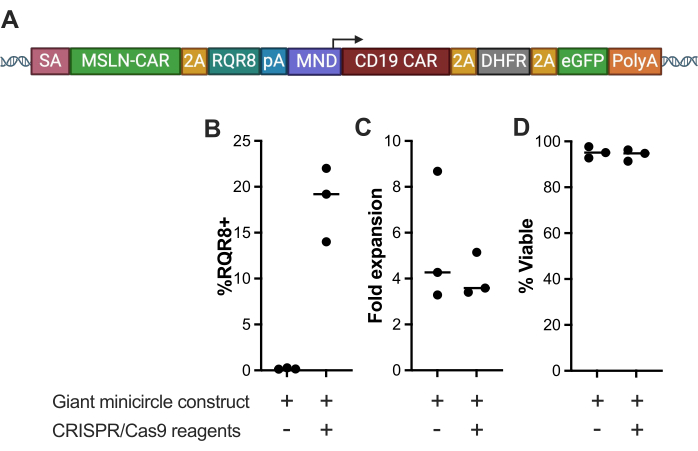
Abbildung 2: Charakterisierung von T-Zellen, die mit dem riesigen Minikreis-Konstrukt elektroporiert wurden. (A) Schematische Darstellung des Riesen-Minicircle-Konstrukts, einer großen (>6,3 kb) multicistronischen Vorlage, die ein Anti-Mesothelin-CAR und RQR8 unter dem TRAC-Promotor mit einem GSG-Linker, einem Anti-CD19-CAR und einem DHFR-Mutein und eGFP unter dem MND-Promotor kodiert. (B) RQR8-Expressionsprozentsatz, (C) Faltenexpansion und (D) Lebensfähigkeit von T-Zellen neun Tage nach der Elektroporation mit dem Giant Minicircle-Konstrukt und CRISPR-Cas9-Reagenzien, verglichen mit einer negativen Kontrolle, die mit dem Giant Minicircle ohne CRISPR-Cas9-Reagenzien elektroporiert wurde. (***p < 0,001). Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
| Plattengröße | Volumen der Küvette | Zellen/Vertiefung | Volumen des Wiederherstellungsmediums | Volumen von 1x TCM mit NAC | Gesamtes Endvolumen |
| 24-Well-Platte | 22/22 μL | 1-3 x 106 cm | 300 μL | 680 μL | 1 mL |
| 6-Well-Platte | 100/110 μL | 4-20 x 106 cm | 1 mL | 2,9 mL | 4 mL |
| 24-Well-G-Rex-Platte | 20-110 μL | 1-20 x 106 cm | 400 μL | 5,6 mL | 6 mL |
Tabelle 1: Zellkonzentrationen für Küvetten und Rückgewinnungsplatten.
| Größe der Küvette | Vorlage Plasmid | gRNA an der Zielstelle | Plasmid-Linearisierung gRNA | Cas9-mRNA |
| ca. 20 μl | 1-2 μg (1 μg) | 1-3 μg (1 μg) | 1-3 μg (1 μg) | 1-3 μg (1,5 μg) |
| 100 μL | 5-10 μg (5 μg) | 5-15 μg (5 μg) | 5-15 μg (5 μg) | 5-15 μg (5 μg) |
Tabelle 2: Menge der benötigten CRISPR/Cas9-Reagenzien und DNA-Vorlage.
Diskussion
Mit der Weiterentwicklung der adoptiven Zelltherapie (ACT) steigt die Nachfrage nach effizienten, nicht-viralen Methoden zur Herstellung von Immunzellen ohne die hohen Kosten und die Komplexität, die mit virusbasierten Vektoren verbunden sind. Ein wichtiges Ziel in diesem Bereich ist die ortsspezifische Integration, die die Konsistenz, Sicherheit und Funktion von technisch hergestellten zellulären Produkten verbessert. Während neuere Studien die erfolgreiche nicht-virale Integration kleiner genetischer Konstrukte wie Reportergene und einzelne CAR- oder TCR-Sequenzen gezeigt haben29, besteht die Notwendigkeit, diese Methoden auf größere Multi-Genexpressionskassetten auszuweiten. Diese größeren Kassetten sind notwendig, um die Funktion der Immunzellen zu verbessern, wie z. B. die Hinzufügung von Chemokinrezeptoren oder Zytokin-Panzerung, die Spezifität zu verbessern (z. B. Logikgattersysteme) oder die Sicherheit mit Kill-Schaltern zu erhöhen. Zu diesem Zweck haben wir versucht, nicht-virale Ansätze zu entwickeln, die in der Lage sind, eine größere genetische Fracht effizient ortsspezifisch zu integrieren25.
Das Protokoll besteht aus mehreren kritischen Schritten. Hochwertige, saubere Nanoplasmide oder Minikreise sind erforderlich, um die Toxizität zu minimieren. In dieser Studie schnitten kommerziell hergestellte Plasmide am besten ab. Der Zeitraum von 36 Stunden zwischen der Aktivierung der T-Zellen und der Elektroporation ist ebenfalls entscheidend für ideale Ergebnisse. Es ist auch wichtig, die Handhabung und Manipulation der Zellen unmittelbar nach der Elektroporation zu minimieren, während sich die Zellen erholen. Es ist auch wichtig, der Kultur nach der Elektroporation wieder neue T-Zell-Aktivierungskügelchen hinzuzufügen, um die bestmögliche Ladungsintegrationsfrequenz und Zellexpansion zu erreichen.
Das Protokoll, wie hier beschrieben, verwendet ein kommerziell erhältliches Elektroporationssystem (siehe Materialtabelle). Andere Elektroporationssysteme könnten ebenfalls geeignet sein, aber sie erfordern wahrscheinlich eine Optimierung der gerätespezifischen Elektroporationsbedingungen und möglicherweise der Handhabung der Zellen nach der Elektroporation.
Es gibt mehrere Einschränkungen bei der HMEJ-vermittelten Entwicklung menschlicher T-Zellen. Das Protokoll kann mit Plasmiden arbeiten, aber optimale Ergebnisse erfordern die Verwendung von Nanoplasmiden oder Minikreisen für die Frachtlieferung, die nicht so einfach herzustellen sind wie Standardplasmide. Obwohl die erfolgreiche Integration einer sehr großen Multicistronic-Kassette demonstriert wurde, gibt es wahrscheinlich eine obere Frachtgrenze, bei der die Häufigkeit der Genintegration abnimmt.
In dieser Studie wird HMEJ als leistungsfähige und effiziente nicht-virale Genom-Engineering-Methode für die Herstellung von gentechnisch veränderten T-Zellen vorgestellt, insbesondere im Rahmen der Krebsimmuntherapie. Durch die Überwindung der Einschränkungen traditioneller viraler Vektoren bietet dieser Ansatz eine kostengünstige, skalierbare und sicherere Alternative zur Erzeugung genetisch veränderter T-Zellen mit verbesserter Funktionalität. Die Fähigkeit, große Multi-Gen-Kassetten präzise zu integrieren, eröffnet neue Möglichkeiten für die Entwicklung von Immunzellen zur Behandlung einer Vielzahl von Krankheiten, darunter Krebs, Infektionskrankheiten und Autoimmunerkrankungen. Darüber hinaus gewährleistet die Kompatibilität der HMEJ-Methode mit klinischen Herstellungsprozessen ihre praktische Anwendbarkeit in realen therapeutischen Umgebungen und ebnet den Weg für zugänglichere und effizientere zellbasierte Therapien in naher Zukunft.
Offenlegungen
B.R.W. und B.S.M. sind Hauptprüfer von Sponsored Research Agreements, die von Intima Biosciences finanziert werden, um die Arbeit in diesem Manuskript zu unterstützen. Es wurden Patente angemeldet, die die in diesem Manuskript beschriebenen Methoden und Ansätze abdecken.
Danksagungen
B.R.W. bedankt sich für die Finanzierung durch das Office of Discovery and Translation, NIH-Zuschüsse R21CA237789, R21AI163731, P01CA254849, P50CA136393, U54CA268069, R01AI146009, die DOD-Zuschüsse HT9425-24-1-1005, HT9425-24-1-1002, HT9425-24-1-0231 und den Children's Cancer Research Fund, den Fanconi Anemia Research Fund und den Randy Shaver Cancer and Community Fund. B.S.M. bedankt sich für die Finanzierung durch das Office of Discovery and Translation, NIH-Zuschüsse R01AI146009, R01AI161017, P01CA254849, P50CA136393, U24OD026641, U54CA232561, P30CA077598, U54 CA268069, DOD-Zuschüsse HT9425-24-1-1005, HT9425-24-1-1002, HT9425-24-1-0231 und den Children's Cancer Research Fund, den Fanconi Anemia Research Fund und den Randy Shaver Cancer and Community Fund.
Materialien
| Name | Company | Catalog Number | Comments |
| Acetic Acid | Millipore Sigma | A6283 | |
| 1x DPBS, no calcium, no magnesium | Thermo Fisher Scientific | 14190144 | |
| 2.5% CTS Immune Cell Serum Replacement | Thermo Fisher Scientific | A2596101 | |
| Amaxa P3 Primary Cell 4D-Nucleofactor X Kit L | Lonza | V4XP-3024 | |
| Amaxa P3 Primary Cell 4D-Nucleofactor X Kit S | Lonza | V4XP-3032 | |
| Bovine Serum Albumin | Thermo Fisher Scientific | 15561020 | |
| Chemically Modified Guide RNAs | Integrated DNA Technologies | na | Custom design |
| CleanCap Cas9 mRNA | Trilink | L-7206 | |
| CTS OpTmizer T cell Expansion Media SFM +OpTmizer T cell Expansion Supplement | Thermo Fisher Scientific | A1048501 | |
| DNase I | Stem Cell Technologies | 07900 | |
| Dynabeads Human T-Activator CD3/CD28 | Thermo Fisher Scientific | 11141D | |
| DynaMag-2 | Thermo Fisher Scientific | 12321D | |
| Human IL15 | PeproTech | 200-15 | |
| Human IL2 | PeproTech | 200-02 | |
| Human IL7 | PeproTech | 200-07 | |
| L-Glutamine | Thermo Scientific | 25030081 | |
| Lonza 4D nucelofector Core | Lonza | AAF-1003B | |
| Lonza 4D nucelofector X Unit | Lonza | AAF-1003X | |
| Minicircle | System Biosciences | MN910A-1 | Custom design |
| N-Acetyl-L-cysteine | MiliporeSigma | A9165 | |
| Nanoplasmid | Aldevron | na | Custom design |
| Penicillin/Streptomycin | Thermo Fisher Scientific | 15-140-122 |
Referenzen
- Sun, L., Su, Y., Jiao, A., Wang, X., Zhang, B. T cells in health and disease. Signal Transduct Target Ther. 8 (1), 235 (2023).
- Takahama, Y. Journey through the thymus: stromal guides for T-cell development and selection. Nature reviews. Immunology. 6 (2), 127-135 (2006).
- Dotti, G., Gottschalk, S., Savoldo, B., Brenner, M. K. Design and development of therapies using chimeric antigen receptor-expressing T cells. Immunol Rev. 257 (1), 107-126 (2014).
- DeRenzo, C., Gottschalk, S. Genetic modification strategies to enhance CAR T cell persistence for patients with solid tumors. Front Immunol. 10, 218 (2019).
- Liu, X., et al. A chimeric switch-receptor targeting PD1 augments the efficacy of second-generation CAR T cells in advanced solid tumors. Cancer Res. 76 (6), 1578-1590 (2016).
- Yeku, O. O., Purdon, T. J., Koneru, M., Spriggs, D., Brentjens, R. J. Armored CAR T cells enhance anti-tumor efficacy and overcome the tumor microenvironment. Sci Rep. 7 (1), 10541 (2017).
- Schober, K., et al. Orthotopic replacement of T-cell receptor α- and β-chains with preservation of near-physiological T-cell function. Nat Biomed Eng. 3 (12), 974-984 (2019).
- Roth, T. L., et al. Reprogramming human T cell function and specificity with non-viral genome targeting. Nature. 559 (7714), 405-409 (2018).
- Chandran, S. S., Klebanoff, C. A. T cell receptor-based cancer immunotherapy: Emerging efficacy and pathways of resistance. Immunol Rev. 290 (1), 127-147 (2019).
- Rosenberg, S. A., Restifo, N. P. Adoptive cell transfer as personalized immunotherapy for human cancer. Science (New York, N.Y.). 348 (6230), 62-68 (2015).
- Campoli, M., Ferrone, S. HLA antigen and NK cell activating ligand expression in malignant cells: a story of loss or acquisition. Semin Immunopathol. 33 (4), 321-334 (2011).
- Walther, W., Stein, U. Viral vectors for gene transfer: a review of their use in the treatment of human diseases. Drugs. 60 (2), 249-271 (2000).
- Bulcha, J. T., Wang, Y., Ma, H., Tai, P. W. L., Gao, G. Viral vector platforms within the gene therapy landscape. Signal Transduct Target Ther. 6 (1), 53 (2021).
- Kumar, M., Keller, B., Makalou, N., Sutton, R. E. Systematic determination of the packaging limit of lentiviral vectors. Hum Gene Ther. 12 (15), 1893-1905 (2001).
- Samulski, R. J., Muzyczka, N. AAV-mediated gene therapy for research and therapeutic purposes. Annu Rev Virol. 1 (1), 427-451 (2014).
- Wang, D., Tai, P. W. L., Gao, G. Adeno-associated virus vector as a platform for gene therapy delivery. Nature reviews. Drug Discov. 18 (5), 358-378 (2019).
- Pomeroy, E. J., et al. a genetically engineered primary human natural killer cell platform for cancer immunotherapy. Mol Ther. 28 (1), 52-63 (2020).
- Johnson, M. J., Laoharawee, K., Lahr, W. S., Webber, B. R., Moriarity, B. S. Engineering of primary human B cells with CRISPR/Cas9 targeted nuclease. Sci Rep. 8 (1), 12144 (2018).
- Nguyen, D. N., et al. Polymer-stabilized Cas9 nanoparticles and modified repair templates increase genome editing efficiency. Nat Biotechnol. 38 (1), 44-49 (2020).
- Lock, D., et al. Automated, scaled, transposon-based production of CAR T cells. J Immunother Cancer. 10 (9), e005189 (2022).
- Semenova, N., et al. Multiple cytosolic DNA sensors bind plasmid DNA after transfection. Nucleic Acids Res. 47 (19), 10235-10246 (2019).
- Wimberger, S., et al. Simultaneous inhibition of DNA-PK and Polϴ improves integration efficiency and precision of genome editing. Nat Comm. 14 (1), 4761 (2023).
- Monjezi, R., et al. Enhanced CAR T-cell engineering using non-viral Sleeping Beauty transposition from minicircle vectors. Leukemia. 31 (1), 186-194 (2017).
- Kay, M. A., He, C. -. Y., Chen, Z. -. Y. A robust system for production of minicircle DNA vectors. Nat Biotechnol. 28 (12), 1287-1289 (2010).
- Webber, B. R., et al. Cas9-induced targeted integration of large DNA payloads in primary human T cells via homology-mediated end-joining DNA repair. Nat Biomed Eng. 8, 1553-1570 (2024).
- Williams, J. A., Paez, P. A. Improving cell and gene therapy safety and performance using next-generation Nanoplasmid vectors. Mol Ther Nucleic Acids. 32, 494-503 (2023).
- Wierson, W. A., et al. Efficient targeted integration directed by short homology in zebrafish and mammalian cells. eLife. 9, 53968 (2020).
- Hendel, A., et al. Chemically modified guide RNAs enhance CRISPR-Cas genome editing in human primary cells. Nat Biotechnol. 33 (9), 985-989 (2015).
- Moretti, A., et al. The past, present, and future of non-viral CAR T cells. Front Immunol. 13, 867013 (2022).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten